JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
siRNA 전달을위한 셀 타입의 특정 안티 HIV gp120 aptamers의 개발
요약
nanomole 친화력과 HIV - 1Ba - L gp120에 대한 몇 가지 2' - 플루오로 RNA의 aptamers는 RNA 라이브러리에서에 의해 고립 아르 체외에서 SELEX 절차. 키메라 새로운 듀얼 억제 기능을 방지 gp120 aptamer - siRNA가 생성되고 체계적 안티 HIV 치료를위한 상당한 약속을 보여줍니다.
초록
HIV 감염의 글로벌 전염병은 antiretroviral 대리인의 새로운 클래스를위한 긴급한 필요를 만들었습니다. 보완 RNA의 성적 표현을 억제하는 작은 간섭 (SI) RNAS의 강력한 기능은 HIV를 포함하여 질병의 다양한 치료제의 새로운 클래스로 악용되고있다. 많은 이전의 보고서 소설 RNAi 기반 anti-HIV/AIDS 치료 전략이 상당한 약속을하는 것으로 나타났습니다, 그러나, 성공적인 치료 응용 프로그램과 siRNAs의 임상 번역에 대한 주요 장애물은 효율적으로 전달됩니다. 특히, RNAi 기반 치료제의 안전성과 효능을 고려하여, 그것은 특정 세포 집단 또는 조직에 타겟 세포 siRNA 전달 방법을 개발하기 위해 매우 바람직합니다. HIV - 1 gp120 단백질, HIV - 1의 표면에 당단백질 봉투는 CD4 세포에 바이러스 항목에 중요한 역할을합니다. HIV - 1 항목을 촉발 요인과 세포 융합을 시작 것을 gp120과 CD4의 상호 작용은 약물 발견을위한 임상 관련 안티 바이러스 전략으로 검증되었습니다.
여기에, 우리는 첫째로 2' - F 수정된 안티 HIV gp120 RNA의 aptamers의 선택과 신분증을 토론합니다. 종래의 nitrocellulose 필터 SELEX 방법을 사용 nanomolar 친화력과 함께 여러 가지 새로운 aptamers는 50 무작위 NT RNA 라이브러리에서 고립되었다. 성공적으로 높은 친화력과 바운드 종 얻기 위해서는, 선택의 엄중는 신중하게 조건을 조정하여 제어됩니다. 선택한 aptamers은 특별히 바인딩할 수 있고 빠르게 HIV - 1 봉투 단백질을 표현하는 세포로 internalized 수 있습니다. 또한, 혼자 aptamers는 HIV - 1 감염을 중화시킬 수 있습니다. 최고의 aptamer A - 1을 기반으로, 우리는 aptamer와 siRNA의 부분이 모두 강력한 항정 HIV 활동을 갖고있는 키메라 소설 이중 억제 기능을 방지 gp120 aptamer - siRNA를 만듭니다. 또한, 우리는 HIV - 1 감염 세포에 siRNA의 세포 유형 특정 배달 gp120 aptamer - siRNA의 chimeras를 활용. 키메라이 듀얼 기능은, 억제 HIV - 1 감염의 다양한 핵산 치료 요원 (aptamer와 siRNA) 결합 매우 적극적인 antiretroviral 치료 (HAART)을 실패한 환자에 대한 aptamer - siRNA의 chimeras에게 매력적인 치료 후보를 만들기위한 상당한 잠재력을 보여줍니다.
프로토콜
1. RNA 라이브러리의 준비
- 시작 DNA 라이브러리는 임의의 순서의 50 세포핵를 포함하고 통합 DNA 기술 (코랄빌 아이오와)에 의해 합성되었다. N 50 - - 전투 편대 ACG ACT CGC CCG A - 3 '(81 NT) AGG ACG ATG CGG 단일 좌초된 DNA oligo 라이브러리 시퀀스는 5' - GGG입니다. 임의의 지역은 T7의 체외의 해독에 대한 발기인 및 RT - PCR을위한 3 '태그를 포함한 지속적인 지역의 어귀이다. 5 '와 3'지속적인 시퀀스는 5는 '- TAA TAC GAC TCA CTA TAG GGA GGA CGA TGC GG - 3'(32 메르) 5 '- TCG GGC 개그 TCG TCT G - 3'(16 메르), 각각. -20 ° C.에 aliquots에 물 저장과 재고 솔루션을 확인하십시오
- PCR 2 MM MgCl 2, 각 dNTP 200 μm의와 함께, 3 μm의 5' -와 3' - primers 각을 사용하여 단일 좌초된 DNA oligo 임의의 라이브러리 (0.4 μm의)를 확대. 원래 DNA 라이브러리의 풍부한을 보존하기 위해, 열 사이클에 PCR을 제한합니다. PCR 반응 (10 반응, 반응 당 100 μL) 후 QIAquick 젤 정화 키트 (QIAGEN)를 사용하여 증폭 dsDNA 풀을 복구할 수 있습니다.
- 제조 업체의 지침에 따라 DuraScription 키트 (Epicentre, 매디슨, WI)를 사용하여 RNA 라이브러리에 결과 dsDNA를 변환합니다. 전사 반응 혼합물에서 ribonuclease 모성 RNA를 만들어 2' - F - CTP 및 2' - F - UTP와 CTP 및 UTP를 교체하십시오. 일반적으로, μL DNA 템플릿, 2 μL 10 × 버퍼, 2 μL dATP, 2 μL dGTP, 2 μL 2' - F - dCTP, 2 2' - F - dUTP, 2 μL DTT 투석을 1 μg를 포함하는 반응을 20 μL를 준비 그리고 2 μL T7 RNA의 상온에서 효소 및 37 품어 ° 6 H. C를 (각 dNTP 50 MM.)
- 이후 8 % polyacrylamide / 7 M의 요소 젤로 템플릿 DNA를 제거하고 정화 DNase I (20 μL T7의 전사 반응 당 1.5 μL)와 반응을 소화. UV 분광 광도법에 의한 정화 RNA 라이브러리를 계량.
aptamers 2.에서는 체외 생성
- 선택하기 전에 선택 버퍼와 refolding 버퍼를 준비합니다. 100 MM Hepes, 산도 7.4를 포함하는 HEPES 버퍼를 준비합니다. 상온에서 산도의 가치와 다음 저장을 조정 NaOH를 사용합니다. 5 MM MgCl 2;, 5 MM CaCl 2; 13.5 MM KCl 50 MM의 Hepes의 산도 7.4, 750 MM NaCl을 포함하는 RNA의 refolding 버퍼를 (5xHBS) 준비합니다. 저염 RNA 결합 버퍼 (10 MM의 HEPES 산도 7.4, 50 MM NaCl, 1 ㎜ CaCl 2, 1 MM MgCl 2, 2.7 MM KCl, 10 MM DTT, 0.01 % BSA와 높은 소금 RNA 결합 버퍼 (10 준비 MM의 HEPES 산도 7.4, 150 MM NaCl, 1 ㎜ CaCl 2, 1 MM MgCl 2, 2.7 MM KCl, 10 MM DTT, 0.01 % BSA)가. -20 ° C.에서 이러한 버퍼를 저장
- 주로로 1-4에서 설명하는 SELEX을 수행합니다. 전에 모든 선택 둥근 1xHBS 버퍼 (10 MM의 HEPES 산도 7.4, 150 MM NaCl, 1 ㎜ CaCl 2, 1 MM MgCl 2, 2.7 MM KCl), 3 분 95 열이 ° C에서 refold RNA 풀 다음 천천히 냉각 37 ° C. 37 배양을 계속 ° C, 10 분.
- 일반적으로, nitrocellulose 필터와 특이 현상이 구속력 사전 adsorb을 최소화하기 위해 대상 HIV - 1 발 gp120 단백질과 사전 부화하기 위해, 30 분 nitrocellulose 필터 (HAWP 필터, 0.45 μm의)에 RNA 풀을 refolded.
- 4 SELEX 반올림 1 30 분 저염 RNA 바인딩을 버퍼에 대상 단백질과 사전 허가 RNA 풀을 품어. SELEX의 네 번째 라운드 후, 높은 소금 RNA 바인딩 버퍼를 사용합니다. SELEX 진행과 함께, gp120 단백질의 양을 줄이고 aptamer 선택의 엄중을 증가하기 위해 경쟁 업체의 효모 tRNA를 높일 수 있습니다.
- 선택의 첫 번째 사이클 들어, 200 μL 낮은 미리 지워 임의의 RNA 수영장 (40 μg, 1.5 nmol, 9x10 14 분자)와 HIV - 1 발 gp120 단백질 (0.23 nmol, RNA / 단백질 비율 6.5 / 1) 알을 품다 30 분 상온에서 회전 플랫폼에 소금 RNA 결합 버퍼.
- 미리 침수 nitrocellulose 필터를 통해 반응을 통과하고 1 ML 구속력이 버퍼로 씻으십시오.
- 95 200 μL 용출 버퍼 (7 M 우레아 5 MM EDTA (에틸렌 다이아 민 테트라 초산)) · Microcon YM - 30 컬럼 페놀 / 클로로포름 추출과 집중에 의해 다음 5 분 C.와 필터에서 바운드 RNA를 Elute
- RT - PCR ThermoScript 시스템 (Invitrogen)를 사용하여 복구 RNA의 수영장을 녹음하고 PCR의 15주기에 대한 증폭을 역방향.
- QIAquick 젤 정화 키트를 사용하여 증폭 dsDNA 수영장 정화와 같은 선택의 다음 라운드를 위해 위에서 설명한 고쳐 쓰다.
3. SELEX 진행 필터 구속력을 분석하여 모니터링
- 필터 구속력을 분석하여 aptamers의 SELEX 진행 상황을 모니터링합니다. T4 폴리 뉴클레오 타이드 키나제와 γ - 32P - ATP와 다음 5' - 삼인산를 시작하고 레이블을 제거하는 CIP와 RNA의 수영장을 처리합니다.
- CIP의 열 10 pmol 다음 ° 5 분 C와이 얼음에 진정 95에서 RNA 라이브러리를 처리. 이후, 2를 추가PNK 버퍼, T4 폴리 뉴클레오 타이드 키나제 1 μL, 20 μL로 감마 - P 1 μL 32 ATP와 물을 μL.
- 37 품어 ° C 30 분, 후 20 ^ 무를 추가, 물을 L과 G - 50 컬럼에 의해 반응을 정화. 마지막으로, 250 nm의 최종 농도에 표시 RNA 40 μL를 구하십시오.
- 분석하기 전에, 1xHBS 버퍼 (10 MM의 HEPES 산도 7.4, 150 MM NaCl, 1 ㎜ CaCl 2, 1 MM MgCl 2, 2.7 MM KCl), 95 열에 refold 표시 RNA 풀 ° C 3 분 후 천천히 냉각을위한 37 ° C. 37 배양을 계속 ° C, 10 분.
- 여기에 예를 들어 바인딩 반응 100 μL를 수행합니다. 부화 최종 표시된 gp120 단백질 (100 NM) 30 분 높은 소금 RNA 결합 버퍼에 특이 현상이 경쟁 tRNA (100 NM)의 10 배 초과 어금니와 RNA 수영장 (10 NM).
- 사전 서부 유럽 표준시 nitrocellulose 필터에 의해 바인딩 반응 50 μL를 분리합니다.
- 2 ML 바인딩 버퍼와 필터를 세척하고 다목적 섬광 카운터 (베크맨 쿨터)를 통해 필터에 보관 방사능을 계산합니다. 입력 컨트롤로서, 동시에 구속력이 반응의 나머지 50 μL를 계산합니다. 바인딩 친화력으로 입력 RNA의 필터에 보관 RNA의 %를 계산합니다.
4. 복제, 시퀀싱 및 정렬
- 더 이상의 농축이 추가 선택 반올림 다음도 관찰되지 않은 경우 11 라운드 후, 다음 RNA 수영장의 최대한의 결합은 잠재적으로 도달했습니다.
- RT - PCR ThermoScript 시스템 (Invitrogen)를 사용하여 매우 풍부한 aptamer 수영장 (12 일 RNA 풀)을 녹음 바꾸게 후 PCR로 cDNA를 증폭 결과. QIAquick 젤 정화 키트 (QIAGEN)를 사용하여 PCR 제품 정화. 클론 젤 - 정화 TA 클로닝 벡터 PCR 2.1 (Invitrogen)에 DNA 제품을. 총, 170 각각의 클론을 예방하고 더욱 개인 시퀀스를 얻을 시퀀싱 DNA에 의해 그들을 식별합니다.
- 개인 aptamer 시퀀스의 정렬에 따라 여섯 가지 그룹으로 각각의 클론을 분류. 왜냐하면 그들의 그룹 내에서 상대적인 풍요의 자세한 특성에 대한 각 그룹 (A - 1, A - 5, A - 9, A - 12, A - 28 B - 68) 한 대표 순서를 선택할 수 있습니다.
5. 체외의 전사에 의해 aptamer와 키메라 RNAS의 생성
- 직접 PCR 2 MM MgCl 2, 각 dNTP 200 μm의와 함께, 2 μm의 5' -와 3' - primers 각을 사용하여 이중 좌초된 DNA 템플릿을 생성하고, QIAquick 젤 정화 키트를 사용하여 결과 PCR 제품을 복구합니다.
- DuraScription 키트 (Epicentre, 매디슨, WI)를 사용하여 생성된 PCR은 DNA의 템플릿에서 키메라 감각의 가닥을 고쳐 쓰다. 전사 반응 혼합물에서 RNase 저하에 강한 RNA를 생산 2' - F - CTP 및 2' - F - UTP와 정식 CTP 및 UTP를 교체하십시오.
- 일반적으로, μL DNA 템플릿 1 μg, 2 ML 10xbuffer, 2 μL dATP, 2 μL dGTP, 2 μL 2' - F - dCTP, 2 μL 2' - F - dUTP, 2 μL DTT 2를 포함하는 반응을 20 μL를 품어 T7 RNA 중합 효소의 37 ° 6 H에 대한 C 및 후 페놀 추출 및 에탄올 침전 다음과 같은 바이오 스핀 30 열 (바이오 래드)로 정화.
- 인터페론 반응을 방지하기 위해 추가로 시작 5' - 삼인산를 제거하는 CIP하여 베꼈는데 RNA를 취급. ° C 60 분 37 성적 3 μg, 버퍼 3 6 μL와 CIP의 0.25 μL를 포함하는 반응이 총 60 μL를 품어. 후 클로로포름 / 페놀 수렴 및 에탄올 침전, 물에 resuspend RNA 펠릿.
- chimeras를 준비에 버퍼, 열을 refolding에 적절한 안티 센스 RNA만을 감지 스트랜드 RNA를 숨겨주 chimeras을 결합 95 ° C 37 냉각 후 3 분 및 대한 ° C 천천히. 37 배양을 계속 ° C, 10 분. finial의 1xHBS 버퍼에서 단계를 refolding 수행합니다. 예를 들어 : 10 μm의 키메라 감각 스트랜드의 혼합 10 μL, 10 μm의의 안티 센스 가닥과 25 μL 시스템에 5 μL refolding 버퍼 (5xHBS) 10 μL.
6. 겔 이동 assays에 의해 해리 상수의 결정
- 결국 P 32 라벨을 대표하는 각 그룹과 chimeras 감각의 가닥에서 aptamer 후 위에서 설명한 refold 1xHBS 버퍼에서 RNA.
- 40 % 아크릴 아미드 / BIS 솔루션, 19.375 ML의 물, 10 % 암모늄 persulfate (APS) 솔루션, 30 μL TEMED 150 μL의 3.125 ML과 10xTBE 버퍼 2.5 ML를 혼합하여 5 % 젤 25 ML을 준비합니다. 겔은 30 분 안에 중합한다. 조심스럽게 빗을 제거하고 실행 버퍼 (1xTBE)와 우물을 씻어 바늘로 장착되어 30 ML 주사기를 사용합니다.
- 젤 단위의 조립을 완료하고 전원 공급 장치에 연결합니다. 겔 4에서 180 V 한 시간 동안 사전 실행할 수 있습니다 ° C.
- 순차적으로 원하는 농도에 바인딩 버퍼와 HIV - 1Bal gp120 단백질을 희석. 최종 Rgp120의 eaction의 농도는 0, 1, 5, 10, 20, 40, 80, 160, 320, 640 NM입니다. 5' - P의 일정한 양의 RNA (10 NM) 30 분 상온에서 회전하는 플랫폼에서 구속력을 버퍼에 gp120 단백질의 농도 증가 (반응 μL 총 20) 32 - 최종 분류를 품어.
- 부화 후 5 %의 비 denaturing polyacrylamide 젤에 5 μL 기본 로딩 버퍼와 부하와 바인딩 반응 20 μL를 섞는다. 1 ㎜ EDTA (에틸렌 다이아 민 테트라 초산), 0.1 % Bromophenol 블루, 0.1 % 크실렌 Cyanol FF, 0.1 % 오렌지 G, 40 % 글리세롤, 10 MM 트리스 - HCL, pH를 7.5를 포함하는 원시 로딩 버퍼 (4X) 준비합니다. -20 ° C.에 aliquots에 저장
- 다음 전기 (4 180 V ° 2 시간 C, 보조 염료가 젤의 중간에서 실행까지)은 형광체 이미지를 화면 젤 노출과 태풍 스캐너를 사용하여 방사성 물질을 계량.
- 그래프 패드 프리즘과 비선형 곡선 회귀를 사용하여 분리 상수를 계산합니다.
7. 유동세포계측법에 의해 세포 표면에 결합 연구
- 소음기의 siRNA 라벨 키트 (앰비온)을 사용하여 형광 aptamer와 chimeras를 생성합니다. 순서대로 다음과 같은 시약을 추가합니다 : 22.5 μL nuclease - 무료 물, 5 ML 10 × 라벨링 버퍼, 15 μL RNA (5 μg), 7.5 μL 라벨 염료.을 37 총 50 μL 라벨 반응 ° C 1 시간을 품어.
- 부화 후, 5.0 μL (0.1 권) 5 M NaCl과 125 μL (2.5 권) 차가운 100 % EtOH를 추가하고 철저히 섞는다. 60 분 -20 ° C에서 알을 품다. 20 분 4 ° C에서 최고 속도로 원심 분리기. 175 μL 70 % EtOH로 뜨는하고 씻어 펠렛을 제거합니다. 다음 공기 건조 어둠 속에 펠렛 및 nuclease없는 물 15 μL가 표시 RNA를 일시 중지합니다.
- 260 nm의에서와 형광 염료에 대한 흡광도 최대 레이블 RNA의 흡광도를 측정. 에서 제공하는 계산기에 따라 염료 비율 및 RNA 농도 : 기본 계산 http://www.ambion.com/techlib/append/base_dye.html을 .
- 위에서 설명한 버퍼를 refolding에 섞어 Cy3 - 표시 chimeras 감각의 가닥과 안티 센스 가닥과 refold.
- 에이즈 연구 및 참조 시약 프로그램 5, 6를 통해 gp160과 CHO - EE 제어 세포를 표현 CHO - WT를 구합니다. GMEM - S 매체에있는 세포 (400 μm의 메티오닌 sulfoximine (MSX)와 글루타민 - 결함 최소 필수 중간) (Gibco, Invitrogen)를 성장. 37 humidified 5% CO 2 배양기에서 문화 세포 ° C.
- prewarmed 세척 버퍼 CHO - WT gp160 또는 CHO - EE 제어 세포를 세척, trypsinize 및 접시에서 분리합니다. 500 μL 바인딩 버퍼와 두 세포를 세척 후, 37 구속력이 버퍼와 부화에있는 세포 알약을 resupend ° C를 30 분. 펠렛 세포 그리고 400 nm의 Cy3 - 라벨 실험 RNAS을 포함 prewarmed 바인딩 버퍼의 50 μL에서 그들을 resuspend.
- 37 부화 후 ° C 40 분, prewarmed 바인딩 버퍼 500 μL와 세포 세 번 씻고, 그리고 마지막으로 바인딩 버퍼 350 μL에 resuspend 37에 prewarmed ° C 즉시 유동세포계측법으로 분석합니다.
8. 라이브 세포 공촛점 현미경에 의해 내면화하고 세포 내 현지화 연구
- 분석 중 하루 전에, 24 H.의 약 70 %의 합류를 허용 2 ML GMEM - S 매체 0.3x10 6 시딩와 35mm 판에서 CHO - WT gp160과 CHO - EE 제어 세포 성장
- 실험 당일, prewarmed PBS 1 ML과 세포를 씻으십시오. 37 30 분 사전 예열 완료 성장 매체 1 ML로 품어 ° C.
- 위에서 설명한대로 Cy3 - 레이블 aptamer - siRNA의 chimeras를 준비합니다. siRNA - 스틱은 위에 설명된대로 aptamer - 스틱 siRNA 복합을 형성 5' - Cy3 - 라벨 감각의 가닥을 포함과 함께 aptamer 스틱 refold을 품어.
- 분석하기 전에, 4 ° C.에 aliquots의 물 저장소에 Hoechst 33342 (라이브 세포 핵 염색, 분자 프로브, Invitrogen, CA)의 0.15 MG / ML 솔루션을 준비
- 37 15 분 0.15 MG / ML Hoechst 33342로 치료하여 세포를 얼룩 ° C. 바로 두 번 1.0 ML 신선한 매체와 염료를 씻어 2 ML 신선한 매체 prewarmed 바꿉니다.
- Cy3 - 라벨 37 5 %의 CO2 인큐베이터 현미경에 살고있는 세포 공촛점 현미경에 대한 언론과 부화에 100 nm의 최종 농도에 aptamer - siRNA 키메라 ° C. 추가
- 이미지에게 40x 배율에 물이 침수 아래의 2 광자 현미경 공촛점 시스템을 인버티드 자이스 혈구 LSM 510 메타를 사용하여 매 15 분를 수집합니다.
9. 체외 HIV - 1 p24 항원 도전과 분석에
- ATCC에서 CCRF - CEM 세포를 구입합니다. 10% 태아 소 혈청 (FBS, HyClone), L - 글루타민과 1xPen - Strep (Gibco, Invitrogen)을 보충 (Cellgro, Mediatech 주) RPMI - 1640으로 성장. 37 humidified 5% CO 2 배양기에서 문화 세포 ° C.
- 말초 혈액 mononucle를 구합니다호프 내셔널 메디컬 센터 (진료소 직원)의 도시에서 건강한 기증자로부터 AR 샘플.
- Ficoll - Hypaque 솔루션을 통해 원심 분리 (Histopaque - 1077, 시그마)에 의해 전체 혈액 PBMCs을 분리. Dynabeads CD8 (Invitrogen, CA)를 사용하여 PBMCs에서 CD8 세포 (T-cytotoxic/suppressor 세포) 비우다.
- T - 세포 활성화 매체 세포를 성장 (BioE, 세인트 폴, MN). 37 humidified 5% CO 2 배양기에서 문화 세포 ° C.
- HIV - 1 IIIB와 에이즈 연구 및 참조 시약 프로그램에서 NL4 - 3 바이러스 및 HIV - 1 발 바이러스를 구합니다. -80에서 aliquots에서 바이러스, 저장 ° C. 전파 후
- HIV 바이러스와 CCRF - CEM 세포 또는 인간 PBMCs (IIIB, NL4 - 3 발)을 (뫄 0.001 또는 0.005) 감염. 포스트 감염 24 시간 후에 부드럽게 무료로 바이러스를 제거하는 PBS로 세 번 세포를 씻으십시오. 문화 37 5 % CO 2 배양기 현미경에 감염된 세포 ° C 4 일간을 계속합니다.
- 치료를 RNA하기 전에, 부드럽게 무료로 바이러스를 제거하는 PBS로 세 번 감염된 세포를 씻어. 함께 품어 2x10 4 감염된 세포와 감염되지 않은 세포 3x10 4 37 ° C (트리플 분석 잘 당 100 μL)에서 96 - 웰 플레이트에 400 nm의 최종 농도를 실험 RNAS을 refolded.
- 다른 시간 (3 D 5 D, 7 D 9 D) 및 p24 분석까지 -20 ° C에 상점에서 문화 supernatants를 (물론 당 10 μL)를 수집합니다.
- HIV - 1 p24 항원 엘리사 키트를 사용하여 p24 항원 분석을 수행합니다.
10. 정량 RT - PCR 분석에 의해 siRNA 기능 검출
- HIV 바이러스 (IIIB, NL4 - 3 발)로 CCRF - CEM 세포 또는 인간 PBMCs를 감염하고 위에 설명된대로 실험 RNAS (400 NM)로 처리합니다.
- 치료, 펠렛 세포 및 7 일 후에 STAT - 60로 총 RNAS을 분리. 게놈 DNA를 제거하고 총 RNA 2 μg을 사용하여 cDNA를 만들어 DNase I으로 총 RNAS을 처리합니다.
- ; 1.5 ML 10 × DNase 버퍼, 4 μL RNA (2 μg) 0.5 μL RNain 억제제와 1 1.0 μL RNase 무료 DNase I. 부화 총 15 μL 반응 37 ° C 8 ML nuclease - 무료 물 다음 시약을 혼합 80 시간 열 ° C 10 분, 바로 DNase I.를 inactivate 얼음에서 반응을 진정합니다.
- 65 다음 2 μL 임의의 프라이머 (μL / 50 NG) 및 1μL dNTP (10 ㎜) 위의 반응 혼합물로, 그리고 열 추가 ° 5 분 C. 바로, 얼음에서 반응을 진정.
- 다음과 같은 시약을 추가 : 5 ML 5xFirst 스트랜드 버퍼, 2.5 ML 0.1 M DTT, 0.5 ML RNain 방지제와 1.0 ML MMLV - RT 있습니다. 25 총 27 ML 반응을 품어 1 시간 ° 10 분 및 37 C ° C. 70 반응, 열 혼합 후 ° C 15 분 얼음에 진정 후 역방향 transcriptase를 inactivate하고하십시오. cDNA는 qRT - PCR 분석을위한 준비가되었습니다.
- 정량 RT - PCR 400 NM의 최종 농도 (트리플 분석) 2 X IQ SyberGreen Mastermix 및 특정 프라이머 세트를 사용하여 대상 유전자의 표현을 분석할 수 있습니다. qPCR 데이터의 정상화를위한 내부 통제로 GAPDH 표현을 사용합니다.
11. 대표 결과 :
1. HIV - 1 gp120에 대한 새로운 발 RNA의 aptamers는 절연 및 특징.
으로 실험 섹션 5 '및 3'종료가 증폭 및 RNA 수영장에 베꼈는데이다에 고정 뇌관 지역 둘러싸인 50 NT 임의의 영역을 포함하는 초기의 DNA oligonucleotide 라이브러리에서 설명했다. 이것은 초기 라이브러리가 다양한 3 - D 구조의 광대한 배열로 접어 10 15 다양한 시퀀스 (1 nmol)로 구성되어 있습니다. 초기 라이브러리의 높은 복잡 성과 다양성이 대상 좋은 바인딩 친화력과 적극적인 구조의 존재를 보장 수도 있습니다.
선택 R5 변형 HIV - 1 gp120 발 봉투 단백질 7 바인딩 2' - fluoropyrimidine 수정 RNA의 aptamers를 선택 체외 SELEX 절차에서 (그림 1)을 사용합니다. 그림 1에 표시된, nitrocellulose 기반의 선택 전략은 구속력이 RNA 분자에서 특정 대상 바인딩 RNAS을 분리 수행됩니다. 단백질은 nitrocellulose에 개 때문에, 오직 RNA / 단백질 단지 또는 집계는 멤브레인에 유지 수 있으며, 무료로 RNAS가 씻겨서 있습니다. denaturing 조건에서 바운드 RNAS가 회복되고 반대 cDNA에 베꼈는데하고 다음 dsDNA로 증폭하고 다음을 선택주기위한 새로운 RNA 풀을 만들 수 베꼈는데 이후의 체외. 선택 엄중는 대상 단백질의 양을 감소와 경쟁 업체의 tRNA의 양을 증가 증가합니다. 각 선택에 사용되는 원형 RNA 수영장, 단백질과 경쟁의 tRNA의 양은 표 1에 표시됩니다.
필터 바인딩 분석하여 각 SELEX 사이클 이후에 선택의 진행 상황을 모니터링합니다. RNA의 %가 fil에서 유지로 바인딩 친화력을 평가총 RNA의 수영장에서 터. 시작 RNA 수영장 (1 - RNA)는 단지 멤브레인에 보관 입력 RNAS의 0.1 %를 보여줍니다. 그러나, 아홉 선택 반올림 후 아홉째 RNA 라이브러리 (9 RNA)는 바운드 입력 RNA의 9.72 %가 있습니다. 추가 선택 순찰 실시 있었지만, 더 이상의 농축은 RNA의 수영장 최대한 결합합니다 (그림 2A)에 도달되었음을 제시, 관찰하지 않습니다. 필터 바인딩 분석과 마찬가지로, 겔 변화 분석도 해리 상수를 결정하는 가장 인기있는 전략 중 하나입니다. 이 절차는 쉽고 편리합니다. 마찬가지로 그림 2B와 같이 겔 시프트 assays 추가 RNA의 수영장의 바인딩 활동을 확인합니다. 이러한 결과는 대상 단백질에 대한 높은 결합 특이성과 어떤 리간드가 연속 이러한 RNA의 웅덩이에 농축되는 입증 ..
복제 및 시퀀스 높은 농축 aptamer 풀 (12 RNA). 개인 복제된 aptamer 시퀀스의 정렬에 따르면, 여섯 가지 그룹으로 표 2에 나타난 분류됩니다. (A / G) TTGAGGGACC (A / G) : 클론의 40 % 정도 (그룹 I과 II는 aptamers)는 보존 순서가 포함되어 있습니다. 왜냐하면 그들의 그룹 내에서 상대적인 풍요의 추가 특성에 대한 : (A - 1, A - 5, A - 9, A - 12, A - 28 B - 68 예) 우리는 한 대표 각 그룹에서 순서를 선택합니다. 기본 겔 이동성 이동 분석을 통해 이러한 대표 aptamers의 해리 상수 (K D)은 (그림 3A) 계산됩니다. 예를 들어, A - 1, aptamers의 최고 52 NM (그림 3B)의 명백한 KD 가치가 있습니다. 마찬가지로 그림 3A와 같이, 이러한 선택 aptamers은 선택 대상 HIV - 1 발 gp120와 결합하지만, HIV gp120의 CM 단백질 수 없습니다.
2. 안티 gp120 aptamer 특별히 바인딩 및 HIV gp160을 표현하는 세포 internalized되고 세포 배양에서 HIV - 1 감염을 억제.
CHO - gp160 안정 HIV 봉투 당단백질 gp160의 표현 세포 선택한 방지 gp120 aptamers의 바인딩 및 국제화를 위해 테스트하는 데 사용됩니다. 그들이 봉투 처리에 필요한 재갈 인코딩 프로 테아제 부족 때문에 이러한 세포가 gp120과 gp41로 gp160을 처리하지 않습니다. 컨트롤로서 우리는 gp160을 표현하지 않는 부모 CHO - EE 세포 라인을 사용합니다. 흐름 cytometric 분석 (그림 4A)는 Cy3 - 라벨 aptamers 특별히 CHO - gp160 세포에 바인딩되는 계시지만 컨트롤 CHO - EE 전지. 또한, 실시간 라이브 세포 Z - 축 공촛점 현미경은 Cy3 - 라벨 aptamer는 선택적으로 배양 2 시간 후 CHO - gp160 전지 (그림 4B 그리고 4C) 내에 internalized는 것을 나타냅니다 아니지만 CHO - EE 제어 세포 ( 그림 4D). 그림 4C도 aptamer는 gp120 aptamers 아마도 수용체 - 매개 endocytosis를 통해 세포의를 입력 제시 세포질 내에 집계 것으로 나타났습니다.
HIV - 1 과제 분석에서 HIV - 1 감염 - PBMC 전지는 aptamers로 취급됩니다. 다른 일에 aptamers와 함께 게시물 치료는 매체의 aliquots은 바이러스성 p24 항원 수준 (그림 4E)에 대한 assayed 있습니다. 결과는 안티 - gp120 aptamers이 (A - 1) 나노 몰라 농도와 HIV - 1 p24 생산을 억제한다는 것을 보여주기.
3. 안티 gp120 aptamer - siRNA 키메라는 설계 및 셀 타입 특정 siRNA 전달 시스템으로서 효능을 평가
마찬가지로 그림 5A, aptamer와 nuclease 방지 2' - 플루오 UTP 및 2' - 플루오로 CTP에 포함된 siRNAs의 감각 스트랜드 세그먼트에 표시하고 체외의 박테리오 파지 전사에 의해 해당 dsDNA 템플릿에서 합성됩니다. 분자의 유연성, 두 염기 링커을 증가하기 위해 (UU) aptamer와 주사위 놀이를하는 사람 기판 부분 사이에 삽입됩니다. chimeras를 포함하는 siRNA를 준비하려면, 체외 베꼈는데 키메라 aptamer - 감지 스트랜드 고분자에 수정되지 않은 안티 센스 RNA 스트랜드의 몰 농도와 annealed 있습니다. 겔 시프트 분석 (그림 5B) 및 유동세포계측법 (그림 5C)에서 이러한 데이터는 chimeras 혼자 aptamers로 거의 동일한 바인딩 동질성을 유지 것을 나타냅니다. 실시간 공촛점 현미경 (그림 5D)에서 시간 코스 이미지는 A - 1 채널 키메라가 성공적으로 세포의 세포질에 internalized 수 있습니다 Cy3 - 레이블 것을 보여줍니다. 예상했던대로, 망상에 대한 이해는 CHO - EE 제어 세포 (그림 5E)와 관찰되지 않습니다.
마찬가지로, RNAS의 항바이러스 가능성은 HIV - 1 과제 분석에 의해 평가됩니다. aptamer와 키메라 모두 p24 생산을 억제하지만, 가장 강한 억제가 키메라 채널 A - 1 치료 관찰되어 HIV p24 항원 분석 (그림 6A 및 6B)의 결과는 표시됩니다.
siRNA 구성 요소가 apta와 함께 작동하는지 확인하려면메르는 감염된 세포에서 A - 1 키메라 채널의 국제화에 따라 우리는 양적 RT - PCR의 표현 assays에 의해 문신 / 레브 유전자 발현의 억제의 상대적 수준을 평가합니다. 혼자 aptamer는 문신 / 레브 유전자 표현 (그림 6C 및 6D)을 영향을주지 않았지만 우리는 chimeras에 감염된 세포의 치료 문신 / 레브 유전자의 입을 유도 수 있습니다 것을 발견했습니다. 이러한 결과는 aptamer 배달 siRNA는 RNAi를 실행되는 추가 지원을 제공합니다.
| 단백질의 양은, RNA 수영장과 tRNA는 선택에 사용 | |||||
| SELEX의 반올림 | 대상 / RNA 비율 | Gp120 단백질 | RNA 수영장 | 경쟁 업체의 tRNA | 선택 버퍼 |
| 1 | 1/6.5 | 229.8 pmol | 1.5 nmol (40.1 μg) | 0 | 낮은 소금 SELEX 버퍼 |
| 2 | 1/6.5 | 114.9 pmol | 0.75 nmol (20.1 μg) | 0.25 nmol (6.6 μg) | |
| 3 | 팔분의 일 | 76.6 pmol | 0.625 nmol (16.7 μg) | 0.25 nmol (6.6 μg) | |
| 4 | 팔분의 일 | 76.6 pmol | 0.625 nmol (16.7 μg) | 0.25 nmol (6.6 μg) | |
| 5 | 팔분의 일 | 38.3 pmol | 0.306 nmol (8.18 μg) | 0.5 nmol (13.2 μg) | 고 염분 SELEX 버퍼 |
| 6 | 팔분의 일 | 38.3 pmol | 0.306 nmol (8.18 μg) | 0.5 nmol (13.2 μg) | |
| 7 | 10분의 1 | 26.8 pmol | 0.268 nmol (7.16 μg) | 0.5 nmol (13.2 μg) | |
| 8 | 10분의 1 | 26.8 pmol | pmol 0.268 nmol (7.16 μg) | 1 nmol (26.4 μg) | |
| 9 | 10분의 1 | 15.3 pmol | 0.153 nmol (4.09 μg) | 1 nmol (26.4 μg) | |
| 10 | 10분의 1 | 15.3 pmol | 0.153 nmol (4.09 μg) | 1.5 nmol (39.6 μg) | |
| 11 | 1/12.5 | 7.66 pmol | 0.096 nmol (2.56 μg) | 2 nmol (52.8 μg) | |
| 12 | 1/12.5 | 7.66 pmol | 0.096 nmol (2.56 μg) | 2 nmol (52.8 μg) | |
표 1. 선택 조건. 단백질의 양은 각각의 선택 및 선택 버퍼에 사용되는 RNA 수영장과 tRNA가 표시됩니다.
| 그룹 | RNA | 랜덤 시퀀스 | 주파수 (140 클론) |
| 그룹 I | A - 1 | AATTGAGGGACCA CGCGCTGCTTGTTGTGATAAGCAG TTTGT CG GATGG | 33 (23.6 %) |
| B - 7 | AATTGAGGGACCA ACGCGAGGATGTGGATAGTGTGTA TTTGC GT GATGG | 3 | |
| A - 32 | AATTGAGGGACCG TTGGTAAAAGCCGGA AATTG AGCT TTTAC GGC GATGG | 5 | |
| B - 55 | AATTGAGGTACCGCG TTATTAGGAACA AATTG GAATTCTAAACGC GATGG | 2 | |
| A - 24 | AATAGAGGGACC CAGATATAGGCTACACGGATGATGGTGTATCTG GATGG | 1 | |
| B - 19 | AATAGAGGAACCG TTTCAGAAGACTACAGGTTAGTCCAATGAAGC GACGG | 1 | |
| B - 31 | AATAGAGGGACCG TGGACAATAATTTATGGTCA TTTATTGGCAC GATGG | 1 | |
| 그룹 II | A - 12 | AGTAGAGGAACCA AGCAATGGATGAATGCAAAAGTGTAAATGCTT GATGG | 10 (7.1 %) |
| 그룹 III | A - 9 | TGAGTTTGGGTAAATTTCCGGTTTCGGTTTACTCACGAAAGATCGGTCGG | 15 (10.7 %) |
| 그룹 IV | A - 28 | TAAAGGAGGGAAGGATGAGACCGCACGAAAAATATCAGCATACG TTTGTG | 10 (7.1 %) |
| 그룹 V | A - 5 | GAAACTAGTTTGAATAATGGTGTAGAGGAGGGTCAATAGTTTCG TTGGTG | 9 (6.4 %) |
| 그룹 VI | B - 68 | ACATAGTAATGACACGGAGGATGGAGAAAAAACAGCCATCTCTTGACGGT | 2 |
| 기타 | 오펀 순서 | 48 |
표 2. 정렬 및 RNA의 aptamers의 식별. 선택의 12 번째 라운드에 이어 선택한 RNA 수영장은 복제와 합성되었다. 모두 140 클론의 정렬 후, 안티 - gp120 aptamers 여섯 그룹이 확인되었습니다. aptamer 핵심 지역에만 무작위 시퀀스 (5' - 3 ') 표시됩니다. 여러 주파수로 발생 분리가 지정됩니다.
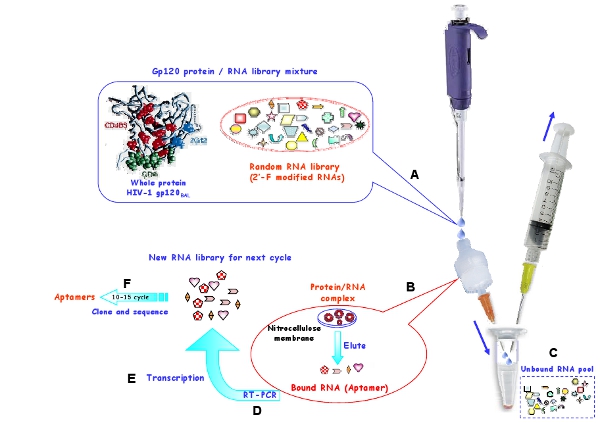
그림 1 : HIV - 1 gp120 단백질에 대한 발 RNA aptamers의 창출을위한, nitrocellulose 막을 사용하여 체외 선택 절차의 도식 표현. (A) RNA 시작 수영장과 대상 단백질은 복잡한 형태로 incubated되었습니다. (B) 바운드 RNA 분자는 세포막에 보유하고 denaturing 조건 하에서 세포막에서 eluted되었습니다. (C) 언바운드 RNAS은 씻어되었습니다. (D) 선택된 RNAS는 반대로 베꼈는데 및 PCR에 의해 증폭되었다. (E) 관련 DNA가 이후 다음 선택 사이클에 대한 새로운 RNA 수영장에 베꼈는데되었습니다. (F) 10-15 선택 반올림 후, 선택된 aptamers 복제 및 시퀀스되었습니다.
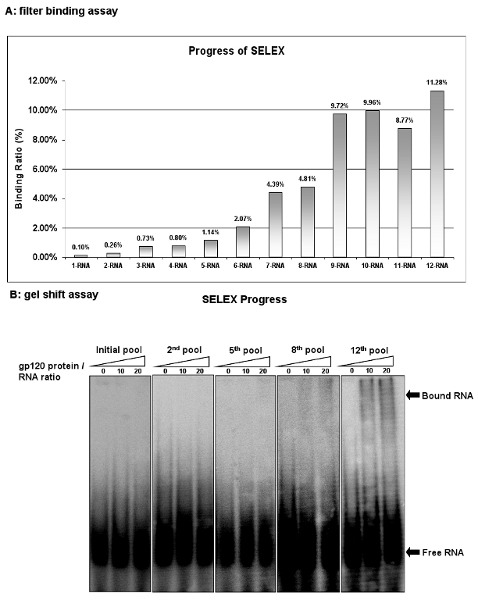
그림 2 : HIV - 1 gp120 aptamers 선택의 진행. (A)의 각 사이클에서 RNA 수영장의 바인딩 활동은 경쟁 업체의 tRNA와 필터를 결합 분석에 의해 분석되었다. 바인딩 활동은 RNA가 단백질 / 복합 RNA의 필터에 보관 입력의 비율로 계산되었다. (B)의 각 사이클에서 RNA 수영장의 바인딩 활동은 겔 변화 분석에 의해 분석되었다. 12 일 RNA 수영장은 가장 높은 바인딩 활동을 보여주었다.
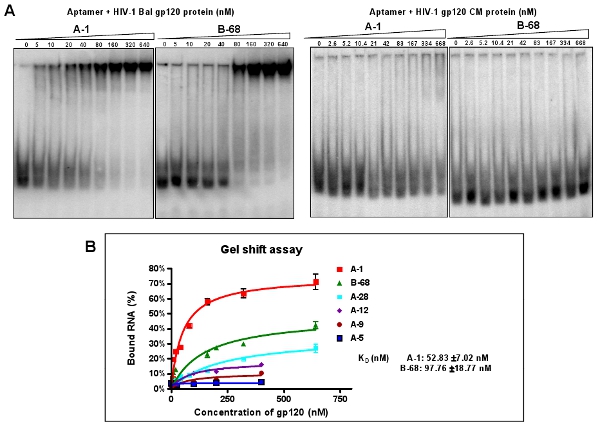
그림 3 : HIV - 1 gp120에 대한 발 선택한 개인 aptamers의 바인딩 활동 분석. (A) 5' 엔드 P 32 라벨 개별 aptamers는 대상 gp120 단백질 또는 불특정 CM 단백질의 양을 증가와 함께 incubated되었습니다. 바인딩 반응 혼합물은 젤의 이동성 이동 분석에 의해 분석되었다. Aptamer A - 1 및 B - 68는 대상 단백질과 함께 최고의 바인딩 친화력을 보여주지만, CM하지 단백질. 데이터 네의 평균 복제 나타냅니다. 겔 변화 분석에서 (B) 바인딩 곡선.
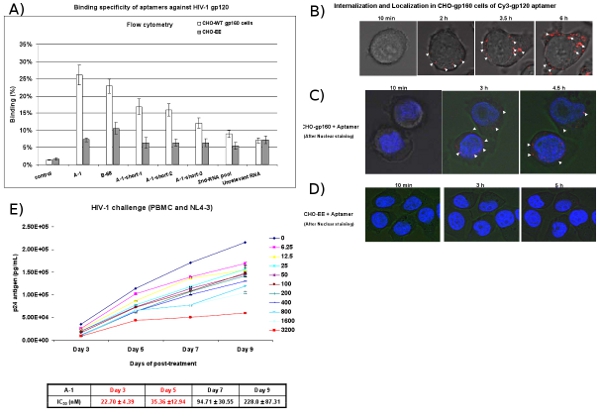
그림 4 : 셀 타입 특정 바인딩과 aptamers의 이해 연구. Cy3 - 라벨 RNAS의 (A) 세포 표면 바인딩은 유동세포계측법에 의해 평가되었다. Cy3 - 라벨 RNAS는 CHO - gp160 세포 CHO - EE 제어 세포에 바인딩을 위해 시험되었다. 선택한 aptamers는 셀 타입의 특정 결합 친화력을 보여주었다. 2 층 RNA 수영장과 관련이없는 RNAS는 부정적인 컨트롤로 사용되었습니다. 데이터는 세의 평균 복제 나타냅니다. (B) 국제화 분석. CHO - gp160 전지는 35mm 판 재배 및 실시간 라이브 셀 - 공촛점 현미경 분석을위한 문화 매체에 A - 1 Cy3 - 라벨의 100 nm의 농도와 incubated했다. 이미지는 15 분에서 수집된 있었다. 40X 배율을 사용하는 간격. (C, D) 현지화 분석. CHO - gp160 세포 CHO - EE 제어 전지는 35mm 접시에서 성장했다. A - 1 Cy3 - 라벨의 100 nm의와 부화하기 전에 전지가 Hoechst 33342 (라이브 세포 핵 염색)와 스테인드 후 실시간 공촛점 현미경을 사용하여 분석했다. (E)을 선택 안티 gp120 aptamers 이전 HIV - 1 NL4 - 3 바이러스에 감염된 인간 PBMCs에서 HIV - 1 복제를 억제. 다른 농도 및 시간 pointes가 소개되었습니다. IC50 값은 지정되었습니다. 데이터는 p24의 세중의 측정의 평균을 나타냅니다.
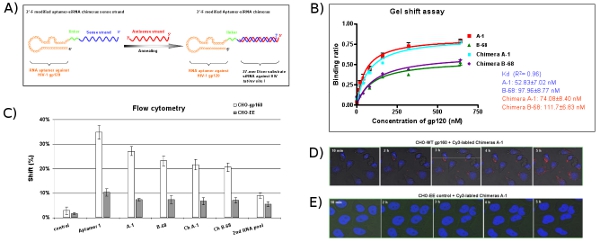
그림 5 : aptamer - siRNA 키메라 전달 시스템의 설계 및 평가. (A) 도식 aptamer - siRNA 키메라 RNAS : 방지 gp120 aptamer의 영역 gp120에 바인딩하고 siRNA는 HIV - 1 두드리는 소리 / 레브의 공통 엑슨를 타겟팅하는 책임이 있습니다. 2' - 플루오가 수정 aptamer - siRNA 감각 단일 가닥이 키메라 분자를 완료하기 위해 상호 보완적인 siRNA의 안티 센스 가닥의 어닐링에 의해 다음, 공동 베꼈는데되었습니다. aptamer와 siRNA 사이의 링커 (UU)는 녹색으로 표시됩니다. (B)뿐만 아니라 부모 aptamers로 비교 KD 가치를 aptamer - siRNA 키메라 RNAS 특별히 HIV 발 gp120 단백질을 바인딩합니다. 데이터는 세의 평균 복제 나타냅니다. (C) aptamers의 셀 타입의 특정 결합 연구. Cy3 - 라벨 RNAS는 CHO - gp160 세포 CHO - EE 제어 세포에 바인딩을 위해 시험되었다. 셀 쉬르Cy3 - 라벨 RNAS의 얼굴 바인딩은 유동세포계측법에 의해 평가되었다. 선택한 aptamers는 셀 타입의 특정 결합 친화력을 보여주었다. 2 층 RNA 수영장과 관련이없는 RNA는 부정적인 컨트롤로 사용되었습니다. 데이터는 두 나라의 평균 복제 나타냅니다. (D, E) 국제화 및 현지화 세포 분석합니다. CHO - gp160 전지는 35mm 접시 재배되었으며 Hoechst 33342 (라이브 세포 핵 염색)와 스테인드되었습니다. 그 후, 세포는 이전에 설명한 것처럼 실시간 라이브 세포 공촛점 현미경 분석 키메라 Cy3 - 라벨의 100 nm의 농도와 문화 매체 incubated되었습니다.
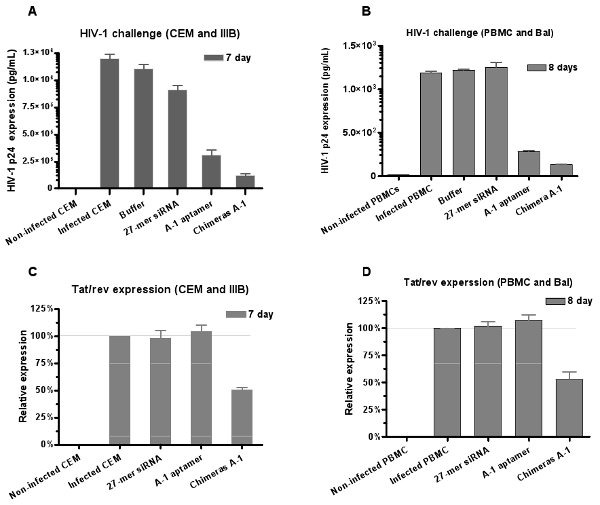
그림 6 : aptamer - siRNA의 chimeras로 인한 HIV - 1 감염의 이중 억제. 안티 gp120 aptamer와 aptamer - siRNA의 chimeras 모두 각각 (A) CEM 세포 (IIIB 변형)에서 HIV - 1 감염을 무력화하고 (B) 인간의 PBMCs (발 변형) 문화. 데이터는 p24의 세중의 측정의 평균을 나타냅니다. chimeras이 표시 혼자 aptamer보다 강한 억제를 보여줬하는 aptamers PBMCs에서 아래쪽 규제 두드리는 소리 / 레브 유전자 발현에 의해 전달 (C, D) siRNA. 데이터는 세의 평균 복제 나타냅니다.
토론
관내는 따라서 타겟 분자 8 매우 구체적이고 꽉 바인딩을 제공하고, 구체적이고 안정 입체 형태를 가정 핵산을 진화에 Aptamers 있습니다. 낮은 nanomolar 구속력 친화력과 목표를 aptamers의 절묘한 특이성은 생체내 이미징 및 치료제 9, 진단을 위해 다양한 도구를합니다. 타겟 siRNA 전달을위한 aptamer 기술의 도래와 함께 이제 siRNAs 10-12의 수용체 매개 end...
공개
감사의 말
우리는 브리타 휸, Guihua 일, 해리스 Soifer 및 도움이 토론에 대한 리사 쉐러 감사합니다. CHO - EE 및 CHO - gp160 셀 :이 작품은 NIH AIDS 연구 및 참조 시약 프로그램, AIDS 학부, NIAID, NIH를 통해 얻은되었습니다 보건 AI29329 다음과 같은 시약을 JJR에게 수여 HL07470 국립 연구소에서 보조금에 의해 지원되었다 라인, pNL4 - 3 륙 벡터, DAIDS, NIAID에서 HIV - 1 gp120 발.
자료
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 | 댓글 (옵션) |
|---|---|---|---|
| MF - Millipore 막 필터 | Millipore | HAWP01300 | 기공 크기는 0.45 μm의 |
| Swinnex 필터 홀더 | Millipore | SX0001300 | 13mm 직경 |
| QIAquick 겔 추출 키트 | QIAGEN | 28,706 | DNA 정화 |
| Microcon YM - 30 칼럼 | Millipore | 42,410 | RNA 농도 |
| 바이오 스핀 30 열 | 바이오 래드 | 732-6250 | RNA 정화 |
| DNA 형성 촉매의 DNA PCR 효소 | 시그마 - 알드리치 | D1806 | |
| RT - PCR ThermoScript 시스템 | Invitrogen | 11146-024 | |
| T7의 전사 키트 DuraScribe | EpiCentre | DS010925 | |
| PCR을위한 dNTP | 로체 | 1 581 295 | |
| Ribonucleic 산, E.coli에서 전송 | 시그마 - 알드리치 | R1753 | tRNA의 경쟁 |
| HIV - 1 바 - L gp120 단백질 | 에이즈 연구 및 참조 시약 프로그램 | 4961 | 타겟 단백질 |
| 소음기의 siRNA 라벨 키트 - Cy3 | 앰비온 | 1632 | |
| 산성 페놀 / 클로로포름 1분의 5 솔루션 (산도 4.5) | 앰비온 | AM9720 | |
| 클로로포름 / 이소프로판올 1분의 24 솔루션 | 시그마 | C0549 | |
| 카프 장 인산 가수 분해 효소 (CIP) | 뉴 잉글랜드 BioLab | M0290L | |
| T4 폴리 뉴클레오 타이드 키나제 | 뉴 잉글랜드 BioLab | M0201L | |
| 글리코겐 | 로체 | 10 901 393 001 | RNA 침전 |
| 감마 - P 32 - ATP | MP 바이오 메디컬 | 013502002 | Radiactivity |
| 40% AccuGel 19시 1분 | 국립 진단 | EC - 850 | |
| 10xTBE | 국립 진단 | EC - 860 | |
| N, N, N, N' - Tetramethylethylenediamine (TMEMD) | 시그마 - 알드리치 | T9281 | |
| 암모늄 persulfate (APS) | 시그마 - 알드리치 | A3678 | |
| L - methioine sulfoximine | 시그마 - 알드리치 | M5379 - 250 MG | |
| RPMI 미디어 1640 | Invitrogen | 11835-030 | |
| 나트륨 비카보네이트 솔루션, 7.5 % W / V | Invitrogen | 25080-094 | |
| 최소 필수 중간 (멤) (10 ) | Invitrogen | 11430-030 | |
| 멤이 아닌 필수 아미노산 (100 ) | Invitrogen | 11140-050 | |
| PCR 2.1 TA 클로닝 키트 | Invitrogen | K2040 - 01 |
참고문헌
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Robertson, D. L., Joyce, G. F. Selection in vitro of an RNA enzyme that specifically cleaves single-stranded DNA. Nature. 344, 467-468 (1990).
- Fitzwater, T., Polisky, B. A SELEX primer. Methods Enzymol. 267, 275-301 (1996).
- Weiss, C. D., White, J. M. Characterization of stable Chinese hamster ovary cells expressing wild-type, secreted, and glycosylphosphatidylinositol-anchored human immunodeficiency virus type 1 envelope glycoprotein. J Virol. 67, 7060-706 (1993).
- Vodicka, M. A. Indicator cell lines for detection of primary strains of human and simian immunodeficiency viruses. Virology. 233, 193-198> (1997).
- Zhou, J. Selection, characterization and application of new RNA HIV gp 120 aptamers for facile delivery of Dicer substrate siRNAs into HIV infected cells. Nucleic Acids Res. , (2009).
- Mayer, G. The chemical biology of aptamers. Angew Chem Int Ed Engl. 48, 2672-2689 (2009).
- Famulok, M., Hartig, J. S., Mayer, G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy. Chem Rev. 107, 3715-3743 (2007).
- Chu, T. C., Twu, K. Y., Ellington, A. D., Levy, M. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34, e73-e73 (2006).
- McNamara, J. O., 2nd, . Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat Biotechnol. 24, 1005-1015 (2006).
- Dassie, J. P. Systemic administration of optimized aptamer-siRNA chimeras promotes regression of PSMA-expressing tumors. Nat Biotechnol. 27, 839-849 (2009).
- Zhou, J., Rossi, J. J. The therapeutic potential of cell-internalizing aptamers. Curr Top Med Chem. 9, 1144-1157 (2009).
- Zhou, J., Li, H., Li, S., Zaia, J., Rossi, J. J. Novel dual inhibitory function aptamer-siRNA delivery system for HIV-1 therapy. Mol Ther. 16, 1481-1489 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유