Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Développement d'un type de cellule spécifique anti-VIH gp120 aptamères pour la livraison de siRNA
Dans cet article
Résumé
Plusieurs 2'-fluoro aptamères d'ARN contre le VIH-1BA-L gp120 avec nanomole affinité sont isolées à partir d'une bibliothèque d'ARN en In vitro La procédure SELEX. Un nouveau double fonction inhibitrice anti-gp120 aptamère-siRNA chimère est créé et montre prometteur pour systémiques thérapie anti-VIH.
Résumé
L'épidémie mondiale d'infection par le VIH a créé un besoin urgent de nouvelles classes d'agents antirétroviraux. La puissante capacité d'interférents (SI) des ARN pour inhiber l'expression des transcrits d'ARN complémentaires est exploitée comme une nouvelle classe d'agents thérapeutiques pour une variété de maladies dont le VIH. Beaucoup de rapports précédents ont montré que les nouvelles stratégies à base RNAi anti-VIH/SIDA thérapeutiques très prometteurs, mais un obstacle majeur à l'application réussie thérapeutiques et la traduction clinique de siRNA est une livraison efficace. En particulier, compte tenu de la sécurité et l'efficacité des traitements à base RNAi, il est hautement souhaitable de développer une approche ciblée intracellulaire livraison de siRNA à des populations spécifiques de cellules ou de tissus. Le VIH-1 protéine gp120, une glycoprotéine de l'enveloppe sur la surface du VIH-1, joue un rôle important dans l'entrée virale dans les cellules CD4. L'interaction de la gp120 et du CD4 qui déclenche le VIH-1 entrée et initie la fusion cellulaire a été validé comme cliniquement pertinente anti-viraux stratégie de découverte de médicaments.
Ici, nous avons d'abord discuter de la sélection et l'identification de la 2'-F modifiés anti-VIH gp120 aptamères d'ARN. En utilisant une méthode SELEX nitrocellulose filtre classique, plusieurs aptamères nouvelle avec une affinité nanomolaire ont été isolés à partir d'un aléatoire 50 bibliothèques nt ARN. Pour réussir à obtenir des espèces lié avec une affinité plus élevée, la rigueur de sélection est soigneusement contrôlée en ajustant les conditions. Les aptamères sélectionnés peuvent se lier spécifiquement et rapidement être internalisés dans les cellules exprimant la protéine d'enveloppe du VIH-1. De plus, les aptamères seul peut neutraliser le VIH-1 infectant. Sur la base des meilleures aptamère A-1, nous créons également un roman à double fonction inhibitrice anti-gp120 aptamère-siRNA chimère dans laquelle les deux l'aptamère et les portions siARN ont une puissante activité anti-VIH. De plus, nous utilisons la gp120 aptamère-siRNA chimères pour un type de cellule spécifique de la livraison de siRNA sur le VIH-1 des cellules infectées. Cette double fonction chimère montre un potentiel considérable pour combiner divers agents acides nucléiques thérapeutiques (aptamère et de siRNA) à supprimer l'infection VIH-1, ce qui rend les chimères aptamère-siRNA attrayants candidats thérapeutiques pour les patients en échec thérapie antirétrovirale hautement active (HAART).
Protocole
1. Préparation de la bibliothèque de l'ARN
- La banque d'ADN de départ contient 50 nucléotides de séquences aléatoires et a été synthétisé par Integrated DNA Technologies (Cedar Rapids, Iowa). La seule séquence bloqués banque d'ADN oligo est 5'-GGG ATG AGG ACG CGG - N 50 - CAG ACG ACT CCG GCC A - 3 '(81 nt). La région est flanquée par hasard régions constantes, qui comprennent le promoteur T7 pour transcription in vitro et une balise de 3 'pour la RT-PCR. Les 5 'et 3' des séquences constantes sont 5 '- TAA TAC GAC TCA CTA GCT GGA GGA CGA TGC GG - 3' (32 mer) et 5 '-TCG TCG TCT GGC GAG G - 3' (16 mer), respectivement. Faire solution mère avec de l'eau et de stocker en aliquots à -20 ° C.
- Amplifier le seul brin d'ADN oligo aléatoire des bibliothèques (0,4 M) par PCR en utilisant 3 uM chacun de 5'-et 3'-amorces, avec 2 mM de MgCl2 et 200 uM de chaque dNTP. Afin de préserver l'abondance de la banque d'ADN d'origine, de limiter à dix cycles de PCR. Après les réactions de PCR (10 réactions, 100 ul par réaction), récupérer le pool ADNdb amplifié en utilisant un kit de purification QIAquick Gel (QIAGEN).
- Convertir le ADNdb résultant d'une bibliothèque de l'ARN en utilisant le kit DuraScription (Epicentre, Madison, WI) en suivant les instructions du fabricant. Dans le mélange réactionnel de transcription, de remplacer CTP et UTP avec 2'-F-CTP et 2'-F-UTP pour produire ribonucléase ARN résistant. Typiquement, préparer 20 ul de réaction contenant 1 ug de protéine purifiée de matrice d'ADN, 2 pl de tampon 10 x, 2 uL dATP, dGTP 2 pl, 2 uL 2'-F-dCTP, 2 pl 2'-F-dUTP, DTT 2 uL et 2 ul T7 RNA polymérase à température ambiante, puis incuber à 37 ° C pendant 6 h. (50 mM de chaque dNTP).
- Par la suite digérer la réaction avec la DNase I (1,5 l par 20 uL réaction de transcription T7) pour enlever la matrice d'ADN et purifier par une polyacrylamide à 8% / 7 M d'urée de gel. Quantifier la bibliothèque de l'ARN purifié par spectrophotométrie UV.
2. Génération in vitro d'aptamères
- Avant la sélection, de préparer le tampon de sélection et de tampon de repliement. Préparer un tampon HEPES contenant 100 mM Hepes, pH 7,4. Utilisez NaOH pour ajuster la valeur du pH et de stocker à température ambiante. Préparer un tampon de repliement d'ARN (5xHBS) contenant 50 mM HEPES pH 7,4, 750 mM NaCl, 5 mM MgCl 2, 5 mM de CaCl2, 13,5 mM de KCl. Préparer un tampon ARN faible teneur en sel de liaison (10 mM HEPES pH 7,4, NaCl 50 mM, 1 mM de CaCl2, MgCl2 1 mM, 2,7 mM de KCl, 10 mM de DTT, 0,01% de BSA et un tampon ARN riche en sel de liaison (10 mM HEPES pH 7,4, NaCl 150 mM, 1 mM de CaCl2, MgCl2 1 mM, 2,7 mM de KCl, 10 mM de DTT, 0,01% de BSA). Stocker ces tampons à -20 ° C.
- Effectuez les SELEX principalement comme décrit 1-4. Avant chaque tour de sélection, repliez les piscines ARN dans 1xHBS tampon (10 mM HEPES pH 7,4, NaCl 150 mM, 1 mM de CaCl2, MgCl2 1 mM, 2,7 mM de KCl), de la chaleur à 95 ° C pendant 3 min, puis refroidir lentement à 37 ° C. Continuer d'incubation à 37 ° C pendant 10 min.
- Généralement, afin de minimiser les liaisons non spécifiques avec les filtres de nitrocellulose, de pré-adsorber les piscines ARN replié à une nitrocellulose (HAWP filtre 0,45 um) filtre pendant 30 minutes, avant l'incubation avec la cible du VIH-1 Bal protéine gp120.
- Incuber la piscine prédédouanés ARN avec la protéine cible dans peu de sel ARN tampon de liaison pendant 30 min pour les rondes SELEX 1 à 4. Après le quatrième tour de SELEX, utiliser un tampon ARN riche en sel contraignant. Avec les progrès de SELEX, réduire la quantité de protéine gp120 et augmenter les ARNt de levure concurrents, afin d'accroître la rigueur de la sélection des aptamères.
- Pour le premier cycle de sélection, incuber les prédédouanés piscine aléatoire ARN (40 ug, 1,5 nmol, 9x10 14 molécules) et du VIH-1 Bal protéine gp120 (0,23 nmol, ARN / protéine 6,5 / 1) dans 200 ul faible sel ARN tampon de liaison sur une plateforme rotative à température ambiante pendant 30 min.
- Passez la réaction à travers un filtre de nitrocellulose pré-mouillé et laver avec 1 ml de tampon de liaison.
- Éluer l'ARN lié au filtre avec 200 uL de tampon d'élution (7 M d'urée et 5 mM d'EDTA) à 95 ° C pendant 5 min, suivie d'une extraction phénol / chloroforme et de concentration avec un Microcon YM-30 de la colonne.
- La transcription inverse de la piscine récupéré l'ARN en utilisant les ThermoScript RT-PCR (Invitrogen) et d'amplifier de 15 cycles de PCR.
- Purifier la piscine ADNdb amplifié en utilisant un kit de purification QIAquick Gel et transcrire tel que décrit ci-dessus pour la prochaine ronde de sélection.
3. SELEX progrès surveillés par dosage de liaison du filtre
- Surveiller les progrès SELEX des aptamères d'essai de liaison du filtre. Traiter le pool d'ARN avec CIP d'enlever l'étiquette initier 5'-triphosphate, puis avec la polynucléotide kinase T4 et γ-32P-ATP.
- Chauffer 10 pmol de CIP traités ARN bibliothèque à 95 ° C pendant 5 min, puis refroidir sur la glace. Par la suite, ajouter 2ul de tampon PNK, 1 pl de T4 polynucléotide kinase, 1 pl de la gamma-P 32-ATP et de l'eau à 20 uL.
- Incuber à 37 ° C pendant 30 min, puis ajouter 20 mu ^; L d'eau et de purifier la réaction en colonne G-50. Enfin, obtenir 40 ul d'ARN marquées à la concentration finale de 250 nM.
- Avant dosage, repliez le étiquetés piscines ARN dans 1xHBS tampon (10 mM HEPES pH 7,4, NaCl 150 mM, 1 mM de CaCl2, MgCl2 1 mM, 2,7 mM de KCl), de la chaleur à 95 ° C pendant 3 min, puis refroidir lentement jusqu'à la 37 ° C. Continuer d'incubation à 37 ° C pendant 10 min.
- Effectuer une uL 100 de réaction de liaison comme par exemple ici. Incuber la fin d'ARN marqué la piscine (10 nM) avec protéine gp120 (100 nM) et un excès de 10 fois molaire de compétiteur non spécifique ARNt (100 nM) dans le tampon d'ARN riche en sel contraignante pour 30 min.
- Séparez une uL 50 de réaction de liaison par un filtre de nitrocellulose pré-mouillé.
- Laver le filtre avec 2 ml de tampon de liaison et de compter la radioactivité retenue sur le filtre à travers un compteur à scintillation multi-usages (Beckman Coulter). Comme un contrôle d'entrée, comptez le solde de 50 uL d'réaction de liaison dans le même temps. Calculer le pour cent de l'ARN retenue sur le filtre dans l'ARN d'entrée comme l'affinité de liaison.
4. Clonage, séquençage et alignements
- Après 11 tours, si aucune enrichissement est observée, même après un tour de sélection supplémentaires, puis liaison maximale de la piscine ARN a potentiellement été atteint.
- La transcription inverse de la piscine hautement enrichi aptamère (12 e ARN piscine) en utilisant les ThermoScript RT-PCR (Invitrogen) et ensuite amplifier l'ADNc obtenu par PCR. Purifier le produit PCR en utilisant un kit de purification QIAquick Gel (QIAGEN). Clone du gel purifié le produit d'ADN dans le vecteur de clonage pCR TA 2.1 (Invitrogen). Au total, inoculer 170 clones individuels et d'autres de les identifier par séquençage d'ADN pour obtenir des séquences individuelles.
- Classer les clones individuels en six groupes différents sur la base des alignements de séquences aptamère individuels. Choisissez une séquence représentant de chaque groupe (A-1, A-5, A-9, A-12, A-28 et B-68) pour une caractérisation plus poussée en raison de leur abondance relative au sein de leur groupe.
5. Génération d'aptamère et d'ARN chimère par transcription in vitro
- Générer directement la matrice d'ADN double brin par PCR en utilisant deux uM chacun de 5'-et 3'-amorces, avec 2 mM de MgCl2 et 200 uM de chaque dNTP, et de récupérer les produits résultant de PCR en utilisant un kit de purification QIAquick Gel.
- Transcrire brin sens chimère de ses modèles d'ADN générés par PCR en utilisant le kit DuraScription (Epicentre, Madison, WI). Dans le mélange réactionnel de transcription, de remplacer le CTP et canoniques UTP avec 2'-F-CTP et 2'-F-UTP pour produire de l'ARN qui est résistant à la dégradation de la RNase A.
- Généralement, incuber 20 ul de réaction contenant 1 ug d'ADN matrice, 2 ml 10xbuffer, 2 uL dATP, dGTP 2 pl, 2 uL 2'-F-dCTP, 2 pl 2'-F-dUTP, DTT 2 uL et 2 ul T7 RNA polymérase à 37 ° C pendant 6 h, et ensuite de le purifier avec Bio-Spin 30 colonnes (Bio-Rad), après extraction au phénol et précipitation à l'éthanol.
- Afin d'éviter la réponse interféron, d'autres traitent l'ARN transcrit par le CIP pour enlever le lancement 5'-triphosphate. Incuber total de 60 ul de réaction contenant 3 mg de transcriptions, 6 pi de tampon de 3 et 0,25 ul de CIP à 37 ° C pendant 60 min. Après phénol / chloroforme et précipitation à l'éthanol exaction, resuspendre culot d'ARN dans l'eau.
- Pour préparer les chimères, combiner les chimères que l'hébergement de l'ARN brin sens avec l'ARN antisens appropriée dans le repliement de tampon, de la chaleur à 95 ° C pendant 3 min, puis refroidir à 37 ° C lentement. Continuer d'incubation à 37 ° C pendant 10 min. Effectuer repliement étape à faîteau 1xHBS un tampon. Par exemple: mélanger 10 uL de 10 uM brin sens chimère, 10 ul de 10 brin antisens uM et 5 uL de tampon de repliement (5xHBS) dans 25 uL du système.
6. Détermination des constantes de dissociation par des essais de retard sur gel
- fin de l'étiquette P 32 l'aptamère représentant de chaque groupe et de brin sens chimères puis repliez l'ARN dans le tampon 1xHBS comme décrit plus haut.
- Préparer 25 ml de gel de 5% en mélangeant 2,5 ml de tampon 10xTBE, avec 3,125 ml de 40% d'acrylamide / bis solution, 19.375 ml d'eau, 150 pi de persulfate d'ammonium à 10% (APS) en solution, et 30 TEMED ul. Le gel doit polymériser dans environ 30 min. Retirer délicatement le peigne et l'utilisation d'une seringue de 30 ml munie d'une aiguille pour laver les puits avec du tampon de fonctionnement (1xTBE).
- Compléter le montage de l'unité de gel et se connecter à une source d'alimentation. Le gel peut être pré-run d'une heure à 180 V à 4 ° C.
- Série diluer le VIH-1BAL protéine gp120 avec le tampon de liaison aux concentrations désirées. Le r finalesLes concentrations eaction de gp120 sont 0, 1, 5, 10, 20, 40, 80, 160, 320, 640 nm. Incuber une quantité constante de 5'-P 32-fin-ARN marqué (10 nm) avec des concentrations accrues de protéine gp120 dans le tampon de liaison (au total 20 ul de réaction) sur une plateforme rotative à température ambiante pendant 30 min.
- Après incubation, mélanger 20 ul de la réaction de liaison avec 5 uL de tampon de charge native et la charger dans un gel de polyacrylamide non dénaturant à 5%. Préparer un tampon de charge native (4x) contenant 10 mM Tris-HCl, pH 7,5, 1 mM EDTA, 0,1% bleu de bromophénol, 0,1% xylène cyanol FF, 0,1% d'Orange G, glycérol 40%. Boutique en aliquots à -20 ° C.
- Après électrophorèse (180 V à 4 ° C pendant 2 heures, jusqu'à ce que le colorant secondaire fonctionne à plein de gel), d'exposer le gel à un écran l'image de phosphore et de quantifier la radioactivité à l'aide d'un scanner Typhoon.
- Calculer les constantes de dissociation à l'aide de régression courbe non linéaire avec un prisme Graph Pad.
7. Cell-surface des études de liaison par cytométrie en flux
- Générer aptamère fluorescentes et chimères en utilisant le kit de marquage Silencieux siRNA (Ambion). Ajouter les réactifs suivants dans l'ordre: 22,5 uL eau sans nucléase; 5 ml 10 x tampon de marquage, 15 uL d'ARN (5 ug); 7,5 Dye Étiquetage ul. Incuber au total 50 uL réaction de marquage à 37 ° C pendant 1 heure.
- Après incubation, ajouter 5,0 ul (0,1 vol) 5 M de NaCl et 125 pi (2,5 vol) EtOH froid 100%, et bien mélanger. Incuber à -20 ° C pendant 60 min. Centrifuger à vitesse maximale à 4 ° C pendant 20 min. Enlever le surnageant et laver culot avec 175 uL EtOH à 70%. Air culot sec dans l'obscurité et de suspendre l'ARN marqués en 15 uL d'eau sans nucléase.
- Mesurer l'absorbance de l'ARN marqués à 260 nm et au maximum d'absorbance pour le colorant fluorescent. Calculer la base: ratio de teinture et de la concentration d'ARN en fonction de la calculatrice fournis par http://www.ambion.com/techlib/append/base_dye.html .
- Mélanger Cy3 marqué brin sens des chimères et brin antisens et repliez dans le repliement de tampon comme décrit ci-dessus.
- Obtenir CHO-WT exprimant la gp160 et les cellules CHO-EE de contrôle à travers la recherche sur le sida et le Programme de réactif de référence 5, 6. Cultiver des cellules dans le GMEM-S moyennes (glutamine-déficientes milieu minimal essentiel avec 400 uM méthionine sulfoximine (MSX)) (Gibco, Invitrogen). Culture des cellules dans un incubateur humidifié à 5% de CO 2 à 37 ° C.
- Laver la gp160 CHO-WT ou des cellules CHO-contrôle EE avec un tampon de lavage préchauffée, trypsiniser et se détacher des plaques. Après le lavage des cellules deux fois avec 500 uL de tampon de liaison, resupend les culots cellulaires dans un tampon de liaison et incuber à 37 ° C pendant 30 min. Cellules Pellet et ensuite les remettre en suspension dans 50 pl de tampon de liaison contenant 400 nM préchauffée Cy3 marqué ARN expérimental.
- Après incubation à 37 ° C pendant 40 min, laver les cellules trois fois avec 500 uL de tampon de liaison préchauffée, et enfin remettre en suspension dans 350 uL de tampon de liaison préchauffé à 37 ° C et analyser immédiatement par cytométrie en flux.
8. L'internalisation et d'études de localisation intracellulaire des cellules vivantes par microscopie confocale
- Avant une journée de test, la croissance de l'gp160 CHO-WT et les cellules CHO-EE de contrôle dans 35 mm avec plaque de l'ensemencement à 0.3x10 6 dans 2 mL GMEM-S moyennes pour permettre à la confluence d'environ 70% en 24 h.
- Le jour de l'expérimentation, se laver les cellules avec 1 ml de PBS préchauffé. Et incuber avec 1 ml de pré-chauffé milieu de croissance complet pendant 30 min à 37 ° C.
- Préparer Cy3 marqué aptamère-siRNA chimères comme décrit ci-dessus. Incuber les replier aptamère-coller avec le siRNA-stick contenant 5'-Cy3 marqué brin sens pour former le conjugué aptamère-stick-siRNA comme décrit ci-dessus.
- Avant dosage, préparation de 0,15 mg / ml solution de Hoechst 33342 (colorant nucléaire pour les cellules vivantes, Molecular Probes, Invitrogen, CA) dans l'eau et de stocker en aliquots à 4 ° C.
- Colorer les cellules par un traitement avec 0,15 mg / mL Hoechst 33342 pendant 15 min à 37 ° C. Immédiatement, rincer la colorant avec 1,0 ml de milieu frais deux fois et remplacer 2 ml préchauffée milieu frais.
- Ajouter le Cy3 marqué aptamère-siRNA chimère à une concentration de 100 nM final dans les médias et les incuber pendant des cellules vivantes microscopie confocale dans un 5% de microscopie incubateur à CO2 à 37 ° C.
- Recueillir les images toutes les 15 min en utilisant un Zeiss LSM 510 Meta Inversé 2 système de photons sous microscope confocal immersion dans l'eau à un grossissement de 40x.
9. In vitro, le VIH-1 Challenge et le dosage d'antigène p24
- Achat cellules CCRF-CEM de l'ATCC. Croître dans RPMI-1640 (Cellgro, Mediatech Inc) complété avec 10% sérum de veau fœtal (SVF, Hyclone), L-glutamine et 1xPen-Strep (Gibco, Invitrogen). Cellules en culture dans un incubateur humidifié à 5% de CO 2 à 37 ° C.
- Obtenir mononucle sang périphériqueéchantillons provenant de donneurs sains ar partir de la ville de Hope National Medical Center (personnel de la clinique).
- Isoler les PBMC du sang total par centrifugation à travers Ficoll-Hypaque solution (Histopaque-1077, Sigma). Appauvrissent les cellules CD8 (cellules T-cytotoxic/suppressor) de PBMC en utilisant Dynabeads CD8 (Invitrogen, CA).
- Cultiver des cellules dans les cellules T milieu actif (BIOE, St. Paul, MN). Cellules en culture dans un incubateur humidifié à 5% de CO 2 à 37 ° C.
- Obtenir le VIH-1 IIIB et NL4-3 virus et virus VIH-1 Bal de la recherche sur le sida et le Programme des réactifs de référence. Après la propagation du virus, stocker en aliquots à -80 ° C.
- Infect cellules CCRF-CEM ou les PBMC humaines par des virus VIH (IIIB, NL4-3 ou de Bal) (0,001 ou 0,005 MOI). Après 24 heures de l'après-infection, lavez doucement les cellules avec du PBS trois fois pour éliminer le virus libre. Continuez à cultiver les cellules infectées dans un incubateur à 5% de CO 2 de microscopie à 37 ° C pendant 4 jours.
- Avant l'ARN traitements, laver délicatement les cellules infectées avec du PBS trois fois pour éliminer le virus libre. Incuber 2x10 4 cellules infectées et 3x10 4 cellules infectées par des ARN replié expérimentale à 400 nm concentration finale dans les plaques 96 puits à 37 ° C (100 ul par puits, le dosage de triplex).
- Recueillir les surnageants de culture (10 pi par puits) à différents moments (3 j, 5 j, 7 j et 9 d) et conserver à -20 ° C jusqu'au dosage de p24.
- Effectuer des analyses antigène p24 en utilisant un antigène p24 du VIH-1 kit ELISA.
10. La fonction de détection siRNA par RT-PCR quantitative de dosage
- Infect cellules CCRF-CEM ou les PBMC humaines avec le VIH virus (IIIB, NL4-3 ou de Bal) et traiter avec les ARN expérimentale (400 nM) tels que décrits ci-dessus.
- Après 7 jours de traitement, les cellules granulés et d'isoler les ARN totaux avec Stat-60. Traiter les ARN totaux à la DNase I pour éliminer l'ADN génomique et produire de l'ADNc en utilisant 2 ug d'ARN total.
- Mélanger les réactifs suivants: 8 ml eau sans nucléase; 1,5 ml de tampon 10 x DNase; 4 ARN uL (2 ug), 0,5 inhibiteur de RNain uL et 1,0 uL RNase DNase I. Incuber totale de réaction 15 uL 37 ° C pendant 1 heures et la chaleur à 80 ° C pendant 10 min pour inactiver la DNase I. Immédiatement, la réaction se détendre sur la glace.
- Ajouter 2 apprêt uL aléatoire (50 ng / uL) et 1 microlitre de dNTP (10 mM) dans le mélange réactionnel ci-dessus, puis chauffer à 65 ° C pendant 5 min. Immédiatement, le refroidissement de la réaction sur la glace.
- Ajouter les réactifs suivants: 5 ml 5xFirst brin tampon; 2,5 ml à 0,1 M de DTT, 0,5 ml RNain inhibiteur et 1,0 mL de MMLV-RT. Incuber total 27 ml de réaction à 25 ° C pendant 10 min et à 37 ° C pendant 1 heure. Après mélange de la chaleur de réaction, à 70 ° C pendant 15 min pour inactiver la transcriptase inverse et puis réfrigérer sur la glace. L'ADNc est prêt pour qRT-PCR.
- Analyser l'expression des gènes cibles par RT-PCR quantitative en utilisant 2 x iQ Sybergreen Mastermix et ensembles d'amorces spécifiques à une concentration finale de 400 nM (test triplex). Utiliser l'expression de GAPDH comme contrôle interne pour la normalisation des données qPCR.
11. Les résultats représentatifs:
1. Nouveau aptamères d'ARN du VIH-1 gp120 BaL sont isolés et caractérisés.
Comme décrit dans la section expérimentale, une banque d'ADN initiale oligonucléotides contenant une région nt 50 aléatoire flanqué de régions primaire fixé sur l'extrémité 5 'et 3' est amplifié et transcrit en ARN d'une piscine. Cette bibliothèque initiale se compose de jusqu'à 10 15 séquences différentes (1 nmol), qui se replient dans un vaste éventail de diverses structures 3-D. La grande complexité et la diversité de la bibliothèque initiale pourrait garantir la présence de structures actives avec une bonne affinité de liaison à la cible.
Employer une procédure SELEX in vitro (figure 1) pour sélectionner 2'-fluoropyrimidine modifiés aptamères d'ARN qui se lient sélectivement la R5 souche VIH-1 en protéines gp120 BaL enveloppe 7. Comme le montre la figure 1, une stratégie de sélection à base de nitrocellulose est réalisée afin d'isoler cible spécifique contraignante ARN à partir des molécules d'ARN non contraignant. Comme la protéine colle à la nitrocellulose, seuls les ARN / protéines complexes ou des agrégats peuvent être retenus sur la membrane et ARN libres sont délavées. Dans des conditions dénaturantes, les ARN liés sont récupérés et sont une transcription inverse en ADNc puis amplifié en ADNdb, et ensuite transcrit in vitro afin de créer un nouveau pool de l'ARN pour le cycle de sélection suivante. La rigueur de sélection est augmenté en réduisant la quantité de protéine cible et augmenter la quantité d'ARNt concurrent. Le montant de pool d'ARN, de protéines et d'ARNt concurrents utilisés dans chaque tour de sélection est présentée au tableau 1.
Surveiller les progrès de la sélection après chaque cycle SELEX par le dosage de filtrage obligatoires. Évaluer l'affinité de liaison que le pour cent de l'ARN retenus sur le filter dans la piscine d'ARN total. La piscine de départ ARN (1-ARN) montre que 0,1% de l'ARN d'entrée retenus sur la membrane. Toutefois, après neuf tours de sélection de la neuvième ARN bibliothèque (9-ARN) a 9,72% de l'ARN d'entrée lié. Bien tours de sélection supplémentaires ont été effectuées, aucun enrichissement est observée, suggérant que la liaison maximale du pool d'ARN a été atteint (figure 2A). Similaires avec le test du filtre obligatoire, le dosage de retard sur gel est également l'une des stratégies les plus populaires pour la détermination de constantes de dissociation. Cette procédure est simple et pratique. Comme le montre la figure 2B, les tests de retard sur gel confirmer davantage les activités de liaison de l'ARN piscines. Ces résultats démontrent que certains ligands avec la spécificité de liaison élevée pour la protéine cible sont successivement enrichi en ces piscines ARN ..
Clone et la séquence des piscines aptamère hautement enrichi (12-ARN). Selon les alignements de séquences individuelles aptamère clonés, six groupes différents sont classés comme le montre le tableau 2. Environ 40% des clones (Groupe I et II aptamères) contiennent une séquence conservée: Un TTGAGGGACC (A / G) (A / G). Nous avons choisi une séquence représentant de chaque groupe (par exemple: A-1, A-5, A-9, A-12, A-28 et B-68) pour une caractérisation plus poussée en raison de leur abondance relative au sein de leur groupe. Grâce à un dosage de retard sur gel natif de mobilité, les constantes de dissociation (Kd) de ces aptamères représentatifs sont calculés (figure 3A). Par exemple, A-1, le meilleur des aptamères, a une valeur Kd apparent de 52 nM (figure 3B). Comme le montre la figure 3A, ces aptamères sélectionnés peuvent se lient sélectivement à la cible du VIH-1 gp120 Bal, mais pas la protéine gp120 du VIH CM.
2. Anti-gp120 aptamère se lie spécifiquement et est internalisé par les cellules exprimant la gp160 du VIH et inhibent l'infection à VIH-1 en culture cellulaire.
CHO-gp160 cellules exprimant de façon stable la gp160 du VIH glycoprotéine d'enveloppe sont utilisés pour tester l'internalisation obligatoire et de l'anti-gp120 sélectionnés aptamères. Ces cellules ne pas traiter en gp160 gp120 et gp41, car ils n'ont pas les protéases gag encodé requis pour l'enveloppe de traitement. Comme un contrôle que nous utilisons la lignée cellulaire CHO-EE parentale qui n'exprime pas la gp160. Analyses par cytométrie en flux (figure 4A) révèlent que le Cy3 marqué aptamères lient spécifiquement aux cellules CHO-gp160, mais pas le contrôle CHO-EE cellules. Par ailleurs, en temps réel des cellules vivantes axe Z microscopie confocale indique que le Cy3 marqué aptamère est sélectivement internalisé dans la gp160-cellules CHO (figure 4B et 4C) après 2 heures d'incubation, mais pas les cellules CHO-EE de contrôle ( Figure 4D). figure 4C montre également que l'aptamère agrégées dans le cytoplasme, ce qui suggère la gp120 aptamères peut pénétrer dans les cellules par l'intermédiaire endocytose.
Dans le test du VIH-1 Challenge, le VIH-1-PBMC cellules sont traitées par les aptamères. A différents jours de post-traitement avec les aptamères, des aliquotes des médias sont dosés pour virale niveaux d'antigène p24 (figure 4E). Les résultats montrent que les aptamères anti-gp120 (A-1) inhibe la production de VIH-1 p24 avec une concentration de nano-molaire.
3. Anti-gp120 aptamère-siRNA chimère est conçu et évalué son efficacité comme un type de cellule système spécifique de livraison de siRNA
Comme le montre la figure 5A, l'aptamère et le segment brin sens de l'ARNsi contenues nucléase résistant 2'-fluoro UTP et 2'-fluoro CTP et sont synthétisés à partir de modèles correspondants ADNdb par transcription in vitro bactériophage. Afin d'augmenter la flexibilité de la molécule, un linker deux nucléotides (UU) est inséré entre l'aptamère et la portion de substrat de Dicer. Pour préparer le siRNA contenant des chimères, transcrit in vitro chimérique polymères brin d'aptamère-sens sont recuits à des concentrations équimolaires d'un ARN non modifiée brin antisens. Ces données de test de retard sur gel (figure 5B) et la cytométrie de flux (figure 5C) indiquent que les chimères maintenir environ les mêmes affinités de liaison que l'aptamères seul. Les images du temps bien sûr du temps réel microscopie confocale (figure 5D) montrent que Cy3 marqué chimère Ch. A-1 peut être avec succès internalisé dans le cytoplasme des cellules. Comme prévu, aucune absorption de la chimère est observée avec les cellules CHO-EE de contrôle (figure 5E).
De même, le potentiel antiviral de l'ARN est évaluée par le VIH-1 test défi. Les résultats des analyses antigène p24 du VIH (figure 6A et 6B) montrent que les deux aptamère et inhiber la production de chimères p24, mais la plus forte inhibition est observé avec la chimère Ch. A-1 du traitement.
Pour confirmer que la composante siARN fonctionne avec l'APTAMer, suite à l'internalisation de l'A-1 chimère dans les cellules infectées Ch., nous avons également d'évaluer les niveaux relatifs de l'inhibition de l'expression du gène TAT / rev par des analyses quantitatives d'expression par RT-PCR. Nous constatons que le traitement de cellules infectées par les chimères est capable d'induire silence des gènes TAT / rev, tandis que l'aptamère seul n'a pas d'incidence sur TAT / l'expression du gène rev (Figure 6C et 6D). Ces résultats fournissent un soutien supplémentaire que les siARN aptamère livré déclenche l'ARNi.
| La quantité de protéines, l'ARN piscine et ARNt utilisés pour la sélection | |||||
| Tours SELEX | Ratio de la cible / ARN | Protéine gp120 | ARN piscine | ARNt Concurrent | Tampon de sélection |
| 1 | 1/6.5 | 229,8 pmol | 1,5 nmol (40,1 mg) | 0 | Faible mémoire tampon SELEX sel |
| 2 | 1/6.5 | 114,9 pmol | 0,75 nmol (20,1 mg) | 0,25 nmol (6,6 mg) | |
| 3 | 1 / 8 | 76,6 pmol | 0.625 nmol (16,7 mg) | 0,25 nmol (6,6 mg) | |
| 4 | 1 / 8 | 76,6 pmol | 0.625 nmol (16,7 mg) | 0,25 nmol (6,6 mg) | |
| 5 | 1 / 8 | 38,3 pmol | 0.306 nmol (8,18 mg) | 0,5 nmol (13,2 mg) | Haute tampon SELEX sel |
| 6 | 1 / 8 | 38,3 pmol | 0.306 nmol (8,18 mg) | 0,5 nmol (13,2 mg) | |
| 7 | 1 / 10 | 26,8 pmol | 0.268 nmol (7,16 mg) | 0,5 nmol (13,2 mg) | |
| 8 | 1 / 10 | 26,8 pmol | pmol 0.268 nmol (7,16 mg) | 1 nmol (26,4 mg) | |
| 9 | 1 / 10 | 15,3 pmol | 0.153 nmol (4,09 mg) | 1 nmol (26,4 mg) | |
| 10 | 1 / 10 | 15,3 pmol | 0.153 nmol (4,09 mg) | 1,5 nmol (39,6 mg) | |
| 11 | 1/12.5 | 7,66 pmol | 0.096 nmol (2,56 mg) | 2 nmol (52,8 mg) | |
| 12 | 1/12.5 | 7,66 pmol | 0.096 nmol (2,56 mg) | 2 nmol (52,8 mg) | |
Tableau 1. Les conditions de sélection. La quantité de protéines, l'ARN piscine et ARNt utilisé pour chaque sélection et tampon de sélection sont indiqués.
| Groupe | ARN | Séquences aléatoires | Fréquence (140 clones) |
| Groupe I | A-1 | AATTGAGGGACCA CGCGCTGCTTGTTGTGATAAGCAG TTTGT CG GATGG | 33 (23,6%) |
| B-7 | AATTGAGGGACCA ACGCGAGGATGTGGATAGTGTGTA TTTGC GT GATGG | 3 | |
| A-32 | AATTGAGGGACCG TTGGTAAAAGCCGGA AATTG AGCT TTTAC GGC GATGG | 5 | |
| B-55 | AATTGAGGTACCGCG TTATTAGGAACA AATTG GAATTCTAAACGC GATGG | 2 | |
| A-24 | AATAGAGGGACC CAGATATAGGCTACACGGATGATGGTGTATCTG GATGG | 1 | |
| B-19 | AATAGAGGAACCG TTTCAGAAGACTACAGGTTAGTCCAATGAAGC GACGG | 1 | |
| B-31 | AATAGAGGGACCG TGGACAATAATTTATGGTCA TTTATTGGCAC GATGG | 1 | |
| Groupe II | A-12 | AGTAGAGGAACCA AGCAATGGATGAATGCAAAAGTGTAAATGCTT GATGG | 10 (7,1%) |
| Groupe III | A-9 | TGAGTTTGGGTAAATTTCCGGTTTCGGTTTACTCACGAAAGATCGGTCGG | 15 (10,7%) |
| Groupe IV | A-28 | TAAAGGAGGGAAGGATGAGACCGCACGAAAAATATCAGCATACG TTTGTG | 10 (7,1%) |
| Groupe V | A-5 | GAAACTAGTTTGAATAATGGTGTAGAGGAGGGTCAATAGTTTCG TTGGTG | 9 (6,4%) |
| Groupe VI | B-68 | ACATAGTAATGACACGGAGGATGGAGAAAAAACAGCCATCTCTTGACGGT | 2 |
| D'autres | Séquence orphelins | 48 |
Tableau 2. L'alignement et l'identification des aptamères d'ARN. Après la 12 e ronde de sélection, la piscine sélectionnés ARN a été cloné et séquencé. Après l'alignement de tous les clones 140, six groupes d'anti-gp120 aptamères ont été identifiés. Seules les séquences aléatoires des principales régions aptamère (5'-3 ') sont indiqués. Les isolats produisent à des fréquences multiples sont spécifiés.
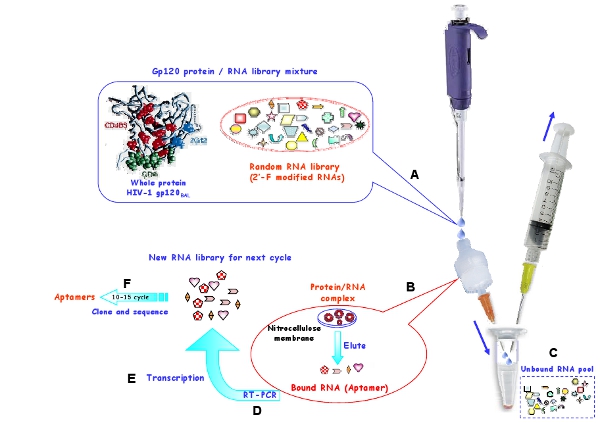
Figure 1: Représentation schématique de la procédure de sélection en vitro en utilisant une membrane de nitrocellulose, pour générer des aptamères d'ARN du VIH-1 Bal protéine gp120. (A) La piscine de départ de l'ARN et des protéines cibles ont été incubées à former des complexes. (B) Les molécules d'ARN liés ont été retenus sur la membrane et élues de la membrane sous condition de dénaturation. (C) L'ARN non liées ont été emportés. (D) Les ARN ont été sélectionnés par transcription inverse et amplifié par PCR. (E) L'ADN a ensuite été pertinentes transcrit en ARN nouvelle piscine pour les cycles de sélection suivante. (F) Après 10-15 tours de sélection, les aptamères sélectionnés ont été clonés et la séquence.
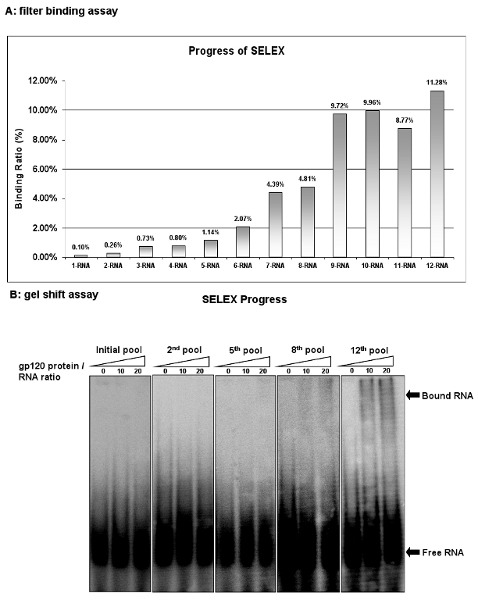
Figure 2: La progression du VIH-1 gp120 sélection des aptamères. (A) L'activité de liaison de la piscine à chaque cycle de l'ARN a été analysée par dosage de liaison filtre avec ARNt concurrent. Activités de liaison ont été calculés comme le pourcentage de l'apport d'ARN retenue sur le filtre en protéines / ARN complexes. (B) L'activité de liaison de la piscine à chaque cycle de l'ARN a été analysée par un test de retard sur gel. La piscine 12 ème ARN a montré la plus grande activité de liaison.
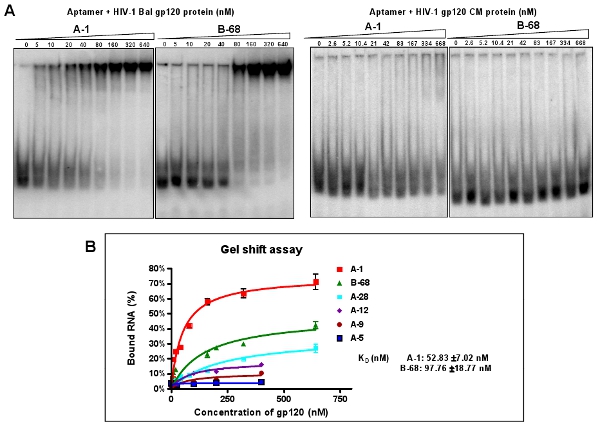
Figure 3: dosage de l'activité de liaison de certains aptamères individuels contre le VIH-1 gp120 Bal. (A) L'extrémité 5 'P 32 étiquetés aptamères individuelles ont été incubées avec des quantités croissantes de protéine gp120 cibles ou de protéines CM non-spécifiques. Les mélanges de réaction de liaison ont été analysés par un test de retard sur gel de la mobilité. Aptamères A-1 et B-68 a montré la meilleure affinité de liaison avec la protéine cible, mais pas de protéines CM. Les données représentent la moyenne de quatre répétitions. (B) courbe de liaison à partir d'un test de retard sur gel.
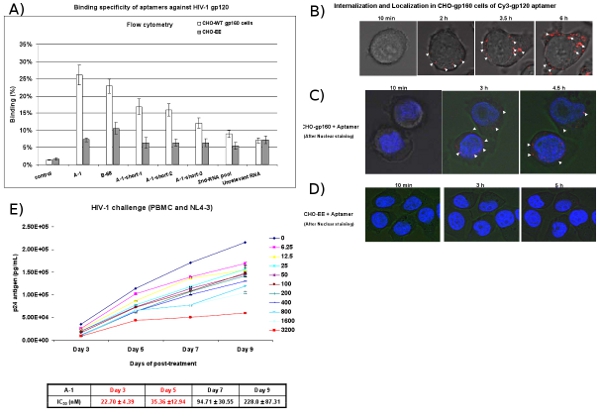
Figure 4: type de cellule liaison spécifique et études sur l'absorption des aptamères. (A) liant la surface cellulaire de Cy3-étiquetés ARN a été évaluée par cytométrie en flux. ARN Cy3 marquées ont été testés pour la liaison à CHO-gp160 et les cellules CHO-EE de contrôle. Les aptamères sélectionnés ont montré un type de cellule affinité de liaison spécifique. Le 2 ème ARN ARN piscine et non pertinentes ont été utilisés comme témoins négatifs. Les données représentent la moyenne de trois répétitions. Internalisation d'analyse (B). CHO-gp160 cellules ont été cultivées dans 35 plaques mm et incubées avec une concentration de 100 nM de Cy3-étiquetés A-1 dans les milieux de culture pour les analyses en temps réel la microscopie des cellules vivantes-confocale. Les images ont été recueillies dans 15 min. intervalles en utilisant un grossissement de 40X. L'analyse de localisation (C, D). CHO-gp160 et les cellules CHO-EE de contrôle ont été cultivées dans 35 plaques mm. Avant l'incubation avec 100 nM de Cy3-étiquetés A-1, les cellules ont été colorés avec Hoechst 33342 (colorant nucléaire pour les cellules vivantes), puis analysées à l'aide en temps réel la microscopie confocale. (E) Les sélectionnés anti-gp120 aptamères inhibent la réplication du VIH-1 dans des PBMC humaines préalablement infectés par le VIH-1 NL4-3 du virus. Différentes concentrations et le temps de pointes ont été présentés. IC50 valeur a été répertoriés. Les données représentent la moyenne des trois mesures de p24.
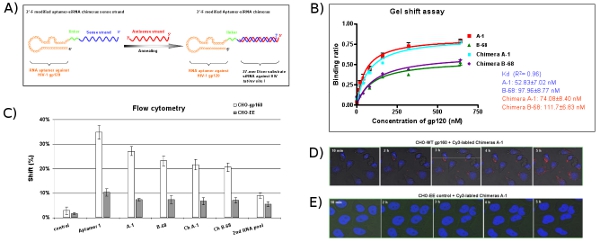
Figure 5: La conception et l'évaluation du système aptamère-siRNA de livraison chimère. (A) Schéma aptamère-siRNA ARN chimérique: la région de l'aptamère anti-gp120 est responsable pour la liaison à la gp120 et le siRNA ciblant un exon est commun de VIH-1 TAT / rev. La 2'-fluoro modifiés brin sens aptamère-siRNA simple a été co-transcrits, suivi par un recuit de le brin antisens de siRNA complémentaires pour compléter la molécule chimérique. Un linker (UU) entre l'aptamère et de siRNA est indiquée en vert. (B) L'ARN aptamère-siRNA chimériques qui ont des valeurs comparables Kd ainsi que les aptamères parentale se lient spécifiquement le VIH Bal protéine gp120. Les données représentent la moyenne de trois répétitions. (C) de type cellulaire particulier des études de liaison des aptamères. ARN Cy3 marquées ont été testés pour la liaison à CHO-gp160 et les cellules CHO-EE de contrôle. Cellulaire surreliures visage de Cy3-étiquetés ARN ont été évalués par cytométrie en flux. Les aptamères sélectionnés ont montré un type de cellule affinité de liaison spécifique. Le 2 ème ARN piscine et ARN non pertinents ont été utilisés comme témoins négatifs. Les données représentent la moyenne des deux répétitions. (D, E) Internalisation et analyses localisation intracellulaire. CHO-gp160 cellules ont été cultivées dans 35 plaques mm et ont été colorés avec Hoechst 33342 (colorant nucléaire pour les cellules vivantes). Par la suite, les cellules ont été incubées dans un milieu de culture avec une concentration de 100 nM de Cy3-étiquetés chimère en temps réel des cellules vivantes analyse par microscopie confocale, comme décrit précédemment.
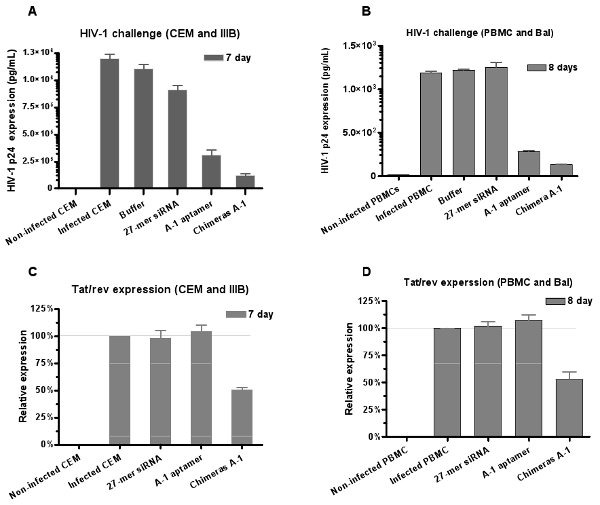
Figure 6: la double inhibition du VIH-1 médiée par aptamère-siRNA chimères. Les deux anti-gp120 aptamère et aptamère-siRNA chimères neutralisé infection VIH-1 dans les cellules CEM (A) (souche IIIB) et (B) culture de PBMC humaines (souche LBA), respectivement. Les données représentent la moyenne des trois mesures de p24. Les chimères ont montré une inhibition plus forte que aptamère seul qui indique que (C, D) le siRNA livrés par les aptamères régulé à la baisse l'expression du gène TAT / rev dans les PBMC. Les données représentent la moyenne de trois répétitions.
Discussion
Les aptamères sont des acides nucléiques in vitro évolué qui supposent spécifique et stable des formes tridimensionnelles, offrant ainsi très spécifique, obligatoire serré pour 8 molécules ciblées. La faible affinité nanomolaire liaison et la spécificité exquise d'aptamères à leurs cibles en font des outils polyvalents pour le diagnostic, l'imagerie in vivo, et de la thérapeutique 9. Avec l'avènement de la technologie pour aptamère l...
Déclarations de divulgation
Remerciements
Nous remercions Britta Hoehn, Guihua Soleil, Harris Soifer et Lisa Scherer pour les discussions utiles. Ce travail a été soutenu par des subventions des National Institutes of Health et de AI29329 HL07470 attribué à JJR Les réactifs suivants ont été obtenus par la recherche des NIH sida et Référence Reagent Program, Division du sida, du NIAID, NIH: Le CHO-EE et CHO-gp160 cellulaire ligne; l'pNL4-3 Luc vecteur; VIH-1 gp120 du DAIDS BaL, le NIAID.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | Commentaires (optionnel) |
|---|---|---|---|
| MF-Millipore filtre à membrane | Millipore | HAWP01300 | La taille des pores 0,45 pm |
| Swinnex Porte-filtre | Millipore | SX0001300 | 13 mm de diamètre |
| QIAquick Gel Extraction Kit | QIAGEN | 28706 | Purification d'ADN |
| Microcon YM-30 colonne | Millipore | 42410 | Concentration d'ARN |
| Bio-spin 30 colonnes | Bio-Rad | 732-6250 | Purification d'ARN |
| Taq polymérase ADN PCR | Sigma-Aldrich | D1806 | |
| ThermoScript RT-PCR | Invitrogen | 11146-024 | |
| DuraScribe Kit de transcription T7 | EpiCentre | DS010925 | |
| dNTP pour la PCR | Roche | 1 581 295 | |
| L'acide ribonucléique, le transfert de E.coli | Sigma-Aldrich | R1753 | compétiteur ARNt |
| VIH-1 Ba-L protéine gp120 | l'AIDS Research and Référence Reagent Program | 4961 | Protéine cible |
| Kit de marquage Silencieux siARN - Cy3 | Ambion | 1632 | |
| Acide phénol / chloroforme 5 / 1 solution (pH 4,5) | Ambion | AM9720 | |
| Chloroforme / isopropanol 24 / 1 solution | Sigma | C0549 | |
| Phosphatase intestinale de veau (CIP) | La Nouvelle-Angleterre BioLab | M0290L | |
| T4 polynucléotide kinase | La Nouvelle-Angleterre BioLab | M0201L | |
| Le glycogène | Roche | 10 901 393 001 | ARN précipitations |
| Gamma-P 32-ATP | MP Biomédical | 013502002 | Radiactivity |
| 40% AccuGel 19:01 | Diagnostic national | CE-850 | |
| 10xTBE | Diagnostic national | CE-860 | |
| N, N, N, N'-tétraméthyléthylènediamine (TMEMD) | Sigma-Aldrich | T9281 | |
| Le persulfate d'ammonium (APS) | Sigma-Aldrich | A3678 | |
| L-methioine sulfoximine | Sigma-Aldrich | M5379-250 mg | |
| RPMI 1640 avec les médias | Invitrogen | 11835-030 | |
| Une solution de bicarbonate de sodium, 7,5% p / v | Invitrogen | 25080-094 | |
| Minimum Essential Medium (MEM) (10 ) | Invitrogen | 11430-030 | |
| MEM non acides aminés essentiels (100 ) | Invitrogen | 11140-050 | |
| TA cloning kit avec pCR 2.1 | Invitrogen | K2040-01 |
Références
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Robertson, D. L., Joyce, G. F. Selection in vitro of an RNA enzyme that specifically cleaves single-stranded DNA. Nature. 344, 467-468 (1990).
- Fitzwater, T., Polisky, B. A SELEX primer. Methods Enzymol. 267, 275-301 (1996).
- Weiss, C. D., White, J. M. Characterization of stable Chinese hamster ovary cells expressing wild-type, secreted, and glycosylphosphatidylinositol-anchored human immunodeficiency virus type 1 envelope glycoprotein. J Virol. 67, 7060-706 (1993).
- Vodicka, M. A. Indicator cell lines for detection of primary strains of human and simian immunodeficiency viruses. Virology. 233, 193-198> (1997).
- Zhou, J. Selection, characterization and application of new RNA HIV gp 120 aptamers for facile delivery of Dicer substrate siRNAs into HIV infected cells. Nucleic Acids Res. , (2009).
- Mayer, G. The chemical biology of aptamers. Angew Chem Int Ed Engl. 48, 2672-2689 (2009).
- Famulok, M., Hartig, J. S., Mayer, G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy. Chem Rev. 107, 3715-3743 (2007).
- Chu, T. C., Twu, K. Y., Ellington, A. D., Levy, M. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34, e73-e73 (2006).
- McNamara, J. O., 2nd, . Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat Biotechnol. 24, 1005-1015 (2006).
- Dassie, J. P. Systemic administration of optimized aptamer-siRNA chimeras promotes regression of PSMA-expressing tumors. Nat Biotechnol. 27, 839-849 (2009).
- Zhou, J., Rossi, J. J. The therapeutic potential of cell-internalizing aptamers. Curr Top Med Chem. 9, 1144-1157 (2009).
- Zhou, J., Li, H., Li, S., Zaia, J., Rossi, J. J. Novel dual inhibitory function aptamer-siRNA delivery system for HIV-1 therapy. Mol Ther. 16, 1481-1489 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon