A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
פיתוח נייד מסוג ספציפי נגד HIV gp120 aptamers למסירה siRNA
In This Article
Summary
מספר 2'-Fluoro aptamers נגד HIV-RNA 1Ba-L gp120 עם זיקה nanomole מבודדים מספריית RNA על ידי במבחנה הליך Selex. חדש כפול פונקציה מעכבות אנטי gp120 aptamer-siRNA הכימרה נוצר מראה הבטחה רבה לטיפול אנטי HIV מערכתית.
Abstract
מגפה עולמית של זיהום על ידי HIV יצרה צורך דחוף כיתות חדשות של סוכנים תרופתי. היכולת חזק של קטן (SI) RNAs התערבות כדי לבלום את הביטוי של תעתיקי רנ"א משלימים המנוצלת כמו סוג חדש של הרפוי עבור מגוון רחב של מחלות כולל איידס. דיווחים קודמים רבים הראו כי הרומן RNAi מבוסס anti-HIV/AIDS אסטרטגיות טיפוליות יש הבטחה רבה, עם זאת, מכשול מרכזי ליישום טיפולית מוצלחת תרגום הקליניים של siRNAs היא מסירה יעילה. במיוחד, בהתחשב הבטיחות והיעילות של RNAi מבוסס הרפוי, רצוי מאוד לפתח משלוח ממוקד תאיים siRNA הגישה אוכלוסיות תאים או רקמות ספציפיות. HIV-1 gp120 חלבון, מעטפה גליקופרוטאין על פני השטח של HIV-1, ממלא תפקיד חשוב בכניסה ויראלי לתאי CD4. האינטראקציה של gp120 ו CD4 שמפעיל HIV-1 כניסה יוזם היתוך תא יאומת כאסטרטגיה אנטי ויראלית רלוונטיות קלינית לגילוי סמים.
בזאת, אנחנו קודם כל לדון הבחירה וזיהוי של 2'-F שונה אנטי HIV aptamers gp120-RNA. בשיטה הקונבנציונלית nitrocellulose לסנן Selex, aptamers חדש מספר עם זיקה nanomolar בודדו מתוך 50 מקרי NT-RNA הספרייה. על מנת להשיג בהצלחה המינים קשורים עם זיקה גבוהה יותר, חומרא הבחירה נשלטת בקפידה על ידי התאמת התנאים. Aptamers שנבחרו במיוחד יכולים להיקשר ולהיות הפנימו במהירות לתוך תאים המבטאים את חלבון ה-HIV-1 המעטפה. בנוסף, aptamers לבדו יכול לנטרל HIV-1 infectivity. בהתבסס על aptamer מיטב A-1, יש לנו גם ליצור רומן כפול פונקציה מעכבות אנטי gp120 aptamer-siRNA הכימרה שבו הן aptamer ואת מנות siRNA יש עוצמה אנטי HIV פעילויות. יתר על כן, אנו מנצלים את gp120 aptamer-siRNA מפלצות למסירה התא סוג מסוים של siRNA לתוך HIV-1 תאים נגועים. זה תפקיד כפול הכימרה מראה פוטנציאל רב לשילוב שונים סוכני חומצות גרעין טיפולית (aptamer ו siRNA) ב-HIV-1 דיכוי, מה שהופך את aptamer-siRNA מפלצות מועמדים אטרקטיביים טיפולית לחולים באי טיפול תרופתי פעיל מאוד (HAART).
Protocol
1. הכנת הספרייה RNA
- ספריית DNA החל הכיל 50 נוקלאוטידים של רצפים אקראיים היה מסונתז על ידי DNA משולב טכנולוגיות (Coralville, איווה). ה-DNA חד גדילי אוליגו רצף הספריה 5'-GGG AGG ACG ATG CGG - N 50 - החטיבה ACG ACT CGC CCG - 3 '(81 NT). האזור אקראי מוקף אזורים קבועים, הכוללים את האמרגן T7 שכן שעתוק במבחנה תג '3 עבור RT-PCR. 5 'ו 3' רצפים קבועים של 5 - TAA TAC GAC TCA CTA TAG GGA GGA CGA TGC ג"ג - 3 '(32 mer) ו 5 "-TCG GGC GAG TCG TCT G - 3' (16 mer), בהתאמה. בצע פתרון מניות במים חנות aliquots ב -20 ° C.
- להגביר את אחד גדילי הדנ"א אוליגו אקראי בספרייה (0.4 מיקרומטר) על ידי PCR באמצעות 3 מיקרומטר אחד 5'-ו-3'primers, יחד עם 2 מ"מ MgCl 2 ו 200 מיקרומטר dNTP של כל אחד. כדי לשמר את השפע של ספריית ה-DNA המקורי, להגביל PCR עד עשר מחזורים. לאחר PCR תגובות (10 תגובות, 100 μL לכל התגובה), לשחזר את הבריכה dsDNA מוגבר באמצעות טיהור ג'ל QIAquick Kit (QIAGEN).
- המרת dsDNA כתוצאה לספרייה RNA באמצעות ערכת DuraScription (Epicentre, מדיסון, ויסקונסין) לפי הוראות היצרן. בתערובת התגובה שעתוק, להחליף CTP ו UTP עם 2'-F-CTP ו 2'-F-UTP לייצר ribonuclease RNA עמיד. בדרך כלל, להכין 20 μL התגובה המכיל 1 מיקרוגרם של מטוהרים תבנית ה-DNA, 2 חיץ μL 10 x, 2 dATP μL, 2 dGTP μL, 2 μL 2'-F-dCTP, 2 μL 2'-F-dUTP, 2 DTT μL ו 2 T7-RNA פולימראז μL בטמפרטורת החדר ואז לדגור על 37 מעלות צלזיוס במשך 6 ח (50 מ"מ dNTP כל אחד.)
- בהמשך לעכל את התגובה עם DNase אני (1.5 μL לכל תגובה T7-20 μL שעתוק) כדי להסיר את תבנית ה-DNA ולטהר ידי polyacrylamide 8% / 7 ג'ל M אוריאה. לכמת את RNA מטוהרים ספריית ידי לספקטרופוטומטריה UV.
2. דור במבחנה של aptamers
- לפני הבחירה, להכין את החיץ הבחירה חיץ refolding. הכן חיץ HEPES המכיל 100 Hepes מ"מ, pH 7.4. השתמש NaOH להתאים את ערך ה-pH ולאחר מכן לאחסן בטמפרטורת החדר. הכן חיץ refolding RNA (5xHBS) המכיל 50 Hepes mM pH 7.4, 750 mM NaCl, 5 מ"מ MgCl 2: 5 מ"מ CaCl 2; 13.5 mM KCl. הכן חיץ דלת מלח RNA מחייב (10 mM HEPES pH 7.4, 50 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2.7 mM KCl, 10 mM DTT, 0.01% ו BSA חיץ גבוה מלח RNA מחייב (10 HEPES mM pH 7.4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2.7 mM KCl, 10 mM DTT, BSA 0.01%). Store מאגרים אלה ב -20 ° C.
- בצע את Selex בעיקר כמתואר 1-4. לפני כל סיבוב הבחירה, ולקפל את בריכות רנ"א 1xHBS חיץ (10 mM HEPES pH 7.4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2.7 mM KCl), חום עד 95 מעלות צלזיוס במשך 3 דקות ולאחר מכן לצנן לאט עד 37 ° C. המשך דגירה על 37 מעלות צלזיוס למשך 10 דקות.
- בדרך כלל, על מנת למזער מחייב ספציפי עם מסנני nitrocellulose, מראש לספוג וקיפלתי בריכות RNA ל nitrocellulose מסנן (פילטר HAWP, 0.45 מיקרומטר) למשך 30 דקות, לפני הדגירה עם יעד HIV-1 באל gp120 חלבון.
- דגירה הבריכה מראש פינה RNA עם חלבון המטרה במאגר RNA נמוך מחייב מלח למשך 30 דקות של סיבובים Selex 1-4. אחרי הסיבוב הרביעי של Selex, להשתמש חיץ גבוה מלח RNA מחייב. עם התקדמות Selex, להפחית את כמות החלבון gp120 ולהגדיל את tRNA שמרים מתחרה על מנת להגביר את ההחמרה של מבחר aptamer.
- עבור המחזור הראשון של הבחירה, דגירה מראש פינה אקראי בריכה RNA (40 מיקרוגרם, 1.5 nmol, 9x10 14 מולקולות) ו-HIV-1 באל gp120 חלבון (0.23 nmol, RNA / חלבון יחס 6.5 / 1) 200 μL נמוכה מלח RNA חיץ מחייב על פלטפורמה מסתובבת בטמפרטורת החדר למשך 30 דקות.
- תעביר את התגובה דרך מסנן מראש הרטובות nitrocellulose ולשטוף עם חיץ מ"ל 1 מחייב.
- Elute רנ"א מחויב מן המסנן עם חיץ 200 elution μL (7 מ אוריאה ו - 5 mM EDTA) בשעה 95 ° C במשך 5 דקות, ואחריו מיצוי פנול / כלורופורם וריכוז עם טור YM-30 Microcon.
- הפוך לתמלל את הבריכה התאושש RNA באמצעות RT-PCR ThermoScript המערכת (Invitrogen) ולהגביר במשך 15 מחזורים של PCR.
- לטהר את הבריכה dsDNA מוגבר באמצעות ערכת טיהור QIAquick ג'ל ו לתמלל כמתואר לעיל לסיבוב הבא של הבחירה.
3. התקדמות Selex פיקוח על ידי assay מחייב מסנן
- מעקב אחר ההתקדמות של Selex aptamers על ידי assay מחייב הסינון. פנקו את הבריכה RNA עם CIP להסיר את התווית ייזום 5'-טריפוספט ולאחר מכן עם קינאז polynucleotide T4 ו γ-32P-ATP.
- חום של 10 pmol CIP שטופלו RNA הספרייה 95 מעלות צלזיוס למשך 5 דקות ולאחר מכן לצנן על הקרח. לאחר מכן, להוסיף 2μL של PNK חיץ, 1 μL של קינאז polynucleotide T4, 1 μL של gamma-32-P-ATP ומים μL 20.
- לדגור על 37 מעלות צלזיוס למשך 30 דקות, ולאחר מכן להוסיף 20 mu ^; L מים לטהר את התגובה לפי עמודה G-50. לבסוף, להשיג 40 μL של רנ"א מסומן בריכוז הסופי של 250 ננומטר.
- לפני assay, ולקפל שכותרתו RNA בריכות 1xHBS חיץ (10 mM HEPES pH 7.4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2.7 mM KCl), חום עד 95 מעלות צלזיוס במשך 3 דקות ולאחר מכן לצנן לאט 37 ° C. המשך דגירה על 37 מעלות צלזיוס למשך 10 דקות.
- לבצע μL 100 התגובה מחייב כמו למשל כאן. דגירה הקצה שכותרתו RNA בריכה (10 ננומטר) עם חלבון gp120 (100 ננומטר) עודף של פי 10 טוחנת של המתחרה tRNA ספציפי (100 ננומטר) למאגר RNA גבוהה מלח מחייב למשך 30 דקות.
- הפרד μL 50 התגובה מחייב ידי לסנן מראש רטוב nitrocellulose.
- שטפו את המסנן עם חיץ 2 מחייב מ"ל ולספור את הרדיואקטיביות שמרו על המסנן דרך דלפק רב תכליתי scintillation (Beckman Coulter). כמו שליטה קלט, לספור את הנותרים 50 μL התגובה מחייב באותו זמן. חישוב אחוזים של RNA שמרו על המסנן ב RNA קלט כמו הזיקה מחייב.
4. , שיבוט רצף מערכים
- לאחר 11 סיבובים, אם לא העשרה נוסף הוא ציין גם לאחר סבבי בחירה נוספת ואז מחייב המקסימלי של הבריכה RNA יש פוטנציאל הושג.
- הפוך לתמלל את הבריכה מועשר aptamer (12 בריכת ה-RNA) באמצעות RT-PCR ThermoScript המערכת (Invitrogen) ולאחר מכן להגביר את cDNA וכתוצאה מכך על ידי PCR. לטהר את מוצר ה-PCR באמצעות טיהור ג'ל QIAquick Kit (QIAGEN). Clone ג'ל טיהר את מוצר ה-DNA לתוך ת"א וקטור שיבוט PCR 2.1 (Invitrogen). בסך הכל, לחסן 170 שיבוטים הפרט נוספת לזהות אותם על ידי רצפי DNA כדי לקבל רצפים נפרדים.
- לסווג את שיבוטים הפרט לשש קבוצות שונות בהתבסס על מערכים של רצפי aptamer בודדים. בחרו רצף אחד נציג של כל קבוצה (A-1, A-5, A-9, A-12, A-28 ו-B-68) לאפיון נוסף בגלל השפע היחסי שלהם בתוך הקבוצה שלהם.
5. הדור של aptamer ו RNAs הכימרה על ידי ב שעתוק במבחנה
- ישירות ליצור פעמיים תקועים תבנית ה-DNA על ידי ה-PCR באמצעות 2 מיקרומטר אחד 5'-ו-3'primers, יחד עם 2 מ"מ MgCl 2 ו 200 מיקרומטר של dNTP כל, ולשחזר את המוצרים כתוצאה PCR באמצעות ערכת טיהור QIAquick ג'ל.
- לתמלל גדיל תחושה תעתוע מתבניות שלה PCR DNA שנוצר באמצעות ערכת DuraScription (Epicentre, מדיסון, ויסקונסין). בתערובת התגובה שעתוק, להחליף את CTP ו UTP הקנונית עם 2'-F-CTP ו 2'-F-UTP לייצר RNA כי הוא עמיד השפלה RNase.
- בדרך כלל, דגירה 20 μL התגובה המכיל 1 מיקרוגרם של תבנית ה-DNA, 2 מ"ל 10xbuffer, 2 dATP μL, 2 dGTP μL, 2 μL 2'-F-dCTP, 2 μL 2'-F-dUTP, 2 DTT μL ו 2 μL T7-RNA פולימראז על 37 מעלות צלזיוס במשך 6 שעות, ולאחר מכן לטהר אותו עם Bio-Spin 30 עמודות (Bio-Rad) לאחר מיצוי פנול משקעים אתנול.
- כדי למנוע תגובה אינטרפרון, נוסף לטיפול RNA עיבד ידי CIP להסיר את ייזום 5'-טריפוספט. דגירה μL הכולל 60 של התגובה המכיל 3 מיקרוגרם של תמלילי, 6 μL של הצפת 3 ו 0.25 μL של CIP על 37 מעלות צלזיוס למשך 60 דקות. לאחר פנול / כלורופורם מעשקות ומשקעים אתנול, resuspend RNA גלולה לתוך המים.
- כדי להכין את מפלצות, לשלב את מפלצות מחסה רק גדיל RNA חוש עם RNA antisense המתאים refolding, חום חיץ עד 95 מעלות צלזיוס במשך 3 דקות ולאחר מכן מגניב 37 ° C לאט. המשך דגירה על 37 מעלות צלזיוס למשך 10 דקות. בצע refolding צעד אחר צעד חיץ עיטורי 1xHBS. לדוגמה: לערבב 10 μL של 10 מיקרומטר גדיל תחושה תעתוע, 10 μL של גדיל 10 מיקרומטר antisense ו 5 חיץ refolding μL (5xHBS) למערכת 25 μL.
6. קביעת קבועי דיסוציאציה על ידי מבחני משמרת ג'ל
- סוף P 32 התווית aptamer נציג מהקבוצה כל גדיל מפלצות תחושה ואז לקפל RNA במאגר 1xHBS כמתואר לעיל.
- הכן 25 מ"ל של ג'ל 5% על ידי ערבוב של 2.5 מ"ל של חיץ 10xTBE, עם 3.125 מ"ל של תמיסת acrylamide / bis 40%, 19.375 מ"ל מים, 150 μL של persulfate אמוניום 10% (APS) פתרון, ו 30 TEMED μL. הג'ל צריך לפלמר ב 30 דקות בערך. הסר בזהירות את המסרק ולהשתמש מזרק 30 מ"ל מצויד במחט לשטוף את הבארות עם חיץ ריצה (1xTBE).
- השלם את הרכבה של יחידת ג'ל ולהתחבר החשמל. הג'ל ניתן מראש לרוץ במשך שעה אחת על V 180 ב 4 ° C.
- סדרתי לדלל את HIV-gp120 חלבון 1Bal עם חיץ מחייב הריכוזים הרצוי. R הסופיריכוזים eaction של gp120 הם 0, 1, 5, 10, 20, 40, 80, 160, 320, 640 ננומטר. דגירה כמות קבועה של 5'-P-32 בסוף שכותרתו RNA (10 ננומטר) עם ריכוז מוגבר של החלבון gp120 במאגר מחייב (סה"כ 20 μL התגובה) על במה מסתובבת בטמפרטורת החדר למשך 30 דקות.
- לאחר דגירה, לערבב 20 μL התגובה מחייב עם חיץ טעינת μL 5 יליד לטעון לתוך ג'ל שאינו denaturing 5% polyacrylamide. הכן חיץ טעינת יליד (4x) המכיל 10 mM טריס-HCl, pH 7.5, 1 mM EDTA, כחול Bromophenol 0.1%, 0.1% קסילן Cyanol FF, 0.1% אורנג' G, גליצרול 40%. אחסן aliquots ב -20 ° C.
- לאחר אלקטרופורזה (180 V על 4 מעלות צלזיוס למשך 2 שעות, עד לצבוע משני רץ באמצע של ג'ל), לחשוף את הג'ל על המסך תמונה זרחן ולכמת את הרדיואקטיביות באמצעות סורק טייפון.
- חשב את קבועי דיסוציאציה שימוש שאינו ליניארי עקומת רגרסיה עם פריזמה Pad גרף.
7. על פני קרום התא מחקרים מחייב על ידי זרימת cytometry
- צור aptamer פלורסנט מפלצות באמצעות siRNA משתיק תיוג Kit (Ambion). מוסיפים את ריאגנטים הבאים לפי סדר: 22.5 מים μL nuclease ללא; 5 מ"ל 10 x הצפת תיוג: 15-RNA μL (5 מיקרוגרם); 7.5 דיי תיוג μL. דגירה התגובה הכולל 50 תיוג μL על 37 מעלות צלזיוס במשך שעה 1.
- לאחר דגירה, להוסיף 5.0 μL (0.1 כרך) 5 M NaCl ו 125 μL (2.5 כרך) EtOH 100% קרים, ומערבבים היטב. דגירה ב -20 מעלות צלזיוס למשך 60 דקות. צנטריפוגה במהירות שיא על 4 מעלות צלזיוס למשך 20 דקות. הסר supernatant ולשטוף עם גלולה EtOH 175 70% μL. האוויר יבש גלולה בחושך ואז להשעות RNA מסומן μL 15 מים nuclease חינם.
- מדוד את ספיגת של רנ"א מסומן ב 260 ננומטר ב המרבי עבור ספיגת צבע פלואורסצנטי. חישוב הבסיס: יחס לצבוע וריכוז RNA על פי המחשבון מסופק על ידי http://www.ambion.com/techlib/append/base_dye.html .
- מערבבים Cy3 שכותרתו מפלצות גדיל לחוש antisense גדיל ולקפל ב refolding חיץ כמתואר לעיל.
- השג CHO-WT להביע gp160 ו CHO-EE תאים לשלוט באמצעות מחקר האיידס מגיב הפניה תוכנית 5, 6. לגדל תאים בינוניים GMEM-S (גלוטמין מחסר מדיום חיוני מינימלי עם 400 מיקרומטר sulfoximine מתיונין (MSX)) (Gibco, Invitrogen). התרבות התאים humidified 5% CO 2 באינקובטור ב 37 ° C.
- שטפו את CHO-WT gp160 או CHO-EE תאים מלאה עם חיץ כביסה prewarmed, trypsinize ולנתק מן הצלחות. לאחר שטיפת התאים פעמיים עם חיץ 500 מחייב μL, resupend את כדורי תא חיץ דגירה מחייב ב 37 מעלות צלזיוס למשך 30 דקות. תאים גלולה ואז resuspend אותם μL 50 של חיץ מחייב prewarmed המכיל 400 nm Cy3 שכותרתו RNAs ניסיוני.
- לאחר הדגרה ב 37 מעלות צלזיוס למשך 40 דקות, לשטוף תאים שלוש פעמים עם 500 μL של חיץ מחייב prewarmed ו resuspend לבסוף μL 350 של חיץ מחייב prewarmed ל 37 ° C ומיד לנתח ידי cytometry הזרימה.
8. הפנמה ומחקרים לוקליזציה תאיים על ידי תאים חיים במיקרוסקופ confocal
- לפני יום אחד assay, לגדל את CHO-WT gp160 ו CHO-EE תאים שליטה צלחת 35 מ"מ עם זריעת ב 0.3x10 6 בינוני 2 GMEM-S מ"ל לאפשר מפגש כ - 70% ב 24 ח
- ביום של הניסויים, לשטוף תאים עם 1 מ"ל של prewarmed PBS. וגם דגירה עם 1 מ"ל של מדיום מראש חימם צמיחה המלא ל 30 דקות ב 37 ° C.
- הכן Cy3 שכותרתו aptamer-siRNA מפלצות כמתואר לעיל. דגירה ולקפל aptamer מקל עם siRNA מקל המכיל 5'-Cy3 שכותרתו גדיל תחושה לטופס aptamer מקל-siRNA מצומדות כפי שתואר לעיל.
- לפני assay, להכין 0.15 מ"ג / מ"ל פתרון של Hoechst 33,342 (צבע גרעיני תאים חיים, בדיקות מולקולריות, Invitrogen, CA) במים חנות aliquots ב 4 ° C.
- הכתם התאים על ידי טיפול Hoechst 0.15 מ"ג / מ"ל 33,342 עבור 15 דקות בשעה 37 ° C. מיד לשטוף את צבען בינוני עם 1.0 מ"ל פעמיים טריים ולהחליף 2 מ"ל prewarmed בינוני טרי.
- מוסיפים את Cy3 שכותרתו aptamer-siRNA הכימרה בריכוז סופי 100 ננומטר לתוך התקשורת דגירה עבור מיקרוסקופיה לחיות תאים confocal במיקרוסקופ של 5% החממה CO2 ב 37 ° C.
- איסוף התמונות כל 15 דקות באמצעות LSM Zeiss 510 Meta הפוך 2 מערכת פוטון תחת מיקרוסקופ confocal טבילה במים בהגדלה של 40X.
9. לאתגר ב HIV-1 במבחנה assay אנטיגן p24
- רכישה CCRF-CEM תאים ATCC. לגדול RPMI-1640 (Cellgro, Mediatech Inc) בתוספת 10% בסרום שור העובר (FBS, HyClone), L-גלוטמין ו-1xPen סטרפטוקוקוס (Gibco, Invitrogen). התרבות התאים humidified 5% CO 2 באינקובטור ב 37 ° C.
- השג mononucle דם היקפייםדגימות ar מתורמים בריאים מהעיר התקווה הלאומית המרכז הרפואי (אנשי המרפאה).
- בודד PBMCs מן הדם על ידי צנטריפוגה כל דרך הפתרון Ficoll-Hypaque (Histopaque-1077, סיגמא). לכלות CD8 תאים (תאים T-cytotoxic/suppressor) מ PBMCs באמצעות Dynabeads CD8 (Invitrogen, CA).
- לגדל תאים בינוניים T-התא הפעיל (BioE, סנט פול, מינסוטה). התרבות התאים humidified 5% CO 2 באינקובטור ב 37 ° C.
- השג HIV-1 ו IIIB NL4-3 הנגיף HIV-1 וירוס באל מן המחקר איידס מגיב תוכנית הפניה. לאחר התפשטות של וירוס חנות, ב aliquots ב -80 ° C.
- להדביק CCRF-CEM תאים או PBMCs אדם עם וירוסים HIV (IIIB, NL4-3 או באל) (משרד הפנים 0.001 או 0.005). לאחר 24 שעות של זיהום פוסט, לשטוף בעדינות תאים עם PBS שלוש פעמים להסיר וירוס בחינם. המשך התרבות תאים נגועים ב 5% CO 2 מיקרוסקופיה החממה ב 37 מעלות צלזיוס למשך 4 ימים.
- לפני RNA טיפולים, לשטוף בעדינות את התאים הנגועים PBS שלוש פעמים להסיר וירוס בחינם. דגירה 2x10 4 תאים נגועים נגוע 3x10 4 תאים עם וקיפלתי RNAs ניסיוני 400 nm הריכוז הסופי 96-היטב צלחות ב 37 ° C (100 μL היטב לכל assay טריפלקס,).
- אסוף את supernatants תרבות (10 μL לכל היטב) במועדים שונים (3 ד, ד 5, 7 ו - 9 ד ד) ולאחסן ב -20 ° C עד assay p24.
- ביצוע ניתוחים אנטיגן p24 באמצעות Antigen HIV-1 p24 ערכת ELISA.
10. פונקציית זיהוי siRNA ידי assay RT-PCR כמותי
- להדביק CCRF-CEM תאים או PBMCs אדם עם HIV וירוסים (IIIB, NL4-3 או באל) ולטפל עם RNAs ניסיוני (400 ננומטר) כפי שתואר לעיל.
- אחרי 7 ימי טיפול, תאים גלולה ולבודד RNAs מוחלטת עם STAT-60. התייחס RNAs מוחלטת עם DNase אני להסיר את הדנ"א הגנומי לייצר cDNA באמצעות 2 מיקרוגרם של רנ"א מוחלט.
- מערבבים את ריאגנטים הבאים: 8 מ"ל nuclease ללא מים; 1.5 מ"ל 10 x DNase חוצץ; 4 RNA μL (2 מיקרוגרם); 0.5 מעכב RNain μL ו 1.0 μL RNase ללא DNase I. דגירה התגובה הכולל 15 μL 37 ° C עבור 1 שעה חום 80 מעלות צלזיוס למשך 10 דקות כדי להשבית DNase י מיד לצנן את התגובה על הקרח.
- הוסף 2 פריימר אקראי μL (50 ng / μL) ו 1μL dNTP (10 mM) לתוך התערובת את התגובה לעיל, ולאחר מכן חום 65 מעלות צלזיוס למשך 5 דקות. מיד לצנן את התגובה על הקרח.
- מוסיפים את ריאגנטים הבאים: 5 מ"ל 5xFirst גדיל חוצץ; 2.5 מ"ל 0.1 M DTT: 0.5 מ"ל RNain מעכב ו 1.0 מ"ל MMLV-RT. דגירה התגובה הכולל 27 מ"ל ב 25 ° C במשך 10 דקות ו - ב 37 מעלות צלזיוס במשך שעה 1. לאחר התערובת, חום התגובה על 70 מעלות צלזיוס למשך 15 דקות כדי להשבית transcriptase הפוכה ואז לצנן על הקרח. CDNA הוא מוכן לניתוח qRT-PCR.
- ניתוח ביטוי גנים יעד כמותי על ידי RT-PCR באמצעות 2 x IQ SyberGreen Mastermix וקובע פריימר ספציפי בריכוז הסופי של 400 ננומטר (assay טריפלקס). השתמש בביטוי GAPDH כמו שליטה פנימי לנורמליזציה של הנתונים qPCR.
11. נציג התוצאות:
1. חדש aptamers RNA נגד HIV-1 gp120 באל מבודדות מאופיין.
כפי שתואר בסעיף הניסוי, ה-DNA הראשונית oligonucleotide ספרייה המכילה אזור 50 nt אקראי מוקף אזורים תחל קבוע על 5 'ו 3' מסתיים מוגבר עיבד לבריכה RNA. זו ספריה הראשוני המורכב של עד 10 15 רצפים שונים (1 nmol), אשר לקפל לתוך מערך עצום של 3-D מבנים שונים. המורכבות והגיוון גבוהה של הספרייה הראשונית עשויה להבטיח את קיומם של מבנים פעילים עם זיקה מחייבת טוב אל המטרה.
להעסיק בהליך Selex חוץ גופית (איור 1) כדי לבחור 2'-fluoropyrimidine שונה aptamers RNA אשר נקלטות באופן סלקטיבי את המתח R5 HIV-1 באל gp120 חלבון המעטפת 7. כפי שניתן לראות בתרשים 1, אסטרטגיה nitrocellulose מבוססי הבחירה מתבצעת לבודד ספציפית יעד מחייב RNAs מ בלתי מחייב מולקולות RNA. מאז החלבון נדבק nitrocellulose, רק RNA / חלבון מתחמי או אגרגטים ניתן להיעזר על הממברנה ו RNAs חינם נשטפים החוצה. בתנאים denaturing, RNAs כבול הם התאושש מתועתקים הפוכה cDNA ואז מוגבר לתוך dsDNA, ולאחר מכן במבחנה עיבד כדי ליצור מאגר חדש רנ"א עבור מחזור הבחירה הבאה. חומרא הבחירה הוא גדל על ידי הפחתת כמות של חלבון המטרה להגדיל את כמות tRNA מתחרה. הסכום של הבריכה רנ"א, חלבונים tRNA מתחרה להשתמש בכל סיבוב הבחירה מוצג בטבלה 1.
מעקב אחר ההתקדמות של הבחירה לאחר כל מחזור Selex ידי assay מחייב הסינון. הערכת זיקה מחייבת את אחוז RNA שמרו על filter בבריכה RNA מוחלט. הבריכה החל RNA (1-RNA) מציג רק 0.1% מכלל RNAs קלט שמר על הממברנה. עם זאת, לאחר תשעה סיבובים הבחירה RNA ninth ספריה (9-RNA) יש 9.72% של תשומה RNA כבול. למרות סיבובים בחירה נוספים שנערכו, לא העשרה נוסף הוא ציין, דבר המצביע על כך מחייב מקסימלי של הבריכה RNA הושג (איור 2 א). דומים עם assay מחייב לסנן את assay משמרת ג'ל גם הוא אחת האסטרטגיות הפופולריות ביותר לקביעת קבועי דיסוציאציה. פרוצדורה זו היא קלה ונוחה. כפי שמוצג באיור 2B, מבחני ג'ל משמרת נוספת לאשר את הפעילויות מחייב של בריכות RNA. תוצאות אלו מראות כי חלק ligands עם סגוליות גבוהה מחייבת עבור חלבון המטרה מועשרים ברציפות אלה בריכות RNA ..
Clone ורצף בריכות aptamer מועשר (12-RNA). לפי מערכים של הפרט רצפים aptamer משובטים, שש קבוצות שונות מסווגים כפי שמוצג בטבלה 2. כ -40% של שיבוטים (קבוצה I ו-II aptamers) מכילים רצף משומר: (A / G) TTGAGGGACC (A / G). אנחנו בוחרים רצף אחד נציג של כל קבוצה (למשל: A-1, A-5, A-9, A-12, A-28 ו-B-68) לאפיון נוסף בגלל השפע היחסי שלהם בתוך הקבוצה שלהם. באמצעות assay ג'ל יליד ניידות במשמרת, קבועי דיסוציאציה (K ד) אלה aptamers נציג מחושבים (איור 3A). לדוגמה, A-1, את הטוב ביותר של aptamers, יש ערכים Kd לכאורה של 52 ננומטר (איור 3B). כפי שמוצג באיור 3 א, אלה aptamers נבחרים באופן סלקטיבי יכול להיקשר עם יעד HIV-1 באל gp120, אך לא את החלבון gp120 CM-HIV.
2. Anti-gp120 aptamer במיוחד קושר מופנם על ידי תאים להביע HIV gp160 ומעכבות HIV-1 זיהום בתרבית תאים.
CHO-gp160 בתאי ביציבות המבטא את gp160 גליקופרוטאין HIV המעטפה משמשים לבדיקת מחייב הפנמה של אנטי gp120 aptamers שנבחר. תאים אלה אינם תהליך gp160 אל gp120 ו - gp41 שכן הם חסרים את פרוטאזות איסור פרסום מקודד נדרש עיבוד המעטפה. כביקורת אנו משתמשים בקו ההורים CHO-EE תא שאינו להביע gp160. ניתוח תזרים cytometric (איור 4 א) עולה כי Cy3 שכותרתו aptamers במיוחד לאגד את CHO-gp160 לתאים אך לא את השליטה CHO-EE תאים. יתר על כן, בזמן אמת לחיות תאים ציר Z-מיקרוסקופיה confocal עולה כי aptamer-Cy3 שכותרתו היא הפנימה באופן סלקטיבי לאחר 2 שעות של דגירה בתוך-gp160 בתאי CHO (איור 4B ו - 4C), אך לא CHO-EE תאים שליטה ( איור 4D). איור 4C גם מראה כי aptamer מצטברים בתוך הציטופלסמה, המציעה את gp120 aptamers אולי להיכנס לתאים באמצעות אנדוציטוזה קולטן בתיווך.
ב assay HIV-1 אתגר, HIV-1-נגועים PBMC תאים מטופלים עם aptamers. באותו יום אחר טיפול פוסט עם aptamers, aliquots של התקשורת הם assayed עבור רמות האנטיגן הנגיפי p24 (איור 4E). התוצאות מראות כי אנטי gp120 aptamers (A-1) מעכב ייצור HIV-1 p24 עם ריכוז ננו שן טוחנת.
3. Anti-gp120 aptamer-siRNA הכימרה מתוכנן להעריך את יעילותו כמו תאים מסוג מסוים של מערכת המסירה siRNA
כפי שמוצג באיור 5A, ו aptamer גדיל תחושה קטע של siRNAs הכלול nuclease עמידים 2'-Fluoro UTP ו 2'-Fluoro CTP ו מסונתזים מתבניות dsDNA המקביל ידי בתעתיק bacteriophage חוץ גופית. על מנת להגדיל את הגמישות של המולקולה, והמקשר two נוקלאוטיד (UU) מוכנס בין aptamer ואת חלק המצע דייסר. כדי להכין את siRNA המכיל מפלצות, במבחנה עיבד aptamer תחושת פולימרים chimeric גדיל הם annealed עם ריכוזים equimolar של גדיל RNA antisense ללא שינוי. אלה נתונים מ assay ג'ל משמרת (איור 5 ב) ו cytometry הזרימה (איור 5 ג) עולה כי מפלצות לשמור בערך זיקות מחייבות זהה aptamers לבד. הפעם, כמובן תמונות מיקרוסקופיה בזמן אמת confocal (איור 5D) מראים Cy3 שכותרתו הכימרה Ch-1 יכול להיות מופנמת בהצלחה לתוך הציטופלסמה של תאים. כצפוי, אין ספיגת של כימרה הוא ציין עם התאים CHO-EE הביקורת (איור 5E).
כמו כן, הפוטנציאל של נגיפים RNAs מוערך על ידי assay-HIV-1 אתגר. התוצאות של ניתוח HIV אנטיגן p24 (איור 6 א ו - 6B) מראים כי הן aptamer ותעתועים מעכבים ייצור p24, אך עיכוב החזק הוא ציין עם Ch הכימרה A-1 לטיפול.
כדי לאשר את הרכיב siRNA פועל יחד עם aptamer, בעקבות הפנמה של Ch-1 הכימרה בתאים נגועים, יש לנו גם להעריך את רמות יחסית של עיכוב ביטוי מידה / rev הגן על ידי מבחני כמותי RT-PCR הביטוי. אנו מוצאים כי הטיפול של תאים נגועים מפלצות הוא מסוגל לגרום השתקה של הגן מידה / rev, בעוד aptamer לבד לא משפיעים על ביטוי מידה / rev גן (איור 6C ו - 6 ד). תוצאות אלה מספקות תמיכה נוספת כי siRNA aptamer נמסר מעורר RNAi.
| כמות חלבון, המשמש RNA בריכה tRNA לבחירה | |||||
| Selex סיבובים | יחס של יעד / RNA | Gp120 חלבון | RNA בריכה | מתחרה tRNA | בחירת הצפת |
| 1 | 1/6.5 | 229.8 pmol | 1.5 nmol (40.1 מיקרוגרם) | 0 | מלח נמוכה Selex חיץ |
| 2 | 1/6.5 | 114.9 pmol | 0.75 nmol (20.1 מיקרוגרם) | 0.25 nmol (6.6 מיקרוגרם) | |
| 3 | 1 / 8 | 76.6 pmol | 0.625 nmol (16.7 מיקרוגרם) | 0.25 nmol (6.6 מיקרוגרם) | |
| 4 | 1 / 8 | 76.6 pmol | 0.625 nmol (16.7 מיקרוגרם) | 0.25 nmol (6.6 מיקרוגרם) | |
| 5 | 1 / 8 | 38.3 pmol | 0.306 nmol (8.18 מיקרוגרם) | 0.5 nmol (13.2 מיקרוגרם) | מלח גבוהה Selex חיץ |
| 6 | 1 / 8 | 38.3 pmol | 0.306 nmol (8.18 מיקרוגרם) | 0.5 nmol (13.2 מיקרוגרם) | |
| 7 | 1 / 10 | 26.8 pmol | 0.268 nmol (7.16 מיקרוגרם) | 0.5 nmol (13.2 מיקרוגרם) | |
| 8 | 1 / 10 | 26.8 pmol | pmol 0.268 nmol (7.16 מיקרוגרם) | 1 nmol (26.4 מיקרוגרם) | |
| 9 | 1 / 10 | 15.3 pmol | 0.153 nmol (4.09 מיקרוגרם) | 1 nmol (26.4 מיקרוגרם) | |
| 10 | 1 / 10 | 15.3 pmol | 0.153 nmol (4.09 מיקרוגרם) | 1.5 nmol (39.6 מיקרוגרם) | |
| 11 | 1/12.5 | 7.66 pmol | 0.096 nmol (2.56 מיקרוגרם) | 2 nmol (52.8 מיקרוגרם) | |
| 12 | 1/12.5 | 7.66 pmol | 0.096 nmol (2.56 מיקרוגרם) | 2 nmol (52.8 מיקרוגרם) | |
טבלה 1. התנאים הבחירה. כמות חלבון, RNA בריכה tRNA המשמשים מבחר כל חיץ מבחר מצוינים.
| קבוצה | RNA | רצפים אקראיים | תדירות (140 שיבוטים) |
| קבוצת אני | A-1 | AATTGAGGGACCA CGCGCTGCTTGTTGTGATAAGCAG TTTGT CG GATGG | 33 (23.6%) |
| B-7 | AATTGAGGGACCA ACGCGAGGATGTGGATAGTGTGTA TTTGC GT GATGG | 3 | |
| A-32 | AATTGAGGGACCG TTGGTAAAAGCCGGA AATTG AGCT TTTAC GGC GATGG | 5 | |
| B-55 | AATTGAGGTACCGCG TTATTAGGAACA AATTG GAATTCTAAACGC GATGG | 2 | |
| A-24 | AATAGAGGGACC CAGATATAGGCTACACGGATGATGGTGTATCTG GATGG | 1 | |
| B-19 | AATAGAGGAACCG TTTCAGAAGACTACAGGTTAGTCCAATGAAGC GACGG | 1 | |
| B-31 | AATAGAGGGACCG TGGACAATAATTTATGGTCA TTTATTGGCAC GATGG | 1 | |
| הקבוצה השנייה | A-12 | AGTAGAGGAACCA AGCAATGGATGAATGCAAAAGTGTAAATGCTT GATGG | 10 (7.1%) |
| קבוצה III | A-9 | TGAGTTTGGGTAAATTTCCGGTTTCGGTTTACTCACGAAAGATCGGTCGG | 15 (10.7%) |
| קבוצה IV | A-28 | TAAAGGAGGGAAGGATGAGACCGCACGAAAAATATCAGCATACG TTTGTG | 10 (7.1%) |
| קבוצה V | A-5 | GAAACTAGTTTGAATAATGGTGTAGAGGAGGGTCAATAGTTTCG TTGGTG | 9 (6.4%) |
| קבוצת VI | B-68 | ACATAGTAATGACACGGAGGATGGAGAAAAAACAGCCATCTCTTGACGGT | 2 |
| אחרים | יתום רצף | 48 |
טבלה 2. יישור וזיהוי של aptamers RNA. בעקבות הסיבוב ה -12 של מבחר, הבריכה שנבחר RNA שוכפלה ולאחר רצף. לאחר היישור של כל 140 שיבוטים, שש קבוצות של אנטי gp120 aptamers זוהו. רק רצפים אקראיים של האזורים הליבה aptamer (5'-3 ") מצוינים. מבודד המתרחשים עם תדירויות מרובות שצוינו.
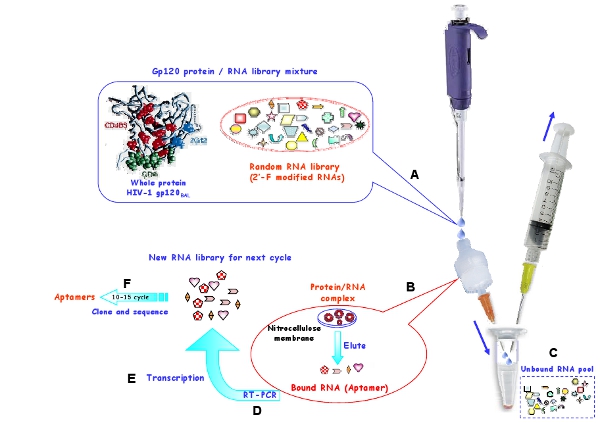
איור 1: ייצוג סכמטי של הליך הבחירה במבחנה באמצעות קרום nitrocellulose, להפקת aptamers הרנ"א לחלבון HIV-1 gp120 באל. (א) הבריכה החל RNA וחלבון היעד הודגרו כדי ליצור מורכבות. (ב) על מולקולות RNA מחויב נשמרו על הממברנה ואת eluted מן הממברנה בתנאי denaturing. (ג) RNAs מאוגד נשטפו. (ד) RNAs שנבחר תועתקו הפוך מוגבר על ידי PCR. (ה) הדנ"א הרלוונטי היה עיבד לאחר מכן לתוך הבריכה RNA חדש מחזורים הבחירה הבאה. (ו) אחרי 10-15 סיבובי הבחירה, aptamers הנבחרת היו משובטים ורצף.
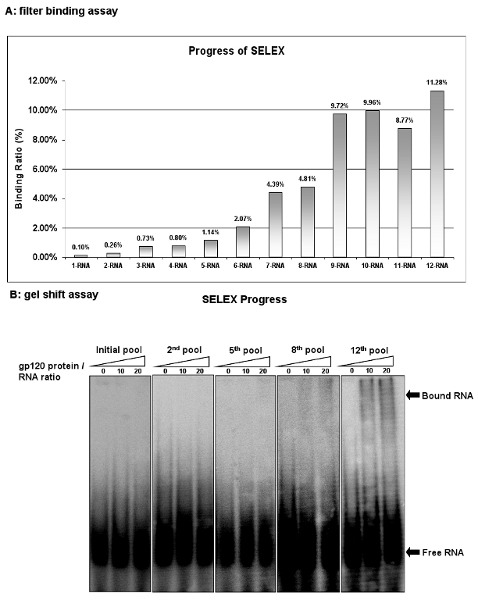
איור 2: התקדמות של בחירה HIV-1 aptamers gp120. (א) פעילות הכריכה של הבריכה RNA על כל מחזור נותח על ידי assay מחייב לסנן tRNA עם מתחרה. פעילות איגוד חושבו כאחוז קלט RNA שמרו על המסנן בחלבון / RNA מורכב. (ב) פעילות הכריכה של הבריכה RNA על כל מחזור נותח על ידי assay משמרת ג'ל. בריכת 12 ה RNA הראה את פעילות מחייב הגבוהה ביותר.
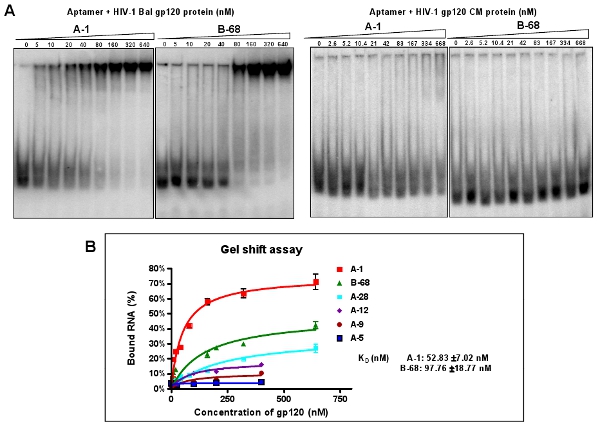
איור 3: assay פעילות עקידת aptamers בודדים נבחרים נגד HIV-1 באל gp120. (א) 5'-end P 32 aptamers הפרט שכותרתו הודגרו עם כמויות גדלות והולכות של חלבון המטרה gp120 או שאינם ספציפיים לחלבון CM. תערובות התגובה מחייב נותחו על ידי assay ג'ל ניידות במשמרת. Aptamer A-1 ו-B-68 הראה זיקה מחייבת הטובה ביותר עם חלבון המטרה, אבל החלבון לא CM. הנתונים מייצגים את הממוצע של ארבעה משכפל. (ב) עקומת עקידת מ assay שינוי ג'ל.
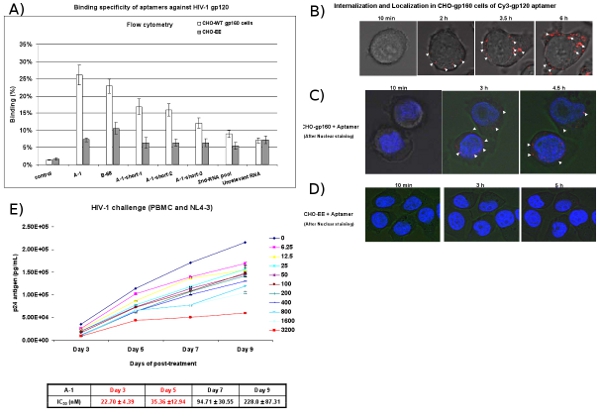
איור 4: נייד מסוג ספציפי מחייב מחקרים קליטת aptamers. (א) פני השטח של תא מחייב Cy3 שכותרתו RNAs הוערכה על ידי cytometry הזרימה. RNAs Cy3 שכותרתו נבדקו עבור מחייב CHO-gp160 לתאים CHO-EE תאים שליטה. Aptamers נבחר הראו תא מסוג זיקה מחייבת ספציפיים. 2 nd RNA RNAs בריכה רלוונטי שימשו שולטת שלילית. הנתונים מייצגים את הממוצע של שלוש משכפל. (ב) ניתוח הפנמה. CHO-gp160 התאים גדלו ב 35 צלחות מ"מ מודגרות עם ריכוז של 100 ננומטר של Cy3 שכותרתו A-1 בתקשורת תרבות לניתוח לחיות התא confocal בזמן אמת במיקרוסקופ. תמונות נאספו ב 15 דקות. מרווחי באמצעות הגדלה 40X. (C, D) לוקליזציה ניתוח. CHO-gp160 לתאים CHO-EE תאים בקרת גדלו ב 35 צלחות מ"מ. לפני הדגירה עם 100 ננומטר של Cy3 שכותרתו A-1, התאים היו מוכתמות Hoechst 33,342 (צבע גרעיני תאים חיים) ולאחר מכן נותחו באמצעות בזמן אמת מיקרוסקופיה confocal. (ה) הנבחר אנטי gp120 aptamers לעכב HIV-1 שכפול PBMCs אדם נדבקו עד כה בנגיף HIV-1-3 NL4. ריכוזים שונים וזמן pointes הוצגו. IC50 ערך היה רשום. נתונים מייצגת את הממוצע של מדידות בשלושה עותקים של p24.
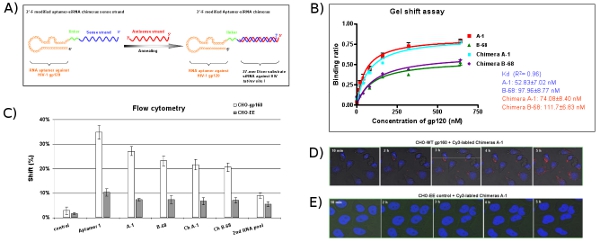
איור 5: תכנון והערכה של המערכת aptamer-siRNA המסירה הכימרה. (א) סכמטי aptamer-siRNA RNAs chimeric: האזור של aptamer אנטי gp120 אחראי מחייב gp120 ו - siRNA הוא מיקוד אקסון משותפת של HIV-1 מידה / rev. 2'-Fluoro שונה aptamer-siRNA גדיל יחיד במובן היה שותף עיבד, ואחריו חישול של גדיל משלים siRNA antisense כדי להשלים את המולקולה chimeric. מקשר (UU) בין aptamer ו siRNA מסומן בירוק. (ב) aptamer-siRNA RNAs chimeric בעלי ערכים דומים Kd וכן aptamers ההורים במיוחד לאגד את החלבון gp120 באל HIV. הנתונים מייצגים את הממוצע של שלוש משכפל. (ג) תאים מסוג מסוים של מחקרים מחייב aptamers. RNAs Cy3 שכותרתו נבדקו עבור מחייב CHO-gp160 לתאים CHO-EE תאים שליטה. תא סוראיגודי פני Cy3 שכותרתו RNAs הוערכו על ידי cytometry הזרימה. Aptamers נבחר הראו תא מסוג זיקה מחייבת ספציפיים. 2 nd RNA בריכה RNA רלוונטי שימשו שולטת שלילית. הנתונים מייצגים את הממוצע של שני משכפל. (D, E) הפנמה ומנתח לוקליזציה תאיים. CHO-gp160 התאים גדלו ב 35 צלחות מ"מ הוכתמו Hoechst 33,342 (צבע גרעיני תאים חיים). כתוצאה מכך, התאים הודגרו במדיום תרבות עם ריכוז של 100 ננומטר Cy3 שכותרתו הכימרה לניתוח לחיות תאים בזמן אמת מיקרוסקופיה confocal כפי שתואר לעיל.
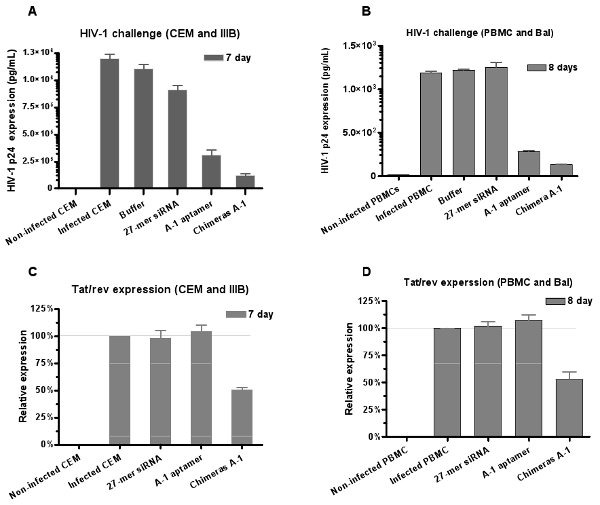
איור 6: עיכוב כפול של הידבקות ב-HIV-1 בתיווך aptamer-siRNA מפלצות. שניהם אנטי gp120 aptamer aptamer ו-siRNA מפלצות לנטרל זיהום HIV-1 ב (א) בתאים CEM (זן IIIB) ו (ב) תרבות PBMCs האנושי (זן BAL), בהתאמה. הנתונים מייצגים את הממוצע של מדידות בשלושה עותקים של p24. מפלצות הראו עיכוב חזק יותר aptamer לבד המציין (C, D) siRNA נמסר על ידי aptamers למטה מוסדר מידה / rev ביטוי גנים PBMCs. הנתונים מייצגים את הממוצע של שלוש משכפל.
Discussion
Aptamers הן במבחנה התפתח חומצות גרעין להניח כי ספציפיים יציב צורות תלת ממדיות, ובכך לספק מאוד ספציפיות, וכפותים מולקולות ממוקדות 8. הזיקה נמוך מחייב nanomolar וספציפיות מעודן של aptamers אל המטרות שלהם להפוך אותם כלים תכליתי עבור אבחון, in vivo הדמיה ותרופות 9. ע...
Disclosures
Acknowledgements
אנו מודים בריטה Hoehn, Guihua Sun, האריס Soifer וליסה שרר לדיונים מועילים. עבודה זו נתמכה על ידי מענקים מהמוסד הלאומי לבריאות AI29329 ו HL07470 הוענק JJR ריאגנטים הבאים התקבלו באמצעות האיידס NIH מחקר מגיב תוכנית הפניה, אגף איידס, NIAID, NIH: CHO CHO-EE ו-gp160 תא קו; pNL4-3 לוק וקטור; HIV-1 באל gp120 מ DAIDS, NIAID.
Materials
| Name | Company | Catalog Number | Comments |
| שם מגיב | חברה | מספר קטלוגי | הערות (אופציונלי) |
|---|---|---|---|
| MF-Millipore קרום מסנן | Millipore | HAWP01300 | גודל הנקבוביות 0.45 מיקרומטר |
| Swinnex בעל מסנן | Millipore | SX0001300 | קוטר 13 מ"מ |
| QIAquick ג'ל הפקת Kit | QIAGEN | 28706 | דנ"א לטיהור |
| Microcon YM-30 טור | Millipore | 42410 | RNA ריכוז |
| ביו ספין 30 טור | Bio-Rad | 732-6250 | RNA טיהור |
| תקי PCR DNA פולימראז | סיגמא אולדריץ | D1806 | |
| ThermoScript RT-PCR מערכת | Invitrogen | 11146-024 | |
| DuraScribe T7 ערכת שעתוק | EpiCentre | DS010925 | |
| dNTP עבור ה-PCR | רוש | 1 581 295 | |
| חומצה ריבונוקלאית, העברת E.coli | סיגמא אולדריץ | R1753 | tRNA מתחרה |
| HIV-1 Ba-L gp120 חלבון | המחקר איידס מגיב תוכנית הפניה | 4961 | יעד חלבון |
| SiRNA משתיק ערכת תיוג - Cy3 | Ambion | 1632 | |
| חומצה פנול / כלורופורם 01/05 פתרון (pH 4.5) | Ambion | AM9720 | |
| כלורופורם / Isopropanol 24 / 1 פתרון | סיגמא | C0549 | |
| עגל phosphatase מעיים (CIP) | ניו אינגלנד BioLab | M0290L | |
| קינאז polynucleotide T4 | ניו אינגלנד BioLab | M0201L | |
| גליקוגן | רוש | 10 901 393 001 | RNA משקעים |
| Gamma-32-P-ATP | MP ביו | 013502002 | Radiactivity |
| 40% AccuGel 19:01 | אבחון הלאומי | EC-850 | |
| 10xTBE | אבחון הלאומי | EC-860 | |
| N, N, N, N'-Tetramethylethylenediamine (TMEMD) | סיגמא אולדריץ | T9281 | |
| חנקתי persulfate (APS) | סיגמא אולדריץ | A3678 | |
| L-methioine sulfoximine | סיגמא אולדריץ | M5379-250 מ"ג | |
| RPMI מדיה 1640 | Invitrogen | 11835-030 | |
| פתרון נתרן ביקרבונט, 7.5% w / v | Invitrogen | 25080-094 | |
| בינוני Essential Minimum (ממ) (10 ) | Invitrogen | 11430-030 | |
| ממ שאינם חיוניים חומצת אמינו (100 ) | Invitrogen | 11140-050 | |
| ת"א ערכה עם שיבוט PCR 2.1 | Invitrogen | K2040-01 |
References
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Robertson, D. L., Joyce, G. F. Selection in vitro of an RNA enzyme that specifically cleaves single-stranded DNA. Nature. 344, 467-468 (1990).
- Fitzwater, T., Polisky, B. A SELEX primer. Methods Enzymol. 267, 275-301 (1996).
- Weiss, C. D., White, J. M. Characterization of stable Chinese hamster ovary cells expressing wild-type, secreted, and glycosylphosphatidylinositol-anchored human immunodeficiency virus type 1 envelope glycoprotein. J Virol. 67, 7060-706 (1993).
- Vodicka, M. A. Indicator cell lines for detection of primary strains of human and simian immunodeficiency viruses. Virology. 233, 193-198> (1997).
- Zhou, J. Selection, characterization and application of new RNA HIV gp 120 aptamers for facile delivery of Dicer substrate siRNAs into HIV infected cells. Nucleic Acids Res. , (2009).
- Mayer, G. The chemical biology of aptamers. Angew Chem Int Ed Engl. 48, 2672-2689 (2009).
- Famulok, M., Hartig, J. S., Mayer, G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy. Chem Rev. 107, 3715-3743 (2007).
- Chu, T. C., Twu, K. Y., Ellington, A. D., Levy, M. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34, e73-e73 (2006).
- McNamara, J. O., 2nd, . Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat Biotechnol. 24, 1005-1015 (2006).
- Dassie, J. P. Systemic administration of optimized aptamer-siRNA chimeras promotes regression of PSMA-expressing tumors. Nat Biotechnol. 27, 839-849 (2009).
- Zhou, J., Rossi, J. J. The therapeutic potential of cell-internalizing aptamers. Curr Top Med Chem. 9, 1144-1157 (2009).
- Zhou, J., Li, H., Li, S., Zaia, J., Rossi, J. J. Novel dual inhibitory function aptamer-siRNA delivery system for HIV-1 therapy. Mol Ther. 16, 1481-1489 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved