È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sviluppo di Cell-tipo specifico anti-HIV gp120 aptameri per la consegna di siRNA
In questo articolo
Riepilogo
Diversi 2'-Fluoro aptameri contro l'HIV-RNA 1BA-L gp120 con nanomole affinità sono isolati da una libreria di RNA In vitro Procedura SELEX. Un nuovo doppio inibitoria funzione anti-gp120 aptamer-siRNA chimera si crea e mostra la promessa considerevole sistemico terapia anti-HIV.
Abstract
L'epidemia globale di infezione da HIV ha creato un urgente bisogno di nuove classi di farmaci antiretrovirali. La capacità potente di small interfering (si) RNA di inibire l'espressione di trascrizioni di RNA complementare viene sfruttato come una nuova classe di terapie per una varietà di malattie tra cui l'HIV. Molti rapporti precedenti hanno dimostrato che nuove strategie a base di RNAi anti-HIV/AIDS terapeutiche sono molto promettente, ma uno dei principali ostacoli per l'applicazione di successo terapeutico e la traduzione clinica di siRNA è la consegna efficiente. In particolare, considerando la sicurezza e l'efficacia di terapie a base di RNAi, è altamente auspicabile per sviluppare un approccio mirato intracellulare consegna di siRNA a popolazioni di cellule o tessuti specifici. L'HIV-1 proteina gp120, una busta glicoproteina sulla superficie del virus HIV-1, svolge un ruolo importante in ingresso del virus nelle cellule CD4. L'interazione di gp120 e CD4 che innesca l'HIV-1 ingresso e avvia la fusione cellulare è stato convalidato come clinicamente rilevante anti-virale strategia di scoperta di nuovi farmaci.
Qui, abbiamo innanzitutto discutere la selezione e l'individuazione di 2'-F modificato anti-HIV gp120 aptameri RNA. Utilizzando un metodo convenzionale SELEX filtro di nitrocellulosa, diversi aptameri nuovo con affinità nanomolare sono stati isolati da un 50 casuale nt RNA biblioteca. Al fine di ottenere con successo le specie legate con maggiore affinità, il rigore selezione viene accuratamente controllato regolando le condizioni. Il aptameri selezionato può legano specificamente ed essere rapidamente internalizzato nelle cellule che esprimono la proteina di HIV-1 busta. Inoltre, il aptameri sola può neutralizzare l'infettività di HIV-1. Sulla base della miglior aptamer A-1, abbiamo anche creato un romanzo a doppia funzione inibitoria anti-gp120 aptamer-siRNA chimera in cui sia il aptamer e le porzioni siRNA hanno potente attività anti-HIV. Inoltre, utilizziamo la gp120 aptamer-siRNA chimere per cellulari tipo di consegna specifiche del siRNA HIV-1 in cellule infette. Questa doppia funzione chimera indica un notevole potenziale per la combinazione di diversi agenti terapeutici degli acidi nucleici (aptamero e siRNA), nella soppressione da HIV-1, rendendo il aptamer-siRNA chimere attraente candidati terapeutici per i pazienti non aver terapia antiretrovirale altamente attiva (HAART).
Protocollo
1. Preparazione della libreria di RNA
- La libreria di DNA di partenza conteneva 50 nucleotidi di sequenze casuali ed è stato sintetizzato da Integrated Technologies DNA (Iowa City, Iowa). Il singolo filamento di DNA sequenza biblioteca oligo è 5'-GGG AGG ATG ACG CGG - N 50 - CAG ACG ATTO CGC CCG A - 3 '(81 nt). La regione a caso è fiancheggiato da regioni costanti, tra cui il promotore T7 per la trascrizione in vitro e tag a 3 'per RT-PCR. 5 'e 3' delle sequenze costanti sono 5 '- TAA TAC GAC TCA CTA TAG GGA GGA CGA TGC GG - 3' (32 mer) e 5 'TCG GGC-GAG TCG TCT G - 3' (16 mer), rispettivamente. Fai la soluzione madre con acqua e conservare in aliquote a -20 ° C.
- Amplificare il singolo filamento di DNA oligo casuale biblioteca (0,4 mM) mediante PCR utilizzando 3 mM ciascuno di 5'-e 3'-primer, insieme con 2 mM MgCl 2 e 200 mM di ogni dNTP. Al fine di preservare la ricchezza della biblioteca del DNA originale, limite PCR a dieci cicli. Dopo le reazioni di PCR (10 reazioni, 100 l per reazione), il recupero della piscina dsDNA amplificato utilizzando un kit di Gel QIAquick purificazione (QIAGEN).
- Convertire il dsDNA conseguente a una libreria di RNA utilizzando il kit DuraScription (Epicentre, Madison, WI) secondo le istruzioni del produttore. Nella miscela di reazione di trascrizione, sostituire CTP e UTP con 2'-F-CTP e 2'-F-UTP per la produzione di RNA ribonucleasi resistenti. Tipicamente, preparare 20 l di reazione contenente 1 mg di purificato modello di DNA, 2 microlitri di buffer x 10, 2 dATP microlitri, 2 dGTP microlitri, 2 microlitri 2'-F-dCTP, 2 microlitri 2'-F-dUTP, 2 DTT microlitri e 2 microlitri T7 RNA polimerasi a temperatura ambiente e poi incubare a 37 ° C per 6 ore (50 mM di ogni dNTP).
- Successivamente digerire la reazione con DNasi I (1,5 microlitri per 20 microlitri reazione trascrizione T7) per rimuovere il DNA stampo e purificare da un poliacrilamide 8% / 7 M urea gel. Quantificare l'RNA purificato libreria spettrofotometria UV.
2. Generazione in vitro di aptameri
- Prima di selezione, preparare il buffer di selezione e di buffer di ripiegamento. Preparare un buffer HEPES contenente 100 Hepes mM, pH 7.4. Utilizzare NaOH per regolare il valore pH e poi conservare a temperatura ambiente. Preparare un buffer ripiegamento dell'RNA (5xHBS) contenente 50 mM pH Hepes 7,4, 750 mM NaCl, 5 mM MgCl 2; 5 mM CaCl 2; 13,5 mm KCl. Preparare una povera di sale buffer di legame RNA (10 HEPES mM pH 7,4, 50 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2.7 mM KCl, 10 mM DTT, 0,01% BSA e un alto contenuto di sale buffer di RNA binding (10 HEPES mM pH 7,4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2.7 mM KCl, 10 mM DTT, 0,01% BSA). Conservare questi buffer a -20 ° C.
- Eseguire la SELEX principalmente come descritto 1-4. Prima di ogni tornata di selezione, ripiegare le piscine RNA in 1xHBS tampone (10 HEPES mM pH 7,4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2.7 mM KCl), il calore a 95 ° C per 3 minuti e poi raffreddare lentamente a 37 ° C. Continua l'incubazione a 37 ° C per 10 min.
- In generale, al fine di ridurre al minimo legame aspecifico con i filtri di nitrocellulosa, pre-adsorbire le piscine RNA ripiegato ad un filtro di nitrocellulosa (HAWP filtro, 0,45 micron) per 30 minuti, prima di incubazione con l'obiettivo di HIV-1 Bal proteina gp120.
- Incubare la pre-cancellati piscina RNA con la proteina bersaglio in condizioni di scarsa sale tampone RNA vincolante per 30 minuti per round SELEX 1 a 4. Dopo il quarto giro di SELEX, utilizzare un alto contenuto di sale tampone legame RNA. Con il progresso SELEX, ridurre la quantità di proteina gp120 e aumentare il tRNA di lievito concorrente al fine di aumentare la severità delle aptamer selezione.
- Per il primo ciclo di selezione, incubare il pre-cancellati pool random RNA (40 mg, 1,5 nmol, 9x10 14 molecole) e HIV-1 Bal proteina gp120 (0,23 nmol, RNA / proteine di 6,5 / 1) a 200 l bassa sale RNA tampone vincolante su una piattaforma rotante a temperatura ambiente per 30 min.
- Passare la reazione attraverso un pre-filtro bagnato nitrocellulosa e lavare con 1 mL di tampone vincolanti.
- Eluire l'RNA legato dal filtro con 200 microlitri di buffer di eluizione (7 M urea e 5 mM EDTA) a 95 ° C per 5 minuti, seguita da estrazione con fenolo / cloroformio e la concentrazione con un YM-30 microcontrollore colonna.
- Reverse trascrivere la piscina recuperato RNA utilizzando la RT-PCR ThermoScript sistema (Invitrogen) e amplificare per 15 cicli di PCR.
- Purificare la piscina dsDNA amplificato utilizzando un kit di Gel QIAquick purificazione e di trascrivere, come descritto sopra per la prossima tornata di selezione.
3. Progresso SELEX monitorato da filtro analisi vincolante
- Monitorare l'andamento della SELEX aptameri da filtro analisi vincolanti. Trattare la piscina RNA con CIP di rimuovere l'etichetta di iniziare 5'-trifosfato e poi con T4 chinasi polinucleotide e γ-32P-ATP.
- Calore di 10 pmol CIP trattati RNA biblioteca a 95 ° C per 5 minuti e poi raffreddare sul ghiaccio. Successivamente, aggiungere 2l di PNK tampone, 1 l di T4 chinasi polinucleotide, 1 l di gamma P-32-ATP e acqua a 20 l.
- Incubare a 37 ° C per 30 minuti, poi aggiungere 20 mu ^; L di acqua e purificare la reazione da G-50 colonna. Infine, ottenere 40 ml di RNA marcato a concentrazione finale di 250 nM.
- Prima del test, ripiegare le piscine etichettato RNA in 1xHBS tampone (10 HEPES mM pH 7,4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl2, 2,7 mM KCl), il calore a 95 ° C per 3 minuti e poi raffreddare lentamente a 37 ° C. Continua l'incubazione a 37 ° C per 10 min.
- Effettuare una 100 l di reazione di legame come ad esempio qui. Incubare alla fine marcati RNA piscina (10 nm) con le proteine gp120 (100 nM) e di 10 volte l'eccesso molare di concorrente tRNA aspecifici (100 nM) nel settore high-sale buffer di RNA vincolante per 30 min.
- Separato da 50 l di reazione vincolante da un pre-filtro di nitrocellulosa bagnato.
- Lavare il filtro con 2 mL di tampone di legame e contare la radioattività trattenuta dal filtro attraverso un multi-purpose contatore a scintillazione (Beckman Coulter). Come un controllo di input, contano i restanti 50 ml di reazione di legame al tempo stesso. Calcolare la percentuale del RNA trattenuta dal filtro nell'RNA ingresso come l'affinità di legame.
4. La clonazione, la sequenza e allineamenti
- Dopo 11 turni, se non ulteriore arricchimento si osserva anche a seguito di ulteriori turni di selezione poi vincolante massima del pool di RNA è potenzialmente stato raggiunto.
- Reverse trascrivere la piscina altamente arricchito aptamero (12 ° piscina RNA) mediante RT-PCR ThermoScript sistema (Invitrogen) e successivamente amplificare il cDNA risultante mediante PCR. Purificare il prodotto di PCR utilizzando un kit di Gel QIAquick purificazione (QIAGEN). Clonare il gel purificato il prodotto DNA nel vettore di clonazione TA pCR 2.1 (Invitrogen). In totale, inoculare 170 cloni di persona, e li identificano con il sequenziamento del DNA per ottenere singole sequenze.
- Classificare i singoli cloni in sei diversi gruppi in base al allineamenti di sequenze aptamero individuali. Ha scelto una sequenza rappresentante di ogni gruppo (A-1, A-5, A-9, A-12, A-28 e B-68) per la caratterizzazione ulteriore a causa della loro abbondanza relativa all'interno del loro gruppo.
5. Generazione di aptamero e RNA da chimera nella trascrizione in vitro
- Generare direttamente a doppio filamento stampo di DNA mediante PCR utilizzando 2 mM ciascuno di 5'-e 3'-primer, insieme con 2 mM MgCl 2 e 200 mM di ogni dNTP, e recuperare i prodotti risultanti PCR utilizzando un kit di gel QIAquick purificazione.
- Trascrivere filo chimera senso dai suoi modelli di PCR DNA generati utilizzando il kit DuraScription (Epicentre, Madison, WI). Nella miscela di reazione di trascrizione, sostituire la canonica CTP e UTP con 2'-F-CTP e 2'-F-UTP per la produzione di RNA che è resistente alla RNasi A degradazione.
- Tipicamente, incubare 20 l di reazione contenente 1 mg di DNA, 2 ml 10xbuffer, 2 dATP microlitri, 2 dGTP microlitri, 2 microlitri 2'-F-dCTP, 2 microlitri 2'-F-dUTP, 2 DTT microlitri e 2 microlitri T7 RNA polimerasi a 37 ° C per 6 ore, e successivamente purificare con Bio-Spin 30 colonne (Bio-Rad) a seguito di estrazione fenolo e precipitazione in etanolo.
- Al fine di evitare la risposta all'interferone, ulteriormente trattare i RNA trascritto dal CIP per rimuovere l'inizio 5'-trifosfato. Incubare totale 60 l di reazione contenente 3 mg di trascrizioni, 6 ml di Tampone 3 e 0,25 ml di PIC a 37 ° C per 60 min. Dopo fenolo / cloroformio esazione e precipitazione in etanolo, risospendere l'RNA a pellet in acqua.
- Per preparare le chimere, unire 95 ° C per 3 minuti e poi raffreddare a 37 ° C lentamente. Chimere ospitare solo il filamento di RNA senso con l'RNA antisenso appropriato in ripiegamento del buffer, il calore di Continua l'incubazione a 37 ° C per 10 min. Eseguire ripiegamento passo alla tampone finial 1xHBS. Per esempio: mescolare 10 ml di 10 micron filamento senso chimera, 10 ml di 10 fili mM antisenso e 5 microlitri di buffer ripiegamento (5xHBS) in 25 microlitri sistema.
6. Determinazione delle costanti di dissociazione da saggi di turno gel
- fine P 32 etichettare il aptamer rappresentante di ogni gruppo e il senso del filo chimere e poi ripiegare RNA nel buffer 1xHBS come descritto sopra.
- Preparare 25 mL del 5% gel mescolando 2,5 ml di tampone 10xTBE, con 3,125 ml di 40% di acrilammide / bis soluzione, 19,375 ml di acqua, 150 ml di persolfato di ammonio 10% (APS) soluzione, e 30 TEMED microlitri. Il gel dovrebbe polimerizza in circa 30 min. Rimuovere con attenzione il pettine e utilizzare una siringa da 30 ml con ago per lavare i pozzetti con tampone di corsa (1xTBE).
- Completare il montaggio dell'unità di gel e la connessione a un alimentatore. Il gel può essere pre-corsa per un'ora a 180 V a 4 ° C.
- Serie diluire l'HIV-1Bal proteina gp120 con tampone vincolante per le concentrazioni desiderate. La r finaleconcentrazioni eaction di gp120 sono 0, 1, 5, 10, 20, 40, 80, 160, 320, 640 nM. Incubare una quantità costante di 5'-P 32-fine-marcato RNA (10 nm) con aumento delle concentrazioni di proteina gp120 nel buffer di legame (totale 20 l di reazione) su una piattaforma rotante a temperatura ambiente per 30 min.
- Dopo l'incubazione, mescolare 20 l di reazione di legame con buffer di 5 microlitri di carico nativo e caricare in un 5% in assenza di gel di poliacrilammide denaturante. Preparare un tampone nativo di carico (4x) contenente 10 mM Tris-HCl, pH 7,5, 1 mM EDTA, 0.1% blu di bromofenolo, 0,1% Xilene Cyanol FF, 0,1% Arancia G, 40% glicerolo. Conservare in aliquote a -20 ° C.
- Elettroforesi seguente (180 V a 4 ° C per 2 ore, fino a quando il colorante secondario funziona a mezzo di gel), esporre il gel per uno schermo l'immagine di fosforo e quantificare la radioattività utilizzando uno scanner Typhoon.
- Calcolare le costanti di dissociazione utilizzando una regressione non lineare curva con Prism Pad grafico.
7. Superficie cellulare studi di binding mediante citometria a flusso
- Generare aptamero fluorescenti e chimere utilizzando il siRNA Kit silenziatore Labeling (Ambion). Aggiungere i seguenti reagenti in ordine: 22,5 microlitri acqua priva di nucleasi, 5 ml 10 x Buffer Etichettatura, 15 microlitri di RNA (5 mg); 7,5 Dye Etichettatura microlitri. Incubare totale 50 microlitri di etichettatura reazione a 37 ° C per 1 ora.
- Dopo l'incubazione, aggiungere 5,0 microlitri (0,1 vol) 5 M di NaCl e 125 microlitri (2,5 vol) freddo EtOH 100%, e mescolare accuratamente. Incubare a -20 ° C per 60 min. Centrifugare a velocità massima a 4 ° C per 20 min. Rimuovere il surnatante e lavare pellet con 175 microlitri EtOH 70%. Aria secca pellet al buio e poi sospendere RNA etichettati in 15 l di acqua priva di nucleasi.
- Misurare l'assorbanza della RNA marcato a 260 nm e al massimo assorbimento per il colorante fluorescente. Calcolare la base: rapporto di tintura e la concentrazione di RNA in base al calcolatore fornito da http://www.ambion.com/techlib/append/base_dye.html .
- Mix Cy3 marcato senso e antisenso chimere filo filo e ripiegare in ripiegamento tampone come descritto sopra.
- Ottenere CHO-WT esprimere gp160 e CHO-EE cellule di controllo attraverso la ricerca AIDS e reagente Reference Program 5, 6. Crescere le cellule in GMEM-S medio (Glutamina con deficit terreno minimo indispensabile con 400 mM metionina sulfoximine (MSX)) (Gibco, Invitrogen). Coltivare le cellule in una umidificata al 5% CO 2 incubatore a 37 ° C.
- Lavare i CHO-WT gp160 o CHO-EE cellule di controllo preriscaldata con tampone di lavaggio, trypsinize e staccare dalle piastre. Dopo aver lavato le cellule due volte con 500 microlitri di buffer vincolante, resupend il pellet di cellule in un tampone di legame e incubare a 37 ° C per 30 min. Cellule pellet e poi risospendere in 50 ml di tampone preriscaldata vincolante contenente 400 nM Cy3 marcato RNA sperimentale.
- Dopo incubazione a 37 ° C per 40 minuti, lavare le cellule tre volte con 500 ml di tampone preriscaldata vincolanti, e, infine, risospendere in 350 ml di buffer di legame preriscaldata a 37 ° C e immediatamente l'analisi mediante citometria a flusso.
8. Interiorizzazione e studi di localizzazione intracellulare di Live-cell microscopia confocale
- Prima di un giorno di test, crescono i CHO-WT gp160 e CHO-EE cellule di controllo in 35 mm con piastra di semina a 0.3x10 6 in 2 ml di mezzo GMEM-S per permettere circa il 70% confluenza in 24 ore
- Il giorno degli esperimenti, lavare le cellule con 1 ml di PBS preriscaldata. E incubare con 1 ml di pre-riscaldato terreno di coltura completo per 30 minuti a 37 ° C.
- Preparare Cy3-marcato aptamer-siRNA chimere come descritto sopra. Incubare la ripiegare aptamer-stick con il siRNA-stick contenente 5'-Cy3 marcato senso del filo per formare il aptamer-stick-siRNA coniugati come descritto sopra.
- Prima del test, preparare 0,15 mg / ml soluzione di Hoechst 33342 (colorante nucleare di cellule vive, Molecular Probes, Invitrogen, CA) in acqua e conservare in aliquote a 4 ° C.
- Macchiare le cellule da un trattamento con 0,15 mg / ml Hoechst 33342 per 15 minuti a 37 ° C. Immediatamente, sciacquare la tintura con 1,0 ml di mezzo fresco due volte e sostituire 2 ml preriscaldata mezzo fresco.
- Aggiungere il Cy3 marcato aptamer-siRNA chimera ad una concentrazione 100 nM finale nei media e incubare per live-cell microscopia confocale in microscopia incubatore al 5% CO2 a 37 ° C.
- Raccogliere le immagini ogni 15 minuti utilizzando un Zeiss LSM 510 Meta invertito 2 Sistema microscopio confocale fotone in immersione con ingrandimento 40x.
9. In vitro di HIV-1 sfida e test antigene p24
- Acquisto CCRF-CEM cellule ATCC. Crescere in RPMI-1640 (Cellgro, Mediatech Inc.) supplementato con 10% siero fetale bovino (FBS, Hyclone), L-glutamina e 1xPen-Strep (Gibco, Invitrogen). Cellule cultura in un umidificata al 5% CO 2 incubatore a 37 ° C.
- Ottenere mononucle sangue perifericocampioni ar da donatori sani appartenenti alla City of Hope National Medical Center (personale della clinica).
- Isolare PBMC dal sangue intero mediante centrifugazione attraverso Ficoll-Hypaque soluzione (HISTOPAQUE-1077, Sigma). Impoveriscono cellule CD8 (cellule T-cytotoxic/suppressor) da PBMC utilizzando Dynabeads CD8 (Invitrogen, CA).
- Crescere le cellule in cellule T mezzo attivo (BioE, St. Paul, MN). Cellule cultura in un umidificata al 5% CO 2 incubatore a 37 ° C.
- Ottenere da HIV-1 IIIB e NL4-3 del virus e virus HIV-1 Bal dalla ricerca AIDS e reagente Reference Program. Dopo la propagazione del virus, conservare in aliquote a -80 ° C.
- Infettare CCRF-CEM cellule o PBMC umane con virus HIV (IIIB, NL4-3 o Bal) (MOI 0,001 o 0,005). Dopo 24 ore di post-infezione, lavare delicatamente le cellule con PBS per tre volte per rimuovere minacce. Continuare a coltivare le cellule infette in un 5% di microscopia incubatore CO 2 a 37 ° C per 4 giorni.
- Prima di RNA trattamenti, lavare delicatamente le cellule infette con PBS per tre volte per rimuovere minacce. Incubare 2x10 4 cellule infette e 3x10 4 cellule non infettate con ripiegata RNA sperimentale a 400 nM concentrazione finale in piastre da 96 pozzetti a 37 ° C (100 ul per pozzetto, saggio triplex).
- Raccogliere i surnatanti cultura (10 L per pozzetto) in tempi diversi (3 d, d 5, 7 e 9 d d), e conservare a -20 ° C fino al dosaggio p24.
- Effettuare le analisi antigene p24 utilizzando un antigene p24 di HIV-1 kit ELISA.
10. La funzione di rilevamento siRNA da quantitativa RT-PCR
- Infettare CCRF-CEM cellule o PBMC umane con virus HIV (IIIB, NL4-3 o Bal) e trattare con la RNA sperimentale (400 Nm), come descritto in precedenza.
- Dopo 7 giorni di trattamento, le cellule pellet e isolare RNA totale con STAT-60. Trattare l'RNA totale con DNasi I di rimuovere il DNA genomico e produrre cDNA con 2 mg di RNA totale.
- Mescolare i seguenti reagenti: 8 ml acqua priva di nucleasi; 1,5 ml 10 x DNasi buffer, 4 microlitri di RNA (2 mg), inibitore RNain 0,5 microlitri e 1,0 microlitri RNasi-free DNasi I. Incubare totale di 15 microlitri di reazione a 37 ° C per 1 ora e calore a 80 ° C per 10 minuti per inattivare DNasi I. Immediatamente, chill la reazione sul ghiaccio.
- Aggiungere 2 microlitri fondo casuale (50 ng / mL) e 1ml dNTP (10 mM) nella miscela di reazione di cui sopra, e poi il calore a 65 ° C per 5 min. Immediatamente, chill la reazione sul ghiaccio.
- Aggiungere i seguenti reagenti: 5 ml 5xFirst filone tampone; 2,5 ml 0,1 M DTT; 0,5 ml RNain inibitore e 1,0 mL MMLV-RT. Incubare reazione totale 27 ml a 25 ° C per 10 minuti ea 37 ° C per 1 ora. Dopo la reazione, la miscela di calore a 70 ° C per 15 minuti per inattivare della trascrittasi inversa e poi raffreddare sul ghiaccio. Il cDNA è pronto per qRT-PCR.
- Analizzare l'espressione dei geni bersaglio di RT-PCR quantitativa con 2 x iQ SyberGreen Mastermix e set di primer specifico ad una concentrazione finale di 400 Nm (saggio triplex). Usare l'espressione GAPDH come controllo interno per la normalizzazione dei dati qPCR.
11. Rappresentante dei risultati:
1. New aptameri RNA contro l'HIV-1 BAL gp120 sono isolati e caratterizzati.
Come descritto nella sezione sperimentale, una prima biblioteca oligonucleotidi DNA contenente un 50 regione casuale nt fiancheggiato da regioni fondo fisso sul 5 'e 3' viene amplificato e trascritto in un pool di RNA. Questa libreria iniziale consiste di un massimo di 10 15 sequenze diverse (1 nmol), che piega in una vasta gamma di varie strutture 3-D. L'elevata complessità e la diversità della biblioteca iniziale potrebbe garantire la presenza di strutture attive con una buona affinità di legame al bersaglio.
Impiegare una procedura in vitro SELEX (Figura 1) per selezionare 2'-fluoropirimidina modificato aptameri RNA che si legano selettivamente la R5 ceppo di HIV-1 Bal proteina gp120 busta 7. Come mostrato nella Figura 1, una a base di nitrocellulosa strategia di selezione viene effettuata per isolare specifici target-RNA vincolante da non vincolante molecole di RNA. Dal momento che la proteina si attacca alla nitrocellulosa, solo l'RNA / proteine complessi o aggregati può essere mantenuto sulla membrana e RNA libero sono sbiaditi. In condizioni di denaturazione, gli RNA legati vengono recuperati e sono contrario trascritto in cDNA e amplificato in dsDNA, e successivamente trascritto in vitro per creare un nuovo pool di RNA per il ciclo di selezione successiva. Il rigore selezione è aumentato riducendo la quantità di proteina bersaglio e aumentando la quantità di tRNA concorrente. La quantità di pool di RNA, proteine e tRNA concorrente utilizzati in ogni fase di selezione è mostrato nella Tabella 1.
Monitorare l'andamento della selezione dopo ogni ciclo di SELEX dal saggio filtro vincolanti. Valutare l'affinità di legame, come la percentuale del RNA mantenuto sul filter in piscina RNA totale. Il pool di RNA di partenza (1-RNA) mostra solo lo 0,1% del RNA ingresso trattenuto sulla membrana. Tuttavia, dopo nove tornate di selezione il nono RNA biblioteca (9-RNA) è 9,72% dell'ingresso RNA legato. Sebbene turni di selezione aggiuntivi sono stati condotti, nessun ulteriore arricchimento è osservato, suggerendo che il legame massima del pool di RNA è stato raggiunto (Figura 2A). Simili con il saggio filtro vincolante, il saggio spostamento gel è anche una delle strategie più importante per determinare costanti di dissociazione. Questa procedura è facile e conveniente. Come mostrato nella Figura 2B, saggi di turno gel confermano ulteriormente l'attività vincolante delle piscine RNA. Questi risultati dimostrano che alcuni ligandi ad alta specificità di legame per la proteina target sono successivamente arricchito in queste piscine RNA ..
Clonare e sequenziare le piscine aptamer altamente arricchito (12-RNA). Secondo gli allineamenti delle singole sequenze aptamero clonate, sei diversi gruppi sono classificati come indicato nella Tabella 2. Circa il 40% dei cloni (gruppo I e II aptameri) contengono una sequenza conservata: A (A / G) TTGAGGGACC (A / G). Abbiamo scelto una sequenza rappresentante di ogni gruppo (ad esempio: A-1, A-5, A-9, A-12, A-28 e B-68) per la caratterizzazione ulteriore a causa della loro abbondanza relativa all'interno del loro gruppo. Attraverso un test nativo gel mobility shift, le costanti di dissociazione (K d) di questi aptameri rappresentative sono calcolati (Figura 3A). Per esempio, A-1, il meglio del aptameri, ha un valore apparente Kd di 52 nM (Figura 3B). Come mostrato nella Figura 3A, questi aptameri selezionati possono legarsi selettivamente con l'obiettivo di HIV-1 Bal gp120, ma non il CM proteina HIV gp120.
2. Anti-gp120 aptamero lega in modo specifico ed è interiorizzato da cellule che esprimono l'HIV gp160 e inibiscono HIV-1 nelle cellule in coltura.
CHO-gp160 cellule che esprimono stabilmente la glicoproteina gp160 envelope di HIV sono usati per testare interiorizzazione vincolante e del selezionati anti-gp120 aptameri. Queste cellule non sono in processo di gp160 gp120 e gp41, perché manca il bavaglio proteasi codificate necessari per l'elaborazione busta. Come controllo si usa il parentali CHO-EE linea cellulare che non esprime gp160. Analisi di citometria a flusso (Figura 4A) rivelano che il Cy3 marcato aptameri si legano specificamente alle cellule CHO-gp160, ma non il controllo CHO-EE cellule. Inoltre, in tempo reale dal vivo di cellule Z microscopia confocale indica che l'etichetta-Cy3 aptamero è selettivamente internalizzati nel CHO-gp160 cellule (Figura 4B e 4C) dopo 2 ore di incubazione, ma non il CHO-EE cellule di controllo ( Figura 4D). Figura 4C mostra anche che il aptamer aggregati all'interno del citoplasma, che suggerisce la gp120 aptameri forse entrare nelle cellule attraverso endocitosi mediata da recettori.
Nel test HIV-1 sfida, HIV-1 infetti-PBMC cellule vengono trattate con il aptameri. In giorni diversi post-trattamento con la aptameri, aliquote dei media sono stati analizzati per i livelli di antigene virale p24 (4E Figura). I risultati mostrano che l'anti-gp120 aptameri (A-1) inibisce la produzione di HIV-1 p24 con un nano-concentrazione molare.
3. Anti-gp120 aptamer-siRNA chimera è stata progettata e valutata la sua efficacia come cellule di tipo specifico sistema di consegna di siRNA
Come mostrato nella Figura 5A, il aptamer e filo segmento senso del siRNA contenute nucleasi resistente 2'-fluoro UTP e 2'-fluoro CTP e sono sintetizzati da modelli dsDNA corrispondente da trascrizione in vitro batteriofago. Al fine di aumentare la flessibilità della molecola, a due linker nucleotide (UU) è inserito tra il aptamer e la parte del substrato Dicer. Per preparare il siRNA contenenti chimere, in vitro trascritto chimerico aptamer senso polimeri sono filo ricotto con concentrazioni equimolari di un filamento di RNA antisenso non modificato. Questi dati dai dati analitici gel shift (Figura 5B) e citometria a flusso (Figura 5C) indicano che le chimere mantenere circa le affinità stesso valore vincolante delle aptameri solo. Il tempo impiegato per le immagini in tempo reale la microscopia confocale (Figura 5D) mostrano che Cy3 marcato chimera Ch A-1 può essere successo interiorizzate nel citoplasma delle cellule. Come previsto, nessun assorbimento della chimera è osservato con il CHO-EE cellule di controllo (Fig. 5E).
Allo stesso modo, il potenziale antivirale di RNA è valutata da HIV-1 saggio sfida. I risultati delle analisi dell'antigene p24 dell'HIV (Figura 6A e 6B) mostrano che sia aptamero chimera e inibire la produzione di p24, ma la più forte inibizione è osservato con la chimera Ch A-1 di trattamento.
Per confermare che il componente siRNA funziona insieme al APTAmer, a seguito di interiorizzazione della A-1 nelle cellule infettate chimera Ch, valutiamo anche i livelli relativi di inibizione di tat / giro di espressione genica da RT-PCR quantitativa saggi di espressione. Troviamo che il trattamento delle cellule infettate con le chimere è in grado di indurre il silenziamento del tat / giro gene, mentre la aptamer da solo non ha influito tat / espressione genica giro (Figura 6C e 6D). Questi risultati forniscono ulteriore supporto che l'siRNA aptamer consegnato trigger RNAi.
| La quantità di proteine, RNA piscina e tRNA utilizzati per la selezione | |||||
| SELEX round | Rapporto di Target / RNA | Proteina gp120 | RNA piscina | Concorrente tRNA | Selezione Buffer |
| 1 | 1/6.5 | 229,8 pmol | 1,5 nmol (40,1 mg) | 0 | Iposodica buffer di SELEX |
| 2 | 1/6.5 | 114,9 pmol | 0,75 nmol (20,1 mg) | 0,25 nmol (6,6 mg) | |
| 3 | 1 / 8 | 76,6 pmol | 0,625 nmol (16,7 mg) | 0,25 nmol (6,6 mg) | |
| 4 | 1 / 8 | 76,6 pmol | 0,625 nmol (16,7 mg) | 0,25 nmol (6,6 mg) | |
| 5 | 1 / 8 | 38,3 pmol | 0,306 nmol (8.18 mg) | 0,5 nmol (13,2 mg) | Alta sale tampone SELEX |
| 6 | 1 / 8 | 38,3 pmol | 0,306 nmol (8.18 mg) | 0,5 nmol (13,2 mg) | |
| 7 | 1 / 10 | 26,8 pmol | 0,268 nmol (7,16 mg) | 0,5 nmol (13,2 mg) | |
| 8 | 1 / 10 | 26,8 pmol | pmol 0,268 nmol (7,16 mg) | 1 nmol (26,4 mg) | |
| 9 | 1 / 10 | 15,3 pmol | 0,153 nmol (4,09 mg) | 1 nmol (26,4 mg) | |
| 10 | 1 / 10 | 15,3 pmol | 0,153 nmol (4,09 mg) | 1,5 nmol (39,6 mg) | |
| 11 | 1/12.5 | 7,66 pmol | 0,096 nmol (2,56 mg) | 2 nmol (52,8 mg) | |
| 12 | 1/12.5 | 7,66 pmol | 0,096 nmol (2,56 mg) | 2 nmol (52,8 mg) | |
Tabella 1. Le condizioni di selezione. La quantità di proteine, RNA piscina e tRNA utilizzato per ogni selezione e di buffer di selezione sono indicati.
| Gruppo | RNA | Sequenze casuali | Frequenza (140 cloni) |
| Gruppo I | A-1 | AATTGAGGGACCA CGCGCTGCTTGTTGTGATAAGCAG TTTGT CG GATGG | 33 (23,6%) |
| B-7 | AATTGAGGGACCA ACGCGAGGATGTGGATAGTGTGTA TTTGC GT GATGG | 3 | |
| A-32 | AATTGAGGGACCG TTGGTAAAAGCCGGA AATTG AGCT TTTAC GGC GATGG | 5 | |
| B-55 | AATTGAGGTACCGCG TTATTAGGAACA AATTG GAATTCTAAACGC GATGG | 2 | |
| A-24 | AATAGAGGGACC CAGATATAGGCTACACGGATGATGGTGTATCTG GATGG | 1 | |
| B-19 | AATAGAGGAACCG TTTCAGAAGACTACAGGTTAGTCCAATGAAGC GACGG | 1 | |
| B-31 | AATAGAGGGACCG TGGACAATAATTTATGGTCA TTTATTGGCAC GATGG | 1 | |
| Gruppo II | A-12 | AGTAGAGGAACCA AGCAATGGATGAATGCAAAAGTGTAAATGCTT GATGG | 10 (7,1%) |
| Gruppo III | A-9 | TGAGTTTGGGTAAATTTCCGGTTTCGGTTTACTCACGAAAGATCGGTCGG | 15 (10,7%) |
| Gruppo IV | A-28 | TAAAGGAGGGAAGGATGAGACCGCACGAAAAATATCAGCATACG TTTGTG | 10 (7,1%) |
| Gruppo V | A-5 | GAAACTAGTTTGAATAATGGTGTAGAGGAGGGTCAATAGTTTCG TTGGTG | 9 (6,4%) |
| Gruppo VI | B-68 | ACATAGTAATGACACGGAGGATGGAGAAAAAACAGCCATCTCTTGACGGT | 2 |
| Altrui | Sequenza orfani | 48 |
Tabella 2. L'allineamento e l'identificazione di aptameri RNA. Dopo il 12 ° turno di selezione, il pool selezionato RNA è stato clonato e sequenziato. Dopo l'allineamento di tutti i 140 cloni, sei gruppi di anti-gp120 aptameri sono stati identificati. Solo le sequenze casuali di regioni centrali aptamer (5'-3 ') sono indicati. Gli isolati che si verificano con frequenze multiple sono specificati.
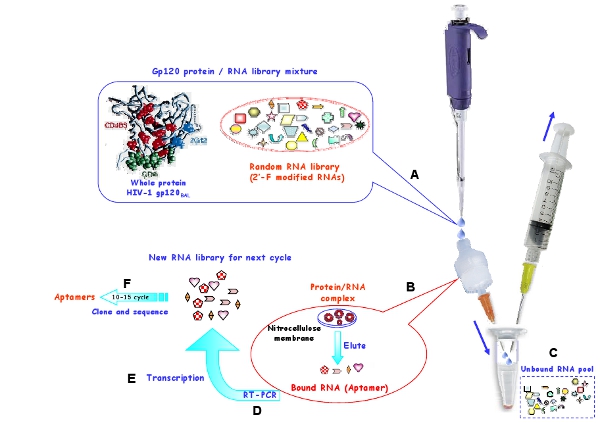
Figura 1: Rappresentazione schematica della procedura di selezione in vitro utilizzando una membrana di nitrocellulosa, per la generazione di aptameri RNA di HIV-1 Bal proteina gp120. (A) La piscina di partenza RNA e proteine bersaglio sono state incubate per formare complessi. (B) Le molecole di RNA legato sono stati mantenuti sulla membrana e eluito dalla membrana in condizioni di denaturazione. (C) L'RNA non legato sono state spazzate via. (D) L'RNA selezionate sono state trascritte e invertire amplificato mediante PCR. (E) Il DNA rilevante è stato successivamente trascritto in RNA nuova piscina per i cicli di selezione successiva. (F) Dopo 10-15 giri di selezione, il aptameri selezionati sono stati clonati e sequenza.
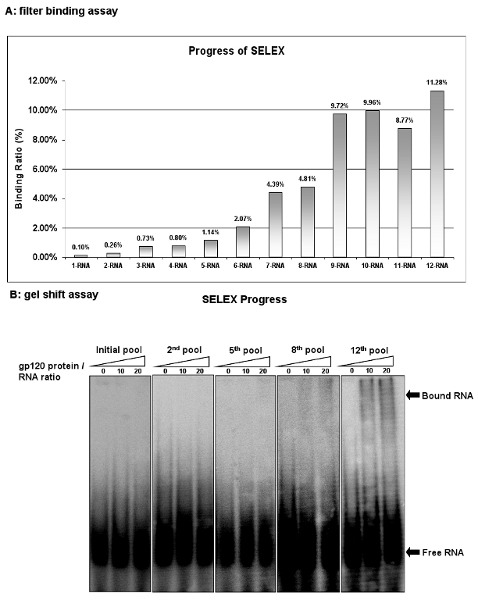
Figura 2: Lo stato di avanzamento del virus HIV-1 gp120 selezione aptameri. (A) L'attività di legame del pool di RNA ad ogni ciclo è stato analizzato dal filtro test vincolante con tRNA concorrente. Attività di rilegatura sono stati calcolati come percentuale di ingresso dell'RNA trattenuta dal filtro di proteine / RNA complesso. (B) L'attività di legame del pool di RNA ad ogni ciclo è stato analizzato mediante test gel shift. Il pool di 12 ° RNA hanno mostrato la più alta attività di legame.
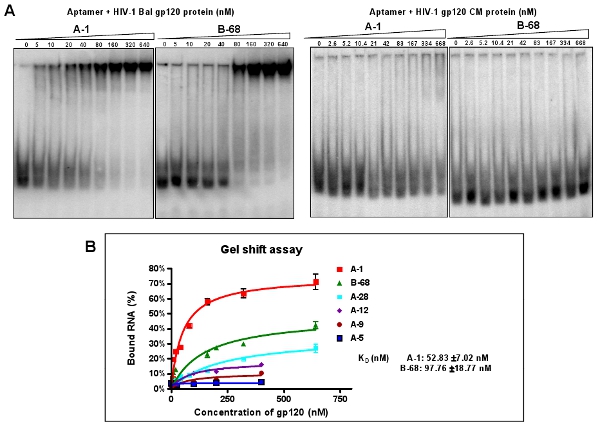
Figura 3: saggio di attività legame di selezionati aptameri individuale contro l'HIV-1 Bal gp120. (A) La 5'-end P 32 aptameri etichettati individuali sono stati incubati con l'obiettivo di quantità crescenti di proteina gp120 o non specifica proteina CM. Le miscele di reazione di legame sono stati analizzati da un saggio di turno gel mobilità. Aptamer A-1 e B-68 ha mostrato la migliore affinità di legame con la proteina bersaglio, ma le proteine non CM. Dati rappresentano la media di quattro replicati. (B) Curva Binding da un saggio di turno gel.
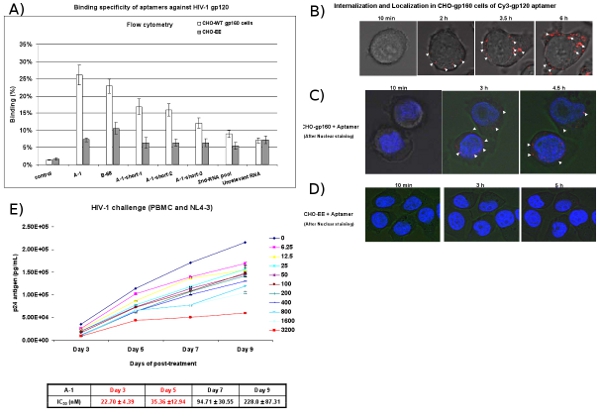
Figura 4: Cell-tipo di rilegatura e studi specifici assorbimento di aptameri. (A) vincolanti superficie cellulare di Cy3-marcato RNA è stata valutata mediante citometria a flusso. RNA Cy3 marcati sono stati testati per il legame di CHO-gp160 cellule e CHO-EE cellule di controllo. Il aptameri selezionati hanno mostrato cellule tipo di affinità specifiche vincolanti. Il 2 ° RNA RNA piscina e irrilevanti sono stati utilizzati come controlli negativi. Dati rappresentano la media di tre repliche. (B) Internalizzazione analisi. CHO-gp160 cellule sono state coltivate in piastre di 35 mm e incubate con una concentrazione di 100 Nm di Cy3-etichetta A-1 in terreni di coltura per il real-time live-cell-analisi di microscopia confocale. Le immagini sono state raccolte in 15 min. intervalli con ingrandimento 40x. (C, D) localizzazione analisi. CHO-gp160 e cellule CHO-EE cellule di controllo sono state coltivate in 35 piastre mm. Prima di incubazione con 100 Nm di Cy3-etichetta A-1, le cellule sono state colorate con Hoechst 33342 (colorante nucleare per cellule vive) e poi analizzati utilizzando in tempo reale la microscopia confocale. (E) La scelta anti-gp120 aptameri inibire la replicazione dell'HIV-1 nelle PBMC umane precedentemente infettati con HIV-1-3 NL4 virus. Diverse concentrazioni e ora punta sono stati presentati. IC50 valore è stato elencato. Dati rappresenta la media delle misurazioni triplice copia di p24.
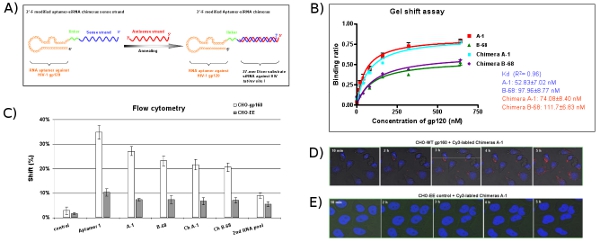
Figura 5: La progettazione e la valutazione del aptamer-siRNA sistema di consegna chimera. (A) Schema aptamer-siRNA RNA chimerico: la regione del movimento anti-gp120 aptamero è responsabile per il legame di gp120 e il siRNA si rivolge ad un esone comune di HIV-1 TAT / giro. La 2'-Fluoro modificato aptamer-siRNA filamento senso unico è stato co-trascritta, seguito da ricottura del filamento complementare antisenso siRNA per completare la molecola chimerica. Un linker (UU) tra il aptamer e siRNA è indicato in verde. (B) Il aptamer-siRNA RNA chimerico che sono confrontabili valori di Kd così come aptameri genitori si legano specificamente la proteina gp120 dell'HIV Bal. Dati rappresentano la media di tre repliche. (C) Cell-tipo specifici studi di legame aptameri. RNA Cy3 marcati sono stati testati per il legame di CHO-gp160 cellule e CHO-EE cellule di controllo. Cellula surassociazioni di fronte a Cy3-marcato RNA sono stati valutati mediante citometria a flusso. Il aptameri selezionati hanno mostrato cellule tipo di affinità specifiche vincolanti. Il 2 ° RNA piscina e RNA irrilevanti sono stati utilizzati come controlli negativi. Dati rappresentano la media di due repliche. (D, E) Internalizzazione ed analisi localizzazione intracellulare. CHO-gp160 cellule sono state coltivate in piastre di 35 mm e sono state colorate con Hoechst 33342 (colorante nucleare di cellule vive). Successivamente, le cellule sono state incubate in terreno di coltura con una concentrazione di 100 Nm di Cy3 marcato chimera in tempo reale live-cella di analisi di microscopia confocale come descritto in precedenza.
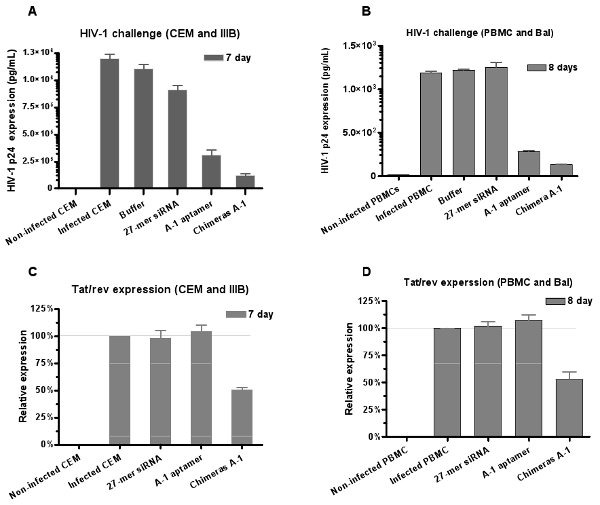
Figura 6: La doppia inibizione di HIV-1 mediata da aptamer-siRNA chimere. Entrambi anti-gp120 aptamer e aptamer-siRNA chimere neutralizzato da HIV-1 in (A), le cellule CEM (ceppo IIIB) e (B) cultura PBMC umane (ceppo BAL), rispettivamente. Dati rappresentano la media delle misurazioni triplice copia di p24. Le chimere hanno mostrato forte inibizione di aptamero solo indicare che (C, D) il siRNA consegnato dal aptameri down-regolato tat / giro di espressione genica in PBMC. Dati rappresentano la media di tre repliche.
Discussione
Aptameri sono evoluti in vitro degli acidi nucleici che assumono specifiche e stabili forme tridimensionali, fornendo in tal modo altamente specifico, legame stretto di molecole mirate 8. La bassa affinità nanomolare legame e specificità squisita aptameri ai loro obiettivi li rendono strumenti versatili per la diagnostica, imaging in vivo, e terapie 9. Con l'avvento della tecnologia per la consegna aptamer siRNA mirate è ora possibile utilizzare la funzione aptamer vincolan...
Divulgazioni
Riconoscimenti
Ringraziamo Britta Hoehn, Guihua Sole, Harris Soifer e Lisa Scherer per utili discussioni. Questo lavoro è stato sostenuto dalle concessioni dal National Institutes of Health AI29329 e HL07470 assegnato a JJR I seguenti reagenti sono stati ottenuti attraverso la ricerca e l'AIDS NIH reagente Reference Program, Division of AIDS, NIAID, NIH: La CHO-EE e CHO-gp160 cellulare linea, il pNL4-3 luc vettoriale; HIV-1 BAL gp120 da DAIDS, NIAID.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
|---|---|---|---|
| MF-Millipore membrana del filtro | Millipore | HAWP01300 | Dimensione dei pori 0,45 micron |
| Swinnex portafiltro | Millipore | SX0001300 | 13 millimetri di diametro |
| QIAquick Gel Extraction Kit | QIAGEN | 28706 | Purificazione del DNA |
| Microcontrollore YM-30 colonna | Millipore | 42410 | RNA concentrazione |
| Bio-spin 30 colonne | Bio-Rad | 732-6250 | RNA purificazione |
| PCR Taq DNA polimerasi | Sigma-Aldrich | D1806 | |
| ThermoScript RT-PCR sistema | Invitrogen | 11146-024 | |
| DuraScribe Kit di trascrizione T7 | Epicentre | DS010925 | |
| dNTP per PCR | Roche | 1 581 295 | |
| Acido ribonucleico, trasferimento da E.coli | Sigma-Aldrich | R1753 | tRNA concorrente |
| HIV-1 Ba-L proteina gp120 | la ricerca AIDS e reagente Reference Program | 4961 | Proteina bersaglio |
| Silenziatore etichettatura kit siRNA - Cy3 | Ambion | 1632 | |
| Acido fenolo / cloroformio 5 / 1 soluzione (pH 4,5) | Ambion | AM9720 | |
| Cloroformio / isopropanolo 24 / 1 soluzione | Sigma | C0549 | |
| Vitello fosfatasi intestinali (CIP) | New England BioLab | M0290L | |
| T4 chinasi polinucleotide | New England BioLab | M0201L | |
| Glicogeno | Roche | 10 901 393 001 | RNA precipitazioni |
| Gamma-P 32-ATP | MP Biomedica | 013502002 | Radiactivity |
| 40% AccuGel 19:01 | Nazionale di diagnostica | CE-850 | |
| 10xTBE | Nazionale di diagnostica | CE-860 | |
| N, N, N, N-Tetramethylethylenediamine (TMEMD) | Sigma-Aldrich | T9281 | |
| Di ammonio persolfato (APS) | Sigma-Aldrich | A3678 | |
| L-methioine sulfoximine | Sigma-Aldrich | M5379-250 mg | |
| RPMI media 1640 | Invitrogen | 11835-030 | |
| Bicarbonato di sodio, 7,5% w / v | Invitrogen | 25080-094 | |
| Minimo Medium (MEM) (10 ) | Invitrogen | 11430-030 | |
| MEM non essenziali aminoacido (100 ) | Invitrogen | 11140-050 | |
| TA clonazione kit con pCR 2,1 | Invitrogen | K2040-01 |
Riferimenti
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Robertson, D. L., Joyce, G. F. Selection in vitro of an RNA enzyme that specifically cleaves single-stranded DNA. Nature. 344, 467-468 (1990).
- Fitzwater, T., Polisky, B. A SELEX primer. Methods Enzymol. 267, 275-301 (1996).
- Weiss, C. D., White, J. M. Characterization of stable Chinese hamster ovary cells expressing wild-type, secreted, and glycosylphosphatidylinositol-anchored human immunodeficiency virus type 1 envelope glycoprotein. J Virol. 67, 7060-706 (1993).
- Vodicka, M. A. Indicator cell lines for detection of primary strains of human and simian immunodeficiency viruses. Virology. 233, 193-198> (1997).
- Zhou, J. Selection, characterization and application of new RNA HIV gp 120 aptamers for facile delivery of Dicer substrate siRNAs into HIV infected cells. Nucleic Acids Res. , (2009).
- Mayer, G. The chemical biology of aptamers. Angew Chem Int Ed Engl. 48, 2672-2689 (2009).
- Famulok, M., Hartig, J. S., Mayer, G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy. Chem Rev. 107, 3715-3743 (2007).
- Chu, T. C., Twu, K. Y., Ellington, A. D., Levy, M. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34, e73-e73 (2006).
- McNamara, J. O., 2nd, . Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat Biotechnol. 24, 1005-1015 (2006).
- Dassie, J. P. Systemic administration of optimized aptamer-siRNA chimeras promotes regression of PSMA-expressing tumors. Nat Biotechnol. 27, 839-849 (2009).
- Zhou, J., Rossi, J. J. The therapeutic potential of cell-internalizing aptamers. Curr Top Med Chem. 9, 1144-1157 (2009).
- Zhou, J., Li, H., Li, S., Zaia, J., Rossi, J. J. Novel dual inhibitory function aptamer-siRNA delivery system for HIV-1 therapy. Mol Ther. 16, 1481-1489 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon