Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Celular del cáncer colorrectal superficie de la proteína perfiles con un microarray de anticuerpos y la multiplexación de fluorescencia
En este artículo
Erratum Notice
Resumen
Se describe un procedimiento para la desagregación de cáncer colorrectal (CCR) para producir células viables único, que son capturados en microarrays de anticuerpos a medida el reconocimiento de los antígenos de superficie (CRC DotScan microarrays). Subpoblaciones de células obligado a los microarrays pueden ser perfiladas por multiplexación de fluorescencia utilizando anticuerpos monoclonales marcados con colorantes fluorescentes.
Resumen
El pronóstico actual y la clasificación de la Convención se basa en la puesta en escena de sistemas que se integran los hallazgos histopatológicos y clínicos. Sin embargo, en la mayoría de los casos de CCR, disfunción de las células es el resultado de numerosas mutaciones que modifican la expresión de proteínas y modificaciones post-traduccionales 1.
Una serie de antígenos de superficie celular, incluyendo cluster de diferenciación (CD) antígenos, han sido identificados como potenciales de pronóstico o marcadores metastásicos en el CCR. Estos antígenos hacer biomarcadores ideal como su expresión cambia a menudo con la progresión del tumor o interacciones con otros tipos de células, como linfocitos infiltrantes de tumor (TIL) y los macrófagos asociados al tumor (TAM).
El uso de la inmunohistoquímica (IHC) para el cáncer de sub-clasificación y el pronóstico está bien establecido para algunos tipos de tumores 2,3. Sin embargo, no solo "marcador" ha demostrado importancia pronóstica superior clínico-patológicas de ensayo o de ganado amplia aceptación para su uso en la presentación de informes de rutina de todos los casos la patología CRC.
Un enfoque más reciente de la estratificación pronóstica de los fenotipos de la enfermedad se basa en los perfiles de proteínas de superficie con varios "marcadores". Mientras que los perfiles de expresión de los tumores mediante técnicas proteómicas como iTRAQ es una poderosa herramienta para el descubrimiento de biomarkers4, no es el óptimo para su uso rutinario en los laboratorios de diagnóstico y no se pueden distinguir diferentes tipos de células en una población mixta. Además, grandes cantidades de tejido tumoral se requieren para la elaboración de perfiles de las glicoproteínas de la membrana plasmática purificada por estos métodos.
En este video se describe un método simple para crear perfiles de la superficie del proteoma de células viables de muestras desglosados CRC utilizando un microarray de anticuerpos DotScan CRC. Los microarrays 122-anticuerpo consiste en un estándar de 82-anticuerpo reconoce una amplia región del linaje de los marcadores específicos de leucocitos, las moléculas de adhesión, los receptores y los marcadores de inflamación y respuesta inmune 5, junto con una región por satélite para la detección de 40 marcadores potencialmente pronóstico de CRC . Las células son capturados sólo en los anticuerpos para los que expresan el antígeno correspondiente. La densidad de las células por punto, determinado por lectura óptica, refleja la proporción de células que expresan ese antígeno, el nivel de expresión del antígeno y la afinidad de los anticuerpos 6.
Para el tejido de la Convención o en la mucosa intestinal normal, las exploraciones ópticas reflejan el inmunofenotipo de la población mixta de células. Multiplexación de fluorescencia se puede utilizar con el perfil seleccionado sub-poblaciones de células de interés capturado en la matriz. Por ejemplo, Alexa molécula de adhesión celular 647-anti-epiteliales (EpCAM, CD326), es un antígeno de diferenciación pan-epiteliales que se usa para detectar células CRC y también las células epiteliales de la mucosa intestinal normal, mientras que ficoeritrina anti-CD3, se utilizó para detectar la infiltración de células T 7. El CRC DotScan microarrays debe ser el prototipo de una alternativa de diagnóstico para el sistema de clasificación anatómica basada en CRC.
Protocolo
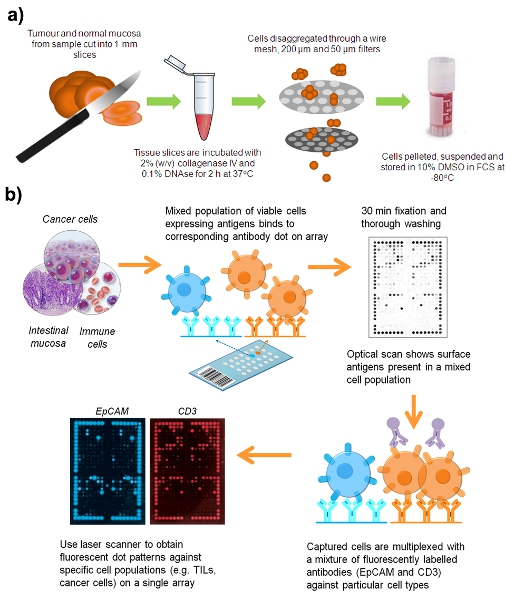
Figura 1. Flujo de trabajo para la preparación de una suspensión de células vivas de una muestra quirúrgica del CRC.
1. Desagregación de la muestra clínica
Todas las muestras fueron recolectadas en el Hospital Royal Prince Alfred (Camperdown, NSW, Australia) y el Hospital Concord de Repatriación (Concord West, NSW, Australia) con el consentimiento informado en el Protocolo N º X08-164.
- Recoger el cáncer fresco colorrectal (CCR) o muestras de adenoma, y en la mucosa intestinal normal por lo menos 10 cm del tumor. Almacene las muestras en Hank equilibrado de pH de la solución salina 7,3 (HBSS) a 4 ° C durante 12 h después de la resección.
- Siga las normas de seguridad de los patógenos humanos, procesar todas las muestras clínicas en un gabinete de seguridad biológica de clase II. Diseccionar las muestras en dos cubos mm en una placa de Petri con dos hojas de bisturí.
- Incubar tumor y el tejido normal por separado en tubos Eppendorf con ocasionales mezcla suave durante 60 minutos a 37 ° C con un volumen igual de medio RPMI 1640 que contiene 2% (v / v) colagenasa tipo 4 (Worthington, Lakewood, NJ, EE.UU.) y 0,1 % (w / v) desoxirribonucleasa I de páncreas bovino (DNAsa I, Sigma-Aldrich).
- Fuerza semi-digeridos de tejidos a través de un colador de alambre de malla fina con un émbolo de una jeringa de 10 ml, células de lavado a través de HBSS.
- Pase resultante suspensión de células a través de 200 micras y 50 micras filtros Filcon (BD Biosciences) para eliminar los agregados celulares. La mayoría de los agregados de ADN, moco y células se extraen de esta serie de filtraciones.
- Centrifugar las suspensiones de células a 400 xg a 20 º durante 5 minutos.
- Pellets resuspender celular en FCS inactivado por calor con 10% dimetilsulfóxido (DMSO), la congelación lenta en crioviales y almacenar a -80 °. El proceso de congelación tiende a reducir la mucosidad en la muestra y la lisis de glóbulos rojos.
2. Preparación de muestras para la captura de células
- Descongelar las muestras de forma rápida en un baño de agua a 37 ° y suspender las células en 10 ml de HBSS para lavar el DMSO.
- Centrifugar las suspensiones de células a 410 xg a 20 º durante 5 minutos.
- Se decanta el sobrenadante y resuspender el botón celular en 500 l de HBSS.
- Tratar la muestra con 0,1% (w / v) ADNasa I durante 20 minutos a temperatura ambiente.
- Mezcla de 10 l de cada suspensión celular con un volumen igual de azul de tripano y de carga de 10 l de la mezcla en un hemocitómetro. El uso de un microscopio de luz a 100 veces de aumento, el recuento de células viables, lo que parece claro debido a la exclusión azul de tripano, mientras que las células muertas de tomar el colorante. Un mínimo de 4 x 10 6 células viables se requiere para la captura de células en el microarray.
- Después del tratamiento con DNAsa, resuspender la suspensión celular en 10 ml de HBSS y centrifugar a 410 xg a 20 º durante 5 minutos.
- Decantar el sobrenadante y resuspender pellets de células en RPMI 1640 con un volumen final de 200 mL.
3. Microarrays de anticuerpos de captura de la célula
- Humedezca los microarrays de anticuerpos DotScan por inmersión de la sección de nitrocelulosa en tampón fosfato salino (PBS) durante unos 20 s. Limpie con cuidado los bordes del vidrio de la micromatriz con Kimwipes cruzados, evitando tocar la sección de nitrocelulosa.
- Agregue el agua a la bandeja de incubación de microarrays para proporcionar una cámara húmeda. Coloque los microarrays en la cámara y la pipeta la suspensión de células en RPMI 1640 en la sección de nitrocelulosa húmeda. Pipeta gotas en cada esquina de la nitrocelulosa para garantizar un reparto equilibrado de las células.
- Incubar los microarrays a 37 ° C durante 1 h. El período de incubación permite que las células puedan establecerse y entrar en contacto con los anticuerpos en el microarray. Las células que expresan antígenos de superficie que corresponde a los anticuerpos que la tierra en que va a capturar.
- Después de la incubación, sumerja los microarrays suave y verticalmente en tres pilas que contienen al menos 15 ml de PBS para lavar las células no unidas (20 s por lavado).
- Prepare un 3,7% (w / v) de formaldehído en PBS para fijar las células y anticuerpos de entrecruzamiento. Pipeta suavemente alrededor de 1 mL para cubrir la sección de nitrocelulosa de la micromatriz. Incubar durante 20 min a temperatura ambiente.
- Próxima inmersión microarrays en 3 cambios de PBS (15 ml, 30 s cada uno) para vaciar el exceso de formaldehído.
- Limpie los bordes y la parte posterior de la lámina de vidrio con Kimwipes y explorar el uso de microarrays del escáner mientras DotScan, la sección de nitrocelulosa está húmedo. La tomografía óptica proporciona el patrón de expresión de los antígenos de una mezcla de células las células de la población por ejemplo, CRC, leucocitos y otras células del estroma del tumor.
4. Fluorescencia de multiplexación
- Quitar el microarray del escáner y se aplican 200 l de tampón de bloqueo (2% w / v BSA, 2% inactivado por calor suero humano AB, PBS, pH 7,3). Incubar en la bandeja de microarrays a temperatura ambiente durante 20 min.
- Prepare la multíparaléxico solución en un tubo Eppendorf, cubiertas con papel de aluminio: 20 l ficoeritrina anti-CD3 (Beckman Coulter, Gladesville, NSW, Australia, # IM12824; 1/7.5 dilución final), 10 l Alexa Fluor 647-anti-EpCAM (Biolegend, San Diego, California, EE.UU., 1 / 15 de dilución), 2 l de suero inactivado por calor humano AB (Sigma-Aldrich, Castle Hill, NSW, Australia) y 118 l de tampón de bloqueo.
- Escurrir el exceso de tampón de bloqueo de los microarrays y la pipeta la solución de multiplexación en la sección de nitrocelulosa, extendiendo uniformemente. Incubar durante 30 minutos en la oscuridad a temperatura ambiente.
- Sumerja los microarrays verticalmente en tres canales de 15 ml de PBS fresco (30 s cada uno).
- Deje secar los microarrays en la oscuridad y se almacenan a 4 ° C en una caja de diapositivas. Los microarrays se pueden almacenar en la oscuridad durante un máximo de 3 meses sin pérdida de fluorescencia.
- Busque en la micromatriz seco con un tifón FLA 9000 escáner (GE Healthcare, Rydalmere, NSW, Australia) con la resolución ajustada a 50 (532 nm láser, 580 BP30 emisión de filtro para el PE. 633 nm láser y 670 BP30 emisión de filtro para Alexa 647). Los microarrays son escaneados con el lado de nitrocelulosa hacia abajo en la bandeja de cristal del escáner.
- Guardar las imágenes fluorescentes como archivos TIFF y Tamaño de la imagen de conjunto de Photoshop 17 x 25 cm y una resolución de 72 píxeles / cm. Importar la imagen en el software de análisis DotScan para analizar la intensidad de los puntos.
- El DotReader captura una imagen digital del patrón de unión de puntos y cuantifica la densidad de la unión de células en cada punto de anticuerpos en una escala de gris de 8 bits (1-256 U). Vez en cuando la unión no específica de control de isotipo se restará de los valores de unión de los anticuerpos con los isotipos de inmunoglobulinas correspondientes. La intensidad de fluorescencia de puntos para cada uno de microarrays se normalizaron en contra de la más brillante punto fijado en la intensidad del 100%. Señal / spot fuerza fue grabado en un archivo. Xml (datos brutos) o representado como un gráfico de barras en un archivo. Pdf (informe final)
- Microarrays heatsmaps y la agrupación jerárquica se realizaron utilizando Multiexperiment Viewer (MeV) la versión 4.4 de la suite TM4 Microarrays Software ( http://www.tm4.org/mev.html ). Agrupación jerárquica se realizó en los antecedentes-ajustado por los MeV con el análisis de ligamiento completo. Distancia euclídea fue utilizado para medir la similitud. El estudiante de 2 colas de la t-test con una variación igual se utiliza para determinar la significación estadística de los resultados.
5. Los resultados representativos:
Los resultados de los microarrays DotScan debe mostrar patrones consistentes de células de unión entre los conjuntos de duplicados. Fuerte alineación de puntos de unión (CD44/CD29) permite a una red que se coloca sobre el área de la matriz. La figura 2 muestra un ejemplo de captura óptima de la célula y la multiplexación. La Figura 3 muestra algunos problemas comunes encontrados durante la captura de células y las posibles soluciones.
Los resultados de microarrays de células de unión se puede cuantificar mediante la medición de la intensidad del punto expresado en una escala de gris que van de 1 a 256. La Figura 4 muestra los datos numéricos a partir de 58 muestras quirúrgicas CRC, se tiñeron con EpCAM-Alexa 647 anticuerpo, como un mapa de calor con la agrupación jerárquica. A pesar de que el número de muestras es limitado, CRC de la misma etapa tienden a agruparse en el mismo grupo.
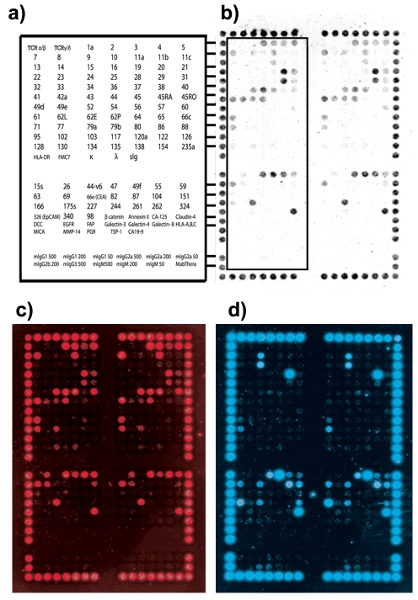
Figura 2. Patrón de unión de células tumorales clínicos del cáncer colorrectal (Australia Clínica Patológica Andamios, ACP escenario B1). (A) DotScan clave de anticuerpos con la ubicación de los anticuerpos de la mitad izquierda de la micromatriz duplicado (contorno). La sección superior contiene el original de 82 anticuerpos de los microarrays de la leucemia DotScan. Un adicional de 40 anticuerpos, lo que corresponde a los antígenos de superficie específica resultó ser regulado hasta en la literatura, se han añadido como los microarrays de "satélite" de un CRC. La sección inferior se compone de anticuerpos isotipo de control (b) la imagen óptica de las células CRC vinculante para los microarrays. (C) CD3 imagen fluorescente que muestra células-T. (D) EpCAM fluorescencia imagen que muestra las células CRC.

Figura 3. Ejemplos de los pobres resultados DotScan y sus posibles soluciones. (A) de células de baja obligatoria; solución: asegúrese de que al menos 4x106 células viables se encuentran en la matriz (b) células control de isotipo vinculantes y no vinculantes específicos; solución: añadir inactivado por calor suero humano AB a la muestra antes de la incubación de microarrays para reducir al mínimo isotipo de control obligatorio. De vez en cuando, una pequeña cantidad de la unión no específica de las células de la nitrocelulosa que ocurre con las muestras de CRC y no afectan significativamente los resultados. (C) La nitrocelulosa se seque durante la incubación, la solución: asegurar la muestra cubre toda la sección de nitrocelulosa y microarrays se incuba en una superficie plana. (D) los artefactos de alta de fondo, solución: asegurar lamicroarrays se lava bien después de incubación.
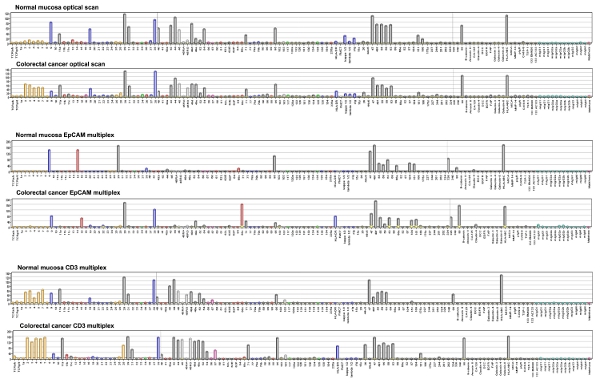
Figura 4. DotScan software de análisis genera gráficos de barras que representan las densidades de células de unión en una escala de gris que van de 1 a 256. Los números del eje se refieren a los antígenos CD. Otras abreviaturas han sido TCR, receptor de células T, κ, λ, cadenas ligeras de inmunoglobulinas; sIg, inmunoglobulina de superficie, DCC, eliminado en la proteína del cáncer colorrectal, el EGFR, receptor del factor de crecimiento epidérmico; FAP, la proteína de activación de fibroblastos, HLA-A, B, C HLA-DR, DR antígenos de leucocitos humanos y A, B y C, respectivamente, MICA, MHC de clase I de la cadena relacionados con la proteína A, MMP-14, metalopeptidasa matriz de 14; pIgR, el receptor de inmunoglobulina polimérica; TSP-1, trombospondina-1; Mabthera, anti-CD20 humanizado. Haga clic aquí para ampliar la imagen .
Discusión
En este video, que demuestran cómo los microarrays de anticuerpos DotScan se puede utilizar en una forma simple, semi-cuantitativo de manera de estudiar los perfiles de antígeno de superficie de las poblaciones de células del tejido CRC.
La obtención de una suspensión de células viables solo a partir de tejido es esencial para el éxito del experimento, ya que depende de la energía los procesos (por ejemplo, el antígeno de recubrimiento y / o la formación de pseudópodos) parecen se...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Damos las gracias al personal del Anatómico laboratorios de patología de la Royal Prince Alfred y Hospitales de Concordia repatriación de recogida de muestras frescas de la CRC y la mucosa intestinal normal. El trabajo fue financiado por una beca del Instituto del Cáncer de Nueva Gales del Sur Programa traslacional.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo o equipo | Empresa | Número de catálogo | Comentarios |
|---|---|---|---|
| Solución salina balanceada de Hanks | Sigma-Aldrich | H6136-10X1L | Tamponado con Hepes 25 mM (Sigma # H3375) |
| Airpure gabinete de seguridad biológica de clase II | Westinghouse | 1687-2340/612 | |
| Hojas de bisturí | Livingstone | 090609 | Paquete de 100 |
| RPMI 1640 con 2 mM Hepes | Sigma-Aldrich | R4130-10X1L | |
| Colagenasa tipo 4 | Worthington | 4188 | |
| Desoxirribonucleasa 1 | Sigma-Aldrich | DN25-1G | |
| Terumo jeringa (10 ml) | Terumo | SS 10 L | Caja de 100 |
| Filcon filtro (200 m) | BD Biosciences | 340615 | |
| Filcon filtro (50 micras) | Filcon filtro (50 micras) | Filcon filtro (50 micras) Filcon filtro (50 micras) y 340.603 | |
| Suero fetal bovino | Gibco / Invitrogen | 10099-141 | |
| Centrífuga 5810 R | Eppendorf | 7017 | |
| Dimetilsulfóxido | Sigma-Aldrich | D2650 | |
| El azul tripán | Sigma-Aldrich | T8154 | |
| Hemocymeter Technocolor Neubar | Hirschmann | no está disponible | |
| Microscopio de luz | Nikon | Nikon TMS | |
| Tubos Cyrovial | Greiner Bio-One | 121278 | |
| Cryo contrainer congelación | Nalgene | 5100-0001 | |
| DotScan anticuerpos microarrays kit | Medsaic | no está disponible | |
| DotScan microarrays bandeja de lavado | Medsaic | no está disponible | |
| Kimwipes | Kimberly-Clark | 4103 | |
| Formaldehído 37% | Sigma-Aldrich | F1635-500ML | |
| DotReaderTM | Medsaic | no está disponible | |
| Albúmina de suero bovino | Sigma-Aldrich | A9418-10G | |
| Inactivado por calor suero AB 2% | Invitrogen | 34005100 | |
| Ficoeritrina conjugado CD3 | Beckman Coulter | ET386 | |
| AlexaFluor647 conjugado EpCAM | BioLegend | 324212 | |
| Tifón FLA 9000 | GE Healthcare | 28-9558-08 | 532 nm láser, 580 BP30 emisión de filtro para el PE. 633 nm láser y 670 BP30 emisión de filtro para Alexa647 |
| Multiexperiment Visor v4.4 | TM4 Microarrays Software Suite | Abierto - el software de código (Ref 11) |
Referencias
- Steinert, R., Buschmann, T., vander Linden, M., Fels, L. M., Lippert, H., Reymond, M. A. The role of proteomics in the diagnosis and outcome prediction in colorectal cancer. Technol. Cancer. Res. Treat. 1, 297 (2002).
- Eifel, P., Axelson, J. A., Costa, J., Crowley, J., Curran, W. J., Deshler, A., Fulton, S., Hendricks, C. B., Kemeny, M., Kornblith, A. B., Louis, T. A., Markman, M., Mayer, R., Roter, D. National Institutes of Health Consensus Development Conference Statement: adjuvant therapy for breast cancer. J. Natl. Canc. Inst. 93, 979 (2001).
- Swerdlow, S. H., Campo, E., Harris, H. L., Jaffe, E. S., Pileri, S. A., Stein, H., Thiele, J., Vardiman, J. W. WHO classification of tumour of haematopoietic and lymphoid tissues. IARC WHO Classification of Tumours. 2, (2008).
- Xiao, G. G., Recker, R. R., Deng, H. W. Recent advances in proteomics and cancer biomarker discovery. Clin. Med. Oncol. , (2008).
- Belov, L., Mulligan, S. P., Barber, N., Woolfson, A., Scott, M., Stoner, K., Chrisp, J. S., Sewell, W. A., Bradstock, K. F., Bandall, L., Pascovici, D. S., Thomas, M., Erber, W., Huang, P., et al. Analysis of human leukaemias and lymphomas using extensive immunophenotypes from an antibody microarray. Br. J. Haematol. 135, 184 (2006).
- Belov, L., Huang, P., Barber, N., Mulligan, S. P., Christopherson, R. I. Identification of repertories of surface antigens on leukemias using an antibody microarray. Proteomics. 3, 2147 (2003).
- Zhou, J., Belov, L., Huang, P. Y., Shin, J., Solomon, M. J., Chapuis, P. H., Bokey, L., Chan, C., Clarke, C., Clarke, S. J., Christopherson, R. I. Surface antigen profiling of colorectal cancer using antibody microarrays with fluorescence multiplexing. J. Immunol. Methods. 355 (1-2), 40-51 (2010).
- Ellmark, P., Belov, L., Huang, P., Lee, C. S., Solomon, M. J., Morgan, D. K., Christopherson, R. I. Multiplex detection of surface molecules on colorectal cancers. Proteomics. 6, 1791 (2006).
- Pearson, J. P., Allen, A., Hutton, D. A. Rheology of mucin. Methods Mol. Biol. 125, 99 (2000).
- Yang, Y. H., Dudoit, S., Luu, P., Lin, D. M., Peng, V., Ngai, J., Speed, T. P. Normalization for cDNA microarray data: a robust composite method addressing single and multiple slide systematic variation. Nucleic Acids Res. 30, 15 (2002).
- Al Saeed, ., et al. TMA: A free, open-source system for microarray data management and analysis. BioTechniques. 34, 374-378 (2003).
Erratum
Formal Correction: Erratum: Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing
Posted by JoVE Editors on 7/03/2015. Citeable Link.
The author's email has been corrected in the publication of Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing. There was an error with the author, Jerry Zhou's, email. The author's email has been updated to:
j.zhou@uws.edu.au
from:
jzho7551@mail.usyd.edu.au
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados