É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Câncer Colorretal Profiling proteína de superfície celular Usando um Microarray de anticorpos e Multiplexação de fluorescência
Neste Artigo
Erratum Notice
Resumo
Nós descrevemos um procedimento para a desagregação de câncer colorretal (CRC) para produzir células viáveis única, que são, então, capturado em microarrays de anticorpos personalizado reconhecer antígenos de superfície (CRC DotScan microarray). Sub-populações de células vinculada ao microarray pode ser perfilado por multiplexação de fluorescência usando anticorpos monoclonais marcados com corantes fluorescentes.
Resumo
O prognóstico atual e classificação do CRC conta com encenação de sistemas que integram achados histopatológicos e clínicos. No entanto, na maioria dos casos CRC, disfunção das células é o resultado de numerosas mutações que modificam a expressão da proteína e pós-translacionais modificação 1.
Um número de antígenos de superfície celular, incluindo cluster de diferenciação (CD) de antígenos, foram identificados como prognósticos ou potenciais biomarcadores metastático em CRC. Estes antígenos fazer biomarcadores ideal como sua expressão muitas vezes muda com a progressão do tumor ou interações com outros tipos de células, como linfócitos tumor infiltrantes (TILs) e tumor associado macrófagos (TAMs).
O uso da imuno-histoquímica (IHQ) para o câncer de sub-classificação e prognóstico é bem estabelecida para alguns tipos de tumor 2,3. No entanto, não 'marcador' single mostrou significado prognóstico maior do que clínico-patológica de teste ou ganhou ampla aceitação para uso em relatórios de patologia de rotina de todos os casos CRC.
Uma abordagem mais recente para a estratificação prognóstica de fenótipos da doença depende de perfis de proteínas de superfície usando vários "marcadores". Enquanto perfil de expressão de tumores através de técnicas proteômicas como iTRAQ é uma poderosa ferramenta para a descoberta de biomarkers4, não é ideal para uso rotineiro nos laboratórios de diagnóstico e não pode distinguir diferentes tipos de células em uma população mista. Além disso, grandes quantidades de tecido tumoral são necessários para a caracterização de glicoproteínas da membrana plasmática purificada por estes métodos.
Neste vídeo nós descrevemos um método simples para criar perfis de superfície proteoma de células viáveis a partir de amostras desagregadas CRC usando um anticorpo DotScan microarray CRC. O microarray 122-anticorpo consiste em uma região de anticorpos 82-standard reconhecendo uma série de marcadores de linhagem específica de leucócitos, moléculas de adesão, receptores e marcadores de inflamação e resposta imune 5, bem como uma região satélite para a detecção de 40 marcadores potencialmente prognóstico para CRC . Células são capturadas apenas em anticorpos para os quais eles expressam o antígeno correspondente. A densidade de células por ponto, determinado pelo escaneamento óptico, reflete a proporção de células que expressam esse antígeno, o nível de expressão do antígeno e afinidade do anticorpo 6.
CRC de tecido ou mucosa intestinal normal, scans óptica refletem o imunofenótipo de populações mistas de células. Multiplexação de fluorescência pode então ser usada para perfil selecionado sub-populações de células de interesse capturado no array. Por exemplo, Alexa molécula de adesão 647-anti-células epiteliais (EpCAM; CD326), é um antígeno de diferenciação pan-epiteliais que foi usado para detectar células CRC e também células epiteliais da mucosa intestinal normal, enquanto Ficoeritrina-anti-CD3, foi usado para detectar infiltração de células T-7. O CRC DotScan microarray deve ser o protótipo de uma alternativa de diagnóstico para o sistema de estadiamento baseado anatomicamente CRC.
Protocolo
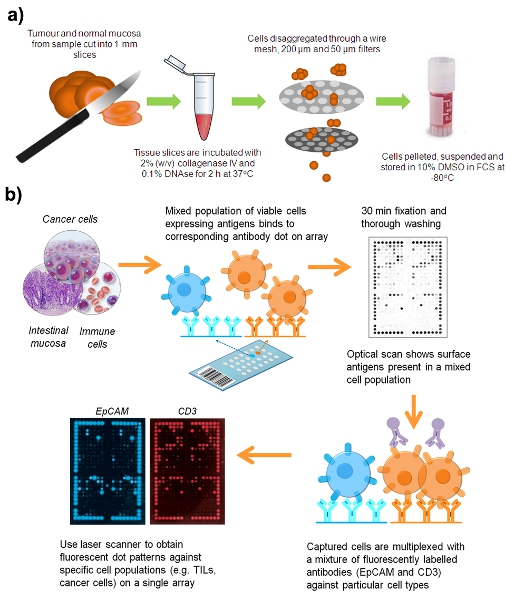
O fluxo de trabalho figura 1. Para a preparação de uma suspensão de células vivas a partir de uma amostra cirúrgica do CRC.
1. Desagregação amostra clínica
Todas as amostras foram coletadas do Príncipe Real Alfred Hospital (Camperdown, NSW, Austrália) e Hospital Repatriação Concord (Concord West, NSW, Austrália) com o consentimento informado nos termos do Protocolo n º X08-164.
- Coletar câncer colorretal fresco (CRC) ou espécimes adenoma, e mucosa intestinal normal, pelo menos, 10 cm do tumor. Armazene as amostras em pH equilibrado Hank solução salina 7,3 (HBSS) a 4 ° C por até 12 h após a ressecção.
- Siga as normas de segurança para patógenos humanos, processo de todas as amostras clínicas em um gabinete de segurança biológica classe II. Dissecar as amostras em dois cubos mm em uma placa de Petri usando duas lâminas de bisturi.
- Incubar tumor e tecido normal em tubos separados Eppendorf com ocasionais mistura suave por 60 min a 37 ° C com um volume igual de meio RPMI 1640 contendo 2% (v / v) colagenase tipo 4 (Worthington, Lakewood, NJ, EUA) e 0,1 % (w / v) desoxirribonuclease I de pancrease bovina (DNAse I; Sigma-Aldrich).
- Força semi-digerida tecido através de uma peneira fina malha de arame usando um êmbolo de uma seringa de 10 mL, as células através de lavagem com HBSS.
- Passe resultando suspensão celular através de 200 mm e 50 filtros Filcon mM (BD Biosciences) para remover agregados celulares. A maioria dos agregados muco DNA, células e são removidos nesta série de filtrações.
- Centrífuga suspensões de células a 400 xg a 20 ° por 5 min.
- Pellets celulares ressuspender em inativado pelo calor FCS contendo 10% de dimetil sulfóxido (DMSO), congelar lentamente em criotubos e armazenar a -80 °. O processo de congelamento tende a reduzir o muco na amostra e lise de células vermelhas do sangue.
2. Preparação de amostras para a captura de células
- Descongelar as amostras rapidamente em banho-maria 37 ° e células ressuspender em 10 ml de HBSS para lavar o DMSO.
- Centrífuga suspensões de células a 410 xg a 20 ° por 5 min.
- Decantar o sobrenadante e ressuspender o pellet celular em 500 mL de HBSS.
- Tratar a amostra com 0,1% (w / v) DNAse I por 20 min em temperatura ambiente.
- Misturar 10 mL de cada suspensão de células com um volume igual de azul de tripano e carga de 10 L da mistura em um hemocitômetro. Usando um microscópio de luz em 100 vezes de ampliação, a contagem de células viáveis, que aparecem claros devido à exclusão de tripan azul, enquanto as células mortas assumir o corante. Um mínimo de 4 x 10 6 células viáveis é necessária para a captura de células na microarray.
- Após o tratamento DNAse, ressuspender a suspensão de células em 10 mL HBSS e centrifugar a 410 xg a 20 ° por 5 min.
- Decantar o sobrenadante e ressuspender pellets de células em RPMI 1640 para um volume final de 200 mL.
3. Anticorpo de captura de células microarray
- Umedecer o microarray de anticorpos DotScan por imersão a seção de nitrocelulose em tampão fosfato salino (PBS) por aproximadamente 20 s. Limpe cuidadosamente as bordas de vidro do microarray com Kimwipes dobrado, evitando tocar a seção de nitrocelulose.
- Adicionar água à bandeja de incubação microarray para fornecer uma câmara úmida. Coloque o microarray para a câmara e pipetar a suspensão de células em RPMI 1640 para a seção de nitrocelulose úmida. Pipeta gotas em cada canto da nitrocelulose para assegurar uma repartição equilibrada das células.
- Incubar os microarrays, a 37 ° C por 1 h. A incubação possibilita que as células para resolver e entrar em contato com os anticorpos no microarray. Células que expressam antígenos de superfície correspondentes aos anticorpos que pousar em será capturada.
- Após a incubação, mergulhe a microarrays delicadamente e verticalmente em três calhas contendo pelo menos 15 mL PBS para lavar as células não ligado (20 s por lavagem).
- Prepare 3,7% (w / v) de formaldeído em PBS para consertar as células e anticorpos pelo cross-linking. Pipeta suavemente cerca de 1 mL para cobrir a seção de nitrocelulose do microarray. Incubar por 20 min em temperatura ambiente.
- Em seguida mergulhe microarrays em 3 trocas de PBS (15 mL; 30 s cada) para lavar o excesso de formol.
- Limpe as bordas e parte de trás da lâmina de vidro com Kimwipes e digitalizar o microarray usando o scanner DotScan enquanto, a seção de nitrocelulose é úmido. A digitalização óptica fornece o padrão de expressão de antígenos de uma mistura de células população células por exemplo, CRC, leucócitos e outras células do estroma do tumor.
4. Fluorescência de multiplexação
- Remover o microarray do scanner e aplicar 200 mL de tampão de bloqueio (2% w / v BSA, 2% inativado pelo calor humano AB soro, PBS, pH 7.3). Incube-o na bandeja microarray em temperatura ambiente por 20 min.
- Prepare o multiplexing solução em um tubo Eppendorf coberto com folha de alumínio: 20 mL Ficoeritrina-anti-CD3 (Beckman Coulter, Gladesville, NSW, Austrália, # IM12824; 1/7.5 diluição final), 10 mL Alexa Fluor 647-anti-EpCAM (Biolegend, San Diego, CA, EUA; 15/01 diluição), 2 mL da inativado pelo calor humano soro AB (Sigma-Aldrich, Castle Hill, NSW, Austrália) e 118 mL de tampão de bloqueio.
- Drenar o excesso de tampão de bloqueio do microarray e pipetar a solução de multiplexação para a seção de nitrocelulose, espalhando de maneira uniforme. Incubar por 30 min no escuro à temperatura ambiente.
- Mergulhe o microarray verticalmente em três bebedouros de 15 ml PBS fresco; (30 s cada).
- Deixe o microarrays seca no escuro e armazenar a 4 ° C em uma caixa de slides. O microarray pode ser armazenado no escuro por até 3 meses sem perda de fluorescência.
- Digitalizar o microarray seco usando um scanner Typhoon FLA 9000 (GE Healthcare, Rydalmere, NSW, Austrália) com a resolução definida para 50 (532 nm laser, 580 BP30 emissão filtro para PE. Laser de 633 nm e 670 BP30 emissão filtro para Alexa 647). Os microarrays são verificados com o lado de nitrocelulose virada para baixo na bandeja de vidro do scanner.
- Guardar as imagens fluorescentes como arquivos TIFF e usando Photoshop tamanho de imagem ajustado para 17 x 25 cm e resolução de 72 pixels / cm. Importar a imagem para o software de análise DotScan para analisar a intensidade de pontos.
- O DotReader capta uma imagem digital do padrão de pontos de ligação e quantifica a densidade de ligação de célula em cada ponto de anticorpos em escala cinza de 8 bits (1-256 U). Ocasional ligação não específica de controle isotipo foi subtraído dos valores de ligação para os anticorpos com isotipos de imunoglobulina correspondente. Intensidades de fluorescência de pontos para cada microarray foram normalizados contra os mais brilhantes dot fixada em 100% de intensidade. Sinal / spot força foi gravada em um arquivo xml. (Dados brutos) ou representado como um gráfico de barras em um arquivo. Pdf (relatório final)
- Microarray heatsmaps e agrupamento hierárquico foram realizados utilizando MultiExperiment Visualizador de versão (MeV) 4.4 do TM4 Microarray Software Suite ( http://www.tm4.org/mev.html ). Agrupamento hierárquico foi realizada sobre dados de fundo ajustado usando MeV com análise de ligação completa. Distância euclidiana foi utilizada para a medida de similaridade. O Estudante 2-tailed t-teste com variância igual foi usada para determinar a significância estatística dos resultados.
5. Resultados representativos:
Os resultados do microarray DotScan deve mostrar padrões consistentes de células de ligação entre matrizes duplicado. Forte alinhamento de pontos de ligação (CD44/CD29) permite uma grade para ser colocado sobre a área da matriz. A Figura 2 mostra um exemplo de captura de célula ideal e multiplexação. A Figura 3 mostra alguns problemas comuns encontrados durante a captura de células e as possíveis soluções.
Os resultados das células microarray ligação pode ser quantificado através da medição intensidades dot expressa numa escala de cinza que varia de 1 a 256. A Figura 4 mostra dados numéricos de 58 peças cirúrgicas CRC, corados com anticorpo Alexa EpCAM-647, como um heatmap com agrupamento hierárquico. Mesmo que o número de amostras é limitada, CRCs do mesmo estágio tendem a se agrupar em um mesmo grupo.
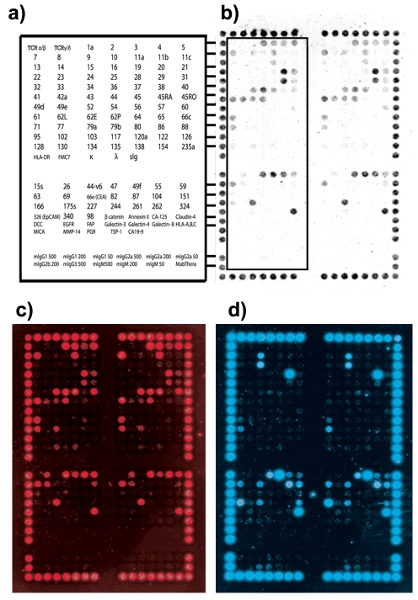
Figura 2. Padrão de ligação de celular do tumor do câncer colorretal clínicas (estadiamento clínico-patológico da Austrália, ACP estágio B1). (A) chave de anticorpos DotScan mostrando a localização de anticorpos para a metade esquerda do microarray duplicados (delineado). A seção superior contém o original 82 anticorpos do microarray DotScan leucemia. Um adicional de 40 anticorpos, correspondentes a antígenos de superfície específicos encontrados para ser sobre-regulada na literatura, foram adicionados como microarray 'satélite' a CRC. A seção inferior consiste de anticorpos controle isotipo (b) imagem óptico de células CRC ligação ao microarray. (C) imagem da fluorescência CD3 mostrando células-T. (D) EpCAM imagem de fluorescência mostrando células CRC.

Figura 3. Exemplos de maus resultados DotScan e possíveis soluções. (A) célula de baixa taxa de ligação; solução: certifique-se, pelo menos, 4x106 células viáveis sobre a matriz (b) Isotype célula de controle de ligação e ligação não específica; solução: adicionar inativado pelo calor humano AB soro a amostra antes da incubação de microarray para minimizar isotipo controle de ligação. Ocasionalmente, uma pequena quantidade de ligação não específica de células para a nitrocelulose ocorre com amostras de CRC e não afetar significativamente os resultados. (C) nitrocelulose secar durante a incubação; solução: garantir a amostra abrange a seção de nitrocelulose todo e microarray é incubada em uma superfície plana. (D) artefatos de fundo de alta; solução: garantir amicroarray é cuidadosamente lavados de incubação seguinte.
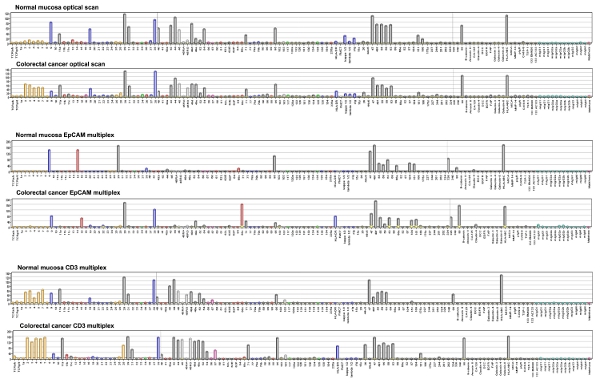
Figura 4. DotScan software de análise gerados gráficos de barras que representam as densidades de células de ligação em uma escala de cinza que varia de 1 a 256. Números no eixo referem-se aos antígenos CD. Outras abreviações são TCR, receptor de células T; κ, λ, as cadeias de imunoglobulina luz; sig, imunoglobulina de superfície; DCC, apagados em proteínas câncer colorretal; EGFR, receptor do fator de crescimento epidérmico; FAP, proteína de ativação de fibroblastos; HLA-A, B, C HLA-DR, DR antígenos leucocitários humanos e A, B, C, respectivamente; MICA, MHC classe I relacionadas à cadeia de proteína A; MMP-14, matriz metallopeptidase 14; PIGR, receptor de imunoglobulina poliméricos; TSP-1, trombospondina-1; Mabthera, humanizado anti-CD20. Clique aqui para ver a imagem ampliada .
Discussão
Neste vídeo, demonstramos como o microarray de anticorpos DotScan pode ser usado em uma forma simples e semi-quantitativa para estudar perfis de antígeno de superfície para as populações de células do tecido CRC.
Obtenção de uma suspensão de células viáveis único a partir do tecido é fundamental para o sucesso do experimento, porque a energia dependente de processos (por exemplo, antígeno de nivelamento e / ou formação de pseudópodos) parecem ser necessárias para a li...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos a equipe do Laboratório de Anatomia Patológica do Príncipe Real Alfred e Hospitais Repatriação Concord para a recolha de amostras frescas de CRC e na mucosa intestinal normal. O trabalho foi financiado por um New South Wales Instituto do Câncer Grant Programa translacional.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente ou equipamento | Companhia | Número de catálogo | Comentários |
|---|---|---|---|
| Solução de Hanks salina balanceada | Sigma-Aldrich | H6136-10X1L | Tamponado com Hepes 25 mM (Sigma # H3375) |
| Airpure gabinete de segurança biológica classe II | Westinghouse | 1687-2340/612 | |
| Lâminas cirúrgicas | Livingstone | 090609 | Pacote de 100 |
| RPMI 1640 com 2 mM Hepes | Sigma-Aldrich | R4130-10X1L | |
| Colagenase tipo 4 | Worthington | 4188 | |
| Desoxirribonuclease 1 | Sigma-Aldrich | DN25-1G | |
| Terumo Seringa (10 mL) | Terumo | SS L 10 | Caixa de 100 |
| Filcon filtro (200 mm) | BD Biosciences | 340615 | |
| Filcon filtro (50 mm) | Filcon filtro (50 mm) | Filcon filtro (50 mm) Filcon filtro (50 mm) 340603 | |
| Soro fetal bovino | Gibco / Invitrogen | 10099-141 | |
| Centrífuga 5810 R | Eppendorf | 7017 | |
| Dimetilsulfóxido | Sigma-Aldrich | D2650 | |
| Azul de tripano | Sigma-Aldrich | T8154 | |
| Hemocymeter Technocolor Neubar | Hirschmann | não disponível | |
| Microscópio de luz | Nikon | Nikon TMS | |
| Tubos Cyrovial | Greiner bio-one | 121278 | |
| Cryo contrainer congelamento | Nalgene | 5100-0001 | |
| DotScan anticorpos microarray kit | Medsaic | não disponível | |
| DotScan bandeja de lavar microarray | Medsaic | não disponível | |
| KimWipes | Kimberly-Clark | 4103 | |
| Formol 37% | Sigma-Aldrich | F1635-500ML | |
| DotReaderTM | Medsaic | não disponível | |
| Albumina de soro bovino | Sigma-Aldrich | A9418-10G | |
| Inativado pelo calor soro AB 2% | Invitrogen | 34005100 | |
| Ficoeritrina conjugada CD3 | Beckman Coulter | ET386 | |
| AlexaFluor647 conjugada EpCAM | BioLegend | 324212 | |
| Typhoon FLA 9000 | GE Healthcare | 28-9558-08 | 532 nm do laser, 580 BP30 emissão filtro para PE. 633 nm do laser e 670 BP30 emissão filtro para Alexa647 |
| MultiExperiment Visualizador v4.4 | TM4 Microarray Software Suite | Open - software de código (Ref 11) |
Referências
- Steinert, R., Buschmann, T., vander Linden, M., Fels, L. M., Lippert, H., Reymond, M. A. The role of proteomics in the diagnosis and outcome prediction in colorectal cancer. Technol. Cancer. Res. Treat. 1, 297 (2002).
- Eifel, P., Axelson, J. A., Costa, J., Crowley, J., Curran, W. J., Deshler, A., Fulton, S., Hendricks, C. B., Kemeny, M., Kornblith, A. B., Louis, T. A., Markman, M., Mayer, R., Roter, D. National Institutes of Health Consensus Development Conference Statement: adjuvant therapy for breast cancer. J. Natl. Canc. Inst. 93, 979 (2001).
- Swerdlow, S. H., Campo, E., Harris, H. L., Jaffe, E. S., Pileri, S. A., Stein, H., Thiele, J., Vardiman, J. W. WHO classification of tumour of haematopoietic and lymphoid tissues. IARC WHO Classification of Tumours. 2, (2008).
- Xiao, G. G., Recker, R. R., Deng, H. W. Recent advances in proteomics and cancer biomarker discovery. Clin. Med. Oncol. , (2008).
- Belov, L., Mulligan, S. P., Barber, N., Woolfson, A., Scott, M., Stoner, K., Chrisp, J. S., Sewell, W. A., Bradstock, K. F., Bandall, L., Pascovici, D. S., Thomas, M., Erber, W., Huang, P., et al. Analysis of human leukaemias and lymphomas using extensive immunophenotypes from an antibody microarray. Br. J. Haematol. 135, 184 (2006).
- Belov, L., Huang, P., Barber, N., Mulligan, S. P., Christopherson, R. I. Identification of repertories of surface antigens on leukemias using an antibody microarray. Proteomics. 3, 2147 (2003).
- Zhou, J., Belov, L., Huang, P. Y., Shin, J., Solomon, M. J., Chapuis, P. H., Bokey, L., Chan, C., Clarke, C., Clarke, S. J., Christopherson, R. I. Surface antigen profiling of colorectal cancer using antibody microarrays with fluorescence multiplexing. J. Immunol. Methods. 355 (1-2), 40-51 (2010).
- Ellmark, P., Belov, L., Huang, P., Lee, C. S., Solomon, M. J., Morgan, D. K., Christopherson, R. I. Multiplex detection of surface molecules on colorectal cancers. Proteomics. 6, 1791 (2006).
- Pearson, J. P., Allen, A., Hutton, D. A. Rheology of mucin. Methods Mol. Biol. 125, 99 (2000).
- Yang, Y. H., Dudoit, S., Luu, P., Lin, D. M., Peng, V., Ngai, J., Speed, T. P. Normalization for cDNA microarray data: a robust composite method addressing single and multiple slide systematic variation. Nucleic Acids Res. 30, 15 (2002).
- Al Saeed, ., et al. TMA: A free, open-source system for microarray data management and analysis. BioTechniques. 34, 374-378 (2003).
Erratum
Formal Correction: Erratum: Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing
Posted by JoVE Editors on 7/03/2015. Citeable Link.
The author's email has been corrected in the publication of Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing. There was an error with the author, Jerry Zhou's, email. The author's email has been updated to:
j.zhou@uws.edu.au
from:
jzho7551@mail.usyd.edu.au
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados