Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Darmkrebs Zelloberflächenprotein Profiling mit einem Antikörper Microarray und Fluoreszenz Multiplexing
In diesem Artikel
Erratum Notice
Zusammenfassung
Wir beschrieben ein Verfahren für die Zerlegung von Darmkrebs (CRC), lebensfähige einzelne Zellen, die dann auf kundenspezifische Antikörper Microarrays erkennen Oberflächen-Antigene (DotScan CRC-Microarray) erfasst zu produzieren. Sub-Populationen von Zellen gebunden, die Microarray kann durch Fluoreszenz-Multiplexing mit monoklonalen Antikörpern mit Fluoreszenzfarbstoffen markiert profiliert werden.
Zusammenfassung
Die aktuelle Prognose und Klassifikation von CRC basiert auf Staging-Systeme, die histopathologischen und klinischen Befunden zu integrieren. Doch in der Mehrzahl der CRC Fällen ist Zelldysfunktion das Ergebnis zahlreicher Mutationen, die Protein-Expression und posttranslationale Modifikation 1 ändern.
Eine Reihe von Zell-Oberflächen-Antigene, einschließlich Cluster der Differenzierung (CD)-Antigene wurden als potenzielle prognostische oder metastasiertem Biomarker in CRC identifiziert worden. Diese Antigene bilden ideale Biomarker als ihren Ausdruck oft ändert sich mit Tumorprogression oder Wechselwirkungen mit anderen Zelltypen, wie zB Tumor-infiltrierenden Lymphozyten (TIL) und Tumor-assoziierte Makrophagen (TAM).
Die Verwendung von Immunhistochemie (IHC) für Krebs sub-Klassifikation und Prognose ist gut für einige Tumorarten 2,3 etabliert. Es wurde jedoch keine einzige "Marker" prognostische Signifikanz gezeigt größer als klinisch-pathologischen Staging-oder gewonnen breite Akzeptanz für den Einsatz in der Routine-Pathologie Meldung aller CRC Fällen.
Ein neuerer Ansatz, um prognostische Stratifizierung Krankheitsphänotypen setzt auf Oberflächenprotein Profile mit mehreren "Marker". Während Expression Profiling von Tumoren mit Proteom-Techniken wie iTRAQ ist ein leistungsfähiges Werkzeug für die Entdeckung der biomarkers4, ist es nicht optimal für den routinemäßigen Einsatz in diagnostischen Laboratorien und kann nicht unterscheiden, verschiedene Zelltypen in eine gemischte Bevölkerung. Darüber hinaus werden große Mengen von Tumorgewebe für die Profilierung des gereinigten Plasmamembran Glykoproteine durch diese Methoden erforderlich.
In diesem Video haben wir beschrieben, eine einfache Methode zur Oberfläche Proteom-Profiling von lebensfähigen Zellen aus aufgeschlüsselte CRC-Proben mit einem DotScan CRC Antikörper Microarray. Die 122-Antikörper Microarray besteht aus einem Standard-82-Antikörper Region die Anerkennung einer Reihe von Lineage-spezifische Leukozyten-Marker, Adhäsionsmoleküle, Rezeptoren und Marker der Entzündung und Immunantwort 5, zusammen mit einem Satelliten-Region für den Nachweis von 40 potentiell prognostischer Marker für CRC . Die Zellen sind nur auf Antikörper, für die sie ausdrücken, das entsprechende Antigen erfasst. Die Zelldichte pro Punkt, durch optische Abtastung ermittelt, spiegelt der Anteil der Zellen, die das Antigen, das Niveau der Expression des Antigens und der Affinität des Antikörpers 6.
Für CRC Gewebe oder normalen Darmschleimhaut, spiegeln optische Scans der Immunphänotyp von gemischten Populationen von Zellen. Fluoreszenz-Multiplexing können dann verwendet werden, um ausgewählte Sub-Populationen von Zellen von Interesse auf dem Array erfasst Profil. Zum Beispiel, Alexa 647-anti-Epithelial Cell Adhesion Molecule (EpCAM, CD326) ist ein pan-epitheliales Differenzierungsantigen, die zur CRC-Zellen und auch Epithelzellen der normalen Darmschleimhaut erkennen war, während Phycoerythrin-anti-CD3, wurde verwendet, zu erkennen infiltrierenden T-Zellen 7. Das DotScan CRC Microarray sollte der Prototyp für eine diagnostische Alternative zu den anatomisch-basierte CRC Staging-System sein.
Protokoll
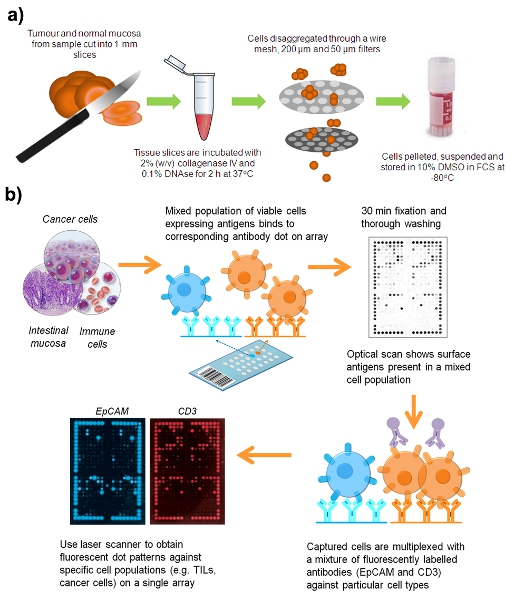
Abbildung 1. Arbeitsablauf für die Herstellung einer Suspension von lebenden Zellen aus einem chirurgischen Stichprobe von CRC.
1. Klinische Probe Disaggregation
Alle Proben wurden von der Royal Prince Alfred Hospital (Camperdown, NSW, Australien) und Concord Rückführung Hospital (Concord West, NSW, Australien) mit Einwilligung im Rahmen des Protokolls Nr. X08-164 gesammelt.
- Sammeln Sie frische Kolorektalkarzinom (CRC) oder Adenom Proben und normalen Darmschleimhaut mindestens 10 cm aus dem Tumor. Shop-Proben in balanced salt Hank-Lösung pH 7,3 (HBSS) bei 4 ° C für bis zu 12 h nach der Resektion.
- Follow Sicherheitsvorschriften für den menschlichen Krankheitserreger, verarbeiten alle klinischen Proben in einer biologischen Sicherheitswerkbank der Klasse II. Präparieren Sie die Proben in 2 mm Würfel in einer Petrischale mit zwei Skalpellklingen.
- Inkubieren Tumor-und Normalgewebe in separaten Eppendorf-Röhrchen mit gelegentlichen vorsichtig mischen und 60 min bei 37 ° C mit dem gleichen Volumen RPMI 1640-Medium mit 2% (v / v) Kollagenase Typ 4 (Worthington, Lakewood, NJ, USA) und 0,1 % (w / v) Desoxyribonuclease I aus Rinder-pancrease (DNAse I; Sigma-Aldrich).
- Force-Gewebe durch ein feines Drahtgeflecht Sieb mit einem Kolben aus einer 10 ml-Spritze halb verdaute; waschen Sie die Zellen durch mit HBSS.
- Pass resultierende Zellsuspension bis 200 um und 50 um Filcon Filter (BD Biosciences), um Zellaggregate zu entfernen. Die meisten der DNA, Schleim und Zell-Aggregate sind in dieser Reihe von Filtrationen entfernt.
- Centrifuge Zellsuspensionen bei 400 xg bei 20 ° für 5 min.
- Resuspendieren Zellpellets in hitzeinaktiviertem FCS mit 10% Dimethylsulfoxid (DMSO), langsam einfrieren in Kryoröhrchen und bei -80 °. Das Einfrieren Prozess neigt dazu, Schleim in der Probe zu reduzieren und Lyse roter Blutkörperchen.
2. Probenvorbereitung für die Zelle zu erfassen
- Auftauen Proben schnell in einem 37 ° Wasserbad und die Zellen in 10 ml HBSS zum Auswaschen der DMSO.
- Centrifuge Zellsuspensionen bei 410 xg bei 20 ° für 5 min.
- Den Überstand und Zellpellet in 500 ul HBSS.
- Behandeln Sie die Probe mit 0,1% (w / v) DNAse I für 20 min bei Raumtemperatur.
- Mix 10 ul jeder Zellsuspension mit dem gleichen Volumen Trypanblau und Last 10 ul der Mischung in einer Zählkammer. Mit einem Lichtmikroskop bei 100-facher Vergrößerung, count lebensfähigen Zellen, die deutlich durch Trypanblau-Ausschluss erscheinen, während tote Zellen nehmen den Farbstoff. Ein mindestens 4 x 10 6 lebensfähige Zellen ist für die Zelle zu erfassen auf dem Mikroarray erforderlich.
- Nach DNAse Behandlung, resuspendieren die Zellsuspension in 10 mL HBSS und Zentrifuge bei 410 xg bei 20 ° für 5 min.
- Dekantieren der Überstände und resuspendieren Zellpellets in RPMI 1640 auf ein Volumen von 200 ul.
3. Antikörper Microarray-Zelle zu erfassen
- Feuchten Sie die DotScan Antikörper Microarray durch Eintauchen der Nitrozellulose Abschnitt in Phosphat-gepufferter Kochsalzlösung (PBS) für etwa 20 s. Wischen Sie die Glaskanten des Mikroarrays mit gefalteten Kimwipes, Vermeidung Berühren Sie die Nitrozellulose Abschnitt.
- Wasser hinzu, bis die Microarray Inkubationswanne um eine feuchte Kammer stellen. Setzen Sie den Microarray in die Kammer und Pipette wurde die Zellsuspension in RPMI 1640 auf die feuchte Nitrocellulose Abschnitt. Pipette Tropfen an jeder Ecke der Nitrocellulose, um sicherzustellen, eine noch von Zellen verteilt.
- Inkubieren Sie die Microarrays bei 37 ° C für 1 h. Die Inkubationszeit kann Zellen, sich niederzulassen und in Kontakt kommen mit Antikörpern auf dem Microarray. Zellen, die Oberflächen-Antigene entsprechend der Antikörper landen sie auf aufgenommen wird.
- Nach der Inkubation, tauchen die Microarrays sanft und vertikal in drei Tröge mit mindestens 15 mL PBS abwaschen ungebundenen Zellen (20 s pro Waschgang).
- Bereiten Sie 3,7% (w / v) Formaldehyd in PBS zu den Zellen und Antikörper durch Vernetzung zu beheben. Gently Pipette etwa 1 mL der Nitrozellulose Abschnitt des Microarray-Abdeckung. Inkubieren für 20 min bei Raumtemperatur.
- Next dip-Microarrays in 3 Änderungen der PBS (15 ml, 30 s pro Stück) zum Auswaschen überschüssigen Formaldehyd.
- Wischen Sie die Kanten-und Rückseite der Glasplatte mit Kimwipes und scannen Sie die Microarray mit dem DotScan Scanner Zeit wird die Nitrocellulose Abschnitt feucht. Die optische Abtastung bietet das Antigen Expressionsmuster einer gemischten Zellpopulation zB CRC-Zellen, Leukozyten und andere Stromazellen des Tumors.
4. Fluoreszenz-Multiplexing
- Entfernen Sie die Microarray vom Scanner und anwenden 200 ul Blocking-Puffer (2% w / v BSA, 2% hitzeinaktiviertem humanem AB-Serum, PBS, pH 7,3). Inkubieren es in der Microarray-Fach bei Raumtemperatur für 20 min.
- Bereiten Sie die multiplexing Lösung in ein Eppendorf-Röhrchen mit Alufolie abgedeckt: 20 ul Phycoerythrin-anti-CD3 (Beckman Coulter, Gladesville, NSW, Australien, # IM12824; 1/7.5 Endverdünnung), 10 ul Alexa Fluor 647-anti-EpCAM (Biolegend, San Diego, CA, USA; 1 / 15 Verdünnung), 2 ul der hitzeinaktiviertem humanem AB-Serum (Sigma-Aldrich, Castle Hill, NSW, Australien) und 118 ul Blocking-Puffer.
- Ablassen über Blockierungspuffer aus dem Mikroarray und Pipette das Multiplexing-Lösung auf die Nitrozellulose Abschnitt Verbreitung gleichmäßig. Inkubieren für 30 min im Dunkeln bei Raumtemperatur.
- Tauchen Sie den Microarray vertikal in drei Mulden von 15 ml frischem PBS; (30 s pro Stück).
- Lassen Sie die Microarrays trocken im Dunkeln und bei 4 ° C in einer Diashow angezeigt. Die Microarray kann in der Dunkelheit für bis zu 3 Monaten ohne Verlust der Fluoreszenz gespeichert.
- Scan der trockenen Mikroarray mit einem Typhoon FLA 9000 Scanner (GE Healthcare, Rydalmere, NSW, Australien) mit einer Auflösung von 50 (532 nm Laser, 580 BP30 Emissionsfilter für PE. 633 nm-Laser und 670 BP30-Emission-Filter für Alexa 647). Die Mikroarrays werden mit der Nitrozellulose-Seite nach unten auf das Glas Scanner Fach gescannt.
- Speichern Sie die fluoreszierende Bilder im TIFF-Dateien und mit Photoshop gesetzt Bildgröße 17 x 25 cm und eine Auflösung von 72 Pixel / cm. Importieren Sie die Grafik in das DotScan Analyse-Software, um die Intensität von Punkten zu analysieren.
- Die DotReader fängt ein digitales Bild des Punktes Bindungsmuster und quantifiziert die Dichte der Zelle bindet jeder Antikörper Punkt auf einem 8-Bit Grau Skala (1-256 U). Gelegentliche unspezifische Isotyp-Kontroll-Bindung wurde die Bindung von Werten für Antikörper mit entsprechenden Immunglobulin-Isotypen abgezogen. Dot Fluoreszenzintensitäten für jeden Microarray wurden gegen den hellsten Punkt bei 100% Intensität normalisiert. Signal / spot Stärke wurde in einem. Xml-Datei (Rohdaten) aufgezeichnet oder dargestellt als Balkendiagramm in einer. Pdf-Datei (Abschlussbericht)
- Microarray heatsmaps und hierarchische Clustering wurden mit MultiExperiment Viewer (MeV) Version 4.4 aus dem TM4 Microarray Software Suite ( http://www.tm4.org/mev.html ). Hierarchical Clustering wurde am Hintergrund bereinigte Daten mit MeV mit komplettem Linkage-Analyse durchgeführt. Euklidische Distanz wurde für Ähnlichkeitsmaß verwendet. Die 2-tailed Student-t-Test mit gleicher Varianz wurde verwendet, um die statistische Signifikanz der Ergebnisse zu bestimmen.
5. Repräsentative Ergebnisse:
Ergebnisse aus dem DotScan Microarray soll zeigen konsistente Zelle Bindungsmuster zwischen doppelten Arrays. Starke Ausrichtung dot Bindung (CD44/CD29) ermöglicht ein Raster über das Array platziert werden. Abbildung 2 zeigt ein Beispiel für eine optimale Zell-Abscheidung und-Multiplexing. Abbildung 3 zeigt einige häufig auftretende Probleme während der Zellteilung erfassen und die möglichen Lösungen begegnet.
Die Microarray-Zell-Bindung Ergebnisse können durch die Messung dot Intensitäten auf einem grauen Skala von 1 bis 256 ausgedrückt quantifiziert werden. Abbildung 4 zeigt numerische Daten aus 58 chirurgischen CRC-Proben, mit EpCAM-Alexa 647 Antikörper gefärbt, als Heatmap mit hierarchischen Clustering. Auch wenn die Anzahl der Proben ist beschränkt, meist CRCs der gleichen Bühne, um die Cluster in der gleichen Gruppe.
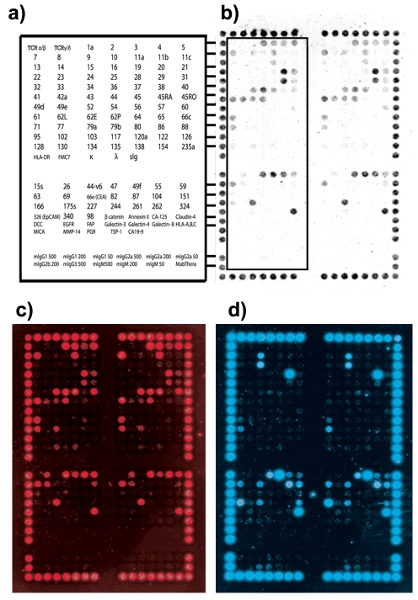
Abbildung 2. Zellbindung Muster der klinischen Darmkrebs Tumor (Australian Clinic-pathologische Staging, ACP Bühne B1). (A) DotScan Antikörper Schlüssel mit Standorten der Antikörper für die linke Hälfte der doppelten Microarray (umrissen). Der obere Abschnitt enthält den ursprünglichen 82-Antikörpern aus dem DotScan Leukämie-Mikroarray. Eine weitere 40-Antikörper, entsprechend spezifische Oberflächenantigene festgestellt, dass in der Literatur hoch reguliert, wurden als CRC "Satelliten" Microarray aufgenommen. Der untere Teil besteht aus Isotypkontrolle Antikörper (b) Optische Aufnahme CRC-Zellen die Bindung an den Microarray. (C) CD3 Abbildungsfunktion T-Zellen. (D) EpCAM Abbildungsfunktion CRC-Zellen.

Abbildung 3. Beispiele für schlechte DotScan Ergebnisse und mögliche Lösungen. (A) Niedrige Zellbindung; Lösung: Vergewissern Sie sich mindestens 4x106 lebensfähige Zellen sind auf dem Array (b) Isotypkontrolle verbindlich und unspezifische Zellbindung; Lösung: add hitzeinaktiviertem humanem AB-Serum zur Probe vor der Inkubation auf Mikroarray zu minimieren Isotypkontrolle bindend. Gelegentlich tritt eine kleine Menge an nicht-spezifische Bindung von Zellen an die Nitrozellulose nicht mit CRC-Proben und keinen signifikanten Einfluss auf die Ergebnisse. (C) Nitrocellulose Austrocknen während der Inkubation; Lösung: Sicherstellung der Probe deckt den gesamten Bereich Nitrocellulose und Microarray wird auf eine flache Oberfläche inkubiert. (D) High Hintergrund Artefakte; Lösung: Sicherstellung derMicroarray wird gründlich nach Inkubation gewaschen.
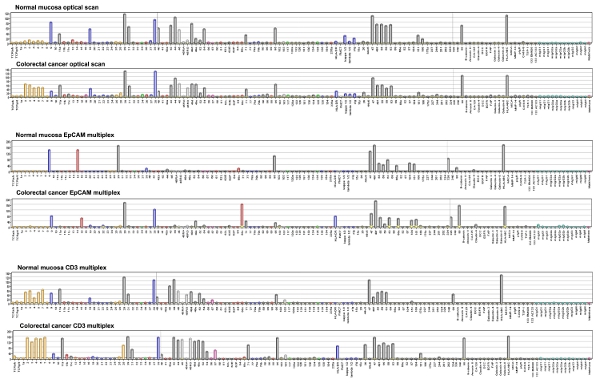
Abbildung 4. DotScan Analyse-Software generierten Balkendiagramme darstellen Zellbindung Dichten auf einem grauen Skala von 1 bis 256. Die Zahlen auf der Achse beziehen sich auf CD-Antigene. Andere Abkürzungen werden TCR, T-Zell-Rezeptor, κ, λ, Immunglobulin leichten Ketten; sIg, Oberflächen-Immunglobulin, DCC, in der Darmkrebs-Protein gestrichen EGFR, epidermal growth factor receptor, FAP, Fibroblasten-Aktivierungs-Protein, HLA-A, B, C HLA-DR, humane Leukozyten-Antigene DR und A, B, C nicht überschreiten; MICA, MHC-Klasse-I-Kette-related protein A; MMP-14, Matrix Metallopeptidase 14; pIgR, polymere Immunglobulin Rezeptor; TSP-1, Thrombospondin-1; Mabthera, humanisierter Anti-CD20. Klicken Sie hier für eine größere Ansicht .
Diskussion
In diesem Video zeigen wir, wie das DotScan Antikörper Microarray in einer einfachen, semi-quantitative Weg, um Oberflächen-Antigen-Profile für Zellpopulationen aus CRC Gewebe Studie verwendet werden kann.
Die Erteilung einer tragfähigen Einzel Zellsuspension aus Gewebe ist entscheidend für den Erfolg des Experiments, weil die Energie-abhängige Prozesse (zB, Antigen Begrenzung und / oder Pseudopodien-Bildung), für feste Bindung von ganzen Zellen zu Antikörper Punkte während der Inku...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Wir danken Mitarbeiter am Anatomischen Pathologie Laboratories des Royal Prince Alfred und Concord Rückführung Krankenhäuser zum Sammeln frischer Proben von CRC und normalen Darmschleimhaut. Die Arbeit wurde durch einen Cancer Institute New South Wales Translational-Zuschüssen finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagens oder Ausrüstung | Firma | Katalog-Nummer | Kommentare |
|---|---|---|---|
| Hanks 'balanced salt solution | Sigma-Aldrich | H6136-10X1L | Buffered mit 25 mM Hepes (Sigma # H3375) |
| Airpure biologischen Sicherheitswerkbank der Klasse II | Westinghouse | 1687-2340/612 | |
| Chirurgische Klingen | Livingstone | 090609 | Packung mit 100 Stück |
| RPMI 1640 mit 2 mM Hepes | Sigma-Aldrich | R4130-10X1L | |
| Collagenase Typ 4 | Worthington | 4188 | |
| Deoxyribonuclease 1 | Sigma-Aldrich | DN25-1G | |
| Terumo Spritze (10 ml) | Terumo | MS +10 L | Box von 100 |
| Filcon Filter (200 um) | BD Biosciences | 340615 | |
| Filcon Filter (50 um) | Filcon Filter (50 um) | Filcon Filter (50 um) Filcon Filter (50 um) 340603 | |
| Fötalem Kälberserum | Gibco / Invitrogen | 10099-141 | |
| Centrifuge 5810 R | Eppendorf | 7017 | |
| Dimethylsulfoxid | Sigma-Aldrich | D2650 | |
| Trypanblau | Sigma-Aldrich | T8154 | |
| Hemocymeter Technocolor Neubar | Hirschmann | nicht verfügbar | |
| Lichtmikroskop | Nikon | Nikon TMS | |
| Cyrovial Rohre | Greiner Bio-One | 121278 | |
| Cryo Einfrieren contrainer | Nalgene | 5100-0001 | |
| DotScan Antikörper Microarray-Kit | Medsaic | nicht verfügbar | |
| DotScan Microarray Waschküvette | Medsaic | nicht verfügbar | |
| Kimwipes | Kimberly-Clark | 4103 | |
| Formaldehyd 37% | Sigma-Aldrich | F1635-500ML | |
| DotReaderTM | Medsaic | nicht verfügbar | |
| Rinderserumalbumin | Sigma-Aldrich | A9418-10G | |
| Heat-inaktivierten AB-Serum 2% | Invitrogen | 34005100 | |
| Phycoerythrin-konjugierten CD3 | Beckman Coulter | ET386 | |
| AlexaFluor647-konjugierten EpCAM | BioLegend | 324212 | |
| Typhoon FLA 9000 | GE Healthcare | 28-9558-08 | 532 nm-Laser, 580 BP30 Emissionsfilter für PE. 633 nm-Laser und 670 BP30 Emissionsfilter für Alexa647 |
| MultiExperiment-Viewer v4.4 | TM4 Microarray Software Suite | Open - Source-Software (Ref 11) |
Referenzen
- Steinert, R., Buschmann, T., vander Linden, M., Fels, L. M., Lippert, H., Reymond, M. A. The role of proteomics in the diagnosis and outcome prediction in colorectal cancer. Technol. Cancer. Res. Treat. 1, 297 (2002).
- Eifel, P., Axelson, J. A., Costa, J., Crowley, J., Curran, W. J., Deshler, A., Fulton, S., Hendricks, C. B., Kemeny, M., Kornblith, A. B., Louis, T. A., Markman, M., Mayer, R., Roter, D. National Institutes of Health Consensus Development Conference Statement: adjuvant therapy for breast cancer. J. Natl. Canc. Inst. 93, 979 (2001).
- Swerdlow, S. H., Campo, E., Harris, H. L., Jaffe, E. S., Pileri, S. A., Stein, H., Thiele, J., Vardiman, J. W. WHO classification of tumour of haematopoietic and lymphoid tissues. IARC WHO Classification of Tumours. 2, (2008).
- Xiao, G. G., Recker, R. R., Deng, H. W. Recent advances in proteomics and cancer biomarker discovery. Clin. Med. Oncol. , (2008).
- Belov, L., Mulligan, S. P., Barber, N., Woolfson, A., Scott, M., Stoner, K., Chrisp, J. S., Sewell, W. A., Bradstock, K. F., Bandall, L., Pascovici, D. S., Thomas, M., Erber, W., Huang, P., et al. Analysis of human leukaemias and lymphomas using extensive immunophenotypes from an antibody microarray. Br. J. Haematol. 135, 184 (2006).
- Belov, L., Huang, P., Barber, N., Mulligan, S. P., Christopherson, R. I. Identification of repertories of surface antigens on leukemias using an antibody microarray. Proteomics. 3, 2147 (2003).
- Zhou, J., Belov, L., Huang, P. Y., Shin, J., Solomon, M. J., Chapuis, P. H., Bokey, L., Chan, C., Clarke, C., Clarke, S. J., Christopherson, R. I. Surface antigen profiling of colorectal cancer using antibody microarrays with fluorescence multiplexing. J. Immunol. Methods. 355 (1-2), 40-51 (2010).
- Ellmark, P., Belov, L., Huang, P., Lee, C. S., Solomon, M. J., Morgan, D. K., Christopherson, R. I. Multiplex detection of surface molecules on colorectal cancers. Proteomics. 6, 1791 (2006).
- Pearson, J. P., Allen, A., Hutton, D. A. Rheology of mucin. Methods Mol. Biol. 125, 99 (2000).
- Yang, Y. H., Dudoit, S., Luu, P., Lin, D. M., Peng, V., Ngai, J., Speed, T. P. Normalization for cDNA microarray data: a robust composite method addressing single and multiple slide systematic variation. Nucleic Acids Res. 30, 15 (2002).
- Al Saeed, ., et al. TMA: A free, open-source system for microarray data management and analysis. BioTechniques. 34, 374-378 (2003).
Erratum
Formal Correction: Erratum: Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing
Posted by JoVE Editors on 7/03/2015. Citeable Link.
The author's email has been corrected in the publication of Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing. There was an error with the author, Jerry Zhou's, email. The author's email has been updated to:
j.zhou@uws.edu.au
from:
jzho7551@mail.usyd.edu.au
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten