Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Kolorektal Kanser Hücre Yüzey Protein Profil Antikor Mikroarray ve Floresans Multiplexing
Bu Makalede
Erratum Notice
Özet
Kolorektal kanser (CRC) uygulanabilir tek bir hücre üretmek için, daha sonra yüzey antijenleri (DotScan ÇHS mikroarray) tanıyan özelleştirilmiş antikor mikrodizinlerinde yakalanır ayrıştırılmasıyla için bir prosedür nitelendirdi. Mikroarray bağlı hücrelerin alt popülasyonlarda floresan boya ile monoklonal antikorlar kullanılarak floresans çoklama profilli olabilir.
Özet
ÇHS geçerli prognoz ve sınıflandırma sistemleri evreleme histopatolojik ve klinik bulgular entegre dayanır. Ancak, ÇHS vakaların çoğunda protein ekspresyonu ve post-translasyonel değişiklik 1 değişiklik sayısız mutasyonlar, hücre disfonksiyonu sonucu.
CRC potansiyel prognostik veya metastatik biyobelirteçleri farklılaşmasını (CD) antijenlerinin küme de dahil olmak üzere hücre yüzey antijenleri, bir dizi olarak tespit edilmiştir. Bu antijenler genellikle kendi ifadesi olarak tümör progresyonu veya diğer hücre türleri gibi ile etkileşim değişiklikleri ideal biyobelirteçleri yapmak tümör infiltre eden lenfositler (TILs) ve tümör ilişkili makrofajlar (TAM).
Kanseri alt sınıflandırma ve prognoz tahmini için immünhistokimyasal (İHK) kullanımı bazı tümör tiplerinde 2,3 için kurulmuştur. Ancak, tek bir 'işaretleyici' den büyük klinik-patolojik prognostik önemi gösterilen veya rutin patoloji raporlama tüm CRC durumlarda kullanılmak üzere geniş bir kabul görmüştür.
Hastalık fenotipleri prognostik tabakalaşma bir daha yeni bir yaklaşım, birden çok 'işaretleyicileri kullanarak yüzey protein profilleri dayanmaktadır. ITRAQ olarak proteomik teknikler kullanarak tümörlerin ifade profil biomarkers4 keşfi için güçlü bir araç olmasına rağmen, teşhis laboratuvarlarında rutin kullanımı için en uygun değildir ve karışık bir nüfus farklı hücre tipleri ayırt edemez. Buna ek olarak, tümör dokusu büyük miktarda bu yöntemlerle saflaştırılmış plazma zarı glikoproteinler profil için gereklidir.
Bu video DotScan CRC antikor mikroarray kullanarak ayrıştırılmış CRC örneklerinden canlı hücreler yüzey proteom profil için basit bir yöntem tanımlanmıştır. 122-antikor mikroarray KRK için 40 potansiyel prognostik belirteçlerin tespiti için bir uydu bölge ile birlikte, bir dizi özel soyundan lökosit belirteçler, adezyon moleküllerinin, reseptörleri ve enflamasyon ve immün yanıt 5 belirteçleri tanıyan standart bir 82-antikor bölgesi oluşur . Hücreler, yalnızca ilgili antijen ifade antikorlar yakalanır. Nokta başına optik tarama tarafından belirlenen hücre yoğunluğu, bu antijen, antijen ve antikor 6 yakınlık ifade düzeyi ifade hücrelerinin oranını yansıtır.
CRC doku ya da normal bağırsak mukoza hücrelerinin karışık nüfusu, optik taramaları immünfenotip yansıtmaktadır. Floresan çoğullama sonra dizi yakalanan ilgi hücreleri seçilen alt popülasyonlarda profil kullanılabilir. Örneğin, Alexa 647 anti-epitel hücre adezyon molekülü (EpCAM CD326) Fikoeritrin-anti-CD3, kullanılmış iken, CRC hücreleri ve normal bağırsak mukozasında epitel hücreleri algılamak için kullanılan bir pan-epitelyal farklılaşma antijen T-hücrelerinin 7 infiltre algılamak için. DotScan CRC mikroarray anatomik tabanlı CRC evreleme sistemi için bir tanı alternatif prototip olmalıdır.
Protokol
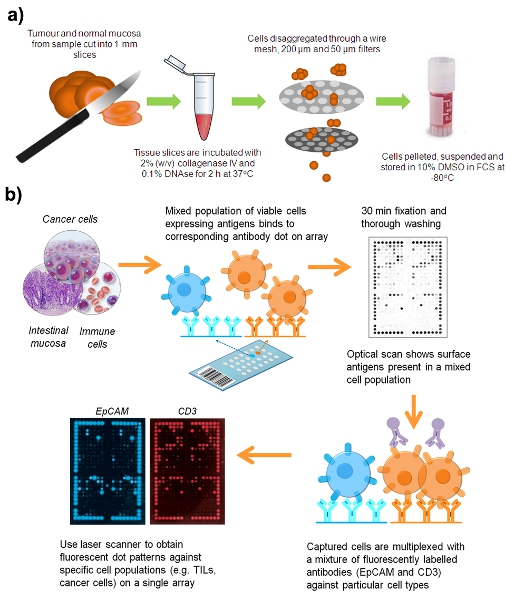
Şekil 1: CRC cerrahi örnek canlı hücreleri süspansiyon hazırlamak için İş akışı.
1. Klinik örnek ayrıştırılmasıyla
Tüm numuneler X08-164 No'lu Protokol'ün altında bilgilendirilmiş onam ile Royal Prince Alfred Hastanesi (Camperdown, NSW, Avustralya) ve Concord Repatriation Hastanesi (Concord Batı, NSW, Avustralya) toplanmıştır.
- Kolorektal kanser (CRC), taze veya adenom numuneler ve normal bağırsak mukozasında tümörün en az 10 cm toplayın. 4 Hank dengeli tuz solüsyonu pH 7,3 (HBSS) ° C kadar rezeksiyon sonrası 12 saat için saklayın örnekleri.
- Biyolojik güvenlik kabini sınıf II, tüm klinik örneklerde süreci, insan patojenler için emniyet kurallarına uyunuz. Iki neşter bıçak kullanarak bir Petri kabındaki 2 mm küp numuneler parçalara ayır.
- 37 az 60 dakika zaman zaman nazik karıştırma ayrı Eppendorf tüpleri tümör ve normal doku inkübe ° C,% 2 kollajenaz tip 4 (Worthington, Lakewood, New Jersey, ABD) ve 0.1 (v / v) içeren RPMI 1640 orta eşit hacimde sığır pankreas% (w / v) deoksiribonükleaz (DNAz, Sigma-Aldrich).
- Force 10 ml şırınga pistonu kullanarak, bir ince tel örgü süzgeç ile doku yarı sindirilmiş HBSS ile yıkama hücreleri yoluyla.
- Hücre agrega kaldırmak için 200 mikron ve 50 mikron Filcon filtreler (BD Biosciences) ile hücre süspansiyonu sonucu iletin. DNA, mukus ve hücre agrega çoğu sızmalarından bu dizi kaldırılır.
- Hücre süspansiyonları, 5 dakika boyunca 20 ° 400 xg'de santrifüjleyin.
- % 10 dimetil sulphoxide (DMSO) içeren ısı ile inaktive edilmiş FCS yeniden süspanse hücre pelletleri, -80 ° cryovials ve mağaza yavaş dondurma. Dondurma işlemi örnek mukus azaltmak eğilimindedir ve kırmızı kan hücreleri lyses.
2. Hücre yakalamak için örnek hazırlama
- 37 ° 10 ml DMSO yıkayın HBSS bir su banyosu ve tekrar süspansiyon hücreleri hızla örnekleri çözülme.
- 5 dakika boyunca 20 ° 410 xg'de hücre süspansiyonları santrifüjleyin.
- Süpernatantı Durusu ve HBSS 500 mcL hücre pelletini tekrar süspansiyon haline getirin.
- % 0,1 (w / v) oda sıcaklığında 20 dakika DNAz I ile örnek davranın.
- Tripan mavi eşit hacmi ile her bir hücre süspansiyonu 10 mcL karıştırın ve karışımı 10 mcL hemasitometre içine yüklemek. 100 kat büyütme bir ışık mikroskobu kullanarak ölü hücrelerden boya alırken, tripan mavi dışlanma nedeniyle net görünen canlı hücreleri, saymak. En az 4 x 10 6 canlı hücreler mikroarray hücre yakalamak için gereklidir.
- DNAz tedaviden sonra, 5 dakika boyunca 20 ° 410 xg'de 10 ml HBSS ve santrifüj hücre süspansiyonu tekrar süspansiyon haline getirin.
- RPMI 1640 süpernatantlar ve tekrar süspansiyon hücre pelletleri son hacim 200 mcL Durusu.
3. Antikor mikroarray hücre yakalama
- DotScan antikor mikroarray nitroselüloz bölümünde fosfat içine batırarak ıslatın yaklaşık 20 s için tamponlu salin (PBS) Dikkatle nitroselüloz bölümünde dokunmaktan kaçınarak, katlanmış Kimwipes mikroarray cam kenarları silin.
- Nemli bir oda sağlamak için mikroarray inkübasyon tepsisine su ekleyin. Mikroarray odasına yerleştirin ve nemli nitroselüloz bölümünde üzerine RPMI 1640 yılında hücre süspansiyonu pipetle. Pipet bile hücrelerinin yayılmasını sağlamak için nitroselüloz her köşesinde düşer.
- 1 saat boyunca 37 ° C'de inkübe mikroarray'ler Kuluçka hücrelerinin mikroarray antikorlar ile temas yerleşmek ve gelecek sağlar. Arazi antikorlar karşılık gelen yüzey antijenlerini eksprese eden hücrelerin Çekilecek.
- Inkübasyondan sonra, bağlanmamış hücreleri (yıkama başına 20 sn) yıkayın için en az 15 ml PBS içeren üç olukları içine yavaşça ve dikey mikroarray'ler daldırma.
- Çapraz bağlayarak hücreleri ve antikor düzeltmek için% 3.7 (w / v) PBS içinde formaldehit hazırlayın. Yavaşça mikroarray nitroselüloz bölümü kapsayan yaklaşık 1 ml pipetle. Oda sıcaklığında 20 dakika inkübe edin.
- Sonraki daldırma mikroarray'ler PBS (15 ml, her biri) 30 s 3 değişiklik içine fazla formaldehit yıkayın.
- Kenarlar ve arka cam slayt Kimwipes ile silin ve mikroarray DotScan tarayıcı süre kullanarak tarama, nitroselüloz bölümünde nemli. Optik tarama, karışık bir hücre popülasyonu örneğin CRC hücreleri, lökositler ve diğer stromal hücreler tümör antijeni ifade desen sağlar.
4. Çoğullama Floresan
- Mikroarray tarayıcıdan çıkarın ve engelleme tampon (% 2 w / v BSA ısı ile inaktive edilmiş insan AB serum, PBS, pH 7.3,% 2) 200 mcL uygulayın. Mikroarray tepsi 20 dakika oda sıcaklığında inkübe edin.
- Multip hazırlayın(Biolegend, Eppendorf tüp, 10 mcL Alexa Fluor 647-anti-EpCAM, 20 mcL Fikoeritrin-anti-CD3 (1/7.5 son seyreltme Beckman Coulter, Gladesville, NSW, Avustralya, # IM12824): alüminyum folyo ile kaplanmış bir çözüm lexing San Diego, CA, ABD; 1 / 15 dilüsyon), ısı ile inaktive edilmiş insan AB serumu (Sigma-Aldrich, Castle Hill, NSW, Avustralya) ve engelleme tampon 118 mcL 2 mcL.
- Mikroarray fazla engelleme tampon boşaltın ve düzgün yayılan nitroselüloz bölüm üzerine çoğullama çözüm pipetle. Oda sıcaklığında karanlıkta 30 dakika inkübe edin.
- 15 ml taze PBS üç olukları içine dikey mikroarray batırın (her biri 30 sn).
- Karanlık ve mağaza mikroarray'ler kurumasını bekleyin, 4 ° C slayt kutusunda. Mikroarray floresan kaybı olmadan, 3 ay kadar karanlık saklanabilir.
- Typhoon FLA 9000 - 50 çözünürlüğe sahip bir tarayıcı (GE Healthcare, Rydalmere, NSW, Avustralya) kullanılarak kuru mikroarray Tarama (532 nm lazer, PE 580 BP30 emisyon filtresi. Alexa 647 633 nm lazer ve 670 BP30 emisyon filtresi). Mikroarray'ler nitroselüloz tarafı cam tarayıcı tepsi üzerindeki aşağı bakacak şekilde taranır.
- TIFF dosyaları olarak floresan görüntü ve Photoshop seti kullanarak görüntü boyutu kaydet 17 x 25 cm ve çözünürlüğü 72 piksel / cm. Nokta yoğunluğu ile analiz etmek, DotScan analiz yazılımı görüntü alma.
- DotReader nokta bağlayıcı deseni dijital bir görüntüsünü yakalar ve bir 8 bit grilik skalası (1-256 arası U) her bir antikor nokta hücre bağlanma yoğunluğu rakamlarla. Ara sıra non-spesifik izotip kontrol bağlanma ilgili immünglobulin isotypes antikorları için bağlayıcı değerler çıkarılır. % 100 yoğunlukta parlak nokta karşı her mikroarray Nokta floresan yoğunlukları normalize edildi. Sinyal / spot gücü bir xml dosyası (ham veri) ya da bir çubuk grafik olarak temsil edildi. Pdf dosyası (son rapor)
- Mikroarray heatsmaps ve hiyerarşik kümeleme TM4 Mikroarray Software Suite (MultiExperiment Viewer (MeV) sürüm 4.4 kullanılarak yapılmıştır http://www.tm4.org/mev.html ). Hiyerarşik kümeleme tam bağlantı analizi ile MeV kullanarak arka plan arındırılmış veriler yapıldı. Öklid mesafe benzerlik ölçmek için kullanılır oldu. Eşit varyans ile 2-kuyruklu Student t-test sonuçlarının istatistiksel önemini belirlemek için kullanılmıştır.
5. Temsilcisi Sonuçlar:
DotScan mikroarray Sonuçlar yinelenen dizileri arasında tutarlı hücre bağlayıcı desen göstermelidir. Güçlü hizalama nokta bağlama (CD44/CD29) dizi alana yerleştirilmiş olmak için bir kılavuz sağlar. Şekil 2, optimum hücre yakalama ve çoğullama bir örnek gösterir. Şekil 3 hücre yakalama ve olası çözümleri sırasında karşılaşılan bazı ortak sorunları gösterir.
Mikroarray hücre bağlayıcı sonuçlar 1 ile 256 arasında değişen bir grilik ölçekte ifade nokta yoğunlukları ölçerek belirlenebilir. Şekil 4 hiyerarşik kümeleme ile İlgi haritası EpCAM-Alexa 647 antikor ile boyandı 58 cerrahi CRC örneklerinden sayısal veriler, gösterir. Numune sayısı sınırlı olsa da, aynı sahnede CRC aynı grupta küme eğilimindedir.
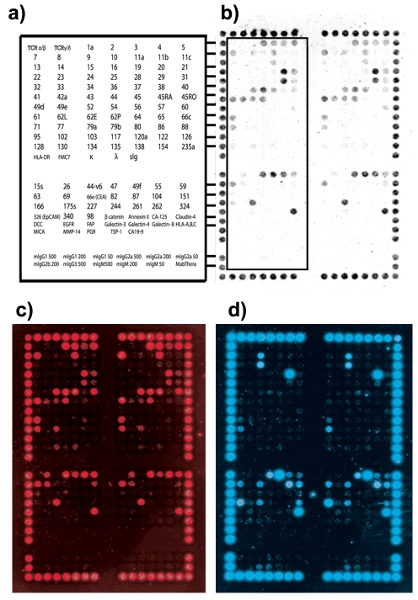
Şekil 2 klinik kolorektal kanser tümörü (Avustralya Klinik-Patolojik Evreleme, ACP evre B1) Hücre bağlayıcı desen. Yinelenen mikroarray (özetlendiği gibi) sol yarısı için antikor yerlerini gösteren (a) DotScan antikor anahtarı. Üst bölümde DotScan lösemi mikroarray orijinal 82 antikorlar içerir. Spesifik yüzey antijenleri ilgili ek bir 40 antikorlar, bir CRC 'uydu' mikroarray olarak eklendi, literatürde up-regüle bulundu. Alt kısmında izotip kontrol antikorlar mikroarray bağlayıcı CRC hücrelerinin (b) Optik görüntü oluşur. T-hücrelerinin (c) CD3 floresan görüntü. (D) CRC hücreleri gösteren EpCAM floresan görüntü.

Şekil 3, kötü DotScan sonuçları ve olası çözümleri örnekleri . (A) Düşük hücre bağlayıcı çözüm:; mikroarray üzerinde kuluçka önce en aza indirmek için örnek ısı ile inaktive edilmiş insan AB serum eklemek çözüm en azından 4x106 canlı hücreler (b) bağlayıcı İzotip kontrolü bağlayıcı ve non-spesifik bir hücre dizisi üzerinde emin olun. izotip kontrol bağlayıcı. Bazen, nitroselüloz hücreleri non-spesifik bağlayıcı bir miktar CRC örnekleri ile ortaya çıkar ve sonuçları önemli ölçüde etkilemez. (C) Selülozik inkübasyon sırasında kuruma; çözüm: örnek tüm nitroselüloz bölümü kapsar ve mikroarray düz bir yüzey üzerinde inkübe sağlamak. (D) Yüksek arka plan eserler; çözüm: sağlamakmikroarray aşağıdaki inkübasyon iyice yıkanır.
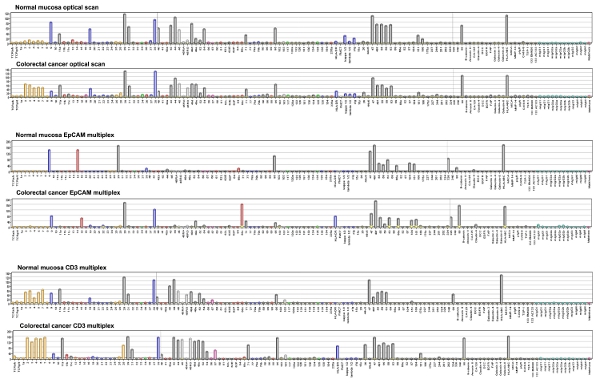
Şekil 4 DotScan analiz yazılımı, 1 ile 256 arasında değişen bir grilik ölçekte bağlayıcı hücre yoğunlukları temsil eden çubuk grafikler oluşturulur. Eksen üzerinde Sayılar CD antijenleri bakın. Κ, λ, immünglobulin hafif zincirleri;; sIg, yüzey immünglobulin; DCC, kolorektal kanser protein silinir; EGFR, epidermal büyüme faktörü reseptörü; FAP, fibroblast aktivasyonu protein, HLA-A, B, Diğer kısaltmalar TCR, T-hücre reseptör A, B, C, sırasıyla C HLA-DR, insan lökosit antijenleri DR ve; MİKA, MHC sınıf I zincir-ilişkili protein A, MMP-14, matris metallopeptidase 14; PIGR, polimerik immünglobulin reseptör; TSP-1, trombospondin-1; Mabthera, insanlara anti-CD20 büyük resim görmek için buraya tıklayın .
Tartışmalar
Bu video, DotScan antikor mikroarray CRC doku hücre popülasyonlarının için yüzey antijeni profillerini incelemek için basit, yarı-kantitatif bir şekilde nasıl kullanılabileceğini göstermektedir.
İnkübasyon sırasında bütün hücrelerin antikor nokta firma bağlanma için gerekli olan enerji bağımlı süreçler (örn., antijen kapak kapatma ve / veya pseudopodia oluşumu) görünür, çünkü doku yaşayabilir bir tek hücre süspansiyonu elde etme, deneyin başarısı içi...
Açıklamalar
Çıkar çatışması ilan etti.
Teşekkürler
Royal Prince Alfred ve Concord ÇHS ve normal bağırsak mukozasında taze örnekleri toplamak için ülkesine geri gönderilme Hastaneler Anatomik Patoloji Laboratuvarları personel teşekkür ederim. Iş Kanser Enstitüsü Yeni Güney Galler Translasyonel Programı Hibe tarafından finanse edildi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Reaktif ya da ekipman Adı | Şirket | Katalog numarası | Yorumlar |
|---|---|---|---|
| Hanks 'dengeli tuz solüsyonu | Sigma-Aldrich | H6136-10X1L | 25 mM HEPES (Sigma # H3375) ile tamponlanmış |
| Airpure biyolojik güvenlik kabini sınıf II | Westinghouse | 1687-2340/612 | |
| Cerrahi bıçaklar | Livingstone | 090609 | 100 adetlik ambalaj |
| 2 mM HEPES RPMI 1640 | Sigma-Aldrich | R4130-10X1L | |
| Kollajenaz tip 4 | Worthington | 4188 | |
| Deoksiribonükleaz 1 | Sigma-Aldrich | DN25-1G | |
| Terumo Şırınga (10 mL) | Terumo | SS 10 L | 100 Kutu |
| Filcon filtre (200 mikron) | BD Biosciences | 340615 | |
| Filcon filtresi (50 mikron) | Filcon filtresi (50 mikron) | Filcon filtresi (50 mikron) Filcon filtresi (50 mikron) 340.603 | |
| Fetal buzağı serumu | Gibco / Invitrogen | 10099-141 | |
| 5810 R Santrifüj | Eppendorf | 7017 | |
| Dimetil sulphoxide | Sigma-Aldrich | D2650 | |
| Tripan mavi | Sigma-Aldrich | T8154 | |
| Hemocymeter Technocolor Neubar | Hirschmann | mevcut değil | |
| Işık mikroskobu | Nikon | Nikon TMS | |
| Cyrovial tüpler | Greiner biyo-bir | 121278 | |
| Cryo dondurma contrainer | Nalgene | 5100-0001 | |
| DotScan antikor mikroarray kiti | Medsaic | mevcut değil | |
| DotScan mikroarray yıkama tepsisi | Medsaic | mevcut değil | |
| Kimwipes | Kimberly-Clark | 4103 | |
| 37% Formaldehit | Sigma-Aldrich | F1635-500ML | |
| DotReaderTM | Medsaic | mevcut değil | |
| Sığır serum albumini | Sigma-Aldrich | A9418-10G | |
| Isı ile inaktive AB serum% 2 | Invitrogen | 34005100 | |
| Fikoeritrin-konjuge CD3 | Beckman Coulter | ET386 | |
| AlexaFluor647-konjuge EpCAM | BioLegend | 324212 | |
| Typhoon FLA 9000 | GE Healthcare | 28-9558-08 | 532 nm lazer, PE 580 BP30 emisyon filtresi. 633 nm lazer ve BP30 Alexa647 için 670 emisyon filtresi |
| MultiExperiment Görüntüleyici v4.4 | TM4 Mikroarray Software Suite | Open - kaynak yazılım (Ref 11) |
Referanslar
- Steinert, R., Buschmann, T., vander Linden, M., Fels, L. M., Lippert, H., Reymond, M. A. The role of proteomics in the diagnosis and outcome prediction in colorectal cancer. Technol. Cancer. Res. Treat. 1, 297 (2002).
- Eifel, P., Axelson, J. A., Costa, J., Crowley, J., Curran, W. J., Deshler, A., Fulton, S., Hendricks, C. B., Kemeny, M., Kornblith, A. B., Louis, T. A., Markman, M., Mayer, R., Roter, D. National Institutes of Health Consensus Development Conference Statement: adjuvant therapy for breast cancer. J. Natl. Canc. Inst. 93, 979 (2001).
- Swerdlow, S. H., Campo, E., Harris, H. L., Jaffe, E. S., Pileri, S. A., Stein, H., Thiele, J., Vardiman, J. W. WHO classification of tumour of haematopoietic and lymphoid tissues. IARC WHO Classification of Tumours. 2, (2008).
- Xiao, G. G., Recker, R. R., Deng, H. W. Recent advances in proteomics and cancer biomarker discovery. Clin. Med. Oncol. , (2008).
- Belov, L., Mulligan, S. P., Barber, N., Woolfson, A., Scott, M., Stoner, K., Chrisp, J. S., Sewell, W. A., Bradstock, K. F., Bandall, L., Pascovici, D. S., Thomas, M., Erber, W., Huang, P., et al. Analysis of human leukaemias and lymphomas using extensive immunophenotypes from an antibody microarray. Br. J. Haematol. 135, 184 (2006).
- Belov, L., Huang, P., Barber, N., Mulligan, S. P., Christopherson, R. I. Identification of repertories of surface antigens on leukemias using an antibody microarray. Proteomics. 3, 2147 (2003).
- Zhou, J., Belov, L., Huang, P. Y., Shin, J., Solomon, M. J., Chapuis, P. H., Bokey, L., Chan, C., Clarke, C., Clarke, S. J., Christopherson, R. I. Surface antigen profiling of colorectal cancer using antibody microarrays with fluorescence multiplexing. J. Immunol. Methods. 355 (1-2), 40-51 (2010).
- Ellmark, P., Belov, L., Huang, P., Lee, C. S., Solomon, M. J., Morgan, D. K., Christopherson, R. I. Multiplex detection of surface molecules on colorectal cancers. Proteomics. 6, 1791 (2006).
- Pearson, J. P., Allen, A., Hutton, D. A. Rheology of mucin. Methods Mol. Biol. 125, 99 (2000).
- Yang, Y. H., Dudoit, S., Luu, P., Lin, D. M., Peng, V., Ngai, J., Speed, T. P. Normalization for cDNA microarray data: a robust composite method addressing single and multiple slide systematic variation. Nucleic Acids Res. 30, 15 (2002).
- Al Saeed, ., et al. TMA: A free, open-source system for microarray data management and analysis. BioTechniques. 34, 374-378 (2003).
Erratum
Formal Correction: Erratum: Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing
Posted by JoVE Editors on 7/03/2015. Citeable Link.
The author's email has been corrected in the publication of Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing. There was an error with the author, Jerry Zhou's, email. The author's email has been updated to:
j.zhou@uws.edu.au
from:
jzho7551@mail.usyd.edu.au
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır