Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Колоректального рака на поверхности клеток белков профилировки с Microarray антитела и флуоресценции мультиплексирования
В этой статье
Erratum Notice
Резюме
Мы описали процедуру разбивки колоректального рака (КРР) для получения жизнеспособных отдельных клеток, которые затем захвачен на индивидуальные микрочипов антител признании поверхностных антигенов (DotScan CRC микрочипов). Суб-популяции клеток, связанных с микрочипов можно профилировать по флуоресценции мультиплексирования с использованием моноклональных антител помеченные флуоресцентными красителями.
Аннотация
The current prognosis and classification of CRC relies on staging systems that integrate histopathologic and clinical findings. However, in the majority of CRC cases, cell dysfunction is the result of numerous mutations that modify protein expression and post-translational modification1.
A number of cell surface antigens, including cluster of differentiation (CD) antigens, have been identified as potential prognostic or metastatic biomarkers in CRC. These antigens make ideal biomarkers as their expression often changes with tumour progression or interactions with other cell types, such as tumour-infiltrating lymphocytes (TILs) and tumour-associated macrophages (TAMs).
The use of immunohistochemistry (IHC) for cancer sub-classification and prognostication is well established for some tumour types2,3. However, no single ‘marker’ has shown prognostic significance greater than clinico-pathological staging or gained wide acceptance for use in routine pathology reporting of all CRC cases.
A more recent approach to prognostic stratification of disease phenotypes relies on surface protein profiles using multiple 'markers'. While expression profiling of tumours using proteomic techniques such as iTRAQ is a powerful tool for the discovery of biomarkers4, it is not optimal for routine use in diagnostic laboratories and cannot distinguish different cell types in a mixed population. In addition, large amounts of tumour tissue are required for the profiling of purified plasma membrane glycoproteins by these methods.
In this video we described a simple method for surface proteome profiling of viable cells from disaggregated CRC samples using a DotScan CRC antibody microarray. The 122-antibody microarray consists of a standard 82-antibody region recognizing a range of lineage-specific leukocyte markers, adhesion molecules, receptors and markers of inflammation and immune response5, together with a satellite region for detection of 40 potentially prognostic markers for CRC. Cells are captured only on antibodies for which they express the corresponding antigen. The cell density per dot, determined by optical scanning, reflects the proportion of cells expressing that antigen, the level of expression of the antigen and affinity of the antibody6.
For CRC tissue or normal intestinal mucosa, optical scans reflect the immunophenotype of mixed populations of cells. Fluorescence multiplexing can then be used to profile selected sub-populations of cells of interest captured on the array. For example, Alexa 647-anti-epithelial cell adhesion molecule (EpCAM; CD326), is a pan-epithelial differentiation antigen that was used to detect CRC cells and also epithelial cells of normal intestinal mucosa, while Phycoerythrin-anti-CD3, was used to detect infiltrating T-cells7. The DotScan CRC microarray should be the prototype for a diagnostic alternative to the anatomically-based CRC staging system.
протокол
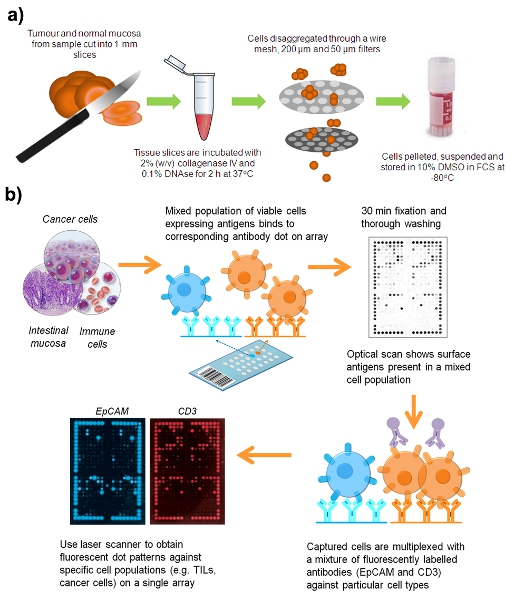
Рисунок 1. Набор операций для приготовления суспензии живых клеток от хирургического образца о правах ребенка.
1. Клинический пример разбивки
Все образцы были собраны из Королевской больницы Принца Альфреда (Camperdown, Новый Южный Уэльс, Австралия) и Согласия Репатриация больница (Конкорд Запад, Новый Южный Уэльс, Австралия) с информированного согласия соответствии с Протоколом № X08-164.
- Сбор свежих колоректального рака (КРР) или аденома образцов, а нормальная слизистая оболочка кишечника не менее 10 см от опухоли. Магазин образцов в сбалансированный солевой раствор Хэнка рН 7,3 (HBSS) при 4 ° С в течение 12 ч после резекции.
- Следуйте правилам безопасности для человека микроорганизмов, процесс всех клинических образцов в кабинете биологической безопасности класса II. Рассеките образцов в 2 мм кубов в чашке Петри с помощью двух скальпель лезвий.
- Инкубируйте опухоли и нормальной ткани в отдельных труб Эппендорф с редкими нежными смешивания в течение 60 мин при 37 ° С с равным объемом RPMI 1640, содержащей 2% (объем / объем) коллагеназы типа 4 (Уортингтон, Лейквуд, штат Нью-Джерси, США) и 0,1 % (м / о) дезоксирибонуклеазы я из бычьего pancrease (ДНКазы I; Sigma-Aldrich).
- Группа полу-переваренной ткани через сито с мелкими отверстиями проволоки использованием плунжера из 10 мл шприца; мыть клетки через с HBSS.
- Pass результате клеточной суспензии через 200 мкм и 50 мкм фильтры Filcon (BD Biosciences) для удаления клеточных агрегатов. Большинство ДНК, слизи и клеточных агрегатов удаляются в этой серии фильтрации.
- Центрифуга клеточные суспензии, при 400 мкг при 20 ° в течение 5 мин.
- Ресуспендируют гранулы ячейку в тепло инактивированной ФТС, содержащей 10% диметилсульфоксида (ДМСО), замораживание медленно криопробирки и хранить при температуре -80 °. Замораживание процесса ведет к снижению слизи в образце и лизирует эритроциты.
2. Подготовка образцов для сотовых захвата
- Оттепель из образцов быстро в 37 ° водяной бане и ресуспендирования клеток в 10 мл HBSS промыть ДМСО.
- Центрифуга клеточные суспензии, при 410 мкг при 20 ° в течение 5 мин.
- Декантируйте супернатант и ресуспендируют осадок клеток в 500 мкл HBSS.
- Лечить образца с 0,1% (м / о) ДНКазы я в течение 20 мин при комнатной температуре.
- Смешать по 10 мкл каждой клеточной суспензии с равным объемом трипанового синий и нагрузка 10 мкл смеси в гемоцитометра. Использование светового микроскопа при 100-кратным увеличением, граф жизнеспособных клеток, которые появляются в связи с ясно трипанового синего исключения, в то время как мертвые клетки занимают красителя. Не менее 4-х 10 6 жизнеспособных клеток требуется для сотовых захвата на микрочипов.
- После лечения ДНКазы, ресуспендируют клеточной суспензии в 10 мл HBSS и центрифуге при 410 мкг при 20 ° в течение 5 мин.
- Декантируйте супернатанты и ресуспендируйте гранулы ячейку в RPMI 1640 до конечного объема 200 мкл.
3. Антитела микрочипов ячейке захвата
- Смочите DotScan микрочипов антител путем погружения нитроцеллюлозы в разделе фосфатным буферным раствором (PBS) в течение примерно 20 секунд Аккуратно протрите стекла края микрочипов со сложенными Kimwipes, избегая касания нитроцеллюлозы разделе.
- Добавьте воду в лоток инкубации микрочипов обеспечить влажной камере. Место микрочипов в камеру и пипетки суспензии клеток в RPMI 1640 на влажном разделе нитроцеллюлозы. Внесите капель на каждом углу нитроцеллюлозы для обеспечения равномерного распределения клеток.
- Инкубируйте микрочипов при 37 ° С в течение 1 ч. Инкубационный позволяет клеткам для проведения расчетов и вступают в контакт с антителами на микрочипов. Клетки, экспрессирующие поверхностные антигены соответствующих антител, они приземляются на будет в плен.
- После инкубации, окуните микрочипов мягко и по вертикали на три желоба содержащих не менее 15 мл PBS, чтобы смыть несвязанных клетки (20 с на мытье).
- Подготовка 3,7% (м / о) формальдегида в PBS исправить клетки и антитела путем сшивания. Аккуратно пипеткой примерно 1 мл на покрытие нитроцеллюлозы разделе микрочипов. Инкубируйте в течение 20 мин при комнатной температуре.
- Следующее падение микрочипов на 3 изменений PBS (15 мл, 30 с каждого), чтобы смыть избыток формальдегида.
- Протрите края и задней стекло с Kimwipes и сканирования микрочипов использованием DotScan в то время как сканер, нитроцеллюлозы разделе влажная. Оптического сканирования дает картину экспрессии антигенов смешанной популяции клеток, например, клетки КПР, лейкоцитов и других стромальных клеток опухоли.
4. Флуоресценция мультиплексирования
- Удалить микрочипов от сканера и применять 200 мкл блокирующего буфера (2% вес / BSA, 2% тепла человеческого инактивированной сыворотки АВ, PBS, рН 7,3). Выдержите его в лоток микрочипов при комнатной температуре в течение 20 мин.
- Подготовка multipлексический раствора в пробирку Эппендорфа покрыта алюминиевой фольгой: 20 мкл фикоэритрин-анти-CD3 (Beckman Coulter, Gladesville, Новый Южный Уэльс, Австралия, # IM12824; 1/7.5 конечное разведение), 10 мкл Alexa Fluor 647-анти-EpCam (Biolegend, Сан - Диего, Калифорния, США; 1 / 15 разбавления), 2 мкл тепла инактивированной человеческой сыворотки АВ (Sigma-Aldrich, Castle Hill, NSW, Австралия) и 118 мкл блокирующего буфера.
- Слить избыток блокирующий буфер из микрочипов и пипетки мультиплексирования раствор на нитроцеллюлозные разделе, распространяясь равномерно. Инкубируйте в течение 30 минут в темном месте при комнатной температуре.
- Dip микрочипов по вертикали на три желоба 15 мл свежего PBS; (30 с каждого).
- Пусть микрочипов сухой в темноте и хранить при 4 ° С в режиме слайд-коробки. Микрочипов может храниться в темноте в течение 3 месяцев без потери флуоресценции.
- Сканирование сухой микрочипов использованием Тайфун FLA-9000 сканер (GE Healthcare, Rydalmere, Новый Южный Уэльс, Австралия) с разрешением равным 50 (532 нм, 580 BP30 выбросов фильтр для PE. 633 лазерных нм и 670 BP30 выбросов фильтр для Alexa 647). Микрочипы будут отсканированы с нитроцеллюлозы стороной вниз на лоток стекло сканера.
- Сохранить флуоресцентные изображения в виде файлов TIFF и используя набор Photoshop изображение размером до 17 х 25 см и разрешением до 72 точек / см. Импорт изображений в DotScan программное обеспечение анализа для анализа интенсивности точек.
- DotReader захватывает цифровое изображение точки обязательным модель и количественная плотность клеточных обязательным для каждого антитела точка на 8 бит серости шкале (1-256 U). Иногда неспецифический изотипа контроль связывание вычитается из обязательного значения для антител с соответствующими изотипов иммуноглобулина. Dot интенсивности флуоресценции для каждого микрочипов были нормализованы против ярких точка установлена на уровне 100% интенсивности. Сигнал / место силы было зафиксировано в. XML-файл (исходные данные) или представлены в виде гистограммы в. PDF-файл (окончательный)
- Microarray heatsmaps и иерархической кластеризации были проведены с использованием MultiExperiment Viewer (МэВ) версии 4.4 от ТМ4 Microarray Software Suite ( http://www.tm4.org/mev.html ). Иерархическая кластеризация проводили на фоне скорректированных данных с использованием МэВ с полным анализом связей. Евклидово расстояние было использовано для меры сходства. 2-хвостами Стьюдента-тест с равной дисперсии был использован для определения статистической значимости результатов.
5. Представитель Результаты:
Результаты микрочипов DotScan должен показать последовательное ячейки обязательным моделей между дублировать массивы. Сильное выравнивание точку привязки (CD44/CD29) позволяет сетке находиться над массивом района. На рисунке 2 показан пример оптимального захвата клеток и мультиплексирование. На рисунке 3 приведены некоторые общие проблемы, возникшие в ходе захвата клеток и возможные решения.
Клетка микрочипов обязательные результаты могут быть количественно путем измерения интенсивности точка, высказанные по серости шкале от 1 до 256. На рисунке 4 показаны цифровые данные с 58 хирургических образцов КПР, окрашивали EpCam-Alexa 647 антитела, как Тепловая карта с иерархической кластеризации. Хотя количество образцов ограничено, контрольных сумм из той же стадии, как правило, кластер в одной группе.
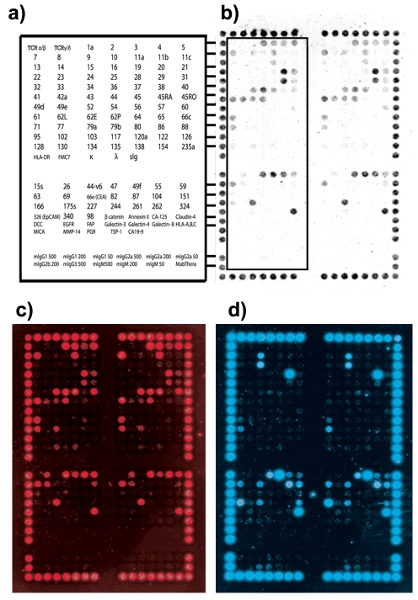
Рисунок 2. Сотовый обязательной структуре клинических колоректального рака опухоли (Австралийский Клинико-патологические Подмости, ACP этапе B1). () DotScan антител ключевым изображением расположения антитела для левой половины дублировать микрочипов (очерчен). Верхняя часть содержит оригинальные 82 антитела микрочипов лейкемии DotScan. Еще 40 антител, соответствующие конкретным антигенам поверхности оказались до регулируемых в литературе, были добавлены в качестве микрочипов "спутник" КПР. Нижняя секция состоит из изотипа антител управления (б) Оптическое изображение клетки КПР привязки к микрочипов. (С) CD3 флуоресценции изображение, показывающее Т-клеток. (Г) EpCam флуоресценции изображение, показывающее CRC клеток.

Рисунок 3. Примеры плохого DotScan результаты и возможные решения. () Низкая ячейки обязательными; Решение: убедитесь, что по крайней мере 4x106 жизнеспособных клеток находятся в массиве (б) Изотип контроль обязательных и необязательных определенной ячейке обязательными; решение: добавить тепла инактивированной человеческой сыворотки АВ образца до инкубации на микрочипов, чтобы минимизировать изотипа контроля обязательными. Иногда небольшое количество неспецифического связывания клеток к нитроцеллюлозы происходит с КПР образцов и не оказывает существенного влияния на результаты. (С) Нитроцеллюлоза высыхания во время инкубации; решение: обеспечить выборка охватывает целый раздел нитроцеллюлозы и микрочипов, инкубируют на плоской поверхности. (Г) высокий артефактов фона; решение: обеспечитьмикрочипов тщательно промывают после инкубации.
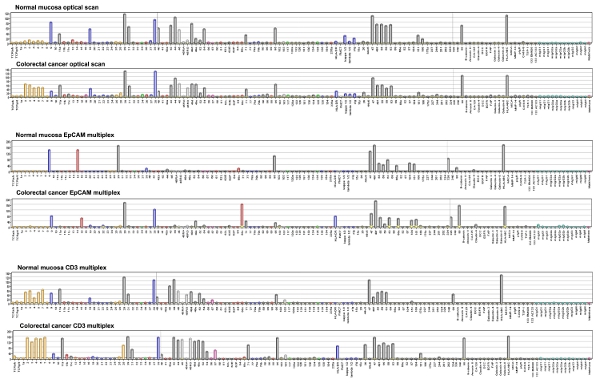
Рисунок 4. DotScan программного обеспечения для анализа порожденных гистограммы представляющие ячейку обязательным плотностей на серость шкале от 1 до 256. Цифры на оси ссылаются на CD антигенов. Другие сокращения TCR, Т-клеточного рецептора; κ, λ, легких цепей иммуноглобулинов; SIG, поверхностных иммуноглобулинов; DCC, удаленные в колоректального рака белка, EGFR, рецептор эпидермального фактора роста, ФАП, белок фибробластов активации; HLA-A, B, С HLA-DR, антигены лейкоцитов человека и DR, B, C соответственно; ММВА, MHC класса I цепь связанных белков, MMP-14, матрица metallopeptidase 14; PIGR, полимерные рецепторов иммуноглобулинов; TSP-1, тромбоспондин-1; Мабтера, гуманизированные анти-CD20. Нажмите здесь, чтобы посмотреть увеличенное изображение .
Обсуждение
В этом видео показано, как DotScan микрочипов антитела могут быть использованы в простых, полуколичественный способ изучения профилей поверхностного антигена для популяции клеток из ткани КПР.
Получение жизнеспособных суспензии отдельных клеток из ткани имеет решающее з...
Раскрытие информации
Нет конфликта интересов объявлены.
Благодарности
Мы благодарим сотрудников Анатомические лаборатории патологии Королевский Принца Альфреда и Согласия Репатриация Больницы для сбора свежих образцов КПР и нормальной слизистой оболочки кишечника. Работа финансировалась института рака Нового Южного Уэльса Поступательное Программа грантов.
Материалы
| Name | Company | Catalog Number | Comments |
| Название реагентов или оборудования | Компания | Номер в каталоге | Комментарии |
|---|---|---|---|
| Сбалансированный солевой Хэнкса решение | Sigma-Aldrich | H6136-10X1L | Буферизацией с 25 мМ Hepes (Sigma # H3375) |
| Airpure биологической безопасности II класса кабинет | Westinghouse | 1687-2340/612 | |
| Хирургические лезвия | Ливингстон | 090609 | Упаковка из 100 |
| RPMI 1640 с 2 мМ Hepes | Sigma-Aldrich | R4130-10X1L | |
| Коллагеназы типа 4 | Уортингтон | 4188 | |
| Дезоксирибонуклеазы 1 | Sigma-Aldrich | DN25-1G | |
| Terumo шприц (10 мл) | Terumo | SS +10 L | Комплект из 100 |
| Filcon фильтр (200 мкм) | BD Biosciences | 340615 | |
| Filcon фильтр (50 мкм) | Filcon фильтр (50 мкм) | Filcon фильтр (50 мкм) Filcon фильтр (50 мкм) 340603 | |
| Фетальной телячьей сыворотки | Gibco / Invitrogen | 10099-141 | |
| Центрифуга 5810 R | Эппендорф | 7017 | |
| Диметилсульфоксид | Sigma-Aldrich | D2650 | |
| Трипановый синий | Sigma-Aldrich | T8154 | |
| Hemocymeter Technocolor Neubar | Hirschmann | недоступный | |
| Оптический микроскоп | Nikon | Nikon TMS | |
| Cyrovial труб | Greiner Bio-One | 121278 | |
| Криогенная заморозка contrainer | Nalgene | 5100-0001 | |
| DotScan антител микрочипов комплект | Medsaic | недоступный | |
| DotScan микрочипов мыть лоток | Medsaic | недоступный | |
| KimWipes | Kimberly-Clark | 4103 | |
| Формальдегид 37% | Sigma-Aldrich | F1635-500ML | |
| DotReaderTM | Medsaic | недоступный | |
| Бычий сывороточный альбумин | Sigma-Aldrich | A9418-10G | |
| Тепло-инактивированной сывороткой AB 2% | Invitrogen | 34005100 | |
| Фикоэритрин-сопряженных CD3 | Beckman Coulter | ET386 | |
| AlexaFluor647-сопряженных EpCam | BioLegend | 324212 | |
| Тайфун FLA-9000 | GE Healthcare | 28-9558-08 | 532 нм, 580 BP30 выбросов фильтр для PE. 633 лазерных нм и 670 BP30 выбросов фильтр для Alexa647 |
| MultiExperiment просмотра v4.4 | ТМ4 Microarray Software Suite | Открыть - программное обеспечение (Ссылка 11) |
Ссылки
- Steinert, R., Buschmann, T., vander Linden, M., Fels, L. M., Lippert, H., Reymond, M. A. The role of proteomics in the diagnosis and outcome prediction in colorectal cancer. Technol. Cancer. Res. Treat. 1, 297 (2002).
- Eifel, P., Axelson, J. A., Costa, J., Crowley, J., Curran, W. J., Deshler, A., Fulton, S., Hendricks, C. B., Kemeny, M., Kornblith, A. B., Louis, T. A., Markman, M., Mayer, R., Roter, D. National Institutes of Health Consensus Development Conference Statement: adjuvant therapy for breast cancer. J. Natl. Canc. Inst. 93, 979 (2001).
- Swerdlow, S. H., Campo, E., Harris, H. L., Jaffe, E. S., Pileri, S. A., Stein, H., Thiele, J., Vardiman, J. W. WHO classification of tumour of haematopoietic and lymphoid tissues. IARC WHO Classification of Tumours. 2, (2008).
- Xiao, G. G., Recker, R. R., Deng, H. W. Recent advances in proteomics and cancer biomarker discovery. Clin. Med. Oncol. , (2008).
- Belov, L., Mulligan, S. P., Barber, N., Woolfson, A., Scott, M., Stoner, K., Chrisp, J. S., Sewell, W. A., Bradstock, K. F., Bandall, L., Pascovici, D. S., Thomas, M., Erber, W., Huang, P., et al. Analysis of human leukaemias and lymphomas using extensive immunophenotypes from an antibody microarray. Br. J. Haematol. 135, 184 (2006).
- Belov, L., Huang, P., Barber, N., Mulligan, S. P., Christopherson, R. I. Identification of repertories of surface antigens on leukemias using an antibody microarray. Proteomics. 3, 2147 (2003).
- Zhou, J., Belov, L., Huang, P. Y., Shin, J., Solomon, M. J., Chapuis, P. H., Bokey, L., Chan, C., Clarke, C., Clarke, S. J., Christopherson, R. I. Surface antigen profiling of colorectal cancer using antibody microarrays with fluorescence multiplexing. J. Immunol. Methods. 355 (1-2), 40-51 (2010).
- Ellmark, P., Belov, L., Huang, P., Lee, C. S., Solomon, M. J., Morgan, D. K., Christopherson, R. I. Multiplex detection of surface molecules on colorectal cancers. Proteomics. 6, 1791 (2006).
- Pearson, J. P., Allen, A., Hutton, D. A. Rheology of mucin. Methods Mol. Biol. 125, 99 (2000).
- Yang, Y. H., Dudoit, S., Luu, P., Lin, D. M., Peng, V., Ngai, J., Speed, T. P. Normalization for cDNA microarray data: a robust composite method addressing single and multiple slide systematic variation. Nucleic Acids Res. 30, 15 (2002).
- Al Saeed, ., et al. TMA: A free, open-source system for microarray data management and analysis. BioTechniques. 34, 374-378 (2003).
Erratum
Formal Correction: Erratum: Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing
Posted by JoVE Editors on 7/03/2015. Citeable Link.
The author's email has been corrected in the publication of Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing. There was an error with the author, Jerry Zhou's, email. The author's email has been updated to:
j.zhou@uws.edu.au
from:
jzho7551@mail.usyd.edu.au
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены