Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Colorectal Cancer Cell Surface Protein profilage utilisant une biopuces d'anticorps et de multiplexage par fluorescence
Dans cet article
Erratum Notice
Résumé
Nous avons décrit une procédure pour la désagrégation du cancer colorectal (CCR) pour produire des cellules viables unique, qui sont ensuite saisies sur biopuces d'anticorps reconnaissant les antigènes de surface sur mesure (CRC DotScan micropuce). Les sous-populations de cellules lié à la puce peut être profilée par multiplexage de fluorescence à l'aide d'anticorps monoclonaux marqués avec des colorants fluorescents.
Résumé
Le pronostic actuel et la classification des CRC repose sur la stadification des systèmes qui intègrent les résultats histopathologiques et cliniques. Cependant, dans la majorité des cas de CCR, le dysfonctionnement cellulaire est le résultat de nombreuses mutations qui modifient l'expression des protéines et des modifications post-traductionnelles 1.
Un certain nombre d'antigènes de surface cellulaire, y compris les clusters de différenciation (CD) antigènes, ont été identifiés comme pronostiques potentiels ou métastatique chez les biomarqueurs CRC. Ces antigènes font biomarqueurs idéal comme leur expression change souvent avec la progression tumorale ou des interactions avec d'autres types cellulaires, comme les lymphocytes infiltrant les tumeurs (TIL) et les macrophages associés aux tumeurs (TAM).
L'utilisation de l'immunohistochimie (IHC) pour le cancer de la sous-classification et le pronostic est bien établi pour certains types de tumeurs 2,3. Cependant, pas un seul «marqueur» a montré l'importance pronostique supérieure clinico-pathologiques ou de mise en scène gagné l'acceptation large pour une utilisation dans les rapports de pathologie de routine de tous les cas, le CRC.
Une approche plus récente à la stratification pronostique des phénotypes de la maladie repose sur les profils protéiques de surface en utilisant de multiples «marqueurs». Alors que le profil d'expression des tumeurs en utilisant des techniques telles que la protéomique iTRAQ est un outil puissant pour la découverte de biomarkers4, il n'est pas optimale pour une utilisation de routine dans les laboratoires de diagnostic et ne peuvent pas distinguer différents types de cellules dans une population mixte. En outre, de grandes quantités de tissus tumoraux sont nécessaires pour le profilage des glycoprotéines purifiées membrane plasmique par ces méthodes.
Dans cette vidéo, nous avons décrit une méthode simple pour le profilage de la surface du protéome de cellules viables à partir d'échantillons CRC ventilées en utilisant une puce à anticorps DotScan CRC. La puce 122-anticorps se compose d'une norme 82-anticorps reconnaissant la région une gamme de la lignée des marqueurs spécifiques des leucocytes, des molécules d'adhésion, les récepteurs et les marqueurs de l'inflammation et la réponse immunitaire 5, avec une région par satellite pour la détection de 40 marqueurs potentiellement pronostique pour le CRC . Les cellules sont capturées uniquement sur les anticorps pour lequel ils expriment l'antigène correspondant. La densité cellulaire par point, déterminé par balayage optique, reflète la proportion de cellules exprimant l'antigène, le niveau d'expression de l'antigène et l'affinité de l'anticorps 6.
Pour les tissus normaux CRC ou la muqueuse intestinale, balayages optiques reflètent l'immunophénotype des populations mixtes de cellules. Multiplexage par fluorescence peut alors être utilisé pour le profil sélectionné sous-populations de cellules d'intérêt capturés sur le tableau. Par exemple, Alexa 647 molécules anti-adhérence des cellules épithéliales (EpCAM; CD326), est un antigène de différenciation pan-épithéliales qui a été utilisé pour détecter les cellules CRC et aussi les cellules épithéliales de la muqueuse intestinale normale, tandis que la phycoérythrine-anticorps anti-CD3, a été utilisé pour détecter l'infiltration de cellules T 7. Le CRC DotScan biopuces devrait être le prototype d'une alternative de diagnostic pour le système anatomique basée sur scène CRC.
Protocole
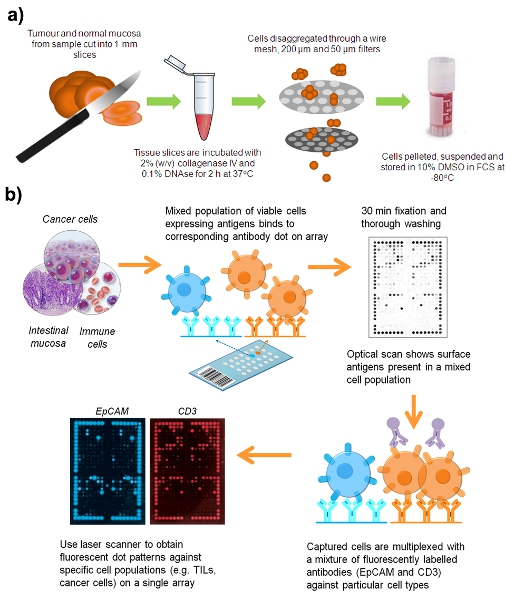
Le flux de travail figure 1. Pour la préparation d'une suspension de cellules vivantes à partir d'un échantillon de chirurgie de la CRC.
1. Désagrégation de l'échantillon clinique
Tous les échantillons ont été prélevés dans l'hôpital Royal Prince Alfred (Camperdown, NSW, Australie) et l'Hôpital de rapatriement Concord (Concord West, NSW, Australie) avec le consentement éclairé du Protocole n ° X08-164.
- Recueillir frais cancer colorectal (CCR) ou adénome spécimens, et la muqueuse intestinale normale d'au moins 10 cm de la tumeur. Conserver les échantillons au pH du sel de Hank solution équilibrée 7.3 (HBSS) à 4 ° C pendant 12 h après la résection.
- Suivez les règles de sécurité pour les agents pathogènes humains, traiter tous les échantillons cliniques dans un coffret de sécurité biologique de classe II. Disséquer les échantillons en cubes de 2 mm dans une boîte de Pétri en utilisant deux lames de bistouri.
- Incuber la tumeur et des tissus normaux dans des tubes Eppendorf séparée avec occasionnellement un mélange doux pendant 60 min à 37 ° C avec un volume égal de milieu RPMI 1640 contenant 2% (v / v) la collagénase de type 4 (Worthington, Lakewood, New Jersey, USA) et 0,1 désoxyribonucléase% (p / v) J'ai du pancréas bovin (DNAse I; Sigma-Aldrich).
- Groupe semi-digérée tissus à travers une passoire fine en utilisant un fil de piston de la seringue de 10 ml, les cellules se laver travers avec HBSS.
- Passe résultant suspension cellulaire à 200 um et 50 um filtres Filcon (BD Biosciences) pour enlever les agrégats de cellules. La plupart des agrégats d'ADN, de mucus et de cellules sont supprimés dans cette série de filtrations.
- Centrifugeuse suspensions cellulaires à 400 xg à 20 ° pendant 5 min.
- Culots cellulaires Remettre en suspension dans la chaleur FCS inactivé, contenant 10% de diméthylsulfoxyde (DMSO), de geler lentement dans cryotubes et conserver à -80 °. Le processus de congélation tend à réduire de mucus dans l'échantillon et lyse des globules rouges.
2. La préparation des échantillons pour la capture de la cellule
- Dégeler rapidement des échantillons dans un bain d'eau à 37 ° et remettre les cellules dans 10 ml de HBSS pour laver le DMSO.
- Centrifugeuse suspensions cellulaires à 410 xg à 20 ° pendant 5 min.
- Décanter le surnageant et remettre le culot cellulaire dans 500 ul de HBSS.
- Traiter l'échantillon avec 0,1% (p / v) DNAse I pendant 20 min à température ambiante.
- Mélanger 10 uL de chaque suspension cellulaire avec un volume égal de bleu de trypan et charger 10 uL du mélange dans un hématimètre. En utilisant un microscope optique à 100 fois le grossissement, le comptage de cellules viables, qui apparaissent clairement en raison d'exclusion du bleu trypan, tandis que les cellules mortes prennent le colorant. Un minimum de 4 x 10 6 cellules viables est requise pour la capture de cellules sur la puce.
- Après traitement à la DNase, remettre en suspension les cellules en suspension dans 10 ml de HBSS et centrifuger à 410 xg à 20 ° pendant 5 min.
- Décanter les surnageants et les culots cellulaires remettre en suspension dans du RPMI 1640 à un volume final de 200 pl.
3. Anticorps de capture cellulaire biopuces
- Humidifiez les biopuces d'anticorps DotScan en plongeant dans la section de nitrocellulose tampon phosphate salin (PBS) pendant environ 20 s. Essuyer soigneusement les bords de verre de la puce à ADN avec Kimwipes plié, en évitant de toucher la section de nitrocellulose.
- Ajouter de l'eau dans le bac d'incubation de microréseaux pour offrir une chambre humide. Placer la puce dans la chambre et la pipette de la suspension cellulaire dans du RPMI 1640 sur la section de nitrocellulose humide. Pipeter gouttes sur chaque coin de la nitrocellulose afin d'assurer une répartition homogène de cellules.
- Incuber les puces à 37 ° C pendant 1 h. L'incubation des cellules permet de s'installer et entrer en contact avec des anticorps sur la puce. Les cellules exprimant les antigènes de surface correspondant à l'anticorps ils débarquent sur sera capturée.
- Après incubation, tremper les microarrays doucement et verticalement en trois bacs contenant au moins 15 ml de PBS à laver les cellules non liée (20 s par lavage).
- Préparer 3,7% (p / v) de formaldéhyde dans du PBS pour fixer les cellules et d'anticorps par réticulation. Délicatement la pipette environ 1 mL pour couvrir la section de nitrocellulose du microréseau. Incuber pendant 20 min à température ambiante.
- Suivant DIP puces en 3 changements de PBS (15 mL; 30 s chacun) pour laver le formaldéhyde en excès.
- Essuyez les bords et le dos de la lame de verre avec Kimwipes et scanner la puce à l'aide du scanner de tout DotScan, la section de nitrocellulose est humide. Le balayage optique fournit le modèle de l'expression des antigènes d'une population de cellules cellules mixtes CRC par exemple, les leucocytes et autres cellules du stroma de la tumeur.
4. Fluorescence multiplexage
- Retirer la puce à partir du scanner et d'appliquer 200 uL de tampon de blocage (2% p / v de BSA, 2% inactivé à la chaleur sérum humain AB, PBS, pH 7,3). Il Incuber dans le plateau de biopuces à température ambiante pendant 20 min.
- Préparer la multiparelexing solution dans un tube Eppendorf recouverts d'une feuille d'aluminium: 20 pl phycoérythrine anti-CD3 (Beckman Coulter, Gladesville, NSW, Australie, # IM12824; 1/7.5 dilution finale), 10 uL Alexa Fluor 647-anti-EpCAM (Biolegend, San Diego, Californie, Etats-Unis; 1 / 15 de dilution), 2 pl de la chaleur sérum humain inactivé AB (Sigma-Aldrich, Castle Hill, NSW, Australie) et 118 uL de tampon de blocage.
- Égoutter l'excès tampon de blocage de la puce et la pipette la solution de multiplexage sur la section de nitrocellulose, répandre uniformément. Incuber pendant 30 min dans le noir à température ambiante.
- Tremper les biopuces verticalement en trois bacs de 15 ml de PBS frais, (30 s chacun).
- Laissez les microarrays sécher dans l'obscurité et stocker à 4 ° C dans une boîte à lames. Les puces à ADN peuvent être stockés dans l'obscurité pendant jusqu'à 3 mois sans perte de fluorescence.
- Numérisez les biopuces à sec en utilisant un typhon FLA 9000 Scanner (GE Healthcare, Rydalmere, NSW, Australie) avec la résolution fixée à 50 (532 nm laser, 580, BP30 émission pour les PE filtre. Laser 633 nm et 670 BP30 filtre d'émission pour Alexa 647). Les biopuces sont scannées avec le côté orienté vers le bas de nitrocellulose sur le plateau de la vitre du scanner.
- Enregistrer les images fluorescentes sous forme de fichiers TIFF et Photoshop en utilisant la taille d'image réglée sur 17 x 25 cm et de résolution de 72 pixels / cm. Importer l'image dans le logiciel d'analyse DotScan d'analyser l'intensité de points.
- Le DotReader capture une image numérique de la configuration de points de liaison et quantifie la densité de la liaison cellulaire sur chaque point d'anticorps sur une échelle grisaille 8 bits (1-256 U). Occasional liaison non spécifique contrôle isotypique a été soustraite de valeurs contraignantes pour les anticorps avec isotypes d'immunoglobulines correspondantes. Dot intensités de fluorescence pour chaque puce ont été normalisés par rapport à la plus brillante de points fixé à 100% d'intensité. Signal / spot force a été enregistré dans un fichier. XML (données brutes), ou représenté comme un graphique à barres dans un fichier. Pdf (rapport final)
- Microarray heatsmaps et le clustering hiérarchique ont été effectuées en utilisant MultiExperiment Viewer (MeV) la version 4.4 de la suite logicielle TM4 Microarray ( http://www.tm4.org/mev.html ). Classification hiérarchique a été réalisée sur des données de base ajustée à l'aide MeV avec l'analyse de liaison complète. Distance euclidienne a été utilisée pour mesure de similarité. L'élève 2-tailed test t de variance égale a été utilisée pour déterminer la signification statistique des résultats.
5. Les résultats représentatifs:
Résultats de la biopuce DotScan devrait montrer les modèles cellulaires compatibles contraignante entre les tableaux en double. Forte d'alignement de points de liaison (CD44/CD29) permet une grille pour être placé sur la zone de tableau. La figure 2 montre un exemple de capture cellulaire optimale et le multiplexage. La figure 3 montre certains problèmes courants rencontrés lors de la capture cellulaire et les solutions possibles.
Les résultats de cellules biopuces liaison peut être quantifiée en mesurant l'intensité de points sur une échelle grisaille allant de 1 à 256. La figure 4 montre les données numériques à partir de 58 échantillons CRC chirurgicale, colorées avec EpCAM-Alexa 647 anticorps, comme un heatmap avec classification hiérarchique. Même si le nombre d'échantillons est limitée, CRC d'un même étage ont tendance à se regrouper dans un même groupe.
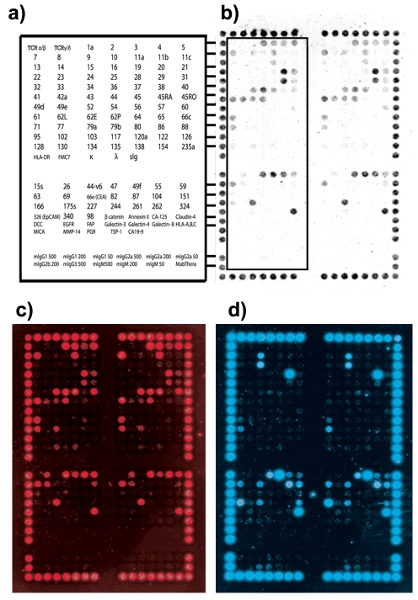
Figure 2. Motif de liaison des cellules tumorales du cancer colorectal cliniques (Australian Clinique-pathologique Staging, ACP étape B1). (A) d'anticorps DotScan clés indiquant les emplacements des anticorps pour la moitié gauche de la puce en double (décrites). La partie supérieure contient les 82 anticorps d'origine du microréseau leucémies DotScan. 40 autres anticorps, correspondant à des antigènes de surface spécifiques jugés sur-régulée dans la littérature, ont été ajoutés comme biopuces «satellite» d'un CRC. La section inférieure se compose d'anticorps isotype contrôle (b) d'image optique de cellules CRC se liant à la puce. (C) image de fluorescence montrant CD3 T-cellules. (D) EpCAM de fluorescence d'image montrant des cellules CRC.

Figure 3. Exemples de mauvais résultats DotScan et les solutions possibles. (A) de cellules à faible liaison; Solution: Assurez au moins 4x106 cellules viables sont sur le tableau (b) la cellule de contrôle isotypique contraignants et non contraignants spécifiques; solution: ajouter de la chaleur inactivé sérum humain AB d'échantillon avant incubation sur microréseaux afin de minimiser isotype contrôle contraignant. Parfois, une petite quantité de liaison non spécifique des cellules de la nitrocellulose se produit avec des échantillons de CRC et n'affecte pas significativement les résultats. (C) La nitrocellulose dessèchement pendant l'incubation; solution: assurer échantillon couvre la section entière et de nitrocellulose est incubée biopuces sur une surface plane. (D) les artefacts de fond élevé; solution: assurer lamicroréseau est soigneusement lavé après incubation.
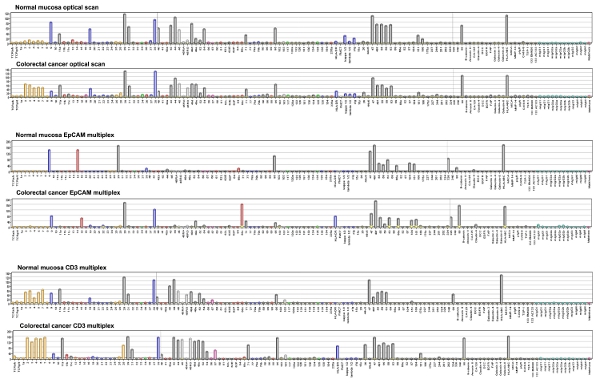
Figure 4. Logiciel d'analyse DotScan généré des graphiques à barres représentant des densités cellulaires contraignant sur une échelle de gris allant de 1 à 256. Numéros sur l'axe se référer à des antigènes CD. D'autres abréviations sont TCR, T cell receptor; κ, λ, chaînes légères d'immunoglobulines, sig, immunoglobulines de surface; CDC, supprimé en protéines du cancer colorectal; EGFR, récepteur épidermique de facteur de croissance; FAP, activation de la protéine fibroblastique; HLA-A, B, C HLA-DR, les antigènes de leucocytes humains DR et A, B, C respectivement; MICA, CMH de classe I liés à la chaîne de la protéine A; MMP-14, métallopeptidase matrice 14; plgR, récepteur des immunoglobulines polymériques; TSP-1, la thrombospondine-1; Mabthera, humanisé anti-CD20. Cliquez ici pour agrandir l'image .
Discussion
Dans cette vidéo, nous démontrons comment les biopuces d'anticorps DotScan peut être utilisé dans un simple, semi-quantitative façon d'étudier les profils d'antigène de surface pour les populations de cellules du tissu CRC.
L'obtention d'une suspension de cellules viables unique à partir de tissus est essentielle à la réussite de l'expérience, parce que dépendant de l'énergie des processus (par exemple, l'antigène le recouvrement et / ou la forma...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Nous remercions le personnel des laboratoires de pathologie anatomique du Royal Prince Alfred et les hôpitaux rapatriement Concord pour la collecte des échantillons frais du CRC et de la muqueuse intestinale normale. Le travail a été financé par une subvention Cancer Institute Nouvelle-Galles du Sud Programme translationnelle.
matériels
| Name | Company | Catalog Number | Comments |
| Nom de réactif ou de l'équipement | Société | Numéro de catalogue | Commentaires |
|---|---|---|---|
| Hanks solution salée équilibrée | Sigma-Aldrich | H6136-10X1L | Tamponné avec 25 mM HEPES (Sigma # H3375) |
| Airpure sécurité biologique de classe II du cabinet | Westinghouse | 1687-2340/612 | |
| Lames chirurgicales | Livingstone | 090609 | Paquet de 100 |
| RPMI 1640 avec 2 mM Hepes | Sigma-Aldrich | R4130-10X1L | |
| Collagénase de type 4 | Worthington | 4188 | |
| Désoxyribonucléase 1 | Sigma-Aldrich | DN25-1G | |
| Terumo Seringue (10 ml) | Terumo | SS 10 L | Boîte de 100 |
| Filcon filtre (200 m) | BD Biosciences | 340615 | |
| Filcon filtre (50 microns) | Filcon filtre (50 microns) | Filcon filtre (50 microns) Filcon filtre (50 microns) 340 603 | |
| Sérum de veau fœtal | Gibco / Invitrogen | 10099-141 | |
| Centrifugeuse 5810 R | Eppendorf | 7017 | |
| Diméthylsulfoxyde | Sigma-Aldrich | D2650 | |
| Le bleu trypan | Sigma-Aldrich | T8154 | |
| Hemocymeter Technocolor Neubar | Hirschmann | pas disponible | |
| Microscope à lumière | Nikon | Nikon TMS | |
| Tubes Cyrovial | Greiner Bio-One | 121278 | |
| Contrainer congélation Cryo | Nalgene | 5100-0001 | |
| DotScan anticorps microarray kit | Medsaic | pas disponible | |
| Bac DotScan laver les biopuces | Medsaic | pas disponible | |
| Kimwipes | Kimberly-Clark | 4103 | |
| Formaldéhyde 37% | Sigma-Aldrich | F1635-500ML | |
| DotReaderTM | Medsaic | pas disponible | |
| L'albumine sérique bovine | Sigma-Aldrich | A9418-10G | |
| Inactivés par la chaleur du sérum AB 2% | Invitrogen | 34005100 | |
| Phycoérythrine conjuguée CD3 | Beckman Coulter | ET386 | |
| AlexaFluor647 conjugué EpCAM | BioLegend | 324212 | |
| Typhon FLA 9000 | GE Healthcare | 28-9558-08 | 532 nm laser, 580, BP30 émission pour les PE filtre. 633 nm laser et 670 BP30 émissions pour Alexa647 filtre |
| MultiExperiment Viewer v4.4 | TM4 Microarray Suite logicielle | Open - source (Réf 11) |
Références
- Steinert, R., Buschmann, T., vander Linden, M., Fels, L. M., Lippert, H., Reymond, M. A. The role of proteomics in the diagnosis and outcome prediction in colorectal cancer. Technol. Cancer. Res. Treat. 1, 297 (2002).
- Eifel, P., Axelson, J. A., Costa, J., Crowley, J., Curran, W. J., Deshler, A., Fulton, S., Hendricks, C. B., Kemeny, M., Kornblith, A. B., Louis, T. A., Markman, M., Mayer, R., Roter, D. National Institutes of Health Consensus Development Conference Statement: adjuvant therapy for breast cancer. J. Natl. Canc. Inst. 93, 979 (2001).
- Swerdlow, S. H., Campo, E., Harris, H. L., Jaffe, E. S., Pileri, S. A., Stein, H., Thiele, J., Vardiman, J. W. WHO classification of tumour of haematopoietic and lymphoid tissues. IARC WHO Classification of Tumours. 2, (2008).
- Xiao, G. G., Recker, R. R., Deng, H. W. Recent advances in proteomics and cancer biomarker discovery. Clin. Med. Oncol. , (2008).
- Belov, L., Mulligan, S. P., Barber, N., Woolfson, A., Scott, M., Stoner, K., Chrisp, J. S., Sewell, W. A., Bradstock, K. F., Bandall, L., Pascovici, D. S., Thomas, M., Erber, W., Huang, P., et al. Analysis of human leukaemias and lymphomas using extensive immunophenotypes from an antibody microarray. Br. J. Haematol. 135, 184 (2006).
- Belov, L., Huang, P., Barber, N., Mulligan, S. P., Christopherson, R. I. Identification of repertories of surface antigens on leukemias using an antibody microarray. Proteomics. 3, 2147 (2003).
- Zhou, J., Belov, L., Huang, P. Y., Shin, J., Solomon, M. J., Chapuis, P. H., Bokey, L., Chan, C., Clarke, C., Clarke, S. J., Christopherson, R. I. Surface antigen profiling of colorectal cancer using antibody microarrays with fluorescence multiplexing. J. Immunol. Methods. 355 (1-2), 40-51 (2010).
- Ellmark, P., Belov, L., Huang, P., Lee, C. S., Solomon, M. J., Morgan, D. K., Christopherson, R. I. Multiplex detection of surface molecules on colorectal cancers. Proteomics. 6, 1791 (2006).
- Pearson, J. P., Allen, A., Hutton, D. A. Rheology of mucin. Methods Mol. Biol. 125, 99 (2000).
- Yang, Y. H., Dudoit, S., Luu, P., Lin, D. M., Peng, V., Ngai, J., Speed, T. P. Normalization for cDNA microarray data: a robust composite method addressing single and multiple slide systematic variation. Nucleic Acids Res. 30, 15 (2002).
- Al Saeed, ., et al. TMA: A free, open-source system for microarray data management and analysis. BioTechniques. 34, 374-378 (2003).
Erratum
Formal Correction: Erratum: Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing
Posted by JoVE Editors on 7/03/2015. Citeable Link.
The author's email has been corrected in the publication of Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing. There was an error with the author, Jerry Zhou's, email. The author's email has been updated to:
j.zhou@uws.edu.au
from:
jzho7551@mail.usyd.edu.au
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon