A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
סרטן המעי הגס Cell Surface חלבון פרופיל שימוש microarray ו ריבוב נוגדן פלואורסצנטי
In This Article
Erratum Notice
Summary
אנו תיאר הליך disaggregation של סרטן המעי (CRC) לייצר תאים בודדים קיימא, אשר נלכדים אז על microarrays נוגדן אישית הכרה אנטיגנים השטח (DotScan microarray CRC). תת אוכלוסיות של תאים חייב microarray ניתן צדודית על ידי ריבוב הקרינה באמצעות נוגדנים חד שבטיים מתוייגים עם צבעי ניאון.
Abstract
הפרוגנוזה הסיווג הנוכחי של CRC מסתמך על היערכות מערכות המשלבים היסטופתולוגיות ממצאים קליניים. עם זאת, ברוב המקרים CRC, תפקוד לקוי של התא היא תוצאה של מוטציות רבים לשנות ביטוי חלבון ופוסט translational שינוי 1.
מספר פני השטח אנטיגנים התא, כולל מקבץ של (CD) אנטיגנים בידול, זוהו סמנים פרוגנוסטיים פוטנציאליים או גרורתי CRC. אנטיגנים אלה הופכים סמנים אידיאלי כביטוי שלהם לעתים קרובות שינויים עם התקדמות הגידול או אינטראקציות עם סוגי תאים אחרים, כגון גידולים הסתננות לימפוציטים (TILs) לבין גידולים הקשורים מקרופאגים (TAMs).
השימוש אימונוהיסטוכימיה (IHC) לטיפול בסרטן תת סיווג התחזית מבוססת היטב של כמה סוגי גידולים 2,3. עם זאת, לא "סמן" יחיד הוכיח משמעות פרוגנוסטית יותר מאשר הזמני המרפאה-פתולוגי או צבר הסכמה רחבה לשימוש דיווח הפתולוגיה שגרתי בכל המקרים CRC.
גישה האחרונות יותר ריבוד פרוגנוסטיים של המחלה פנוטיפים מסתמך על פני השטח באמצעות חלבון פרופילים מרובים "סמנים". בעוד פרופיל הביטוי של גידולים תוך שימוש בטכניקות proteomic כגון iTRAQ הוא כלי רב עוצמה על גילוי biomarkers4, זה לא אופטימלי לשימוש שגרתי במעבדות אבחון אינה יכולה להבחין סוגי תאים שונים באוכלוסייה מעורבת. בנוסף, כמויות גדולות של רקמת הגידול נדרשים עבור פרופיל של קרום גליקופרוטאינים מטוהרים פלזמה בשיטות אלה.
בסרטון הזה תיארנו שיטה פשוטה אפיון פני השטח של תאים proteome קיימא מן disaggregated דגימות CRC באמצעות microarray CRC DotScan נוגדן. Microarray 122-נוגדן מורכב באזור 82-נוגדן תקן להכיר מגוון של השושלת הסמנים הספציפיים ליקוציט, מולקולות הדבקה, קולטנים סמנים של דלקת התגובה החיסונית 5, יחד עם האזור לווין לגילוי של 40 סמנים פרוגנוסטיים פוטנציאלי עבור CRC . תאים הם כבשו רק על נוגדנים שהם מבטאים את האנטיגן המתאים. צפיפות התאים לכל נקודה, נקבע על ידי סורק אופטי, משקף את חלקם של התאים לבטא כי אנטיגן, רמת הביטוי של אנטיגן ואת הזיקה של הנוגדן 6.
עבור רקמת רירית המעי CRC או רגיל, סורק אופטי לשקף את immunophenotype של אוכלוסיות מעורבות של תאים. ריבוב Fluorescence לאחר מכן ניתן להשתמש בפרופיל הנבחר תת אוכלוסיות של תאים בעלי עניין שנתפסו על המערך. לדוגמה, 647-Alexa אנטי אפיתל הידבקות התא מולקולות (EpCAM; CD326), הוא אנטיגן הפאן אפיתל בידול ששימש לזהות תאים CRC גם לתאי האפיתל של רירית המעי נורמלי, בעוד Phycoerythrin אנטי-CD3, שימש כדי לזהות חדירה לתאי T-7. CRC DotScan microarray צריך להיות טיפוס אחר חלופה אבחון למערכת CRC אנטומית מבוססי הזמני.
Protocol
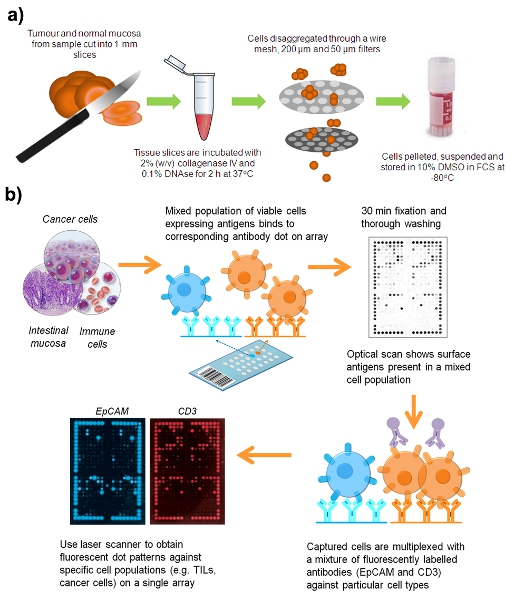
1. תרשים זרימה עבודה להכנת השעיה של תאים חיים ממדגם כירורגית של CRC.
1. מדגם קלינית disaggregation
כל הדגימות נאספו הנסיך המלכותי אלפרד החולים (קמפרדאון, NSW, אוסטרליה) ובית החולים והרפטריאציה קונקורד (Concord המערבית, NSW, אוסטרליה) עם הסכמה מדעת לפי פרוטוקול מס '164-X08.
- איסוף סרטן המעי הגס טרי (CRC) או דגימות אדנומה, ואת רירית המעי נורמלי לפחות 10 ס"מ מן הגידול. חנות דגימות ה-pH מאוזן של האנק מלח פתרון 7.3 (HBSS) בשעה 4 מעלות צלזיוס במשך שעות עד 12 לאחר כריתה.
- עקוב אחר תקנות הבטיחות עבור פתוגנים אנושיים, כל תהליך דוגמאות קליניות בארון ביולוגי בטיחות בכיתה השנייה. מנתחים את הדגימות לתוך 2 קוביות מ"מ בצלחת פטרי באמצעות שני להבי אזמל.
- דגירה הגידול לבין הרקמות הבריאות של צינורות נפרדים Eppendorf עם ערבוב עדין מדי פעם במשך 60 דקות ב 37 מעלות צלזיוס עם נפח שווה של המדיום 1640 RPMI המכיל 2% (v / v) סוג collagenase 4 (וורטינגטון, לייקווד, ניו ג'רסי, ארה"ב) ו - 0.1 (w / v)% deoxyribonuclease אני מ pancrease שור (DNAse אני; סיגמא אולדריץ ').
- חיל מעוכל למחצה רקמה דרך מסננת רשת תיל דק באמצעות הבוכנה של מזרק 10 מ"ל; תאים דרך לשטוף עם HBSS.
- Pass כתוצאה ההשעיה התא דרך 200 מיקרומטר ו 50 פילטרים Filcon מיקרומטר (BD Biosciences) כדי להסיר אגרגטים התא. רוב אגרגטים, DNA ריר תא יוסרו בסדרה זו של filtrations.
- צנטריפוגה השעיות התא XG ב 400 ° 20 דקות 5.
- כדורי תא Resuspend בחום FCS-מומת המכיל sulphoxide דימתיל 10% (DMSO), להקפיא לאט cryovials ולאחסן ב -80 מעלות. תהליך הקפאה נוטה להפחית ליחה המדגם lyses כדוריות דם אדומות.
2. לדוגמא כהכנה ללכוד תאים
- להפשיר דגימות במהירות באמבט 37 מעלות מים ותאי resuspend ב 10 מ"ל של HBSS לשטוף את DMSO.
- צנטריפוגה השעיות התא XG ב 410 ° 20 דקות 5.
- למזוג supernatant ו resuspend התא גלולה ב 500 μL של HBSS.
- פנקו את המדגם עם 0.1% (w / v) DNAse אני עבור 20 דקות בטמפרטורת החדר.
- מערבבים 10 μL ההשעיה כל תא עם נפח שווה של trypan כחול לטעון 10 μL של התערובת לתוך hemocytometer. שימוש במיקרוסקופ אור בהגדלה של פי 100, לספור תאים קיימא, אשר מופיעים ברור עקב הרחקה trypan כחול, בעוד תאים מתים לקחת את צבען. מינימום 4 x 10 6 תאים קיימא נדרש ללכוד תאים על microarray.
- בעקבות טיפול DNAse, resuspend ההשעיה תא HBSS 10 מ"ל ו 410 צנטריפוגות ב XG ב ° 20 במשך 5 דקות.
- למזוג supernatants וכדוריות תא resuspend ב RPMI 1640 עד נפח סופי של 200 μL.
3. Microarray נוגדן תא ללכוד
- לחלח microarray נוגדן DotScan על ידי טבילה בסעיף nitrocellulose לתוך בופר פוספט (PBS) למשך כ 20 שניות לנגב בזהירות את שולי כוס microarray עם Kimwipes מקופל, הימנעות נוגע בסעיף nitrocellulose.
- מוסיפים מים למגש הדגירה microarray לספק קאמרית לח. מניחים את microarray החדרה ו פיפטה ההשעיה תא RPMI 1640 על החלק nitrocellulose לח. פיפטה טיפות בכל פינה של nitrocellulose כדי להבטיח להתפשט גם של תאים.
- דגירה microarrays ב 37 ° C עבור 1 ח הדגירה מאפשר לתאים להתיישב לבוא במגע עם נוגדנים על microarray. תאים להביע את פני השטח אנטיגנים המתאימים את הנוגדנים הם קרקע יהיה בשבי.
- לאחר דגירה, לטבול את microarrays בעדינות אנכית לשלושה שקתות המכיל לפחות 15 מ"ל PBS כדי לשטוף את התאים מאוגד (20 s לכל לשטוף).
- הכן 3.7% (w / v) הפורמלין PBS לתקן את התאים נוגדנים על ידי cross-linking. בעדינות פיפטה כ 1 מ"ל על מנת לכסות את סעיף nitrocellulose של microarray. דגירה של 20 דקות בטמפרטורת החדר.
- הבא לטבול microarrays לתוך 3 שינויים של PBS (15 מ"ל, 30 של כל אחד) כדי לשטוף את עודפי פורמלדהיד.
- נגבו את הקצוות האחוריים של שקופיות הזכוכית עם Kimwipes ולסרוק את microarray באמצעות סורק תוך DotScan, בסעיף nitrocellulose לח. סריקה אופטית מספק את הביטוי אנטיגן דפוס של אוכלוסיה מעורבת תאים תאים למשל CRC, לויקוציטים ותאי סטרומה אחרים של הגידול.
4. Fluorescence ריבוב
- הסר את microarray מהסורק וליישם 200 μL של חיץ חסימה (2% w / v BSA, 2% חום אנושי מומת א.ב. בסרום, PBS, pH 7.3). דגירה זה במגש microarray בטמפרטורת החדר למשך 20 דקות.
- הכן את multiplexing פתרון בצינור Eppendorf מכוסה בנייר אלומיניום: 20 μL Phycoerythrin-אנטי CD3 (Beckman Coulter, Gladesville, NSW, אוסטרליה, # IM12824: 1/7.5 הדילול הסופי), 10 μL אלקסה פלואוריד 647-אנטי EpCAM (Biolegend, בסן דייגו, קליפורניה, ארה"ב; 1 / 15 דילול), 2 μL של חום אנושי מומת א.ב. בסרום (סיגמא אולדריץ, Castle Hill, NSW, אוסטרליה) ו 118 μL של חיץ חסימה.
- מסננים את החיץ חסימת עודף מ microarray ו פיפטה פתרון רבוב על הקטע nitrocellulose, מתפשט באופן אחיד. דגירה למשך 30 דקות בחושך בטמפרטורת החדר.
- טובלים את microarray אנכית לשלושה שקתות של 15 מ"ל PBS טרי; (30 של כל אחד).
- בואו microarrays היבש בחושך חנות ב 4 ° C בתיבת שקופיות. Microarray ניתן לאחסן בחושך עד 3 חודשים ללא הפסד של הקרינה.
- סרוק את microarray יבש באמצעות FLA טייפון 9000 סורק (GE Healthcare, Rydalmere, NSW, אוסטרליה) עם רזולוציה להגדיר עד 50 (532 ננומטר לייזר, 580 BP30 פליטת לסנן PE. הלייזר 633 nm ו 670 BP30 פליטת סינון עבור Alexa 647). Microarrays נסרקים עם הצד nitrocellulose פונה כלפי מטה על מגש זכוכית הסורק.
- שמור את התמונות פלורסנט כקובצי TIFF באמצעות תמונת Photoshop גודל מוגדר 17 x 25 ס"מ ורזולוציה עד 72 פיקסלים / ס"מ. ייבוא התמונה אל התוכנה ניתוח DotScan לנתח את העוצמה של נקודות.
- DotReader לוכדת תמונה דיגיטלית של הדפוס מחייבת נקודה מכמת את צפיפות התאים מחייב על כל נקודה נוגדן בקנה מידה האפרוריות 8 סיביות (1-256 U). השליטה מזדמנים שאינם ספציפיים מחייב אלוטיפ היה מופחתים הערכים מחייב נוגדנים עם isotypes אימונוגלובולינים המקביל. עוצמות הקרינה עבור דוט microarray כל היו מנורמל נגד נקודה הבהיר סט בעוצמה של 100%. האות / נקודה כוח נרשם בקובץ XML. (נתונים גולמיים) או כפי שהוא מיוצג בתרשים בר. קובץ PDF (הדו"ח הסופי)
- Microarray heatsmaps ו באשכולות היררכי נערכו באמצעות MultiExperiment Viewer (MeV) גרסה 4.4 של TM4 microarray Software Suite ( http://www.tm4.org/mev.html ). אשכולות היררכי בוצעה על רקע נתונים מנוכי באמצעות MeV עם ניתוח הצמדה מלאה. המרחק האוקלידית שימש למדידת הדמיון. סטודנט 2 זנב של t-מבחן עם שונות שווה שימש כדי לקבוע את המובהקות הסטטיסטית של התוצאות.
5. נציג תוצאות:
תוצאות מ microarray DotScan צריך להראות דפוסים תא עקבי מחייב בין מערכים כפולים. נקודה חזקה יישור מחייב (CD44/CD29) מאפשרת לרשת להציב על שטח המערך. תרשים 2 מציג דוגמה ללכוד תאים ריבוב אופטימלית. איור 3 מראה כמה בעיות נפוצות נתקל במהלך ללכוד תאים ועל הפתרונות האפשריים.
התוצאות תא microarray מחייב ניתן לכמת על ידי מדידת עוצמות נקודה הביע בקנה מידה האפרוריות בטווח שבין 1 ל 256. איור 4 מראה נתונים מספריים מתוך 58 דגימות כירורגית CRC, מוכתם נוגדן 647 EpCAM-Alexa, כמו Heatmap עם אשכולות היררכי. למרות מספר דגימות מוגבל, CRCs הבמה באותו נוטים אשכול באותה קבוצה.
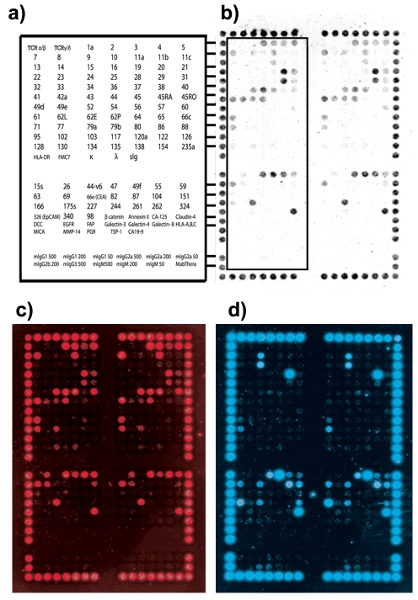
איור 2. Cell דפוס מחייב קלינית של הגידול בסרטן המעי הגס (Staging האוסטרלי Clinic-פתולוגי, ACP הבמה B1). (א) DotScan מפתח נוגדנים מראה מקומות של נוגדנים לחצי השמאלי של microarray כפולות (בקווים כלליים). המקטע העליון מכיל את 82 המקורי נוגדנים של microarray הלוקמיה DotScan. נוסף 40 נוגדנים, המתאים אנטיגנים ספציפיים השטח נמצא למעלה מוסדר בספרות, נוספו כמו microarray "לווין" CRC. המקטע התחתון מכיל נוגדנים לשלוט אלוטיפ (ב) תמונה אופטי של תאים CRC מחייב microarray. (ג) CD3 תמונה הקרינה מראה מתאי T. (ד) EpCAM הקרינה תמונת מראה תאים CRC.

איור 3. דוגמאות של תוצאות DotScan עניים פתרונות אפשריים. (א) תא נמוכה מחייב; פתרון: לוודא לפחות 4x106 קיימא הם תאים במערך (ב) תא אלוטיפ לשלוט מחייב הלא ספציפית מחייב; פתרון: להוסיף החום האנושי מומת א.ב. בסרום לטעום לפני הדגירה על microarray כדי למזער אלוטיפ לשלוט מחייב. מדי פעם, כמות קטנה של הכריכה הלא ספציפית של תאים nitrocellulose מתרחשת עם דגימות CRC ואינו להשפיע באופן משמעותי על התוצאות. (ג) Nitrocellulose להתייבש במהלך הדגירה; הפיתרון: להבטיח מדגם מכסה את סעיף nitrocellulose שלם microarray הוא מודגרות על משטח שטוח. (ד) ממצאים רקע גבוהה; הפיתרון: להבטיח אתmicroarray הוא שטף ביסודיות הדגירה הבאה.
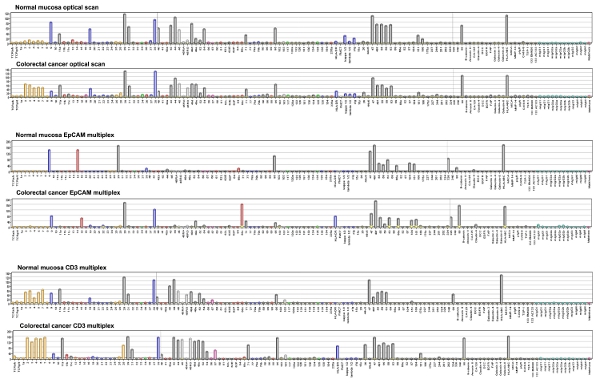
איור 4. DotScan תוכנה לניתוח שנוצר תרשימים בר המייצגים צפיפות תא מחייב בקנה מידה האפרוריות בטווח שבין 1 ל 256. מספרים על ציר מתייחסים אנטיגנים CD. קיצורים אחרים TCR, T-cell receptor, κ, λ, אור אימונוגלובולינים שרשראות, SIG, אימונוגלובולינים פני השטח; DCC, שנמחקו חלבון סרטן המעי הגס; EGFR, אפידרמיס קולטן גורם גדילה, FAP, הפעלת חלבון פיברובלסטים, HLA-A, B, C-HLA-DR, אנטיגנים לויקוציטים אנושיים DR ו A, B, C בהתאמה, מיקה, MHC בכיתה אני שרשרת הקשורות חלבון, MMP-14, metallopeptidase מטריקס 14; PIGR, קולטן אימונוגלובולינים פולימריות, כפית-1, thrombospondin-1; Mabthera, humanised אנטי CD20. לחץ כאן כדי להציג תמונה גדולה יותר .
Discussion
בסרטון הזה אנחנו מדגימים איך microarray נוגדן DotScan ניתן להשתמש דרך פשוטה וכמותיות, ללמוד אנטיגן פרופילים השטח עבור אוכלוסיות תאים מרקמות CRC.
קבלת השעיה בת קיימא תא בודד מרקמת הוא קריטי להצלחה של הניסוי, כי האנרגיה תלויה בתהליכים (למשל, ...
Disclosures
אין ניגודי אינטרסים הכריז.
Acknowledgements
אנו מודים לצוות על מעבדות פתולוגיה אנטומית של הנסיך המלכותי אלפרד חולים קונקורד והרפטריאציה לאיסוף דגימות טריים של CRC ו רירית מעיים רגילה. העבודה מומן על ידי מענק מכון הסרטן בניו דרום ויילס תוכנית Translational.
Materials
| Name | Company | Catalog Number | Comments |
| שם מגיב או ציוד | חברה | מספר קטלוגי | תגובות |
|---|---|---|---|
| הנקס "פתרון מאוזן מלח | סיגמא אולדריץ | H6136-10X1L | שנאגרו עם 25 Hepes מ"מ (Sigma # H3375) |
| Airpure בטיחות ביולוגית ארון מעמד II | Westinghouse | 1687-2340/612 | |
| כירורגי להבי | ליווינגסטון | 090609 | חבילת של 100 |
| RPMI 1640 עם 2 mM Hepes | סיגמא אולדריץ | R4130-10X1L | |
| Collagenase סוג 4 | וורטינגטון | 4188 | |
| Deoxyribonuclease 1 | סיגמא אולדריץ | DN25-1G | |
| Terumo מזרק (10 מ"ל) | Terumo | אס 10 L | קופסא של 100 |
| Filcon מסנן (200 מיקרומטר) | BD Biosciences | 340615 | |
| Filcon מסנן (50 מיקרומטר) | Filcon מסנן (50 מיקרומטר) | Filcon מסנן (50 מיקרומטר) Filcon מסנן (50 מיקרומטר) 340,603 | |
| עגל עוברית בסרום | Gibco / Invitrogen | 10099-141 | |
| צנטריפוגה 5810 R | Eppendorf | 7017 | |
| דימתיל sulphoxide | סיגמא אולדריץ | D2650 | |
| Trypan כחול | סיגמא אולדריץ | T8154 | |
| Hemocymeter Technocolor Neubar | הירשמן | לא זמין | |
| מיקרוסקופ אור | ניקון | Nikon TMS | |
| Cyrovial צינורות | גריינר ביו אחד | 121278 | |
| Cryo contrainer הקפאת | Nalgene | 5100-0001 | |
| DotScan נוגדן microarray ערכת | Medsaic | לא זמין | |
| DotScan microarray מגש לשטוף | Medsaic | לא זמין | |
| KimWipes | קימברלי קלארק | 4103 | |
| פורמלדהיד 37% | סיגמא אולדריץ | F1635-500ml | |
| DotReaderTM | Medsaic | לא זמין | |
| שור בסרום אלבומין | סיגמא אולדריץ | A9418-10G | |
| חום מומת א.ב. בסרום 2% | Invitrogen | 34005100 | |
| Phycoerythrin מצומדות-CD3 | Beckman Coulter | ET386 | |
| AlexaFluor647-מצומדות EpCAM | BioLegend | 324212 | |
| טייפון FLA 9000 | GE Healthcare | 28-9558-08 | לייזר 532 ננומטר, 580 BP30 פליטת לסנן PE. 633 ננומטר לייזר 670 BP30 פליטת לסנן Alexa647 |
| MultiExperiment Viewer v4.4 | TM4 microarray חבילת תוכנה | פתח - תוכנת קוד (אסמכתא 11) |
References
- Steinert, R., Buschmann, T., vander Linden, M., Fels, L. M., Lippert, H., Reymond, M. A. The role of proteomics in the diagnosis and outcome prediction in colorectal cancer. Technol. Cancer. Res. Treat. 1, 297 (2002).
- Eifel, P., Axelson, J. A., Costa, J., Crowley, J., Curran, W. J., Deshler, A., Fulton, S., Hendricks, C. B., Kemeny, M., Kornblith, A. B., Louis, T. A., Markman, M., Mayer, R., Roter, D. National Institutes of Health Consensus Development Conference Statement: adjuvant therapy for breast cancer. J. Natl. Canc. Inst. 93, 979 (2001).
- Swerdlow, S. H., Campo, E., Harris, H. L., Jaffe, E. S., Pileri, S. A., Stein, H., Thiele, J., Vardiman, J. W. WHO classification of tumour of haematopoietic and lymphoid tissues. IARC WHO Classification of Tumours. 2, (2008).
- Xiao, G. G., Recker, R. R., Deng, H. W. Recent advances in proteomics and cancer biomarker discovery. Clin. Med. Oncol. , (2008).
- Belov, L., Mulligan, S. P., Barber, N., Woolfson, A., Scott, M., Stoner, K., Chrisp, J. S., Sewell, W. A., Bradstock, K. F., Bandall, L., Pascovici, D. S., Thomas, M., Erber, W., Huang, P., et al. Analysis of human leukaemias and lymphomas using extensive immunophenotypes from an antibody microarray. Br. J. Haematol. 135, 184 (2006).
- Belov, L., Huang, P., Barber, N., Mulligan, S. P., Christopherson, R. I. Identification of repertories of surface antigens on leukemias using an antibody microarray. Proteomics. 3, 2147 (2003).
- Zhou, J., Belov, L., Huang, P. Y., Shin, J., Solomon, M. J., Chapuis, P. H., Bokey, L., Chan, C., Clarke, C., Clarke, S. J., Christopherson, R. I. Surface antigen profiling of colorectal cancer using antibody microarrays with fluorescence multiplexing. J. Immunol. Methods. 355 (1-2), 40-51 (2010).
- Ellmark, P., Belov, L., Huang, P., Lee, C. S., Solomon, M. J., Morgan, D. K., Christopherson, R. I. Multiplex detection of surface molecules on colorectal cancers. Proteomics. 6, 1791 (2006).
- Pearson, J. P., Allen, A., Hutton, D. A. Rheology of mucin. Methods Mol. Biol. 125, 99 (2000).
- Yang, Y. H., Dudoit, S., Luu, P., Lin, D. M., Peng, V., Ngai, J., Speed, T. P. Normalization for cDNA microarray data: a robust composite method addressing single and multiple slide systematic variation. Nucleic Acids Res. 30, 15 (2002).
- Al Saeed, ., et al. TMA: A free, open-source system for microarray data management and analysis. BioTechniques. 34, 374-378 (2003).
Erratum
Formal Correction: Erratum: Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing
Posted by JoVE Editors on 7/03/2015. Citeable Link.
The author's email has been corrected in the publication of Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing. There was an error with the author, Jerry Zhou's, email. The author's email has been updated to:
j.zhou@uws.edu.au
from:
jzho7551@mail.usyd.edu.au
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved