JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
항체 Microarray 및 형광 멀티플렉싱을 사용하여 대장암 세포 표면 단백질 프로파일
Erratum Notice
요약
그러면 표면 항원 (DotScan CRC microarray)를 인식 맞춤 항체 microarrays에 캡처되어 가능한 단일 세포를 생산하는 대장암 (CRC)의 disaggregation에 대한 절차를 설명했다. microarray에 바인딩된 세포의 하위 인구는 형광 염료와 태그 단클론 항체를 사용하여 형광 멀티플렉싱하여 프로파일 수 있습니다.
초록
CRC의 현재 예측과 분류 histopathologic 및 임상 결과를 통합하는 시스템을 준비에 의존합니다. 그러나, CRC 케이스의 대다수에서, 세포 기능 장애는 단백질 표현과 사후 translational 수정 수정 1 수많은 돌연변이의 결과이다.
차별화 (CD) 항원의 클러스터를 포함하여 세포 표면 항원,의 숫자 CRC 잠재적인 전조 또는 전이성 생체으로 확인되었습니다. 이러한 항원들은 종종 자신의 표현으로 종양의 진행이나 다른 세포 유형, 같은과 상호 작용과 변화를 이상적인 생체를 종양 - 침투 lymphocytes (TILs)와 종양 - 관련 macrophages (리버 탐)를.
암 하위 분류와 예지를위한 immunohistochemistry (IHC)의 사용은 물론 일부 종양 유형 2,3에 대해 설정됩니다. 그러나, 하나의 '표지'는 clinico - 병적인 준비 이상의 전조 중요성을 표시하거나 모든 CRC 케이스의 일상 병리보고에 사용하기 위해 광범위한 수용을 받고있다.
질병 phenotypes의 전조 충화에 더 최근의 접근 방법은 여러 '표시'를 사용하는 표면 단백질 프로파일에 의존합니다. 같은 iTRAQ로 proteomic 기술을 이용하여 종양의 표현 프로 파일링이 biomarkers4의 발견을위한 강력한 도구이지만, 그것은 진단 실험실에서 일상적인 사용을위한 최적되지 않고 혼합 인구의 서로 다른 세포 유형을 구분할 수 없습니다. 또한, 종양 조직의 대량는 이러한 방법으로 정화 플라즈마 막 glycoproteins의 프로파일이 필요합니다.
이 비디오에서 우리는 DotScan CRC의 항체 microarray를 사용하여 disaggregated CRC 샘플에서 가능한 세포의 표면 프로테옴 프로 파일링위한 간단한 방법을 설명했다. 122 - 항체 microarray는 CRC 40 잠재적인 전조 마커의 검출을위한 위성 지역과 함께 혈통 특정 백혈구 마커, 접착 분자, 수용체 및 염증 및 면역 반응 5 마커의 범위를 인식 표준 82 - 항체 영역으로 구성되어 있습니다 . 세포는 그들이 해당 항원을 표현하는 항체에 캡처됩니다. 광학 스캔에 의해 결정 점 당 세포 밀도, 그 항원, 항체 6 항원과 친화력의 표현의 수준을 표현하는 세포의 비율을 반영합니다.
CRC 조직이나 정상적인 창자 점막 들어, 광학 스캔은 세포의 혼합 인구의 immunophenotype을 반영합니다. 형광 멀티플렉싱은 다음 배열에 캡처 관심 세포의 선택된 하위 인구 프로파일에 사용할 수 있습니다. 예를 들어, 알렉사 647 - 안티 상피 세포 유착 분자 (EpCAM, CD326)는 Phycoerythrin - 안티 인 경우에는 3 번 CD에 들어, 사용하는 동안, CRC 세포뿐만 아니라 정상적인 창자 점막의 상피 세포를 감지하는 데 사용한 냄비 - 상피 분화 항원이다 T - 세포 7 침투 감지합니다. DotScan CRC microarray는 해부학적인 몸의 구조 기반 CRC의 준비 시스템 진단 대안에 대한 프로토 타입을해야합니다.
프로토콜
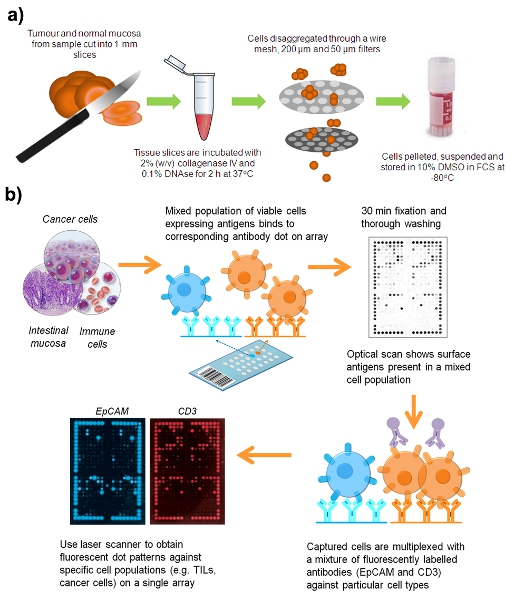
CRC의 수술 표본에서 살고 세포 현탁액의 준비를 위해 그림 1. 워크플로우.
1. 임상 샘플 disaggregation
모든 샘플은 프로토콜 번호 X08 - 164 아래의 정보를 동의를 얻어 로얄 프린스 알프레드 병원 (Camperdown, NSW, 호주)과 콩코드 송환 병원 (콩코드 웨스트, NSW, 호주)에서 수집되었다.
- 신선한 대장암 (CRC) 또는 adenoma의 표본, 그리고 정상적인 창자 점막 종양에서 적어도 10cm를 수집합니다. 절제술 후 최대 12 H 4에서 행크의 균형 소금 솔루션 산도 7.3 (HBSS) ° C에서 저장 샘플.
- 생물 학적 안전 캐비닛 클래스 II의 모든 임상 샘플 처리, 인간의 병원체에 대한 안전 규정을 따르십시오. 이 메스 블레이드를 사용하여 배양 접시에있는 2mm 큐브에 샘플을 해부하다.
- 37 60 분 가끔 부드러운 혼합 별도의 Eppendorf 튜브에 종양과 정상 조직을 품어 ° 2% (V / V) collagenase 타입 4 (워싱턴, 레이크 우드, 뉴저지, 미국)와 0.1이 포함된 RPMI 1640 배지의 동일한 볼륨 C 소 pancrease에서 % (W / V) deoxyribonuclease I (DNAse I, 시그마 - 올드 리치).
- 포스 10 ML의 주사기에서 플런저를 사용하여 미세 와이어 메쉬 스트레이너를 통해 조직을 준 소화, HBSS로 세척을 통해 세포.
- 셀 집계를 제거 200 μm의 50 μm의 Filcon 필터 (BD Biosciences)를 통해 세포 현탁액을 결과 전달합니다. DNA, 점액 및 세포 집계의 대부분은 filtrations이 시리즈 제거됩니다.
- 5 분 20 °에서 400 XG에서 세포 정지를 원심 분리기.
- 10% 디메틸 sulphoxide (DMSO)를 포함하는 열 inactivated FCS에서 Resuspend 세포 산탄은 -80 °에서 cryovials과 가게에서 천천히 냉동. 동결 과정은 샘플에서 점액을 감소하는 경향과 적혈구를 lyses.
2. 세포 캡처를위한 샘플 준비
- DMSO를 씻어 HBSS 10 ML에서 37 ° 물 욕조, resuspend 세포에 신속하게 샘플을 좀 녹여.
- 5 분 20 °에서 410 XG에서 세포 정지를 원심 분리기.
- 뜨는을 가만히 따르다하고 HBSS 500 μL의 세포 펠렛을 resuspend.
- 0.1 % (W / V) 실온에서 20 분 DNAse I로 샘플을 처리합니다.
- trypan 파랑의 동일한 볼륨 각 세포 현탁액의 10 μL를 혼합하고 hemocytometer로 혼합 10 μL를로드합니다. 죽은 세포는 염료를 차지하면서 100 배 배율에서 가벼운 현미경을 사용하여, trypan 파랑 배제로 인해 명확한 표시 가능한 세포를 계산합니다. 최소 4 X 10 6 유력한 세포는 microarray에 셀 캡처가 필요합니다.
- DNAse 처리 다음, 5 분 20 °에서 410 XG 10 ML HBSS와 원심 분리기의 세포 현탁액을 resuspend.
- 200 μL의 최종 볼륨 RPMI 1640에서 supernatants 및 resuspend 세포 알약을 가만히 따르다.
3. 항체 microarray 셀 캡처
- 인산염로 nitrocellulose 섹션을 찍기로 DotScan 항체 microarray를 축축하게하다하면 약 20 S.위한 식염수 (PBS)를 버퍼 조심스럽게 nitrocellulose 섹션을 감동 피하고, 접힌 Kimwipes와 microarray의 유리 가장자리를 닦으십시오.
- 습기 챔버를 제공하기 위해 microarray 보육 트레이에 물을 추가합니다. 챔버에 microarray를 놓고 젖은 nitrocellulose 섹션에 RPMI 1640에 세포 현탁액을 피펫. 피펫은 심지어 세포의 확산하기 위해 nitrocellulose의 각 모서리에 방울.
- 1 H.위한 37 ° C에서 microarrays를 품어 배양은 세포의 microarray에 대한 항체와 접촉에 정착하여 올 수 있습니다. 그들은에 착륙 항체에 해당하는 표면 항원을 표현 전지 캡처됩니다.
- 부화 후, 언바운드 세포 (씻어 당 20의) 씻어 최소 15 ML PBS를 포함하는 세 골짜기로 부드럽게 수직 microarrays을 찍어.
- 교차 연결하여 세포와 항체를 해결하기 위해 3.7 % (W / V) PBS에 포름 알데히드를 준비합니다. 부드럽게 microarray의 nitrocellulose 부분을 충당하기 위해 약 1 ML을 피펫. 상온 20 분 알을 품다.
- 다음 딥 microarrays PBS (15 ML, 30의 각) 3 변화에 초과하는 포름 알데히드를 씻어 수 있습니다.
- 가장자리와 Kimwipes있는 유리 슬라이드 다시 닦으하고 DotScan 스캐너 동안을 사용하여 microarray를 스캔, nitrocellulose 섹션 촉촉한입니다. 광학 스캔 혼합 세포 인구 예 : CRC 세포, leukocytes 및 종양의 다른 stromal 세포의 항원 표현 패턴을 제공합니다.
4. 멀티플렉싱 형광
- 스캐너에서 microarray를 제거하고 차단 버퍼 (2 % W / V BSA, 2퍼센트 열 inactivated 인간 AB 묘약, PBS, pH를 7.3) 200 μL를 적용합니다. 20 분 상온에서 microarray 트레이에 품어.
- multip 준비(Biolegend, 10 μL 알렉사 형석 647 - 안티 EpCAM, 20 μL Phycoerythrin - 안티 인 경우에는 3 번 CD (1/7.5 최종 희석 베크 만 보습 바로 앞에 달린 풀베는 날, Gladesville, NSW, 호주, # IM12824) : 알루미늄 호일로 덮여 Eppendorf 튜브에 솔루션을 lexing 샌디에이고, CA, 미국, 15분의 1 희석), 열 inactivated 인간 AB 혈청 (시그마 - 올드 리치, 성 힐, NSW, 호주)와 차단 버퍼 118 μL 2 μL.
- microarray에서 초과 차단 버퍼를 배수하고 균일하게 확산, nitrocellulose 섹션에 멀티플렉싱 솔루션을 피펫. 상온에서 어두운 곳에 30 분 알을 품다.
- 세로 15 ML 신선한 PBS의 세 골짜기로 microarray를 찍어, (30의 각).
- 4 어둡고 저장소에있는 microarrays가 건조 해 ° 슬라이드 상자에 C. microarray는 형광의 손실없이 최대 3 개월 동안 어둠에 저장할 수 있습니다.
- 50 설정된 해상도 태풍 FLA 9000 스캐너 (GE 헬스케어, Rydalmere, NSW, 호주)를 사용하여 건조 microarray 스캔 (532 nm의 레이저, PE에 대한 580 BP30 방출 필터를. 알렉사 647에 대한 633 NM 레이저와 670 BP30 방출 필터). microarrays은 유리 스캐너 트레이에서 아래쪽으로 직면하고있는 nitrocellulose 측면과 함께 스캔됩니다.
- 17 TIFF 파일과 같은 형광 이미지를 사용하여 포토샵 설정 이미지 크기를 저장하는 것은 X 25cm와 해상도 72 픽셀 / cm합니다. 점들의 강도를 분석하는 DotScan 분석 소프트웨어로 이미지를 가져옵니다.
- DotReader는 점 바인딩 패턴의 디지털 이미지를 캡처하고 8 비트 greyness 규모 (1-256 U) 각 항체 점에서 셀 바인딩의 밀도를 quantifies. 수시 아닌 특정 isotype 컨트롤 바인딩이 해당 면역 글로불린과 항체 isotypes에 대한 구속력이 값에서 빼는되었습니다. 각 microarray에 대한 점 형광 농도 100 %의 강도로 설정 밝은 점에 대한 정상화되었습니다. 신호 / 현장 강도. xml 파일 (원시 데이터)에 기록된 또는에서 막대 차트로 표시했습니다. pdf 파일 (최종 보고서)
- Microarray heatsmaps 및 계층적 클러스터링은 TM4 Microarray 소프트웨어 스위트 (에서 MultiExperiment 뷰어 (MeV) 버전 4.4을 사용하여 수행되었다 http://www.tm4.org/mev.html ). 계층적 클러스터링은 전체 연동 분석 MeV를 사용하여 배경 - 조정 데이터에서 수행되었다. 유클리드 거리는 유사성 측정을 위해 사용되었다. 동일한 분산과 함께 2 꼬리 학생의 T - 테스트 결과의 통계적 중요성을 결정하는 데 사용되었다.
5. 대표 결과 :
DotScan의 microarray의 결과는 중복 배열 사이의 일관성을 셀 바인딩 패턴을 표시합니다. 강력한 정렬 도트 바인딩 (CD44/CD29)는 배열의 영역에 배치 될 수있는 격자 수 있습니다. 그림 2는 최적의 세포 캡처 및 멀티플렉싱의 예를 보여줍니다. 그림 3은 세포 캡처 및 가능한 해결 방법 중에 발생한 몇 가지 일반적인 문제를 보여줍니다.
microarray 셀 바인딩 결과는 1부터 256까지 greyness 규모에 표현 점 농도를 측정하여 계량하실 수 있습니다. 그림 4는 계층 클러스터링과 히트맵로 EpCAM - 알렉사 647 항체 물들일 58 수술 CRC 샘플에서 수치 데이터를 나타냅니다. 샘플의 수가 제한에도 불구하고, 동일한 단계 CRCs 같은 그룹에 클러스터하는 경향이있다.
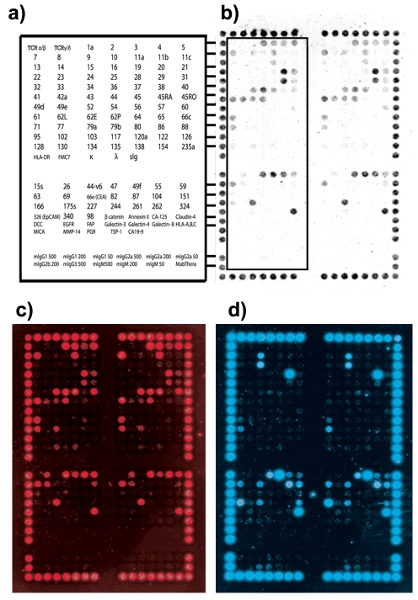
그림 2. 임상 대장암 종양 (호주 클리닉 - 병적 스테이징, ACP 단계 B1)의 세포 결합 패턴. 중복 microarray (설명)의 왼쪽 절반 항체의 위치를 보여주는 (A) DotScan 항체 키. 상단 부분 DotScan의 백혈병 microarray의 원래 82 항체가 포함되어 있습니다. 특정 표면 항원에 해당하는 추가 정보는 40 항체는 CRC '위성'microarray으로 추가되었습니다, 문학에 - 규제로 발견했습니다. 아래 섹션 isotype 제어 항체 microarray에 바인딩 CRC 세포 (B) 광학 이미지로 구성되어 있습니다. T - 세포를 보여주는 (C) 인 경우에는 3 번 CD의 형광 이미지. CRC 세포를 보여주는 (D) EpCAM 형광 이미지.

그림 3. 가난한 DotScan 결과와 가능한 해결 방법의 예. (A) 낮은 셀 바인딩, 솔루션 :; 최소화하기 위해 microarray에 부화하기 전에 샘플로 열 inactivated 인간 AB 혈청을 추가 솔루션 적어도 4x106 가능한 세포 (B) 바인딩 Isotype 컨트롤 바인딩이 아닌 특정 세포가 배열에 있는지 확인하십시오 isotype 제어 바인딩. 때때로, nitrocellulose에 세포가 아닌 특정 바인딩의 작은 금액은 CRC 샘플 발생하고 훨씬 결과에 영향을주지 않습니다. (C) Nitrocellulose이 부화하는 동안 밖으로 건조, 솔루션 : 예제는 전체 nitrocellulose 섹션을 포함하고 microarray는 평평한 표면에 incubated 있는지 확인하십시오. (D) 높은 배경 유물, 솔루션 :을 보장microarray는 철저하게 다음과 부화를 씻어입니다.
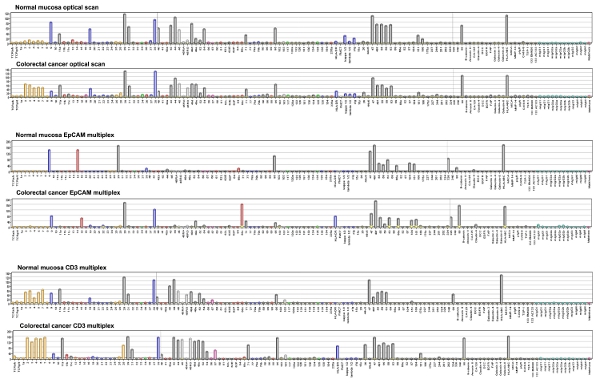
그림 4. DotScan 분석 소프트웨어는 1에서 256까지 greyness 규모 셀 바인딩 밀도를 나타내는 막대 차트를 생성. 축의 숫자는 CD 항원을 참조하십시오. κ, λ, 면역 글로불린 라이트 체인,, 시그마, 표면 면역 글로불린, DCC, 대장암 단백질의 삭제, EGFR, 표피 성장 인자 수용체, FAP, fibroblast 활성화 단백질, HLA - A, B, 기타 약어는 TCR, T - 세포 수용체 아르 A, B, C 각각 C HLA - DR, 인간 백혈구 항원 DR과, 운모, MHC 클래스 I 체인과 관련된 단백질, MMP - 14, 매트릭스 metallopeptidase 14, PIGR, 고분자 면역 글로불린 수용체, TSP - 1, thrombospondin - 1; Mabthera, humanised 안티 CD20. 큰 이미지를 보려면 여기를 클릭하십시오 .
토론
이 비디오에서는, 우리는 DotScan 항체 microarray는 CRC 조직의 세포 집단을위한 표면 항원 프로필을 연구하기위한 간단한 세미 양적 방법으로 사용할 수있는 방법을 보여줍니다.
에너지 종속 프로세스 (예 : 항원이 상한 및 / 또는 pseudopodia 형성) 보육시 항체 점들에 전체 세포의 회사 바인딩에 필요한 것으로 나타납니다 때문에 조직에서 가능한 단일 세포 현탁액 구하기, 실험의 ?...
공개
관심 없음 충돌 선언하지 않습니다.
감사의 말
우리는 로얄 프린스 알프레드와 CRC와 정상적인 장 점막의 신선한 샘플 수집에 대한 콩코드 송환 병원의 해부 병리학 연구소에서 직원을 감사드립니다. 작품은 암 연구소 뉴사우스 웨일즈 Translational 프로그램 권한 부여에 의해 투자되었다.
자료
| Name | Company | Catalog Number | Comments |
| 시약이나 장비의 명칭 | 회사 | 카탈로그 번호 | 댓글 |
|---|---|---|---|
| 행크스 '균형 소금 솔루션 | 시그마 - 올드 리치 | H6136 - 10X1L | 25 MM Hepes (시그마 # H3375)와 버퍼 |
| Airpure 생물 학적 안전 캐비닛 클래스 II | 웨스팅 하우스 | 1687-2340/612 | |
| 외과 블레이드 | 리빙스톤 | 090609 | 100 팩 |
| 이 MM Hepes와 RPMI 1640 | 시그마 - 올드 리치 | R4130 - 10X1L | |
| Collagenase 유형 4 | 워싱턴 | 4188 | |
| Deoxyribonuclease 1 | 시그마 - 올드 리치 | DN25 - 1G | |
| Terumo 주사기 (10 ML) | Terumo | SS 10 L | 100 박스 |
| Filcon 필터 (200 μm의) | BD Biosciences | 340615 | |
| Filcon 필터 (50 μm의) | Filcon 필터 (50 μm의) | Filcon 필터 (50 μm의) Filcon 필터 (50 μm의) 340603 | |
| 태아 송아지 혈청 | Gibco / Invitrogen | 10099-141 | |
| 5810 R을 원심 분리기 | Eppendorf | 7017 | |
| 디메틸 sulphoxide | 시그마 - 올드 리치 | D2650 | |
| Trypan 파랑 | 시그마 - 올드 리치 | T8154 | |
| Hemocymeter Technocolor Neubar | Hirschmann | 사용할 수 없습니다 | |
| 라이트 현미경 | 니콘 | 니콘 TMS | |
| Cyrovial 튜브 | Greiner 바이오 - 원 | 121278 | |
| Cryo의 동결 contrainer | Nalgene | 5100-0001 | |
| DotScan 항체 microarray 키트 | Medsaic | 사용할 수 없습니다 | |
| DotScan microarray 페인트 트레이 | Medsaic | 사용할 수 없습니다 | |
| KimWipes | 킴벌리 - 클락 | 4103 | |
| 37% 포름 알데히드 | 시그마 - 올드 리치 | F1635 - 500ML | |
| DotReaderTM | Medsaic | 사용할 수 없습니다 | |
| 소 혈청 알부민 | 시그마 - 올드 리치 | A9418 - 10G | |
| 열 inactivated AB 혈청 2% | Invitrogen | 34005100 | |
| Phycoerythrin - 복합 인 경우에는 3 번 CD에 들어 | 베크 만 보습 바로 앞에 달린 풀베는 날 | ET386 | |
| AlexaFluor647 - 복합 EpCAM | BioLegend | 324212 | |
| 태풍 FLA 9000 | GE 헬스케어 | 28-9558-08 | 532 NM 레이저, PE에 대한 580 BP30 방출 필터. 633 NM 레이저와 Alexa647에 대한 670 BP30 방출 필터 |
| MultiExperiment 뷰어 v4.4 | TM4 Microarray 소프트웨어 스위트 | 오픈 - 소스 소프트웨어 (참조 11) |
참고문헌
- Steinert, R., Buschmann, T., vander Linden, M., Fels, L. M., Lippert, H., Reymond, M. A. The role of proteomics in the diagnosis and outcome prediction in colorectal cancer. Technol. Cancer. Res. Treat. 1, 297 (2002).
- Eifel, P., Axelson, J. A., Costa, J., Crowley, J., Curran, W. J., Deshler, A., Fulton, S., Hendricks, C. B., Kemeny, M., Kornblith, A. B., Louis, T. A., Markman, M., Mayer, R., Roter, D. National Institutes of Health Consensus Development Conference Statement: adjuvant therapy for breast cancer. J. Natl. Canc. Inst. 93, 979 (2001).
- Swerdlow, S. H., Campo, E., Harris, H. L., Jaffe, E. S., Pileri, S. A., Stein, H., Thiele, J., Vardiman, J. W. WHO classification of tumour of haematopoietic and lymphoid tissues. IARC WHO Classification of Tumours. 2, (2008).
- Xiao, G. G., Recker, R. R., Deng, H. W. Recent advances in proteomics and cancer biomarker discovery. Clin. Med. Oncol. , (2008).
- Belov, L., Mulligan, S. P., Barber, N., Woolfson, A., Scott, M., Stoner, K., Chrisp, J. S., Sewell, W. A., Bradstock, K. F., Bandall, L., Pascovici, D. S., Thomas, M., Erber, W., Huang, P., et al. Analysis of human leukaemias and lymphomas using extensive immunophenotypes from an antibody microarray. Br. J. Haematol. 135, 184 (2006).
- Belov, L., Huang, P., Barber, N., Mulligan, S. P., Christopherson, R. I. Identification of repertories of surface antigens on leukemias using an antibody microarray. Proteomics. 3, 2147 (2003).
- Zhou, J., Belov, L., Huang, P. Y., Shin, J., Solomon, M. J., Chapuis, P. H., Bokey, L., Chan, C., Clarke, C., Clarke, S. J., Christopherson, R. I. Surface antigen profiling of colorectal cancer using antibody microarrays with fluorescence multiplexing. J. Immunol. Methods. 355 (1-2), 40-51 (2010).
- Ellmark, P., Belov, L., Huang, P., Lee, C. S., Solomon, M. J., Morgan, D. K., Christopherson, R. I. Multiplex detection of surface molecules on colorectal cancers. Proteomics. 6, 1791 (2006).
- Pearson, J. P., Allen, A., Hutton, D. A. Rheology of mucin. Methods Mol. Biol. 125, 99 (2000).
- Yang, Y. H., Dudoit, S., Luu, P., Lin, D. M., Peng, V., Ngai, J., Speed, T. P. Normalization for cDNA microarray data: a robust composite method addressing single and multiple slide systematic variation. Nucleic Acids Res. 30, 15 (2002).
- Al Saeed, ., et al. TMA: A free, open-source system for microarray data management and analysis. BioTechniques. 34, 374-378 (2003).
Erratum
Formal Correction: Erratum: Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing
Posted by JoVE Editors on 7/03/2015. Citeable Link.
The author's email has been corrected in the publication of Colorectal Cancer Cell Surface Protein Profiling Using an Antibody Microarray and Fluorescence Multiplexing. There was an error with the author, Jerry Zhou's, email. The author's email has been updated to:
j.zhou@uws.edu.au
from:
jzho7551@mail.usyd.edu.au
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유