Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Células vivas imágenes de la migración de las células que expresan proteínas de fluorescencia etiquetado en una matriz tridimensional
En este artículo
Resumen
Procesos celulares, tales como la migración celular tradicionalmente han sido estudiados en dos dimensiones, las superficies de plástico rígido. Este informe describe una técnica para visualizar de manera directa la localización de proteínas y el análisis de la dinámica de proteínas en las células que migran de una manera más fisiológicamente relevantes, matriz tridimensional.
Resumen
Tradicionalmente, la migración celular se ha estudiado en dos dimensiones, las superficies de plástico rígido. Sin embargo, durante los procesos biológicos como la cicatrización de heridas, regeneración de tejidos y metástasis del cáncer, las células deben navegar a través de complejos de tres dimensiones del tejido extracelular. Para comprender mejor los mecanismos detrás de estos procesos biológicos, es importante examinar las funciones de las proteínas responsables de conducir la migración celular. Aquí, se describe un protocolo para estudiar los mecanismos de migración celular utilizando la línea de células epiteliales (MDCK), y en tres dimensiones, fibroso, la auto-polimerización de la matriz como un sistema modelo. Esta matriz extracelular ópticamente claro es fácilmente susceptible de estudios de imágenes de células vivas y una mejor imita el entorno fisiológico, los tejidos blandos. Este informe demuestra una técnica para visualizar de manera directa la localización de proteínas y la dinámica, y la deformación de los alrededores en tres dimensiones de la matriz.
Examenmen de localización de la proteína y la dinámica en los procesos celulares proporciona información clave en las funciones de las proteínas. Genéticamente codificados etiquetas fluorescentes proporcionan un método único para la observación de localización de la proteína y la dinámica. Usando esta técnica, podemos analizar la acumulación subcelular de la clave, la fuerza generadora de los componentes del citoesqueleto en tiempo real como las maniobras de la célula a través de la matriz. Además, el uso de múltiples etiquetas fluorescentes con diferentes longitudes de onda, podemos examinar la localización de las proteínas de forma simultánea, lo que nos permite poner a prueba, por ejemplo, si las diferentes proteínas tienen funciones similares o divergentes. Además, la dinámica de las proteínas marcadas con fluorescencia se puede cuantificar el uso de recuperación después de photobleaching fluorescente (FRAP) el análisis. Esta medición de la movilidad de los ensayos de proteína y cómo de manera estable las proteínas están obligados a la red del citoesqueleto.
Mediante la combinación de imágenes de células vivas con el tratamiento de los inhibidores de la función de la proteína,podemos analizar en tiempo real los cambios en la distribución de las proteínas y la morfología de las células que migran. Además, también se combinan imágenes de células vivas con el uso de partículas de trazador fluorescente integrado dentro de la matriz para visualizar la deformación de la matriz durante la migración celular. Por lo tanto, podemos visualizar cómo una célula migrar distribuye la generación de la fuerza de las proteínas, y donde las fuerzas de tracción que se ejerce la matriz circundante. A través de estas técnicas, se puede obtener información valiosa sobre el papel de proteínas específicas y sus contribuciones a los mecanismos de migración celular.
Protocolo
1. Generación de línea celular estable (por ejemplo, las células MDCK)
- Células de la placa a 80-90% de confluencia en una placa de p35. No permita que las células del 100% de monocapa, lo que reducirá la eficiencia de transfección.
- Transfectar las células con el plásmido de interés utilizando Lipofectamine 2000. Optimizar las condiciones de transfección utilizando el protocolo del fabricante.
- Al día siguiente, el paso de las células en dos p150 placas de Petri. El plato grande se recomienda dejar suficiente espacio entre las colonias estables.
- Al día siguiente, cambio de los medios de comunicación y añadir 500 g / ml de G418 para cada plato. La concentración G418 deben ser optimizados para las líneas de células individuales.
- Cambiar los medios de comunicación todos los días durante aproximadamente 2 semanas. Después de 2 semanas, G418 resistentes a las colonias que comienzan a formarse, y será visible por el ojo desnudo.
- Utilizando un microscopio de fluorescencia invertido, identificar las colonias de GFP positivos. Marcar estas colonias en la placa con un rotulador.
- A selectivamente trypsinize las colonias de la placa de cultivo de tejidos, aspirar a los medios de comunicación, y se lavan las células dos veces con PBS o solución de tripsina. En el segundo lavado, no aspirar a que toda la solución. Deja una fina capa de líquido en la parte inferior de la placa para evitar que las células se sequen.
- Para cada colonia marcada: usar un hisopo de algodón esterilizados para limpiar lo más cerca posible de todo el borde de la colonia. Esto creará una isla de la zona húmeda que contiene la colonia de células. Pipeta de 10 l de tripsina en la colonia. Repita la operación para todas las colonias, y proceder con rapidez para evitar la desecación. Un investigador con experiencia por lo general puede tomar aproximadamente 12 colonias por placa p150.
- Incubar la placa a 37 ° C durante 5-10 minutos hasta que las células se desprenden de la placa y aparecen todo el año.
- Para cada colonia: Pipetear 10 l de tripsina en la colonia, y la pipeta hacia arriba y abajo un par de veces para separar las células de la placa. A continuación, una pipeta de todas las células de la colonia en un solo pozo en un plato de 24 pocillos.
- Después de colonias estables han crecido, la expresión de proteínas de cada colonia se analiza el uso estándar occidental blot e inmunofluorescencia. Ampliar estas líneas de células para su posterior análisis.
2. Modificación de superficies del plato con fondo de cristal para la unión de colágeno óptima (Opcional)
- Para silanizar el cristal, una pipeta 300 l de 2% 3-aminopropiltrimetoxisilano solución en la parte de vidrio de cada plato p35 con una abertura de 10 mm. Ver Figura 1 para la silanización y entrecruzamiento de esquemas. 3-aminopropiltrimetoxisilano se diluye en el agua filtrada.
- Se incuba durante 1 hora a temperatura ambiente.
- Aspirar la solución de 3 aminopropiltrimetoxisilano y lavar con agua filtrada tres veces durante 10 minutos cada uno.
- Aspirar a los platos de agua y colocar en un plato caliente ajustado a 50 ° C durante 1,5 horas. Tapas lugar de platos un poco del plato para que la humedad en el plato se puede escapar.
- Sacar los recipientes de fuego y se deja arrullarl.
- Pipeta de 300 l de solución de glutaraldehído al 2% en la parte de vidrio de cada plato. El glutaraldehído se diluye en PBS.
- Incubar durante 1 hora.
- Aspirar la solución de glutaraldehído y lavar los platos con PBS tres veces durante 10 minutos cada uno.
- Esterilizar los platos a la exposición a los rayos UV durante 1 hora. Los platos silanizada se puede almacenar a temperatura ambiente.
3. Preparación de gel de colágeno en 3D con partículas de trazador
- Lávese las partículas fluorescentes del marcador al girar por 100 l de acciones partículas trazadoras (10 10 partículas / ml) en una centrífuga a 15.000 rpm durante 5 minutos, se aspira el líquido y añadir 500 l de los medios de comunicación. Repita 5 veces. Tras el último lavado, las partículas se resuspenden en 30 l de los medios de comunicación.
- Trypsinize células GFP expresar, como de costumbre.
- Resuspender pellet de células de aproximadamente 2 x 10 6 células / ml.
- Pipeta de 240 l de medio de cultivo en un tubo Eppendorf.
- Añadir 12,6 l de Hepes 1M, 20 l de agua filtrada, 50 l de solución de la célula, de 10 l de partículas fluorescentes.
- Por último, añadir 167 l de colágeno dermis bovina que la solución (para obtener una concentración de trabajo de 1 mg / ml).
- Mezcle la solución a fondo, después pipeta de 80 l de la solución en la parte de vidrio del plato con fondo de cristal silanizado.
- Introducir la cápsula en la incubadora y permitir que el gel se polimeriza a 37 ° C durante 30 minutos. Ver Figura 1A de gel de colágeno típico en un recipiente de vidrio p35 inferior.
- Añadir 2 ml de medio de crecimiento con cuidado para cada plato.
4. Procedimiento para la adquisición de la imagen de lapso de tiempo
- Encienda el control de la temperatura del recinto microscopio y deja que la cámara alcance equilibre a una temperatura de estado estacionario a 37 ° C. Ver Figura 2 para un ejemplo de sistema de imágenes de células vivas.
- Cambiar los medios de comunicación a los nuevos medios suplementado con 25 mM Hepes para mantener un pH neutro. ParaDIC imágenes, el intercambio de la parte superior del plato con uno que tiene una tapa de cristal. Utilizando una tira fina de parafina, la cubierta del lado del plato para evitar la evaporación de los medios de comunicación.
- Para un objetivo de inmersión en aceite, coloque una gota de líquido de inmersión en el objetivo.
- El gel de colágeno que contiene un plato sobre platina del microscopio, y asegúrese de que el plato se pone en contacto con el líquido de inmersión.
- El enfoque de la muestra y la búsqueda de las células de interés para la imagen. Para minimizar la deriva de la etapa, permitirá plato que conformarse con unos 45 minutos antes de comenzar una captura de largo.
- Especificar los parámetros de adquisición de imágenes. Minimizar la exposición al láser, lo que podría dañar las células, mediante la modulación de la potencia del láser, el tiempo de exposición, la frecuencia y la duración de tiempo de captura. Los parámetros reales pueden variar con el sistema de microscopio y el tipo de células.
- Durante la adquisición de lapso de tiempo, un inhibidor puede ser añadido. Para el experimento además de inhibidores, no sella el recipiente con parafilm.
- Preparaciónse Hepes complementado los medios de comunicación con las drogas a una concentración de trabajo deseada.
- Cuando esté listo para añadir fármacos a los medios de comunicación, una pausa en la captura de la imagen, y retirar con cuidado la parte superior plato sin alterar el plato.
- Aspirar a los medios de comunicación en el plato, y la pipeta de la droga que contienen los medios de comunicación en el plato. A continuación, vuelva a colocar cuidadosamente la parte superior plato.
- La muestra puede estar fuera de foco debido a la adición de los nuevos medios, reajustar el enfoque.
- Reiniciar la captura, a continuación, control de las capturas para los próximos 30 minutos, y reajustar el enfoque según sea necesario. Ver Figura 4 para un ejemplo de experimento de drogas además.
5. FRAP procedimiento y el análisis
- La configuración del FRAP varía entre los sistemas, siga las instrucciones del fabricante.
- Configurar los parámetros de células vivas imágenes.
- Configurar los parámetros de photobleaching. Utilice la potencia del láser suficiente para photobleach la señal fluorescente, sin dañar la célula. Prueba de estos parameters en las células de la práctica.
- Iniciar la captura de imágenes, y permitir por lo menos 5 cuadros de captura de la imagen antes de photobleaching la región de interés.
- Continuar la captura y el tiempo de recuperación debe ser suficiente para captar la recuperación de fluorescencia completa. Véase la Figura 5 muestra un ejemplo de experimento FRAP.
- Analizar la recuperación de fluorescencia mediante la medición de la intensidad media de fluorescencia de la zona photobleached (antes y después de photobleaching) a través del tiempo. El uso de un software de análisis estadístico (por ejemplo, Excel), el ajuste de la curva de recuperación de la ecuación exponencial: I = I f (1-e-kt), donde I es la intensidad, I f es la intensidad final, t es el tiempo, y τ ½ es el tiempo que tarda la intensidad para llegar a la mitad del valor final: τ = ½ ln (2) / k. El medio tiempo es una medida de la tasa de movilidad de la proteína.
- Obtener los parámetros, la mitad de tiempo y la intensidad final de la curva exponencial de ajuste. Calcular ªe móvil fracción tomando la relación de la final para inicial la intensidad de fluorescencia.
6. Resultados representante
Un ejemplo de la imagen en vivo de las células sanas de las células epiteliales dentro de la matriz se muestra en la Figura 3. Las células sanas presentan una membrana suave y continua, y núcleo diferenciado, mientras que las células enfermas a menudo tienen una membrana interrumpido, y un excesivo número de vacuolas. En una matriz 3D, solo las células epiteliales migran 1, y en el transcurso de varios días, las células epiteliales de forma tridimensional, esférico, multicelulares quistes dentro de la matriz 2. Las células también son muy dinámicos, y migran dentro del quiste (Figura 3). La deformación de la matriz como consecuencia de las fuerzas de tracción ejercida por la migración de las células se analiza a través del desplazamiento de las partículas de trazador incrustados en la matriz circundante (Figura 4). Los movimientos de las partículas del trazador se muestran como una imagen de proyección máximo de diferenciadiferentes puntos de tiempo, y cada momento es pseudo-color para indicar el tiempo (Figura 4B). Por otra parte, las imágenes de una partícula de trazador individuales se muestran como un montaje para mostrar el movimiento de la partícula de trazador en el tiempo (Figura 4C). Todos los análisis de imágenes se realizó con ImageJ. Estas evaluaciones cualitativas de la deformación de la matriz, y por lo tanto, las fuerzas ejercidas por las células que migran, son primera aproximación útil de la distribución de fuerza de tracción. La estimación cuantitativa de las fuerzas de tracción ejercida por la migración de células en 3D está fuera del alcance de este protocolo y se describe en otro 3,4.
Un típico de recuperación después de fluorescencia photobleaching experimento se muestra en la Figura 5. La región de interés debe ser photobleached con los ajustes optimizados por láser para que la intensidad de la fluorescencia es visiblemente menor en comparación con los niveles de fondo (para maximizar la relación señal-ruido), mientras que mantiene las células sanas y en buen estado. La configuración óptima que be determinada empíricamente, ya que cada instalación FRAP es diferente (por ejemplo, FRAP mediante láser confocal de barrido del sistema).
La exposición y el intervalo de tiempo de adquisición de imágenes post-FRAP deben ser cuidadosamente controlados para evitar photobleaching fondo. El uso de la potencia del láser más bajo, corto tiempo de exposición, y el uso de la cámara óptica de alta sensibilidad y eficientes son esenciales para obtener imágenes de alta calidad FRAP. Si la fluorescencia de blanqueo durante la adquisición de la imagen es importante, este desvanecimiento de fondo debe ser cuantificado y normalizado a partir de la recuperación de la intensidad de fluorescencia observado. Debido a la naturaleza 3D del medio ambiente, la componente z de la recuperación de la intensidad de fluorescencia puede ser importante. Por lo tanto, es importante definir un volumen photobleached mediante la adopción de pre y post-stack en 3D las imágenes de la región photobleached. Su posterior análisis y modelización de la recuperación de la fluorescencia se discute en otra parte 5,6.
La combinación única de imágenes de células vivas, techniques: las buenas prácticas agrarias para seguir la dinámica de las proteínas del citoesqueleto, la fluorescencia roja partículas de seguimiento para controlar la deformación de la matriz, y la adición de los inhibidores, se pueden utilizar simultáneamente para el análisis de la dinámica de las proteínas, la fuerza de tracción y las vías moleculares.
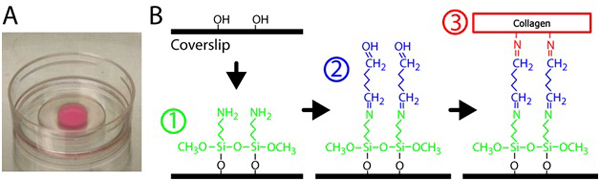
Figura 1. Preparación de gel de colágeno. A) la matriz de colágeno polimerizado en un plato con fondo de vidrio. El color rosado del gel se debe a las partículas fluorescentes incrustados. B) Procedimiento para el tratamiento de los platos de fondo de cristal para entrecruzar el gel de colágeno de la superficie del vidrio. En primer lugar, el plato de fondo de cristal se trata con 3 aminopropiltrimetoxisilano solución, solución de glutaraldehído que crosslinks la matriz de colágeno de la copa.
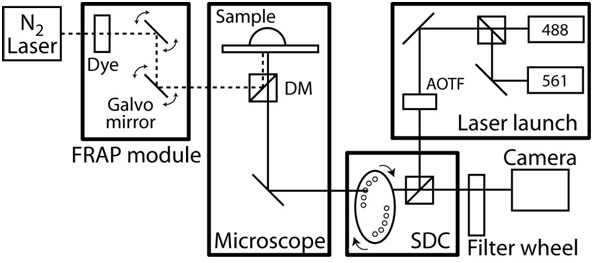
Figura 2. Esquema de la configuración del microscopio confocal / FRAP. El confocalmicroscopio se basa en un AxioObserver Zeiss con un CoolSnap cámara CCD HQ II y completamente automatizado por Slidebook software (Intelligent innovaciones de imagen). La unidad confocal es personalizado y basado en la unidad de disco giratorio CSU10 Yokogawa y dos láseres de estado sólido (488 nm con 50 mW y 561 nm, con 40 mW) con acústico-óptico sintonizable filtro (AOTF) para permitir el cambio entre dos milésimas de segundo láser. Los filtros de emisión nm 525/50 y 620/60 nm (# 118661 # 118085 y, Chroma Technology) y espejo dicroico es 488-568 Brightline banda dual (Semrock). Los objetivos son una larga distancia de trabajo 40x C-Apochromat objetivo con NA 1.1 y distancia de trabajo de 0,62 mm, y un 63x Plan-Apochromat objetivo con NA 1.4 y una distancia de trabajo de 0,19 mm. El microscopio también incluye un sistema de fotoablación FRAP, que consiste en una fibra de bombeo óptico láser de colorante, una posición ordenador haz controlado e intensidad, y un tamaño de spot de difracción limitada. Además, el microscopio está equipado conuna etapa XY motorizado que incluye 0,1 micras codificadores lineales en cada eje. Durante el time-lapse, la temperatura ambiente se mantiene por una cámara del microscopio diseñado y un calentador con control de temperatura de retroalimentación. Para aislar el ruido y las vibraciones, el sistema de microscopio entero es de una vibración libre de la tabla.
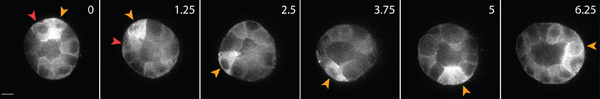
Figura 3. En vivo de imágenes de células de las células epiteliales que expresan GFP-actina. Estas células forman un quiste después de 4 días de cultivo en una matriz de colágeno en 3D. Algunas células se mueven a lo largo de la superficie del quiste (punta de flecha amarilla), mientras que otros emigran en el interior del quiste (rojo punta de flecha). Barra de escala 10 micras, el tiempo en horas.
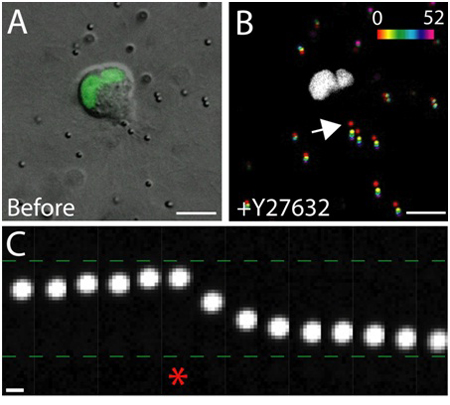
Figura 4. El efecto de la inhibición de la Rho-kinasa en la fuerza de tracción. A) Imagen DIC de una célula de la migración de MDCK expresar GFP etiquetados marcador nuclear. La imagen fue tomada inmediatamente antes del tratamiento de la Rho-kinasa inhibidor de la Y-27632. Barra de escala 10 micras. B) el desplazamiento de partículas resultantes de la adición de Y-27632. Las posiciones de las partículas en diversos puntos temporales (0 - 52 min) fueron pseudo-color de acuerdo a la escala de intensidad, proyecta en una sola imagen. La región de blanca es el núcleo GFP positivos en la migración de las células. Barra de escala 10 micras. Tiempo en minutos. C) La partícula de movimiento en el borde de salida de la celda (ver flecha en B). Asterisco indica el último fotograma capturado antes de Y-27632 además. La partícula de trazador se trasladó hacia y desde fuera del borde de salida de la celda de la migración de antes y después de la adición de Y-27632, respectivamente. Barra de escala 1 micra.
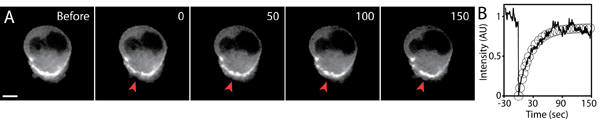
Figura 5. FRAP análisis de GFP-actina expresión de la migración de células en una matriz tridimensional. A) las imágenes Timelapse de la GFP-actina de la célula que expresa antes y después del photobleaching. Una pequeña región de la GFP-actina acumulada en la parte trasera de células se photobleached (rojo punta de flecha) en el punto de tiempo 0. Tiempo en segundos, la barra de escala 5 micras. B) La intensidad media de fluorescencia de la región photobleached (línea continua), y el ajuste exponencial de la recuperación de fluorescencia (círculos) en función con el tiempo. La intensidad de fluorescencia se normalizaron al valor inicial antes de photobleaching.
Discusión
Aquí se describe un método para el uso de células vivas de imagen para estudiar los mecanismos de migración de las células en una matriz tridimensional. El éxito de esta técnica depende de la obtención de "buenos" clones que expresan establemente GFP-etiquetados proteínas. El bajo nivel de las proteínas GFP requerirá una exposición excesiva excitación que compromete la salud de las células, mientras que el nivel demasiado alto, las buenas prácticas agrarias tienen efectos secundarios indeseables...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Damos las gracias al Dr. Grant Sumida para la lectura crítica del manuscrito. Este trabajo fue apoyado por un Premio al Investigador Joven Beckman (SY), el Premio Hellman Familia Nueva Facultad (SY), EUREKA NIH, la Universidad de California de Investigación del Cáncer del Comité de Coordinación.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| Colágeno, bovino, Tipo I | BD Biosciences | 354231 | Archivo es de aproximadamente 3 mg / ml |
| 3-aminopropiltrimetoxisilano | Sigma Aldrich | 281778 | Diluir en agua |
| glutaraldehído | Sigma Aldrich | 340855 | Diluir en PBS |
| Hepes 1M | Invitrogen | 15630-080 | |
| Poliestireno Fluospheres microesferas de 1 micra, fluorescencia roja (580/605) | Invitrogen | F13083 | |
| Geneticina (G418) | Invitrogen | 11811-031 | |
| Los medios de cultivo componentes: | |||
| DMEM | Invitrogen | 31600-034 | |
| El suero fetal bovino | Atlanta Productos Biológicos | S115500 | |
| Penicilina / estreptomicina | Invitrogen | 15140-122 | |
| Kanamicina | Invitrogen | 15160-054 | |
Referencias
- Shih, W., Yamada, S., Myosin, . A dependent retrograde flow drives 3D cell migration. Biophys. J. 98 (8), L29-L31 (2010).
- O'Brien, L. E., Yu, W., Tang, K., Jou, T. S., Zegers, M. M., Mostov, K. E. Morphological and biochemical analysis of Rac1 in three-dimensional epithelial cell cultures. Methods Enzymol. 406, 676-691 (2006).
- Legant, W. R., Miller, J. S., Blakely, B. L., Cohen, D. M., Genin, G. M., Chen, C. S. Measurement of mechanical tractions exerted by cells in three-dimensional matrices. Nat. Methods. 7 (12), 969-971 (2010).
- Del Alamo, J. C., Meili, R., Alonso-Latorre, B., Rodriguez-Rodriguez, J., Aliseda, A., Firtel, R. A., Lasheras, J. C. Spatio-temporal analysis of eurkaryotic cell motility by improved force cytometry. Proc. Natl. Acad. Sci. U.S.A. 104 (33), 13343-13348 (2007).
- Lippincott-Schwartz, J., Snapp, E., Kenworthy, A. Studying protein dynamics in living cells. Nat. Rev. Mol. Cell Biol. 2 (6), 444-456 (2001).
- Phair, R. D., Misteli, T. Kinetic modeling approaches to in vivo imaging. Nat. Rev. Mol. Cell Biol. 2 (12), 898-907 (2001).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. Fluorescent toolbox for assessing protein location and function. Science. 312 (5771), 217-224 (2006).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Methods. 2, 905-909 (2005).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados