É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Em células vivas, imagens de células que expressam proteínas fluorescentes Migrando-marcado em uma matriz tridimensional
Neste Artigo
Resumo
Processos celulares, tais como a migração das células têm sido tradicionalmente estudados em duas dimensões, superfícies de plástico duro. Este relatório descreve uma técnica para a visualização direta localização de proteínas e análise dinâmica de proteínas em células migrando de uma forma mais fisiologicamente relevantes, matriz tridimensional.
Resumo
Tradicionalmente, a migração celular foi estudada em duas dimensões, superfícies de plástico duro. No entanto, durante processos biológicos importantes, tais como a cicatrização da ferida, regeneração de tecidos, e metástase de câncer, as células devem navegar através de complexas, tecido tridimensional extracelular. Para compreender melhor os mecanismos por trás desses processos biológicos, é importante examinar os papéis das proteínas responsáveis por dirigir a migração celular. Aqui, descrevemos um protocolo para estudar os mecanismos de migração celular usando a linha de células epiteliais (MDCK), e uma imagem tridimensional, fibroso, matriz auto-polimerização como um sistema modelo. Esta matriz extracelular opticamente clara se adequa facilmente aos estudos em células vivas, de imagem e melhor imita o fisiológico, meio ambiente dos tecidos moles. Este relatório demonstra uma técnica para a visualização direta localização de proteínas e dinâmica, e deformação das imediações matriz tridimensional.
Exameminação da localização de proteínas e dinâmica durante processos celulares fornece uma visão fundamental em funções da proteína. Geneticamente codificado marcas fluorescentes fornecem um método único para a observação de localização de proteínas e dinâmica. Usando esta técnica, podemos analisar o acúmulo subcelulares de-chave, a força de geração de componentes do citoesqueleto em tempo real como as manobras de células através da matriz. Além disso, utilizando várias marcas fluorescentes com diferentes comprimentos de onda, podemos examinar a localização de proteínas simultaneamente, permitindo-nos testar, por exemplo, se diferentes proteínas têm funções semelhantes ou divergentes. Além disso, a dinâmica das proteínas fluorescentes marcados pode ser quantificada usando a Recuperação Após Fotodegradação Fluorescente (FRAP) análise. Esta ensaios de medição da mobilidade de proteínas e como forma estável as proteínas são vinculados à rede do citoesqueleto.
Ao combinar live-célula de imagem com o tratamento dos inibidores da função da proteína,podemos examinar em tempo real as mudanças na distribuição de proteínas e morfologia das células migrando. Além disso, nós também combinar live-célula de imagem com o uso de partículas fluorescentes tracer incorporados dentro da matriz para visualizar a deformação da matriz durante a migração celular. Assim, podemos visualizar como uma célula migrar distribui força de geração de proteínas, e onde as forças de tração são exercidos com a matriz circundante. Através destas técnicas, podemos obter informações valiosas sobre os papéis de proteínas específicas e suas contribuições para os mecanismos de migração celular.
Protocolo
1. Geração de linha celular estável (por exemplo, células MDCK)
- Células em placas a 80-90% confluência em um prato de p35. Não deixe que as células formam 100% monocamada confluente, que irá reduzir a eficiência de transfecção.
- Transfecção as células com o plasmídeo de interesse usando Lipofectamine 2000. Otimizar as condições de transfecção usando o protocolo do fabricante.
- No dia seguinte passagem, as células em duas p150 placas de Petri. O grande prato é recomendada para permitir o espaçamento suficiente entre as colônias estáveis.
- No dia seguinte, altere a mídia e adicionar 500 mg / ml de G418 para cada prato. A concentração de G418 deve ser otimizado para linhagens de células individuais.
- Alterar mídia todos os dias por aproximadamente duas semanas. Após 2 semanas, G418 colônias resistentes deve começar a se formar, e será visível a olho nu.
- Usando um microscópio invertido fluorescentes, identificar colônias GFP positivas. Mark essas colônias na chapa usando uma caneta Sharpie.
- Para seletivamente trypsinize as colônias da placa de cultura de tecidos, aspirar a mídia, e lavar as células duas vezes com PBS ou solução de tripsina. Na segunda lavagem, não aspire para fora toda a solução. Deixe uma fina camada de líquido no fundo do prato para evitar que as células de secagem.
- Para cada colônia marcada: use um cotonete esterilizado para eliminar o mais próximo possível ao redor da borda da colônia. Isto irá criar uma ilha de área molhada contendo a colônia de células. Pipetar 10 mL de tripsina para a colônia. Repita para cada colônia, e proceder rapidamente para evitar a secagem. Um investigador experiente pode pegar normalmente ~ 12 colônias por placa p150.
- Incubar placa a 37 ° C por 5-10 minutos até retirar as células da placa e aparecem rodada.
- Para cada colônia: Pipetar 10 ml de tripsina para a colônia, e pipetar cima e para baixo algumas vezes para retirar as células da placa. Então pipeta todas as células da colônia em um único poço em um prato de 24 também.
- Depois de colônias estáveis têm crescido, a expressão da proteína de cada colônia é analisada usando western blot padrão e de imunofluorescência. Expandir essas linhas de células para análise posterior.
2. Modificação da superfície do prato fundo de vidro para a ligação de colágeno ideal (Opcional)
- Para o vidro silanizadas, pipetar 300 mL de solução de 2% 3-Aminopropyltrimethoxysilane sobre a parcela de vidro de cada prato p35 com uma abertura 10 mm. Veja a Figura 1 para a silanização e cross-linking esquemático. 3-Aminopropyltrimethoxysilane é diluído em água filtrada.
- Incubar por 1 hora em temperatura ambiente.
- Aspirar a solução de 3-Aminopropyltrimethoxysilane e lavar com água filtrada três vezes durante 10 minutos cada.
- Aspirar a pratos de água e coloque na chapa quente ajustada para 50 ° C por 1,5 horas. Tops lugar de pratos um pouco fora do prato, para que a umidade no prato pode escapar.
- Retire os pratos do fogo e deixe a cool.
- Pipetar 300 mL da solução de glutaraldeído a 2% sobre a parcela de vidro de cada prato. O glutaraldeído é diluído em PBS.
- Incubar por 1 hora.
- Aspirar a solução de glutaraldeído e lavar pratos com PBS três vezes durante 10 minutos cada.
- Esterilizar os pratos com a exposição à luz UV durante 1 hora. Os pratos silanizadas podem ser armazenados em temperatura ambiente.
3. Preparação de gel de colágeno 3D com partículas de traçador
- Lavar partículas traçador fluorescente para baixo, girando 100 l de partículas de ações marcador (10 10 partículas / ml) em uma centrífuga a 15.000 rpm por 5 minutos, aspirar líquido para fora, e adicionar 500 mL de mídia. Repita 5 vezes. Depois última lavagem, as partículas de ressuspender em 30 mL de mídia.
- Trypsinize células GFP expressar como de costume.
- Ressuspender pellet celular a aproximadamente 2 x 10 6 células / ml.
- Pipetar 240 mL de meio de crescimento em um tubo eppendorf.
- Adicionar 12,6 mL de Hepes 1M, 20 l de água filtrada, 50 ml de solução de células, então 10 ml de partículas fluorescentes.
- Por fim, adicionar 167 mL de colágeno da derme bovina Eu solução (para obter uma concentração de trabalho de 1 mg / ml).
- Misturar a solução completa, então pipeta 80 mL da solução para a parte de vidro do prato de vidro silanizadas fundo.
- Prato coloque em incubadora e permitir que o gel para polimerizar a 37 ° C por 30 minutos. Figura 1A para ver gel de colágeno típica em um prato fundo de vidro p35.
- Adicionar 2 ml de meio de crescimento com cuidado para cada prato.
4. Procedimento para lapso de tempo de aquisição de imagem
- Ligue o controle de temperatura do recinto microscópio e deixar a câmara de escopo atingir a uma temperatura em estado estacionário, 37 ° C. Veja a Figura 2 para um exemplo de live-célula do sistema de imagem.
- Mudança de mídia para a nova mídia suplementado com 25 mM Hepes para manter um pH neutro. ParaDIC de imagens, troca a parte superior do prato com um que tem um tampo de vidro. Usando uma fina tira de parafilme, cobrir o lado do prato para evitar a evaporação da mídia.
- Para um objetivo de imersão em óleo, coloque uma gota de fluido de imersão sobre o objectivo.
- Coloque o gel de colágeno contendo prato no palco microscópio, e certifique-se o prato faz contato com o líquido de imersão.
- O foco da amostra e busca de células de interesse para a imagem. Para minimizar oscilações estágio, permita prato que se contentar com cerca de 45 minutos antes de iniciar uma captura muito tempo.
- Especificar os parâmetros de aquisição de imagem. Minimizar a exposição a laser, o que poderia danificar as células, pelo poder do laser modulando o, tempo de exposição, freqüência e duração do tempo de captura. Os parâmetros reais podem variar com o sistema de microscópio e tipo de célula.
- Durante a aquisição de lapso de tempo, um inibidor pode ser adicionado. Para o experimento além inibidor, não selar o prato com parafilme.
- Preparaçãosão complementados Hepes mídia com droga em uma concentração de trabalho desejada.
- Quando estiver pronto para adicionar drogas para a mídia, fazer uma pausa na captura de imagem, e remova cuidadosamente o topo prato sem perturbar o prato.
- Aspirar a mídia no prato, e pipetar a droga contendo mídia no prato. Então, substitua cuidadosamente a parte superior do prato.
- A amostra pode estar fora de foco devido à adição de novas mídias, reajustar o foco.
- Reiniciar a captura, em seguida, monitorar a captura para os próximos 30 minutos, e reajustar o foco, se necessário. Veja a Figura 4 para um exemplo de experimento além de drogas.
5. Procedimento FRAP e análise
- A configuração FRAP varia entre os sistemas, siga as instruções do fabricante.
- Configurar parâmetros para live-célula de imagem.
- Configurar parâmetros para fotobranqueamento. Use a potência do laser suficiente para photobleach o sinal fluorescente sem danificar a célula. Testar essas partro diâmetros em células prática.
- Iniciar a captura de imagem, e permitir que pelo menos 5 quadros de captura de imagem antes de fotodegradação da região de interesse.
- Continue a captura, e tempo de recuperação deve ser suficiente para capturar a recuperação completa de fluorescência. Ver Figura 5 para um exemplo de experimento FRAP.
- Analisar a recuperação de fluorescência através da medição da intensidade média de fluorescência da área de Foto-descoloração (antes e depois fotobranqueamento) ao longo do tempo. Usando um software de análise estatística (por exemplo, Excel), o traçado da curva de recuperação para a equação exponencial: I = I f (1-e-kt), onde I é a intensidade, eu f é a intensidade final, t é o tempo, e τ ½ é o tempo que leva para a intensidade para chegar a metade do valor final: ½ τ = ln (2) / k. O meio-tempo é uma medida da taxa de mobilidade da proteína.
- Obter os parâmetros, metade do tempo e intensidade final da curva exponencial-fit. Calcule ªe fração móvel, tendo a razão entre o final de intensidades de fluorescência inicial.
6. Resultados representante
Um exemplo da imagem ao vivo de células saudáveis de células epiteliais dentro da matriz é mostrada na Figura 3. Células saudáveis apresentam uma membrana lisa, contínua, e núcleo distinto, enquanto que as células saudáveis, muitas vezes têm uma membrana interrompido, e um número excessivo de vacúolos. Em uma matriz 3D, única células epiteliais migram 1, e ao longo de vários dias, as células epiteliais forma tridimensional, esférico, multi-celular cistos dentro da matriz 2. As células também são altamente dinâmicos e migrar dentro do cisto (Figura 3). A deformação da matriz, como resultado das forças de tração exercida pelas células de migrar é analisado através do deslocamento de partículas tracer incorporados na matriz circundante (Figura 4). Os movimentos das partículas tracer são exibidos como uma imagem de projeção máxima de diferentesrentes pontos de tempo, e cada momento é pseudo-coloridos para indicar o tempo (Figura 4B). Alternativamente, as imagens de uma partícula tracer individuais são exibidos como uma montagem para mostrar o movimento da partícula traçador ao longo do tempo (Figura 4C). Toda a análise de imagem foi feita usando ImageJ. Estas avaliações qualitativas da deformação da matriz e, portanto, as forças exercidas por células migratórias, são primeira aproximação útil de distribuição de força de tração. A estimativa quantitativa das forças de tração exercida pelas células migram em 3D está fora do âmbito deste protocolo e é descrito em outros lugares 3,4.
A recuperação típica fluorescência após fotobranqueamento experimento é mostrado na Figura 5. A região de interesse deve ser Foto-descoloração usando as configurações de laser otimizado para que a intensidade de fluorescência está visivelmente diminuída em relação aos níveis de fundo (para maximizar a relação sinal-ruído), mantendo as células saudáveis e sem danos. A configuração ideal deve be determinada empiricamente como cada configuração FRAP é diferente (por exemplo, FRAP sistema usando laser de varredura confocal).
A exposição eo intervalo de tempo de pós-FRAP aquisição da imagem deve ser cuidadosamente controlada para evitar a fotodegradação de fundo. O uso de menor poder laser, curto tempo de exposição, bem como a utilização de câmera sensível e de alta óptica eficientes são essenciais para imagens de alta qualidade FRAP. Se a fluorescência de branqueamento durante a aquisição da imagem é significativa, esta sumindo de fundo devem ser quantificados e normalizados a partir da recuperação de intensidade de fluorescência observada. Devido à natureza do ambiente 3D, o z-componente de recuperação de intensidade de fluorescência pode se tornar significativo. Portanto, é importante definir um volume de Foto-descoloração tomando pré e pós-stack imagens 3D da região Foto-descoloração. Posterior análise e modelagem de recuperação de fluorescência é discutido em outros lugares 5,6.
A combinação única de live-célula de imagem techniques: GFP para acompanhar a dinâmica das proteínas do citoesqueleto, vermelho partículas de fluorescência marcador para monitorar a deformação da matriz, e da adição de inibidores, podem ser usados simultaneamente para a análise da dinâmica de proteínas, força de tração e vias moleculares.
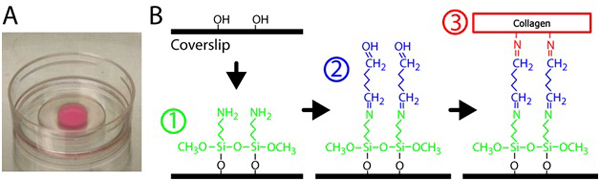
Figura 1. Preparação de gel de colágeno. A) matriz de colágeno polimerizado em um prato fundo de vidro. A cor rosa do gel é devido a partículas fluorescentes embutidas. B) Procedimento para tratar os pratos com fundo de vidro para crosslink o gel de colágeno para a superfície de vidro. Primeiro, o prato fundo de vidro é tratado com 3-Aminopropyltrimethoxysilane solução, então a solução de glutaraldeído que ligações cruzadas da matriz de colágeno para o vidro.
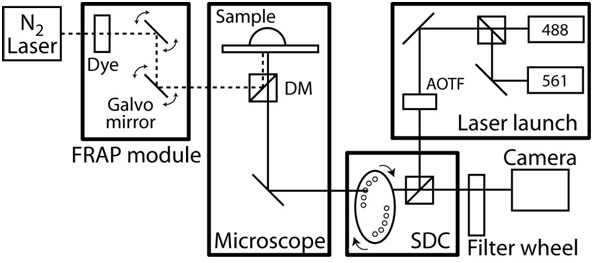
Figura 2. Schematics de confocal / FRAP configuração microscópio. O confocalmicroscópio é baseado em uma AxioObserver Zeiss com uma HQ II CoolSnap câmera CCD e completamente automatizado por Slidebook software (Intelligent Innovations Imaging). A unidade é confocal personalizados e com base na Yokogawa unidade de disco giratório CSU10 e dois lasers de estado sólido (488 nm com 50 mW e 561 nm com 40 mW) com filtro acústico-óptico (AOTF) para permitir a comutação entre dois milissegundos lasers. Os filtros de emissão são nm 525/50 e 620/60 nm (# 118661 e # 118085, Tecnologia Chroma) e espelho dicróico é 488-568 banda BrightLine Dual (Semrock). Os objetivos são uma longa distância de trabalho objetivo C Apochromat-40x com NA 1.1 e distância de trabalho de 0,62 mm, e um objetivo Plano Apochromat-63x com NA 1.4 e uma distância de trabalho de 0,19 mm. O microscópio também inclui um sistema de fotoablação FRAP que consiste em uma fibra óptica de corante bombeado por laser, um computador posição feixe controlado e intensidade, e um tamanho de ponto de difração limitada. Além disso, o microscópio é equipado comxy um estágio motorizado que inclui 0,1 micron encoders lineares em cada eixo. Durante a criação de imagens de lapso de tempo, a temperatura ambiente é mantida por uma câmara de microscópio personalizados e um aquecedor com um controle de temperatura feedback. Para isolar qualquer ruído e vibração, o sistema de microscópio inteira está em uma vibração livre de mesa.
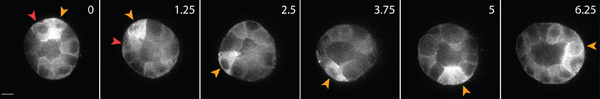
Figura 3. Vivo imagens de células de células epiteliais expressando GFP-actina. Essas células formam um cisto após 4 dias de cultura em uma matriz de colágeno 3D. Algumas células se movem ao longo da superfície de cisto (seta amarela), enquanto outros migram no interior do cisto (seta vermelha). Barra de escala 10 mM, o tempo em horas.
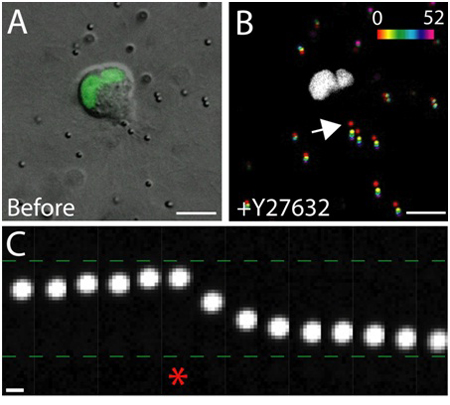
Figura 4. O efeito de inibição da Rho-quinase na força de tração. A) imagem DIC de uma célula migrar MDCK expressar GFP tag marcador nuclear. A imagem foi tomada imediatamente antes do tratamento de Rho-quinase inibidor Y-27632. Barra de escala 10 mM. B) o deslocamento de partículas resultantes da adição de Y-27632. As posições das partículas em várias alturas (0 - 52 min) foram pseudo-colorido de acordo com a escala de intensidade, então projetada em uma única imagem. A região branca é o núcleo GFP positivos nas células migratórias. Barra de escala 10 mM. Tempo em minutos. C) O movimento de partículas no bordo de fuga da célula (ver seta em B). Asterisco representa o último frame capturado antes de Y-27632 adição. A partícula tracer se moveu em direção e para longe da borda posterior da célula migrar antes e após a adição de Y-27632, respectivamente. Barra de escala 1 mícron.
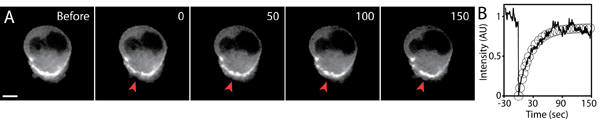
Figura 5. FRAP análise de GFP-actina expressando a migração de células em uma matriz tridimensional. A) imagens Timelapse do GFP-actina expressar célula de antes e depois da fotodegradação. Uma pequena região do GFP-actina acumulada na parte traseira de células foi Foto-descoloração (vermelho ponta de seta) em timepoint 0. Tempo em segundos, a escala de barras 5 mm. B) A intensidade média de fluorescência da região Foto-descoloração (linha cheia), eo ajuste exponencial da recuperação de fluorescência (círculos) desenhados ao longo do tempo. A intensidade de fluorescência foi normalizada para o valor inicial antes de fotodegradação.
Discussão
Aqui nós descrevemos um método para usar live-célula de imagem para estudar os mecanismos de migração celular em uma matriz tridimensional. O sucesso desta técnica depende da obtenção de "bom" clones estavelmente expressando proteínas GFP. O baixo nível de proteínas GFP requer uma exposição de excitação excessiva que compromete a saúde das células, enquanto o nível GFP muito alto vai ter efeitos colaterais indesejáveis na célula. Assim, a escolha do vetor de transfecção genes em cé...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos ao Dr. Grant Sumida para a leitura crítica do manuscrito. Este trabalho foi financiado por um Investigator Award Beckman Young (SY), uma família Hellman Award Faculdade Novo (SY), um EUREKA NIH, da Universidade da Califórnia Cancer Research Comissão Coordenadora.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Colágeno, bovina, tipo I | BD Biosciences | 354231 | Estoque é cerca de 3 mg / ml |
| 3-aminopropyltrimethoxysilane | Sigma Aldrich | 281778 | Diluída em água |
| glutaraldeído | Sigma Aldrich | 340855 | Diluir em PBS |
| Hepes 1M | Invitrogen | 15630-080 | |
| Poliestireno Fluospheres microesferas 1 mícron, fluorescência vermelha (580/605) | Invitrogen | F13083 | |
| Geneticin (G418) | Invitrogen | 11811-031 | |
| Componentes da cultura da mídia: | |||
| DMEM | Invitrogen | 31600-034 | |
| Soro fetal bovino | Atlanta Biologicals | S115500 | |
| Penicilina / estreptomicina | Invitrogen | 15140-122 | |
| Canamicina | Invitrogen | 15160-054 | |
Referências
- Shih, W., Yamada, S., Myosin, . A dependent retrograde flow drives 3D cell migration. Biophys. J. 98 (8), L29-L31 (2010).
- O'Brien, L. E., Yu, W., Tang, K., Jou, T. S., Zegers, M. M., Mostov, K. E. Morphological and biochemical analysis of Rac1 in three-dimensional epithelial cell cultures. Methods Enzymol. 406, 676-691 (2006).
- Legant, W. R., Miller, J. S., Blakely, B. L., Cohen, D. M., Genin, G. M., Chen, C. S. Measurement of mechanical tractions exerted by cells in three-dimensional matrices. Nat. Methods. 7 (12), 969-971 (2010).
- Del Alamo, J. C., Meili, R., Alonso-Latorre, B., Rodriguez-Rodriguez, J., Aliseda, A., Firtel, R. A., Lasheras, J. C. Spatio-temporal analysis of eurkaryotic cell motility by improved force cytometry. Proc. Natl. Acad. Sci. U.S.A. 104 (33), 13343-13348 (2007).
- Lippincott-Schwartz, J., Snapp, E., Kenworthy, A. Studying protein dynamics in living cells. Nat. Rev. Mol. Cell Biol. 2 (6), 444-456 (2001).
- Phair, R. D., Misteli, T. Kinetic modeling approaches to in vivo imaging. Nat. Rev. Mol. Cell Biol. 2 (12), 898-907 (2001).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. Fluorescent toolbox for assessing protein location and function. Science. 312 (5771), 217-224 (2006).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Methods. 2, 905-909 (2005).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados