Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Imagerie des cellules vivantes des cellules exprimant des protéines par fluorescence Migration-étiquetés dans une matrice tridimensionnelle
Dans cet article
Résumé
Processus cellulaires tels que la migration des cellules ont traditionnellement été étudié sur deux dimensions, les surfaces en plastique rigide. Ce rapport décrit une technique pour visualiser directement la localisation des protéines et l'analyse de la dynamique des protéines dans les cellules de migrer dans un plus physiologiquement pertinents, en trois dimensions de la matrice.
Résumé
Traditionnellement, la migration cellulaire a été étudié sur deux dimensions, les surfaces en plastique rigide. Toutefois, au cours des processus biologiques importants tels que la cicatrisation des plaies, la régénération des tissus, et les métastases du cancer, les cellules doivent naviguer à travers complexe, en trois dimensions du tissu extracellulaire. Afin de mieux comprendre les mécanismes derrière ces processus biologiques, il est important d'examiner les rôles des protéines responsables de la conduite de migration cellulaire. Ici, nous présentons un protocole d'étudier les mécanismes de la migration cellulaire en utilisant la lignée cellulaire épithéliale (MDCK), et un en trois dimensions, fibreuse, autopolymérisant matrice comme un système modèle. Cette matrice extracellulaire est optiquement clair facilement s'adapter à des études d'imagerie des cellules vivantes et mieux imite le physiologiques, l'environnement des tissus mous. Ce rapport démontre une technique pour visualiser directement la localisation des protéines et la dynamique, et la déformation de la entourant matrice tridimensionnelle.
Exammination de la localisation des protéines et la dynamique des processus cellulaires au cours donne un aperçu de la clé dans les fonctions des protéines. Génétiquement codés étiquettes fluorescentes offrent une méthode unique pour l'observation de la localisation des protéines et de dynamique. En utilisant cette technique, nous pouvons analyser l'accumulation intracellulaire de la clé, la force génératrice de composants du cytosquelette en temps réel que les manœuvres des cellules à travers la matrice. De plus, en utilisant de multiples balises fluorescentes avec différentes longueurs d'onde, nous pouvons examiner la localisation de protéines multiples simultanément, nous permettant ainsi de tester, par exemple, si différentes protéines ont des rôles similaires ou divergents. Par ailleurs, la dynamique des protéines par fluorescence marqués peuvent être quantifiés à l'aide de récupération fluorescent après photoblanchiment (FRAP) analyse. Cette mesure de la mobilité des dosages des protéines et comment stablement lié les protéines sont sur le réseau du cytosquelette.
En combinant imagerie des cellules vivantes avec le traitement des inhibiteurs de la fonction des protéines,nous pouvons examiner en temps réel l'évolution de la répartition des protéines et la morphologie des cellules migrantes. Par ailleurs, nous aussi combiner imagerie des cellules vivantes avec l'utilisation de particules de traceur fluorescent incorporé dans la matrice pour visualiser la déformation de matrice lors de la migration cellulaire. Ainsi, nous pouvons visualiser comment une cellule migration distribue la force génératrice de protéines, et où les forces de traction sont exercées à la matrice environnante. Grâce à ces techniques, nous pouvons avoir un aperçu précieux sur les rôles des protéines spécifiques et de leurs contributions aux mécanismes de la migration cellulaire.
Protocole
1. Génération d'une lignée cellulaire stable (par exemple des cellules MDCK)
- Cellules de la plaque à 80-90% de confluence dans un plat à p35. Ne laissez pas les cellules sous forme de 100% monocouche confluente, ce qui réduira l'efficacité de la transfection.
- Transfecter les cellules avec le plasmide d'intérêt en utilisant Lipofectamine 2000. Optimiser les conditions de transfection utilisant le protocole du fabricant.
- Le lendemain, le passage des cellules dans deux boîtes de Pétri p150. Le grand plat est recommandé pour permettre à l'espacement suffisant entre les colonies stables.
- Le lendemain, changer les médias et ajouter 500 pg / ml de G418 à chaque plat. La concentration du G418 devrait être optimisé pour les lignées de cellules individuelles.
- Changer les médias chaque jour pendant environ 2 semaines. Après 2 semaines, G418 colonies résistantes devraient commencer à se former, et sera visible à l'œil nu.
- En utilisant un microscope inversé fluorescentes, identifier les colonies GFP-positives. Marquer ces colonies sur la plaque à l'aide d'un stylo Sharpie.
- Pour sélectivement trypsiniser les colonies de la plaque de culture de tissus, aspirer à la presse, et laver les cellules deux fois avec du PBS ou solution de trypsine. Sur le second lavage, ne pas aspirer à toute la solution. Laisser une mince couche de liquide sur le fond de l'assiette pour empêcher les cellules de séchage.
- Pour chaque colonie a marqué: utilisez un coton-tige stérile pour essuyer le plus proche possible sur le pourtour de la colonie. Cela va créer une île de la zone humide contenant de la colonie de cellules. Pipeter 10 pl de la trypsine sur la colonie. Répétez l'opération pour chaque colonie, et de procéder rapidement pour éviter le séchage. Un chercheur expérimenté peut généralement prendre ~ 12 colonies par p150 assiette.
- Incuber la plaque à 37 ° C pendant 5-10 minutes jusqu'à ce cellules se détachent de la plaque et paraître rond.
- Pour chaque colonie: pipette 10 ul de la trypsine sur la colonie, et la pipette monter et descendre une ou deux fois pour détacher les cellules de la plaque. Puis la pipette toutes les cellules de la colonie en un seul puits dans un plat à 24 puits.
- Après colonies stables ont augmenté, l'expression des protéines de chaque colonie est analysée en utilisant la norme western blot et immunofluorescence. Développer ces lignées de cellules pour une analyse ultérieure.
2. Modification de la surface du plat à fond de verre pour la liaison de collagène optimale (facultatif)
- Pour silaniser le verre, pipette 300 pi de 2% 3-aminopropyltriméthoxysilane solution sur la partie vitrée de chaque plat p35 avec une ouverture de 10 mm. Voir Figure 1 pour la silanisation et réticulation schématique. 3-aminopropyltriméthoxysilane est dilué dans de l'eau filtrée.
- Incuber pendant 1 heure à température ambiante.
- Aspirer la solution 3-aminopropyltriméthoxysilane et laver avec de l'eau filtrée trois fois pour 10 minutes chacune.
- Aspirer les plats d'eau et placer sur une plaque chaude réglé à 50 ° C pendant 1,5 heures. Lieu de sommets plats légèrement du plat afin que l'humidité dans le plat ne peut échapper.
- Retirez les plats du feu et laisser à gazouillerl.
- Pipeter 300 pi de solution de glutaraldéhyde à 2% sur la partie en verre de chaque plat. Le glutaraldéhyde est dilué dans du PBS.
- Incuber pendant 1 heure.
- Aspirer la solution de glutaraldéhyde et laver la vaisselle avec du PBS à trois reprises pendant 10 minutes chacun.
- Stériliser les plats par une exposition aux rayons UV pendant 1 heure. Les plats silanisé peut être conservée à température ambiante.
3. Préparation du gel de collagène 3D avec des particules traceurs
- Laver les particules traceur fluorescent en faisant tourner en baisse de 100 ul de particules traceurs de stock (10 10 particules / ml) dans une centrifugeuse à 15 000 rpm pendant 5 minutes, aspirer le liquide, et ajouter 500 ul de médias. Répéter 5 fois. Après dernier lavage, les particules de remettre en suspension dans 30 pl de médias.
- Trypsiniser cellules GFP exprimer comme d'habitude.
- Resuspendre le culot cellulaire d'environ 2 x 10 6 cellules / ml.
- Pipeter 240 ul de milieu de croissance dans un tube Eppendorf.
- Ajouter 12,6 ul d'HEPES 1M, 20 pl d'eau filtrée, 50 pl de solution de la cellule, puis 10 ul de particules fluorescentes.
- Enfin, ajouter 167 l de collagène du derme bovin J'ai solution (pour obtenir une concentration de travail de 1 mg / ml).
- Mélanger une solution à fond, puis la pipette 80 ul de la solution sur la partie en verre du plat à fond de verre silanisé.
- Vaisselle Placer dans l'incubateur et permettre au gel de polymériser à 37 ° C pendant 30 minutes. Voir Figure 1A pour le gel de collagène dans un plat typique de p35 à fond de verre.
- Ajouter 2 ml de milieu de croissance avec soin pour chaque plat.
4. Procédure à suivre pour l'acquisition d'image time-lapse
- Allumez le contrôle de la température de l'enceinte de microscope et de laisser la chambre de la portée s'équilibrer à une température état d'équilibre à 37 ° C. Voir Figure 2 pour un exemple de système d'imagerie des cellules vivantes.
- Changer les médias aux nouveaux médias complété avec 25 mM de HEPES à maintenir un pH neutre. PourDIC imagerie, de l'échange au sommet de la parabole avec un qui a un dessus en verre. En utilisant une mince bande de parafilm, couvrir le côté du plat pour éviter l'évaporation des médias.
- Pour un objectif à immersion d'huile, déposer une goutte de liquide d'immersion sur l'objectif.
- Placer le gel de collagène contenant plat sur la platine du microscope, et assurez le plat en contact avec le liquide d'immersion.
- Axer la recherche d'échantillon et les cellules d'intérêt pour l'image. Pour minimiser la dérive scène, permettent de régler plat pendant environ 45 minutes avant de commencer une capture longtemps.
- Spécifiez les paramètres d'acquisition des images. Minimiser l'exposition au laser, ce qui pourrait endommager les cellules, en modulant la puissance du laser, le temps d'exposition, la fréquence et la durée du temps de capture. Les paramètres réels varient avec le système de microscope et le type de cellule.
- Lors de l'acquisition time-lapse, un inhibiteur peut être ajouté. Pour l'expérience de plus inhibiteur, ne pas sceller le plat avec du parafilm.
- Préparationsont complétées Hepes médias avec la drogue, à une concentration de travail désirée.
- Lorsque vous êtes prêt à ajouter des médicaments aux médias, mettre en pause la capture d'image, et enlever soigneusement le dessus plat sans déranger le plat.
- Aspirer à la presse dans le plat, et une pipette contenant le médicament des médias dans le plat. Puis, soigneusement remplacer le dessus plat.
- L'échantillon peut être hors-de-foyer à cause de l'ajout de nouveaux médias, réajuster la mise au point.
- Redémarrez la capture, puis de surveiller les captures pour les 30 prochaines minutes, et réajuster la mise au point si nécessaire. Voir Figure 4 pour un exemple de l'expérience de plus de drogue.
5. Procédure de FRAP et de l'analyse
- La configuration du PAF varie entre les systèmes, suivre les instructions du fabricant.
- Mettre en place des paramètres pour imagerie des cellules vivantes.
- Mettre en place des paramètres pour photoblanchiment. Utilisez la puissance du laser suffit à photobleach le signal fluorescent sans endommager les cellules. Testez ces nominaleameters sur les cellules pratique.
- Démarrer la capture d'image, et attendre au moins 5 images de la capture d'image avant photoblanchiment la région d'intérêt.
- Continuer la capture, et le temps de récupération devrait être suffisant pour capturer le plein rétablissement de fluorescence. Voir Figure 5 pour un exemple de l'expérience de FRAP.
- Analyser la récupération de fluorescence en mesurant l'intensité moyenne de fluorescence de la zone photobleached (avant et après photoblanchiment) au cours du temps. L'utilisation d'un logiciel d'analyse statistique (Excel par exemple), l'ajustement de la courbe de récupération de l'équation exponentielle: I = f (1-e-kt), où I est l'intensité, je f est l'intensité finale, t le temps, et τ ½ est le temps qu'il faut pour l'intensité pour atteindre la moitié de la valeur finale: τ ½ = ln (2) / k. La mi-temps est une mesure du taux de mobilité de la protéine.
- Obtenir les paramètres, à mi-temps et l'intensité finale de la courbe exponentielle ajustement. Calculer ee fraction mobile en prenant le rapport de la finale à la première intensités de fluorescence.
6. Les résultats représentatifs
Un exemple de l'imagerie des cellules vivantes des cellules épithéliales saines au sein de la matrice est montré dans la figure 3. Les cellules saines présentent une surface lisse, membrane continue, et noyau distinct, tandis que les cellules malsaines ont souvent une membrane perturbé, et un nombre excessif de vacuoles. Dans une matrice 3D, seule la migration des cellules épithéliales 1, et au cours de plusieurs jours, les cellules épithéliales forme tridimensionnelle, sphérique, multi-cellulaire des kystes au sein de la matrice 2. Les cellules sont également très dynamiques, et de migrer dans le kyste (figure 3). La déformation de matrice à la suite des forces de traction exercée par les cellules migrent sont analysées par le déplacement de particules traçantes noyées dans la matrice environnante (figure 4). Les mouvements des particules traceurs sont affichés comme une image de projection maximale de varierpoints de temps différentes, et chaque moment est une pseudo-couleur pour indiquer le temps (figure 4B). Sinon, les images d'une particule traceur individuels sont affichées comme un montage pour montrer le mouvement de la particule traceur au cours du temps (figure 4C). Toutes analyse d'image a été réalisée en utilisant ImageJ. Ces évaluations qualitatives de la déformation matrice, et donc les forces exercées par les cellules migrent, sont utiles en première approximation de la répartition de la force de traction. L'estimation quantitative des forces de traction exercée par les cellules migrent en 3D est au-delà de la portée de ce protocole et est décrit ailleurs 3,4.
Une reprise typique de fluorescence après photoblanchiment expérience est montré dans la figure 5. La région d'intérêt doivent être photobleached utilisant des paramètres laser optimisés de sorte que l'intensité de fluorescence est visiblement diminué par rapport aux niveaux de fond (pour maximiser signal-bruit), tout en préservant les cellules saines et en bon état. Le réglage optimal doit be déterminée empiriquement que chaque configuration est différente FRAP (par exemple, le FRAP en utilisant le système de balayage laser confocal).
L'exposition et intervalle de temps d'acquisition des images post-FRAP doit être soigneusement contrôlée pour éviter photoblanchiment fond. L'utilisation de faible puissance laser, temps d'exposition court, et l'utilisation de la caméra sensible et optique haute efficacité sont essentiels pour une qualité d'image haute FRAP. Si la fluorescence de blanchiment lors de l'acquisition d'image est importante, ce fondu de fond doivent être quantifiés et normalisés de la reprise de l'intensité de fluorescence observée. En raison de la nature 3D de l'environnement, la composante z de la reprise d'intensité de fluorescence peuvent devenir importants. Par conséquent, il est important de définir un volume photobleached en prenant pré-et post-stack images 3D de la région de photobleached. Une analyse plus poussée et la modélisation de la récupération de fluorescence est discutée ailleurs 5,6.
La combinaison unique d'imagerie des cellules vivantes techniques: GFP de suivre la dynamique des protéines du cytosquelette, la fluorescence rouge particules traceur pour surveiller la déformation de matrice, et l'addition d'inhibiteurs, peuvent être utilisés simultanément pour l'analyse de la dynamique des protéines, la force de traction et de voies moléculaires.
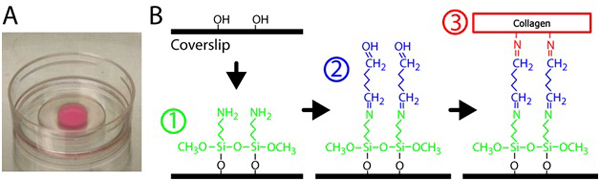
Figure 1. Préparation du gel de collagène. A) la matrice de collagène polymérisé sur un plat à fond de verre. La couleur rose du gel est due à des particules fluorescentes intégré. B) Procédure pour traiter les plats à fond de verre pour réticuler le gel de collagène à la surface du verre. Tout d'abord, le plat à fond de verre est traitée avec 3-aminopropyltriméthoxysilane solution, une solution de glutaraldéhyde alors que la matrice de collagène réticule sur le verre.
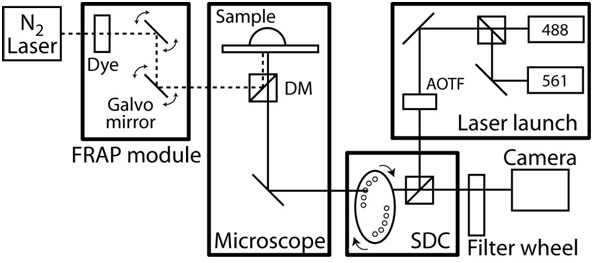
Figure 2. Schéma de configuration microscope confocal / FRAP. Le confocalemicroscope est basé sur un AxioObserver Zeiss avec une caméra CCD Coolsnap HQ II et complètement automatisé par Slidebook logiciels (Intelligent innovations en imagerie). L'unité est conçue sur mesure confocale et basé sur l'unité Yokogawa disque rotatif CSU10 et deux lasers à état solide (488 nm à 50 mW et 561 nm avec 40 mW) avec acousto-optique à filtre accordable (AOTF) pour permettre la commutation entre deux millisecondes lasers. Les filtres d'émission sont nm 525/50 et 620/60 nm (# 118661 et # 118085, technologie Chroma) et miroir dichroïque est Brightline 488-568 bi-bande (Semrock). Les objectifs sont une longue distance de travail 40x C-Apochromat objectif avec NA 1.1 et distance de travail de 0,62 mm, et un 63x Plan-Apochromat objectif avec NA 1,4 et une distance de travail de 0,19 mm. Le microscope comprend également un système de photoablation FRAP qui se compose d'une fibre laser à colorant pompé optiquement, une position du faisceau contrôlé par ordinateur et d'intensité, et une taille de tache de diffraction limitée. Par ailleurs, le microscope est équipéune platine XY motorisée, qui comprend 0,1 microns encodeurs linéaires sur chaque axe. Au cours de l'imagerie time-lapse, la température ambiante est maintenue par une chambre du microscope conçu sur mesure et un appareil de chauffage avec un contrôle de température retour. Pour isoler le bruit et les vibrations, le système de microscope entier est sur une table de vibrations.
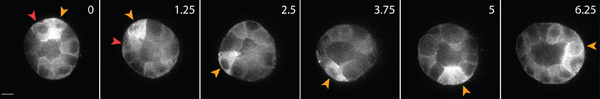
Figure 3. Direct imagerie cellulaire de cellules épithéliales exprimant la GFP-actine. Ces cellules forment un kyste au bout de 4 jours de culture dans une matrice de collagène 3D. Certaines cellules se déplacent le long de la surface du kyste (flèche jaune), tandis que d'autres migrent à l'intérieur de kystes (rouge pointe de flèche). La barre d'échelle 10 microns, le temps en heures.
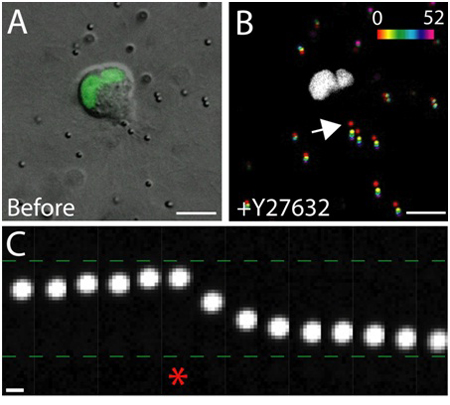
Figure 4. L'effet de l'inhibition de la Rho-kinase sur la force de traction. A) l'image DIC d'une cellule de migration MDCK exprimant GFP taggés marqueur nucléaire. L'image a été prise immédiatement avant le traitement de l'inhibiteur de la Rho-kinase Y-27632. La barre d'échelle 10 microns. B) Le déplacement des particules résultant de l'addition de Y-27632. Les positions des particules à différents temps (0 - 52 min) ont été pseudo-colorée en fonction de l'échelle d'intensité, puis projetée sur une seule image. La zone blanche est le noyau GFP-positives dans les cellules en migration. La barre d'échelle 10 microns. Temps en minutes. C) Le mouvement des particules sur le bord de fuite de la cellule (voir flèche en B). Astérisque indique la dernière image capturée avant Y-27632 plus. La particule traceur déplacé vers et loin du bord de fuite de la cellule migration avant et après l'ajout de Y-27632, respectivement. Échelle barre de 1 um.
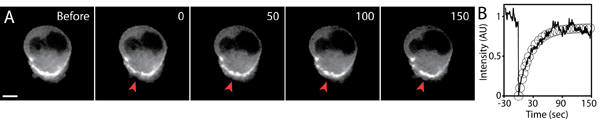
Figure 5. Analyse du PAF de GFP-actine exprimer la migration de cellules dans une matrice tridimensionnelle. Images Timelapse A) de la GFPactine cellule exprimant des avant et après le photoblanchiment. Une petite région de la GFP-actine accumulée à l'arrière des cellules a été photobleached (rouge pointe de flèche) au point de temps 0. Temps en secondes, barre d'échelle 5 um. B) L'intensité moyenne de fluorescence de la région photobleached (ligne continue), et l'ajustement exponentiel de la reprise de fluorescence (cercles) en fonction au cours du temps. L'intensité de fluorescence a été normalisée à la valeur initiale avant photoblanchiment.
Discussion
Nous décrivons ici une méthode pour utiliser imagerie des cellules vivantes pour étudier les mécanismes de la migration de cellules dans une matrice tridimensionnelle. Le succès de cette technique dépend de l'obtention de "bons" clones exprimant de façon stable la GFP-tagged protéines. Le faible niveau de protéines GFP, il faudra une exposition d'excitation en excès qui compromet la santé des cellules, alors que le niveau trop élevé GFP aura des effets secondaires indésirables sur la cell...
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Nous remercions le Dr Grant Sumida pour une lecture critique du manuscrit. Ce travail a été soutenu par une bourse de jeune chercheur Beckman (SY), une famille Hellman Faculté nouveau prix (SY), un EUREKA NIH, l'Université de Californie recherche sur le cancer comité de coordination.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | Commentaires |
| Collagène, bovins, de type I | BD Biosciences | 354231 | Stock est d'environ 3 mg / ml |
| 3-aminopropyltriméthoxysilane | Sigma-Aldrich | 281778 | Diluer dans de l'eau |
| glutaraldéhyde | Sigma-Aldrich | 340855 | Diluer dans du PBS |
| Hepes 1M | Invitrogen | 15630-080 | |
| Polystyrène Fluospheres microsphères 1 pm, fluorescence rouge (580/605) | Invitrogen | F13083 | |
| Généticine (G418) | Invitrogen | 11811-031 | |
| Culture composantes médias: | |||
| DMEM | Invitrogen | 31600-034 | |
| Sérum de veau fœtal | Atlanta Biologicals | S115500 | |
| Pénicilline / streptomycine | Invitrogen | 15140-122 | |
| Kanamycine | Invitrogen | 15160-054 | |
Références
- Shih, W., Yamada, S., Myosin, . A dependent retrograde flow drives 3D cell migration. Biophys. J. 98 (8), L29-L31 (2010).
- O'Brien, L. E., Yu, W., Tang, K., Jou, T. S., Zegers, M. M., Mostov, K. E. Morphological and biochemical analysis of Rac1 in three-dimensional epithelial cell cultures. Methods Enzymol. 406, 676-691 (2006).
- Legant, W. R., Miller, J. S., Blakely, B. L., Cohen, D. M., Genin, G. M., Chen, C. S. Measurement of mechanical tractions exerted by cells in three-dimensional matrices. Nat. Methods. 7 (12), 969-971 (2010).
- Del Alamo, J. C., Meili, R., Alonso-Latorre, B., Rodriguez-Rodriguez, J., Aliseda, A., Firtel, R. A., Lasheras, J. C. Spatio-temporal analysis of eurkaryotic cell motility by improved force cytometry. Proc. Natl. Acad. Sci. U.S.A. 104 (33), 13343-13348 (2007).
- Lippincott-Schwartz, J., Snapp, E., Kenworthy, A. Studying protein dynamics in living cells. Nat. Rev. Mol. Cell Biol. 2 (6), 444-456 (2001).
- Phair, R. D., Misteli, T. Kinetic modeling approaches to in vivo imaging. Nat. Rev. Mol. Cell Biol. 2 (12), 898-907 (2001).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. Fluorescent toolbox for assessing protein location and function. Science. 312 (5771), 217-224 (2006).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Methods. 2, 905-909 (2005).
- Cukierman, E., Pankov, R., Stevens, D. R., Yamada, K. M. Taking cell-matrix adhesions to the third dimension. Science. 294 (5547), 1708-1712 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon