Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Preparación de segmentados microtúbulos para estudiar las mociones impulsadas por las que desmontan microtúbulos Ends
En este artículo
Resumen
Los microtúbulos son polímeros inherentemente inestables, y su conmutación entre el crecimiento y acortamiento es estocástico y difícil de controlar. Aquí se describen los protocolos que utilizan microtúbulos segmentados con tapones estabilizadores fotoescindible. La despolimerización de los microtúbulos segmentados puede ser activado con una alta resolución temporal y espacial, ayudando de este modo el análisis de los movimientos con los extremos de los microtúbulos desmontaje.
Resumen
Despolimerización de microtúbulos puede proporcionar la fuerza para el transporte de diferentes complejos de proteínas y los granos proteicos recubierto in vitro. Los mecanismos subyacentes se cree que desempeñan un papel vital en los movimientos de los cromosomas microtúbulos dependientes durante la división celular, pero las proteínas correspondientes y sus funciones exactas están mal definidos. Por lo tanto, hay una necesidad creciente de desarrollar ensayos con el que estudiar tales motilidad in vitro utilizando componentes purificados y medio bioquímica definida. Los microtúbulos, sin embargo, son inherentemente inestables polímeros; su conmutación entre el crecimiento y acortamiento es estocástico y difícil de controlar. Los protocolos que describimos aquí se aprovechan de los microtúbulos segmentados que se hacen con el fotoescindible estabilizar tapas. La despolimerización de microtúbulos tales segmentados se puede activar con alta resolución temporal y espacial, ayudando así a los estudios de la motilidad en los extremos de los microtúbulos desmontaje. Esta técnica se puede utilizar para CArry a cabo un análisis cuantitativo del número de moléculas en los complejos de proteínas marcadas con fluorescencia, que se mueven processively con los extremos de los microtúbulos dinámica. Para optimizar una relación señal-ruido en este y otros ensayos fluorescentes cuantitativos, cubreobjetos deben ser tratados para reducir la absorción no específica de proteínas marcadas fluorescentemente-solubles. Protocolos detallados se proporcionan a tener en cuenta la falta de uniformidad de la iluminación fluorescente, y determinar la intensidad de un solo fluoróforo utilizando ajuste gaussiano equidistantes. Por último, se describe el uso de los microtúbulos segmentados para estudiar los movimientos de los microtúbulos dependientes de las microperlas de proteína-revestido, proporcionando conocimientos sobre la capacidad de las diferentes proteínas motoras y no motoras para acoplar la despolimerización de microtúbulos al movimiento de carga processive.
Introducción
Los microtúbulos son altamente conservadas estructuras citoesqueléticas que son importantes para la arquitectura celular, la motilidad celular, la división celular, y el transporte intracelular 1. Estos polímeros dinámicos se ensamblan a partir de tubulina en presencia de GTP, y cambian de forma espontánea entre el crecimiento y acortamiento 2. Los microtúbulos son muy delgado (sólo 25 nm de diámetro), por lo tanto las técnicas ópticas especiales para incrementar el contraste debe ser utilizado para observar los microtúbulos con un microscopio óptico. El trabajo previo con estos polímeros examinó su comportamiento dinámico mediante contraste de interferencia diferencial (DIC) 3. Este y otros estudios in vitro revelaron que en condiciones típicas experimentales, los microtúbulos se someten a la catástrofe y cambie a la despolimerización sólo en raras ocasiones, una vez cada 5-15 minutos (esta frecuencia es de 7-15 concentración de tubulina soluble mM examinada en 28-32 ° C ) 4. Así, se han propuesto diferentes técnicas para la induccióne microtúbulos despolimerización de una manera controlada. Acortamiento de microtúbulos puede ser activado mediante el lavado de distancia tubulina soluble en 5,6, corte microtúbulos con un haz de láser 7, o el uso de los microtúbulos segmentados 8, tal como se describe aquí. El trabajo previo utilizando los microtúbulos segmentados, así como polímeros de conmutación estocásticamente, se ha encontrado que pequeñas cargas intracelulares, tales como cromosomas, vesículas, y perlas de proteína recubiertos, se pueden mover en los extremos de los microtúbulos acortamiento 9-13. Este fenómeno se cree que tiene una implicación directa de los movimientos de los cromosomas en las células mitóticas, y los mecanismos subyacentes están actualmente bajo investigación activa 14-16.
Recientemente, las técnicas a base de fluorescentes, incluyendo la reflexión interna total de fluorescencia (TIRF) microscopía, se han empleado para estudiar la motilidad con microtúbulos dinámicos extremos 17-24. La ventaja de este enfoque es que permite el examen de la interaccións entre los microtúbulos y proteínas de unión a microtúbulos en tiempo real utilizando proteínas marcadas con diferentes fluoróforos. Se encontraron varios complejos de proteínas para mover processively con el alargamiento y / o acortamiento de los extremos de los microtúbulos. Ellos incluyen las proteínas asociadas a los microtúbulos Dam1 10,12,18, Ska1 19, y XMAP215 20, así como motores de quinesina Kif18A 21,22, MCAK 23 y CENP-E 24. Estas proteínas exhiben processive punta-de seguimiento, que es fundamentalmente diferente de la de las proteínas de punta de seguimiento de clásicos como EB1 25. Aunque las moléculas EB1 y los socios parecen permanecer de manera estable asociada a los microtúbulos dinámica termina, las moléculas individuales permanecen atados a la punta de los microtúbulos por sólo ~ 0,8 seg, intercambiar rápidamente con la piscina soluble 26. Por el contrario, procesivas punta-trackers, como Dam1, viajan con los microtúbulos termina para muchos micras, y su asociación con las puntas de los microtúbulos pueden durar mningún segundo. La punta tiempo asociación, así como el que resulte de seguimiento, depende en gran medida del número de moléculas que forman la punta de rastreo complejo 27. Grandes conjuntos de proteínas suelen ser mucho mejores de punta-trackers. Por ejemplo, dichos ensamblajes complejos como los cinetocoros de levadura aisladas pueden permanecer acoplado a microtúbulos termina por hora 28. Algunas proteínas de unión a microtúbulos, por ejemplo Ndc80 complejo de proteínas cinetocoro, se han encontrado para ser incapaces de realizar un seguimiento con microtúbulos termina en un solo nivel de molécula, sin embargo Ndc80 es muy eficiente en el acoplamiento del movimiento del talón de carga 19,29-31. Por lo tanto, para entender el mecanismo de la punta de seguimiento por diferentes complejos de proteínas, así como sus funciones biológicas, es importante examinar la punta de seguimiento como una función del número de moléculas en el complejo punta de seguimiento, así como para determinar la capacidad de estos complejos para exhibir la motilidad colectiva en la superficie de la carga de talón.
A continuación ofrecemos protocolos detallados para preparar y llevar a cabo experimentos con los microtúbulos segmentados (Figura 1A). En primer lugar, los portaobjetos de vidrio disponibles comercialmente son modificados para unir tubos de polietileno corto (Protocolo 1). La cámara de flujo microscopía reutilizable se ensambla a partir de una diapositiva tal y el plasma-limpiado y cubreobjetos silanizada (protocolo 2) 32-34 químicamente o. El volumen de la cámara resultante es sólo 20-25 l (o tan pequeño como 15 l, véase la nota 3 en el Protocolo 1), incluyendo el volumen de la tubería de entrada. Cámaras de flujo disponibles comercialmente también se pueden usar, pero su volumen es generalmente más grande, que conduce a la pérdida innecesaria de proteínas. Si se emplea una cámara más grande, el volumen de todas las soluciones en los protocolos a continuación debe ser escalado proporcionalmente. Semillas de microtúbulos continuación, se preparan, por ejemplo, usando GTP analógico lentamente hidrolizable, GMPCPP (guanosina-5'-[(α, β)-methyleno] trifosfato) (Protocolo 3, ver también Hyman et al. 35). Las semillas se inmovilizan sobre un cubreobjetos de limpiado y la superficie es posteriormente bloqueando los comandos para prevenir la absorción no específica de otras proteínas 32 (protocolo de 4 describe semillas inmovilización utilizando digoxigenina). Los microtúbulos segmentados pueden ser preparados usando el Protocolo 5. La razón principal de este enfoque es que los polímeros de microtúbulos dinámicos, que se forman en presencia de GTP, se pueden estabilizar temporalmente mediante la adición de los cortos "topes" de segmentos de tubulina estables, que contienen GMPCPP. Estas tapas también contienen tubulina marcada con rodamina, para que puedan ser retirados simplemente iluminando el campo de visión con un láser de 530-550 nm o lámpara de arco de mercurio (Protocolo 6) 36. La intensidad de fluorescencia de la señal de la punta de seguimiento se puede usar entonces para estimar el número de moléculas que se desplazan con los extremos de los microtúbulos desmontaje, teniendo en cuenta la falta de uniformidad de la iluminación de campo del microscopio (Protocolo 7). Un enfoque similar se puede utilizarpara estudiar las interacciones entre los microtúbulos depolymerizing y perlas de proteína-revestido, preparado como se describe en el 27 (Protocolo de 8). Algunas proteínas se unen fácilmente a las paredes de los microtúbulos segmentados, pero pinzas láser también pueden utilizarse para sostener el cordón cerca de la pared de microtúbulos, promoviendo así su unión.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Equipo necesario: Los experimentos descritos a continuación requieren de un microscopio óptico equipado para DIC y fluorescencia de imagen (Tabla 1). Campo brillante iluminación LED se puede utilizar para mejorar de manera significativa la detección de las semillas de microtúbulos cubreobjetos-adjunto 37, que son difíciles de observar con una lámpara halógena regular. Para controlar el flujo de líquido en las cámaras de microscopía, las soluciones deben ser intercambiados con una bomba peristáltica capaz de velocidades de flujo de 10 a 100 l / min. Una bomba de jeringa también se puede utilizar pero se debe tener cuidado para evitar las burbujas de aire que se pueden formar cuando la velocidad de flujo se cambia bruscamente. Para la manipulación de perlas de proteína-revestido, por ejemplo, para llevarlos cerca de la pared de microtúbulos segmentado, una nm haz de láser de onda continua 1064 se puede introducir en el eje óptico del microscopio y se concentró con un objetivo de abertura numérica alta (1,3 o superior) para producir una trampa. Para el análisis cuantitativo de la fluorescenciaintensidad de las moléculas individuales de la luz de excitación debe ser proporcionada por una fuente láser-base ya que la intensidad de esta fuente de luz es más estable que la generada por una lámpara de mercurio. Para reducir al mínimo las vibraciones mecánicas, el microscopio se debe colocar en una mesa óptica. Se requiere un equipo más sofisticado para estudiar el movimiento de las perlas con la despolimerización de microtúbulos termina bajo una fuerza constante, y para medir las señales de fuerza de disparo único 11,38,39, estos métodos se describen en otra parte.
1. Fabricación reutilizables cámaras de flujo
Los portaobjetos de vidrio para cámaras de flujo reutilizables se pueden pedir a una planta de fabricación de vidrio local utilizando esquemas de la Figura 1B (ver Tabla 2 para obtener información sobre nuestro proveedor). Con la molienda ultrasónica modificar diapositivas normales de microscopio (75 mm x 25 mm, 1,0 mm de grosor) para hacer dos ranuras 15 ± 1 mm de largo, 1,0 ± 0,1 mm de ancho y 0,8 ± 0,05 mm de profundidad.Distancia entre los extremos más cercanos debe ser 14 ± 1 mm; esta distancia es óptima para una cámara montada con 22 mm x 22 mm cubreobjetos. Consulte la Tabla 2 para obtener una lista de otros materiales.
- Coloque un tubo largo de polietileno 100 mm (OD 0.61 mm, Tabla 2) en cada ranura de la diapositiva, dejando ~ 5 mm voladizos en los extremos interiores de las ranuras. Fijar los tubos dentro de las ranuras con adhesivo de cianoacrilato, la incorporación de los tubos completamente dentro de las ranuras.
- Llene las ranuras con pegamento epoxi, evitando derramar el pegamento dentro de los tubos. Deje que el pegamento se seque durante aproximadamente 1 día.
- Con una hoja de afeitar afilada cortar la masa solidificada pegamento de 3-4 mm desde el extremo distal de cada sitio de unión, la eliminación de las partes proximal al centro de la diapositiva. Los tubos deben permanecer dentro de sus ranuras. Retirada de las piezas proximales también cortar y eliminar los voladizos interiores, creando una superficie plana con dos aberturas del tubo.
- Llene una jeringa con agua y pruebasi las trompas están funcionando correctamente. Si el líquido fluye libremente, poner una gota de pegamento epoxi (~ 5 mm de diámetro) en los extremos exteriores de las arboledas y secos por 1 días (Figura 1D). Esto hará que las cámaras más duradera, por lo que puede ser utilizado en varias ocasiones durante muchos meses.
Nota 1: Para hacer una cámara para un microscopio invertido, los portaobjetos deben modificarse adicionalmente para hacer dos agujeros pequeños en los extremos opuestos de las ranuras (Figura 1C). Inserte los tubos a través de los agujeros en la diapositiva, doblar los tubos y que encajen perfectamente dentro de las ranuras (Figura 1E). Siga los pasos 1.2 a 1.4, pero quitar el pegamento epoxi de toda la superficie, que se utiliza para hacer una cámara de flujo.
Nota 2: Para reducir el volumen de la cámara, utilice la molienda para hacer dos muescas 0,050 ± 0,005 mm de profundidad, dejando la parte central de la diapositiva 5.0 ± 0.5 mm de ancho y ligeramente elevada (ver "áreas grabadas" en las figuras 1B y 1C). Cuando tcámara de flujo que se monta (como se describe más adelante), coloque la cinta de doble cara en el interior de estas hendiduras.
Nota 3: Para volver a utilizar estas diapositivas modificados, después de terminar los experimentos quitar el cubreobjetos y la cinta de doble cara con una cuchilla de afeitar. Retire el sellador pelando apagado y limpiando el portaobjetos con etanol al 70%. Colocar el portaobjetos en un recipiente con un 1-2% de un detergente para lavar platos de laboratorio, conecte un tubo a una bomba peristáltica y perfundir 50-70 ml, siga con igual volumen de agua desionizada, se seca y guardar en un compartimento libre de polvo.
2. Preparación de Cubreobjetos
Este protocolo tiene 6-8 horas y ayudará a preparar 12 cubreobjetos. Usted necesitará un titular cubreobjetos cerámica y 3 recipientes de tinción cubreobjetos con tapas; volumen frasco debe ser de 15 ml, por lo que cada uno tendrá 4 cubreobjetos apilados juntos. Un frasco de vidrio con una tapa (250 ml) se debe utilizar para incubar cubreobjetos con silano. Utilice cubreobjetos de vidrio No.1 regulares (22 mm x 22 mm o 22 mm x 30 mm, ver tablas 2 y 3 para una lista de materiales). Todos los pasos deben llevarse a cabo en una campana de extracción, mientras que el uso de guantes.
- Ponga los portaobjetos en los recipientes de tinción cubreobjetos de vidrio y llenar los frascos con acetona. Incubar durante 1 hora, lavar 10 veces con agua desionizada.
- Incubar los cubreobjetos de 10 min con etanol y lavar de nuevo 10 veces con agua desionizada.
- Preparar la solución de "pirañas". Poner 60 ml de solución de peróxido de hidrógeno (30% en agua) en un recipiente de vidrio resistente al calor y añadir lentamente 100 ml de ácido sulfúrico (relación final de ácido a la solución de peróxido de hidrógeno es 05:03). La solucion se calienta, esto es normal, pero tenga cuidado. Solución Piranha es extremadamente corrosivo! Utilice laboratorio grueso abrigo, guantes y gafas de protección!
- Llene los recipientes de tinción cubreobjetos con solución "piraña", cerrar los párpados y colocar los frascos en un baño de agua previamente calentada a 90 ° C durante 1 hora.
- Se retira la soluti "piraña"en y deseche según las instrucciones de las regulaciones de seguridad en su lugar de trabajo. Lave cubreobjetos 10 veces con agua desionizada.
- Llene los recipientes de tinción cubreobjetos con 0,1 M de KOH, incubar 10 minutos, y lavar 10 veces con agua desionizada. Esto neutralizará cualquier residuo ácido que quedan en los cubreobjetos después del tratamiento "pirañas".
- Cubreobjetos secos, uno a la vez manteniendo cada cubreobjetos con unas pinzas plana con bordes recubiertos de teflón (para minimizar el daño a una superficie de vidrio) y mientras se sopla nitrógeno seco comprimido. Asegúrese de que los cubreobjetos se secan por completo, ya que la solución de silano es altamente reactivo con el agua.
- Pila de los cubreobjetos secos en los titulares de cerámica (12 cubreobjetos por titular), que deben ser previamente secado a fondo con nitrógeno. Mantener los titulares de cerámica cubiertos para evitar el polvo se pegue a la superficie de cubreobjetos.
- Cubrir el fondo del tarro de 250 ml de vidrio (6 cm de diámetro) con tamices moleculares, grado 564, para la absorción de agua.
- Llene el frasco con 200 ml deSolución PlusOne Repel Silano y sumergirse lentamente un soporte cerámico con cubreobjetos en un frasco, cierra la tapa y se incuba durante 5 minutos a temperatura ambiente. Esto creará recubrimiento hidrófobo sobre la superficie cubreobjetos.
- Retire lentamente el soporte con cubreobjetos de la jarra y la transferencia cubreobjetos uno a la vez en los recipientes de tinción cubreobjetos llenos de metanol.
- Coloque un metal o pedestal de vidrio en el depósito de agua de un baño de ultrasonidos, de tal manera que la cubeta de tinción cubreobjetos se sumerge durante 2/3 de su altura. Someter a ultrasonidos a 70 W durante 20 min, el cambio de solución de metanol cada 5 min, luego enjuague 10 veces con agua desionizada. Si la silanización funcionaba correctamente, aparecerán en seco los cubreobjetos cuando se retira del agua.
- Eliminar completamente el agua residual utilizando nitrógeno, como anteriormente.
- Interlay los cubreobjetos con Kimwipes para evitar el contacto de superficie a superficie entre los cubreobjetos. Los cubreobjetos se pueden almacenar en un contenedor sellado durante varias semanas a temperatura ambiente.
Nota 1: Los pasos 2.1 a 2.6 se puede sustituir por la limpieza de los cubreobjetos con el limpiador de plasma durante 15 minutos a 30 W, lo que reduce considerablemente el tiempo de preparación total. La presión dentro de la cámara de limpieza se fija en 100-200 mTorr. Tanto el oxígeno atmosférico y comprimido se puede utilizar. Apilar los cubreobjetos plasma limpiado en los titulares de cerámica y vaya al paso 2.7.
3. Preparación de microtúbulos estabilizados-GMPCPP Semillas
Este procedimiento se llevará a ~ 1 hr y las semillas de microtúbulos resultantes son estables durante 1-2 días a temperatura ambiente. Consulte la Tabla 4 para una lista de los reactivos.
- Mezcle en el hielo:
- 10 l no marcados tubulina (100 M, Tabla 4) en BRB-80 tampón (Pipes 80 mM, EGTA 1 mM, MgCl2 4 mM, pH 6,9 con KOH; suplemento con TDT 1-2 mM utilizando alícuota para cada experimento).
- 2,6 l marcada con digoxigenina tubulina (Tabla 4). Ajuste el volumen en función de la preparación,de tal manera que la proporción final de la etiqueta a la tubulina no marcada es ~ 1:10. Mezcle bien con la pipeta.
- 1,4 l mM GMPCPP 10 (concentración final 1 mM)
- Incubar 15 min a 35 ° C, las semillas crecerán 2-3 micras de largo. Ajustar el tiempo si se desea diferente longitud de los microtúbulos.
- Añadir 35 l BRB-80 (precalentado a 35 ° C), se mezcla con la pipeta, y se centrifuga durante 15 minutos a 25.000 xg para sedimentar las semillas a temperatura ambiente.
- Desechar el sobrenadante, lavar el pellet añadiendo con cuidado y quitando 50 l de BRB-80 caliente.
- Resuspender pellet bien en 25 l BRB-80.
4. La unión de los microtúbulos semillas a los Cubreobjetos
Protocolos 4 y 5 requieren 2-3 horas, por lo que dos cámaras de flujo se utilizan por día.
- Montar la cámara de flujo según las instrucciones del fabricante utilizando cubreobjetos silanizadas y vaya al paso 4.2. Si utiliza cubreobjetos medida (Protocolo 1), siga el bel pasosow.
- Coloque dos trozos de cinta adhesiva de doble cara (5 mm x 30 mm) a lo largo de la zona ancha mm ~ 5 central, poner cubreobjetos silanizada encima de la cinta, presione con firmeza.
- Llenar la cámara con BRB-80 a través de uno de los tubos y conecte ambos tubos con palillos de dientes redondos.
- Coloque una pequeña gota de bicolor Kwik sellante moldeada en la parte superior de una pequeña placa Petri de plástico, y mezclar rápidamente, pero a fondo con un palillo de dientes. El sellador se pondrá verde, aplicar de inmediato, sellando cuidadosamente todos los bordes del cubreobjetos. Si el sellador penetra muy profundamente bajo el cubreobjetos, abra uno de los tubos quitando el tapón de palillo de dientes y aplique una presión suave para evitar que el sellador se filtre en el interior de los tubos.
- Deje secar la cámara durante 10 minutos y confirme que el flujo no está restringida antes de seguir adelante.
- Coloque la cámara en una platina del microscopio precalentada a 32 ° C y colocar uno de los tubos a una bomba, que bombear el líquido fuera. La longitud del tubo de entradadebe reducirse al mínimo para evitar la pérdida innecesaria de los reactivos: la longitud recomendada es de 5-7 cm. Sumerja este fin en un vial de 0,5 ml con BRB-80 buffer. Esta y todas las soluciones a continuación debe ser precalentado a 32-35 ° C.
- Aplicar una suave presión con una bomba o simplemente levantar el extremo del tubo de salida para exprimir las burbujas de aire, que pueden formar ocasionalmente cuando se retira el tapón de tubo de entrada.
- Ajuste la velocidad de la bomba a 100 l / min. Lavar con 2 volúmenes de cámara de anticuerpos anti-digoxigenina diluidas 1:30 en BRB-80, incubar 15 min para permitir la adsorción de anticuerpos.
- Lavar con 5-10 volúmenes de cámara de BRB-80 caliente, incubar 10 min con 1% de Pluronic F-127 en BRB-80 para bloquear la superficie hidrofóbica de cubreobjetos silanizada.
- Lavar con 5-10 volúmenes de cámara de tampón de la motilidad (BRB-80 suplementado con 0,4 mg / ml de caseína).
- Reduzca la velocidad de la bomba de 10 l / min y perfundir semillas microtúbulos diluida 1:200-1:600 en 30-40 búfer motilidad l. Incubar 15 millasn para promover la unión de las semillas a los anticuerpos cubreobjetos-adsorbido.
- Lavar la cámara a 100 l / min con 400 l de tampón de la motilidad para eliminar cualquier material no unido.
Nota 1: La densidad resultante de semillas debe ser 10-30 por campo microscópico (Figura 2A). Para solucionar problemas, utilice la tubulina marcado con fluorescencia durante la polimerización (paso 3.1) para la detección fácil de las semillas cubreobjetos inscritos.
Nota 2: axonemas preparados a partir de Chlamydomonas 40 o de otras fuentes biológicas, así como las unas películas de células lisadas Tetrahymena y deciliated 41 también se pueden utilizar como nucleadores de microtúbulos. Estos nucleadores son útiles para crear arreglos pequeños microtúbulos, y se prefieren cuando microtúbulos con número específico de protofilamentos se desean (GMPCPP nuclea semilla uno de los microtúbulos que contiene ≥ 14 protofilamentos 42). Estas estructuras se pueden unir a los cubreobjetos limpiado por la absorción no específica, pero la Attachment suele ser menos estables en comparación con el apego a base de anticuerpos, especialmente cuando se utilizan los cubreobjetos silanizadas.
5. Preparación de Segmented microtúbulos
Todos los volúmenes de solución a continuación son para el volumen de la cámara de 15 a 20 l, aumentar proporcionalmente si se utiliza la cámara más grande.
- Precaliente mezcla tubulina sin marcar (búfer motilidad 45 l suplementado con 1 mM Mg-GTP y con 10-15 M tubulina sin etiqueta) durante 30 segundos a 35 ° C. Perfusión a 30 l / min.
- Controle atentamente el crecimiento de microtúbulos con DIC óptica (Figura 2B, vídeo 1). Durante 5-7 minutos de incubación de los microtúbulos por lo general crecen ~ 10 micras de largo.
- Preparar la mezcla Rodamina-tubulina (65 l de tampón suplementado con motilidad mM GMPCPP 0,5 y 2-5 M de tubulina marcada con rodamina con 0,5-1 relación molar de Rodamina a la tubulina) y calentar la solución a 35 ° C durante 30 seg.
- Perfusión de inmediato a 30 μl / min. Se incuba durante 8-10 minutos para promover la formación de tapones fluorescentes estables en las puntas de los microtúbulos. Segmentos de microtúbulos estables también se nuclean espontáneamente y serán visibles con la óptica DIC.
- Lavar la cámara de bien con 100 l de tampón de la motilidad en 20 l / min para eliminar tubulinas y nucleótidos, así como fragmentos de microtúbulos solubles.
- Con la DIC, confirman que los microtúbulos son visibles (Figura 2D), su número, sin embargo, debería disminuir debido a que muchos microtúbulos desarme durante el taponado (Video 2 muestra un campo típico con los microtúbulos segmentados).
Nota 1: segmentado microtúbulos son muy estables y pueden utilizarse durante al menos 2 horas. Sin embargo, la vida útil de estos microtúbulos disminuye con la solución de intercambio excesiva, o si 2-mercaptoetanol se utiliza en la memoria intermedia de imágenes.
6. Observación experimental de la Proteína de seguimiento con despolimerización de microtúbulos Ends
- IntrodUCE 30-50 l de proteína fluorescente (0,1-20 nM) en la cámara a 10 l / min. Si la proteína se pegue a la cubreobjetos es evidente, complemento a la memoria intermedia de la motilidad con 4-8 mg / ml de BSA. Alexa488-Dam1 punta de seguimiento requiere además mM DTT 10 o 0,5-1% βME 10.
- Limite el campo de iluminación utilizando un diafragma de campo del microscopio para evitar el blanqueo innecesario y desmontaje de los microtúbulos.
- Comience adquisición de vídeo mediante cubo de filtro de las buenas prácticas agrarias, y luego cambiar a Rodamina cubo de filtro sin interrumpir la grabación de imágenes. Los segmentos de color rojo en los extremos de los microtúbulos deben ser claramente visibles, sino que comenzarán a desaparecer y se desintegran rápidamente (Video 3).
- Continuar para iluminar hasta las tapas de casi desaparecen (por lo general de 10 a 20 segundos, pero esta vez serán más largos por las tapas que crecen con una relación de etiquetado Rodamina inferior), y volver al canal para grabar GFP proteína de seguimiento con el desmontaje de microtúbulos.
- Analizar lasecuencias resultantes mediante la construcción de kymographs, es decir, imágenes de dos dimensiones que muestran la intensidad de fluorescencia a lo largo del eje de microtúbulos durante diversos tiempos durante la observación) usando MetaMorph, libremente disponible ImageJ u otro software de procesamiento de imágenes (Figura 2E).
Nota 1: Tasa de adquisición debe ser ajustado en función de la periodicidad de los fenómenos observados. La dosis recomendada es de 2-3 fotogramas por segundo (fps) para el movimiento lento, anillo de tamaño Dam1 complejos 27, pero el tiempo de adquisición de moléculas individuales debe ser> 20 fps 19.
Nota 2: A EMCCD muy sensible, por ejemplo ANDOR iXon3, es necesario para la grabación rápida de eventos punta de seguimiento con los microtúbulos depolymerizing. La configuración recomendada para cámara Andor iXon3 son: 5x ganancia, EM ganar 200, la velocidad de lectura de 1 MHz, el modo de sensor de 16 bits, 80 ms tiempo de exposición.
Nota 3: El uso de la microscopía TIRF será la relación señal-ruido mejora, sin embargo, se deben usar los microtúbulos más cortos, tales thaT las tapas de estabilización fluorescentes permanecen dentro del alcance de campo evanescente.
7. Análisis cuantitativo del tamaño molecular de los microtúbulos Complejos Tip-tracking
La justificación de este enfoque es determinar el número de moléculas en un complejo de punta de seguimiento mediante la búsqueda de la relación de la intensidad de fluorescencia total del complejo punta de seguimiento a la intensidad de un solo fluoróforo. Este enfoque se puede aplicar a fusiones de proteínas-GFP y proteínas marcadas con colorantes fluorescentes, pero puede subestimar el número de moléculas en los complejos punta-de seguimiento si algunas moléculas de proteína en las preparaciones no son fluorescentes.
- Cinética fotoblanqueo récord para las moléculas de proteína marcadas con fluorescencia.
- Montar cámara de microscopía regular usando un portaobjetos de vidrio no modificado, dos tiras de cinta adhesiva de doble cara, y un cubreobjetos limpio, que puede ser preparado usando todo el Protocolo 2, o sólo los pasos 2.1 a 2.6 de esteprotocolo.
- Añadir aproximadamente 50 nM de proteína en tampón de la motilidad, lavar brevemente con tampón de motilidad y sellar la cámara con valap (Tabla 4). Optimizar la concentración de proteína para obtener el campo con manchas uniformemente dispersas (Figura 3A), que representan las moléculas individuales y sus pequeños agregados (trímeros y tetrámeros, que pueden formar espontáneamente en la solución o pueden aparecer cuando varias moléculas individuales están muy juntas y no se pueden resolver) . Este paso es muy importante para la obtención de una distribución multi-pico de pasos de fotoblanqueo y determinación precisa de un tamaño de paso (ver más abajo).
- Minimizar la intensidad del láser de iluminación a la que las manchas fluorescentes individuales siguen siendo visibles; con iluminación inferior se extiende el tiempo de fotoblanqueo, por lo que las trazas de fotoblanqueo ya se pueden obtener. También minimizar el tiempo de exposición para reducir la probabilidad de más de un fluoróforo de blanqueo durante un fotograma. La configuración recomendadapara Andor iXon3 cámara: Ganancia 5.0x, EM ganar 999, la velocidad de lectura de 10 MHz, 50-100 milisegundos el tiempo de exposición.
- Centrarse en la superficie del cubreobjetos, cierre el obturador de iluminación, vaya a un campo nuevo, abra el obturador iluminación y adquirir imágenes hasta que todos los complejos han blanqueado (posteriormente denominado img (x, y)).
- Corregir las imágenes adquiridas para irregularidades de la iluminación (Figura 3B).
- Preparar la solución de cualquier fluoróforo, por ejemplo, 1 M isotiocianato de fluoresceína (FITC) en BRB-80. Tal solución puede ser preparada de antemano, se dividió en alícuotas y se almacenó a -20 ° C.
- Montar una cámara como en la Sección 7.1.1, pero utilizar un cubre regular. Añadir la solución de fluoróforo y sellar la cámara utilizando valap.
- Recoger> 50 imágenes de todo el campo microscópico: mover el escenario para una nueva área sin blanquear mientras el obturador iluminación está cerrada, y adquirir las imágenes inmediatamente después de abrir el obturador.
- Crear proyección media de la pila y el filtro con desenfoque gaussiano con un radio de 5 píxeles con ImageJ u otro software (Figura 3C). La imagen resultante representa la distribución de la intensidad de iluminación del campo (Illum (x, y), donde x e y corresponden a las coordenadas del píxel).
- Determinar el brillo máximo de píxeles de esta imagen (Max (Illum)).
- Con el obturador cerrado y el uso de la iluminación mismos ajustes de la cámara como en la Sección 7.2.3 adquirir una imagen, determinar la intensidad media de píxeles de esta imagen; este valor corresponde a CN, ruido de la cámara.
- Utilice los valores anteriores y la imagen (Illum (x, y)) para normalizar la imagen experimental (img (x, y)) con la siguiente expresión:
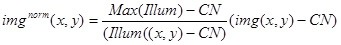
Utilice la norma imagen Img resultante (x, y) para el análisis cuantitativo de la BRIllo de los complejos fluorescentes estacionarias, y también para normalizar las imágenes con los complejos punta-viradas (Figura 3D).
- Determinar la intensidad de un solo fluoróforo.
- El uso de imágenes normalizadas img norma (x, y) y cualquier software de procesamiento de imagen, selecciona una mancha fluorescente con una región circular (5-6 píxeles de diámetro) y determinar su intensidad integral de todas las tramas, la generación de las huellas photobleaching. Evite mismos puntos brillantes (> 5 veces más brillante que los reguladores de intensidad).
- Utilizando la misma herramienta región circular, seleccione al menos 3 áreas libres de manchas, generar las trazas fotoblanqueo correspondientes, promedio y en forma con la función de decaimiento exponencial.
- Tabule este contexto curva de intensidad para que coincida con los puntos de tiempo de experimentación y restarlo de las curvas photobleaching.
- Suavizar las curvas photobleaching (promedio con la ventana deslizante de 3-5 puntos). Inspeccione visualmente las curvas resultantes ydescartar cualquier curva que muestra un aumento brusco en la fluorescencia o la falta de blanqueo evidente (Figura 3E).
- Para cada una de las curvas restantes (generalmente 50-70% del número total de curvas), seleccionar visualmente la meseta final, cuando la mancha fluorescente ha blanqueado. Acortar este segmento para dejar sólo ~ 100 puntos y un promedio de estas intensidades. Restar este valor de la curva de fotoblanqueo acortada para minimizar pequeñas variaciones es los niveles de fondo y para reducir el tamaño del pico de fondo (por debajo).
- Trazar un histograma de las intensidades de todos los puntos de tiempo de 20 o más curvas photobleaching (> 1000 puntos de tiempo). El histograma debe exhibir al menos 4 picos distintos (ver nota 2).
- Montar el histograma con la distribución de Gauss equidistante 10,43 utilizando MATLAB, Mathematica o software similar (Figura 3F):
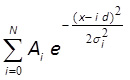
waquí A i y σ i, d y N son parámetros de ajuste. Parámetros A i y σ i corresponden a la amplitud y la anchura de el i-ésimo pico; d es la distancia entre los picos, N es número entero, que se corresponde con el número total de picos en la distribución. Si los centros de los primeros 3 o más picos muestran una visualmente buen partido a la línea ajustada, la distancia entre estos picos (parámetro d) corresponde a una intensidad de fluorescencia de un solo fluoróforo.
Nota 1: El número de puntos examinados (Sección 7.3.6) se debe aumentar si el sistema de microscopía exhibe vibraciones importantes o hay otra fuente de ruido (rayo láser por ejemplo inestable).
Nota 2: Es esencial para obtener> 3 picos para un análisis preciso con un ajuste gaussiano equidistantes. Si se obtienen menos picos, el falso (por ejemplo, doble) tamaño de paso se podría obtenercuando las condiciones de iluminación no son óptimas, por ejemplo, cuando los puntos se blanquean pasos muy rápidos y simples no se resuelven así.
- Determine el tamaño molecular del complejo punta-tracking.
- Utilice las imágenes recogidas con el Protocolo 6 y seleccione los primeros 2-4 marcos, que fueron adquiridos inmediatamente después de abrir el obturador. Si el campo se iluminó durante algún tiempo antes de que se observó la punta de seguimiento, estimar la intensidad original de la cinética de photobleaching en las mismas condiciones experimentales.
- La media de los fotogramas seleccionados y normalizar la imagen resultante que en las secciones 7.2.5-7.2.7.
- Medir la intensidad integral del complejo punta de seguimiento fluorescente utilizando el mismo tamaño de la región como en la Sección 7.3.1.
- Medir la intensidad integral de 3 zonas de fondo cerca del complejo de la punta de seguimiento y el uso de la misma región, el promedio de estos valores y restar de la intensidad del complejo punta-tracking de la Sección 7.4.3.
- Calcular el número de moléculas de fluoróforo en el complejo dividiendo la intensidad de fluorescencia obtenida en la Sección 7.4.4 por la intensidad del fluoróforo sola obtenido en la Sección 7.3.5.
Nota 1: Es deseable que los ajustes de iluminación y de adquisición de protocolo 7.4 son los mismos que en el protocolo 7.1. Si bien el tiempo de exposición o la intensidad del láser se ajustó durante estos pasos, los valores fluorescentes resultantes debe ampliarse en consecuencia. Sin embargo, la exactitud de tales escala debe ser verificada mediante imágenes de la misma muestra (por ejemplo, tiempo fluorescente) bajo estas diferentes condiciones y el cálculo de la relación de las intensidades resultantes.
8. Microtúbulos Tip-tracking por las cuentas de Proteína Coated
- Llevar a cabo experimentos con perlas de punta de seguimiento mediante la activación de desmontaje MT como en el protocolo 6. La intensidad de la fuente de luz DIC debe reducirse para permitir la visualización Rodamina fluorescencia simultáneamente conDe formación de imágenes DIC.
- Preparar las cuentas como en Grishchuk et al. 10 y Asbury et al. 11 Introducir 30-50 l de suspensión de cuentas dentro de la cámara a 10 l / min. La concentración de perlas sugerido es de 10 -16 -10 -17 M.
- Si usa el microscopio en posición vertical, retire la cámara desde la platina del microscopio y de invertirlo durante 5-10 minutos para permitir que los granos de sedimento en el cubreobjetos. Esto promueve una mejor fijación del talón a los microtúbulos atados al cubreobjetos, pero este procedimiento no tiene éxito con 0,5 micras perlas de poliestireno, que muestran poca sedimentación por gravedad durante un tiempo tan corto.
- Seleccionar un cordón que está unido a los microtúbulos cubreobjetos-atado; el talón debe moverse en un arco de clara 44 (Figura 4A). El cordón atado debe estar ubicado 1-3 m de distancia de la superficie cubreobjetos, que es claramente visible debido a las perlas cubreobjetos-adjunta ocasionales que permanecen motionless.
- Cambie a Rodamina cubo filtro y comenzar a recoger imágenes durante el uso de la iluminación DIC.
- Abrir obturador para iluminar el campo de la imagen (restringido con un diafragma de campo) con una lámpara de mercurio o de 530 a 550 nm láser. Continuar el registro hasta que el talón se separa o se mueve con el fin de microtúbulos desmontaje (Figuras 4D, 4F y 4G).
Nota 1: trampa óptica puede utilizarse para promover la interacción entre la pared de los microtúbulos y el talón recubierto de proteína. Esto es especialmente útil cuando se trabaja con perlas recubiertas con motores de quinesina (Figuras 4E-G). Siga mismos protocolos que el anterior pero complementar búfer motilidad con 2 mM Mg-ATP. En la etapa 8.3, la captura de una gota de libre flotación con el rayo láser 1064 nm, mover la etapa de llevar el talón atrapado cerca de la pared de microtúbulos segmentado. Comience de imágenes con poca luz DIC ya través del cubo de filtro de rodamina y esperar a que el cordón para comenzar a caminar hacia el final de los microtúbulos tapado. En popaer se observa el movimiento del talón dirigida, cierre el obturador para atrapar la viga y abrir el obturador para la iluminación fluorescente. Continuar el registro hasta que el talón se separa o pistas con el fin de microtúbulos desmontaje.
Access restricted. Please log in or start a trial to view this content.
Resultados
Proteína seguimiento con depolymerizing microtúbulos termina. Cinetocoro levadura componente Dam1 es, con mucho, el mejor consejo-tracker de los microtúbulos depolymerizing termina 14. Este complejo de 10 subunidad marcada con GFP se puede expresar fácilmente y se purifica a partir de células bacterianas 18,38, por lo que recomendamos su uso como un control positivo para el ensayo de la punta de seguimiento. Una proteína fluorescente que rastrea con el extremo despolimerizaci...
Access restricted. Please log in or start a trial to view this content.
Discusión
Muchos ensayos de una sola molécula hoy en día utilizan habitualmente cubreobjetos especialmente tratadas para reducir drásticamente la adherencia no específica de proteínas. El procedimiento se describe aquí es una modificación del protocolo original desarrollado en Howard laboratorio 32, y nos encontramos con que silanizar los cubreobjetos es bien vale la pena el esfuerzo, incluso con ensayos de cuentas basados en la DIC, que no utilizan la fluorescencia. Cámaras ensambladas con tales cubreobj...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores desean agradecer a FI Ataullakhanov para ayudar a diseñar y fabricar cámaras de flujo reutilizables, N. Dashkevich, N. y A. Gudimchuk Korbalev para proporcionar imágenes de figuras, N. y P. Gudimchuk Zakharov para el desarrollo de un protocolo y proporcionar reactivos para preparar semillas microtúbulos marcadas con digoxigenina, A. Potapenko para obtener ayuda con la edición de texto y otros miembros del laboratorio Grishchuk para consejos y discusiones. Esta obra fue financiada en parte por subvención NIH R01 GM-098389 y una subvención piloto de Pennsylvania Muscle Instituto de ELG, que es un Kimmel Scholar, por RFBR ofrece 12-04-00111-A, 13-04-40190-H y 13 -04-40188-H, Academia Rusa de Ciencias del Presidium de Subvenciones (Mecanismos de Sistemas Molecular Integración y programas de Biología Celular Molecular y Moleculares) a FI Ataullakhanov, NIH subvención GM R01 GM033787 a JR McIntosh, y una beca postdoctoral de la Fundación Dinastía Zimin Dmitry al VAV
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Table 1. Microscopy and other equipment. | |||
| Microscope | Zeiss Nikon | Axio Imager 2 Eclipse Ti | other microscope models capable of DIC and epifluorescence-imaging can be used |
| Objective | Zeiss Nikon | 420490-9900-000 CFI Apo 100x Oil 1.49 | 100X, DIC, 1.3-1.49 NA |
| Objective heater | Bioptechs | 150803, 150819-19 | |
| Fluorescent filter cube | Chroma | 49004 or 49008 41017 or 49020 | optimized for Rhodamine fluorescence optimized for GFP fluorescence |
| Acquisition software | freeware MicroManager Molecular Devices | not applicable MetaMorph 7.5 | http://valelab.ucsf.edu/~MM/MMwiki/ other software can be used to acquire images and for a particle tracking |
| EMCCD camera | Andor | iXon3, DU-897E-cs0-#BV | Highly sensitive EMCCD camera |
| Trapping laser | IPG Photonics | YLR-10-1064-LP | 1,064 nm laser, 10 W |
| Fluorescence excitation lasers | Coherent, Inc. Coherent, Inc. | Sapphire 488 LP Sapphire 552 LP | excitation of green fluorophores excitation of red fluorophores |
| Plasma Cleaner | Harrick Plasma | PDC-001 | |
| Commercial flow chambers | Warner Instruments | RC-20 or RC-30 | |
| Perfusion pump | Cole Palmer Harvard Apparatus | Masterflex 77120-52 Pico Plus | Both pumps provide the required rate of liquid flow but a peristaltic pump may pulse at very slow speed. The flow with a syringe pump is more consistent for a wide range of rates but this pump has inertia. |
| Table 2. Microscopy chamber preparation. | |||
| Modified microscope slides for reusable chambers | Precision Glassblowing of Colorado | Custom order www.precisionglassblowing.com | Sonic slots in slides using schematics in Figure 1 |
| Polyethylene tubing | Intramedic | 427410 | I.D. 0.58 mm, O.D. 0.965 mm; use these tubes to connect assembled chamber to the pump and waste container |
| Polyethylene tubing | Intramedic | 427400 | I.D. 0.28 mm, O.D. 0.61 mm; use these tubes to make the reusable chamber |
| Regular microscope slides | VWR | 48312-003 | Other similar slides can be used |
| Coverslips | VWR | 48393-150, 48366-067 | Other similar coverslips can be used |
| Silicon sealant | World Precision Instruments | KIT, SILICON SEALANT 5 MIN CURE | |
| Epoxy glue | Loctite | 83082 | |
| Cyanoacrylate adhesive | Scotch 3M | AD114 | Or cyanoacrylate adhesive from other manufacturers |
| Table 3. Coverslips cleaning and coating. | |||
| Molecular Sieves, Grade 564 | Macron | 4490-04 | |
| Coverglass Staining Jar | Ted Pella, Inc. | 21036 | |
| Coverslip Ceramic Holder | Thomas Scientific | 8542e40 | |
| PlusOne Repel Silane | GE Healthcare Biosciences | 17-1332-01 | |
| Pluronic F-127 | Sigma-Aldrich | P2443 | |
| Anti-digoxigenin AB | Roche Applied Science | 11093274910 | |
| Table 4. Preparation of seeds and segmented microtubules. | |||
| Tubulin | purified from cow brains Cytoskeleton, Inc | T238P | For purification protocols see 49–51 Unlabeled porcine tubulin |
| Labeled tubulin | Cytoskeleton, Inc Invitrogen Invitrogen | TL590M C1171 (Rhodamine) A-2952 (Digoxigenin) | Rhodamine-labeled porcine tubulin Tubulin can be labeled with any amine-reactive dye as in reference52. |
| GMPCPP | Jena Biosciences | NU-405 | Aliquot and store at -70 °C |
| VALAP | Vaseline, lanolin, and paraffin at 1:1:2 by mass | see reference9 | |
Referencias
- Desai, A., Mitchison, T. J. Microtubule polymerization dynamics. Ann. Rev. Cell Dev. Biol. 13, 83-117 (1997).
- Mitchison, T. M., Kirschner, M. W. Dynamic instability of microtubule growth. Nature. 312 (15), 237-242 (1984).
- Walker, R. A., Brien, O., et al. Dynamic Instability of Individual Microtubules Analyzed by Video Light Microscopy: Rate Constants and Transition Frequencies. J. Cell Biol. 107, 1437-1448 (1988).
- Gardner, M. K., Zanic, M., Gell, C., Bormuth, V., Howard, J. Depolymerizing Kinesins Kip3 and MCAK Shape Cellular Microtubule Architecture by Differential Control of Catastrophe. Cell. 147 (5), 1092-1103 (2011).
- Lombillo, V. A., Stewart, R. J., McIntosh, J. R. Minus-end-directed motion of kinesin-coated microspheres driven by microtubule depolymerization. Nature. 373, 161-164 (1995).
- Franck, A. D., Powers, A. F., Gestaut, D. R., Gonen, T., Davis, T. N., Asbury, C. L. Tension applied through the Dam1 complex promotes microtubule elongation providing a direct mechanism for length control in mitosis. Nat. Cell Biol. 9 (7), 832-837 (2007).
- Tran, P. T., Walker, R. A., Salmon, E. D. A metastable intermediate state of microtubule dynamic instability that differs significantly between plus and minus ends. J. Cell Biol. 138 (1), 105-117 (1997).
- Grishchuk, E. L., Molodtsov, M. I., Ataullakhanov, F. I., McIntosh, J. R. Force production by disassembling microtubules. Nature. 438, 384-388 Forthcoming.
- Coue, M., Lombillo, A., Richard, J. Microtubule Depolymerization Promotes Particle and Chromosome Movement In Vitro. J. Cell Biol. 112 (6), 1165-1175 (1991).
- Grishchuk, E. L., Spiridonov, I. S., et al. Different assemblies of the DAM1 complex follow shortening microtubules by distinct mechanisms. Proc. Natl. Acad. Sci. U.S.A. 105 (19), 6918-6923 (2008).
- Asbury, C. L., Gestaut, D. R., Powers, A. F., Franck, A. D., Davis, T. N. The Dam1 kinetochore complex harnesses microtubule dynamics to produce force and movement. Proc. Natl. Acad. Sci. U.S.A. 103 (26), 9873-9878 (2006).
- Westermann, S., Wang, H. -W., Avila-Sakar, A., Drubin, D. G., Nogales, E., Barnes, G. The Dam1 kinetochore ring complex moves processively on depolymerizing microtubule ends. Nature. 440 (7083), 565-569 (2006).
- Grissom, P. M., Fiedler, T., Grishchuk, E. L., Nicastro, D., West, R. R., Mcintosh, J. R. Kinesin-8 from Fission Yeast A Heterodimeric , Plus-End – directed Motor that Can Couple Microtubule Depolymerization to Cargo Movement. Mol. Biol. Cell. 20, 963-972 (2009).
- McIntosh, J. R., Volkov, V., Ataullakhanov, F. I., Grishchuk, E. L. Tubulin depolymerization may be an ancient biological motor. J. Sci. 123, 3425-3434 (2010).
- Grishchuk, E. L., McIntosh, J. R., Molodtsov, M. I., Ataullakhanov, F. I. Force generation by dynamic microtubule polymers. Compr. Biophys. 4, 93-117 (2012).
- Asbury, C. L., Tien, J. F., Davis, T. N. Kinetochores' gripping feat: conformational wave or biased diffusion. Trends Cell Biol. (1), 38-46 (2011).
- Tien, J. F., Umbreit, N. T., et al. Cooperation of the Dam1 and Ndc80 kinetochore complexes enhances microtubule coupling and is regulated by aurora B. Cell Biol. 189 (4), 713-723 (2010).
- Gestaut, D. R., Graczyk, B., et al. Phosphoregulation and depolymerization-driven movement of the Dam1 complex do not require ring formation. Nat. Biol. 10 (4), 407-414 (2008).
- Schmidt, J. C., Arthanari, H., et al. The Kinetochore-Bound Ska1 Complex Tracks Depolymerizing Microtubules and Binds to Curved Protofilaments. Dev. Cell. 23 (5), 968-980 (2012).
- Brouhard, G. J., Stear, J. H., et al. XMAP215 is a processive microtubule polymerase. Cell. 132 (1), 79-88 (2008).
- Stumpff, J., Du, Y., et al. A Tethering Mechanism Controls the Processivity and Kinetochore-Microtubule Plus-End Enrichment of the Kinesin-8 Kif18A. Mol. Cell. 43 (5), 764-775 (2011).
- Su, X., Qui, W., Gupta, M., Pereira-Leal, J., Reck-Peterson, S. L., Pellman, D. Mechanisms underlying the dual-mode regulation of microtubule dynamics by Kip3/kinesin-8. Mol. Cell. 43 (5), 751-763 (2011).
- Helenius, J., Brouhard, G., Kalaidzidis, Y., Diez, S., Howard, J. The depolymerizing kinesin MCAK uses lattice diffusion to rapidly target microtubule ends. Nature. 441 (7089), 115-119 (2006).
- Gudimchuk, N., Vitre, B., et al. Kinetochore kinesin CENP-E is a processive bi-directional tracker of dynamic microtubule tips. Nat. Cell Biol. 15 (9), 1079-1088 (2013).
- Akhmanova, A., Steinmetz, M. Microtubule +TIPs at a glance. J. Sci. 20 (Pt 20), 3415-3419 (2010).
- Dixit, R., Barnett, B., Lazarus, J., Tokito, M., Goldman, Y., Holzbaur, E. Microtubule plus-end tracking by CLIP-170 requires EB1. Proc. Natl. Acad. Sci. U.S.A. 106 (2), 492-497 (2009).
- Grishchuk, E. L., Efremov, A. K., et al. The Dam1 ring binds microtubules strongly enough to be a processive as well as energy-efficient coupler for chromosome motion. Proc. Natl. Acad. Sci. U.S.A. 105 (40), 15423-15428 (2008).
- Akiyoshi, B., Sarangapani, K. K., et al. Tension directly stabilizes reconstituted kinetochore-microtubule attachments. Nature. 468 (7323), 576-579 (2010).
- McIntosh, J. R., Grishchuk, E. L., et al. Fibrils Connect Microtubule Tips with Kinetochores A Mechanism to Couple Tubulin Dynamics to Chromosome Motion. Cell. 135 (2), 322-333 (2008).
- Powers, A. F., Franck, A. D., et al. The Ndc80 kinetochore complex forms load-bearing attachments to dynamic microtubule tips via biased diffusion. Cell. 136 (5), 865-875 (2009).
- Umbreit, N. T., Gestaut, D. R., et al. The Ndc80 kinetochore complex directly modulates microtubule dynamics. Proc. Natl. Acad. Sci. U.S.A. 109 (40), 16113-16118 (2012).
- Gell, C., Bormuth, V., et al. Microtubule Dynamics Reconstituted In Vitro and Imaged by Single-Molecule Fluorescence Microscopy. Methods Biol. 95, 221-245 (2010).
- Dixit, R., Ross, J. L. Studying Plus-End Tracking at Single Molecule Resolution Using TIRF Microscopy. Methods Cell Biol. 95, 543-554 (2010).
- Beausang, F. J., Sun, Y., Quinlan, E. M., Forkey, N. J., Goldman, Y. Construction of Flow Chambers for Polarized Total Internal Reflection Fluorescence Microscopy (polTIRFM). Cold Spring Harbour Protoc. 6, 712-715 (2012).
- Hyman, A. A., Salser, S., Drechsel, D. N., Unwin, N., Mitchison, T. J. Role of GTP Hydrolysis in Microtubule Dynamics: Information from a Slowly Hydrolyzable Analogue, GMPCPP. Mol. Biol. Cell. 3, 1155-1167 (1992).
- Grishchuk, E. L., Ataullakhanov, F. I. In Vitro Assays to Study the Tracking of Shortening Microtubule Ends and to Measure Associated Forces. Methods Cell Biol. 95, 657-676 (2010).
- Gutiérrez-Medina, B., Block, S. M. Visualizing individual microtubules by bright field microscopy. Am. J. Phys. 78 (11), 1152-1159 (2010).
- Volkov, V. A., Zaytsev, A. V., et al. Long tethers provide high-force coupling of the Dam1 ring to shortening microtubules. Proc. Natl. Acad. Sci. U.S.A. 110 (19), 7708-7713 (2013).
- Laan, L., Pavin, N., et al. Cortical dynein controls microtubule dynamics to generate pulling forces that position microtubule asters. Cell. 148 (3), 502-514 (2012).
- Myster, S. H., Knott, J. A., O'Toole, E., Porter, M. E. The Chlamydomonas Dhc1 gene encodes a dynein heavy chain subunit required for assembly of the I1 inner arm complex. Mol. Biol. Cell. 8, 607-620 (1997).
- Lombillo, V. A., Coue, M., McIntosh, J. R. In vitro motility assays using microtubules tethered to Tetrahymena pellicles. Methods Cell Biol. 39, 149-165 (1993).
- Hyman, A., Chrétien, D., Arnal, I., Wade, R. Structural changes accompanying GTP hydrolysis in microtubules: information from a slowly hydrolyzable analogue guanylyl-(alpha,beta)-methylene-diphosphonate. J. Cell Biol. 128 (1-2), 117-125 (1995).
- Park, M., Kim, H., Kim, D., Song, N. W. Counting the Number of Fluorophores Labeled in Biomolecules by Observing the Fluorescence-Intensity Transient of a Single Molecule. Bull. Chem. Soc. Jap. 78, 1612-1618 (2005).
- Welburn, J. P. I., Grishchuk, E. L., Backer, C. B., Wilson-Kubalek, E. M., Yates, J. R., Cheeseman, I. M. The human kinetochore Ska1 complex facilitates microtubule depolymerization-coupled motility. Dev. Cell. 16 (3), 374-385 (2009).
- Efremov, A., Grishchuk, E. L., Mcintosh, J. R., Ataullakhanov, F. I. In search of an optimal ring to couple microtubule depolymerization to processive chromosome motions. Natl. Acad. Sci. U.S.A. (48), 19017-19022 (2007).
- Itoh, T., Hisanaga, S., Hosoi, T., Kishimoto, T., Hotani, H. Phosphorylation states of microtubule-associated protein 2 (MAP2) determine the regulatory role of MAP2 in microtubule dynamics. Biochemistry. 36 (41), 12574-12582 (1997).
- Oguchi, Y., Uchimura, S., Ohki, T., Mikhailenko, S. V., Ishiwata, S. The bidirectional depolymerizer MCAK generates force by disassembling both microtubule ends. Nat. Biol. (6), 1-8 (2011).
- Kishino, A., Yanagida, T. Force measurements by micromanipulation of a single actin filament by glass needles. Nature. 334, 74-76 (1988).
- Borisy, G. G., Marcum, J. M., Olmsted, J. B., Murphy, D. B., Johnson, K. A. Purification of tubulin and associated high molecular weight proteins from porcine brain and characterization of microtubule assembly in vitro. Ann. NY Acad. Sci. 253, 107-132 (1975).
- Weingarten, M. D., Lockwood, A. H., Hwo, S., Kirschner, M. W. A Protein Factor Essential for Microtubule Assembly. Proc. Natl. Acad. Sci. U.S.A. 72 (5), 1858-1862 (1975).
- Widlund, P. O., Podolski, M., et al. One-step purification of assembly-competent tubulin from diverse eukaryotic sources. Mol. Biol. Cell. 23 (22), 4393-4401 (2012).
- Hyman, A., Drechsel, D., et al. Preparation of Modified Tubulins. Methods Enzymol. 196, 478-485 (1991).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados