Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Préparation des microtubules sectoriels pour étudier les motions Poussé par le démontage microtubules Ends
Dans cet article
Résumé
Les microtubules sont des polymères intrinsèquement instables, et leur commutation entre la croissance et de raccourcissement est stochastique et difficile à contrôler. Nous décrivons ici les protocoles à l'aide de microtubules segmentés avec des bouchons de stabilisation photoablatable. Dépolymérisation des microtubules segmentées peut être déclenchée avec une résolution temporelle et spatiale, aidant ainsi l'analyse de mouvements avec les extrémités des microtubules démontage.
Résumé
Dépolymérisation des microtubules peut fournir une force pour le transport de différents complexes de protéines et de perles revêtus de protéine in vitro. Les mécanismes sous-jacents sont supposés jouer un rôle essentiel dans les mouvements des chromosomes microtubules dépend cours de la division cellulaire, mais les protéines concernées et leurs rôles exacts sont mal définis. Ainsi, il ya un besoin croissant de développer des tests qui permettent d'examiner cette motilité in vitro en utilisant des composants purifiés et milieu biochimique défini. Les microtubules, cependant, sont par nature instables des polymères; leur commutation entre la croissance et le raccourcissement est stochastique et difficile à contrôler. Les protocoles que nous décrivons ici profiter des microtubules segmentées qui sont faites avec le photoablatable stabilisation bouchons. La dépolymérisation des microtubules tels segmentées peut être déclenchée avec une résolution spatiale et temporelle élevée, aidant de ce fait les études de motilité au niveau des extrémités de désassemblage des microtubules. Cette technique peut être utilisée pour carry à une analyse quantitative du nombre de molécules dans les complexes de protéines marquées par fluorescence, qui se déplacent avec processively dynamique des microtubules se termine. Afin d'optimiser un rapport signal sur bruit dans cet et d'autres dosages quantitatifs fluorescentes, des lamelles doivent être traités pour réduire l'absorption non spécifique de protéines solubles marqués par fluorescence. Des protocoles détaillés sont prévus pour prendre en compte les irrégularités de l'illumination fluorescente, et déterminer l'intensité d'un fluorophore unique en utilisant l'ajustement gaussien équidistants. Enfin, nous décrivons l'utilisation des microtubules segmentés pour étudier les mouvements des microtubules dépendant des microbilles de revêtus de protéine, apportant des éclairages sur la capacité des différentes protéines motrices et non motrices à deux dépolymérisation des microtubules au mouvement de la cargaison processive.
Introduction
Les microtubules sont des structures hautement conservées du cytosquelette qui sont importants pour l'architecture cellulaire, la motilité cellulaire, la division cellulaire, le transport intracellulaire et 1. Ces polymères dynamiques assembler à partir de tubuline en présence de GTP, et ils commutent spontanément entre la croissance et le raccourcissement 2. Les microtubules sont très mince (seulement 25 nm de diamètre) donc des techniques optiques spéciales pour renforcer le contraste doit être utilisé pour observer les microtubules avec un microscope optique. Les travaux antérieurs de ces polymères a examiné leur comportement dynamique utilisant contraste interférentiel différentiel (DIC) 3. Et d'autres semblables études in vitro ont montré que dans des conditions expérimentales typiques, les microtubules subissent catastrophe et passer à la dépolymérisation que rarement, une fois tous les 5-15 min (cette fréquence est de 7-15 mM concentration de tubuline soluble examiné à 28-32 ° C ) 4. Différentes techniques ont ainsi été proposés à inductione dépolymérisation des microtubules d'une manière contrôlée. Raccourcissement des microtubules peut être déclenché par élimination par lavage de la tubuline soluble 5,6, coupant microtubules avec un faisceau laser à 7, ou en utilisant des microtubules segmentées 8, comme décrit ici. Des travaux antérieurs utilisant microtubules segmentés, ainsi que les polymères de commutation stochastique, a trouvé que les petites cargaisons intracellulaires, comme les chromosomes, vésicules, et perles de revêtus de protéine, peuvent se déplacer à l'extrémité des microtubules de raccourcissement 9-13. Ce phénomène est supposé avoir une incidence directe sur les mouvements des chromosomes dans les cellules en mitose, et les mécanismes sous-jacents sont actuellement sous enquête 14-16.
Récemment, des techniques fluorescentes base, y compris la fluorescence totale interne de réflexion (FRBR) microscopie, ont été utilisées pour étudier la motilité de microtubules dynamique extrémités 17-24. L'avantage de cette approche est qu'elle permet l'examen de l'interactions entre les microtubules et les protéines des microtubules contraignant en temps réel à l'aide de protéines marquées avec des fluorophores différents. Plusieurs complexes de protéines ont été trouvés pour déplacer processively avec allongement et / ou raccourcissement des extrémités des microtubules. Ils comprennent les protéines associées aux microtubules Dam1 10,12,18, SKA1 19, et XMAP215 20, ainsi que les moteurs de kinésine Kif18A 21,22, MCAK 23 et CENP-E 24. Ces protéines présentent pointe de suivi processive, qui est fondamentalement différente de celle des protéines de pointe de suivi classiques comme EB1 25. Bien que les molécules EB1 et les partenaires associés semblent rester stable associée à des microtubules dynamique se termine, les molécules individuelles restent liés à la pointe des microtubules pour seulement ~ 0,8 sec, échanger rapidement avec la piscine soluble 26. En revanche, la pointe des trackers processives, comme Dam1, voyagent avec des microtubules se termine pour beaucoup de microns, et leur association avec des conseils de microtubules peuvent durer mdes secondes. La pointe de temps de l'association, ainsi que le taux de suivi résultant, dépend fortement du nombre de molécules qui forment la pointe de suivi complexe 27. Plus grands ensembles de protéines sont généralement beaucoup mieux la pointe des trackers. Par exemple, ces ensembles complexes que les kinétochores de levure isolées peuvent rester couplé à des microtubules extrémités pour heures 28. Certaines protéines de microtubules liaison, par exemple Ndc80 complexe protéique kinétochore, ont été trouvés dans l'impossibilité de suivre avec microtubules se termine à un niveau de la molécule unique, encore Ndc80 est très efficace dans le couplage de la motion de perle cargaison 19,29-31. Ainsi, afin de comprendre le mécanisme de la pointe de poursuite par différents complexes protéiques, ainsi que leurs rôles biologiques, il est important d'examiner la pointe de suivi en fonction du nombre de molécules dans le complexe de la pointe de poursuite, ainsi que pour déterminer la capacité de ces complexes à exposer la motilité collective sur la surface de chargement des billes.
Ci-dessous nous fournissons des protocoles détaillés de préparer et de mener des expériences avec les microtubules segmentés (figure 1A). Tout d'abord, les lames de verre sont disponibles dans le commerce modifiés pour attacher court tube en polyéthylène (protocole 1). La chambre d'écoulement de microscopie réutilisable est ensuite assemblée à partir d'un tel coulisseau et le plasma nettoyé et traité au silane lamelle (protocole 2) 32 à 34 chimiquement ou. Le volume de la chambre résultant est seulement 20-25 pi (ou aussi petite que 15 pi, voir la note 3 dans le Protocole 1), y compris le volume de la tubulure d'admission. Disponibles dans le commerce chambres d'écoulement peuvent également être utilisés, mais leur volume est généralement plus grande, ce qui conduit à la perte inutile de protéines. Si une chambre plus grande est utilisé, le volume de toutes les solutions dans les protocoles ci-dessous doit être réduite proportionnellement. graines de microtubules sont ensuite préparées, par exemple en utilisant lentement hydrolysable du GTP analogique, GMPCPP (guanosine-5'-[(α, β)-methyleno] triphosphate) (protocole 3, voir aussi Hyman et al. 35). Les graines sont immobilisés sur une lamelle couvre-objet et la surface nettoyée est ensuite bloquée pour empêcher l'absorption non spécifique d'autres protéines 32 (protocole 4 décrit l'immobilisation de graines à l'aide de la digoxigénine). Les microtubules segmentés peuvent être préparés en utilisant le protocole 5. La raison principale de cette approche est que les polymères de microtubules dynamiques, qui se forment dans la présence de GTP, peuvent être temporairement stabilisée par l'ajout des «caps» de courtes segments de tubuline stables, qui contiennent GMPCPP. Ces capsules contiennent également de la tubuline marqué à la rhodamine, de sorte qu'ils peuvent être éliminés simplement en illuminant le champ de vision avec un laser de 530 à 550 nm ou une lampe à arc au mercure (Protocole 6) 36. L'intensité de fluorescence du signal de suivi de pointe peut alors être utilisée pour estimer le nombre de molécules qui se déplacent avec les extrémités de désassemblage des microtubules, en tenant compte de l'irrégularité de l'éclairement de champ de microscope (Protocole 7). Une approche similaire peut être utiliséepour étudier les interactions entre les microtubules de dépolymérisation et de perles de protéine revêtu, préparé comme décrit dans 27 (protocole n ° 8). Certaines protéines se lient facilement aux parois des microtubules segmentées, mais pinces laser peuvent également être utilisés pour la tenue du bourrelet à proximité de la paroi des microtubules, ce qui favorise sa fixation.
Access restricted. Please log in or start a trial to view this content.
Protocole
Équipement requis: Les expériences décrites ci-dessous requièrent un microscope optique équipé pour DIC et l'imagerie de fluorescence (tableau 1). Champ lumineux éclairage LED peut être utilisé pour améliorer de façon significative la détection des graines des microtubules lamelle-joint 37, qui sont difficiles à observer avec une lampe halogène régulière. Pour contrôler l'écoulement du liquide dans les chambres de microscopie, les solutions doivent être échangés avec une pompe péristaltique capable de vitesses d'écoulement de 10 à 100 l / min. Une pompe à seringue peut également être utilisé, mais il faut prendre soin d'éviter les bulles d'air qui peuvent se former lorsque la vitesse d'écoulement est modifié brusquement. Pour la manipulation des billes de protéine revêtues, par exemple pour leur proximité de la paroi des microtubules segmenté amener, une nm faisceau laser 1064 à onde continue peut être introduit dans l'axe optique du microscope et focalisée par un objectif à ouverture numérique élevée (1.3 ou plus) pour produire un piège. Pour l'analyse quantitative de la fluorescenceintensité de molécules individuelles de la lumière d'excitation doit être fourni par une source laser à base puisque l'intensité de cette source de lumière est plus stable que celle produite par une lampe à mercure. Afin de minimiser les vibrations mécaniques, le microscope doit être placé sur une table optique. Matériel plus sophistiqué est nécessaire pour étudier le mouvement des billes avec le microtubules dépolymérisation se termine sous une force constante, et pour mesurer les signaux de force unique-shot 11,38,39, ces méthodes seront décrites ailleurs.
Une. Fabrication réutilisables flux Chambers
Les lames de verre pour chambres d'écoulement réutilisables peuvent être commandés à partir d'une usine de fabrication de verre local à l'aide des schémas de la figure 1B (voir le tableau 2 pour plus de détails au sujet de notre fournisseur). Fraisage ultrasons modifier des diapositives réguliers de microscope (75 mm x 25 mm, 1,0 mm d'épaisseur) pour faire deux rainures 15 ± 1 mm de long, 1,0 ± 0,1 mm de large et 0,8 ± 0,05 mm de profondeur.Distance entre les extrémités les plus proches doit être de 14 ± 1 mm, ce qui est la distance optimale pour une chambre assemblé avec 22 mm x 22 mm lamelle. Voir le tableau 2 pour une liste d'autres matériaux.
- Placer un long tube de polyéthylène de 100 mm (diamètre extérieur 0,61 mm, le tableau 2) dans chaque rainure de la coulisse, laissant ~ 5 mm saillies aux extrémités intérieures des rainures. Fixer les tubes à l'intérieur des rainures avec de la colle cyanoacrylate, noyant les tubes complètement à l'intérieur des rainures.
- Remplir les rainures avec de la colle époxy, tout en évitant de répandre la colle à l'intérieur des tubes. Laissez la colle sécher pendant environ un jour.
- Avec une lame de rasoir couper la masse solidifiée de la colle 3-4 mm de l'extrémité distale de chaque site de fixation, en enlevant les parties proximale par rapport à l'axe de la diapositive. Les tubes doivent rester à l'intérieur de leurs rainures. Retrait des parties proximales permettra également de réduire et éliminer les surplombs intérieurs, créant une surface plane avec deux ouvertures de tube.
- Remplir une seringue avec de l'eau et essaisi les tubes fonctionnent correctement. Si le liquide s'écoule librement, mettre une goutte de colle époxy (~ 5 mm de diamètre) aux extrémités extérieures des bosquets secs pour 1 jour (figure 1D). Cela rendra chambres plus durable, afin qu'ils puissent être utilisés de façon répétée pendant de nombreux mois.
Note 1: Pour une chambre destinée à un microscope inversé, les lames doivent être modifiées pour faire en outre deux petits trous au niveau des extrémités opposées des rainures (figure 1C). Insérer les tubes dans les trous de la lame, plier les tubes et de les adapter étroitement à l'intérieur des rainures (figure 1E). Suivre les étapes 1.2 à 1.4, mais enlever la colle époxy à partir de la surface, qui sera utilisée pour fabriquer une chambre d'écoulement.
Note 2: Pour réduire le volume de la chambre, utilisez le fraisage de faire deux empreintes 0,050 ± 0,005 mm de profondeur, laissant la partie centrale de la lame 5,0 ± 0,5 mm de large et légèrement surélevée (voir «zones gravées" sur les figures 1B et 1C). Quand til s'écouler chambre est assemblé (tel que décrit ci-dessous), placer le ruban adhésif double face à l'intérieur de ces empreintes.
Note 3: Pour réutiliser ces diapositives modifiés, après avoir terminé les expériences supprimer la lamelle et ruban adhésif double face en utilisant une lame de rasoir. Retirez le mastic par pelant et en essuyant la lame avec 70% d'éthanol. Placer la lame dans un récipient avec 1-2% d'un détergent à vaisselle laboratoire, fixez un tube à une pompe péristaltique et perfuser 50-70 ml, suivi avec un volume égal d'eau déminéralisée, sec et ranger dans un compartiment exempt de poussière.
2. Préparation des lamelles couvre-
Ce protocole prend 6-8 heures et vous aidera à préparer 12 lamelles. Vous aurez besoin d'un support de lamelle de céramique et 3 lamelle pots de coloration avec des couvercles, le volume de la cuve doit être de 15 ml, de sorte que chaque tiendra 4 lamelles empilées. Un pot en verre avec un couvercle (250 ml) doit être utilisé pour incuber avec des lamelles couvre-silane. Utilisez lamelles régulières de verre n ° 1 (22 mm x 22 mm ou 22 mm x 30 mm, voir les tableaux 2 et 3 pour une liste des matériaux). Toutes les mesures doivent être effectuées dans une hotte de laboratoire, tout en portant des gants.
- Mettez les lamelles dans les pots de coloration de lamelle de verre et remplir les pots avec de l'acétone. Incuber pendant 1 heure, laver avec de l'eau déminéralisée 10x.
- Incuber les lamelles 10 min avec de l'éthanol et laver de nouveau 10x avec de l'eau déminéralisée.
- Préparer une solution "piranha". Placer 60 ml de solution de peroxyde d'hydrogène (30% dans l'eau) dans un récipient en verre résistant à la chaleur et ajouter lentement 100 ml d'acide sulfurique (rapport final de l'acide à la solution de peroxyde d'hydrogène est de 05:03). Solution se réchauffer, ce qui est normal, mais soyez prudent. solution de Piranha est extrêmement corrosif! Utilisez laboratoire épais manteau, des gants et des lunettes de protection!
- Remplissez les pots lamelle de coloration avec une solution «piranha», fermez le couvercle et placer les pots dans un bain d'eau préchauffée à 90 ° C pendant 1 heure.
- Verser le "piranha" solutiet abandonnez comme indiqué par les règles de sécurité à votre lieu de travail. Lavage des lamelles couvre-10x avec de l'eau déminéralisée.
- Remplissez les pots de coloration de lamelle de 0,1 M KOH, incuber 10 min, et laver avec de l'eau déminéralisée 10x. Cela neutraliser les résidus acides laissés sur les lamelles après le traitement "piranha".
- Lamelles sèches, un à la fois en maintenant chaque lamelle avec des pincettes plat tranchant revêtus de téflon (pour minimiser les dommages sur une surface de verre) et tout en insufflant de l'azote comprimé sec. Assurez-vous que les lamelles sont séchées complètement, parce solution de silane est très réactif avec de l'eau.
- Empilez les lamelles séchées dans des supports en céramique (12 lamelles couvre par titulaire), qui doit être soigneusement pré-séché avec de l'azote. Gardez les supports en céramique couverts pour éviter la poussière de coller à la surface de la lamelle.
- Couvrir le fond du pot de 250 ml en verre (6 cm de diamètre) avec des tamis moléculaires, de nuance 564, pour l'absorption de l'eau.
- Remplissez le pot avec 200 ml d'Solution PlusOne Repel Silane et plonger lentement un support en céramique avec des lamelles dans un bocal, fermez le couvercle et incuber pendant 5 min à température ambiante. Ceci créera revêtement hydrophobe sur la surface de la lamelle.
- Retirer lentement le support avec des lamelles dans le bocal et le transfert des lamelles couvre-un à la fois dans les pots lamelle de coloration remplis avec du méthanol.
- Placer une colonne de verre ou de métal dans le réservoir d'eau d'un bain sonique, de sorte que la lamelle couvre-pot de coloration est immergé pendant 2/3 de sa hauteur. Soniquer à 70 W pendant 20 min, en changeant solution de méthanol toutes les 5 minutes, puis rincer avec de l'eau déminéralisée 10x. Si la silanisation a fonctionné correctement, les lamelles apparaîtront sec lorsqu'il est retiré de l'eau.
- Bien enlever toute l'eau résiduelle avec de l'azote, comme ci-dessus.
- Interposer les lamelles couvre-objet avec Kimwipes pour éviter le contact de surface à surface entre les lamelles. Les lamelles couvre-peuvent être stockés dans un récipient fermé pendant plusieurs semaines à température ambiante.
Note 1: Les étapes 2.1 à 2.6 peut être remplacé par le nettoyage des lamelles avec le Nettoyant plasma pendant 15 min à 30 W, ce qui réduit considérablement le temps de préparation total. La pression à l'intérieur de la chambre de nettoyage est fixée à 100 à 200 mTorr. À la fois de l'oxygène atmosphérique et le comprimé peut être utilisé. Empilez les lamelles de plasma nettoyé dans des supports en céramique et passez à l'étape 2.7.
3. Préparation de GMPCPP stabilisées Graines de microtubules
Cette procédure prend environ 1 heure et les graines résultantes des microtubules sont stables pendant 1 à 2 jours à la température ambiante. Voir le tableau 4 pour une liste des réactifs.
- Mélanger sur la glace:
- 10 pi non marqués tubuline (100 M, tableau 4) dans BRB-80 (tampon Pipes 80 mM, EGTA 1 mM, 4 mM de MgCl2, pH 6,9 avec KOH; supplément mM DTT 1-2 en utilisant aliquote frais pour chaque expérience).
- 2,6 ul marquée à la digoxigénine tubuline (tableau 4). Ajustez le volume en fonction de la préparation,telles que le rapport final de la tubuline marqué à non marqué est ~ 01:10. Mélangez bien à la pipette.
- 1,4 pi mM GMPCPP 10 (concentration finale 1 mM)
- Incuber 15 min à 35 ° C, les graines pousseront 2-3 m de long. Réglez l'heure si différente longueur des microtubules est souhaitée.
- Ajouter 35 ul BRB-80 (préchauffé à 35 ° C), mélanger par pipetage et centrifuger pendant 15 min à 25 000 xg pour sédimenter les graines à la température ambiante.
- Rejeter le surnageant, laver le culot en ajoutant douceur et la suppression de 50 pi de chaud BRB-80.
- Remettre le culot et dans 25 ul BRB-80.
4. Fixation des microtubules Graines à les lamelles
Protocoles 4 et 5, il faudra 2-3 heures, de sorte que deux chambres d'écoulement sont utilisés par jour.
- Assembler chambre d'écoulement selon les instructions du fabricant en utilisant des lamelles silanisées et passez à l'étape 4.2. Si vous utilisez des lamelles sur mesure (Protocole 1), suivez les étapes below.
- Joindre deux morceaux de ruban adhésif double face (5 mm x 30 mm) le long de la ~ 5 mm zone centrale large, mettre lamelle silanisée au sommet de la bande, appuyez fermement.
- Remplir la chambre avec BRB-80 à travers l'un des tubes et de brancher les deux tubes avec des cure-dents rondes.
- Déposer une petite goutte d'bicolore Kwik Cast étanchéité sur une boîte de Petri en plastique petit et mélanger rapidement mais soigneusement à l'aide d'un cure-dent. Le mastic devient vert; appliquer immédiatement, d'étanchéité soigneusement tous les bords de la lamelle. Si le scellant pénètre trop profondément sous la lamelle, ouvrez l'un des tubes en enlevant le bouchon de cure-dents et appliquer une légère pression pour empêcher les fuites d'étanchéité à l'intérieur des tubes.
- Laissez la chambre sèche pendant 10 minutes et vérifiez que le flux ne se limite pas avant d'aller plus loin.
- Placer la chambre sur une platine de microscope préchauffée à 32 ° C et fixer l'un des tubes d'une pompe, qui pompe le liquide à l'extérieur. La longueur du tube d'entréedoit être minimisée pour éviter la perte inutile de réactifs: la durée recommandée est de 5-7 cm. Plongez cette fin dans un flacon de 0,5 ml avec BRB-80 tampon. Ceci et toutes les solutions ci-dessous doit être préchauffé à 32-35 ° C.
- Appliquer une légère pression avec une pompe ou il suffit de soulever l'extrémité du tube de sortie pour faire sortir les bulles d'air qui peuvent se former, de temps en temps lorsque le bouchon de tube d'entrée est supprimé.
- Régler le débit de la pompe à 100 pi / min. Laver avec 2 volumes de chambre des anticorps anti-digoxigénine dilué 1:30 dans BRB-80, incuber 15 min pour permettre l'adsorption des anticorps.
- Laver avec 5-10 volumes de chambre de chaud BRB-80, incuber 10 min à 1% de Pluronic F-127 dans BRB-80 pour bloquer la surface hydrophobe de lamelle silanisée.
- Laver avec de 5 à 10 volumes de chambre de tampon de motilité (BRB-80 supplémenté avec 0,4 mg / ml de caséine).
- Réduire la vitesse de la pompe à 10 pi / min et perfuser graines des microtubules dilué 1:200-1:600 30-40 tampon de la motilité ul. Incuber 15 kmn pour favoriser la liaison des anticorps aux graines de lamelle-adsorbé.
- Laver la chambre à 100 pi / min avec 400 pi de tampon de la motilité de retirer tout matériel non lié.
Note 1: La densité résultant de graines devrait être 10-30 champ de microscope par (figure 2A). Pour dépanner, utiliser la tubuline marqué par fluorescence lors de la polymérisation (étape 3.1) pour la détection plus facile des graines de lamelle-joint.
Note 2: préparées à partir de Chlamydomonas axonèmes 40 ou d'autres sources biologiques, ainsi que les pellicules de cellules lysées et Tetrahymena deciliated 41 peuvent également être utilisés comme agents de nucléation des microtubules. Ce sont de nucléation utile pour créer des petits réseaux de microtubules, et on préfère quand microtubules certain nombre de protofilaments sont souhaitées (GMPCPP nucléation de semences un des microtubules qui contient ≥ 14 protofilaments 42). Ces structures peuvent être associées aux lamelles nettoyées par absorption non spécifique, mais l'attachment est généralement moins stable par rapport à l'attachement à base d'anticorps, en particulier lors de l'utilisation des lamelles silanisées.
5. Préparation des microtubules sectorielle
Tous les volumes de la solution ci-dessous sont pour le volume de la chambre de 15-20 pi; augmenter proportionnellement si grande chambre est utilisée.
- Préchauffer non marqué tubuline mix (tampon de la motilité de 45 pi complété avec 1 mM de Mg-GTP et 10-15 uM tubuline non marqué) pendant 30 secondes à 35 ° C. Perfuser à 30 pi / min.
- Surveiller la croissance des microtubules avec DIC optique (figure 2B, vidéo 1). Pendant 5-7 minutes d'incubation les microtubules se développent habituellement ~ 10 um de long.
- Préparer mélange Rhodamine-tubuline (65 pi de tampon de la motilité supplémenté avec 0,5 mM GMPCPP et 2-5 uM de tubuline Rhodamine marqué avec 0,5 à 1 proportion molaire de la rhodamine à la tubuline) et chauffer la solution à 35 ° C pendant 30 sec.
- Perfuser immédiatement à 30 μl / min. Incuber pendant 8-10 min pour favoriser la formation de bouchons fluorescents stables à l'extrémité des microtubules. Segments de microtubules stables seront également nucléation spontanée et seront visibles avec optique DIC.
- Laver la chambre et avec 100 pi de tampon de motilité à 20 ul / min pour éliminer les tubulines et des nucléotides, ainsi que les fragments solubles de microtubules.
- Avec DIC, confirment que les microtubules sont visibles (figure 2D), leur nombre devrait toutefois diminuer en raison de nombreux microtubules démonter lors du capsulage (Vidéo 2 montre un champ typique avec microtubules segmentés).
Note 1: segmenté microtubules sont très stables et peuvent être utilisés pendant au moins 2 h. Toutefois, la durée de vie de ces microtubules diminue avec l'échange de solution excessive, ou si le 2-mercaptoéthanol est utilisé dans le tampon d'image.
6. Observation expérimentale de la suivi des protéines avec dépolymérisation microtubules Ends
- Introduce 30 à 50 pi de la protéine fluorescente (0,1 à 20 nM) dans la chambre à 10 pl / min. Si la protéine coller à la lamelle est évident, compléter le tampon de la motilité de 4-8 mg / ml de BSA. Alexa488-Dam1 pointe de suivi requiert en outre 10 mM de DTT ou 0,5-1% ßME 10.
- Limiter le champ d'éclairement au moyen d'un diaphragme de champ de microscope pour éviter le blanchiment inutile et démontage des microtubules.
- Commencez acquisition vidéo en utilisant un filtre de GFP cube, puis passer à la rhodamine filtre cube sans interrompre l'enregistrement de l'image. Les segments rouges aux extrémités des microtubules doivent être clairement visibles; ils vont commencer à s'estomper et se désintègrent rapidement (vidéo 3).
- Continuer à éclairer jusqu'à ce que les bouchons disparaissent presque (généralement 10-20 secondes, mais ce temps sera plus long pour les bouchons cultivées avec un ratio plus faible de l'étiquetage rhodamine), et revenir au canal de la GFP pour enregistrer protéine suivi avec le désassemblage des microtubules.
- Analyser l'séquences résultantes en construisant kymographs, c'est à dire des images bidimensionnelles qui montrent l'intensité de fluorescence le long de l'axe des microtubules pendant divers temps au cours de l'observation) en utilisant MetaMorph, librement disponibles ImageJ ou autre logiciel de traitement d'image (figure 2E).
Remarque 1: taux d'acquisition doit être ajustée en fonction de la chronologie des événements observés. La dose recommandée est de 2-3 images par seconde (fps) pour le lent, anneau de taille Dam1 complexes de 27, mais le temps d'acquisition de molécules simples devrait être> 20 fps 19.
Note 2: Un EMCCD très sensible, par exemple ANDOR iXon3, est nécessaire pour l'enregistrement rapide des événements de pointe de suivi avec les microtubules dépolymérisation. Les paramètres recommandés pour caméra Andor iXon3 sont: 5x de gain, EM gagner 200, 1 MHz vitesse de lecture, le mode de capteur 16 bits, 80 ms de temps d'exposition.
Note 3: En utilisant la microscopie TIRF permettra d'améliorer le rapport signal-sur-bruit, cependant, les microtubules courts doivent être utilisés, tels that les bouchons de stabilisation fluorescentes restent à la portée de champ évanescent.
7. Analyse quantitative de la taille moléculaire des microtubules Complexes Conseil de suivi
La justification de cette approche est de déterminer le nombre de molécules dans un complexe de pointe de suivi par trouver le rapport de l'intensité de fluorescence totale du complexe à l'intensité d'un seul fluorophore pointe de suivi. Cette approche peut être appliquée à des fusions GFP-protéine et les protéines marquées avec des colorants fluorescents, mais il peut sous-estimer le nombre de molécules dans les complexes pointe de suivi si certaines molécules de protéines dans les préparations ne sont pas fluorescent.
- Records cinétique de photoblanchiment pour les molécules de protéines marquées par fluorescence.
- Assembler chambre de microscopie régulière en utilisant une lame de verre non modifié, deux bandes de ruban adhésif double face, et une lamelle propre, qui peut être préparé à l'aide de tout le Protocole 2, ou seulement les étapes 2.1-2.6 de cetteprotocole.
- Ajouter environ 50 nM de protéine dans un tampon de la motilité, laver rapidement avec du tampon de la motilité et de sceller la chambre avec valap (tableau 4). Optimiser la concentration de protéines pour obtenir le champ avec des taches réparties de façon égale (figure 3A), représentant des molécules simples et leurs petits agrégats (trimères et tétramères, qui peuvent se former spontanément dans la solution ou peuvent apparaître lorsque plusieurs molécules simples sont rapprochés et ne peuvent être résolus) . Cette étape est très important pour l'obtention d'une distribution multi-pic des étapes de photoblanchiment et une détermination précise de la taille de pas (voir ci-dessous).
- Réduire l'intensité du laser d'éclairage à laquelle les taches fluorescentes individuelles sont encore visibles; avec un éclairage plus le temps de photoblanchiment est prolongé, donc des traces plus photoblanchiment peuvent être obtenus. Réduire également le temps d'exposition afin de réduire la probabilité de plus d'un fluorophore de blanchiment pendant une trame. Le réglage recommandépour Andor iXon3 caméra: Gain 5.0x, EM gagner 999, 10 MHz vitesse de lecture, 50-100 ms de temps d'exposition.
- Focus sur la surface de la lamelle, fermer l'obturateur d'éclairage, passer à un champ de frais, ouvrir le volet d'éclairage et d'acquérir des images jusqu'à ce que tous les complexes ont blanchi (ci-après dénommé img (x, y)).
- Corrigez les images acquises pour irrégularité de l'éclairage (figure 3B).
- Préparer la solution de tout fluorophore, par exemple, 1 uM isothiocyanate de fluorescéine (FITC) dans BRB-80. Une telle solution peut être préparée à l'avance, aliquoté et conservé à -20 ° C.
- Monter une chambre que dans la section 7.1.1, mais utiliser une lamelle régulière. Ajouter la solution de fluorophore et sceller la chambre à l'aide valap.
- Recueillir> 50 images du champ de microscope ensemble: déplacer la scène pour une nouvelle zone écru tandis que l'obturateur d'éclairage est fermé, et d'acquérir les images immédiatement après l'ouverture de l'obturateur.
- Créer projection moyenne de cette pile et le filtre avec flou gaussien avec 5 rayon de pixel en utilisant ImageJ ou un autre logiciel (figure 3C). L'image résultante représente la répartition de l'intensité d'éclairage du champ (Illum (x, y), où x et y correspondent à des coordonnées de pixels).
- Déterminer la luminosité maximale en pixels de cette image (Max (Illum)).
- Avec l'obturateur fermé et d'illumination utilisant les mêmes réglages de l'appareil comme dans la Section 7.2.3 acquérir une image dans déterminer l'intensité moyenne des pixels de cette image; cette valeur correspond à CN, le bruit de la caméra.
- Utiliser les valeurs ci-dessus et de l'image (Illum (x, y)) pour normaliser l'image expérimental (img (x, y)) en utilisant l'expression suivante:
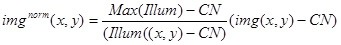
Utilisez l'image résultante img norme (x, y) pour l'analyse quantitative de la brightness des complexes fluorescents fixes, et aussi pour normaliser les images avec des complexes de pointe-vireur (figure 3D).
- Déterminer l'intensité d'un seul fluorophore.
- En utilisant des images normalisées img norme (x, y) et un logiciel de traitement d'image sélectionner une tache fluorescente avec une région circulaire (5-6 pixels de diamètre) et de déterminer son intensité intégrale pour tous les cadres, la génération des traces de photoblanchiment. Évitez mêmes points lumineux (> 5 fois plus lumineux que les gradateurs ceux).
- En utilisant le même outil de région circulaire, sélectionner au moins trois zones sans taches, générer les traces de photoblanchiment correspondant, en moyenne et en forme avec la fonction de décroissance exponentielle.
- Compiler cette courbe d'intensité de fond pour assortir les points de temps expérimentales et soustraire des courbes de photoblanchiment.
- Lisser les courbes de photoblanchiment (moyenne de la fenêtre coulissante de 3-5 points). Inspecter visuellement les courbes obtenues etrejeter toute courbe qui montre une brusque augmentation de la fluorescence ou de l'absence de blanchiment évidente (figure 3E).
- Pour chacune des courbes restantes (généralement 50-70% du nombre total de courbes), sélectionnez visuellement le palier final, lorsque la tache fluorescente a blanchi. Raccourcir ce segment pour ne laisser que ~ 100 points, et la moyenne de ces intensités. Soustraire cette valeur à partir de la courbe de photoblanchiment raccourcie pour réduire au minimum les petites variations est le bruit de fond et de réduire la taille de la crête d'arrière-plan (en bas).
- Tracer un histogramme des intensités de tous les points de temps de 20 ou plusieurs courbes de photoblanchiment (> 1000 points de temps). L'histogramme doit présenter au moins quatre pics distincts (voir la note 2).
- Monter l'histogramme avec équidistant distribution gaussienne 10,43 utilisant MATLAB, Mathematica ou logiciels similaires (figure 3F):
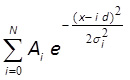
wici A i et σ i, d et n sont des paramètres d'ajustement. Paramètres a i et σ i correspondent à l'amplitude et la largeur des i-ème sommet, d est la distance entre les sommets; N est nombre entier, qui correspond au nombre total de pics dans la distribution. Si les centres des trois ou plusieurs premiers pics montrent un bon match visuellement à la ligne ajustée, la distance entre ces pics (paramètre d) correspond à une intensité de fluorescence d'un fluorophore unique.
Remarque 1: Nombre de points examinés (section 7.3.6) devrait être augmentée si le système de microscopie présente d'importantes vibrations ou il ya une autre source de bruit (de faisceau laser par exemple instable).
Note 2: Il est essentiel d'obtenir> 3 pics pour une analyse précise avec un ajustement gaussien équidistants. Si le nombre de pics sont obtenus, le faux (par exemple, double) taille d'étape pourrait être obtenuelorsque les conditions d'éclairage ne sont pas optimales, par exemple lorsque les points blanchissent pas trop rapides et simples ne sont pas bien résolu.
- Déterminer la taille moléculaire du complexe de pointe de suivi.
- Utiliser des images recueillies avec le protocole 6 et sélectionner les 2-4 premières images, qui ont été acquises immédiatement après l'ouverture de l'obturateur. Si le champ a été allumé pendant un certain temps avant que la pointe de suivi a été observée, estimer l'intensité d'origine de la cinétique de photoblanchiment dans les mêmes conditions expérimentales.
- La moyenne des images sélectionnées et normaliser l'image résultante comme dans les sections 7.2.5-7.2.7.
- Mesurer l'intensité intégrale de l'ensemble d'embout de suivi fluorescent en utilisant la même taille de la région que dans la section 7.3.1.
- Mesurer l'intensité intégrale de 3 zones de fond situées à proximité du complexe de pointe de suivi et à l'aide de la même région, la moyenne de ces valeurs et soustraire de l'intensité du complexe de la section 7.4.3 pointe de suivi.
- Calculer le nombre de molécules de fluorophore dans le complexe en divisant l'intensité de fluorescence obtenue dans la section 7.4.4 de l'intensité du fluorophore unique obtenue dans la Section 7.3.5.
Note 1: Il est souhaitable que les illumination et d'acquisition des paramètres pour le protocole 7.4 sont les mêmes que dans le protocole 7.1. Si soit le temps d'exposition ou de l'intensité du laser a été ajustée au cours de ces étapes, les valeurs de fluorescence résultantes doivent être réduites en conséquence. Cependant, l'exactitude de cette mise à l'échelle doit être vérifiée par l'imagerie du même échantillon (par exemple de temps fluorescent) dans ces différentes conditions et en calculant le rapport des intensités résultant.
8. Microtubules Tip-suivi par les perles de protéines couché
- Réaliser des expériences avec des perles pointe de suivi par le déclenchement MT démontage comme dans le protocole 6. Intensité de la source lumineuse DIC devrait être réduite pour permettre la visualisation de la rhodamine fluorescence simultanément avecImagerie DIC.
- Préparer les perles comme dans Grishchuk et al. Asbury et 10 et al. Introduire 11 pl de 30 à 50 de la suspension de billes dans la chambre à 10 pl / min. La concentration de billes suggéré est de 10 -16 -10 -17 M.
- Si vous utilisez microscope droit, retirer la chambre de la platine du microscope et inverser pendant 5-10 min pour permettre à billes sédimentent à la lamelle. Cela favorise une meilleure liaison de la perle aux microtubules attachés à la lamelle, mais cette procédure n'est pas de succès avec 0,5 um billes de polystyrène, qui montrent peu de sédimentation par gravité pendant un temps si court.
- Sélectionner un bourrelet qui est attaché à la microtubules lamelle-attaché; la perle doit se déplacer dans un arc clair 44 (figure 4A). Le bourrelet attaché doit être situé à une distance de 1 à 3 um de la surface de la lamelle couvre-objet, ce qui est clairement visible grâce à des billes de lamelle-jointe occasionnels qui restent motionless.
- Passez à la rhodamine filtre cube et commencer à recueillir des images en utilisant un éclairage DIC.
- Ouvrir le volet pour éclairer le champ de formation d'image (limité par un diaphragme de champ) avec une lampe à mercure de 530 à 550 nm ou à laser. L'enregistrement jusqu'à la perle se détache ou se déplace avec la fin démontage des microtubules (figures 4D, 4F, 4G et).
Note 1: Le piège optique peut être utilisé pour favoriser l'interaction entre la paroi et des microtubules bourrelet revêtu de protéine. Ceci est particulièrement utile lorsque vous travaillez avec des billes recouvertes avec des moteurs de kinésine (figures 4E-G). Suivez mêmes protocoles que ci-dessus mais compléter tampon de motilité avec 2 mM de Mg-ATP. A l'étape 8.3, capturer une bille flottante libre avec le faisceau laser de 1064 nm, déplacer la platine pour amener la bille piégée plus proche de la paroi des microtubules segmenté. Commencez imagerie à faible lumière DIC et à travers le filtre rhodamine cube et attendre la perle de commencer à marcher vers la fin des microtubules plafonné. À l'arrièreer le mouvement de perle réalisé est observée, fermer l'obturateur pour piéger faisceau et ouvrir l'obturateur pour l'éclairage fluorescent. L'enregistrement jusqu'à la perle détache ou pistes avec la fin des microtubules démontage.
Access restricted. Please log in or start a trial to view this content.
Résultats
Suivi de la dépolymérisation des microtubules protéines se termine. Levure kinetochore composant Dam1 est de loin le meilleur conseil-tracker de la dépolymérisation des microtubules se termine 14. Ce complexe de 10 sous-unité marquée par la GFP peut être facilement exprimée et purifiée à partir de cellules bactériennes 18,38, nous vous recommandons donc l'utiliser comme un contrôle positif pour le dosage de la pointe de suivi. Une protéine fluorescente qui permet ...
Access restricted. Please log in or start a trial to view this content.
Discussion
De nombreux essais de molécules simples de nos jours utilisent couramment des lamelles spécialement traitées pour réduire considérablement non spécifique de protéines de collage. La procédure que nous décrivons ici est une modification du protocole original développé en laboratoire Howard 32, et nous constatons que silaniser les lamelles vaut bien l'effort même avec les dosages de perles à base de DIC, qui n'utilisent pas de fluorescence. Chambers assemblés avec ces lamelles présentent ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Les auteurs tiennent à remercier FI Ataullakhanov pour aider à concevoir et fabriquer des chambres d'écoulement réutilisables, N. Dachkevitch, N. et A. Gudimchuk Korbalev pour fournir des images de figures, N. et P. Gudimchuk Zakharov pour l'élaboration d'un protocole et de fournir des réactifs à préparer les graines des microtubules marquées à la digoxigénine, A. Potapenko de l'aide pour l'édition de texte et d'autres membres de Grishchuk laboratoire pour obtenir des conseils et des discussions. Ce travail a été financé en partie par des subventions du NIH GM R01-098 389 et une subvention pilote de Pennsylvanie Muscle Institut ELG, qui est un Kimmel Scholar, par RFBR accorde 12-04-00111-a, le 13-04-40190-H et 13 -04-40188-H, Académie russe des Sciences subventions Présidium (mécanismes de l'intégration des systèmes moléculaires et les programmes de biologie cellulaire et moléculaire et moléculaires) à FI Ataullakhanov, NIH GM R01 GM033787 à JR McIntosh, et une bourse de recherche postdoctorale de la Fondation Dmitry dynastie Zimin à VAV
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Table 1. Microscopy and other equipment. | |||
| Microscope | Zeiss Nikon | Axio Imager 2 Eclipse Ti | other microscope models capable of DIC and epifluorescence-imaging can be used |
| Objective | Zeiss Nikon | 420490-9900-000 CFI Apo 100x Oil 1.49 | 100X, DIC, 1.3-1.49 NA |
| Objective heater | Bioptechs | 150803, 150819-19 | |
| Fluorescent filter cube | Chroma | 49004 or 49008 41017 or 49020 | optimized for Rhodamine fluorescence optimized for GFP fluorescence |
| Acquisition software | freeware MicroManager Molecular Devices | not applicable MetaMorph 7.5 | http://valelab.ucsf.edu/~MM/MMwiki/ other software can be used to acquire images and for a particle tracking |
| EMCCD camera | Andor | iXon3, DU-897E-cs0-#BV | Highly sensitive EMCCD camera |
| Trapping laser | IPG Photonics | YLR-10-1064-LP | 1,064 nm laser, 10 W |
| Fluorescence excitation lasers | Coherent, Inc. Coherent, Inc. | Sapphire 488 LP Sapphire 552 LP | excitation of green fluorophores excitation of red fluorophores |
| Plasma Cleaner | Harrick Plasma | PDC-001 | |
| Commercial flow chambers | Warner Instruments | RC-20 or RC-30 | |
| Perfusion pump | Cole Palmer Harvard Apparatus | Masterflex 77120-52 Pico Plus | Both pumps provide the required rate of liquid flow but a peristaltic pump may pulse at very slow speed. The flow with a syringe pump is more consistent for a wide range of rates but this pump has inertia. |
| Table 2. Microscopy chamber preparation. | |||
| Modified microscope slides for reusable chambers | Precision Glassblowing of Colorado | Custom order www.precisionglassblowing.com | Sonic slots in slides using schematics in Figure 1 |
| Polyethylene tubing | Intramedic | 427410 | I.D. 0.58 mm, O.D. 0.965 mm; use these tubes to connect assembled chamber to the pump and waste container |
| Polyethylene tubing | Intramedic | 427400 | I.D. 0.28 mm, O.D. 0.61 mm; use these tubes to make the reusable chamber |
| Regular microscope slides | VWR | 48312-003 | Other similar slides can be used |
| Coverslips | VWR | 48393-150, 48366-067 | Other similar coverslips can be used |
| Silicon sealant | World Precision Instruments | KIT, SILICON SEALANT 5 MIN CURE | |
| Epoxy glue | Loctite | 83082 | |
| Cyanoacrylate adhesive | Scotch 3M | AD114 | Or cyanoacrylate adhesive from other manufacturers |
| Table 3. Coverslips cleaning and coating. | |||
| Molecular Sieves, Grade 564 | Macron | 4490-04 | |
| Coverglass Staining Jar | Ted Pella, Inc. | 21036 | |
| Coverslip Ceramic Holder | Thomas Scientific | 8542e40 | |
| PlusOne Repel Silane | GE Healthcare Biosciences | 17-1332-01 | |
| Pluronic F-127 | Sigma-Aldrich | P2443 | |
| Anti-digoxigenin AB | Roche Applied Science | 11093274910 | |
| Table 4. Preparation of seeds and segmented microtubules. | |||
| Tubulin | purified from cow brains Cytoskeleton, Inc | T238P | For purification protocols see 49–51 Unlabeled porcine tubulin |
| Labeled tubulin | Cytoskeleton, Inc Invitrogen Invitrogen | TL590M C1171 (Rhodamine) A-2952 (Digoxigenin) | Rhodamine-labeled porcine tubulin Tubulin can be labeled with any amine-reactive dye as in reference52. |
| GMPCPP | Jena Biosciences | NU-405 | Aliquot and store at -70 °C |
| VALAP | Vaseline, lanolin, and paraffin at 1:1:2 by mass | see reference9 | |
Références
- Desai, A., Mitchison, T. J. Microtubule polymerization dynamics. Ann. Rev. Cell Dev. Biol. 13, 83-117 (1997).
- Mitchison, T. M., Kirschner, M. W. Dynamic instability of microtubule growth. Nature. 312 (15), 237-242 (1984).
- Walker, R. A., Brien, O., et al. Dynamic Instability of Individual Microtubules Analyzed by Video Light Microscopy: Rate Constants and Transition Frequencies. J. Cell Biol. 107, 1437-1448 (1988).
- Gardner, M. K., Zanic, M., Gell, C., Bormuth, V., Howard, J. Depolymerizing Kinesins Kip3 and MCAK Shape Cellular Microtubule Architecture by Differential Control of Catastrophe. Cell. 147 (5), 1092-1103 (2011).
- Lombillo, V. A., Stewart, R. J., McIntosh, J. R. Minus-end-directed motion of kinesin-coated microspheres driven by microtubule depolymerization. Nature. 373, 161-164 (1995).
- Franck, A. D., Powers, A. F., Gestaut, D. R., Gonen, T., Davis, T. N., Asbury, C. L. Tension applied through the Dam1 complex promotes microtubule elongation providing a direct mechanism for length control in mitosis. Nat. Cell Biol. 9 (7), 832-837 (2007).
- Tran, P. T., Walker, R. A., Salmon, E. D. A metastable intermediate state of microtubule dynamic instability that differs significantly between plus and minus ends. J. Cell Biol. 138 (1), 105-117 (1997).
- Grishchuk, E. L., Molodtsov, M. I., Ataullakhanov, F. I., McIntosh, J. R. Force production by disassembling microtubules. Nature. 438, 384-388 Forthcoming.
- Coue, M., Lombillo, A., Richard, J. Microtubule Depolymerization Promotes Particle and Chromosome Movement In Vitro. J. Cell Biol. 112 (6), 1165-1175 (1991).
- Grishchuk, E. L., Spiridonov, I. S., et al. Different assemblies of the DAM1 complex follow shortening microtubules by distinct mechanisms. Proc. Natl. Acad. Sci. U.S.A. 105 (19), 6918-6923 (2008).
- Asbury, C. L., Gestaut, D. R., Powers, A. F., Franck, A. D., Davis, T. N. The Dam1 kinetochore complex harnesses microtubule dynamics to produce force and movement. Proc. Natl. Acad. Sci. U.S.A. 103 (26), 9873-9878 (2006).
- Westermann, S., Wang, H. -W., Avila-Sakar, A., Drubin, D. G., Nogales, E., Barnes, G. The Dam1 kinetochore ring complex moves processively on depolymerizing microtubule ends. Nature. 440 (7083), 565-569 (2006).
- Grissom, P. M., Fiedler, T., Grishchuk, E. L., Nicastro, D., West, R. R., Mcintosh, J. R. Kinesin-8 from Fission Yeast A Heterodimeric , Plus-End – directed Motor that Can Couple Microtubule Depolymerization to Cargo Movement. Mol. Biol. Cell. 20, 963-972 (2009).
- McIntosh, J. R., Volkov, V., Ataullakhanov, F. I., Grishchuk, E. L. Tubulin depolymerization may be an ancient biological motor. J. Sci. 123, 3425-3434 (2010).
- Grishchuk, E. L., McIntosh, J. R., Molodtsov, M. I., Ataullakhanov, F. I. Force generation by dynamic microtubule polymers. Compr. Biophys. 4, 93-117 (2012).
- Asbury, C. L., Tien, J. F., Davis, T. N. Kinetochores' gripping feat: conformational wave or biased diffusion. Trends Cell Biol. (1), 38-46 (2011).
- Tien, J. F., Umbreit, N. T., et al. Cooperation of the Dam1 and Ndc80 kinetochore complexes enhances microtubule coupling and is regulated by aurora B. Cell Biol. 189 (4), 713-723 (2010).
- Gestaut, D. R., Graczyk, B., et al. Phosphoregulation and depolymerization-driven movement of the Dam1 complex do not require ring formation. Nat. Biol. 10 (4), 407-414 (2008).
- Schmidt, J. C., Arthanari, H., et al. The Kinetochore-Bound Ska1 Complex Tracks Depolymerizing Microtubules and Binds to Curved Protofilaments. Dev. Cell. 23 (5), 968-980 (2012).
- Brouhard, G. J., Stear, J. H., et al. XMAP215 is a processive microtubule polymerase. Cell. 132 (1), 79-88 (2008).
- Stumpff, J., Du, Y., et al. A Tethering Mechanism Controls the Processivity and Kinetochore-Microtubule Plus-End Enrichment of the Kinesin-8 Kif18A. Mol. Cell. 43 (5), 764-775 (2011).
- Su, X., Qui, W., Gupta, M., Pereira-Leal, J., Reck-Peterson, S. L., Pellman, D. Mechanisms underlying the dual-mode regulation of microtubule dynamics by Kip3/kinesin-8. Mol. Cell. 43 (5), 751-763 (2011).
- Helenius, J., Brouhard, G., Kalaidzidis, Y., Diez, S., Howard, J. The depolymerizing kinesin MCAK uses lattice diffusion to rapidly target microtubule ends. Nature. 441 (7089), 115-119 (2006).
- Gudimchuk, N., Vitre, B., et al. Kinetochore kinesin CENP-E is a processive bi-directional tracker of dynamic microtubule tips. Nat. Cell Biol. 15 (9), 1079-1088 (2013).
- Akhmanova, A., Steinmetz, M. Microtubule +TIPs at a glance. J. Sci. 20 (Pt 20), 3415-3419 (2010).
- Dixit, R., Barnett, B., Lazarus, J., Tokito, M., Goldman, Y., Holzbaur, E. Microtubule plus-end tracking by CLIP-170 requires EB1. Proc. Natl. Acad. Sci. U.S.A. 106 (2), 492-497 (2009).
- Grishchuk, E. L., Efremov, A. K., et al. The Dam1 ring binds microtubules strongly enough to be a processive as well as energy-efficient coupler for chromosome motion. Proc. Natl. Acad. Sci. U.S.A. 105 (40), 15423-15428 (2008).
- Akiyoshi, B., Sarangapani, K. K., et al. Tension directly stabilizes reconstituted kinetochore-microtubule attachments. Nature. 468 (7323), 576-579 (2010).
- McIntosh, J. R., Grishchuk, E. L., et al. Fibrils Connect Microtubule Tips with Kinetochores A Mechanism to Couple Tubulin Dynamics to Chromosome Motion. Cell. 135 (2), 322-333 (2008).
- Powers, A. F., Franck, A. D., et al. The Ndc80 kinetochore complex forms load-bearing attachments to dynamic microtubule tips via biased diffusion. Cell. 136 (5), 865-875 (2009).
- Umbreit, N. T., Gestaut, D. R., et al. The Ndc80 kinetochore complex directly modulates microtubule dynamics. Proc. Natl. Acad. Sci. U.S.A. 109 (40), 16113-16118 (2012).
- Gell, C., Bormuth, V., et al. Microtubule Dynamics Reconstituted In Vitro and Imaged by Single-Molecule Fluorescence Microscopy. Methods Biol. 95, 221-245 (2010).
- Dixit, R., Ross, J. L. Studying Plus-End Tracking at Single Molecule Resolution Using TIRF Microscopy. Methods Cell Biol. 95, 543-554 (2010).
- Beausang, F. J., Sun, Y., Quinlan, E. M., Forkey, N. J., Goldman, Y. Construction of Flow Chambers for Polarized Total Internal Reflection Fluorescence Microscopy (polTIRFM). Cold Spring Harbour Protoc. 6, 712-715 (2012).
- Hyman, A. A., Salser, S., Drechsel, D. N., Unwin, N., Mitchison, T. J. Role of GTP Hydrolysis in Microtubule Dynamics: Information from a Slowly Hydrolyzable Analogue, GMPCPP. Mol. Biol. Cell. 3, 1155-1167 (1992).
- Grishchuk, E. L., Ataullakhanov, F. I. In Vitro Assays to Study the Tracking of Shortening Microtubule Ends and to Measure Associated Forces. Methods Cell Biol. 95, 657-676 (2010).
- Gutiérrez-Medina, B., Block, S. M. Visualizing individual microtubules by bright field microscopy. Am. J. Phys. 78 (11), 1152-1159 (2010).
- Volkov, V. A., Zaytsev, A. V., et al. Long tethers provide high-force coupling of the Dam1 ring to shortening microtubules. Proc. Natl. Acad. Sci. U.S.A. 110 (19), 7708-7713 (2013).
- Laan, L., Pavin, N., et al. Cortical dynein controls microtubule dynamics to generate pulling forces that position microtubule asters. Cell. 148 (3), 502-514 (2012).
- Myster, S. H., Knott, J. A., O'Toole, E., Porter, M. E. The Chlamydomonas Dhc1 gene encodes a dynein heavy chain subunit required for assembly of the I1 inner arm complex. Mol. Biol. Cell. 8, 607-620 (1997).
- Lombillo, V. A., Coue, M., McIntosh, J. R. In vitro motility assays using microtubules tethered to Tetrahymena pellicles. Methods Cell Biol. 39, 149-165 (1993).
- Hyman, A., Chrétien, D., Arnal, I., Wade, R. Structural changes accompanying GTP hydrolysis in microtubules: information from a slowly hydrolyzable analogue guanylyl-(alpha,beta)-methylene-diphosphonate. J. Cell Biol. 128 (1-2), 117-125 (1995).
- Park, M., Kim, H., Kim, D., Song, N. W. Counting the Number of Fluorophores Labeled in Biomolecules by Observing the Fluorescence-Intensity Transient of a Single Molecule. Bull. Chem. Soc. Jap. 78, 1612-1618 (2005).
- Welburn, J. P. I., Grishchuk, E. L., Backer, C. B., Wilson-Kubalek, E. M., Yates, J. R., Cheeseman, I. M. The human kinetochore Ska1 complex facilitates microtubule depolymerization-coupled motility. Dev. Cell. 16 (3), 374-385 (2009).
- Efremov, A., Grishchuk, E. L., Mcintosh, J. R., Ataullakhanov, F. I. In search of an optimal ring to couple microtubule depolymerization to processive chromosome motions. Natl. Acad. Sci. U.S.A. (48), 19017-19022 (2007).
- Itoh, T., Hisanaga, S., Hosoi, T., Kishimoto, T., Hotani, H. Phosphorylation states of microtubule-associated protein 2 (MAP2) determine the regulatory role of MAP2 in microtubule dynamics. Biochemistry. 36 (41), 12574-12582 (1997).
- Oguchi, Y., Uchimura, S., Ohki, T., Mikhailenko, S. V., Ishiwata, S. The bidirectional depolymerizer MCAK generates force by disassembling both microtubule ends. Nat. Biol. (6), 1-8 (2011).
- Kishino, A., Yanagida, T. Force measurements by micromanipulation of a single actin filament by glass needles. Nature. 334, 74-76 (1988).
- Borisy, G. G., Marcum, J. M., Olmsted, J. B., Murphy, D. B., Johnson, K. A. Purification of tubulin and associated high molecular weight proteins from porcine brain and characterization of microtubule assembly in vitro. Ann. NY Acad. Sci. 253, 107-132 (1975).
- Weingarten, M. D., Lockwood, A. H., Hwo, S., Kirschner, M. W. A Protein Factor Essential for Microtubule Assembly. Proc. Natl. Acad. Sci. U.S.A. 72 (5), 1858-1862 (1975).
- Widlund, P. O., Podolski, M., et al. One-step purification of assembly-competent tubulin from diverse eukaryotic sources. Mol. Biol. Cell. 23 (22), 4393-4401 (2012).
- Hyman, A., Drechsel, D., et al. Preparation of Modified Tubulins. Methods Enzymol. 196, 478-485 (1991).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon