Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La investigación de las proteínas priónicas, como la extensión y la toxicidad de Uso del Metazoan Organismo modelo
En este artículo
Resumen
Prion-like propagation of protein aggregates has recently emerged as being implicated in many neurodegenerative diseases. The goal of this protocol is to describe, how to use the nematode C. elegans as a model system to monitor protein spreading and to investigate prion-like phenomena.
Resumen
Prions are unconventional self-propagating proteinaceous particles, devoid of any coding nucleic acid. These proteinaceous seeds serve as templates for the conversion and replication of their benign cellular isoform. Accumulating evidence suggests that many protein aggregates can act as self-propagating templates and corrupt the folding of cognate proteins. Although aggregates can be functional under certain circumstances, this process often leads to the disruption of the cellular protein homeostasis (proteostasis), eventually leading to devastating diseases such as Alzheimer’s disease (AD), Parkinson’s disease (PD), Amyotrophic lateral sclerosis (ALS), or transmissible spongiform encephalopathies (TSEs). The exact mechanisms of prion propagation and cell-to-cell spreading of protein aggregates are still subjects of intense investigation. To further this knowledge, recently a new metazoan model in Caenorhabditis elegans, for expression of the prion domain of the cytosolic yeast prion protein Sup35 has been established. This prion model offers several advantages, as it allows direct monitoring of the fluorescently tagged prion domain in living animals and ease of genetic approaches. Described here are methods to study prion-like behavior of protein aggregates and to identify modifiers of prion-induced toxicity using C. elegans.
Introducción
Muchas de las enfermedades neurodegenerativas, como la enfermedad de Alzheimer (EA), enfermedad de Parkinson (EP), la esclerosis lateral amiotrófica (ELA), y las encefalopatías espongiformes transmisibles (EET), se asocian con proteínas de agregación propensos y están, por tanto, colectivamente conocidos como proteínas misfolding trastornos (PMD ). EET o enfermedades priónicas constituyen una clase única de PMD en que pueden ser infecciosas en seres humanos y animales 1. A nivel molecular, los priones se replican mediante el reclutamiento y la conversión-rica α-hélice PrP celular codificada por el hospedador monomérico (PrP C) en el rico en β-hoja de PrP Sc conformación patológica 2,3. Agregados de proteínas autopropagante también se han identificado en los hongos, que comparten características importantes con priones mamíferos 4,5. Además, priones mamíferos son capaces de pasar de célula a célula e infectan células de los animales 6,7.
Mientras PMD other de EET no son infecciosas, comparten un principio patógena común con las enfermedades priónicas 8,9. Aunque las proteínas vinculadas a cada uno de los PMD no están relacionadas en estructura o función, que todos los agregados formulario a través de un proceso de cristalización como llaman nucleado o polimerización sembraron; Además semillas proteicas crecen mediante la contratación de sus isoformas solubles 2,10,11. La eficiencia a la auto-propagan in vivo varía, dependiendo de las propiedades intrínsecas de la proteína, que junto con factores celulares adicionales, tales como chaperones moleculares en última instancia determinan las tasas de nucleación agregada, la siembra, la fragmentación y la difusión de 12-15. Por lo tanto, debe existir un equilibrio entre estos factores que permiten la propagación eficiente de la agregación de proteínas. Esto también podría explicar por qué sólo algunos agregados amiloidogénicos albergan las características de un prión, y por lo tanto no todos los PMD son infecciosos. Los priones parecen representar o 'top artistas intérpretes o ejecutantesfa amplio espectro de agregados proteicos auto-replicantes, que los convierte en una poderosa herramienta para estudiar PMD 8,13.
Curiosamente, la toxicidad asociada con los agregados relacionados con la enfermedad tiene a menudo un componente autónomo no celular 16,17. Esto significa que afectan a las células vecinas que no expresan el gen correspondiente, en contraste con un efecto de célula autónoma estrictamente, lo que implica que sólo las células que expresan el gen de la exposición fenotipo específico. Esto fue convincentemente demostrado por la expresión específica de tejido o derribar las respectivas proteínas en numerosos modelos de enfermedades neurodegenerativas 18-26. Diversos mecanismos se han sugerido como una base para esta toxicidad autónoma no celular en PMD, incluyendo el suministro de nutrientes disminuido, el desequilibrio en la señalización neuronal, excitotoxicidad del glutamato, y la neuroinflamación 16,27,28. Además, un movimiento de priones como de agregados de enfermedades vinculadas entre las células Might contribuyen a este aspecto 29,30. La evidencia creciente sugiere que las inclusiones proteína distinta de priones pueden transmitir de célula a célula, lo que puede explicar la característica de propagación de la patología observado en muchos PMD 30-36. Sin embargo, aún no se ha determinado si existe una clara relación causal entre el movimiento intercelular de proteínas de la enfermedad y el efecto tóxico sobre las células vecinas. Por lo tanto, una mejor comprensión de las vías celulares que subyacen a la transmisión de célula a célula y la toxicidad celular autónomo no es necesario y esencial para el desarrollo de nuevas terapias. Sin embargo, muchos aspectos de priones como la difusión y celulares factores que influyen en la transmisión de célula a célula de proteínas mal plegadas en metazoos no se conocen bien, en particular a nivel del organismo.
El nematodo Caenorhabditis elegans tiene varias ventajas que ofrecen el potencial de descubrir nuevas facetas de priones como spreading en metazoos 17. Es transparente, lo que permite el seguimiento in vivo de las proteínas fluorescentemente marcadas en el organismo vivo. Además, muchos procesos celulares y fisiológicas afectadas por la enfermedad se conservan de gusanos para la salud humana, y C. elegans también es susceptible a una amplia variedad de manipulaciones genéticas y análisis moleculares y bioquímicos 37-39. Exactamente 959 células somáticas conforman el adulto hermafrodita con un plan de cuerpo simple que todavía tiene varios tipos de tejidos distintos, incluyendo los músculos, neuronas y delgado.
Para establecer un nuevo modelo de prión en C. elegans, se optó por expresar de forma exógena la glutamina / asparagina bien caracterizado (Q / N) ricos en priones dominio NM de la proteína priónica de levadura citosólica Sup35, ya que no hay proteínas priónicas endógenos conocidos gusanos 4,40. Los priones de levadura han sido invaluables en la aclaración de los mecanismos básicos de la replicación de priones 41-44. Además, Nuevo México es el abetocitosólica proteína prión como st que se ha demostrado para recapitular el ciclo de vida completo de un prión en cultivos celulares de mamíferos 45,46. Del mismo modo, cuando se expresa en C. elegans, el dominio prión Sup35 adoptado notablemente bien a los diferentes requisitos para la propagación en células de metazoos en comparación con células de levadura y de las características clave de la biología expuestas prión agregación 40. NM se asoció con una profunda fenotipo tóxicos, incluyendo la interrupción de la integridad mitocondrial y la apariencia de diversas vesículas autofagia relacionados en el nivel celular, así como la detención embrionario y larval, retraso del desarrollo, y una alteración generalizada del medio ambiente plegamiento de la proteína en el nivel del organismo. Sorprendentemente, el dominio prion celular exhibe toxicidad celular autónoma y no autónoma, que afecta a los tejidos vecinos en la que no se expresó el transgén. Además, el transporte vesicular del dominio prión dentro de y entre las células se monitoriza en tiempo real in vivo 40.
Aquí se describe cómo examinar difusión-prión como en C. elegans. Vamos a explicar cómo supervisar el transporte intra e intercelular de vesículas que contienen el dominio de priones usando microscopía de fluorescencia de lapso de tiempo. Haremos hincapié en el uso de sensores de plegado específicos de tejido y de expresión ubicua reporteros de estrés para evaluar los teléfonos celulares efectos autónomas autónomas y no sobre la aptitud celular. Por último, vamos a describir el procedimiento de una pantalla recientemente realizado genoma amplia ARN de interferencia (RNAi) para identificar nuevos modificadores de la toxicidad inducida por priones. En combinación, estos métodos pueden ayudar a desmenuzar las vías genéticas implicadas en el movimiento intercelular de proteínas y su toxicidad autónoma no celular.
Protocolo
1. Seguimiento transcelular Difusión de proteínas priónicas-como por En Vivo Time-lapse de imágenes
NOTA: Crecer C. elegans de tipo salvaje (WT) (N2) y las líneas transgénicas de acuerdo con métodos estándar y controlar cuidadosamente la temperatura de cultivo 47.
- Generar líneas transgénicas de C. elegans expresar la proteína prión como, etiquetados con la proteína fluorescente de color rojo monomérica (mRFP). Mira este video que muestra cómo utilizar la microinyección 48. Para más información y métodos que describen cómo integrar estas líneas extracromosómicos, ver 49.
- Preparar poblaciones sincronizadas por la puesta de huevos o de blanqueo de acuerdo con métodos estándar 50.
- Sincronización de la puesta de huevos
- Transferencia 10-20 adultos grávidas en un plato y dejar que ellos ponen huevos por 1 - 2 horas. Retire los adultos de la placa y dejar que la progenie crecer hasta la edad deseada.
- Sincronización por blanqueo
- Recoger una población no sincronizada de los adultos grávidas y blanquear con solución de hipoclorito alcalino (NaOH 250 mM y 1: 4 (v / v) dilución de lejía comercial en H 2 O). Lavar los huevos dos veces (218 g durante 1 min) con tampón M9 47 (21 MMNA 2 HPO 4 · 7H 2 O, 22 mMKH 2 PO 4, 86 mMNaCl, 1 mMMgSO 4 · 7H 2 O, agregue dH2O hasta 1 L).
- Deje que se traman en tampón M9 con suave agitación O / N a 20 ° C. Desarrollo gusano arresto en la fase L1 en ausencia de una fuente de alimento, dejando una población sincronizada. Traslado L1s en crecimiento de Medios (NGM) placas fresco nematodos sembradas con OP50 E. bacterias coli y permiten la progenie se desarrolla hasta la edad de 47 años que desee.
- Sincronización de la puesta de huevos
- Prepare 2% agarosa almohadillas (en H 2 O) en un portaobjetos de microscopio como se describe 50.
- Prepare dos millasdiapositivas croscope con la cinta de etiquetado se coloca sobre toda su longitud para ser utilizados como espaciadores. Coloque una tercera portaobjetos de microscopio entre ellos.
- Disolver 2% de agarosa en H 2 O y una pipeta una gota sobre el portaobjetos limpio.
- Coloque cuarto de diapositivas perpendicular a los otros tres se desliza sobre la parte superior de la caída de agar. Presione suavemente hacia abajo para aplanar la almohadilla en el mismo grosor que la cinta de etiquetar.
- Deje que se seque durante 1 minuto antes de retirar los espaciadores y tirando suavemente de las diapositivas aparte. La almohadilla de agar se apegará a uno de ellos.
- Pipeta ~ 10 l anestésico (levamisol 2 mM en tampón M9) en la superficie y la transferencia de ~ 10 animales utilizando un pico alambre de platino. Cubra con una hoja de cubierta (~ 22 x 22 mm) y tomar imágenes dentro de 1 hora.
- Alternativamente, para adquirir películas durante un período de tiempo más largo o para reducir aún más la posibilidad de cualquier movimiento de los animales, utilizar una combinación de anestésico y el talón de inmovilización 51.
- Preparar 10% agaalmohadillas rosas (en tampón M9) como se describe 51 y agregar gusanos a solución normas de tamaño nanoesfera 3 l (perlas de poliestireno, 100 nm), además de 3 l anestésicos (levamisol 4 mM en tampón M9). Cubra ligeramente con un cubreobjetos. Para evitar la desecación, sellar la hoja de la cubierta con valap (mezcla de cantidades iguales de vaselina, lanolina y cera de parafina).
- Image inmovilizado gusanos usando un microscopio confocal.
Nota: Los resultados se obtienen utilizando un Spinning Disc AF Microscopio Confocal equipado con una cámara EM-CCD y un software de Microscopía Automatización y Análisis de imagen tales como MetaMorph y describen los detalles a continuación, pero otros sistemas de formación de imágenes confocal comparable también se pueden utilizar.- Utilice el objetivo aceite 63X o 100X / 1.4NA y colocar el portaobjetos de microscopio que contiene gusanos en el soporte de portaobjetos de microscopio.
- Abrir el software. Ajustar la potencia del láser y filtros para imágenes mRFP. Utilice el láser de 561 nm en el 10% de energía y la emisión de filtro> 600 nm.
- Abrir "MultiDimensional Adquisición". En la pestaña "Principal", active las casillas de "Timelapse" y "Run Journals" (Hardware enfoque automático: desactivado).
- Bajo la pestaña "Saving" seleccionar o crear la carpeta del directorio donde los archivos deben ser guardados. Asigne un nombre al archivo.
- Bajo la pestaña "Longitud de onda", seleccione la iluminación adecuada y ajustar la exposición y ganancia. Use "YokoQuad Red" (o una configuración de iluminación equivalente para formación de imágenes mRFP) con una exposición de entre 100 y 300 mseg y una ganancia de la cámara entre 100 y 300, dependiendo de la muestra individual (evaluado mediante el uso de la imagen en vivo).
- Bajo la pestaña "Timelapse" seleccione "Número de puntos de tiempo" = 301 "Duración" = 5 min y "Tiempo / Intervalo" = 1 seg.
- En el marco del "Revistas" pestaña seleccione Diario: "AFC SET Z HOLD" y "AFC Volver a Z HOLD", Tipo: &# 8220; Especial "(dos veces), Punto inicial:" Inicio de la hora punta "y" Fin de punto en el tiempo ". Esta opción de enfoque automático es importante para la imagen de la misma sección durante un período de tiempo más largo.
- Una vez finalizada la configuración, pulse "Adquirir". El vídeo timelapse se safed como archivos TIFF independientes.
- Abre todos los archivos .tiff de una serie de tiempo dada en ImageJ. Diríjase a "Imagen" → "Stacks" → "Imágenes Stacks". Opcionalmente, ajustar el brillo y el contraste, añadir la barra de tamaño, etc. Exportar la película en "Archivo" → "Guardar como" → "AVI ..." (por ejemplo, ver video 1 y 2 correspondientes a la Figura 1).
2. Usando sensores plegables y Reporteros Estrés encargado de investigar la célula autónoma y no de células Autónoma Afecto sobre Proteostasis y Toxicidad
- Generar líneas transgénicas que coexpresan un sensor de plegado o Reporte estrésr junto con la proteína prión-similares. Para los métodos sobre cómo establecer cruces o generar líneas transgénicas, consulte 48,49,52. Ver Tabla 1 para una lista de C. elegans cepas que se pueden utilizar.
- Preparar poblaciones sincronizados de animales transgénicos y crecer hasta la edad deseada como se describió anteriormente (sección 1.2) 50.
- El uso de un microscopio estereoscópico, examine el respectivo fenotipo del sensor de plegado o reportero estrés.
- Para el sensor de plegado, determinar el número de animales que albergan agregados de cada día después de la sincronización (por ejemplo, véase la Figura 2E y F).
- Para el reportero estrés, prueba si la coexpresión de las proteínas priónicas resultados similares en un aumento de fluorescencia del informador (por ejemplo, véase la Figura 3).
3. Pantalla de todo el genoma para la supresión de la toxicidad inducida por priones en C. elegans
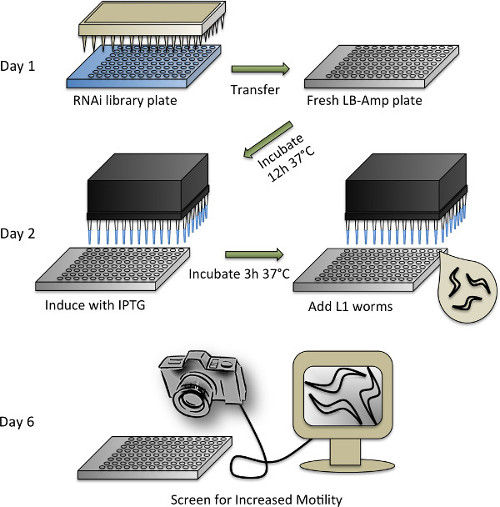
Figura 4. Representación esquemática del protocolo de cribado RNAi. Vea la sección protocolo 3 para una descripción detallada de los pasos individuales.
- Sincronización de C. gusanos elegans y la duplicación de RNAi biblioteca
- Para la pantalla de RNAi, utilizar la biblioteca Ahringer RNAi (o la biblioteca Vidal RNAi) 53,54. Rearray la biblioteca RNAi desde el formato de pozo original 384 en placas de 96 pocillos llenando las placas con 100 l de medios LB-Amp (50 mg / ml de ampicilina en LB) suplementado con 10% de glicerol y inocular utilizando un replicador de 96 pines. Crece a 37 ° CO / N con agitación y se almacena a -80 ° C.
- Mantener C. elegans WT y líneas transgénicas a 20 ° C en NGM placas sembradas con OP50 E. bacterias coli de acuerdo con métodos estándar 47.
- Sincronizar transgénico dominio prión y WT (control) nematodos por el blanqueamiento (ver sección 1.2).
- El mismo día que los gusanos se blanquean, preparar medio LB suplementado con 50 mg / ml de ampicilina. Utilice un dispensador de reactivos automatizado para dispensar (o una pipeta manual) 65 l de los medios de comunicación LB-amp en cada pocillo de una placa de 96 pocillos.
- Retire 96 y placas de biblioteca Ahringer RNAi de -80 ° C y llevarlos a una campana estéril forrada con papel de cocina. Retire inmediatamente la junta cinta manteniendo la placa boca abajo mientras que las placas todavía están congelados, teniendo cuidado de evitar la contaminación de hielo en el exterior de las placas. Reinvertir las placas y dejar que se descongelen durante aproximadamente 30 minutos.
- Sumerja una estéril replicador de 96 pines en una placa RNAi biblioteca, y luego en una placa LB-amp fresco. Use un replicador estéril nueva para cada plato. Selle todas las placas con cinta de papel adhesivo y devolver las placas de biblioteca a -80 ° C. Los replicadores pueden ser remojados en lejía, enjuagarse,y en autoclave para ser utilizado de nuevo.
- Permitir a las placas de RNAi duplicados para crecer O / N en una incubadora a 37 ° C y 300 rpm. Para utilizar HT115 E. coli bacterias que albergan el vector vacío (L4440) como un control, que crecen por separado en un tubo de cultivo y después de pipetear 65 l de cultivo con una pipeta multicanal en un cuarto de dos placas de 96 pocillos.
- La inducción de la producción bacteriana dsRNA y la adición de gusanos
- Diluir isopropil-β-D-thiogalactopyranosid (IPTG) a una concentración 5 mM en ddH 2 O. Utilice una estación de trabajo automatizado con una cabeza multicanal para dispensar (o pipeta manualmente) 10 l de IPTG diluido en cada pocillo de las bacterias RNAi y las placas de control. Volver a sellar y colocar las placas de nuevo en la incubadora. Deja que sacuden durante 3 horas a 37 ° C.
- Mientras que las bacterias están temblando, preparar los gusanos. Mezclar el / suspensión gusano M9 bien, y la pipeta una muestra pequeña (~ 5-10 l) a un portaobjetos de vidrio. Contarel número de gusanos bajo un microscopio estereoscópico y calcular el número de gusanos por l.
- Utilizando una técnica estéril, preparar una solución de M9 suplementado (M9 +) con las siguientes concentraciones finales: 0,20 mg / ml de IPTG, 8,0 g / ml de colesterol, 50 mg / ml de ampicilina, 9,6 g / ml de tetraciclina, 0.0835 g / ml de Fungizone, y 15 gusanos por 50 l.
- Haga soluciones separadas para transgénico dominio prión y gusanos WT. El volumen final de la solución gusano M9 + necesaria dependerá del número de placas de RNAi copiados (ver más abajo), además de ~ 30 ml de volumen muerto que permanecerá en el depósito, si se utiliza un dispensador automatizado.
- Después de la inducción 3 h de producción bacteriana dsRNA, tome las placas de la incubadora y dejar que se enfríe a temperatura ambiente (~ 30 min) para evitar el calor perturbe a los animales.
- Dispensar 50 l M9 + dominio prión solución gusano transgénico a cada pocillo de las placas de RNAi y una de las placas de control de vector vacío. HacerAsegúrese de mezclar la solución gusano antes de cada paso como los animales tienden a sedimentar al fondo del depósito.
- Prescindir gusanos WT en la segunda placa de control. Deje las placas sin sellar para permitir la aireación. Para mantener el cultivo líquido se evapore, apilar 4-5 platos juntos y envolver con una toalla de papel húmeda y papel de aluminio. Incubar a 200 rpm a 20 ° C durante 4 días.
- Tanteo
NOTA: Después de 4 días en la incubadora, los gusanos estarán en el segundo día de la edad adulta y están listos para defender. Deje que los animales se adaptan a condiciones no sacuden durante 30 minutos antes de cribado para asegurar golear sin ser molestados.- Utilizando una cámara Falcon 4M60 conectado a un ordenador con una pantalla del monitor visual para aumentar la thrashing en comparación con tratados con el control de dominio prión animales transgénicos.
- Compilar una lista de éxitos preliminares confirmar utilizando wrMTrck (véase la sección siguiente).
4. Confirmación de preliminarPantalla Golpea
- Ensayo de la motilidad en placas sólidas
- Preparar placas con NGM suplementadas con 100 g / ml de ampicilina, 12,5 mg / ml de tetraciclina y 1 mM IPTG, de acuerdo con métodos estándar 47. Si es posible, utilice una máquina de placa de vertido para asegurar todas las placas tienen la misma altura de los medios de comunicación, lo que permitirá un proceso de adquisición de vídeo más ágil.
- Cultivar las diferentes clones RNAi en ~ 1 ml de LB + 50 mg / ml de ampicilina, O / N a 37 ° C y 250 rpm.
- El día siguiente, inducir la expresión de la dsRNA con 1 mM IPTG durante 3 horas. Sembrar las placas con 150 l de cada clon bacteriano RNAi, que se distribuyen en una capa delgada. Deje que las bacterias secas durante 2 días a temperatura ambiente en la oscuridad. Preparar 3 planchas por RNAi clon.
- Sincronizar la población de gusanos por blanqueo de acuerdo con los métodos estándar 50 (ver sección 1.2), y dejar que los huevos eclosionan O / N en M9 medios.
- Tomar una muestra de la suspensión de gusano y determinar la cantidad de nematodes por l en el microscopio estereoscópico. Entonces, pipetear el volumen adecuado de M9 más gusanos en cada placa experimental para que contenga de 25 - 30 gusanos L1. Crecer los nematodos a 20 ° C durante 4 días hasta que los gusanos alcanzan día 2 de la edad adulta.
- El análisis cuantitativo de la motilidad gusano con el plugin wrMTrck para ImageJ
NOTA: Los videos fueron grabados utilizando un microscopio estereoscópico a 10 aumentos con un Hamamatsu Orca-R2 cámara digital C10600-10B y el software de imágenes PCI simple Hamamatsu.- Encienda la cámara y el software de imágenes PCI simple. Haga clic en "Live" para permitir el ajuste de las condiciones de la imagen.
- Configurar las condiciones de vídeo de la siguiente manera: Ganancia = 0; Modo Luz = Alta; Velocidad Índice = 1; Intervalos = 2. Haga clic en "Auto Expose" y luego ajustar las condiciones de luz, mover los espejos de microscopio y los diales de brillo y contraste.
NOTA: El video tiene que tener un alto contraste sin ser Sobreexpcerradas de la, por lo que los animales aparecen como formas negras sobre un fondo brillante. - Haga clic en "Scan Time" y seleccione una carpeta y un nombre de archivo. Ajuste "Delay Campo" a 20 ms y "Parada en el tiempo" a 30 seg. Presione "Revisión Live" con el fin de elegir el área de la placa para grabar (donde la mayoría de los gusanos son). Toque en la placa de 3 o 4 veces en el escenario, de forma rápida confirmar su posición en el campo de visión y presione "Start".
- Después de la película termine de grabar, haga clic derecho sobre la imagen y selecciona "Exportar Montaje Secuencia" para exportar la película a partir de un formato .cxd a un .avi.
- El análisis de los vídeos de la motilidad
- Abra el software ImageJ, vaya a la pestaña "Plugins", luego "wrmtrck" y selecciona "Lote wrMTrck". Seleccione el directorio que contiene todos los archivos a analizar.
- En la ventana de entrada principal de wrMTrck_Batch, cargue los valores de entrada como se detalla en Figure 5C. Explicación para cada uno de los parámetros se puede encontrar en las instrucciones que acompañan el plugin. Haga clic en "Aceptar" y dejar que el análisis del movimiento de ejecución.
- El comisariado de los resultados y confirmar que todas las pistas detectadas son de real C. elegans y eliminar los artefactos, se abren cada uno de los archivos .txt creados para cada película y copiar la información a un archivo de software de análisis de datos. Abra el "*" _labels.zip archivos creados y ejecutar el resultante "* _labels.tif" para comprobar y eliminar pistas falsas gusano manualmente.
Resultados
Monitoreo intercelular propagación de proteínas priónicas como in vivo por time-lapse
Transgénicos C. elegans líneas que expresan el dominio de priones son particularmente adecuados para el análisis de ciertos aspectos de proteínas priónicas-como, por ejemplo, la transmisión de célula a célula y la toxicidad no autónoma de la célula. La transparencia de los animales permite el seguimiento de fluorescencia proteínas etiquetadas desde d...
Discusión
Los métodos descritos aquí ayudan a ilustrar la difusión y la toxicidad celular autónoma la célula compleja autónomos y no de las proteínas priones similares. Recientemente hemos descubierto que un dominio citosólico prión agregación propensos se recoge en vesículas de membrana en un proceso de autofagia relacionados. Un subconjunto específico de estas vesículas transporta el dominio prión dentro de y entre las células y los tejidos 40. La clave para controlar su movimiento en el animal vivo es...
Divulgaciones
The authors declare no competing financial interests.
Agradecimientos
We thank Cindy Voisine and Yoko Shibata for helpful discussion and critical comments on the manuscript. We acknowledge the High Throughput Analysis Laboratory (HTAL) and the Biological Imaging Facility (BIF) at Northwestern University for their assistance. This work was funded by grants from the National Institutes of Health (NIGMS, NIA, NINDS), the Ellison Medical Foundation, and the Daniel F. and Ada L. Rice Foundation (to R.I.M.). C.I.N.-K. was supported by the Deutsche Forschungsgemeinschaft (KR 3726/1-1).
Materiales
| Name | Company | Catalog Number | Comments |
| Reagent | |||
| Nanosphere size standards 100 nm | ThermoScientific | 3100A | |
| Levamisole | Sigma | L-9756 | |
| IPTG | Sigma | 15502-10G | |
| Ahringer RNAi library | Source BioScience LifeSciences | http://www.lifesciences.sourcebioscience .com/clone-products/non-mammalian/c-elegans/c-elegans-rnai-library/ | |
| Equipment | |||
| Sorvall Legend XTR Refrigerated Centrifuge, 120VAC | ThermoScientific | 75004521 | http://www.coleparmer.com/Product/Thermo_Scientific_Sorvall_Legend_ XTR_Refrigerated_Centrifuge_120 VAC/EW-17707-60 |
| 96 pin replicator | Scionomix | http://www.scinomix.com/all-products/96-pin-replicator/ | |
| HiGro high-capacity, incubating shaker | Digilab | http://www.digilabglobal.com/higro | |
| Multidrop Combi Reagent Dispenser | Titertrek | http://groups.molbiosci.northwestern.edu/hta/titertek.htm | |
| Biomek FX AP96 Automated Workstation | Beckman Coulter | http://groups.molbiosci.northwestern.edu/hta/biomek_multi.htm | |
| Innova44 shaker | New Brunswick | http://www.eppendorf.com/int///index.php?sitemap=2.3&pb=d78efbc05310ec 04&action=products&contentid=1& catalognode=83389 | |
| M205 FA | Leica | http://www.leica-microsystems.com/de/produkte/stereomikroskope-makroskope/fluoreszenz/details/product/leica-m205-fa/ | |
| ORCA-R2 C10600-10BDigital CCD camera | Hamamatsu | http://www.hamamatsu.com/jp/en/community/life_science_camera/product/search/C10600-10B/index.html | |
| Spinning Disc AF Confocal Microscope | Leica | http://www.leica-microsystems.com/products/light-microscopes/life-science-research/fluorescence-microscopes/details/product/leica-sd-af/ | |
| Falcon 4M60 camera | Teledyne Dalsa | http://www.teledynedalsa.com/imaging/products/cameras/area-scan/falcon/PT-41-04M60/ | |
| Software | |||
| MetaMorph Microscopy Automation & Image Analysis Software | Molecular Devices | http://www.moleculardevices.com/products/software/meta-imaging-series/metamorph.html | |
| Hamamatsu SimplePCI Image Analysis Software | Meyer Instruments | http://meyerinst.com/imaging-software/hamamatsu/index.htm | |
| ImageJ | NIH | http://rsbweb.nih.gov/ij/download.html | |
| wrMTrck plugin for ImageJ | http://www.phage.dk/plugins/wrmtrck.html | ||
| C. elegans strains | |||
| N2 (WT) | Caenorhabditis Genetics Center (CGC) | http://www.cgc.cbs.umn.edu/strain.php?id=10570 | |
| AM815 rmIs323[myo-3p::sup35(r2e2)::rfp] | Morimoto lab | available from our laboratory | |
| See table 1 for a source for folding sensor and stress reporter strains |
Referencias
- Prusiner, S. B. Novel proteinaceous infectious particles cause scrapie. Science. 216 (4542), 136-144 (1982).
- Jarrett, J. T., Lansbury, P. T. Seeding 'one-dimensional crystallization' of amyloid: a pathogenic mechanism in Alzheimer's disease and scrapie. Cell. 73 (6), 1055-1058 (1993).
- Caughey, B., Kocisko, D. A., Raymond, G. J., Lansbury, P. T. Aggregates of scrapie-associated prion protein induce the cell-free conversion of protease-sensitive prion protein to the protease-resistant state. Chem Biol. 2 (12), 807-817 (1995).
- Wickner, R. B. [URE3] as an altered URE2 protein: evidence for a prion analog in Saccharomyces cerevisiae. Science. 264 (5158), 566-569 (1994).
- Chien, P., Weissman, J. S., DePace, A. H. Emerging principles of conformation-based prion inheritance. Annu Rev Biochem. 73, 617-656 (2004).
- Kimberlin, R. H., Walker, C. A. Pathogenesis of mouse scrapie: patterns of agent replication in different parts of the CNS following intraperitoneal infection. J R Soc Med. 75 (8), 618-624 (1982).
- Beekes, M., McBride, P. A., Baldauf, E. Cerebral targeting indicates vagal spread of infection in hamsters fed with scrapie. J Gen Virol. 79 (3), 601-607 (1998).
- Jucker, M., Walker, L. C. Self-propagation of pathogenic protein aggregates in neurodegenerative diseases. Nature. 501 (7465), 45-51 (2013).
- Aguzzi, A. Cell biology: Beyond the prion principle. Nature. 459 (7249), 924-925 (2009).
- Scherzinger, E., et al. Self-assembly of polyglutamine-containing huntingtin fragments into amyloid-like fibrils: implications for Huntington's disease pathology. Proc Natl Acad Sci U S A. 96 (8), 4604-4609 (1999).
- Wood, S. J., et al. alpha-synuclein fibrillogenesis is nucleation-dependent. Implications for the pathogenesis of Parkinson's disease. J Biol Chem. 274 (28), 19509-19512 (1999).
- Wang, Y. Q., et al. Relationship between prion propensity and the rates of individual molecular steps of fibril assembly. J Biol Chem. 286 (14), 12101-12107 (2011).
- Cushman, M., Johnson, B. S., King, O. D., Gitler, A. D., Shorter, J. Prion-like disorders: blurring the divide between transmissibility and infectivity. J Cell Sci. 123 (8), 1191-1201 (2010).
- Tanaka, M., Collins, S. R., Toyama, B. H., Weissman, J. S. The physical basis of how prion conformations determine strain phenotypes. Nature. 442 (7102), 585-589 (2006).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. J Struct Biol. 179 (2), 152-160 (2012).
- Ilieva, H., Polymenidou, M., Cleveland, D. W. Non-cell autonomous toxicity in neurodegenerative disorders: ALS and beyond. J Cell Biol. 187 (6), 761-772 (2009).
- Nussbaum-Krammer, C. I., Morimoto, R. I. Caenorhabditis elegans as a model system for studying non-cell-autonomous mechanisms in protein-misfolding diseases. Dis Model Mech. 7 (1), 31-39 (2014).
- Lino, M. M., Schneider, C., Caroni, P. Accumulation of SOD1 mutants in postnatal motoneurons does not cause motoneuron pathology or motoneuron disease. J Neurosci. 22 (12), 4825-4832 (2002).
- Li, J. Y., et al. Lewy bodies in grafted neurons in subjects with Parkinson's disease suggest host-to-graft disease propagation. Nat Med. 14 (5), 501-503 (2008).
- Desplats, P., et al. Inclusion formation and neuronal cell death through neuron-to-neuron transmission of alpha-synuclein. Proc Natl Acad Sci U S A. 106 (31), 13010-13015 (2009).
- Clement, A. M., et al. Wild-type nonneuronal cells extend survival of SOD1 mutant motor neurons in ALS mice. Science. 302 (5642), 113-117 (2003).
- Gu, X., et al. Pathological cell-cell interactions elicited by a neuropathogenic form of mutant Huntingtin contribute to cortical pathogenesis in HD mice. Neuron. 46 (3), 433-444 (2005).
- Yamanaka, K., et al. Mutant SOD1 in cell types other than motor neurons and oligodendrocytes accelerates onset of disease in ALS mice. Proc Natl Acad Sci U S A. 105 (21), 7594-7599 (2008).
- Garden, G. A., et al. Polyglutamine-expanded ataxin-7 promotes non-cell-autonomous purkinje cell degeneration and displays proteolytic cleavage in ataxic transgenic mice. J Neurosci. 22 (12), 4897-4905 (2002).
- Raeber, A. J., et al. Astrocyte-specific expression of hamster prion protein (PrP) renders PrP knockout mice susceptible to hamster scrapie. EMBO J. 16 (20), 6057-6065 (1997).
- Yazawa, I., et al. Mouse model of multiple system atrophy alpha-synuclein expression in oligodendrocytes causes glial and neuronal degeneration. Neuron. 45 (6), 847-859 (2005).
- Lobsiger, C. S., Cleveland, D. W. Glial cells as intrinsic components of non-cell-autonomous neurodegenerative disease. Nat Neurosci. 10 (11), 1355-1360 (2007).
- Sambataro, F., Pennuto, M. Cell-autonomous and non-cell-autonomous toxicity in polyglutamine diseases. Prog Neurobiol. 97 (2), 152-172 (2012).
- Polymenidou, M., Cleveland, D. W. Prion-like spread of protein aggregates in neurodegeneration. J Exp Med. 209 (5), 889-893 (2012).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nat Rev Mol Cell Biol. 11 (4), 301-307 (2010).
- Braak, H., Braak, E., Bohl, J. Staging of Alzheimer-related cortical destruction. Eur Neurol. 33 (6), 403-408 (1993).
- Meyer-Luehmann, M., et al. Exogenous induction of cerebral beta-amyloidogenesis is governed by agent and host. Science. 313 (5794), 1781-1784 (2006).
- Luk, K. C., et al. Pathological alpha-synuclein transmission initiates Parkinson-like neurodegeneration in nontransgenic mice. Science. 338 (6109), 949-953 (2012).
- Clavaguera, F., et al. Transmission and spreading of tauopathy in transgenic mouse brain. Nat Cell Biol. 11 (7), 909-913 (2009).
- Nonaka, T., et al. Prion-like Properties of Pathological TDP-43 Aggregates from Diseased Brains. Cell Rep. 4 (1), 124-134 (2013).
- Lundmark, K., et al. Transmissibility of systemic amyloidosis by a prion-like mechanism. Proc Natl Acad Sci U S A. 99 (10), 6979-6984 (2002).
- Lai, C. H., Chou, C. Y., Ch'ang, L. Y., Liu, C. S., Lin, W. Identification of novel human genes evolutionarily conserved in Caenorhabditis elegans by comparative proteomics. Genome Res. 10 (5), 703-713 (2000).
- Xu, X., Kim, S. K. The early bird catches the worm: new technologies for the Caenorhabditis elegans toolkit. Nat Rev Genet. 12 (11), 793-801 (2011).
- Boulin, T., Hobert, O. From genes to function: the C. elegans genetic toolbox. Wiley Interdiscip Rev Dev Biol. 1 (1), 114-137 (2012).
- Nussbaum-Krammer, C. I., Park, K. W., Li, L., Melki, R., Morimoto, R. I. Spreading of a prion domain from cell-to-cell by vesicular transport in Caenorhabditis elegans. PLoS Genet. 9 (3), e1003351 (2013).
- Chernoff, Y. O., Lindquist, S. L., Ono, B., Inge-Vechtomov, S. G., Liebman, S. W. Role of the chaperone protein Hsp104 in propagation of the yeast prion-like factor [psi. Science. 268 (5212), 880-884 (1995).
- Liu, J. J., Lindquist, S. Oligopeptide-repeat expansions modulate 'protein-only' inheritance in yeast. Nature. 400 (6744), 573-576 (1999).
- Halfmann, R., et al. Prions are a common mechanism for phenotypic inheritance in wild yeasts. Nature. 482 (7385), 363-368 (2012).
- Tyedmers, J., Madariaga, M. L., Lindquist, S. Prion switching in response to environmental stress. PLoS Biol. 6 (11), e294 (2008).
- Krammer, C., et al. The yeast Sup35NM domain propagates as a prion in mammalian cells. Proc Natl Acad Sci U S A. 106 (2), 462-467 (2009).
- Hofmann, J. P., et al. Cell-to-cell propagation of infectious cytosolic protein aggregates. Proc Natl Acad Sci U S A. 110 (15), 5951-5956 (2013).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , (2006).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C. elegans Using Microinjection. J. Vis. Exp. (18), e833 (2008).
- Evans, T. C. Transformation and microinjection. WormBook. , (2006).
- Shaham, S. Methods in cell biology. Wormbooks. , (2006).
- Kim, E., Sun, L., Gabel, C. V., Fang-Yen, C. Long-term imaging of Caenorhabditis elegans using nanoparticle-mediated immobilization). PLoS One. 8 (1), e53419 (2013).
- Fay, D. Genetic mapping and manipulation: Chapter 1-Introduction and basics. WormBook. , (2006).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Rual, J. F., et al. Toward improving Caenorhabditis elegans phenome mapping with an ORFeome-based RNAi library. Genome Res. 14 (10B), 2162-2168 (2004).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Kern, A., Ackermann, B., Clement, A. M., Duerk, H., Behl, C. HSF1-controlled and age-associated chaperone capacity in neurons and muscle cells of C. elegans. PLoS One. 5 (1), e8568 (2010).
- Becker, J., Walter, W., Yan, W., Craig, E. A. Functional interaction of cytosolic hsp70 and a DnaJ-related protein, Ydj1p, in protein translocation in vivo. Mol Cell Biol. 16 (8), 4378-4386 (1996).
- Salvaterra, P. M., McCaman, R. E. Choline acetyltransferase and acetylcholine levels in Drosophila melanogaster: a study using two temperature-sensitive mutants. J Neurosci. 5 (4), 903-910 (1985).
- Goloubinoff, P., Mogk, A., Zvi, A. P., Tomoyasu, T., Bukau, B. Sequential mechanism of solubilization and refolding of stable protein aggregates by a bichaperone network. Proc Natl Acad Sci U S A. 96 (24), 13732-13737 (1999).
- Schroder, H., Langer, T., Hartl, F. U., Bukau, B. D. n. a. K. DnaJ and GrpE form a cellular chaperone machinery capable of repairing heat-induced protein damage. EMBO J. 12 (11), 4137-4144 (1993).
- Rampelt, H., et al. Metazoan Hsp70 machines use Hsp110 to power protein disaggregation. EMBO J. 31 (21), 4221-4235 (2012).
- Gupta, R., et al. Firefly luciferase mutants as sensors of proteome stress. Nat Methods. 8 (10), 879-884 (2011).
- Gidalevitz, T., Ben-Zvi, A., Ho, K. H., Brignull, H. R., Morimoto, R. I. Progressive disruption of cellular protein folding in models of polyglutamine diseases. Science. 311 (5766), 1471-1474 (2006).
- Ben-Zvi, A., Miller, E. A., Morimoto, R. I. Collapse of proteostasis represents an early molecular event in Caenorhabditis elegans aging. Proc Natl Acad Sci U S A. 106 (35), 14914-14919 (2009).
- Karady, I., et al. Using Caenorhabditis elegans as a model system to study protein homeostasis in a multicellular organism. J Vis Exp. (82), e50840 (2013).
- Gidalevitz, T., Krupinski, T., Garcia, S., Morimoto, R. I. Destabilizing protein polymorphisms in the genetic background direct phenotypic expression of mutant SOD1 toxicity. PLoS Genet. 5 (3), e1000399 (2009).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proc Natl Acad Sci U S A. 99 (16), 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine proteins at the pathogenic threshold display neuron-specific aggregation in a pan-neuronal Caenorhabditis elegans model. J Neurosci. 26 (29), 7597-7606 (2006).
- Mohri-Shiomi, A., Garsin, D. A. Insulin signaling and the heat shock response modulate protein homeostasis in the Caenorhabditis elegans intestine during infection. J Biol Chem. 283 (1), 194-201 (2008).
- Libina, N., Berman, J. R., Kenyon, C. Tissue-specific activities of C. elegans DAF-16 in the regulation of lifespan. Cell. 115 (4), 489-502 (2003).
- Schatzl, H. M., et al. A hypothalamic neuronal cell line persistently infected with scrapie prions exhibits apoptosis. J Virol. 71 (11), 8821-8831 (1997).
- Keith, S. A., Amrit, F. R., Ratnappan, R., Ghazi, A. The C. elegans healthspan and stress-resistance assay toolkit. Methods. , (2014).
- Pierce-Shimomura, J. T., et al. Genetic analysis of crawling and swimming locomotory patterns in C. elegans. Proc Natl Acad Sci U S A. 105 (52), 20982-20987 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados