É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Investigando as proteínas príon-like espalhando e de toxicidade de usar o Metazoan Organismo Modelo
Neste Artigo
Resumo
Prion-like propagation of protein aggregates has recently emerged as being implicated in many neurodegenerative diseases. The goal of this protocol is to describe, how to use the nematode C. elegans as a model system to monitor protein spreading and to investigate prion-like phenomena.
Resumo
Prions are unconventional self-propagating proteinaceous particles, devoid of any coding nucleic acid. These proteinaceous seeds serve as templates for the conversion and replication of their benign cellular isoform. Accumulating evidence suggests that many protein aggregates can act as self-propagating templates and corrupt the folding of cognate proteins. Although aggregates can be functional under certain circumstances, this process often leads to the disruption of the cellular protein homeostasis (proteostasis), eventually leading to devastating diseases such as Alzheimer’s disease (AD), Parkinson’s disease (PD), Amyotrophic lateral sclerosis (ALS), or transmissible spongiform encephalopathies (TSEs). The exact mechanisms of prion propagation and cell-to-cell spreading of protein aggregates are still subjects of intense investigation. To further this knowledge, recently a new metazoan model in Caenorhabditis elegans, for expression of the prion domain of the cytosolic yeast prion protein Sup35 has been established. This prion model offers several advantages, as it allows direct monitoring of the fluorescently tagged prion domain in living animals and ease of genetic approaches. Described here are methods to study prion-like behavior of protein aggregates and to identify modifiers of prion-induced toxicity using C. elegans.
Introdução
Muitas doenças neurodegenerativas, como a doença de Alzheimer (DA), doença de Parkinson (DP), a esclerose lateral amiotrófica (ELA), e encefalopatias espongiformes transmissíveis (EET), estão associados a proteínas de agregação propensas e são, portanto, conhecidos coletivamente como proteína misfolding transtornos (EPM ). EET ou doenças provocadas por priões constituem uma classe única de EPM em que eles podem ser infecciosas em seres humanos e animais 1. A nível molecular, prions replicar recrutando e convertendo monomérica α-hélice-rico codificado-host PrP celular (PrP C) para o patológico β-folha-rico conformação PrP Sc 2,3. Agregados de proteína auto-propagação também foram identificados em fungos, que partilham características importantes de mamíferos com priões 4,5. Além disso, os priões mamíferos são capazes de mover a partir de célula-a-célula e infectar células naive 6,7.
Enquanto EPM other do TSE não são infecciosos, eles compartilham um princípio patogênico comum com doenças de príon 8,9. Embora as proteínas ligadas a cada um dos EPM não estão relacionados em termos de estrutura ou função, eles todos formam agregados por meio de um processo de cristalização, chamado nucleada ou semeada de polimerização; Além disso sementes proteicos crescer através do recrutamento de suas isoformas solúveis 2,10,11. A eficiência para a auto-propagam in vivo varia, dependendo das propriedades intrínsecas da proteína, que, em conjunto com factores celulares adicionais, tais como as chaperonas moleculares, em última análise determinam as taxas de nucleação agregado, a sementeira, a fragmentação e espalhamento 12-15. Por isso, deve haver um bom equilíbrio entre esses fatores que permite a propagação eficiente de agregação de proteínas. Isso também pode explicar por que apenas alguns agregados amiloidogénicas abrigar as características de um prião, e, assim, nem todos os EPM são infecciosas. Os príons parecem representar o 'top-performers'fa amplo espectro de agregados proteicos de auto-replicação, o que os torna uma ferramenta poderosa para estudar EPM 8,13.
Curiosamente, a toxicidade associada com agregados relacionados com a doença muitas vezes tem um componente autônomo não célula 16,17. Isto significa que eles afectam as células vizinhas que não expressam o gene correspondente, em contraste com um efeito estritamente autónoma célula, o que implica que apenas as células que expressam o gene exibem o fenótipo específico. Este foi convincentemente demonstrada pela expressão específica de tecido ou derrubar das respectivas proteínas em vários modelos de doenças neurodegenerativas 18-26. Vários mecanismos têm sido sugeridos como uma base para esta não-toxicidade celular autônomo na EPM, incluindo o fornecimento de nutrientes diminuído, o desequilíbrio na sinalização neuronal, excitotoxicidade do glutamato, e neuroinflammation 16,27,28. Além disso, um movimento de prião semelhante de agregados ligados de doenças entre as células might contribuir para este aspecto 29,30. Uma evidência crescente sugere que outras inclusões de proteína que pode transmitir priões a partir de célula-para-célula, o que pode explicar a característica de difusão na patologia observada em muitos EPM 30-36. No entanto, ele ainda não foi determinado se há um claro nexo de causalidade entre o movimento intercelular de proteínas doença eo efeito tóxico sobre as células vizinhas. Por conseguinte, um melhor entendimento das vias celulares que estão subjacentes a transmissão célula-a-célula e célula não autónomo toxicidade é necessária e essencial para o desenvolvimento de novas terapias. No entanto, muitos aspectos do prião como espalhando-celulares e factores que influenciam a transmissão célula a célula de proteínas mal enroladas em metazoários não são bem compreendidos, em particular ao nível dos organismos.
O nemátodo Caenorhabditis elegans tem várias vantagens que oferecem o potencial de descobrir novas facetas de spreadi prion-likeng em metazoans 17. É transparente, permitindo a rastrear in vivo de proteínas marcadas fluorescentemente no organismo vivo. Além disso, muitos processos celulares e fisiológicas afectadas pela doença são conservados de vermes para a saúde humana, e C. elegans é também susceptível a uma grande variedade de manipulações genéticas e análises moleculares e bioquímicos 37-39. Exatamente 959 células somáticas compõem o hermafrodita adulto com um plano corporal simples, que ainda tem vários tipos de tecidos diferentes, incluindo músculos, neurônios e intestino.
Para estabelecer um novo modelo de príon em C. elegans, que escolheu para expressar exogenamente a glutamina bem caracterizada / asparagina (Q / N) rico em prião NM domínio do cytosolic proteína de levedura prion Sup35, já que não há proteínas príon endógenas conhecidas em vermes 4,40. Prions levedura foram inestimáveis para elucidar os mecanismos básicos de prion replicação 41-44. Além disso, NM é o abetoproteína prião como citosólica-r que tem sido mostrado para recapitular o ciclo de vida completo de um prião em cultura de células de mamífero 45,46. Da mesma forma, quando expresso em C. elegans, o domínio prion Sup35 adotado muito bem com as diferentes necessidades para propagação em células metazoários em comparação com células de levedura e principais características expostas da biologia prion agregação 40. NM foi associada com um fenótipo tóxico profunda, incluindo a ruptura da integridade mitocondrial e aparecimento de várias vesículas autofagia relacionados sobre o nível celular, bem como prisão embrionário e larval, atraso do desenvolvimento, e uma perturbação generalizada do ambiente dobramento de proteínas ao nível do organismo. Surpreendentemente, o domínio príon celular apresenta toxicidade autônoma e não autônomos celular, afetando tecidos vizinhos em que o transgene não foi expressa. Além disso, o transporte vesicular do domínio prião dentro e entre as células é monitorado em tempo real in vivo 40.
Aqui nós descrevemos como examinar divulgação prion-like em C. elegans. Vamos explicar como monitorar o transporte intra e intercelular de vesículas contendo o domínio prion usando microscopia de fluorescência de lapso de tempo. Vamos enfatizar o uso de sensores de dobradura de tecidos específicos e universalmente expressa repórteres de estresse para avaliar Cell efeitos autônomos autónomos e não sobre fitness celular. Finalmente, vamos descrever o procedimento de uma grande tela recentemente realizada genoma RNA de interferência (RNAi) para identificar novos modificadores de toxicidade induzida por prion. Em conjunto, estes métodos podem ajudar a provocar uma separação de caminhos genéticos envolvidos no movimento intercelular de proteínas e sua toxicidade autónoma non celular.
Protocolo
1. Monitoramento transcelular Espalhando de proteínas príon-like por In Vivo tempo-lapso
NOTA: Crescer C. elegans do tipo selvagem (WT) (N2) e linhas transgénicas de acordo com métodos padrão e controlar cuidadosamente a temperatura de cultivo 47.
- Gerar linhagens transgênicas de C. elegans que expressa a proteína prion-like, marcado com proteína fluorescente vermelha monomérica (MRFP). Assista a este vídeo que demonstra como usar a microinjeção 48. Para mais detalhes e métodos que descrevem como integrar estas linhas extracromossómicos, consulte 49.
- Prepare populações sincronizadas por colocação de ovo ou branqueamento de acordo com métodos padronizados 50.
- Sincronização de postura de ovos
- Transferência de 10-20 adultos grávidas em um prato e deixe que eles põem ovos por 1-2 horas. Remover adultos a partir da placa e deixar a crescer até a progenitura idade desejada.
- Sincronização por branqueamento
- Recolha uma população de adultos grávidos não sincronizado e branquear com uma solução de hipoclorito alcalino (NaOH a 250 mM e 1: 4 (v / v) diluição da lixívia comercial em H 2 O). Lavar os ovos duas vezes (218 xg durante 1 min) com tampão M9 47 (21 MMNA 2 HPO 4 · 7H 2 O, 22 mMKH 2 PO 4, 86 mMNaCl, 1 mMMgSO · 4 7H 2 O, adicionar dH2O até 1 L).
- Permita-lhes a eclodir em tampão M9 com suave agitação O / N a 20 ° C. Desenvolvimento verme vai prender na fase L1 na ausência de uma fonte de alimento, deixando uma população sincronizada. Transfira para L1s fresco nematóides Crescimento Media (NGM) placas semeadas com OP50 E. coli bactérias e deixar a prole se desenvolver até a idade desejada 47.
- Sincronização de postura de ovos
- Prepare 2% almofadas de agarose (em H 2 O) em uma lâmina de microscópio como descrito 50.
- Prepare dois microscope com lâminas de fita de rotulagem colocados ao longo de todo o seu comprimento para ser usado como espaçadores. Colocar uma terceira lâmina de microscópio entre eles.
- Dissolve-se 2% de agarose em H 2 O e uma pipeta de queda sobre a lâmina limpa.
- Coloque uma quarta corrediça perpendicular aos outros três lâminas sobre o topo da gota de agar. Pressione levemente para baixo para achatar a almofada para a mesma espessura que a fita de rotulagem.
- Deixe secar por 1 min antes de retirar os espaçadores e puxando os slides separados. A almofada de agar vai ficar com um deles.
- Pipeta ~ 10 ul anestésico (2 levamisol mM em tampão M9) para a almofada de transferência e ~ 10 animais utilizando-se uma escolha de fio de platina. Cubra com uma lamela (~ 22 x 22 mm) e tirar fotos dentro de 1 hora.
- Alternativamente, para adquirir os filmes ao longo de um longo período de tempo ou para reduzir ainda mais a possibilidade de uma deslocação dos animais, usar uma combinação de anestésico e talão de imobilização 51.
- Prepare 10% agaalmofadas aumentou (em tampão M9), conforme descrito 51 e adicionar minhocas a 3 ul solução padrões de tamanho nanosphere (esferas de poliestireno, 100 nm), mais 3 anestésicos ul (4 levamisol mM em tampão M9). Cubra delicadamente com uma lamela. Para evitar a desidratação, selar a lamínula com valap (mistura de quantidades iguais de vaselina, lanolina e cera de parafina).
- Imagem vermes imobilizada utilizando um microscópio confocal.
Nota: Os resultados são obtidos utilizando um disco giratório AF confocal microscópio equipado com uma câmera de EM-CCD e um Software Microscopia Automação e Análise de Imagem, como MetaMorph e delinear os detalhes abaixo, mas outros sistemas de imagem confocal comparável também pode ser usado.- Use o objetivo de óleo 63X ou 100X / 1.4NA e coloque a lâmina de microscópio contendo vermes no suporte da lâmina de microscópio.
- Abra o software. Ajuste de potência do laser e filtros para MRFP imagem. Use o laser 561 nm a 10% de energia e emissão de filtro> 600 nm.
- Open "MultiDimensional Aquisição". Na aba "Main", marque as caixas de "Timelapse" e "Run Journals" (Hardware Auto Foco: off).
- Sob a guia "Saving" selecionar ou criar a pasta do diretório onde os arquivos devem ser salvos. Atribua um nome para o arquivo.
- No separador "Comprimento de onda", selecione a iluminação adequada e ajustar a exposição e ganho. Use "YokoQuad Red" (ou uma configuração de iluminação equivalente para MRFP de imagem) com uma exposição de entre 100 e 300 ms e um ganho da câmera entre 100 e 300, dependendo da amostra individual (avaliada usando a imagem ao vivo).
- No separador "Timelapse" selecionar "Número de pontos de tempo" = 301, "Duração" = 5 min e "Time / Interval" = 1 seg.
- Sob a "Journals" tab selecione Journal: "AFC SET Z HOLD" e "AFC Return to Z HOLD", Tipo: &# 8220; Special "(duas vezes), Ponto inicial:" Inicio do horário de ponto "e" Fim do ponto de tempo ". Esta opção de focagem automática é importante para a imagem da mesma secção ao longo de um período mais longo de tempo.
- Quando a instalação for concluída, pressione "Adquirir". O vídeo timelapse é safed como arquivos TIFF separados.
- Abra todas ficheiros.tiff de uma determinada série tempo em ImageJ. Vá em "Imagem" → "Stacks" → "Imagens para Stacks". Opcionalmente, ajustar brilho e contraste, adicionar bar tamanho, etc. Exportar o filme em "File" → "salvar como" → "AVI ..." (por exemplo, ver vídeo 1 e 2 correspondente à Figura 1).
2. Usando dobráveis Sensores e Repórteres de estresse para Investigar celular autónomos e não celular Autónoma afeta em Proteostase e Toxicidade
- Gerar linhagens transgênicas que co-expressar um sensor de dobragem ou estresse reporter em conjunto com a proteína prião semelhante. Para os métodos sobre como estabelecer cruzes ou gerar linhagens transgênicas, ver 48,49,52. Ver Tabela 1 para uma lista de C. elegans estirpes que podem ser utilizadas.
- Prepare populações sincronizados de animais transgênicos e cultivá-las até a idade desejada, conforme descrito acima (seção 1.2) 50.
- Usando um microscópio estereoscópico, examinar o respectivo fenótipo do sensor de dobragem ou estresse repórter.
- Para o sensor de dobragem, determina o número de animais que albergam agregados em cada dia depois da sincronização (por exemplo, ver Figura 2E e F).
- Para o repórter tensão, se o ensaio de co-expressão dos resultados priões do tipo de proteína em uma fluorescência aumentada do repórter (para um exemplo, ver Figura 3).
3. Tela Genome-wide para Repressão de toxicidade induzida por Prion em C. elegans
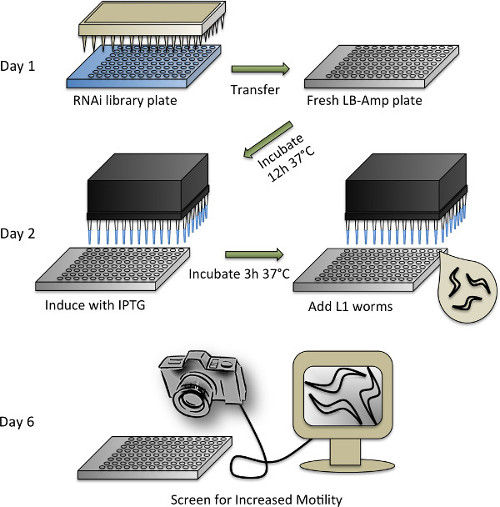
Figura 4. Representação esquemática do protocolo de triagem RNAi. Consulte a seção de protocolo 3 para uma descrição detalhada das etapas individuais.
- Sincronização de C. vermes elegans e duplicação de biblioteca RNAi
- Para a tela de RNAi, usar a biblioteca Ahringer RNAi (ou a biblioteca Vidal RNAi) 53,54. Rearray a biblioteca de RNAi do original 384 formato bem em placas de 96 poços, preenchendo as placas com 100 ul meio LB-amp (50 ug / ml de ampicilina em LB) suplementado com 10% de glicerol e inocular utilizando um replicador de 96 pinos. Crescer a 37 ° CO / N com agitação e armazenar a -80 ° C.
- Manter C. linhagens transgênicas em 20 ° C em placas NGM elegans WT e semeado com OP50 E. bactérias coli de acordo com métodos padrão 47.
- Sincronize transgênico domínio prion e WT (controle) nematóides pelo branqueamento (ver secção 1.2).
- No mesmo dia em que os vermes são branqueados, preparar meio LB suplementado com 50 ug / ml de ampicilina. Usar um dispensador automático para distribuir o reagente (ou pipeta manualmente) 65 ul dos meios LB-amp em cada poço de uma placa de 96 poços.
- Retirar 96 poços placas biblioteca Ahringer RNAi de -80 ° C e trazê-los para um capuz estéril forrado com papel toalha. Remover imediatamente o selo-tape, segurando a placa de cabeça para baixo, enquanto as placas ainda estão congelados, tendo o cuidado para evitar a contaminação de gelo do lado de fora das placas. Reinverter as placas e deixá-los descongelar por cerca de 30 min.
- Mergulhe uma estéril 96 pin replicador em uma placa de RNAi biblioteca, e, em seguida, em uma placa de LB-amp fresco. Use um replicador estéril nova para cada prato. Vede todas as placas com fita adesiva e papel alumínio retornar as placas da biblioteca para -80 ° C. Os replicadores pode ser embebido em água sanitária, enxaguado,e autoclavado para ser usado novamente.
- Permitir placas duplicadas de ARNi para crescer O / N em uma incubadora ajustada a 37 ° C e 300 rpm. Para usar HT115 E. coli bactérias que albergam o vector vazio (L4440) como um controlo, crescem-lo separadamente num tubo de cultura e, em seguida, pipeta 65 ul da cultura com uma pipeta de canais múltiplos dentro de um quarto de duas placas de 96 poços.
- A indução da produção bacteriana ARNcd e adição de vermes
- Dilui-Isopropil-β-D-thiogalactopyranosid (IPTG) para uma concentração de 5 mM em ddH 2 O. Utilize uma estação de trabalho automatizada com uma cabeça para dispensar múltiplos canais (ou pipeta manualmente) 10 ul de IPTG diluída em todos os poços das bactérias RNAi e as placas de controlo. Voltar a vedar e colocar as placas de novo na incubadora. Deixá-los agitar durante 3 horas a 37 ° C.
- Enquanto as bactérias estão tremendo, preparar os vermes. Misture a suspensão M9 / verme bem, e pipetar uma pequena amostra (~ 5 - 10 ul) para uma lâmina de microscópio de vidro. Contaro número de vermes sob microscópio estereoscópico e calcular o número de vermes por ul.
- Utilizando uma técnica estéril, preparar uma solução de M9 suplementado (M9 +) com as seguintes concentrações finais: 0,20 mg / ml de IPTG, 8,0 ug / ml de colesterol, 50 ug / ml de ampicilina, 9,6 ug / ml de tetraciclina, 0,0835 ug / ml de Fungizone, e 15 vermes por 50 ul.
- Faça soluções separadas para transgênica domínio prion e vermes WT. O volume final da solução verme M9 + necessária irá depender do número de placas de ARNi copiados (ver abaixo), além de ~ 30 ml de volume morto que vai permanecer no reservatório, se um distribuidor automático é usado.
- Após a indução de 3 horas de produção dsRNA bacteriana, tirar as placas fora da incubadora e deixe-os arrefecer à RT (~ 30 min) para evitar calor perturbar os animais.
- Dispensar 50 ul de domínio de prião solução verme transgénico M9 + para cada poço das placas de ARNi e uma das placas de controlo do vector vazio. FazerCertifique-se de misturar a solução verme antes de cada passo como os animais tendem a sedimentar para o fundo do reservatório.
- Dispensar vermes WT para a segunda placa de controle. Deixar as placas não vedadas para permitir o arejamento. Para manter a cultura líquido evapore, empilhe 4-5 placas juntos e enrole com uma toalha de papel úmido e papel alumínio. Incubar a 200 rpm a 20 ° C durante 4 dias.
- Scoring
NOTA: Após 4 dias na incubadora, os vermes estará no segundo dia de vida adulta e estão prontos para o rastreio. Deixe os animais se adaptar às condições não agitam durante 30 minutos antes de triagem para garantir surra sem serem incomodados.- Usando uma câmara Falcon 4M60 ligado a um computador com um monitor, para o aumento da tela visualmente debulha, em comparação com o domínio de prião animais transgénicos de controlo tratados.
- Compilar uma lista de visitas preliminares para confirmar usando wrMTrck (veja a próxima seção).
4. Confirmação da PreliminarBatida de tela
- Ensaio de motilidade em placas sólidas
- Preparar placas com NGM suplementadas com 100 ug / ml de ampicilina, 12,5 ug / ml de tetraciclina e 1 mM de IPTG, de acordo com métodos padrão 47. Se possível, use uma máquina de despejar placa para assegurar todas as placas têm a mesma altura dos meios de comunicação, o que permitirá um processo de aquisição de vídeo mais simplificado.
- Crescer as diferentes clones de ARNi em ~ 1 ml de LB + 50 ug / ml de ampicilina, O / N a 37 ° C e 250 rpm.
- No dia seguinte, induzir a expressão do ARNcd com IPTG 1 mM durante 3 h. Semente as placas com 150 mL de cada clone bacteriano RNAi, distribuídos em uma camada fina. Deixe as bactérias secas durante 2 dias à TA no escuro. Prepare 3 placas por RNAi clone.
- Sincronize a população sem-fim por branqueamento de acordo com métodos padronizados 50 (ver secção 1.2), e deixe os ovos eclodem O / N em M9 mídia.
- Tomar uma amostra da suspensão verme e determinar a quantidade de NEMatodes por ul no microscópio estereoscópico. Em seguida, pipetar o volume adequado de meio M9 mais vermes em cada placa experimental de modo que contenha 25 - 30 vermes L1. Crescer as nemátodos a 20 ° C durante 4 dias até que os vermes atingir dia 2 da fase adulta.
- A análise quantitativa da motilidade verme com o plugin wrMTrck para ImageJ
NOTA: Os vídeos foram gravados usando um microscópio estereoscópico com aumento de 10x com um Hamamatsu Orca-R2 câmera digital C10600-10B eo software PCI imaging Simples Hamamatsu.- Ligue a câmera eo software PCI imagem simples. Clique em "Live" para permitir o ajuste das condições de imagem.
- Configurar as condições de vídeo como se segue: Ganho = 0; Modo Luz = alta; Índice de Velocidade = 1; Binning = 2. Clique em "Auto Exposé" e, em seguida, ajustar as condições de iluminação, movendo-se os espelhos microscópio e do brilho e contraste mostradores.
NOTA: O vídeo precisa ter um alto contraste sem ser OVEREXPosed, de modo que os animais aparecem como formas pretas em um fundo brilhante. - Clique em "Scan Time" e escolha uma pasta e um nome de arquivo. Defina "Campo Delay" para 20 ms e "Pare no Time" a 30 seg. Pressione "Review Live", a fim de escolher a área da chapa para gravar (onde a maioria dos worms são). Toque na placa 3 ou 4 vezes no palco, rapidamente confirmar a sua posição no campo de visão e pressione "Start".
- Depois que o filme termina de gravar, clique direito sobre a imagem e escolha "Exportar Montage Sequence" para exportar o filme a partir de um formato .cxd a um .avi.
- Analisando os vídeos de motilidade
- Abra o software ImageJ, vá para a aba "Plugins" e depois em "wrmtrck" e selecione "Batch wrMTrck". Selecione o diretório que contém todos os arquivos a serem analisados.
- Na janela de entrada principal do wrMTrck_Batch, carregar os valores de entrada, conforme detalhado na Figure 5C. Explicação para cada um dos parâmetros podem ser encontradas no manual de instruções que acompanham o plugin. Clique em "OK" e deixar a análise do movimento de execução.
- Para a curadoria os resultados e confirmar que todas as faixas detectadas são de real C. elegans e eliminar artefatos, abra cada um dos arquivos .txt criados para cada filme e copiar as informações em um arquivo de software de análise de dados. Abra o "* _labels.zip" arquivos criados e executar o que resulta "* _labels.tif" para verificar e eliminar faixas vermes falsos manualmente.
Resultados
Monitoramento intercelular difusão de proteínas príon-like in vivo por time-lapse imaging
Transgênicos C. linhas elegans que expressam o domínio prião são particularmente bem adaptados para a análise de certos aspectos de proteínas priões do tipo, por exemplo, transmissão de célula para célula e a toxicidade celular não autónomo. A transparência dos animais permite o acompanhamento de fluorescente etiquetado proteínas de d...
Discussão
Os métodos descritos aqui ajudam a ilustrar espalhando e celular toxicidade autônoma da célula complexo autônomo e não de proteínas príon-like. Descobrimos recentemente que um domínio de prion cytosolic agregação propensas é retomado em vesículas ligadas à membrana em um processo de autofagia relacionados. Um subconjunto específico dessas vesículas transporta o domínio de prião dentro e entre as células e tecidos 40. A chave para controlar o seu movimento no animal vivo é que a proteína te...
Divulgações
The authors declare no competing financial interests.
Agradecimentos
We thank Cindy Voisine and Yoko Shibata for helpful discussion and critical comments on the manuscript. We acknowledge the High Throughput Analysis Laboratory (HTAL) and the Biological Imaging Facility (BIF) at Northwestern University for their assistance. This work was funded by grants from the National Institutes of Health (NIGMS, NIA, NINDS), the Ellison Medical Foundation, and the Daniel F. and Ada L. Rice Foundation (to R.I.M.). C.I.N.-K. was supported by the Deutsche Forschungsgemeinschaft (KR 3726/1-1).
Materiais
| Name | Company | Catalog Number | Comments |
| Reagent | |||
| Nanosphere size standards 100 nm | ThermoScientific | 3100A | |
| Levamisole | Sigma | L-9756 | |
| IPTG | Sigma | 15502-10G | |
| Ahringer RNAi library | Source BioScience LifeSciences | http://www.lifesciences.sourcebioscience .com/clone-products/non-mammalian/c-elegans/c-elegans-rnai-library/ | |
| Equipment | |||
| Sorvall Legend XTR Refrigerated Centrifuge, 120VAC | ThermoScientific | 75004521 | http://www.coleparmer.com/Product/Thermo_Scientific_Sorvall_Legend_ XTR_Refrigerated_Centrifuge_120 VAC/EW-17707-60 |
| 96 pin replicator | Scionomix | http://www.scinomix.com/all-products/96-pin-replicator/ | |
| HiGro high-capacity, incubating shaker | Digilab | http://www.digilabglobal.com/higro | |
| Multidrop Combi Reagent Dispenser | Titertrek | http://groups.molbiosci.northwestern.edu/hta/titertek.htm | |
| Biomek FX AP96 Automated Workstation | Beckman Coulter | http://groups.molbiosci.northwestern.edu/hta/biomek_multi.htm | |
| Innova44 shaker | New Brunswick | http://www.eppendorf.com/int///index.php?sitemap=2.3&pb=d78efbc05310ec 04&action=products&contentid=1& catalognode=83389 | |
| M205 FA | Leica | http://www.leica-microsystems.com/de/produkte/stereomikroskope-makroskope/fluoreszenz/details/product/leica-m205-fa/ | |
| ORCA-R2 C10600-10BDigital CCD camera | Hamamatsu | http://www.hamamatsu.com/jp/en/community/life_science_camera/product/search/C10600-10B/index.html | |
| Spinning Disc AF Confocal Microscope | Leica | http://www.leica-microsystems.com/products/light-microscopes/life-science-research/fluorescence-microscopes/details/product/leica-sd-af/ | |
| Falcon 4M60 camera | Teledyne Dalsa | http://www.teledynedalsa.com/imaging/products/cameras/area-scan/falcon/PT-41-04M60/ | |
| Software | |||
| MetaMorph Microscopy Automation & Image Analysis Software | Molecular Devices | http://www.moleculardevices.com/products/software/meta-imaging-series/metamorph.html | |
| Hamamatsu SimplePCI Image Analysis Software | Meyer Instruments | http://meyerinst.com/imaging-software/hamamatsu/index.htm | |
| ImageJ | NIH | http://rsbweb.nih.gov/ij/download.html | |
| wrMTrck plugin for ImageJ | http://www.phage.dk/plugins/wrmtrck.html | ||
| C. elegans strains | |||
| N2 (WT) | Caenorhabditis Genetics Center (CGC) | http://www.cgc.cbs.umn.edu/strain.php?id=10570 | |
| AM815 rmIs323[myo-3p::sup35(r2e2)::rfp] | Morimoto lab | available from our laboratory | |
| See table 1 for a source for folding sensor and stress reporter strains |
Referências
- Prusiner, S. B. Novel proteinaceous infectious particles cause scrapie. Science. 216 (4542), 136-144 (1982).
- Jarrett, J. T., Lansbury, P. T. Seeding 'one-dimensional crystallization' of amyloid: a pathogenic mechanism in Alzheimer's disease and scrapie. Cell. 73 (6), 1055-1058 (1993).
- Caughey, B., Kocisko, D. A., Raymond, G. J., Lansbury, P. T. Aggregates of scrapie-associated prion protein induce the cell-free conversion of protease-sensitive prion protein to the protease-resistant state. Chem Biol. 2 (12), 807-817 (1995).
- Wickner, R. B. [URE3] as an altered URE2 protein: evidence for a prion analog in Saccharomyces cerevisiae. Science. 264 (5158), 566-569 (1994).
- Chien, P., Weissman, J. S., DePace, A. H. Emerging principles of conformation-based prion inheritance. Annu Rev Biochem. 73, 617-656 (2004).
- Kimberlin, R. H., Walker, C. A. Pathogenesis of mouse scrapie: patterns of agent replication in different parts of the CNS following intraperitoneal infection. J R Soc Med. 75 (8), 618-624 (1982).
- Beekes, M., McBride, P. A., Baldauf, E. Cerebral targeting indicates vagal spread of infection in hamsters fed with scrapie. J Gen Virol. 79 (3), 601-607 (1998).
- Jucker, M., Walker, L. C. Self-propagation of pathogenic protein aggregates in neurodegenerative diseases. Nature. 501 (7465), 45-51 (2013).
- Aguzzi, A. Cell biology: Beyond the prion principle. Nature. 459 (7249), 924-925 (2009).
- Scherzinger, E., et al. Self-assembly of polyglutamine-containing huntingtin fragments into amyloid-like fibrils: implications for Huntington's disease pathology. Proc Natl Acad Sci U S A. 96 (8), 4604-4609 (1999).
- Wood, S. J., et al. alpha-synuclein fibrillogenesis is nucleation-dependent. Implications for the pathogenesis of Parkinson's disease. J Biol Chem. 274 (28), 19509-19512 (1999).
- Wang, Y. Q., et al. Relationship between prion propensity and the rates of individual molecular steps of fibril assembly. J Biol Chem. 286 (14), 12101-12107 (2011).
- Cushman, M., Johnson, B. S., King, O. D., Gitler, A. D., Shorter, J. Prion-like disorders: blurring the divide between transmissibility and infectivity. J Cell Sci. 123 (8), 1191-1201 (2010).
- Tanaka, M., Collins, S. R., Toyama, B. H., Weissman, J. S. The physical basis of how prion conformations determine strain phenotypes. Nature. 442 (7102), 585-589 (2006).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. J Struct Biol. 179 (2), 152-160 (2012).
- Ilieva, H., Polymenidou, M., Cleveland, D. W. Non-cell autonomous toxicity in neurodegenerative disorders: ALS and beyond. J Cell Biol. 187 (6), 761-772 (2009).
- Nussbaum-Krammer, C. I., Morimoto, R. I. Caenorhabditis elegans as a model system for studying non-cell-autonomous mechanisms in protein-misfolding diseases. Dis Model Mech. 7 (1), 31-39 (2014).
- Lino, M. M., Schneider, C., Caroni, P. Accumulation of SOD1 mutants in postnatal motoneurons does not cause motoneuron pathology or motoneuron disease. J Neurosci. 22 (12), 4825-4832 (2002).
- Li, J. Y., et al. Lewy bodies in grafted neurons in subjects with Parkinson's disease suggest host-to-graft disease propagation. Nat Med. 14 (5), 501-503 (2008).
- Desplats, P., et al. Inclusion formation and neuronal cell death through neuron-to-neuron transmission of alpha-synuclein. Proc Natl Acad Sci U S A. 106 (31), 13010-13015 (2009).
- Clement, A. M., et al. Wild-type nonneuronal cells extend survival of SOD1 mutant motor neurons in ALS mice. Science. 302 (5642), 113-117 (2003).
- Gu, X., et al. Pathological cell-cell interactions elicited by a neuropathogenic form of mutant Huntingtin contribute to cortical pathogenesis in HD mice. Neuron. 46 (3), 433-444 (2005).
- Yamanaka, K., et al. Mutant SOD1 in cell types other than motor neurons and oligodendrocytes accelerates onset of disease in ALS mice. Proc Natl Acad Sci U S A. 105 (21), 7594-7599 (2008).
- Garden, G. A., et al. Polyglutamine-expanded ataxin-7 promotes non-cell-autonomous purkinje cell degeneration and displays proteolytic cleavage in ataxic transgenic mice. J Neurosci. 22 (12), 4897-4905 (2002).
- Raeber, A. J., et al. Astrocyte-specific expression of hamster prion protein (PrP) renders PrP knockout mice susceptible to hamster scrapie. EMBO J. 16 (20), 6057-6065 (1997).
- Yazawa, I., et al. Mouse model of multiple system atrophy alpha-synuclein expression in oligodendrocytes causes glial and neuronal degeneration. Neuron. 45 (6), 847-859 (2005).
- Lobsiger, C. S., Cleveland, D. W. Glial cells as intrinsic components of non-cell-autonomous neurodegenerative disease. Nat Neurosci. 10 (11), 1355-1360 (2007).
- Sambataro, F., Pennuto, M. Cell-autonomous and non-cell-autonomous toxicity in polyglutamine diseases. Prog Neurobiol. 97 (2), 152-172 (2012).
- Polymenidou, M., Cleveland, D. W. Prion-like spread of protein aggregates in neurodegeneration. J Exp Med. 209 (5), 889-893 (2012).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nat Rev Mol Cell Biol. 11 (4), 301-307 (2010).
- Braak, H., Braak, E., Bohl, J. Staging of Alzheimer-related cortical destruction. Eur Neurol. 33 (6), 403-408 (1993).
- Meyer-Luehmann, M., et al. Exogenous induction of cerebral beta-amyloidogenesis is governed by agent and host. Science. 313 (5794), 1781-1784 (2006).
- Luk, K. C., et al. Pathological alpha-synuclein transmission initiates Parkinson-like neurodegeneration in nontransgenic mice. Science. 338 (6109), 949-953 (2012).
- Clavaguera, F., et al. Transmission and spreading of tauopathy in transgenic mouse brain. Nat Cell Biol. 11 (7), 909-913 (2009).
- Nonaka, T., et al. Prion-like Properties of Pathological TDP-43 Aggregates from Diseased Brains. Cell Rep. 4 (1), 124-134 (2013).
- Lundmark, K., et al. Transmissibility of systemic amyloidosis by a prion-like mechanism. Proc Natl Acad Sci U S A. 99 (10), 6979-6984 (2002).
- Lai, C. H., Chou, C. Y., Ch'ang, L. Y., Liu, C. S., Lin, W. Identification of novel human genes evolutionarily conserved in Caenorhabditis elegans by comparative proteomics. Genome Res. 10 (5), 703-713 (2000).
- Xu, X., Kim, S. K. The early bird catches the worm: new technologies for the Caenorhabditis elegans toolkit. Nat Rev Genet. 12 (11), 793-801 (2011).
- Boulin, T., Hobert, O. From genes to function: the C. elegans genetic toolbox. Wiley Interdiscip Rev Dev Biol. 1 (1), 114-137 (2012).
- Nussbaum-Krammer, C. I., Park, K. W., Li, L., Melki, R., Morimoto, R. I. Spreading of a prion domain from cell-to-cell by vesicular transport in Caenorhabditis elegans. PLoS Genet. 9 (3), e1003351 (2013).
- Chernoff, Y. O., Lindquist, S. L., Ono, B., Inge-Vechtomov, S. G., Liebman, S. W. Role of the chaperone protein Hsp104 in propagation of the yeast prion-like factor [psi. Science. 268 (5212), 880-884 (1995).
- Liu, J. J., Lindquist, S. Oligopeptide-repeat expansions modulate 'protein-only' inheritance in yeast. Nature. 400 (6744), 573-576 (1999).
- Halfmann, R., et al. Prions are a common mechanism for phenotypic inheritance in wild yeasts. Nature. 482 (7385), 363-368 (2012).
- Tyedmers, J., Madariaga, M. L., Lindquist, S. Prion switching in response to environmental stress. PLoS Biol. 6 (11), e294 (2008).
- Krammer, C., et al. The yeast Sup35NM domain propagates as a prion in mammalian cells. Proc Natl Acad Sci U S A. 106 (2), 462-467 (2009).
- Hofmann, J. P., et al. Cell-to-cell propagation of infectious cytosolic protein aggregates. Proc Natl Acad Sci U S A. 110 (15), 5951-5956 (2013).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , (2006).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C. elegans Using Microinjection. J. Vis. Exp. (18), e833 (2008).
- Evans, T. C. Transformation and microinjection. WormBook. , (2006).
- Shaham, S. Methods in cell biology. Wormbooks. , (2006).
- Kim, E., Sun, L., Gabel, C. V., Fang-Yen, C. Long-term imaging of Caenorhabditis elegans using nanoparticle-mediated immobilization). PLoS One. 8 (1), e53419 (2013).
- Fay, D. Genetic mapping and manipulation: Chapter 1-Introduction and basics. WormBook. , (2006).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Rual, J. F., et al. Toward improving Caenorhabditis elegans phenome mapping with an ORFeome-based RNAi library. Genome Res. 14 (10B), 2162-2168 (2004).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Kern, A., Ackermann, B., Clement, A. M., Duerk, H., Behl, C. HSF1-controlled and age-associated chaperone capacity in neurons and muscle cells of C. elegans. PLoS One. 5 (1), e8568 (2010).
- Becker, J., Walter, W., Yan, W., Craig, E. A. Functional interaction of cytosolic hsp70 and a DnaJ-related protein, Ydj1p, in protein translocation in vivo. Mol Cell Biol. 16 (8), 4378-4386 (1996).
- Salvaterra, P. M., McCaman, R. E. Choline acetyltransferase and acetylcholine levels in Drosophila melanogaster: a study using two temperature-sensitive mutants. J Neurosci. 5 (4), 903-910 (1985).
- Goloubinoff, P., Mogk, A., Zvi, A. P., Tomoyasu, T., Bukau, B. Sequential mechanism of solubilization and refolding of stable protein aggregates by a bichaperone network. Proc Natl Acad Sci U S A. 96 (24), 13732-13737 (1999).
- Schroder, H., Langer, T., Hartl, F. U., Bukau, B. D. n. a. K. DnaJ and GrpE form a cellular chaperone machinery capable of repairing heat-induced protein damage. EMBO J. 12 (11), 4137-4144 (1993).
- Rampelt, H., et al. Metazoan Hsp70 machines use Hsp110 to power protein disaggregation. EMBO J. 31 (21), 4221-4235 (2012).
- Gupta, R., et al. Firefly luciferase mutants as sensors of proteome stress. Nat Methods. 8 (10), 879-884 (2011).
- Gidalevitz, T., Ben-Zvi, A., Ho, K. H., Brignull, H. R., Morimoto, R. I. Progressive disruption of cellular protein folding in models of polyglutamine diseases. Science. 311 (5766), 1471-1474 (2006).
- Ben-Zvi, A., Miller, E. A., Morimoto, R. I. Collapse of proteostasis represents an early molecular event in Caenorhabditis elegans aging. Proc Natl Acad Sci U S A. 106 (35), 14914-14919 (2009).
- Karady, I., et al. Using Caenorhabditis elegans as a model system to study protein homeostasis in a multicellular organism. J Vis Exp. (82), e50840 (2013).
- Gidalevitz, T., Krupinski, T., Garcia, S., Morimoto, R. I. Destabilizing protein polymorphisms in the genetic background direct phenotypic expression of mutant SOD1 toxicity. PLoS Genet. 5 (3), e1000399 (2009).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proc Natl Acad Sci U S A. 99 (16), 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine proteins at the pathogenic threshold display neuron-specific aggregation in a pan-neuronal Caenorhabditis elegans model. J Neurosci. 26 (29), 7597-7606 (2006).
- Mohri-Shiomi, A., Garsin, D. A. Insulin signaling and the heat shock response modulate protein homeostasis in the Caenorhabditis elegans intestine during infection. J Biol Chem. 283 (1), 194-201 (2008).
- Libina, N., Berman, J. R., Kenyon, C. Tissue-specific activities of C. elegans DAF-16 in the regulation of lifespan. Cell. 115 (4), 489-502 (2003).
- Schatzl, H. M., et al. A hypothalamic neuronal cell line persistently infected with scrapie prions exhibits apoptosis. J Virol. 71 (11), 8821-8831 (1997).
- Keith, S. A., Amrit, F. R., Ratnappan, R., Ghazi, A. The C. elegans healthspan and stress-resistance assay toolkit. Methods. , (2014).
- Pierce-Shimomura, J. T., et al. Genetic analysis of crawling and swimming locomotory patterns in C. elegans. Proc Natl Acad Sci U S A. 105 (52), 20982-20987 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados