このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
拡散とプリオン様タンパク質の毒性は後生動物モデル生物を用いて調査
要約
Prion-like propagation of protein aggregates has recently emerged as being implicated in many neurodegenerative diseases. The goal of this protocol is to describe, how to use the nematode C. elegans as a model system to monitor protein spreading and to investigate prion-like phenomena.
要約
Prions are unconventional self-propagating proteinaceous particles, devoid of any coding nucleic acid. These proteinaceous seeds serve as templates for the conversion and replication of their benign cellular isoform. Accumulating evidence suggests that many protein aggregates can act as self-propagating templates and corrupt the folding of cognate proteins. Although aggregates can be functional under certain circumstances, this process often leads to the disruption of the cellular protein homeostasis (proteostasis), eventually leading to devastating diseases such as Alzheimer’s disease (AD), Parkinson’s disease (PD), Amyotrophic lateral sclerosis (ALS), or transmissible spongiform encephalopathies (TSEs). The exact mechanisms of prion propagation and cell-to-cell spreading of protein aggregates are still subjects of intense investigation. To further this knowledge, recently a new metazoan model in Caenorhabditis elegans, for expression of the prion domain of the cytosolic yeast prion protein Sup35 has been established. This prion model offers several advantages, as it allows direct monitoring of the fluorescently tagged prion domain in living animals and ease of genetic approaches. Described here are methods to study prion-like behavior of protein aggregates and to identify modifiers of prion-induced toxicity using C. elegans.
概要
アルツハイマー病(AD)、パーキンソン病(PD)、筋萎縮性側索硬化症(ALS)、および伝染性海綿状脳症(TSE)を含む多くの神経変性疾患は、凝集しやすいタンパク質と会合している、したがって、集合的にタンパク質の誤った折りたたみに起因する疾患(PMDのとして知られている)。それらはヒトおよび動物の両方1に感染することができるという点でのTSEまたはプリオン病は、PMDののユニークなクラスを構成する。分子レベルでは、プリオンは募集および病理学的βシートが豊富なPrP Scの高次構造2,3に単量体のαヘリックスが豊富なホストでエンコードされた細胞のプリオン(PRP C)を変換することによって複製する。自己増殖タンパク質凝集体は、哺乳動物のプリオン4,5との重要な特徴を共有する真菌、で同定されている。さらに、哺乳類プリオンは細胞間の移動が可能であり、ナイーブ細胞6,7-感染する。
のPMD OTHながらERのTSEが感染性はないよりも、彼らは、プリオン病8,9と共通の病原性の原則を共有しています。のPMDのそれぞれにリンクされたタンパク質は、構造または機能に関連していないが、これらは結晶化のようなプロセスを経由してすべてのフォームの凝集体は核と呼ばれるまたは重合を播種。さらに、タンパク質性種子はそれらの可溶性アイソ2,10,11を補充することによって成長する。自己増殖に効率が一緒にそのような分子シャペロンなどの追加の細胞因子と最終的に集計核、播種、断片化および12〜15の拡散率を決定するタンパク質の固有の特性に応じて、 生体内で変化する。したがって、タンパク質凝集を効率的に伝播することができ、これらの要素の間で微妙なバランスが存在しなければならない。唯一のいくつかのアミロイド形成凝集体はプリオンの特性を保有するため、すべてのPMDが感染性ではない理由も説明するかもしれない。プリオンは、「トップパフォーマー 'Oを表しているようだそれらのPMD 8,13を研究するための強力なツールになり自己複製タンパク性凝集体のFA広いスペクトル、。
興味深いことに、疾患関連凝集に関連する毒性は、多くの場合、非細胞自律的な構成要素16,17を持っています。これは、厳密にのみ細胞が遺伝子を示すの特定の表現型を発現することを意味している自律的な効果を、細胞とは対照的に、対応する遺伝子を発現しない隣接細胞に影響を与えることを意味する。これは説得力組織特異的発現によって実証または神経変性疾患18-26の多数のモデルにおけるそれぞれのタンパク質のノックダウンした。様々なメカニズムが減少した栄養供給、ニューロンのシグナル伝達における不均衡、グルタミン酸興奮毒性、および神経炎症16,27,28を含むのPMDで、この非細胞自律的毒性、の基礎として提案されている。また、セル間の疾患関連プリオン凝集体のような動きは、mighTこの側面29,30に貢献する。増加する証拠は、プリオン以外のタンパク質含有物特性は、多くのPMD 30-36で観察病理の広がりを説明し得る、細胞間で伝達することができることを示唆している。しかし、まだ疾患タンパク質の細胞間移行及び隣接セルに対する毒性効果の間に明確な因果関係が存在するか否かを決定されなければならない。したがって、細胞間の伝送と非細胞自律的な毒性の根底にある細胞経路のより良い理解は、新規治療薬の開発のために必要不可欠である。しかし、後生動物において、ミスフォールドタンパク質の細胞から細胞への伝達に影響を広げ、細胞因子プリオン様の多くの側面はよく生物レベルで、特に理解されていない。
線虫(Caenorhabditis elegans)線虫は、電位を提供するいくつかの利点を有する プリオン様spreadiの新しい面を発見後生17 ngの。これは、in vivoで生体内の蛍光タグ化タンパク質の追跡を可能にする、透明である。さらに、病気の影響を受け、多くの細胞および生理学的プロセスは、人間のワームから保存されており、C.されている虫も遺伝子操作および分子および生化学的解析37-39幅広い種類のに適している。正確には959体細胞はまだ筋肉、神経細胞および腸を含む、いくつかの異なる組織タイプがあり、シンプルなボディプランに大人の雌雄同体を構成している。
C.の新しいプリオンモデルを確立するために、 虫は、我々はワーム4,40には知られている内因性プリオンタンパク質が存在しないので、外因的に、よく特徴付けグルタミン/アスパラギン(Q / N)細胞質の酵母プリオンタンパク質Sup35のリッチプリオンドメインNMを表現することにしました。酵母プリオンはプリオン複製41-44の基本的なメカニズムの解明に貴重だった。さらに、NMはモミです哺乳動物細胞培養45,46におけるプリオンの完全なライフサイクルを再現することが示されている第サイトゾルプリオン様タンパク質。同様に、Cで発現した場合虫は 、Sup35プリオンドメインは、酵母細胞と比較して、後生動物細胞内での増殖のためのさまざまな要件に非常によく採用され、プリオン生物学40の主要な機能を示した。NM凝集は、ミトコンドリアの完全性の破壊との外観を含め、深遠な有毒な表現型と関連していた細胞レベルで、様々なオートファジー関連小胞、ならびに胚および幼虫停止、発達遅延、および生物レベルでのタンパク質の折り畳み環境の広範な乱れ。驚くべきことに、プリオンドメインは、導入遺伝子が発現していないもので、隣接組織に影響を与え、細胞自律的かつ非細胞自律的な毒性を示す。さらに、内および細胞間のプリオンドメインの小胞輸送をリアルタイムに監視されるの生体内40で。
ここでは、Cのプリオンのような普及を検査する方法を説明します虫 。私たちは、タイムラプス蛍光顕微鏡を用いてプリオンドメインを含む小胞の細胞内および細胞間輸送を監視する方法について説明します。私たちは、組織特異的な折り畳み式センサの使用を強調し、普遍的に細胞のフィットネス上の細胞自律的かつ非細胞自律的な効果を評価するためのストレスレポーターを表現します。最後に、プリオン誘発毒性の新たな変性剤を同定するために最近行われたゲノムワイドRNA干渉(RNAi)は、画面の手順を説明する。組み合わせて、これらの方法は、タンパク質の細胞間移行及びそれらの非細胞自律的な毒性に関与する遺伝的経路を離れていじめるのを助けることができる。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1.モニタリング経細胞は、in vivoタイムラプスイメージングにより、プリオン様タンパク質の広がり
注:C.を育てる注意深くエレガンス野生型(WT)(N2)とトランスジェニック系統の標準的な方法に従ってとは、培養温度47を制御する。
- C.のトランスジェニック系統を生成しますエレガンス単量体赤色蛍光タンパク質(mRFPと)でタグ付けされたプリオン様タンパク質を発現する。マイクロインジェクション48を使用する方法を示し、このビデオを見る。これらの染色体外の行を統合する方法を説明し、さらに詳細及び方法については、49を参照してください。
- 卵は、標準的な方法50に従って敷設又は漂白によって同期集団を準備します。
- 産卵による同期
- 転送10から20妊娠プレート上の大人とそれらの1のために卵を産むましょう - 2時間。プレートから大人を削除し、子孫が希望歳までに成長しましょう。
<漂白によるLI>同期 - 妊娠大人の非同期の人口を収集し、アルカリ性の次亜塩素酸ナトリウム溶液(250 mMのNaOHおよび1:H 2 O中の市販の漂白剤の4(v / v)で希釈)でそれらを漂白。二回(1分間218×gで)M9バッファ47(21 MMNA 2 HPO 4·7H 2 O、22 mMKH 2 PO 4、86 mMNaCl、1 mMMgSO 4·7H 2 O、1までのdH 2 Oを追加すると、卵を洗うL)。
- 彼らは20℃で穏やかに撹拌し、O / NでM9バッファーに孵化することを許可する。ワームの開発が同期集団を残し、食料源の非存在下でL1段階で阻止するであろう。 OP50 E.を播種し、新鮮な線虫の成長メディア(NGM)プレート上になL1を転送大腸菌とは、子孫が希望する47歳まで開発してみましょう。
- 産卵による同期
- 50を説明するように顕微鏡スライド上に(H 2 O)2%アガロースパッドを準備します。
- 2マイルを準備その全長にわたって配置された表示用テープとcroscopeスライドをスペーサーとして使用する。それらの間の第三の顕微鏡スライドを置きます。
- H 2 Oおよびクリーンスライド上にピペット1滴の2%アガロースを溶解する。
- 寒天ドロップの上に他の3つのスライドに垂直な第四のスライドを置きます。静かにラベルテープと同じ厚さにパッドを平らにするためにそれを押し下げます。
- スペーサを取り外し、静かに離れてスライドを引いて前に1分間、それが乾燥してみましょう。寒天パッドは、それらのいずれかに固執します。
- 白金線のピックを使って、パッドと転送〜10匹の動物にピペット〜10μlの麻酔薬(M9バッファーで2 mMのレバミゾール)。カバースリップ(〜22×22 mm)ので覆い、1時間以内に画像を撮影する。
- あるいは、より長い期間にわたって動画を取得するか、さらに、動物の任意の動きの可能性を低減するために、麻酔薬およびビーズ固定化51の組み合わせを使用。
- 10%のAGAを準備(M9バッファ内の)51を説明し、3μlのナノスフェアサイズ標準溶液へのワーム(ポリスチレンビーズ、100nm)を加えた3μlの麻酔薬(M9バッファーで4 mMのレバミゾール)を追加すると、パッドの増加となりました。カバースリップで優しくカバー。乾燥を避けるために、VALAP(ワセリン、ラノリン、パラフィンワックスの等量混合物)を用いてカバースリップをシールする。
- 画像は、共焦点顕微鏡を使用してワームを固定化した。
注:結果は、EM-CCDカメラとのMetaMorphなどの顕微鏡オートメーションおよび画像解析ソフトウェアを備えたスピニングディスクAF共焦点顕微鏡を用いて得られ、以下の仕様の概要を説明するが、他の同等の共焦点撮像システムも使用することができるされている。- 63Xまたは100X / 1.4NA油浸対物を使用して、顕微鏡スライドホルダーにワームを含む顕微鏡スライドを配置。
- ソフトウェアを開きます。 mRFPとイメージング用のレーザパワーとフィルタを調整します。 10%の電力とエミッションフィルタ> 600 nmで561 nmのレーザーを使用してください。
- 開く "多次元取得」。 「メイン」タブで、「微速度撮影」と「ファイル名を指定して実行ジャーナル」(:オフハードウェアオートフォーカス)のチェックボックスをオン。
- 「保存」タブで選択したり、ファイルを保存するディレクトリのフォルダを作成します。ファイルに名前を割り当てます。
- 「ウェーブ長」タブの下で適切な照明を選択して、露出とゲインを調整します。 (ライブ画像を用いて評価)個々のサンプルに応じて、100と300ミリ秒と100と300の間で、カメラのゲインと露出で「YokoQuadレッド」(またはmRFPを撮像のための同等の照明設定)を使用します。
- 「微速度撮影」タブで「時間ポイント数」= 301、 "時間" = 5分および「時間/間隔」= 1秒を選択します。
- 「AFCのSET ZのHOLD」と「AFCリターンのZ HOLDに」、種類::& "ジャーナル」タブでジャーナルを選択#8220;スペシャル」(2回)、初期点:「スタート時点」と「時点の終わり」。このオートフォーカスオプションは、画像より長期間にわたって同じセクションに重要です。
- セットアップが終了したら、Enterキーを押して「取得」。タイムラプスビデオは、独立したTIFFファイルとしてリコールセーフされている。
- ImageJの中で与えられた時系列のすべて.TIFFファイルを開きます。 「イメージ」→「スタック」→「スタックへのイメージ」に進みます。必要に応じて、→「ファイル」の下にムービーを書き出すなど、サイズバーを追加、明るさとコントラストを調整→「AVI ...」「名前を付けて保存」(例えば、ビデオ1と2は図1を対応する参照)。
2。細胞自律と非細胞自律を調査するために折りたたみセンサーおよびストレスレポーターを使用すると、タンパク質恒常性と毒性に影響
- 折りたたみセンサやストレスreporteを同時発現するトランスジェニック系統を生成しますプリオン様タンパク質と一緒にrを。十字架を確立するか、トランスジェニック系統を生成する方法の方法については、48,49,52を参照してください。 C.のリストについては、表1を参照してください使用することができる。エレガンス株 。
- トランスジェニック動物の同期化された集団を準備し、(セクション1.2)50上記のように希望歳までに成長します。
- 実体顕微鏡を用いて、折り畳み式センサやストレスレポーターのそれぞれの表現型を調べる。
- 折り畳みセンサーは、同期後に各日の凝集体を保有する動物の数を決定する(例えば、 図2EおよびF参照)。
- ストレスレポーターのために、テストレポーターの増加した蛍光のプリオン様タンパク質の共発現の結果(例えば、 図3を参照)場合。
C.におけるプリオン誘導毒性の抑制3.ゲノムワイドスクリーンエレガンス
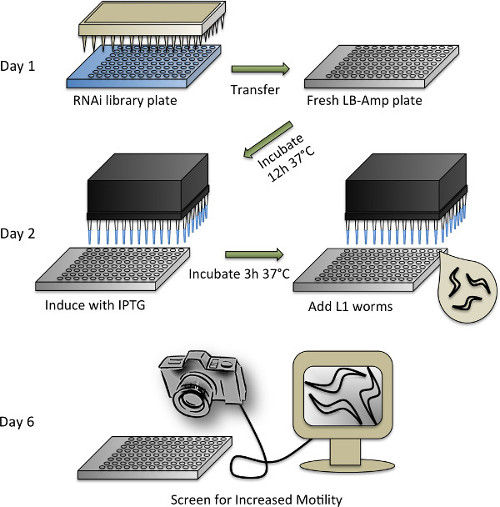
のRNAiスクリーニングプロトコルの4略図を図。個々のステップの詳細については、プロトコルセクション3を参照してください。
- C.の同期虫ワームおよびRNAiライブラリの重複
- RNAiスクリーニングのために、AhringerのRNAiライブラリ(またはビダルのRNAiライブラリ)53,54を使用しています。 10%グリセロールを補った100μlのLB-アンプメディアとプレート(LB中の50μg/ mlのアンピシリン)を充填することにより、96ウェルプレートにオリジナルの384ウェルフォーマットからのRNAiライブラリをRearrayと96ピンレプリケーターを使用して接種する。 -80℃で攪拌し、店舗で37℃CO / Nで成長する。
- C.を維持虫 WT及び20℃のNGMプレート上でのトランスジェニック系統をOP50 E.を播種標準的な方法47に従って大腸菌 。
- 漂白によってプリオンドメイントランスジェニックおよびWT(コントロール)線虫を同期する(セクション1.2を参照)。
- ワームが漂白されていることを同じ日に、50μg/ mlのアンピシリンを補充したLBメディアを準備。分配するために、自動試薬ディスペンサーを使用する(またはピペット手動で)、96ウェルプレートの各ウェルにLB-アンプ培地65μlの。
- -80℃から96ウェルAhringerのRNAiライブラリプレートを取り外し、ペーパータオルで並んで無菌フードにそれらをもたらす。すぐにプレートがまだ凍結されている間、プレートの外側に氷からの汚染を避けるように注意しながら、上下逆さまにプレートを保持することにより、シールテープを取り除く。プレートを再反転し、それらを約30分間解凍しましょう。
- 1のRNAiライブラリのプレートに1無菌の96ピン·レプリケータを浸し、その後1新鮮なLB-Ampプレートに。各プレートのために新鮮な無菌のレプリケータを使用してください。接着剤箔テープですべてのプレートを密封し、-80℃にライブラリプレートを返す。リプリケータは、漂白剤に浸しすすぐことができ、そして再使用するオートクレーブ処理。
- 複製されたのRNAiプレートを37℃、300rpmに設定したインキュベーター中でO / Nを成長させる。 HT115 Eを使用するには大腸菌対照として空のベクター(L4440)を保有する細菌は、培養チューブに分けて、それを成長した後、2枚の96ウェルプレートの四分の一にマルチチャンネルピ ペットとの文化の65μlのピペット。
- 細菌のdsRNA生産やワームの追加の誘導
- 希釈しイソプロピルβ-D- thiogalactopyranosid(IPTG)のddH 2 Oで5 mMの濃度まで希釈されたIPTG10μlのは、RNAi細菌と対照プレートの各ウェルに(手動またはピペット)分配するマルチチャンネルヘッドを自動化されたワークステーションを使用してください。バックインキュベーターにプレートを再シールして配置します。それらは、37℃で3時間振盪しましょう。
- 細菌が揺れている間、ワームを準備します。ガラス顕微鏡スライドに - (10μL〜5)少量のサンプルをよくM9 /ワームのサスペンションを混ぜ、ピペット。カウント実体顕微鏡下でのワームの数と、1μl当たりのワームの数を計算する。
- 0.20 mg / mlのIPTG、8.0 / mlのコレステロール、50μg/ mlのアンピシリン、9.6 / mlのテトラサイクリン、0.0835 / mlのファンギゾン、および:無菌技術を用いて、以下の最終濃度を補充したM9(M9 +)の溶液を調製する50μLあたり15ワーム。
- プリオンドメイントランスジェニックおよびWTワームのための別々の溶液を作る。必要なM9 +ワーム溶液の最終体積は、RNAiコピープレート(下記参照)に加え〜自動化ディスペンサーが使用される場合、リザーバ内に残るデッドボリュームを30mlの数に依存する。
- 細菌のdsRNA生産の3時間の誘導後、培養器の外板を取り、動物にストレスを熱を避けるために、RT(〜30分)にそれらを冷ます。
- 50μlのM9 +プリオンドメインジェニックワームのRNAiプレートの各ウェルに溶液および空ベクター対照プレートのいずれかを分配する。作る動物は貯水池の底に沈降する傾向があるので、各ステップの前にワームの溶液を混合するようにしてください。
- 第二制御板へのWTワームを分注する。通気を可能にするために開封プレートのままにしておきます。一緒に5皿や湿ったペーパータオルとアルミホイルで包む - 蒸発する液体培養を維持するには、4スタック。 4日間、20℃、200rpmでインキュベートする。
- 得点
注:インキュベーターで4日後、ワームは、成人期の二日目にも、画面の準備ができています。動物が邪魔されずにスラッシングを確実にするためにスクリーニングする前に30分間ノン揺れ条件に合わせて調整しましょう。- 対照処置プリオンドメイントランスジェニック動物と比較して増加したスラッシングを目視でモニターとコンピュータに接続されたファルコン4M60カメラを用いて、スクリーン。
- wrMTrck(次のセクションを参照)を使用して確認するために、予備的ヒットのリストをコンパイルします。
予備の4.確認画面のヒット
- 固体プレート上の運動アッセイ
- 標準的な方法47に従って、アンピシリン100μg/ ml、12.5 / mlのテトラサイクリンおよび1mM IPTGを補ったNGMでプレートを準備します。可能な場合は、より合理映像取得プロセスを可能にするメディアの同じ高さを、持っているすべてのプレートを確実にするために、板注ぐマシンを使用しています。
- 37℃、250 rpmで〜1ミリリットルのLB +アンピシリン50μg/ ml、O / Nで異なるRNAiのクローンを育てる。
- 次の日、3時間、1mMのIPTGでdsRNAの発現を誘導する。薄い層に広げ、それぞれのRNAi細菌クローン150μlでプレートをシード。暗闇の中で室温で2日間乾燥細菌をしてみましょう。 RNAiのクローン当たり3プレートを準備します。
- 標準的な方法50(セクション1.2を参照)に応じて漂白することによりワームの人口を同期して、M9培地中で卵が孵化O / Nを聞かせて。
- ワーム懸濁液のサンプルを取り、NEMの量を決定する実体顕微鏡で1μl当たりatodes。 30のL1ワーム - それは25を含むように続いて、各実験プレートにM9プラスワームの適切な量をピペット。ワームは成人期の2日目に達するまで4日間、20℃で線虫を成長させる。
- ImageJのためのwrMTrckのプラグインとのワームの運動性の定量分析
注:ビデオは浜松オルカ-R2デジタルカメラC10600-10Bと浜松シンプルPCIイメージングソフトウェアと10倍の倍率で立体顕微鏡を用いて記録した。- カメラとシンプルなPCIイメージングソフトウェアをオンにします。画像形成条件の調整を可能にする「ライブ」をクリックする。
- 次のようにビデオ条件を設定します。ゲイン= 0;ハイライトモード=;高速インデックス= 1;ビニング= 2.「自動公開」した後、顕微鏡鏡と明るさとコントラストダイヤルを動かし、照明条件を調整します。
NOTE:ビデオoverexpことなく、高いコントラストを有する必要がある動物は明るい背景に黒い図形として表示されるように、osed。 - 「リアルタイム検索」をクリックし、フォルダとファイル名を選択します。 30秒20ミリ秒と「時の停止」に「フィールド遅延」に設定します。 (ほとんどのワームがどこにあるか)を記録する板の面積を選択するために押して「ライブレビュー "。ステージ上でプレートを3または4回タップして、すぐに「開始」ビューとプレスの分野での地位を確認する。
- 映画は記録を終了した後、画像の上で右クリックして、.AVIに.cxd形式からムービーを書き出すために「エクスポートモンタージュシーケンス」を選択します。
- 運動性のビデオを分析する
- ImageJソフトウェアを開き、「プラグイン」タブに移動し、その後「wrmtrck」と選択して "wrMTrckバッチ」。分析対象のすべてのファイルを含むディレクトリを選択します。
- FIGURで詳述するようにwrMTrck_Batchの主入力ウィンドウでは、入力値をロードE 5C。各パラメータの説明は、プラグインに付属の説明書に記載されています。 「OK」をクリックし、運動解析の実行をしてみましょう。
- 結果をキュレーションし、検出されたすべてのトラックが実際のCからであることを確認するエレガンスと、アーチファクトを排除する各ムービーの作成.txtファイルのそれぞれを開き、データ解析ソフトウェアファイルに情報をコピーする。手動で偽のワームのトラックをチェックし、排除するために作成された「* _labels.zip」ファイルを開き、得られた「* _labels.tif "を実行。
Access restricted. Please log in or start a trial to view this content.
結果
in vivoでのタイムラプスイメージングによるプリオン様タンパク質の広がり間のモニタリング
トランスジェニックC.プリオンドメインを発現する線虫株は、例えば 、細胞から細胞への伝達および非細胞自律的な毒性、プリオン様タンパク質の特定の側面を分析するために特に適している。動物の透明性は、蛍光人生のあらゆる段階?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
ここに記載されている方法は、拡散とプリオン様タンパク質の複雑な細胞自律的かつ非細胞自律的毒性を説明するのに役立つ。我々は最近、凝集しやすいサイトゾルプリオンドメインはオートファジー関連プロセスでの膜結合小胞に取り込まれることを発見した。これらの小胞の特定のサブセット内および細胞及び組織40との間のプリオンドメインを輸送する。生きている動物に彼ら...
Access restricted. Please log in or start a trial to view this content.
開示事項
The authors declare no competing financial interests.
謝辞
We thank Cindy Voisine and Yoko Shibata for helpful discussion and critical comments on the manuscript. We acknowledge the High Throughput Analysis Laboratory (HTAL) and the Biological Imaging Facility (BIF) at Northwestern University for their assistance. This work was funded by grants from the National Institutes of Health (NIGMS, NIA, NINDS), the Ellison Medical Foundation, and the Daniel F. and Ada L. Rice Foundation (to R.I.M.). C.I.N.-K. was supported by the Deutsche Forschungsgemeinschaft (KR 3726/1-1).
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Reagent | |||
| Nanosphere size standards 100 nm | ThermoScientific | 3100A | |
| Levamisole | Sigma | L-9756 | |
| IPTG | Sigma | 15502-10G | |
| Ahringer RNAi library | Source BioScience LifeSciences | http://www.lifesciences.sourcebioscience .com/clone-products/non-mammalian/c-elegans/c-elegans-rnai-library/ | |
| Equipment | |||
| Sorvall Legend XTR Refrigerated Centrifuge, 120VAC | ThermoScientific | 75004521 | http://www.coleparmer.com/Product/Thermo_Scientific_Sorvall_Legend_ XTR_Refrigerated_Centrifuge_120 VAC/EW-17707-60 |
| 96 pin replicator | Scionomix | http://www.scinomix.com/all-products/96-pin-replicator/ | |
| HiGro high-capacity, incubating shaker | Digilab | http://www.digilabglobal.com/higro | |
| Multidrop Combi Reagent Dispenser | Titertrek | http://groups.molbiosci.northwestern.edu/hta/titertek.htm | |
| Biomek FX AP96 Automated Workstation | Beckman Coulter | http://groups.molbiosci.northwestern.edu/hta/biomek_multi.htm | |
| Innova44 shaker | New Brunswick | http://www.eppendorf.com/int///index.php?sitemap=2.3&pb=d78efbc05310ec 04&action=products&contentid=1& catalognode=83389 | |
| M205 FA | Leica | http://www.leica-microsystems.com/de/produkte/stereomikroskope-makroskope/fluoreszenz/details/product/leica-m205-fa/ | |
| ORCA-R2 C10600-10BDigital CCD camera | Hamamatsu | http://www.hamamatsu.com/jp/en/community/life_science_camera/product/search/C10600-10B/index.html | |
| Spinning Disc AF Confocal Microscope | Leica | http://www.leica-microsystems.com/products/light-microscopes/life-science-research/fluorescence-microscopes/details/product/leica-sd-af/ | |
| Falcon 4M60 camera | Teledyne Dalsa | http://www.teledynedalsa.com/imaging/products/cameras/area-scan/falcon/PT-41-04M60/ | |
| Software | |||
| MetaMorph Microscopy Automation & Image Analysis Software | Molecular Devices | http://www.moleculardevices.com/products/software/meta-imaging-series/metamorph.html | |
| Hamamatsu SimplePCI Image Analysis Software | Meyer Instruments | http://meyerinst.com/imaging-software/hamamatsu/index.htm | |
| ImageJ | NIH | http://rsbweb.nih.gov/ij/download.html | |
| wrMTrck plugin for ImageJ | http://www.phage.dk/plugins/wrmtrck.html | ||
| C. elegans strains | |||
| N2 (WT) | Caenorhabditis Genetics Center (CGC) | http://www.cgc.cbs.umn.edu/strain.php?id=10570 | |
| AM815 rmIs323[myo-3p::sup35(r2e2)::rfp] | Morimoto lab | available from our laboratory | |
| See table 1 for a source for folding sensor and stress reporter strains |
参考文献
- Prusiner, S. B. Novel proteinaceous infectious particles cause scrapie. Science. 216 (4542), 136-144 (1982).
- Jarrett, J. T., Lansbury, P. T. Seeding 'one-dimensional crystallization' of amyloid: a pathogenic mechanism in Alzheimer's disease and scrapie. Cell. 73 (6), 1055-1058 (1993).
- Caughey, B., Kocisko, D. A., Raymond, G. J., Lansbury, P. T. Aggregates of scrapie-associated prion protein induce the cell-free conversion of protease-sensitive prion protein to the protease-resistant state. Chem Biol. 2 (12), 807-817 (1995).
- Wickner, R. B. [URE3] as an altered URE2 protein: evidence for a prion analog in Saccharomyces cerevisiae. Science. 264 (5158), 566-569 (1994).
- Chien, P., Weissman, J. S., DePace, A. H. Emerging principles of conformation-based prion inheritance. Annu Rev Biochem. 73, 617-656 (2004).
- Kimberlin, R. H., Walker, C. A. Pathogenesis of mouse scrapie: patterns of agent replication in different parts of the CNS following intraperitoneal infection. J R Soc Med. 75 (8), 618-624 (1982).
- Beekes, M., McBride, P. A., Baldauf, E. Cerebral targeting indicates vagal spread of infection in hamsters fed with scrapie. J Gen Virol. 79 (3), 601-607 (1998).
- Jucker, M., Walker, L. C. Self-propagation of pathogenic protein aggregates in neurodegenerative diseases. Nature. 501 (7465), 45-51 (2013).
- Aguzzi, A. Cell biology: Beyond the prion principle. Nature. 459 (7249), 924-925 (2009).
- Scherzinger, E., et al. Self-assembly of polyglutamine-containing huntingtin fragments into amyloid-like fibrils: implications for Huntington's disease pathology. Proc Natl Acad Sci U S A. 96 (8), 4604-4609 (1999).
- Wood, S. J., et al. alpha-synuclein fibrillogenesis is nucleation-dependent. Implications for the pathogenesis of Parkinson's disease. J Biol Chem. 274 (28), 19509-19512 (1999).
- Wang, Y. Q., et al. Relationship between prion propensity and the rates of individual molecular steps of fibril assembly. J Biol Chem. 286 (14), 12101-12107 (2011).
- Cushman, M., Johnson, B. S., King, O. D., Gitler, A. D., Shorter, J. Prion-like disorders: blurring the divide between transmissibility and infectivity. J Cell Sci. 123 (8), 1191-1201 (2010).
- Tanaka, M., Collins, S. R., Toyama, B. H., Weissman, J. S. The physical basis of how prion conformations determine strain phenotypes. Nature. 442 (7102), 585-589 (2006).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. J Struct Biol. 179 (2), 152-160 (2012).
- Ilieva, H., Polymenidou, M., Cleveland, D. W. Non-cell autonomous toxicity in neurodegenerative disorders: ALS and beyond. J Cell Biol. 187 (6), 761-772 (2009).
- Nussbaum-Krammer, C. I., Morimoto, R. I. Caenorhabditis elegans as a model system for studying non-cell-autonomous mechanisms in protein-misfolding diseases. Dis Model Mech. 7 (1), 31-39 (2014).
- Lino, M. M., Schneider, C., Caroni, P. Accumulation of SOD1 mutants in postnatal motoneurons does not cause motoneuron pathology or motoneuron disease. J Neurosci. 22 (12), 4825-4832 (2002).
- Li, J. Y., et al. Lewy bodies in grafted neurons in subjects with Parkinson's disease suggest host-to-graft disease propagation. Nat Med. 14 (5), 501-503 (2008).
- Desplats, P., et al. Inclusion formation and neuronal cell death through neuron-to-neuron transmission of alpha-synuclein. Proc Natl Acad Sci U S A. 106 (31), 13010-13015 (2009).
- Clement, A. M., et al. Wild-type nonneuronal cells extend survival of SOD1 mutant motor neurons in ALS mice. Science. 302 (5642), 113-117 (2003).
- Gu, X., et al. Pathological cell-cell interactions elicited by a neuropathogenic form of mutant Huntingtin contribute to cortical pathogenesis in HD mice. Neuron. 46 (3), 433-444 (2005).
- Yamanaka, K., et al. Mutant SOD1 in cell types other than motor neurons and oligodendrocytes accelerates onset of disease in ALS mice. Proc Natl Acad Sci U S A. 105 (21), 7594-7599 (2008).
- Garden, G. A., et al. Polyglutamine-expanded ataxin-7 promotes non-cell-autonomous purkinje cell degeneration and displays proteolytic cleavage in ataxic transgenic mice. J Neurosci. 22 (12), 4897-4905 (2002).
- Raeber, A. J., et al. Astrocyte-specific expression of hamster prion protein (PrP) renders PrP knockout mice susceptible to hamster scrapie. EMBO J. 16 (20), 6057-6065 (1997).
- Yazawa, I., et al. Mouse model of multiple system atrophy alpha-synuclein expression in oligodendrocytes causes glial and neuronal degeneration. Neuron. 45 (6), 847-859 (2005).
- Lobsiger, C. S., Cleveland, D. W. Glial cells as intrinsic components of non-cell-autonomous neurodegenerative disease. Nat Neurosci. 10 (11), 1355-1360 (2007).
- Sambataro, F., Pennuto, M. Cell-autonomous and non-cell-autonomous toxicity in polyglutamine diseases. Prog Neurobiol. 97 (2), 152-172 (2012).
- Polymenidou, M., Cleveland, D. W. Prion-like spread of protein aggregates in neurodegeneration. J Exp Med. 209 (5), 889-893 (2012).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nat Rev Mol Cell Biol. 11 (4), 301-307 (2010).
- Braak, H., Braak, E., Bohl, J. Staging of Alzheimer-related cortical destruction. Eur Neurol. 33 (6), 403-408 (1993).
- Meyer-Luehmann, M., et al. Exogenous induction of cerebral beta-amyloidogenesis is governed by agent and host. Science. 313 (5794), 1781-1784 (2006).
- Luk, K. C., et al. Pathological alpha-synuclein transmission initiates Parkinson-like neurodegeneration in nontransgenic mice. Science. 338 (6109), 949-953 (2012).
- Clavaguera, F., et al. Transmission and spreading of tauopathy in transgenic mouse brain. Nat Cell Biol. 11 (7), 909-913 (2009).
- Nonaka, T., et al. Prion-like Properties of Pathological TDP-43 Aggregates from Diseased Brains. Cell Rep. 4 (1), 124-134 (2013).
- Lundmark, K., et al. Transmissibility of systemic amyloidosis by a prion-like mechanism. Proc Natl Acad Sci U S A. 99 (10), 6979-6984 (2002).
- Lai, C. H., Chou, C. Y., Ch'ang, L. Y., Liu, C. S., Lin, W. Identification of novel human genes evolutionarily conserved in Caenorhabditis elegans by comparative proteomics. Genome Res. 10 (5), 703-713 (2000).
- Xu, X., Kim, S. K. The early bird catches the worm: new technologies for the Caenorhabditis elegans toolkit. Nat Rev Genet. 12 (11), 793-801 (2011).
- Boulin, T., Hobert, O. From genes to function: the C. elegans genetic toolbox. Wiley Interdiscip Rev Dev Biol. 1 (1), 114-137 (2012).
- Nussbaum-Krammer, C. I., Park, K. W., Li, L., Melki, R., Morimoto, R. I. Spreading of a prion domain from cell-to-cell by vesicular transport in Caenorhabditis elegans. PLoS Genet. 9 (3), e1003351(2013).
- Chernoff, Y. O., Lindquist, S. L., Ono, B., Inge-Vechtomov, S. G., Liebman, S. W. Role of the chaperone protein Hsp104 in propagation of the yeast prion-like factor [psi. Science. 268 (5212), 880-884 (1995).
- Liu, J. J., Lindquist, S. Oligopeptide-repeat expansions modulate 'protein-only' inheritance in yeast. Nature. 400 (6744), 573-576 (1999).
- Halfmann, R., et al. Prions are a common mechanism for phenotypic inheritance in wild yeasts. Nature. 482 (7385), 363-368 (2012).
- Tyedmers, J., Madariaga, M. L., Lindquist, S. Prion switching in response to environmental stress. PLoS Biol. 6 (11), e294(2008).
- Krammer, C., et al. The yeast Sup35NM domain propagates as a prion in mammalian cells. Proc Natl Acad Sci U S A. 106 (2), 462-467 (2009).
- Hofmann, J. P., et al. Cell-to-cell propagation of infectious cytosolic protein aggregates. Proc Natl Acad Sci U S A. 110 (15), 5951-5956 (2013).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , (2006).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C. elegans Using Microinjection. J. Vis. Exp. (18), e833(2008).
- Transformation and microinjection. WormBook. Evans, T. C. , (2006).
- Methods in cell biology. Wormbooks. Shaham, S. , (2006).
- Kim, E., Sun, L., Gabel, C. V., Fang-Yen, C. Long-term imaging of Caenorhabditis elegans using nanoparticle-mediated immobilization). PLoS One. 8 (1), e53419(2013).
- Fay, D. Genetic mapping and manipulation: Chapter 1-Introduction and basics. WormBook. , (2006).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Rual, J. F., et al. Toward improving Caenorhabditis elegans phenome mapping with an ORFeome-based RNAi library. Genome Res. 14 (10B), 2162-2168 (2004).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Kern, A., Ackermann, B., Clement, A. M., Duerk, H., Behl, C. HSF1-controlled and age-associated chaperone capacity in neurons and muscle cells of C. elegans. PLoS One. 5 (1), e8568(2010).
- Becker, J., Walter, W., Yan, W., Craig, E. A. Functional interaction of cytosolic hsp70 and a DnaJ-related protein, Ydj1p, in protein translocation in vivo. Mol Cell Biol. 16 (8), 4378-4386 (1996).
- Salvaterra, P. M., McCaman, R. E. Choline acetyltransferase and acetylcholine levels in Drosophila melanogaster: a study using two temperature-sensitive mutants. J Neurosci. 5 (4), 903-910 (1985).
- Goloubinoff, P., Mogk, A., Zvi, A. P., Tomoyasu, T., Bukau, B. Sequential mechanism of solubilization and refolding of stable protein aggregates by a bichaperone network. Proc Natl Acad Sci U S A. 96 (24), 13732-13737 (1999).
- Schroder, H., Langer, T., Hartl, F. U., Bukau, B. D. naK. DnaJ and GrpE form a cellular chaperone machinery capable of repairing heat-induced protein damage. EMBO J. 12 (11), 4137-4144 (1993).
- Rampelt, H., et al. Metazoan Hsp70 machines use Hsp110 to power protein disaggregation. EMBO J. 31 (21), 4221-4235 (2012).
- Gupta, R., et al. Firefly luciferase mutants as sensors of proteome stress. Nat Methods. 8 (10), 879-884 (2011).
- Gidalevitz, T., Ben-Zvi, A., Ho, K. H., Brignull, H. R., Morimoto, R. I. Progressive disruption of cellular protein folding in models of polyglutamine diseases. Science. 311 (5766), 1471-1474 (2006).
- Ben-Zvi, A., Miller, E. A., Morimoto, R. I. Collapse of proteostasis represents an early molecular event in Caenorhabditis elegans aging. Proc Natl Acad Sci U S A. 106 (35), 14914-14919 (2009).
- Karady, I., et al. Using Caenorhabditis elegans as a model system to study protein homeostasis in a multicellular organism. J Vis Exp. (82), e50840(2013).
- Gidalevitz, T., Krupinski, T., Garcia, S., Morimoto, R. I. Destabilizing protein polymorphisms in the genetic background direct phenotypic expression of mutant SOD1 toxicity. PLoS Genet. 5 (3), e1000399(2009).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proc Natl Acad Sci U S A. 99 (16), 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine proteins at the pathogenic threshold display neuron-specific aggregation in a pan-neuronal Caenorhabditis elegans model. J Neurosci. 26 (29), 7597-7606 (2006).
- Mohri-Shiomi, A., Garsin, D. A. Insulin signaling and the heat shock response modulate protein homeostasis in the Caenorhabditis elegans intestine during infection. J Biol Chem. 283 (1), 194-201 (2008).
- Libina, N., Berman, J. R., Kenyon, C. Tissue-specific activities of C. elegans DAF-16 in the regulation of lifespan. Cell. 115 (4), 489-502 (2003).
- Schatzl, H. M., et al. A hypothalamic neuronal cell line persistently infected with scrapie prions exhibits apoptosis. J Virol. 71 (11), 8821-8831 (1997).
- Keith, S. A., Amrit, F. R., Ratnappan, R., Ghazi, A. The C. elegans healthspan and stress-resistance assay toolkit. Methods. , (2014).
- Pierce-Shimomura, J. T., et al. Genetic analysis of crawling and swimming locomotory patterns in C. elegans. Proc Natl Acad Sci U S A. 105 (52), 20982-20987 (2008).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved