Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Untersuchung der Verbreitung und Toxizität von Prion-ähnlichen Proteine Mit dem Metazoan Modellorganismus
In diesem Artikel
Zusammenfassung
Prion-like propagation of protein aggregates has recently emerged as being implicated in many neurodegenerative diseases. The goal of this protocol is to describe, how to use the nematode C. elegans as a model system to monitor protein spreading and to investigate prion-like phenomena.
Zusammenfassung
Prions are unconventional self-propagating proteinaceous particles, devoid of any coding nucleic acid. These proteinaceous seeds serve as templates for the conversion and replication of their benign cellular isoform. Accumulating evidence suggests that many protein aggregates can act as self-propagating templates and corrupt the folding of cognate proteins. Although aggregates can be functional under certain circumstances, this process often leads to the disruption of the cellular protein homeostasis (proteostasis), eventually leading to devastating diseases such as Alzheimer’s disease (AD), Parkinson’s disease (PD), Amyotrophic lateral sclerosis (ALS), or transmissible spongiform encephalopathies (TSEs). The exact mechanisms of prion propagation and cell-to-cell spreading of protein aggregates are still subjects of intense investigation. To further this knowledge, recently a new metazoan model in Caenorhabditis elegans, for expression of the prion domain of the cytosolic yeast prion protein Sup35 has been established. This prion model offers several advantages, as it allows direct monitoring of the fluorescently tagged prion domain in living animals and ease of genetic approaches. Described here are methods to study prion-like behavior of protein aggregates and to identify modifiers of prion-induced toxicity using C. elegans.
Einleitung
Viele neurodegenerative Erkrankungen, einschließlich Alzheimer-Krankheit (AD), Parkinson-Krankheit (PD), Amyotrophe Lateralsklerose (ALS), und übertragbaren spongiformen Enzephalopathien (TSE) werden mit Aggregation neigende Proteine verbunden und werden daher allgemein als Proteinfehlfaltungserkrankungen (PMDs bekannt ). TSE oder Prionenerkrankungen stellen eine einzigartige Klasse von PMDs indem sie Infektions in Mensch und Tier 1 sein kann. Auf molekularer Ebene, replizieren Prionen durch die Rekrutierung und Umwandlung von monomeren α-Helix reiche Wirts kodierten zellulären PrP (PrP C) in die pathologische β-Faltblatt-reiche PrP Sc Konformation 2,3. Selbstausbreiteproteinaggregate wurden auch in Pilzen, die wichtige Eigenschaften mit Säugetier Prionen 4,5 austauschen können. Zusätzlich sind Säuger Prionen bewegen kann, von Zelle zu Zelle und infizieren naiven Zellen 6,7.
Während PMDs other als TSE nicht infektiös sind, teilen sie sich eine gemeinsame pathogene Prinzip mit Prionenerkrankungen 8,9. Obwohl die Proteine, die jeder der PMDs verknüpft sind nicht in der Struktur oder Funktion bezogen, sie bilden alle Aggregate über eine Kristallisationsartigen Prozess namens nukleierten oder impft Polymerisation; außerdem proteinhaltigen Samen wachsen durch die Einstellung ihrer löslichen Isoformen 2,10,11. Die Effizienz der Selbst Propagate variiert in vivo in Abhängigkeit von den intrinsischen Eigenschaften des Proteins, die zusammen mit zusätzlichen zellulären Faktoren wie molekulare Chaperone bestimmen letztlich Raten Aggregat Nukleation, Säen, Fragmentierung und Ausbreiten 12-15. Daher muss es eine feine Balance zwischen diesen Faktoren, die eine effiziente Vermehrung von Proteinaggregation ermöglicht existieren. Dies könnte auch erklären, warum nur einige amyloidogenen Aggregate Hafen der Eigenschaften eines Prion und damit nicht alle PMDs sind ansteckend. Prionen scheint "Top-Performer" o vertretenfa breites Spektrum von sich selbst replizierenden proteinartige Aggregate, die ihnen ein leistungsfähiges Werkzeug zur PMDs 8,13 zu studieren.
Interessanterweise hat die Toxizität mit krankheitsbedingten Aggregaten assoziiert oft ein nicht zellautonomen Komponenten 16,17. Dies bedeutet, dass sie Nachbarzellen, die das entsprechende Gen nicht exprimieren, im Gegensatz zu einem rein Zelle autonome Wirkung, was bedeutet, daß nur die Zellen, die das Gen weisen die spezifischen Phänotyp beeinflussen. Dies wurde überzeugend von gewebespezifische Expression nachgewiesen oder knock down der entsprechenden Proteine in zahlreichen Modellen neurodegenerativer Erkrankungen 18-26. Verschiedene Mechanismen wurden als Grundlage für diese nicht-zellautonomen Toxizität PMDs einschließlich vermindert Nährstoffversorgung Ungleichgewicht in neuronalen Signalisierung Glutamatexzitotoxizität und neuroinflammation 16,27,28 vorgeschlagen. Zusätzlich ist ein Prion artige Bewegung der Krankheit gebundenen Aggregate zwischen Zellen might diesem Aspekt 29,30 tragen. Zunehmende Hinweise darauf, dass andere Proteineinschlüsse als Prionen von Zelle-zu-Zelle zu übertragen, was erklären kann, die charakteristische Verteilung der Pathologie in vielen PMDs 30-36 beobachtet. Es hat sich jedoch noch nicht bestimmt werden, ob es einen klaren ursächlichen Zusammenhang zwischen interzelluläre Bewegung Krankheit Proteine und die toxische Wirkung auf Nachbarzellen. Daher ist ein besseres Verständnis der zellulären Wege, die Zell-zu-Zell-Übertragung und nicht zellautonomen Toxizität zugrunde notwendig und wesentlich für die Entwicklung neuartiger Therapeutika. Allerdings sind viele Aspekte der Prionartigen Ausbreitung und zellulären Faktoren, die Zell-zu-Zell-Übertragung von fehlgefalteten Proteinen Metazoen beeinflussen nicht gut verstanden, insbesondere auf der Ebene der Organismen.
Der Nematode Caenorhabditis elegans hat mehrere Vorteile, die das Potential bieten entdecken Sie neue Facetten des Prion-ähnlichen spreading in Metazoen 17. Es ist transparent, so dass für in vivo-Tracking von fluoreszenzmarkierten Proteine im lebenden Organismus. Darüber hinaus werden viele zelluläre und physiologische Prozesse durch Krankheit betroffen von Würmern die menschliche konserviert und C. elegans eignet sich auch für eine Vielzahl von genetischen Manipulationen und molekularen und biochemischen Analysen 37-39. Genau 959 Körperzellen bilden die erwachsenen Hermaphroditen mit einem einfachen Körper Plan, der noch mehrere verschiedene Gewebearten, einschließlich Muskeln, Neuronen und Darm hat.
Um eine neue Prion-Modell in C zu etablieren elegans, entschieden wir uns für exogen drücken die gut charakterisiert Glutamin / Asparagin (Q / N) -reichen Prion Domain NM der cytosolischen Hefe-Prion-Protein Sup35, da gibt es keine bekannten endogenen Prion-Proteine in Würmern 4,40. Hefe Prionen waren von unschätzbarem Wert bei der Aufklärung grundlegender Mechanismen von Prionenreplikation 41-44. Darüber hinaus ist die Tanne NMst cytosolische Prion-ähnliches Protein, die gezeigt wurde, um den gesamten Lebenszyklus eines Prion in Säugetierzellkulturen 45,46 rekapitulieren. Ebenso wird, wenn in C exprimiert elegans, die hervorragend an die unterschiedlichen Anforderungen für die Vermehrung in Metazoen-Zellen im Vergleich zu Hefezellen und wiesen Merkmale von Prion biology 40. NM Aggregation wurde mit einem tiefen toxischen Phänotyp verbunden sind, einschließlich der Störung der mitochondrialen Integrität und das Aussehen angenommen Sup35 Prion Domäne verschiedene Autophagie bezogenen Vesikel auf zellulärer Ebene sowie embryonale und larvale Stillstand, Entwicklungsverzögerung, und eine weit verbreitete Störung der Proteinfaltung Umgebung auf der organismischen Ebene. Auffallend ist, dass das Prion-Domain weist Zell autonomen und nicht autonomen Zelltoxizität, Auswirkungen auf benachbarte Gewebe, bei denen das Transgen nicht ausgedrückt. Ferner ist der vesikulären Transport des Prion Domäne innerhalb und zwischen den Zellen in Echtzeit überwacht In-vivo-40.
Hier beschreiben wir, wie man Prion-ähnlichen Verbreitung in C prüfen elegans. Wir werden erklären, wie die intra- und interzellulären Transport von Vesikeln, die das Prion-Domäne mit Zeitraffer-Fluoreszenzmikroskopie zu überwachen. Wir werden die Verwendung von gewebespezifischen Klapp Sensoren unterstreichen und ubiquitär exprimiert Stress Reportern zu Zelle autonomen und nicht autonomen Zell Effekte auf zellulärer Fitness zu bewerten. Schließlich werden wir den Ablauf einer vor kurzem durchgeführt genomweiten RNA-Interferenz (RNAi) Bildschirm beschreiben, neue Modifikatoren von Prion-induzierte Toxizität zu identifizieren. In Kombination können diese Methoden helfen, necken neben genetischen Pfade in der interzellulären Bewegung von Proteinen und ihre nicht zellautonomen Toxizität beteiligt.
Protokoll
1. Überwachung transzelluläre Verbreitung des Prion-ähnlichen Proteine durch In-vivo-Zeitraffer-Imaging
HINWEIS: Wachsen C. elegans Wildtyp (WT) (N2) und transgenen Linien nach Standardmethoden und die Kultivierungstemperatur 47 sorgfältig zu kontrollieren.
- Gene transgenen Linien von C elegans Expression des Prion-ähnlichen Protein, mit monomeren rot fluoreszierendes Protein (mRFP) markiert. In diesem Video, das, wie man die Mikroinjektion 48 demonstriert. Weitere Details und die Methoden beschreiben, wie Sie diese extrachromosomale Linien zu integrieren, finden 49.
- Bereiten synchronisiert Populationen Eiablage oder Bleichen nach Standardverfahren 50.
- Synchronisation durch Eiablage
- Über 10-20 gravid Erwachsenen auf einem Teller und lassen Sie sie Eier legen für 1-2 Std. Erwachsene entfernen von der Platte und lassen Sie die Nachkommen wachsen, bis die gewünschte Zeit.
- Synchronisation von Bleich
- Sammeln Sie eine unsynchronisierte Bevölkerung von schwangeren Erwachsenen und bleichen sie mit alkalischen Natriumhypochlorit-Lösung (250 mM NaOH und 1: 4 (v / v) Verdünnung der kommerziellen Bleichmittel in H 2 O). Zweimal (218 · g für 1 min) Waschen Sie die Eier mit M9-Puffer 47 (21 MMNA 2 HPO 4 · 7 H 2 O, 22 MMKH 2 PO 4, 86 mM NaCl, 1 mMMgSO 4 · 7 H 2 O, fügen dH 2 O bis zu 1 L).
- Es ihnen ermöglichen, in M9-Puffer unter leichtem Schütteln O bei 20 ° C schlüpfen / N. Worm Entwicklung wird im L1 Stufe in Abwesenheit eines Nahrungsmittels zu verhaften, so dass eine synchronisierte Bevölkerung. Übertragen L1s auf frische Nematode Wachstumsmedien (NGM) Platten mit OP50 E. ausgesät coli-Bakterien und lassen Sie die Nachkommen entwickeln, bis die gewünschte 47 Jahren.
- Synchronisation durch Eiablage
- Planen 2% Agarose-Pads (in H 2 O) auf einen Objektträger, wie beschrieben 50.
- Bereiten Sie zwei mikop Folien mit Etikettenband über ihre ganze Länge angeordnet, um als Abstandshalter verwendet werden. Platzieren Sie eine dritte Objektträger zwischen ihnen.
- Löse 2% Agarose in H 2 O und Pipette ein Tropfen auf die saubere Folie.
- Platzieren eines vierten Schieber senkrecht zu den drei anderen Folien oben auf den Agar Abfall. Drücken Sie ihn vorsichtig nach unten, um das Kissen in die gleiche Dicke wie die Etikettierung Band zu glätten.
- Lassen Sie es trocken für 1 Minute, bevor Sie die Abstandshalter und leichtes Ziehen die Folien auseinander. Die Agar-Pad, einen von ihnen zu bleiben.
- Pipette ~ 10 ul Anästhetikum (2 mM Levamisol in M9-Puffer) mit dem Pad und Übertragungs ~ 10 Tieren unter Verwendung einer Platindraht-Pick. Deckel mit einem Deckglas (~ 22 x 22 mm) und nehmen Sie Bilder in 1 Std.
- Alternativ, um Filme über einen längeren Zeitraum hinweg zu erwerben oder die Möglichkeit einer Bewegung der Tiere weiter zu reduzieren, verwenden eine Kombination von Betäubung und Ruhigstellung Wulst 51.
- Bereiten Sie 10% agaRose-Pads (in M9-Puffer), wie beschrieben, 51 und fügen Würmer zu 3 ul Nanokugel Größenstandards Lösung (Polystyrol-Kügelchen, 100 nm) und 3 ul Betäubung (4 mM Levamisol in M9-Puffer). Bedecken Sie vorsichtig mit einem Deckglas. Um ein Austrocknen zu verhindern, versiegeln das Deckglas mit VALAP (Mischung aus gleichen Teilen Vaseline, Lanolin und Paraffinwachs).
- Bild immobilisiert Würmer mit einem konfokalen Mikroskop.
HINWEIS: Die Ergebnisse werden mit einem Spinning Disc AF Konfokalmikroskop mit einem EM-CCD-Kamera und einem Mikroskopie Automatisierung und Bildanalyse-Software wie MetaMorph ausgestattet erhalten und stellen die Besonderheiten unter, aber vergleichbaren konfokalen Bildgebungssysteme können ebenfalls verwendet werden.- Verwenden Sie das 63X oder 100X / 1.4NA Öl-Objektiv und setzen Sie den Objektträger, die Würmer in den Mikroskopobjektträgerhalter.
- Öffnen Sie die Software. Passen Laserleistung und Filter für mRFP Bildgebung. Verwenden Sie die 561-nm-Laser mit 10% Leistung und Emissionsfilter> 600 nm.
- Öffnen "Mehrdimensionale Bildaufnahme". Unter der Registerkarte "Allgemein" die Kontrollkästchen für "Timelapse" und "Run Journals" (Hardware-Autofokus: aus).
- Auf der Registerkarte "Speichern" auswählen oder das Verzeichnis Ordner, in dem die Dateien gespeichert werden sollen. Benennen Sie die Datei.
- Unter dem Reiter "Wellenlänge", wählen Sie die entsprechende Beleuchtung und die Belichtungszeit und Verstärkung. Mit "YokoQuad Red" (oder eine äquivalente Beleuchtungseinstellung für mRFP imaging) mit einer Einwirkungszeit zwischen 100 und 300 ms und einer Kameraverstärkung zwischen 100 und 300, abhängig von der einzelnen Probe (beurteilt unter Verwendung der Livebild).
- Unter dem Reiter "Timelapse" wählen Sie "Anzahl der Zeitpunkte" = 301, "Dauer" = 5 min und "Zeit / Intervall" = 1 Sek.
- Im Rahmen des "Journals" Tab wählen Journal: "AFC SET Z HOLD" und "AFC Zurück zur Z HOLD", Typ: &# 8220; Special "(zweimal), Ausgangspunkt:" Start Zeitpunkt "und" End of Zeitpunkt ". Das Autofokus-Option ist wichtig, um Bild die gleiche Abschnitt über einen längeren Zeitraum.
- Wenn das Setup abgeschlossen ist, klicken Sie auf "Erfassen". Die Zeitraffer-Video wird als separate TIFF-Files abgelegt.
- Öffnen Sie alle TIFF-Dateien eines bestimmten Zeitreihen in ImageJ. Gehen Sie auf "Bild" → "Stacks" → "Bilder zu Stacks". Optional können Helligkeit und Kontrast, fügen Größe Bar, etc. Exportieren Sie den Film unter "Datei" → "Speichern als" → "AVI ..." (zum Beispiel siehe Video 1 und 2 entsprechend Figur 1).
2. Mit Klapp Sensoren und Spannungs Reporter Cell Autonome und Nichtzellen Autonome Wirkt auf Proteostase und Toxizität Untersuchen
- Gene transgenen Linien, die einen Klappsensor oder Stress reporte coexprimierenr zusammen mit der Prion-ähnlichen Proteins. Für Methoden, wie man Kreuze errichten oder zu generieren transgenen Linien finden 48,49,52. Siehe Tabelle 1 für eine Liste von C elegans-Stämme, die verwendet werden können.
- Bereiten synchronisierten Populationen transgener Tiere und wachsen, bis die gewünschte Alters wie oben (Abschnitt 1.2) 50 beschrieben.
- Mit einem Stereomikroskop, überprüfen Sie die entsprechende Phänotyp des Faltungssensor oder Stress Reporter.
- Für die Faltung Sensor, bestimmen die Anzahl der Tiere beherbergen Aggregate an jedem Tag nach der Synchronisation (ein Beispiel finden Sie in Abbildung 2 E und F).
- Für die Stress-Reporter, Test, wenn die Co-Expression der Prion-ähnlichen Protein führt zu einer erhöhten Fluoreszenz des Reporter (für ein Beispiel siehe Abbildung 3).
3. Genomweite Bildschirm Unterdrückung von Prion-induzierte Toxizität in C. elegans
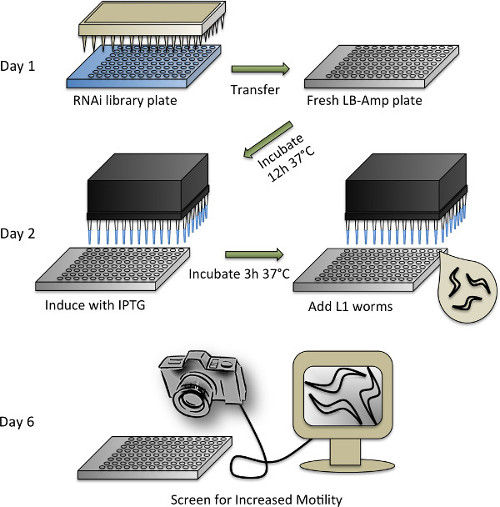
Abbildung 4. Schematische Darstellung des RNAi-Screening-Protokoll. Siehe Protokoll Abschnitt 3 für eine detaillierte Beschreibung der einzelnen Schritte.
- Synchronisation von C. elegans Würmer und Vervielfältigung von RNAi-Bibliothek
- Für die RNAi Bildschirm aus den Ahringer RNAi-Bibliothek (oder die Vidal RNAi-Bibliothek) 53,54. Rearray das RNAi-Bibliothek aus dem ursprünglichen 384-Well-Format in ein 96-Well-Platten, indem die Platten mit 100 & mgr; l LB-Amp-Medium (50 ug / ml Ampicillin in LB) mit 10% Glycerin ergänzt und Beimpfen mit einem 96-Pin-Replikator. Wachsen bei 37 ° CO / N unter Rühren und bei -80 ° C.
- Pflegen C. elegans WT und transgenen Linien bei 20 ° C auf NGM-Platten ausgesät mit OP50 E. coli Bakterien nach Standardverfahren 47.
- Synchronisieren Prion Domain transgene und WT (Kontrolle) Nematoden durch Bleichen (siehe Abschnitt 1.2).
- Am selben Tag, dass die Würmer gebleicht werden, bereiten LB-Medium mit 50 ug / ml Ampicillin. Verwenden Sie eine automatisierte Reagenzspender zu verzichten (oder Pipette manuell) 65 ul der LB-Amp-Medium in jede Vertiefung einer Platte mit 96 Vertiefungen.
- Entfernen Sie 96-Loch Ahringer RNAi Bibliothek Platten von -80 ° C und bringt sie zu einer sterilen Haube mit Küchenpapier ausgekleidet. Unverzüglich entfernen Sie die Dichtungsband, indem Sie die Platte auf den Kopf, während Platten noch gefroren sind, wobei darauf geachtet, um eine Kontamination von Eis auf der Außenseite der Platten zu vermeiden. Reinvert die Teller und lassen Sie sie für etwa 30 Minuten auftauen.
- Tauchen Sie ein steriles 96 Pin-Replikator in einem RNAi-Bibliothek Platte und dann in eine frische LB-Amp-Platte. Verwenden Sie eine neue sterile Replikator für jede Platte. Dichten Sie alle Platten mit Klebefolienband und geben die Bibliotheksplatten bis -80 ° C. Die Replikatoren in Bleichmittel eingeweicht werden, gespült,und autoklaviert, um wieder verwendet werden.
- Lassen duplizierten RNAi Platten zu O / N in einem Brutschrank auf 37 ° C und 300 Umdrehungen pro Minute eingestellt wachsen. Um HT115 E. verwenden coli Bakterien beherbergen, die einen leeren Vektor (L4440) als Kontrolle, wachsen sie einzeln in ein Kulturröhrchen pipettieren und 65 ul der Kultur mit einer Mehrkanalpipette in ein Viertel der zwei Platten mit 96 Vertiefungen.
- Die Induktion der Bakterien dsRNA Produktion und Zugabe von Würmern
- Verdünne Isopropyl-β-D-thiogalactopyranosid (IPTG) zu einer 5 mM Konzentration in ddH 2 O. Verwenden Sie eine automatisierte Workstation mit einem Mehrkanal-Kopf zu verzichten (oder Pipette manuell) 10 ul verdünnt IPTG in jede Vertiefung der RNAi Bakterien und der Steuerplatten. Verschließen Sie und legen Sie die Platten wieder in den Inkubator. Lassen Sie sie schütteln für 3 Stunden bei 37 ° C.
- Während Bakterien zittern, bereiten die Würmer. Mischen Sie die M9 / Wurm Federung gut und Pipette eine kleine Probe (~ 5-10 ul) auf einen Glasobjektträger. Zählendie Zahl der Würmer unter einem Stereomikroskop und berechnen Sie die Anzahl der Würmer pro ul.
- Unter Einhaltung steriler Techniken bereiten eine Lösung ergänzt M9 (M9 +) mit den folgenden Endkonzentrationen: 0,20 mg / ml IPTG, 8,0 ug / ml Cholesterin, 50 ug / ml Ampicillin, 9,6 ug / ml Tetracyclin, 0,0835 g / ml Fungizone und 15 Würmer pro 50 ul.
- Tätigen Sie Lösungen für Prionen Domain transgene und WT Würmer. Das Endvolumen M9 + benötigt wird von der Anzahl der RNAi Platten kopiert werden (siehe unten) abhängig Schneckenlösung plus ~ 30 ml Totvolumen, das in dem Reservoir verbleiben, wenn eine automatisierten Dispenser verwendet.
- Nach 3 Stunden Induktion der Bakterien dsRNA Produktion, nehmen die Platten aus dem Inkubator und lassen Sie sie auf Raumtemperatur (~ 30 min) zu kühlen, um Wärme zu belasten die Tiere.
- Dispense 50 ul M9 + Prion Domain transgenen Wurm-Lösung in jede Vertiefung der RNAi-Platten und einer der leeren Vektor Kontrollplatten. Machensicher, dass die Schnecken Lösung vor jedem Schritt zu mischen, wie die Tiere dazu neigen, auf den Boden des Behälters absetzen.
- Dispense WT Würmer in die zweite Steuerplatte. Lassen Sie die Platten nicht abgedichtet, um die Belüftung zu ermöglichen. Um die Flüssigkultur verdunstet halten, Stack 4-5 Platten zusammen und wickeln mit einem feuchten Papiertuch und Aluminiumfolie. Inkubation bei 200 rpm bei 20 ° C für 4 Tage.
- Wertung
HINWEIS: Nach 4 Tagen im Brutschrank werden die Würmer am zweiten Tag Erwachsenenalter sein und bereit sind, zu untersuchen. Lassen Sie die Tiere nicht schütteln Bedingungen anpassen 30 min vor Screening ungestört Prügel zu gewährleisten.- Mit einem Falcon 4M60 Kamera an einen Computer mit Monitor, Bildschirm visuell erhöht Prügel verbunden, verglichen mit Kontrolle behandelt Prion Domain transgenen Tieren.
- Stellen Sie eine Liste der vorläufigen Ergebnisse zu bestätigen, mit wrMTrck (siehe nächster Abschnitt).
4. Bestätigung der vorläufigenBildschirm Hits
- Motilität Assay auf festen Platten
- Bereiten Platten mit NGM mit 100 ug / ml Ampicillin, 12,5 ug / ml Tetracyclin und 1 mM IPTG ergänzt, nach Standardverfahren 47. Verwenden Sie nach Möglichkeit einen platten Gießmaschinen, um sicherzustellen, alle Platten haben die gleiche Höhe von Medien, die für eine schlankere Videoakquisitionsprozess ermöglicht.
- Wachsen die verschiedenen RNAi-Klone in ~ 1 ml LB + 50 ug / ml Ampicillin, O / N bei 37 ° C und 250 Umdrehungen pro Minute.
- Am nächsten Tag, induziert die Expression der dsRNA mit 1 mM IPTG für 3 Stunden. Samen, die Platten mit 150 ul jeder RNAi bakterielle Klon, zu einer dünnen Schicht ausgebreitet. Lassen Sie die Bakterien trocken 2 Tage bei RT im Dunkeln. Bereiten Sie 3 Platten pro RNAi-Klon.
- Synchronisieren Sie die Wurmpopulation durch Bleichen nach Standardverfahren 50 (siehe Abschnitt 1.2), und lassen Sie die Eier ausbrüten O / N in M9-Medium.
- Nehmen Sie eine Probe des Wurms Federung und bestimmen die Höhe der nematodes pro ul bei dem Stereomikroskop. Dann pipettieren das richtige Volumen der M9 und Würmer in jedem Versuchsplatte, so dass es 25 enthält - 30 L1 Würmer. Wachsen die Nematoden bei 20 ° C für 4 Tage, bis die Würmer erreichen Tag 2 des Erwachsenseins.
- Die quantitative Analyse der Wurm Motilität mit der wrMTrck Plugin für ImageJ
HINWEIS: Die Videos wurden mit einem Stereomikroskop bei 10-facher Vergrößerung mit einem Hamamatsu Orca-R2 Digitalkamera C10600-10B und Hamamatsu einfache PCI-Imaging-Software erfasst.- Schalten Sie die Kamera und das Simple PCI-Imaging-Software. Klicken Sie auf "Live", um zur Einstellung der Bilderzeugungsbedingungen zu ermöglichen.
- Richten Sie die Videobedingungen wie folgt: Gain = 0; Light Mode = High; Speedindex = 1; Binning = 2. Klicken Sie auf "Auto Expose" und passen Sie die Lichtverhältnisse, Bewegung der Mikroskopspiegel und die Helligkeits- und Kontrastwahl.
HINWEIS: Das Video muss einen hohen Kontrast ohne ÜBERBEL habenosed, so dass die Tiere wie schwarze Gestalten auf einem hellen Hintergrund erscheinen. - Klicken Sie auf "Zeit Scan" und wählen Sie einen Ordner und einen Dateinamen ein. Set "Feld Delay" auf 20 ms und "Stopp bei Time" um 30 Sekunden. Drücken Sie auf "Live Review", um den Bereich der Platte wählen zu erfassen (wo die meisten Würmer sind). Tippen Sie auf die Platte 3 oder 4 mal auf der Bühne, schnell bestätigen ihre Position im Sichtfeld ein und drücken Sie "Start".
- Nachdem der Film endet die Aufzeichnung der rechten Maustaste auf das Bild und wählen Sie "Export Montage Sequence", um den Film von einem Format in ein .cxd .avi exportieren.
- Die Analyse der Motilität Videos
- Öffnen Sie die ImageJ-Software, gehen Sie zur Registerkarte "Plugins", dann "wrmtrck" und wählen Sie "wrMTrck Batch". Wählen Sie das Verzeichnis alle Dateien, die analysiert werden soll.
- In der Haupteingabefenster wrMTrck_Batch, laden Sie die Eingabewerte wie in Figur detaillierte 5C. Erläuterung der einzelnen Parameter finden Sie in den Anweisungen, die das Plugin begleiten zu finden. Klicken Sie auf "OK", und lassen Sie die Bewegungsanalyselauf.
- Um die Ergebnisse zu kuratieren und bestätigen Sie, dass alle erkannten Tracks sind von den tatsächlichen C. elegans und beseitigen Artefakte, öffnen Sie jede der TXT-Dateien für jeden Film geschaffen und kopieren Sie die Daten auf eine Datenanalyse-Software-Datei. Öffnen Sie die "* _labels.zip" erstellten Dateien und führen Sie die daraus resultierende "* _labels.tif" von Hand überprüfen und zu beseitigen falsche Wurmspuren.
Ergebnisse
Überwachung interzelluläre Ausbreitung von Prionen-ähnlichen Proteinen durch In-vivo-Zeitraffer-Bildgebung
Transgenen C. elegans Linien die Prion-Domäne exprimieren, sind besonders gut für die Analyse von bestimmten Aspekten der Prion-ähnlichen Proteine, zB Zell-zu-Zell-Übertragung und nicht zellautonomen Toxizität geeignet. Die Transparenz der Tiere ermöglicht Verfolgung von fluoreszenzmarkierten Proteine aus dem lebenden Organismus...
Diskussion
Die hier beschriebenen Methoden helfen, um zu veranschaulichen die Verbreitung und der komplexen Zell autonomen und nicht autonomen Zelltoxizität von Prion-ähnlichen Proteinen. Wir haben vor kurzem festgestellt, dass eine Aggregation neigende cytosolische Prion-Domäne wird in membrangebundene Vesikel in einer Autophagie bezogenen Prozess übernommen. Eine spezielle Untergruppe dieser Vesikel transportiert das Prion-Domäne innerhalb und zwischen den Zellen und Gewebe 40. Der Schlüssel, um ihre Bewegung im...
Offenlegungen
The authors declare no competing financial interests.
Danksagungen
We thank Cindy Voisine and Yoko Shibata for helpful discussion and critical comments on the manuscript. We acknowledge the High Throughput Analysis Laboratory (HTAL) and the Biological Imaging Facility (BIF) at Northwestern University for their assistance. This work was funded by grants from the National Institutes of Health (NIGMS, NIA, NINDS), the Ellison Medical Foundation, and the Daniel F. and Ada L. Rice Foundation (to R.I.M.). C.I.N.-K. was supported by the Deutsche Forschungsgemeinschaft (KR 3726/1-1).
Materialien
| Name | Company | Catalog Number | Comments |
| Reagent | |||
| Nanosphere size standards 100 nm | ThermoScientific | 3100A | |
| Levamisole | Sigma | L-9756 | |
| IPTG | Sigma | 15502-10G | |
| Ahringer RNAi library | Source BioScience LifeSciences | http://www.lifesciences.sourcebioscience .com/clone-products/non-mammalian/c-elegans/c-elegans-rnai-library/ | |
| Equipment | |||
| Sorvall Legend XTR Refrigerated Centrifuge, 120VAC | ThermoScientific | 75004521 | http://www.coleparmer.com/Product/Thermo_Scientific_Sorvall_Legend_ XTR_Refrigerated_Centrifuge_120 VAC/EW-17707-60 |
| 96 pin replicator | Scionomix | http://www.scinomix.com/all-products/96-pin-replicator/ | |
| HiGro high-capacity, incubating shaker | Digilab | http://www.digilabglobal.com/higro | |
| Multidrop Combi Reagent Dispenser | Titertrek | http://groups.molbiosci.northwestern.edu/hta/titertek.htm | |
| Biomek FX AP96 Automated Workstation | Beckman Coulter | http://groups.molbiosci.northwestern.edu/hta/biomek_multi.htm | |
| Innova44 shaker | New Brunswick | http://www.eppendorf.com/int///index.php?sitemap=2.3&pb=d78efbc05310ec 04&action=products&contentid=1& catalognode=83389 | |
| M205 FA | Leica | http://www.leica-microsystems.com/de/produkte/stereomikroskope-makroskope/fluoreszenz/details/product/leica-m205-fa/ | |
| ORCA-R2 C10600-10BDigital CCD camera | Hamamatsu | http://www.hamamatsu.com/jp/en/community/life_science_camera/product/search/C10600-10B/index.html | |
| Spinning Disc AF Confocal Microscope | Leica | http://www.leica-microsystems.com/products/light-microscopes/life-science-research/fluorescence-microscopes/details/product/leica-sd-af/ | |
| Falcon 4M60 camera | Teledyne Dalsa | http://www.teledynedalsa.com/imaging/products/cameras/area-scan/falcon/PT-41-04M60/ | |
| Software | |||
| MetaMorph Microscopy Automation & Image Analysis Software | Molecular Devices | http://www.moleculardevices.com/products/software/meta-imaging-series/metamorph.html | |
| Hamamatsu SimplePCI Image Analysis Software | Meyer Instruments | http://meyerinst.com/imaging-software/hamamatsu/index.htm | |
| ImageJ | NIH | http://rsbweb.nih.gov/ij/download.html | |
| wrMTrck plugin for ImageJ | http://www.phage.dk/plugins/wrmtrck.html | ||
| C. elegans strains | |||
| N2 (WT) | Caenorhabditis Genetics Center (CGC) | http://www.cgc.cbs.umn.edu/strain.php?id=10570 | |
| AM815 rmIs323[myo-3p::sup35(r2e2)::rfp] | Morimoto lab | available from our laboratory | |
| See table 1 for a source for folding sensor and stress reporter strains |
Referenzen
- Prusiner, S. B. Novel proteinaceous infectious particles cause scrapie. Science. 216 (4542), 136-144 (1982).
- Jarrett, J. T., Lansbury, P. T. Seeding 'one-dimensional crystallization' of amyloid: a pathogenic mechanism in Alzheimer's disease and scrapie. Cell. 73 (6), 1055-1058 (1993).
- Caughey, B., Kocisko, D. A., Raymond, G. J., Lansbury, P. T. Aggregates of scrapie-associated prion protein induce the cell-free conversion of protease-sensitive prion protein to the protease-resistant state. Chem Biol. 2 (12), 807-817 (1995).
- Wickner, R. B. [URE3] as an altered URE2 protein: evidence for a prion analog in Saccharomyces cerevisiae. Science. 264 (5158), 566-569 (1994).
- Chien, P., Weissman, J. S., DePace, A. H. Emerging principles of conformation-based prion inheritance. Annu Rev Biochem. 73, 617-656 (2004).
- Kimberlin, R. H., Walker, C. A. Pathogenesis of mouse scrapie: patterns of agent replication in different parts of the CNS following intraperitoneal infection. J R Soc Med. 75 (8), 618-624 (1982).
- Beekes, M., McBride, P. A., Baldauf, E. Cerebral targeting indicates vagal spread of infection in hamsters fed with scrapie. J Gen Virol. 79 (3), 601-607 (1998).
- Jucker, M., Walker, L. C. Self-propagation of pathogenic protein aggregates in neurodegenerative diseases. Nature. 501 (7465), 45-51 (2013).
- Aguzzi, A. Cell biology: Beyond the prion principle. Nature. 459 (7249), 924-925 (2009).
- Scherzinger, E., et al. Self-assembly of polyglutamine-containing huntingtin fragments into amyloid-like fibrils: implications for Huntington's disease pathology. Proc Natl Acad Sci U S A. 96 (8), 4604-4609 (1999).
- Wood, S. J., et al. alpha-synuclein fibrillogenesis is nucleation-dependent. Implications for the pathogenesis of Parkinson's disease. J Biol Chem. 274 (28), 19509-19512 (1999).
- Wang, Y. Q., et al. Relationship between prion propensity and the rates of individual molecular steps of fibril assembly. J Biol Chem. 286 (14), 12101-12107 (2011).
- Cushman, M., Johnson, B. S., King, O. D., Gitler, A. D., Shorter, J. Prion-like disorders: blurring the divide between transmissibility and infectivity. J Cell Sci. 123 (8), 1191-1201 (2010).
- Tanaka, M., Collins, S. R., Toyama, B. H., Weissman, J. S. The physical basis of how prion conformations determine strain phenotypes. Nature. 442 (7102), 585-589 (2006).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. J Struct Biol. 179 (2), 152-160 (2012).
- Ilieva, H., Polymenidou, M., Cleveland, D. W. Non-cell autonomous toxicity in neurodegenerative disorders: ALS and beyond. J Cell Biol. 187 (6), 761-772 (2009).
- Nussbaum-Krammer, C. I., Morimoto, R. I. Caenorhabditis elegans as a model system for studying non-cell-autonomous mechanisms in protein-misfolding diseases. Dis Model Mech. 7 (1), 31-39 (2014).
- Lino, M. M., Schneider, C., Caroni, P. Accumulation of SOD1 mutants in postnatal motoneurons does not cause motoneuron pathology or motoneuron disease. J Neurosci. 22 (12), 4825-4832 (2002).
- Li, J. Y., et al. Lewy bodies in grafted neurons in subjects with Parkinson's disease suggest host-to-graft disease propagation. Nat Med. 14 (5), 501-503 (2008).
- Desplats, P., et al. Inclusion formation and neuronal cell death through neuron-to-neuron transmission of alpha-synuclein. Proc Natl Acad Sci U S A. 106 (31), 13010-13015 (2009).
- Clement, A. M., et al. Wild-type nonneuronal cells extend survival of SOD1 mutant motor neurons in ALS mice. Science. 302 (5642), 113-117 (2003).
- Gu, X., et al. Pathological cell-cell interactions elicited by a neuropathogenic form of mutant Huntingtin contribute to cortical pathogenesis in HD mice. Neuron. 46 (3), 433-444 (2005).
- Yamanaka, K., et al. Mutant SOD1 in cell types other than motor neurons and oligodendrocytes accelerates onset of disease in ALS mice. Proc Natl Acad Sci U S A. 105 (21), 7594-7599 (2008).
- Garden, G. A., et al. Polyglutamine-expanded ataxin-7 promotes non-cell-autonomous purkinje cell degeneration and displays proteolytic cleavage in ataxic transgenic mice. J Neurosci. 22 (12), 4897-4905 (2002).
- Raeber, A. J., et al. Astrocyte-specific expression of hamster prion protein (PrP) renders PrP knockout mice susceptible to hamster scrapie. EMBO J. 16 (20), 6057-6065 (1997).
- Yazawa, I., et al. Mouse model of multiple system atrophy alpha-synuclein expression in oligodendrocytes causes glial and neuronal degeneration. Neuron. 45 (6), 847-859 (2005).
- Lobsiger, C. S., Cleveland, D. W. Glial cells as intrinsic components of non-cell-autonomous neurodegenerative disease. Nat Neurosci. 10 (11), 1355-1360 (2007).
- Sambataro, F., Pennuto, M. Cell-autonomous and non-cell-autonomous toxicity in polyglutamine diseases. Prog Neurobiol. 97 (2), 152-172 (2012).
- Polymenidou, M., Cleveland, D. W. Prion-like spread of protein aggregates in neurodegeneration. J Exp Med. 209 (5), 889-893 (2012).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nat Rev Mol Cell Biol. 11 (4), 301-307 (2010).
- Braak, H., Braak, E., Bohl, J. Staging of Alzheimer-related cortical destruction. Eur Neurol. 33 (6), 403-408 (1993).
- Meyer-Luehmann, M., et al. Exogenous induction of cerebral beta-amyloidogenesis is governed by agent and host. Science. 313 (5794), 1781-1784 (2006).
- Luk, K. C., et al. Pathological alpha-synuclein transmission initiates Parkinson-like neurodegeneration in nontransgenic mice. Science. 338 (6109), 949-953 (2012).
- Clavaguera, F., et al. Transmission and spreading of tauopathy in transgenic mouse brain. Nat Cell Biol. 11 (7), 909-913 (2009).
- Nonaka, T., et al. Prion-like Properties of Pathological TDP-43 Aggregates from Diseased Brains. Cell Rep. 4 (1), 124-134 (2013).
- Lundmark, K., et al. Transmissibility of systemic amyloidosis by a prion-like mechanism. Proc Natl Acad Sci U S A. 99 (10), 6979-6984 (2002).
- Lai, C. H., Chou, C. Y., Ch'ang, L. Y., Liu, C. S., Lin, W. Identification of novel human genes evolutionarily conserved in Caenorhabditis elegans by comparative proteomics. Genome Res. 10 (5), 703-713 (2000).
- Xu, X., Kim, S. K. The early bird catches the worm: new technologies for the Caenorhabditis elegans toolkit. Nat Rev Genet. 12 (11), 793-801 (2011).
- Boulin, T., Hobert, O. From genes to function: the C. elegans genetic toolbox. Wiley Interdiscip Rev Dev Biol. 1 (1), 114-137 (2012).
- Nussbaum-Krammer, C. I., Park, K. W., Li, L., Melki, R., Morimoto, R. I. Spreading of a prion domain from cell-to-cell by vesicular transport in Caenorhabditis elegans. PLoS Genet. 9 (3), e1003351 (2013).
- Chernoff, Y. O., Lindquist, S. L., Ono, B., Inge-Vechtomov, S. G., Liebman, S. W. Role of the chaperone protein Hsp104 in propagation of the yeast prion-like factor [psi. Science. 268 (5212), 880-884 (1995).
- Liu, J. J., Lindquist, S. Oligopeptide-repeat expansions modulate 'protein-only' inheritance in yeast. Nature. 400 (6744), 573-576 (1999).
- Halfmann, R., et al. Prions are a common mechanism for phenotypic inheritance in wild yeasts. Nature. 482 (7385), 363-368 (2012).
- Tyedmers, J., Madariaga, M. L., Lindquist, S. Prion switching in response to environmental stress. PLoS Biol. 6 (11), e294 (2008).
- Krammer, C., et al. The yeast Sup35NM domain propagates as a prion in mammalian cells. Proc Natl Acad Sci U S A. 106 (2), 462-467 (2009).
- Hofmann, J. P., et al. Cell-to-cell propagation of infectious cytosolic protein aggregates. Proc Natl Acad Sci U S A. 110 (15), 5951-5956 (2013).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , (2006).
- Berkowitz, L. A., Knight, A. L., Caldwell, G. A., Caldwell, K. A. Generation of Stable Transgenic C. elegans Using Microinjection. J. Vis. Exp. (18), e833 (2008).
- Evans, T. C. Transformation and microinjection. WormBook. , (2006).
- Shaham, S. Methods in cell biology. Wormbooks. , (2006).
- Kim, E., Sun, L., Gabel, C. V., Fang-Yen, C. Long-term imaging of Caenorhabditis elegans using nanoparticle-mediated immobilization). PLoS One. 8 (1), e53419 (2013).
- Fay, D. Genetic mapping and manipulation: Chapter 1-Introduction and basics. WormBook. , (2006).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30 (4), 313-321 (2003).
- Rual, J. F., et al. Toward improving Caenorhabditis elegans phenome mapping with an ORFeome-based RNAi library. Genome Res. 14 (10B), 2162-2168 (2004).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Kern, A., Ackermann, B., Clement, A. M., Duerk, H., Behl, C. HSF1-controlled and age-associated chaperone capacity in neurons and muscle cells of C. elegans. PLoS One. 5 (1), e8568 (2010).
- Becker, J., Walter, W., Yan, W., Craig, E. A. Functional interaction of cytosolic hsp70 and a DnaJ-related protein, Ydj1p, in protein translocation in vivo. Mol Cell Biol. 16 (8), 4378-4386 (1996).
- Salvaterra, P. M., McCaman, R. E. Choline acetyltransferase and acetylcholine levels in Drosophila melanogaster: a study using two temperature-sensitive mutants. J Neurosci. 5 (4), 903-910 (1985).
- Goloubinoff, P., Mogk, A., Zvi, A. P., Tomoyasu, T., Bukau, B. Sequential mechanism of solubilization and refolding of stable protein aggregates by a bichaperone network. Proc Natl Acad Sci U S A. 96 (24), 13732-13737 (1999).
- Schroder, H., Langer, T., Hartl, F. U., Bukau, B. D. n. a. K. DnaJ and GrpE form a cellular chaperone machinery capable of repairing heat-induced protein damage. EMBO J. 12 (11), 4137-4144 (1993).
- Rampelt, H., et al. Metazoan Hsp70 machines use Hsp110 to power protein disaggregation. EMBO J. 31 (21), 4221-4235 (2012).
- Gupta, R., et al. Firefly luciferase mutants as sensors of proteome stress. Nat Methods. 8 (10), 879-884 (2011).
- Gidalevitz, T., Ben-Zvi, A., Ho, K. H., Brignull, H. R., Morimoto, R. I. Progressive disruption of cellular protein folding in models of polyglutamine diseases. Science. 311 (5766), 1471-1474 (2006).
- Ben-Zvi, A., Miller, E. A., Morimoto, R. I. Collapse of proteostasis represents an early molecular event in Caenorhabditis elegans aging. Proc Natl Acad Sci U S A. 106 (35), 14914-14919 (2009).
- Karady, I., et al. Using Caenorhabditis elegans as a model system to study protein homeostasis in a multicellular organism. J Vis Exp. (82), e50840 (2013).
- Gidalevitz, T., Krupinski, T., Garcia, S., Morimoto, R. I. Destabilizing protein polymorphisms in the genetic background direct phenotypic expression of mutant SOD1 toxicity. PLoS Genet. 5 (3), e1000399 (2009).
- Morley, J. F., Brignull, H. R., Weyers, J. J., Morimoto, R. I. The threshold for polyglutamine-expansion protein aggregation and cellular toxicity is dynamic and influenced by aging in Caenorhabditis elegans. Proc Natl Acad Sci U S A. 99 (16), 10417-10422 (2002).
- Brignull, H. R., Moore, F. E., Tang, S. J., Morimoto, R. I. Polyglutamine proteins at the pathogenic threshold display neuron-specific aggregation in a pan-neuronal Caenorhabditis elegans model. J Neurosci. 26 (29), 7597-7606 (2006).
- Mohri-Shiomi, A., Garsin, D. A. Insulin signaling and the heat shock response modulate protein homeostasis in the Caenorhabditis elegans intestine during infection. J Biol Chem. 283 (1), 194-201 (2008).
- Libina, N., Berman, J. R., Kenyon, C. Tissue-specific activities of C. elegans DAF-16 in the regulation of lifespan. Cell. 115 (4), 489-502 (2003).
- Schatzl, H. M., et al. A hypothalamic neuronal cell line persistently infected with scrapie prions exhibits apoptosis. J Virol. 71 (11), 8821-8831 (1997).
- Keith, S. A., Amrit, F. R., Ratnappan, R., Ghazi, A. The C. elegans healthspan and stress-resistance assay toolkit. Methods. , (2014).
- Pierce-Shimomura, J. T., et al. Genetic analysis of crawling and swimming locomotory patterns in C. elegans. Proc Natl Acad Sci U S A. 105 (52), 20982-20987 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten