Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un modelo de la célula endotelial planar for Imaging inmunológica Synapse Dinámica
En este artículo
Resumen
Adaptive immunity is controlled by dynamic 'immunological synapses' formed between T cells and antigen presenting cells. This protocol describes methods for investigating endothelial cells both as understudied physiologic APCs and as a novel type of 'planar cellular APC model'.
Resumen
La inmunidad adaptativa está regulado por las interacciones dinámicas entre las células T y las células presentadoras de antígeno ("APC") refiere como 'sinapsis inmunológicas. Dentro de estas interfaces de célula-célula íntimos grupos subcelulares discretos de MHC / Ag-TCR, F-actina, adhesión y moléculas de señalización forman y remodelar rápidamente. Estas dinámicas se cree que son determinantes críticos de la eficiencia y la calidad de las respuestas inmunes que se desarrollan y, por tanto, de protección contra la inmunidad patológica. El conocimiento actual de las sinapsis inmunológicas con fisiológica APC está limitada por la insuficiencia de la resolución de las imágenes se puede obtener. Aunque los modelos de sustratos artificiales (por ejemplo, bicapas lipídicas planas) ofrecen una excelente resolución y han sido herramientas muy valiosas, que son inherentemente no fisiológica y simplificada. Las células endoteliales vasculares y linfáticos se han convertido en un importante tejido periférico (o estroma) Compartimiento de "semi-profesionalal APC '. Estas APCs (que expresan la mayor parte de la maquinaria molecular de APCs profesional) tienen la característica única de la formación de la superficie celular prácticamente planar y están fácilmente transfectable (por ejemplo, con los reporteros fluorescentes de proteínas). Aquí un enfoque básico para implementar las células endoteliales como una novela y fisiológica 'planar modelo APC celular' para mejorar la formación de imágenes y el interrogatorio de los procesos de señalización antigénicos fundamentales se describirá.
Introducción
Los linfocitos T son una rama del sistema inmune adaptativo que se caracteriza por la capacidad de reconocer eficientemente péptido antígeno (Ag) unida a complejo mayor de histocompatibilidad (MHC) moléculas a través de sus receptores de células T (TCR) 1. Linfocitos ingenuos constitutivamente migran y escanear 'Ag células presentadoras profesionales' (APC; por ejemplo, células dendríticas) en los ganglios linfáticos, mientras que las células de memoria / T efectoras necesitan para estudiar con eficacia una muy amplia gama de vehículos blindados y potenciales células diana dentro de los tejidos periféricos.
En el min tras el reconocimiento inicial de cognado Ag en un APC, linfocitos detener su migración y comienzan a formar una interfaz célula-célula íntima especializado denominado 'sinapsis inmunológica "(IS). Sostenida (es decir, 30 a 60 min) ES se requieren contactos para ampliar y mantener la señalización 2-7. Estudios emergentes identificar que dentro del IS, que es la formación continua y rápida remodeling de señalización subcelular micro-grupos discretos (es decir, que contienen MHC / Ag-TCR, F-actina, la adhesión y moléculas de señalización) que determinan la fuerza y la calidad de la que resulta la respuesta inmune 2-7. Sin embargo, los detalles dinámicos y mecanismo de regulación de este proceso se conocen por completo 8,9. Esto se debe en gran parte de los desafíos técnicos asociados con topologías irregulares de las superficies de APC y la orientación de un mal control de los planos de interacción célula-célula, las cuestiones que limitan profundamente la imagen espacio-temporal necesaria acerca 10.08 (Figure1A).
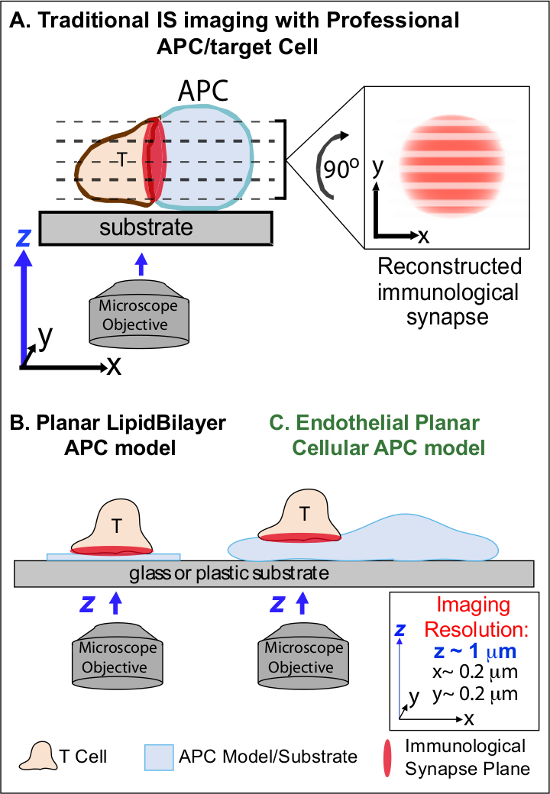
Figura 1. Un fisiológica planar Modelo APC Cell for Imaging inmunológica Synapse Dynamics. El esquema ilustra la imagen tradicional de la sinapsis inmunológica entre una célula T y un professio nal de células APC (A) y T y un modelo tradicional de lípidos planar APC bicapa (B) en comparación con este novedoso modelo de APC planar endotelial (C). APCs profesionales proporcionan sinapsis inmunológicas fisiológicas pero ofrecen interfaz mal orientada célula-célula (es decir, con respecto al plano óptima de imagen xy; resolución ~ 0,2 micras), lo que compromete drásticamente espacial (z plano de la imagen de resolución ~ 1 m) y temporal (es decir, debido a la necesidad de escanear repetidamente a través de todos los planos de formación de imágenes z) resolución de la imagen. Modelos bicapa tienen una topología plana que ofrece óptima de imágenes de resolución espacio-temporal, pero también son muy simplificada, no fisiológico y rígido. Este modelo de células endoteliales combina la topología planar de bicapas lipídicas con el sustrato fisiológico de un APC clásico para entregar óptima de imágenes de resolución espacial y temporal en un entorno fisiológico.m / files / ftp_upload / 53288 / 53288fig1large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
El trabajo previo ha eludido parcialmente estos obstáculos mediante el desarrollo de modelos sustrato plano (es decir, bicapas lipídicas y las superficies recubiertas de anticuerpo) que proporcionan resolución espacio-temporal óptima (es decir, a través de la fijación de la superficie de activación de las células T en un único plan que es paralela a la proyección de imagen óptima xy avión) 11 a 15 (Figura 1B). Estos modelos han facilitado importantes conocimientos sobre la dinámica subcelulares / moleculares que controlan la señalización antigénico en las células T, incluyendo el descubrimiento de actina dinámica / TCR señalización micro-clusters 7,11-14. Sin embargo, tales modelos están inherentemente simplificaron, así como rígido (que impide el desarrollo / estudio de características topológicas 3 dimensiones) (Figura 1B). Por lo tanto, sigue siendo incierto cómo relacionar estos hallazgos a physiologic célula-célula vigilancia inmune.
Aunque todavía poco estudiado, vascular y las células endoteliales linfáticas están emergiendo como un grande (es decir, mayor en número que todos los vehículos blindados profesionales, por ~ 1000 veces) compartimento periférico de "semi-profesional" APC 16-18. Estas células expresan MHC-I, MHC-II- y una multitud de moléculas co-estimuladoras (por ejemplo, CD40, LFA3, ICOSL, el 4-1BB, OX40L, TL1A, PD-L1; pero no CD80 y CD86) y son estratégicamente posicionado en la interfase sangre-tejido donde sirven funciones especializadas centinela 16-18. Estudios previos demostraron que las células endoteliales pueden efectivamente re-estimular efectoras / memoria, pero no ingenuo, las células T 19-25. Así, las células endoteliales es probable que desempeñen un papel único de APC en la fase efectora de la respuesta inmune adaptativa dentro de los tejidos periféricos, tales como la influencia local sobre la activación de células T, la diferenciación, la memoria y la tolerancia 16-17,26. Cricamente, cuando se cultivan in vitro, las células endoteliales forman superficies celulares prácticamente planas y están fácilmente transfectable (por ejemplo, con los reporteros fluorescentes de proteínas). Estas características son ideales para imágenes de alta resolución espacio-temporal de la dinámica topológicos durante las interacciones célula-célula 19,27. Por lo tanto las células endoteliales podrían servir como un 'celular plana APC modelo fisiológica claramente adecuado para el estudio de los mecanismos de remodelación subcelulares / molecular que impulsan el reconocimiento de antígenos y regulan las respuestas (Figura 1C) 19,20.
Previamente establecidos técnicas de imagen complementarios (incluyendo la transfección de las células endoteliales con los fabricantes de proteínas fluorescentes de la membrana plasmática y citosol) para estudiar los detalles de la interacción de leucocitos endotelial durante la adhesión y migración transendotelial 27, mostraron que los leucocitos sondean activamente la superficie del endotelio por Dynamic una inserciónd retracción de la sub-escala micrométrica, protuberancias cilíndricas de actina-ricos (~ 200-1.000 nm de diámetro y profundidad) denomina-invadosome como salientes (es decir, 'ILPS') 27,28. Estos enfoques de imagen se han ampliado aún más, junto con la creación de protocolos para tomar ventaja de la función endotelial de APC para desarrollar los primeros métodos de alta resolución de imagen espacio-temporal de la célula endotelial sinapsis inmunológica T según lo informado 19,20 y describir con más detalle en el presente documento. Un hallazgo central derivado de esta novela plana celular modelo de APC es que programas de vida independiente de células T funcionan tanto en la promoción de la detección inicial Ag y en el mantenimiento de la señalización posterior. De hecho, las matrices de múltiples programas de vida independiente (que fueron estabilizados y devengados en respuesta a sus iniciales en el flujo de calcio) muestran un enriquecimiento en TCR y moléculas sugestivos de señalización activa tal PKC-Q, ZAP-70, fosfotirosina y HS1. Por lo tanto, programas de vida independiente parecen representar un equivalente fisiológico tridimensional a la micro TCR de señalizaciónracimos visto en los modelos de dos capas planas. Este enfoque, por lo tanto, revela sensibilidad / informes dinámica molecular y arquitectónicos (y biomecánico implícitas) de otro modo no detectable.
El método descrito en este documento deberá ser útil para reducir la brecha entre los modelos de sustratos artificiales APC APC profesional y con el fin de mejorar nuestra capacidad de interrogar a los mecanismos básicos de la respuesta inmune adaptativa. Mientras que aquí la atención se centra en la activación de tipo Th1 CD4 + efectoras / célula de memoria, este enfoque básico puede ser modificado fácilmente para estudiar una amplia gama de tipos de células T y Ags, como se discute a continuación.
Protocolo
Todos los experimentos descritos en este protocolo se llevan a cabo con las células T humanas primarias y células endoteliales disponibles comercialmente primarios humanos (dérmica o microvascular pulmonar EC) protocolo de investigación Cualquie en seres humanos debe ser aprobada por una junta de revisión institucional y el consentimiento informado por escrito se debe proporcionar de cada donante de sangre. Los experimentos llevados a cabo usando este protocolo fue aprobado por el IRB de Beth Israel Deaconess Medical Center.
1. Preparación de células CD4 + Th1 Humano efector / T de memoria
- Aplicar torniquete en el brazo del donante, limpie la vena con el alcohol, e insertar la aguja. Lentamente dibujar 15 ml de sangre en vacutainer utilizando EDTA como anticoagulante. Cuando la sangre se ha elaborado, desate el torniquete antes de retirar la aguja. Inmediatamente aplicar presión a la herida con gasa estéril cuando se retira la aguja.
- Transferencia de sangre a un tubo de 50 ml. Añadir RPMI-1640 a temperatura ambiente en una dilución 1: 1 (volumen final 30 ml). Superponer con cuidado el diluidod sangre en dos tubos de 50 ml que contienen 15 ml de medio de aislamiento pre-filtrada linfocitos tales como Ficoll-Paque a TA.
- Centrifugar el gradiente a TA durante 30 min a 1.200 xg en un rotor de cubeta oscilante. Mientras centrifugación preparar el medio de células T (500 ml de RPMI-1640, 50 ml de FCS, 5 ml de penicilina / estreptomicina).
- Tras la retirada del tubo de 50 ml de la centrífuga, observar cuatro capas: un sedimento de células rojas de la sangre en la parte inferior, el paque, una capa de células que contiene células blancas de la sangre (incluyendo linfocitos) y el plasma. Retire cuidadosamente la capa de células blancas de la sangre con una pipeta pasteur y la transferencia en un tubo Falcon 50 ml.
- Se lava la capa de células blancas de la sangre mediante la adición de RT RPMI-1460 (hasta 20 ml) y centrifugando a TA durante 5 min a 1200 x g. Resuspender las células blancas de la sangre en 1 ml de medio de células T. Añadir 5 l de la suspensión 1 ml de células a 250 l medio de células T en un tubo de 1,5 ml de centrífuga y mezclar suavemente arriba y abajo con una pipeta.
- Añadir 25 l diluird suspensión celular a 25 l 0,4% de azul de tripano. Añadir 10 l de mezcla a cada lado de un hemocitómetro estándar.
- Coloque en un hemocitómetro bajo microscopio de luz de energía. El uso de un objetivo de 10X, cuente el número de células que han excluido del colorante azul de tripano y están presentes en la plaza central de ambos lados del hemocitómetro vivir.
- Para el cálculo de la concentración de células, multiplicar el promedio de las 2 plazas por 100 (factor de dilución) y luego se multiplica por 10 4 para dar el número de células / ml.
- Ajustar la concentración final de 0,5 x 10 6 células / ml en medio de células T. Añadir una concentración final de 1 mg / ml de cada uno de los superantígenos bacterianos enterotoxina estafilocócica B (SEB) y síndrome de shock tóxico de la toxina 1 (TSST) a las células. Cultura durante 72 horas (37 ° C y 5% de CO 2) para ampliar la población de células T CD4 +.
- Pellet células T (1.200 xg, 5 min) y se resuspenden en 0.5 x 10 6 células / ml en medio de células T con laAdemás de la IL-15 (20 ng / ml). Transferencia de los linfocitos a un matraz T150. Continuar ampliar / dividir celdas en pleno medio-IL-15 cada 24 a 48 horas, según sea necesario (basado en el color de los medios de comunicación, es decir, cada vez que los medios de comunicación resulta de rosa a ligeramente amarillo) a partir de entonces. Mantener la población de linfocitos que resulta para un máximo de 15 días.
NOTA: Por diseño, este protocolo se activará y ampliar específicamente subconjunto de células T CD4 + que son reactivos a SEB y TSST y luego conducirlos hacia un fenotipo efector / memoria tipo Th1. Otras células blancas de la sangre no pueden sobrevivir y crecer en estas condiciones, de tal manera que a paso 1.10 las células serán al menos 95% CD4 +, T CD45RO + células 19, como puede ser fácilmente evaluada por citometría de flujo. Si se desea, purificación adicional se puede conseguir fácilmente a través de kits de selección positivos o negativos basados en anticuerpos disponibles comercialmente / magnético de talón.
2. A partir primaria endoteliales humanas Cell Culture
- Escudo un matraz T25 con fibronectina (FN) 20 g / ml en PBS en condiciones estériles. Deja a temperatura ambiente durante 30 a 60 min. Quitar FN y añadir 5 ml de medio completo (medio basal endotelial (EBM-2) medio suplementado con medio de crecimiento endotelial (EGM-2) SingleQuots). Pre-incubado en 37 ° C incubadora de cultivo celular durante al menos 30 min.
- Descongelar un vial de congelados de pulmón o dérmica microvasculares humana células endoteliales (HLMVECs o HDMVECs) en un baño de agua a 37 ° C con agitación suave ocasional para ~ 2-3 min. Transferir inmediatamente células para matraz T25 que contiene los medios de comunicación pre-calentado. Girar y colocar en la incubadora a 37 ° C con cuidado.
- Cambie los medios de comunicación después de ~ 4.6 hr. Continuar para cambiar medios aproximadamente cada 48 horas (o cuando los medios de comunicación se vuelve ligeramente amarillo) hasta que la placa alcanza ~ 90-95% de confluencia.
3. La división de General y expansión de las células endoteliales
- Crecer células para el 90-95 ~ confluencia. Este proceso puede tardar 2-5 días. Para la división,retirar el medio y enjuagar con PBS. Retire PBS y reemplazarlo con un volumen mínimo de tripsina 1x fresco (0,5 ml para T25 o 1.5 ml para T75). Hacer girar suavemente para cubrir toda la superficie con tripsina. Incubar a 37 ° C durante ~ 5 min. Supervisar el desprendimiento de las células de la placa utilizando un microscopio de luz de bajo poder.
- Cuando mayoría de células aparecen redondeada o extraído, añadir 5 volumen (es decir, en comparación con el volumen añadido de tripsina) de medio de pre-calentado completa EGM-2 y la pipeta suavemente sobre la superficie del matraz para separar todas las células.
- Recuento de células endoteliales con un hemocitómetro como se describe en 1.6 a 1.7. Sedimentar las células por centrifugación (5 min, 1.200 xg). Eliminar el sobrenadante. Ajustar la concentración a 0,5 millones de células por ml mediante la adición de EGM-2 completa MV medios pre-calentado.
- Transferir alícuotas de las células a los platos o matraces recubiertos-FN apropiadas para el mantenimiento. Hacer girar suavemente y colocar en la incubadora. Cambie los medios de comunicación dentro de 6 a 12 horas de la siembra. Sho MediosULD ser cambiado aproximadamente cada 48 h a partir de entonces.
4. endotelial transfección de células
NOTA: Primaria células endoteliales son refractarios a la transfección por la mayoría de los métodos químicos y electroporación común. El método basado en la transfección nuclear se describe a continuación permite la relativamente alta eficacia de transfección (~ 50-70%). Un método alternativo eficaz es el uso de la infección por vectores virales apropiados (véanse los comentarios en la Tabla Materiales).
- Preparar matraces T25 o T75 (según sea necesario) de HLMVECs o HDMVECs a una densidad final de 90-95% confluency.Coat con fibronectina (FN) 20 ug / ml en PBS en condiciones estériles o bien placas de cultivo de microscopio tales como placas de Delta-T (por paso 5) o 12 mm cubreobjetos de vidrio circulares colocados dentro de un pocillo de una placa de cultivo celular de 24 pocillos (para el paso 6) con como se describió anteriormente (2,1).
- Añadir 1 ml de EGM-2 completa medios de cultivo a la cultura microscopio platesor 0,5 ml a cada uno de 24 pocillos y equilibrar en placasun humidificado 37 ° C / 5% de CO 2 incubadora.
- Cosecha y contar las células endoteliales como en los pasos 3.1-3.3.Centrifuge el volumen requerido de células (0,5 millones de células por muestra) a 1200 xg durante 5 min a TA. Resuspender el sedimento celular cuidadosamente en 100 l solución de transfección nuclear RT por muestra.
- Combinar 100 l de suspensión celular con 1-5 g de ADN. Traslado celular / suspensión ADN en cubeta certificada; muestra debe cubrir el fondo de la cubeta sin burbujas de aire.
NOTA: Las construcciones diana YFP o DsRed a la membrana celular (a través de los N-terminales 20 aminoácidos de neuromodulin que contiene una señal para palmitoylation postraduccional) se utilizaron solo (sola membrana-YFP o membrana-DsRed solo) o co-transfectadas con un marcador volumétrica citoplasmática (por ejemplo, la membrana-YFP y DsRed soluble). Muchos permutaciones de los marcadores de proteínas fluorescentes se pueden utilizar. - Cierre la cubeta con su tapa. Inserte la cubeta consuspensión de células / ADN en el soporte de cubetas del electroporador y aplicar programas electroporación S-005. Tome la cubeta del soporte una vez finalizado el programa.
- Añadir ~ 500 l de los medios de cultivo pre-equilibrado a la cubeta y retirar suavemente la suspensión de células de la cubeta usando las pipetas de transferencia de plástico proporcionados en el kit de transfección nuclear.
- Para los experimentos utilizando placas de cultivo de microscopio partición de la suspensión de células de una reacción igualmente entre dos platos que contienen los medios de comunicación pre-calentado (Pasos 4,2-4,3). Para experimentos con 24 pocillos / placa, una partición de reacción igual entre 3 pozos.
- Se incuban las células en un humidificado 37 ° C / 5% de CO 2 incubadora y cambiar los medios de comunicación 4-6 horas, y de nuevo en 12-16 horas después de la transfección.
5. Imágenes de células vivas y Análisis
- Endotelio Preparación
- Día 0: cotransfectar HLMVECs primarias con membrana-YFP y DsRed soluble a través de un nucleofetecnología cción como se describe en el paso 4 y la placa en placas de cultivo de células de imágenes en directo.
- Día 1: Reemplazar medio con medio fresco que contiene IFN-γ (100 ng / ml) para inducir la expresión de MHC-II. En el Día 2. Estimular las células transfectadas mediante la adición de 20 ng ml de TNF-α / a los medios de comunicación existentes.
- En el Día 3, se incuba el endotelio con 1 g / ml cada uno de los superantígenos bacterianos enterotoxina estafilocócica B (SEB) y la toxina del síndrome de choque tóxico 1 (TSST) a 37 ° C durante 30-60 minutos inmediatamente antes de los experimentos. Omita este paso para las condiciones de control '-ag'.
- Preparación de linfocitos
- En paralelo con el paso 5.1.3, preparar Buffer A (libre de rojo fenol HBSS) suplementado con Hepes 20 mM, pH 7,4 y 0,5% v / v de albúmina de suero humano pre-calienta a 37 DO. Tomar una muestra de linfocitos cultivados y determinar la densidad contando con un hemocitómetro (Paso 1.12 a 1.17).
- Centrifugar 2 millones de células por muestra de 1,200 xg durante 5 min a temperatura ambiente en un tubo cónico de 15 ml. Medios Aspirar y sedimento celular suavemente resuspender en 2 ml de Tampón A de tal manera que no hay grupos de células permanecen.
- Retirar un alícuota fresca de Fura-2 tinte de calcio y resuspender en DMSO para hacer una concentración madre de 1 mM.
- Añadir 2 l de Fura-2 stock solución a la suspensión de células T (2 mM de concentración final), tapar el tubo y mezclar inmediatamente invirtiendo el tubo para asegurar una dispersión de tinte. Incubar a 37 ° C durante 30 min.
- Centrifugar como en el paso 5.2.2. Aspirar Buffer-A y linfocitos suave pero completamente resuspender en 20 a 40 l de fresca Buffer-A.
- Configuración de Células vivas imágenes y Adquisición
NOTA: Una amplia variedad de sistemas puede ser empleado para imágenes de fluorescencia de células vivas en los microscopios de luz verticales e invertidos. Los requisitos básicos son una fuente de fluorescencia luz y filtros, una cámara CCD, la conmutación de filtro motorizado y persianas, una etapa calienta (o Microsccámara caliente ope-montable) y software para la adquisición de imágenes automatizado. Para este protocolo de alta apertura numérica, de gran aumento (es decir, 40X, 63X) se requieren lentes de inmersión en aceite para lograr la resolución espacial necesaria. Especial cuidado debe tenerse en la elección de la fuente de fluorescencia apropiado y lentes de imágenes de calcio a base de Fura-2 ya que no todos son compatibles con las longitudes de onda de excitación necesarias 340/380 nm. Un enfoque alternativo (compatible con conjuntos de verde y rojo estándar de filtro de fluorescencia) se puede utilizar con los colorantes de calcio sensibles no ratiomentic (por ejemplo, Fluo-4, Rhod-3), aunque éstas no pueden cuantificar con precisión el flujo de calcio y sólo proporcionan un pariente / lectura cuantitativa.- Encienda el sistema de microscopio (PC para la operación, microscopio, cámara CCD, rueda de filtros y la lámpara de xenón).
- Abra el software designada.
- Establecer microscopio / software para time-lapse multicanal automatizada. Incluya adquisición secuencial de anunciocontraste ifferential interferencia (DIC), fluorescencia verde estándar, fluorescencia roja estándar y estándar de excitación 340 y 380 nm Fura-2 imágenes. Establezca el intervalo para la adquisición de 10 a 30 segundos y una duración total de ~ 20 a 60 min.
- Establecer tiempos de exposición para Fura-2 de formación de imágenes.
- Añadir aceite de objetivo fresco y montar un plato de microscopio que contiene sólo 0,5 ml de Tampón-A en el adaptador etapa de calentamiento y gire inmediatamente a equilibrar a 37 ° C (se llevará a ~ 2-3 min).
- Añadir descansando linfocitos cargados-Fura2 a la cámara de plato microscopio montado usando una pipeta de 20 l.
- Encienda imágenes de campo claro. Seleccione la trayectoria de la luz a los oculares. Utilice el botón de enfoque grueso para llevar objetivo en contacto con la parte inferior del plato miscoscope. Utilice el ocular y la perilla de enfoque fino para centrarse en las células T asentado en el fondo del plato.
- Utilice los controles de etapa xy para seleccionar un campo que contiene al menos 10 células. Evitardemasiado concurrida campos y grupos de células ya que estos crean artefactos de imagen.
- Cambiar de campo claro a la fuente de luz fluorescente. Cambiar de imagen ocular para cámara CCD. Establecer los parámetros de adquisición (por ejemplo, tiempo de exposición, ganancia del detector y binning). Usando el software de adquisición y descansando Fura2-340 imágenes Fura2-380 (empezando con el tiempo de exposición idéntica para cada uno, por lo general en el rango de 200-1.000 ~ mseg).
- Utilizar los métodos descritos en el Paso 5.4.1 para calcular el Fura2-340 / Fura2-380 para cada linfocitos. Realizar iteraciones repetidas de ajuste de la Fura2-340 y tiempos de exposición Fura2-380, la adquisición de imágenes y el cálculo de relaciones hasta que los valores promedio están cerca de 1.
- Establecer los tiempos de exposición para mem-YFP y DsRed.
- Vuelva a colocar el plato microscopio usado en el paso 5.3.3.1 con un plato miscroscope contiene transfectadas, activado y SAg tratada (o sin tratamiento; control) HLMVECs o HDMVECs de incubadora de cultivo celular (Pasos 4.5.1). Utilice una pipeta de transferencia desechable para eliminar rápidamente los medios de comunicación, aclarar de una vez por adición de ~ 1 ml de pre-calentado Buffer-A. Aspirar y luego añadir 0,5 ml de Tampón-A.
- Identificar ámbitos en los cuales las células endoteliales transfectantes positivos brillantes fluorescentes están presentes y aparecen saludable con uniones intercelulares bien formados.
- Ajustar parámetros de adquisición (por ejemplo, tiempo de exposición, ganancia del detector y binning) para mem-YFP y DsRed. Asegúrese de que la intensidad media de señal de fluorescencia en cada canal se sitúa entre 25% y 75% del rango dinámico del detector.
- Establecer tiempos de exposición para Fura-2 de formación de imágenes.
- Realizar en vivo de células experimento de imágenes.
- Utilice el software automatizado para comenzar la adquisición de imágenes y capturar a varios intervalos de imágenes para establecer la línea de base.
- Durante la adquisición de aplicar ~ 5 l de linfocitos cargados con Fura-2 concentradas (del paso 5.2) para el centro de la campo de la imagen plato microscopio mediante la inserción de la punta de un pequeño volumen (P-5o pipeta en los medios de comunicación cerca del centro del objetivo y expulsión lentamente P-20).
- Como los linfocitos se asientan en el campo de la imagen, hacer ajustes finos en el foco para asegurar que la interfaz de célula a célula-T endotelios (sinapsis inmunológicas) se mantienen en el plano focal. Con 40 y objetivo 63X, ~ 10-20 células por campo son óptimas. Si se observan menos células en el campo de la imagen repita el paso 5.3.4.2.
- Después de que se compitió el intervalo de observación deseada de experimento, continuar de formación de imágenes y la pipeta inmediatamente ionomicina directamente en el plato miscoscope (utilizando la técnica como en 5.3.4.2) a una concentración final de 2 mM para inducir el flujo de calcio máxima de la señal de señalización / Fura-2 (es decir, , un medio de calibración, ver Análisis 5.4.).
- A medida que se compitió una alternativa a 5.3.4.4after el intervalo de observación deseada de experimento, aspirar inmediatamente el Buffer-A y reemplazarlo con 0,5 ml solución de fijación (3,7% de formaldehído en PBS) durante 5 min aRT, seguido de un enjuague tres veces con PBS. Luego continúe con el paso 6.
- Análisis de Células vivas imágenes
NOTA: Después de guardar los archivos adquiridos, pueden ser analizados directamente o exportados para su análisis por una amplia variedad de aplicaciones de software de análisis de imágenes fuera de línea. ImageJ es una particularmente valioso paquete, de gran versatilidad que está disponible gratuitamente y es compatible con casi todos los paquetes de software de adquisición. El diseño de los experimentos de imagen descritos en los pasos 5.1-5.3 rendirá alta dinámica espacial y temporal de resolución de las interacciones entre los linfocitos y endotelial APC en ausencia y presencia de cognado SAg. Casi análisis ilimitadas son posibles al dirigirse a los datos de imagen de la dinámica morfométricos / señalización celular. Los objetivos específicos que se describen en este protocolo en particular son de cuantificar de forma coordinada la migración de linfocitos y de señalización (es decir, la cinética y los niveles de flujo de calcio intracelular) junto con la dinámicaotros cambios en la arquitectura de la sinapsis inmunológica con el foco en las características específicas (por ejemplo, programas de vida independiente / Podo-grabados). Los siguientes son ejemplos de norma separada y no estándar (es decir, desarrollados específicamente para estos experimentos / preguntas) analiza.- Medida de linfocitos flujo de calcio
- Seleccione la inicial Fura2-340 (340 EX-510 EM nm nm) y Fura2-380 (380 nm EX-510 nm EM) imágenes que fueron adquiridos antes de la adición de los linfocitos. Utilice estos como imágenes de fondo y digital restarlos de la totalidad de las imágenes de la serie de tiempo para cada canal respectivo.
- Para cada punto de tiempo de crear una imagen digital de la relación de fondo-resta Fura2-340 y las imágenes Fura2-380 fondo-resta (es decir, 340 nm 510 nm EX-EM-fondo / 380 nm 510 nm EX-EM-fondo).
- Seleccione la herramienta de dibujo software apropiado. Haga clic en la pantalla para dibujar una región circular de interés (ROI) en torno a cada uno de los linfocitos. Estos generarán un valor de la relación de píxeles promediada para cada cuadro de linfocitos y el tiempo.
- Trazar la relación calculada como una función del tiempo usando una aplicación de software apropiado (por ejemplo, el programa de hoja de cálculo). Calcular el flujo de calcio promediado para el experimento sumando los valores de la relación de todos los linfocitos por campo y dividiendo ese valor por el número total de linfocitos para cada punto de tiempo
- Realizar lympocyte análisis de seguimiento de la migración.
- Analizar celular migración de células T usando una aplicación apropiada de seguimiento de celda de software (por ejemplo, ImageJ) utilizando el canal de DIC de cada video. El uso de ImageJ célula de seguimiento Plugin, identificar en cada cuadro del centro de gravedad de cada célula de forma manual haciendo clic en él en cada trama progresiva.
- Utilice las coordenadas xy serie resultantes (es decir, los rastros de las rutas de migración) para calcular la velocidad media (distancia total migrado intervalo / o total def formación de imágenes) y la tortuosidad (distancia total migraron / la distancia lineal de extremo a extremo entre la ubicación de la celda en la primera y última imagen).
- Cross-correlacionar estos parámetros con el flujo de calcio (5.4.1) dinámica sobre una base de celda por celda.
- Evaluar la densidad Podo-print / ILP dentro de la IS
- Contar el número total de ILP formado en cada célula T intervalos se define (por ejemplo, después de la adición de 5 minutos de las células T). Estos pueden ser identificados fluorescentes anillos membrana-YFP discreta micras escala que co-localizar con las ojeras en citoplasmática DsRed en el endotelio de APC en un sitio de adhesión de linfocitos.
- Cross-correlacionar los índices resultantes '' ILP con el flujo de calcio (5.4.1) dinámica sobre una base de celda por celda.
- Mida la podo-print / vidas ILP.
- Para cada podo-print / ILP en un determinado es el cálculo de sus vidas como el último punto de tiempo cuando una ILP era visible - el punto de tiempo cuando ese podo-print / ILP fiprimero apareció. Promedio ILP vidas tanto por ES.
- Cross-correlacionar estos tiempos de vida con el flujo de calcio (5.4.1) dinámica sobre una base de celda por celda.
- Evaluar la relación temporal entre la formación inicial y la ILP flujo de calcio.
- Para cada linfocito identificar el punto de tiempo (marco) a la que el calcio se levanta primero encima del valor inicial 19.
- Para cada linfocito identificar el punto de tiempo en el cual / ILP aparece la primera impresión podo.
- Para cada linfocito calcular un "tiempo de compensación 'restando el punto de tiempo de flujo de calcio inicial de la de la podo-imprimir / ILP inicial. La media de los valores de todos los linfocitos en un campo. Los valores positivos indican la formación ILP precede el flujo de calcio, mientras que los números negativos indican lo contrario.
NOTA: Estos experimentos fácilmente se puede realizar bajo flujo de cizallamiento laminar fisiológicamente relevante utilizando comercialmente disponibles paralelo pared cámaras de formación de imágenes de flujo como se describe 19.
- Medida de linfocitos flujo de calcio
6.-Imaging celular fijo y análisis
- Punto final fijo de células T-endotelial es la formación y tinción
- Preparar las células T como se describe en 1. Preparar las células endoteliales (- / + SAG) como en el paso 5.1 (transfección, Paso 5.1.1 es opcional). Tomar una muestra de linfocitos cultivados y determinar la densidad contando con un hemocitómetro (Sección 1).
- Transferir un volumen de cultivo de linfocitos equivalente a al menos 3 x 10 5 linfocitos / muestra a un tubo cónico de 15 ml y centrifugar a 200 xg durante 3 min. Eliminar el sobrenadante y resuspender el botón de linfocitos en Tampón A (Paso 5.2.1) la concentración de 6 x 10 5 linfocitos / ml.
- Retire las células endoteliales de la incubadora de cultivo celular y (proceder de una muestra a la vez), sucesivamente, los medios de comunicación de aspirado y reemplazar con 0,5 ml de suspensión de linfocitos (3 x 10 5 linfocitos / muestra) y reemplazar a 37 I ° Cncubator.
- Después de tiempos de incubación apropiados, se ha indicado anteriormente, el medio aspirado de los pocillos y añadir suficiente solución de fijación (3,7% de formaldehído en PBS) para cubrir completamente la muestra (por ejemplo, en placa de 24 ~ de 300-500 l). Incubar a temperatura ambiente durante 5-10 min.
- Enjuague muestra 3 veces con PBS. Teñir añadiendo anticuerpo primario durante 60 minutos a temperatura ambiente (ver lista). Si el anticuerpo primario está dirigida a una proteína intracelular, un paso de permeabilización adicional necesita ser realizada mediante la adición de solución de permeabilización suficiente (0,01% Triton X-100 en PBS) para cubrir completamente la muestra durante 1 min a TA. Enjuague muestra 3 veces con PBS. Añadir anticuerpos secundarios apropiados (en algunos casos de forma concomitante con faloidina fluorescente) durante 60 min a TA.
- Monte el cubreobjetos de vidrio circular en una diapositiva de imagen. Punto final fijo de células T-endotelial es la formación y la tinción. Para empezar lugar de imágenes de la diapositiva de imágenes que contiene muestras de la platina del microscopio y seleccione el aceite 63Xobjetivo. Aplicar aceite de inmersión fresca.
- Utilice el modo de imagen de campo brillante y lentes oculares para localizar el plano focal. Cambie a epifluorescencia e inspeccionar la muestra a través de las lentes oculares. Seleccione un campo de interés. Cambiar al modo de escaneo láser.
- Usando el modo de exploración rápida y de trabajo en el campo más amplio posible ajustar individualmente la potencia del láser y la ganancia de cada canal para optimizar la señal de tal modo que, idealmente, la intensidad de la señal específica alcance al menos el ~ 25% y no más del 75% de la gama dinámica de los detectores.
- El uso de los controles de enfoque manual, escanear rápidamente a través del eje Z y determinar los límites superior e inferior de seccionamiento (por lo general toda la información debe estar contenida dentro de un espesor de ~ 15 micras). Seleccione el espesor de la sección del eje Z en el intervalo de 0,2 a 1,0 ~ m.
- Por último, el zoom / recortar el campo de la imagen de la región específica de interés y exploración conducta. Además de tener muy luminoso y específica si la fluorescenciagnal en la muestra, la adquisición de alta resolución de imagen 3D-requiere iteraciones de exploración y hacer los ajustes a los parámetros de adquisición. El objetivo es obtener xy máxima y resolución del eje z, sin apreciable de foto-blanqueo.
- Analizar cualitativamente la topología de la topología 3D de la SI mediante la realización de la reconstrucción digital 3D de las secciones ópticas resultantes a través de una aplicación de software apropiado (por ejemplo, Imagen J). A través de aplicaciones similares evaluar cuantitativamente la distribución de la señal de fluorescencia (por ejemplo, co-localización y la intensidad de fluorescencia correlativa análisis de línea de exploración de Pearson y la visualización ortogonal).
Resultados
Un enfoque de formación de imágenes novedoso utilizando células endoteliales y la combinación de las ventajas resolución de la planar bicapas lipídicas modelo con la complejidad fisiológica y la deformabilidad de las APC profesional fue desarrollado (Figura 1). La Figura 2 proporciona ejemplos de migración típica, el flujo de calcio y la dinámica topológicos observado con esta enfoque. En ausencia de la SAG en el endotelio, los linfocitos SAG-...
Discusión
En general, este protocolo describe métodos para la investigación de las células endoteliales como i) APC fisiológicos poco estudiada y ii) como un nuevo tipo de "modelo APC celular plana '. Con respecto a lo primero, se ha vuelto cada vez más apreciada que la APC periférica no hematopoyéticas (o 'estroma') juegan un papel crítico, no redundantes (es decir, en comparación con hematopoyético APC) en la conformación de la respuesta inmune adaptativa 16-18. Entre tales "...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
We thank Dr. Peter T. Sage for his assistance in generating some of the representative images. This work was supported by an NIH R01 grant to C.V.C. (HL104006).
Materiales
| Name | Company | Catalog Number | Comments |
| BD Vacutainer stretch latex free tourniquet | BD Biosciences | 367203 | |
| BD alcohol swabs | BD Biosciences | 326895 | |
| BD Vacutainer Safety-Lok | BD Biosciences | 367861 | K2 EDTA |
| BD Vacutainer Push Button Blood Collection Set | BD Biosciences | 367335 | |
| RPMI-1640 | Sigma-Aldrich | R8758-1L | |
| Ficoll-Paque | Sigma-Aldrich | GE17-1440-02 | Bring to RT before use |
| FCS-Optima | Atlanta Biologics | s12450 | Heat inactivated |
| Penicillin-Streptomycin | Sigma-Aldrich | P4458-100ML | |
| Trypan blue | Sigma-Aldrich | T8154-20ML | |
| staphylococcal enterotoxin B | Toxin Technology | BT202RED | Stock solution 1mg/ml in PBS |
| toxic shock syndrome toxin 1 | Toxin Technology | TT606RED | Stock solution 1mg/ml in PBS |
| human IL-15 | R&D Systems | 247-IL-025 | Stock solution 50ug/ml in PBS |
| PBS | Life Technologies | 10010-049 | |
| Fibronectin | Life Technologies | 33016-015 | Stock solution 1mg/ml in H20 |
| HMVEC-d Ad-Dermal MV Endo Cells | Lonza | CC-2543 | Other Human Microvascular ECs can be used, i.e. HLMVECs |
| EGM-2 MV bullet kit | Lonza | CC-3202 | |
| Trypsin-EDTA | Sigma-Aldrich | T-4174 | Stock solution 10x, dilute in PBS |
| amaxa-HMVEC-L Nucleofector Kit | Lonza | vpb1003 | Required Kit for step 4 |
| IFN-g | Sigma-Aldrich | I3265 | Stock solution 1mg/ml in H20 |
| TNF-alpha 10ug, human | Life Technologies | PHC3015 | Stock solution 1mg/ml in H20 |
| phenol Red-free HBSS | Life Technologies | 14175-103 | |
| Hepes | Fisher Scientific | BP299-100 | |
| Calcium Chloride | Sigma-Aldrich | C1016-100G | Stock solution 1M in H20 |
| Magnesium chloride | Sigma-Aldrich | 208337 | Stock solution 1M in H20 |
| Human Serum albumin | Sigma-Aldrich | A6909-10ml | |
| Immersol 518 F fluorescence free Immersion oil | Fisher Scientific | 12-624-66A | |
| Fura-2 AM 20x50ug | Life Technologies | F1221 | Stock solution 1mM in DMSO |
| pEYFP-Mem (Mem-YFP) | Clontech | 6917-1 | |
| pDsRed-Monomer (Soluble Cytoplasmic DsRed) | Clontech | 632466 | |
| pDsRed-Monomer Membrane (Mem-DsRed) | Clontech | 632512 | |
| pEGFP-Actin | Clontech | 6116-1 | |
| Alexa Fluor 488 Phalloidin | Life Technologies | A12379 | |
| Formaldehyde solution 37% | Fisher Scientific | BP531-500 | Toxic, use fumehood |
| Triton X-100 | Sigma-Aldrich | X100-5ML | |
| Falcon 15mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-70C | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-49A | |
| Falcon Tissue Culture Treated Flasks T25 | Fisher Scientific | 10-126-9 | |
| Falcon Tissue Culture Treated Flasks T75 | Fisher Scientific | 13-680-65 | |
| Corning Cell Culture Treated T175 | Fisher Scientific | 10-126-61 | |
| Glass coverslips | Fisher Scientific | 12-545-85 | 12 mm diameter |
| Falcon Tissue Culture Plates 24-well | Fisher Scientific | 08-772-1 | |
| Delta-T plates | Bioptechs | 04200415B | |
| Wheaton Disposable Pasteur Pipets | Fisher Scientific | 13-678-8D | |
| 1.5 ml Eppendorf tube | Fisher Scientific | 05-402-25 | |
| ICAM1 mouse anti-human | BD Biosciences | 555509 | |
| HS1 mouse anti-human | BD Biosciences | 610541 | |
| Anti-Human CD11a (LFA-1alpha) Purified | ebioscience | BMS102 | |
| Anti-Human CD3 Alexa Fluor® 488 | ebioscience | 53-0037-41 | |
| Anti-MHC Class II antibody | Abcam | ab55152 | |
| Anti-Talin 1 antibody | Abcam | ab71333 | |
| Anti-PKC theta antibody | Abcam | ab109481 | |
| phosphotyrosine (4G10 Platinum) | Millipore | 50-171-463 | |
| Nucleofector II | Amaxa Biosystems | Required electroporator for step 4 | |
| Zeiss Axiovert | Carl Zeiss MicroImaging | ||
| Zeiss LSM510 | Carl Zeiss MicroImaging | ||
| Zeiss Axiovison Software | Carl Zeiss MicroImaging | ||
| NU-425 (Series 60) Biological Safety Cabinet | NuAIRE | Nu-425-600 | |
| Forma STRCYCLE 37 °C, 5% CO2 Cell culture Incubator | Fisher Scientific | 202370 | |
| Centrifuge 5810 | Eppendorf | EW-02570-02 | |
| Hemocytometer | Sigma-Aldrich | Z359629 | Bright-Line Hemocytometer |
| Isotemp Waterbath model 202 | Fisher Scientific | 15-462-2 |
Referencias
- von Andrian, U. H., Mackay, C. R. T-cell function and migration. Two sides of the same coin. N. Engl. J. Med. 343 (14), 1020-1034 (2000).
- Springer, T. A. Adhesion receptors of the immune system. Nature. 346 (6283), 425-434 (1990).
- Shaw, A. S., Dustin, M. L. Making the T cell receptor go the distance: a topological view of T cell activation. Immunity. 6 (4), 361-369 (1997).
- Monks, C. R., Freiberg, B. A., Kupfer, H., Sciaky, N., Kupfer, A. Three-dimensional segregation of supramolecular activation clusters in T cells. Nature. 395 (6697), 82-86 (1998).
- Delon, J., Stoll, S., Germain, R. N. Imaging of T-cell interactions with antigen presenting cells in culture and in intact lymphoid tissue. Immunol Rev. 189, 51-63 (2002).
- Brossard, C., et al. Multifocal structure of the T cell - dendritic cell synapse. Eur J Immunol. 35 (6), 1741-1753 (2005).
- Dustin, M. L. The cellular context of T cell signaling. Immunity. 30 (4), 482-492 (2009).
- Balagopalan, L., Sherman, E., Barr, V. A., Samelson, L. E. Imaging techniques for assaying lymphocyte activation in action. Nat Rev Immunol. 11 (1), 21-33 (2011).
- Cebecauer, M., Spitaler, M., Serge, A., Magee, A. I. Signalling complexes and clusters: functional advantages and methodological hurdles. J Cell Sci. 123, 309-320 (2010).
- Oddos, S., et al. High-speed high-resolution imaging of intercellular immune synapses using optical tweezers. Biophys J. 95 (10), L66-L68 (2008).
- Grakoui, A., et al. The immunological synapse: a molecular machine controlling T cell activation. Science. 285 (5425), 221-227 (1999).
- Bunnell, S. C., et al. T cell receptor ligation induces the formation of dynamically regulated signaling assemblies. J Cell Biol. 158 (7), 1263-1275 (2002).
- Yokosuka, T., et al. Newly generated T cell receptor microclusters initiate and sustain T cell activation by recruitment of Zap70 and SLP-76. Nat Immunol. 6 (12), 1253-1262 (2005).
- Seminario, M. C., Bunnell, S. C. Signal initiation in T-cell receptor microclusters. Immunol Rev. 221, 90-106 (2008).
- Dustin, M. L. Supported bilayers at the vanguard of immune cell activation studies. J Struct Biol. 168 (1), 152-160 (2009).
- Martinelli, R., Carman, C. V., Bradshaw, R. A., Stahl, P. Lymphocyte Endothelilal Interactions. Encyclopedia of Cell Biology. , (2015).
- Choi, J., Enis, D. R., Koh, K. P., Shiao, S. L., Pober, J. S. T lymphocyte-endothelial cell interactions. Annu Rev Immunol. 22, 683-709 (2004).
- Marelli-Berg, F. M., Jarmin, S. J. Antigen presentation by the endothelium: a green light for antigen-specific T cell trafficking?. Immunol Lett. 93 (2-3), 109-113 (2004).
- Sage, P. T., et al. Antigen recognition is facilitated by invadosome-like protrusions formed by memory/effector T cells. J Immunol. 188 (8), 3686-3699 (2012).
- Kumari, S., et al. Actin foci facilitate activation of the phospholipase C-gama in primary T lymphocytes via the WASP pathway . eLife. , (2015).
- Marelli-Berg, F. M., et al. Major histocompatibility complex class II-expressing endothelial cells induce allospecific nonresponsiveness in naive T cells. J Exp Med. 183 (4), 1603-1612 (1996).
- Ma, W., Pober, J. S. Human endothelial cells effectively costimulate cytokine production by, but not differentiation of, naive CD4+ T cells. J Immunol. 161 (5), 2158-2167 (1998).
- Perez, V. L., Henault, L., Lichtman, A. H. Endothelial antigen presentation: stimulation of previously activated but not naive TCR-transgenic mouse T cells. Cell Immunol. 189 (1), 31-40 (1998).
- Epperson, D. E., Pober, J. S. Antigen-presenting function of human endothelial cells. Direct activation of resting CD8 T cells. J Immunol. 153 (12), 5402-5412 (1994).
- Shiao, S. L., et al. Human effector memory CD4+ T cells directly recognize allogeneic endothelial cells in vitro and in vivo. J Immunol. 179 (7), 4397-4404 (2007).
- Marelli-Berg, F. M., Okkenhaug, K., Mirenda, V. A two-signal model for T cell trafficking. Trends Immunol. 28 (6), 267-273 (2007).
- Carman, C. V., et al. Transcellular diapedesis is initiated by invasive podosomes. Immunity. 26 (6), 784-797 (2007).
- Carman, C. V. Mechanisms for transcellular diapedesis: probing and pathfinding by 'invadosome-like protrusions'. J Cell Sci. 122 ((Pt 17)), 3025-3035 (2009).
- Dustin, M. L., Tseng, S. Y., Varma, R., Campi, G. T. T cell-dendritic cell immunological synapses. Curr Opin Immunol. 18 (4), 512-516 (2006).
- Saito, T., Yokosuka, T. Immunological synapse and microclusters: the site for recognition and activation of T cells. Curr Opin Immunol. 18 (3), 305-313 (2006).
- Gomez, T. S., et al. HS1 functions as an essential actin-regulatory adaptor protein at the immune synapse. Immunity. 24 (6), 741-752 (2006).
- Burbach, B. J., Medeiros, R. B., Mueller, K. L., Shimizu, Y. T-cell receptor signaling to integrins. Immunol Rev. 218, 65-81 (2007).
- Vicente-Manzanares, M., Sanchez-Madrid, F. Role of the cytoskeleton during leukocyte responses. Nat Rev Immunol. 4 (2), 110-122 (2004).
- Ma, Z., Janmey, P. A., Finkel, T. H. The receptor deformation model of TCR triggering. Faseb J. 22 (4), 1002-1008 (2008).
- Ma, Z., Sharp, K. A., Janmey, P. A., Finkel, T. H. Surface-anchored monomeric agonist pMHCs alone trigger TCR with high sensitivity. PLoS Biol. 6 (2), e43 (2008).
- Groves, J. T. Bending mechanics and molecular organization in biological membranes. Annu Rev Phys Chem. 58, 697-717 (2007).
- Xu, C., et al. Regulation of T cell receptor activation by dynamic membrane binding of the CD3epsilon cytoplasmic tyrosine-based motif. Cell. 135 (4), 702-713 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados