É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um modelo de celular endotelial Planar for Imaging imunológica Synapse Dynamics
Neste Artigo
Resumo
Adaptive immunity is controlled by dynamic 'immunological synapses' formed between T cells and antigen presenting cells. This protocol describes methods for investigating endothelial cells both as understudied physiologic APCs and as a novel type of 'planar cellular APC model'.
Resumo
A imunidade adaptativa é regulada por interações dinâmicas entre as células T e células apresentadoras de antígenos ('APCs') conhecido como 'sinapses imunológicas. Dentro destas interfaces de célula-célula íntimos agrupamentos sub-celular discretas de MHC / Ag-TCR, F-actina, adesão e moléculas de sinalização formar e remodelar rapidamente. Essas dinâmicas são pensados para ser determinantes críticos tanto da eficiência e qualidade das respostas imunes que se desenvolvem e, portanto, de proteção contra a imunidade patológica. Compreensão atual das sinapses imunológicas com fisiológico APCs é limitado pela inadequação da resolução de imagem obtidos. Embora modelos substrato artificial (por exemplo, bicamadas lipídicas planas) oferecem excelente resolução e têm sido ferramentas extremamente valiosas, eles são inerentemente não-fisiológica e simplista. As células endoteliais vasculares e linfáticas surgiram como um tecido periférico importante (ou estroma) compartimento de "semi-profissãoai APCs '. Estas APCs que expressam (a maioria da maquinaria molecular de APCs profissionais) têm a característica única de formação da superfície celular praticamente plana e são facilmente transfectáveis (por exemplo, com os repórteres de proteína fluorescente). Aqui uma abordagem básica para implementar as células endoteliais como uma nova e fisiológico "planar modelo celular da APC 'para melhorar a imagiologia de interrogação e de processos fundamentais de sinalização antigénicos irá ser descrito.
Introdução
Os linfócitos T são um ramo do sistema imunitário adaptativo, caracterizado por a capacidade de reconhecer eficientemente antigénio peptídico (Ag) ligado ao complexo principal de histocompatibilidade (MHC) através dos seus receptores de células T (TCRs) 1. Linfócitos naive constitutivamente migrar e digitalizar 'Ag células apresentadoras de profissionais "(APCs; por exemplo, células dendríticas) dentro de gânglios linfáticos, enquanto as células de memória / T efetoras precisa fazer um levantamento efetivamente uma gama extremamente ampla de APCs e células alvo em potencial dentro tecidos periféricos.
Na min após o reconhecimento inicial de cognato Ag em um APC, linfócitos prender sua migração e começam a formar uma interface célula-célula íntimo especializado denominado "sinapse imunológica" (IS). Sustentada (ie, 30-60 min) É contatos são necessários para ampliar e sustentar a sinalização 2-7. Estudos emergentes identificar que no seja, é a formação contínua e rápida remodeling de micro-aglomerados discretos sinalização sub-celular (ou seja, contendo MHC / Ag-TCR, F-actina, adesão e moléculas de sinalização) que determinam a força e qualidade dos resultando respostas imunes 2-7. No entanto, detalhes dinâmicos e mecanismo de regulação deste processo são completamente compreendidas 8,9. Isso decorre, em grande parte dos desafios técnicos associados com topologias de superfícies irregulares APC e orientação mal controlada dos planos de interação célula-célula, as questões que limitam profundamente a imagem espaço-temporal requisito aproxima 8-10 (Figura 1A).
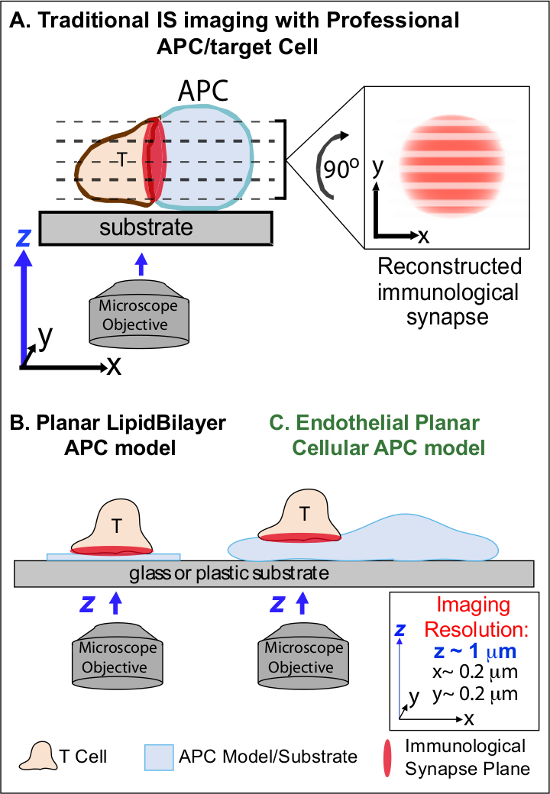
Figura 1. Um Fisiológica Planar Modelo APC celular for Imaging imunológica Synapse Dynamics. O esquema ilustra imagem tradicional da sinapse imunológica entre uma célula T e um professio nal APC (A) e de células T e um lipídicas planas modelo tradicional bicamada APC (B) em comparação com este modelo planar APC endotelial novel (C). APCs profissionais fornecer sinapses imunológicos fisiológicos, mas oferecem interface de mal orientadas célula-célula (isto é, em relação ao plano de imagem XY óptima; resolução ~ 0,2 ^ M), o que compromete dramaticamente espacial (plano de imagem z resolução ~ 1 mm) e temporal (isto é, devido à necessidade de digitalizar repetidamente através de todos os planos de imagem z) resolução de imagem. Modelos de duas camadas tem uma topologia plana que fornece melhor resolução de imagem espaço-temporal, mas também são altamente simplificado, não-fisiológica e rígida. Este modelo combina células endoteliais a topologia planar de camadas duplas lipídicas com o substrato fisiológico de um APC clássico para oferecer resolução de imagem espacial e temporal ideal em um ambiente fisiológico.m / files / ftp_upload / 53288 / "target =" _ blank 53288fig1large.jpg "> Clique aqui para ver uma versão maior desta figura.
Trabalhos anteriores contornada parcialmente estes obstáculos através do desenvolvimento de modelos de substrato planar (isto é, as bicamadas lipídicas e das superfícies revestidas com anticorpo) que fornecem resolução espaço-temporal óptimo (ou seja, através de fixação superfície a activação das células T num único plano que é paralelo ao imagiologia XY óptima avião) 11-15 (Figura 1B). Estes modelos têm facilitado importantes insights sobre a dinâmica subcelulares / molecular que controlam a sinalização antigênico nas células T, incluindo a descoberta de actina dinâmica / TCR sinalização micro-aglomerados 7,11-14. No entanto, tais modelos são inerentemente muito simplificado, assim como rígida (impedindo o desenvolvimento / estudo de características topológicas 3-dimensional) (Figura 1B). Portanto, permanece incerto como relacionar esses achados para phycélula-célula siologic vigilância imunológica.
Embora ainda pouco estudadas, vascular e células endoteliais linfáticas estão emergindo como uma grande (ou seja, maior em número do que todos os APCs profissionais, por ~ 1.000 vezes) compartimento periférico de "semi-profissional" APCs 16-18. Estas células expressam MHC-I, MHC-II e uma multiplicidade de moléculas co-estimuladoras (por exemplo, CD40, LFA3, ICOSL, 4-1BB, OX40L, TL1A, PD-L1, mas não CD80 e CD86) e são estrategicamente posicionada na interface de sangue-tecido onde servem funções especializadas sentinela 16-18. Estudos anteriores demonstraram que as células endoteliais podem efetivamente re-estimular efector / memória, mas não naïve, células T 19-25. Assim, as células endoteliais são susceptíveis de desempenhar papéis exclusivos da APC na fase efectora da resposta imune adaptativa no interior dos tecidos periféricos, tais como a influência local sobre a activação de células T, a diferenciação, a memória e a tolerância 16,17,26. Criticalmente, quando cultivadas in vitro, as células endoteliais formam superfícies celulares praticamente planares e estão facilmente transfectáveis (por exemplo, com os repórteres de proteína fluorescente). Estas características são ideais para imagens de alta resolução espacial e temporal da dinâmica topológica durante as interações célula-célula 19,27. Assim, as células endoteliais pode servir como um fisiológico "planar celular da APC modelo extremamente adequado para o estudo dos mecanismos de remodelação subcelulares / moleculares que conduzem o reconhecimento do antigénio e regulam as respostas (Figura 1C) 19,20.
Previamente estabelecida técnicas de imagem complementares (incluindo a transfecção de células endoteliais com fabricantes de proteína fluorescente da membrana de plasma e citossol) para estudar os detalhes da interacção de leucócitos-endotélio durante a adesão e a migração transendotelial de 27, mostraram que os leucócitos sondar activamente a superfície do endotélio por dinâmico inserção de umd retração de sub-mícron-escala, saliências cilíndricas ricas em actina (~ 200-1.000 nm de diâmetro e profundidade) denominado saliências invadosome-like (ou seja, 'ILPS') 27,28. Estas abordagens de imagem têm sido expandida, juntamente com a criação de protocolos para tirar proveito da função endotelial APC para desenvolver os primeiros métodos para imagens de alta resolução espacial e temporal da célula endotelial sinapse imunológica T como relatado 19,20 e ainda descrever aqui. Uma constatação fundamental derivado dessa modelo APC celular romance planar é que procedimentos ILP células T funcionar tanto na promoção da detecção Ag inicial e na manutenção de sinalização subsequente. Na verdade, matrizes de múltiplos procedimentos ILP (que foram estabilizados e acumulados em resposta a rubricar o fluxo de cálcio) mostram enriquecimento em TCR e moléculas de sinalização sugestivos activa, tais PKC-Q, ZAP-70, fosfotirosina e HS1. Portanto, procedimentos ILP parecem representar um equivalente fisiológico tridimensional para o micro-TCR sinalizaçãoaglomerados observados nos modelos de duas camadas planas. Esta abordagem, assim, com sensibilidade revela / relatórios dinâmica molecular e arquitetônicos (e implícitas biomecânico) de outra forma não detectável.
O método descrito aqui deve ser útil para fazer a ponte entre APC profissional e modelos artificiais de substrato APC a fim de aumentar a nossa capacidade de interrogar mecanismos básicos da resposta imune adaptativa. Embora aqui o foco é sobre a activação das células T CD4 + do tipo Th1 efector / célula de memória, esta abordagem básica pode ser facilmente modificado para estudar uma ampla gama de tipos de células T e Ags, como discutido abaixo.
Protocolo
Todas as experiências descritas neste protocolo são realizadas com células T humanas primárias e células humanas primárias disponíveis comercialmente endoteliais (dérmica ou microvascular pulmonar ECs) protocolo de pesquisa .any envolvendo seres humanos deve ser aprovado por um conselho de revisão institucional e consentimento informado por escrito deve ser fornecida a partir de cada doador de sangue. Experimentos conduzidos utilizando este protocolo foi aprovado pelo IRB do Beth Israel Deaconess Medical Center.
1. Preparar as células CD4 + Th1 Humano Effector / T de memória
- Aplicar torniquete no braço do doador, limpe veia com álcool, e inserir a agulha. Desenhar lentamente 15 ml de sangue para vacutainer usando EDTA como anticoagulante. Quando o sangue tiver sido tirada, desatar o torniquete antes de retirar a agulha. Aplicar imediatamente pressão para ferida com gaze estéril quando a agulha é removida.
- Transferir o sangue para um tubo de 50 ml. Adicionar RPMI-1640 à TA a uma diluição 1: 1 (volume final 30 mL). Sobrepor cuidadosamente a diluídad sangue em dois tubos de 50 ml contendo 15 ml de pré-filtrada linfócitos meio de isolamento, tais como Ficoll-Paque à TA.
- Centrifuga-se o gradiente a TA durante 30 min a 1.200 x g num rotor de balde oscilante. Enquanto centrifugação preparar o meio de células T (500 ml de RPMI-1640, FCS a 50 ml, 5 ml penicilina / estreptomicina).
- Após a remoção do tubo de 50 ml de centrífuga, observar quatro camadas: um sedimento de glóbulos vermelhos no fundo, o paque, uma camada de células que contém células brancas do sangue (incluindo linfócitos) e o plasma. Remova cuidadosamente a camada de células brancas do sangue com uma pipeta de pasteur e transferência em um tubo Falcon 50 ml.
- Lava-se a camada de células brancas do sangue, adicionando RT RPMI-1460 (até 20 ml) e centrifugação à TA durante 5 min a 1200 x g. Ressuspender as células brancas do sangue, em 1 ml de meio de células T. Adiciona-se 5 mL da suspensão de 1 ml de células para meio de células T de 250 ul num tubo de centrífuga de 1,5 ml, e misturar suavemente cima e para baixo com uma pipeta.
- Adicionar 25 ul diluird suspensão de células a 25 uL de 0,4% azul de Tripano. Adicionar 10 ul de mistura a cada um dos lados de um hemocitómetro padrão.
- Coloque hemocit�etro em uma luz baixa potência microscópio. Utilizando uma objectiva de 10X contar o número de células que excluem o corante azul de tripano e que se encontram presentes no quadrado do meio de ambos os lados do hemocitómetro vivo.
- Para calcular a concentração de células, multiplicar a média dos quadrados por 2 100 (factor de diluição) e, em seguida, multiplicar por 10 4 para se obter o número de células / ml.
- Ajustar a concentração final de 0,5 x 10 6 células / ml em meio de células T. Adicionar uma concentração final de 1 ug / ml de cada um dos superantigénios bacterianas enterotoxina B de estafilococos (SEB) e toxina de síndroma de choque tóxico 1 (TSST) para as células. Cultura durante 72 horas (37 ° C e 5% de CO 2) para expandir a população de células T CD4 +.
- As células T da pelota (1200 xg, 5 minutos) e ressuspender em 0,5 x 10 6 células / ml em meio de células T com oadição de IL-15 humana (20 ng / mL). Transferir para um balão de linfócitos T150. Continuar a expandir / células divididas em pleno meio-IL-15 a cada 24-48 horas, conforme necessário (com base na cor da mídia, ou seja, sempre que a mídia passa de rosa a ligeiramente amarelo) depois. Manter a população de linfócitos resultante por até 15 dias.
NOTA: Por design, este protocolo irá ativar e ampliar especificamente subconjunto de células T CD4 + que são reativos a SEB e TSST e, em seguida, levá-los em direção a um tipo Th1 fenótipo efetor / memória. Outras células brancas do sangue não conseguem sobreviver e crescer sob estas condições, de tal modo que a passo 1.10 as células irão ser, pelo menos, 95% de CD4 +, CD45RO + de células T 19, como pode ser prontamente avaliada por citometria de fluxo. Se desejado, a purificação pode ser facilmente conseguida por meio de kits de selecção baseados positivos ou negativos de anticorpos disponível comercialmente / magnética-grânulo.
2. A partir Cultura de células endoteliais humanas Primária
- Revestimento um frasco T25 com fibronectina (FN) 20 ug / ml em PBS em condições estéreis. Deixar a TA durante 30-60 min. Remover FN e adicionar 5 ml de meio completo (Basal Endotelial Médio (EBM-2) meio suplementado com Meio de Crescimento Endotelial (EGM-2) singlequots). Pré-incubar em 37 ° C incubadora de cultura de células durante pelo menos 30 min.
- Descongelar um frasco de pulmão humano ou dérmica microvasculares endoteliais células congeladas (HLMVECs ou HDMVECs C) em um banho de água a 37 ° C com agitação suave ocasional durante ~ 2-3 min. Imediatamente para transferir células frasco T25 contendo meio de pré-aquecido. Agite cuidadosamente e coloque em incubadora a 37 ° C.
- Altere a mídia depois de ~ 4-6 horas. Continuar a mudar media aproximadamente a cada 48 horas (ou quando a mídia se torna um pouco amarelo) até placa atinge ~ 90-95% de confluência.
3. Divisão Geral e Expansão das células endoteliais
- Crescer as células a ~ 90-95 confluência. Isso pode demorar 2-5 dias. Para rachar,remova a mídia e enxaguar com PBS. Remover PBS e substituir com volume mínimo de tripsina 1x fresco (0,5 ml de T25 ou 1,5 ml para T75). Agite suavemente para cobrir toda a superfície com tripsina. Incubar a 37 ° C para ~ 5 min. Monitorar o desprendimento das células da placa utilizando uma luz baixa potência microscópio.
- Quando maioria das células parecem arredondado ou individual, adicionam-se 5 volumes (ou seja, em comparação com o volume adicionado tripsina) de meio completo pré-aquecido EGM-2 e pipeta suavemente sobre a superfície do balão para separar todas as células.
- Contar as células endoteliais com um hemocitómetro, como descrito em 1,6-1,7. Agregar as células por centrifugação (5 min, 1,200 xg). Remover o sobrenadante. Ajustar a concentração para 0,5 milhões de células por ml pela adição de completa EGM-MV meios 2 de pré-aquecido.
- Transferir alíquotas de células aos pratos ou frascos apropriados FN-revestidos para manutenção. Agite cuidadosamente e colocar na incubadora. Troque a mídia dentro de 6-12 horas de chapeamento. Sho mídiauld ser alterado aproximadamente a cada 48 horas depois disso.
4. Célula Endotelial Transfecção
NOTA: Primário células endoteliais são refratários à transfecção pela maioria dos métodos químicos e eletroporação comum. O método baseado na transfecção nuclear descrito abaixo permite a eficiência de transfecção relativamente elevada (~ 50-70%). Um método alternativo eficaz é o uso de infecção por vectores virais adequados (ver comentários em Materiais Tabela).
- Prepare T25 ou T75 frascos (conforme necessário) de HLMVECs ou HDMVECs para uma densidade final de 90-95% confluency.Coat com fibronectina (FN) 20 ug / ml em PBS, em condições estéreis, quer microscópio placas de cultura, tais como placas de delta-T (por Passo 5) ou lamelas de vidro de 12 mm circulares colocados no interior de um poço de uma placa de 24 poços de cultura de células (para o passo 6) como descrito acima com (2.1).
- Adicionar 1 ml de AGE-2 meios de cultura completos para cultura microscópio platesor 0,5 ml a cada um de 24 poços e equilibrar em placasnuma atmosfera humidificada a 37 ° C / 5% de CO2.
- Colheita e contagem de células endoteliais tal como nos passos 3.1-3.3.Centrifuge o volume necessário de células (0,5 milhões de células por amostra) a 1.200 xg durante 5 min à temperatura ambiente. Ressuspender o sedimento celular em 100 ul com cuidado solução de transfecção nuclear RT por amostra.
- Combinar 100 ul de suspensão de células com 1-5 ug de ADN. Transferência de células em suspensão / ADN em cuvete certificado; amostra deve cobrir o fundo da cuvete sem bolhas de ar.
NOTA: As construções de direccionamento YFP ou DsRed à membrana celular (por meio da N-terminal de 20 aminoácidos de neuromodulina que contém um sinal de palmitoilação pós-tradução) foram utilizados sozinhos (membrana-YFP sozinho ou membrana-DsRed sozinho) ou co-transfectadas com um marcador volumétrica citoplasmático (por exemplo, YFP-membrana e DsRed solúvel). Muitos permutações de marcadores de proteína fluorescente pode ser utilizado. - Feche a cuvete com a tampa. Coloque a célula comsuspensão de células / DNA no suporte da cuvete da electroporador e aplicar programa eletroporação S-005. Tome a tina para fora do suporte quando o programa for concluído.
- Adicionar ~ 500 ul de meio de cultura pré-equilibrada para a cuvete e remover suavemente a suspensão de células a partir da cuvete usando as pipetas de transferência de plástico fornecidos com o kit de transfecção nuclear.
- Para experiências utilizando placas de cultura de microscópio partição da suspensão de células a partir de uma reacção entre dois pratos igualmente contendo meio pré-aquecido (Passos 4,2-4,3). Para experiências com 24 poços / placa, uma partição reação igualmente entre 3 poços.
- Incubam-se as células numa atmosfera humidificada com 37 ° C / 5% de CO2 e mudar de suporte 4-6 horas, e de novo em 12-16 horas após a transfecção.
5. imagens de células vivas e Análise
- Preparando endotélio
- Dia 0: Co-transfectar HLMVECs primárias com membrana-YFP e DsRed solúvel através de um nucleofetecnologia ction conforme descrito na etapa 4 e placa para em células vivas, placas de cultura de imagem.
- Dia 1: Substituir meio com meio fresco contendo IFN-γ (100 ng / ml) para induzir a expressão de MHC-II. No Dia 2. Estimular células transfectadas por adição de 20 ng ml TNF-α / para a mídia existente.
- No Dia 3, o endotélio incubar com 1 ug / ml de cada um dos superantigénios de enterotoxina B de estafilococos bacterianas (SEB) e toxina da síndroma do choque tóxico 1 (TSST) a 37 ° C durante 30-60 minutos imediatamente antes das experiências. Omitir esta etapa para as condições de controlo dos -AG '.
- Preparando Linfócitos
- Em paralelo com a etapa 5.1.3, preparar Tampão A (sem vermelho de fenol HBSS), suplementado com Hepes a 20 mM, pH 7,4 e 0,5% v / v de albumina de soro humano pré-aquecido a 37 ° C. Retirar uma amostra de linfócitos em cultura e determinar a densidade por contagem com um hemocitómetro (Passo 1,12-1,17).
- Centrífuga 2 milhões de células por amostra em 1,200 xg durante 5 min à temperatura ambiente num tubo cónico de 15 ml. Aspirar e meios sedimento de células suavemente ressuspender em 2 ml de tampão A de tal modo que não há grupos de células permanecem.
- Remover uma nova porção de Fura-2 corante de cálcio e ressuspender em DMSO para fazer uma concentração de estoque de 1 mM.
- Adicionar 2 mL de solução de estoque de Fura-2 para a suspensão de células T (2 uM de concentração final), a tampa do tubo e misturar imediatamente por inversão do tubo para assegurar a dispersão uniforme do corante. Incubar a 37 ° C durante 30 min.
- Centrifugar como no passo 5.2.2. Aspirar Tampão-A e linfócitos suave mas completamente ressuspender em 20-40 ul de tampão fresco-A.
- Live-célula Imaging and Acquisition Setup
NOTA: Uma grande variedade de sistemas pode ser utilizado para imagiologia de fluorescência de células ao ar em cima e para baixo microscópios de luz. Os requisitos básicos incluem uma fonte de fluorescência de luz e filtros, uma câmera CCD, comutação de filtro motorizado e estores, uma fase aquecida (ou microscope-montável câmara aquecida) e software para aquisição de imagem automatizado. Para este protocolo de alta abertura numérica, alta ampliação (ou seja, 40X, 63X) lentes de imersão em óleo são necessários para conseguir a resolução espacial necessária. Um cuidado especial deve ser tomado na escolha da fonte de fluorescência apropriado e lentes para geração de imagens de cálcio com base em Fura-2 como nem todos são compatíveis com as exigidas 340/380 comprimentos de onda de excitação nm. Uma abordagem alternativa (compatível com o padrão de conjuntos de filtros de fluorescência verde e vermelho) podem ser usados com corantes não-ratiomentic sensível ao cálcio (por exemplo, Fluo-4, Rhod-3), embora estes não pode quantificar com precisão o fluxo de cálcio e apenas fornecem um parente / leitura quantitativa.- Ligue sistema de microscópio (PC para a operação, microscópio, câmera CCD, roda de filtros e lâmpadas de xénon).
- Abra o software designado.
- Configure microscópio / software para multicanal automatizada de lapso de tempo de imagem. Incluem a aquisição sequencial de anúncioifferential contraste de interferência (DIC), fluorescência verde padrão, fluorescência vermelha padrão e padrão de excitação 340 e 380 nm Fura-2 imagens. Defina o intervalo para a aquisição de 10-30 seg e uma duração total de ~ 20-60 min.
- Definir tempos de exposição para Fura-2 imagem.
- Adicione o óleo fresco e objetivo montar um prato microscópio contendo apenas 0,5 ml de Tampão-A no adaptador fase de aquecimento e imediatamente ligar para equilibrar a 37 ° C (~ levará 2-3 min).
- Adicionar descansando linfócitos Fura2-carregados para a câmara de microscópio prato montado utilizando uma pipeta de 20 mL.
- Ligue brilhante imaging campo. Selecione o caminho de luz para as oculares. Use o botão de foco aproximado para trazer objetivo em contato com fundo do prato miscoscope. Use a ocular e o botão de foco fino para se concentrar nas células T se instalaram no fundo do prato.
- Use os controles de palco xy para selecionar um campo que contenha pelo menos 10 células. Evitarsuperlotado campos e aglomerados de células como estes irão criar artefatos de imagem.
- Mudar de campo claro a fluorescente fonte de luz. Mudar de imagem para a câmera CCD ocular. Definir parâmetros de aquisição (por exemplo, tempo de exposição, ganho de detector e binning). Utilizando o software de aquisição de imagens e de repouso Fura2-340 Fura2-380 (começando com o tempo de exposição idêntico para cada um, geralmente na gama de 200-1.000 ~ mseg).
- Utilizar os métodos descritos no Passo 5.4.1 para calcular o Fura2-340 / Fura2-380 para cada linfócitos. Execute repetidas iterações do ajuste da Fura2-340 e tempos de exposição Fura2-380, aquisição de imagens e cálculo dos rácios até que os valores médios estão próximos de 1.
- Definir tempos de exposição para mem-YFP e DsRed.
- Substitua o prato microscópio usado no passo 5.3.3.1 com um prato contendo miscroscope transfectadas, ativado e sag tratada (ou não tratada; controle) HLMVECs ou HDMVECs de cultura de células incubadora (Passos 4.5.1). Usar uma pipeta de transferência descartável para remover rapidamente meios, lavar uma vez por adição de ~ 1 ml de tampão de pré-aqueceu-A. Aspirar e, em seguida, adicionar 0,5 ml de tampão-A.
- Identificar áreas em que as células endoteliais transfectantes positivos brilhantemente fluorescentes estão presentes e parecem saudáveis com junções intercelulares bem formadas.
- Ajustar os parâmetros de aquisição (por exemplo, tempo de exposição, ganho de detector e binning) para mem-YFP e DsRed. Certifique-se de que a intensidade de sinal de fluorescência média em cada canal se situe entre 25% e 75% da gama dinâmica do detector.
- Definir tempos de exposição para Fura-2 imagem.
- Realizar experimento de imagem de células vivas.
- Use um software automatizado para começar a aquisição de imagem e capturar vários intervalos de imagens para estabelecer linha de base.
- Durante a aquisição de aplicar ~ 5 jil de linfócitos de Fura-2-carregados concentradas a partir do passo (5.2) para o centro do campo de imagem de microscópio prato, inserindo a ponta de um pequeno volume (P-5ou P-20) da pipeta na mídia, perto do centro do objetivo e ejetar lentamente.
- Como linfócitos resolver no campo de imagem, fazer ajustes finos no foco para assegurar que a interface celular de células-T (endotélios sinapses imunológicas) são mantidas no plano focal. Com 40 e objetiva 63X, ~ 10-20 células por campo são ideais. Se menos células são observados no campo de imagem repita o passo 5.3.4.2.
- Após o intervalo de observação desejada de experiência é competiu, continuar a imagiologia imediatamente ionomicina e pipeta directamente para o prato miscoscope (utilizando uma técnica tal como no 5.3.4.2) a uma concentração final de 2 ^ M para induzir o fluxo máximo de cálcio sinal de sinalização / Fura-2 (ou seja , um meio de calibragem, Ver análise 5.4.).
- Como uma alternativa para o intervalo de observação 5.3.4.4after desejado de experiência é competiu, aspirar imediatamente o tampão A e substituí-la com 0,5 ml de solução fixadora (formaldeído a 3,7% em PBS) durante 5 min aRT, seguido por lavagem três vezes com PBS. Em seguida, avance para a etapa 6.
- Análise de imagem Live-célula
NOTA: Depois de salvar os arquivos adquiridos, eles podem ser analisados diretamente ou exportados para análise por uma ampla variedade de aplicações de software de análise de imagem off-line. ImageJ é um particularmente valioso pacote, altamente versátil que é livremente disponível e compatível com quase todos os pacotes de software de aquisição. O desenho dos experimentos de imagem descritos nos passos 5,1-5,3 produzirá dinâmica de resolução espacial e temporal elevadas de interacções entre linfócitos e APC endotelial na ausência e na presença de cognato ceder. Quase análises ilimitadas são possíveis quando abordando dados de imagem da dinâmica morfométricas / sinalização celular. Os objectivos específicos descritos neste protocolo específico são quantificar coordenadamente migração de linfócitos e sinalização (ou seja, cinética e níveis de fluxo de cálcio intracelular), juntamente com dinâmicaal mudanças na arquitetura sinapse imunológica com foco em características específicas (ou seja, ILPS / PODO-prints). Os seguintes são exemplos de padrão separado e não-padrão (ou seja, desenvolvido especificamente para estas experiências / perguntas) analisa.- Medir Linfócitos fluxo de cálcio
- Seleccione a inicial Fura2-340 (340 nm-510 nm EX EM) imagens e Fura2-380 (380 nm EX-510 nm EM) que tenham sido adquiridos antes da adição de linfócitos. Use-os como imagens de fundo e subtrai-los digitalmente a partir do todas as imagens na série de tempo para cada respectivo canal.
- Para cada ponto de tempo, criar uma relação da imagem digital do subtraído-fundo Fura2-340 e as imagens Fura2-380 subtraído-fundo (ou seja, 340 nm EX-510 nm EM-fundo / 380 nm EX-510 nm EM-fundo).
- Selecione a ferramenta de desenho do software apropriado. Clique na tela para desenhar uma região circular de interest (ROI) em torno de cada linfócito. Estes irão gerar um valor de proporção média-pixel para cada linfócito e prazo.
- Traça-se a proporção calculada como uma função do tempo utilizando uma aplicação de software apropriado (por exemplo, programa de folha de cálculo). Calcule a média do fluxo de cálcio para o ensaio pela soma dos valores da relação de todos os linfócitos por campo e dividindo esse valor por o número total de linfócitos para cada ponto de tempo
- Realizar lympocyte análise de monitoramento de migração.
- Analisar a migração de células T de células utilizando uma aplicação adequada de rastreamento de pilha de software (por exemplo, ImageJ), utilizando o canal DIC de cada vídeo. Usando ImageJ celular Rastreamento Plugin, identificar em cada frame o centróide de cada célula manualmente, clicando sobre ela em cada quadro progressivo.
- Use as coordenadas xy de série resultantes (ou seja, traços de caminhos de migração) para calcular a velocidade média (distância total migraram intervalo total / of de imagem) e a tortuosidade (distância total migrado / a distância linear de ponta a ponta entre o local de célula na primeira e na última imagem).
- Cross-correlacionar estes parâmetros com o fluxo de cálcio (5.4.1) dinâmica numa base de célula-por-célula.
- Avaliar a densidade Podo-print / ILP dentro do IS
- Contagem total de ILP formada em cada célula T intervalos é definido (por exemplo, 5 min após a adição de células T). Estes podem ser identificados fluorescentes anéis discretos escala mícron membrana-YFP que co-localizam com círculos escuros em citoplasmática DsRed na APC endotelial num local de adesão de linfócitos.
- Cross-correlacionar os índices de PLI resultantes '' com o fluxo de cálcio (5.4.1) dinâmica numa base célula-a-célula.
- Medir o podo-print / vidas de PLI.
- Para cada podo-print / ILP em uma dada é calcular suas vidas como o último ponto de tempo quando um ILP era visível - o ponto de tempo quando que podo-print / ILP fiapareceu primeiro. Média ILP vidas tanto por IS.
- Cross-correlacionar estas vidas com o fluxo de cálcio (5.4.1) dinâmica numa base célula-a-célula.
- Avaliar a relação temporal entre a formação inicial e ILP fluxo de cálcio.
- Para cada linfócito identificar o ponto de tempo (frame) na qual o cálcio nasce primeiro acima da linha de base 19.
- Para cada linfócitos identificar o ponto temporal em que o primeiro Podo-print / ILP aparece.
- Para cada linfócitos calcular um "tempo de deslocamento 'subtraindo o ponto inicial de tempo o fluxo de cálcio a partir do inicial que Podo-impressão / PLI. Calcular a média dos valores para todos os linfócitos em um campo. Os valores positivos indicam a formação ILP precede o fluxo de cálcio enquanto que números negativos indicam o oposto.
NOTA: Estas experiências podem facilmente ser realizado sob fluxo laminar cisalhamento fisiologicamente relevante usando câmaras de imagem de fluxo de parede paralela comercialmente disponíveis como descrito 19.
- Medir Linfócitos fluxo de cálcio
Imagem e Análise 6. células fixo
- Endpoint fixo T célula endotelial é a formação e coloração
- Preparar células T tal como descrito em 1. Preparar células endoteliais (- / + SAG) como no Passo 5.1 (transfecção, Passo 5.1.1 é opcional). Tomar uma amostra de linfócitos em cultura e determinar a densidade, contando com um hemocitômetro (Seção 1).
- Transferir um volume de cultura de linfócitos equivalente a pelo menos 3 x 10 5 linfócitos / amostra para um tubo de 15 ml e centrifugar a 200 xg durante 3 min. Remover o sobrenadante e ressuspender o sedimento em tampão de linfócitos-A (Passo 5.2.1) concentração de 6 x 10 5 / ml de linfócitos.
- Remover as células endoteliais da incubadora de cultura de células e (Procedendo uma amostra de cada vez) meios sucessivamente aspirados e substituir com 0,5 ml de suspensão de linfócitos (3 x 10 5 linfócitos / amostra) e para substituir 37 ° C incubator.
- Após períodos de incubação adequados, observado acima, o meio aspirado dos poços e adicionar solução suficiente fixador (formaldeído a 3,7% em PBS) para cobrir completamente a amostra (por exemplo, na placa de 24 poços ~ 300-500 uL). Incubar à temperatura ambiente durante 5-10 min.
- Lavar a amostra 3 vezes com PBS. Manchas por adição de anticorpo primário durante 60 min à temperatura ambiente (ver a lista). Se o anticorpo primário é dirigido a uma proteína intracelular, um passo adicional de permeabilização precisa de ser realizada por adição de solução de permeabilização suficiente (0,01% Triton X-100 em PBS) para cobrir completamente a amostra durante 1 min à temperatura ambiente. Lavar a amostra 3 vezes com PBS. Adicionar anticorpos secundários adequados (em alguns casos faloidina fluorescente concomitantemente) durante 60 min à temperatura ambiente.
- Monte a lamela de vidro circular em um slide de imagem. Endpoint fixo T célula endotelial é a formação e coloração. Para começar lugar de imagem do slide de imagem contendo amostras no palco microscópio e selecione o óleo 63Xobjetivo. Aplicar óleo de imersão fresco.
- Use o modo de imagem de campo brilhante e lentes oculares para localizar o plano focal. Mudar para epifluorescência e inspecionar amostra através das lentes oculares. Selecione um campo de interesse. Alternar para o modo de varredura a laser.
- Usando o modo rápido-scan e trabalhando no maior campo possível ajustar individualmente a potência do laser e ganho de cada canal para optimização sinal de tal forma que, idealmente, a intensidade do sinal específico atinge pelo menos ~ 25% e não mais de 75% da gama dinâmica dos detectores.
- O uso de controles de foco manual, varredura rapidamente através do eixo Z e identificar os limites superior e inferior de corte (normalmente todas as informações devem ser contidos dentro de uma espessura de ~ 15 mm). Seleccionar a espessura de corte do eixo Z na gama de ~ 0,2-1,0 uM.
- Finalmente, zoom / recortar o campo de imagem para a região específica de interesse e conduta digitalização. Além de ter muito brilhante e específica si fluorescêncianal na amostra, a aquisição de alta resolução 3D-imaging requer iterações de digitalização e fazer ajustes para os parâmetros de aquisição. O objetivo é obter xy máxima e resolução do eixo z, sem apreciável foto-branqueamento.
- Qualitativamente analisar a topologia da topologia 3D da IS através da realização de reconstrução digital 3D das seções ópticas resultantes através de um aplicativo de software adequado (por exemplo, J Imagem). Através de aplicações semelhantes avaliar quantitativamente a distribuição do sinal de fluorescência (por exemplo, co-localização e intensidade de fluorescência correlativo linha-scan análise de Pearson e visualização ortogonal).
Resultados
Uma nova abordagem de imagem usando células endoteliais e que combina as vantagens de resolução do cartão planar bicamadas lipídicas modelo com a complexidade fisiológica e deformabilidade da APC profissional foi desenvolvido (Figura 1). A Figura 2 proporciona exemplos de migração típica, fluxo de cálcio e dinâmica topológica observada com este abordagem. Na ausência de retracção no endotélio, os linfócitos T CD4 + Th1 SAg esp...
Discussão
No geral, este protocolo descreve métodos para a investigação de células endoteliais como i) APCs fisiológicos pouco estudado e ii) como um novo tipo de "modelo APC celular planar". Com relação ao primeiro, tornou-se cada vez mais apreciado que APCs não hematopoiéticas periférica (ou 'estromais') desempenham papéis, não redundantes críticos (ou seja, em comparação com hematopoiéticas APCs) na formulação de respostas imunes adaptativas 16-18. Entre tais "semi-p...
Divulgações
The authors have nothing to disclose.
Agradecimentos
We thank Dr. Peter T. Sage for his assistance in generating some of the representative images. This work was supported by an NIH R01 grant to C.V.C. (HL104006).
Materiais
| Name | Company | Catalog Number | Comments |
| BD Vacutainer stretch latex free tourniquet | BD Biosciences | 367203 | |
| BD alcohol swabs | BD Biosciences | 326895 | |
| BD Vacutainer Safety-Lok | BD Biosciences | 367861 | K2 EDTA |
| BD Vacutainer Push Button Blood Collection Set | BD Biosciences | 367335 | |
| RPMI-1640 | Sigma-Aldrich | R8758-1L | |
| Ficoll-Paque | Sigma-Aldrich | GE17-1440-02 | Bring to RT before use |
| FCS-Optima | Atlanta Biologics | s12450 | Heat inactivated |
| Penicillin-Streptomycin | Sigma-Aldrich | P4458-100ML | |
| Trypan blue | Sigma-Aldrich | T8154-20ML | |
| staphylococcal enterotoxin B | Toxin Technology | BT202RED | Stock solution 1mg/ml in PBS |
| toxic shock syndrome toxin 1 | Toxin Technology | TT606RED | Stock solution 1mg/ml in PBS |
| human IL-15 | R&D Systems | 247-IL-025 | Stock solution 50ug/ml in PBS |
| PBS | Life Technologies | 10010-049 | |
| Fibronectin | Life Technologies | 33016-015 | Stock solution 1mg/ml in H20 |
| HMVEC-d Ad-Dermal MV Endo Cells | Lonza | CC-2543 | Other Human Microvascular ECs can be used, i.e. HLMVECs |
| EGM-2 MV bullet kit | Lonza | CC-3202 | |
| Trypsin-EDTA | Sigma-Aldrich | T-4174 | Stock solution 10x, dilute in PBS |
| amaxa-HMVEC-L Nucleofector Kit | Lonza | vpb1003 | Required Kit for step 4 |
| IFN-g | Sigma-Aldrich | I3265 | Stock solution 1mg/ml in H20 |
| TNF-alpha 10ug, human | Life Technologies | PHC3015 | Stock solution 1mg/ml in H20 |
| phenol Red-free HBSS | Life Technologies | 14175-103 | |
| Hepes | Fisher Scientific | BP299-100 | |
| Calcium Chloride | Sigma-Aldrich | C1016-100G | Stock solution 1M in H20 |
| Magnesium chloride | Sigma-Aldrich | 208337 | Stock solution 1M in H20 |
| Human Serum albumin | Sigma-Aldrich | A6909-10ml | |
| Immersol 518 F fluorescence free Immersion oil | Fisher Scientific | 12-624-66A | |
| Fura-2 AM 20x50ug | Life Technologies | F1221 | Stock solution 1mM in DMSO |
| pEYFP-Mem (Mem-YFP) | Clontech | 6917-1 | |
| pDsRed-Monomer (Soluble Cytoplasmic DsRed) | Clontech | 632466 | |
| pDsRed-Monomer Membrane (Mem-DsRed) | Clontech | 632512 | |
| pEGFP-Actin | Clontech | 6116-1 | |
| Alexa Fluor 488 Phalloidin | Life Technologies | A12379 | |
| Formaldehyde solution 37% | Fisher Scientific | BP531-500 | Toxic, use fumehood |
| Triton X-100 | Sigma-Aldrich | X100-5ML | |
| Falcon 15mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-70C | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-49A | |
| Falcon Tissue Culture Treated Flasks T25 | Fisher Scientific | 10-126-9 | |
| Falcon Tissue Culture Treated Flasks T75 | Fisher Scientific | 13-680-65 | |
| Corning Cell Culture Treated T175 | Fisher Scientific | 10-126-61 | |
| Glass coverslips | Fisher Scientific | 12-545-85 | 12 mm diameter |
| Falcon Tissue Culture Plates 24-well | Fisher Scientific | 08-772-1 | |
| Delta-T plates | Bioptechs | 04200415B | |
| Wheaton Disposable Pasteur Pipets | Fisher Scientific | 13-678-8D | |
| 1.5 ml Eppendorf tube | Fisher Scientific | 05-402-25 | |
| ICAM1 mouse anti-human | BD Biosciences | 555509 | |
| HS1 mouse anti-human | BD Biosciences | 610541 | |
| Anti-Human CD11a (LFA-1alpha) Purified | ebioscience | BMS102 | |
| Anti-Human CD3 Alexa Fluor® 488 | ebioscience | 53-0037-41 | |
| Anti-MHC Class II antibody | Abcam | ab55152 | |
| Anti-Talin 1 antibody | Abcam | ab71333 | |
| Anti-PKC theta antibody | Abcam | ab109481 | |
| phosphotyrosine (4G10 Platinum) | Millipore | 50-171-463 | |
| Nucleofector II | Amaxa Biosystems | Required electroporator for step 4 | |
| Zeiss Axiovert | Carl Zeiss MicroImaging | ||
| Zeiss LSM510 | Carl Zeiss MicroImaging | ||
| Zeiss Axiovison Software | Carl Zeiss MicroImaging | ||
| NU-425 (Series 60) Biological Safety Cabinet | NuAIRE | Nu-425-600 | |
| Forma STRCYCLE 37 °C, 5% CO2 Cell culture Incubator | Fisher Scientific | 202370 | |
| Centrifuge 5810 | Eppendorf | EW-02570-02 | |
| Hemocytometer | Sigma-Aldrich | Z359629 | Bright-Line Hemocytometer |
| Isotemp Waterbath model 202 | Fisher Scientific | 15-462-2 |
Referências
- von Andrian, U. H., Mackay, C. R. T-cell function and migration. Two sides of the same coin. N. Engl. J. Med. 343 (14), 1020-1034 (2000).
- Springer, T. A. Adhesion receptors of the immune system. Nature. 346 (6283), 425-434 (1990).
- Shaw, A. S., Dustin, M. L. Making the T cell receptor go the distance: a topological view of T cell activation. Immunity. 6 (4), 361-369 (1997).
- Monks, C. R., Freiberg, B. A., Kupfer, H., Sciaky, N., Kupfer, A. Three-dimensional segregation of supramolecular activation clusters in T cells. Nature. 395 (6697), 82-86 (1998).
- Delon, J., Stoll, S., Germain, R. N. Imaging of T-cell interactions with antigen presenting cells in culture and in intact lymphoid tissue. Immunol Rev. 189, 51-63 (2002).
- Brossard, C., et al. Multifocal structure of the T cell - dendritic cell synapse. Eur J Immunol. 35 (6), 1741-1753 (2005).
- Dustin, M. L. The cellular context of T cell signaling. Immunity. 30 (4), 482-492 (2009).
- Balagopalan, L., Sherman, E., Barr, V. A., Samelson, L. E. Imaging techniques for assaying lymphocyte activation in action. Nat Rev Immunol. 11 (1), 21-33 (2011).
- Cebecauer, M., Spitaler, M., Serge, A., Magee, A. I. Signalling complexes and clusters: functional advantages and methodological hurdles. J Cell Sci. 123, 309-320 (2010).
- Oddos, S., et al. High-speed high-resolution imaging of intercellular immune synapses using optical tweezers. Biophys J. 95 (10), L66-L68 (2008).
- Grakoui, A., et al. The immunological synapse: a molecular machine controlling T cell activation. Science. 285 (5425), 221-227 (1999).
- Bunnell, S. C., et al. T cell receptor ligation induces the formation of dynamically regulated signaling assemblies. J Cell Biol. 158 (7), 1263-1275 (2002).
- Yokosuka, T., et al. Newly generated T cell receptor microclusters initiate and sustain T cell activation by recruitment of Zap70 and SLP-76. Nat Immunol. 6 (12), 1253-1262 (2005).
- Seminario, M. C., Bunnell, S. C. Signal initiation in T-cell receptor microclusters. Immunol Rev. 221, 90-106 (2008).
- Dustin, M. L. Supported bilayers at the vanguard of immune cell activation studies. J Struct Biol. 168 (1), 152-160 (2009).
- Martinelli, R., Carman, C. V., Bradshaw, R. A., Stahl, P. Lymphocyte Endothelilal Interactions. Encyclopedia of Cell Biology. , (2015).
- Choi, J., Enis, D. R., Koh, K. P., Shiao, S. L., Pober, J. S. T lymphocyte-endothelial cell interactions. Annu Rev Immunol. 22, 683-709 (2004).
- Marelli-Berg, F. M., Jarmin, S. J. Antigen presentation by the endothelium: a green light for antigen-specific T cell trafficking?. Immunol Lett. 93 (2-3), 109-113 (2004).
- Sage, P. T., et al. Antigen recognition is facilitated by invadosome-like protrusions formed by memory/effector T cells. J Immunol. 188 (8), 3686-3699 (2012).
- Kumari, S., et al. Actin foci facilitate activation of the phospholipase C-gama in primary T lymphocytes via the WASP pathway . eLife. , (2015).
- Marelli-Berg, F. M., et al. Major histocompatibility complex class II-expressing endothelial cells induce allospecific nonresponsiveness in naive T cells. J Exp Med. 183 (4), 1603-1612 (1996).
- Ma, W., Pober, J. S. Human endothelial cells effectively costimulate cytokine production by, but not differentiation of, naive CD4+ T cells. J Immunol. 161 (5), 2158-2167 (1998).
- Perez, V. L., Henault, L., Lichtman, A. H. Endothelial antigen presentation: stimulation of previously activated but not naive TCR-transgenic mouse T cells. Cell Immunol. 189 (1), 31-40 (1998).
- Epperson, D. E., Pober, J. S. Antigen-presenting function of human endothelial cells. Direct activation of resting CD8 T cells. J Immunol. 153 (12), 5402-5412 (1994).
- Shiao, S. L., et al. Human effector memory CD4+ T cells directly recognize allogeneic endothelial cells in vitro and in vivo. J Immunol. 179 (7), 4397-4404 (2007).
- Marelli-Berg, F. M., Okkenhaug, K., Mirenda, V. A two-signal model for T cell trafficking. Trends Immunol. 28 (6), 267-273 (2007).
- Carman, C. V., et al. Transcellular diapedesis is initiated by invasive podosomes. Immunity. 26 (6), 784-797 (2007).
- Carman, C. V. Mechanisms for transcellular diapedesis: probing and pathfinding by 'invadosome-like protrusions'. J Cell Sci. 122 ((Pt 17)), 3025-3035 (2009).
- Dustin, M. L., Tseng, S. Y., Varma, R., Campi, G. T. T cell-dendritic cell immunological synapses. Curr Opin Immunol. 18 (4), 512-516 (2006).
- Saito, T., Yokosuka, T. Immunological synapse and microclusters: the site for recognition and activation of T cells. Curr Opin Immunol. 18 (3), 305-313 (2006).
- Gomez, T. S., et al. HS1 functions as an essential actin-regulatory adaptor protein at the immune synapse. Immunity. 24 (6), 741-752 (2006).
- Burbach, B. J., Medeiros, R. B., Mueller, K. L., Shimizu, Y. T-cell receptor signaling to integrins. Immunol Rev. 218, 65-81 (2007).
- Vicente-Manzanares, M., Sanchez-Madrid, F. Role of the cytoskeleton during leukocyte responses. Nat Rev Immunol. 4 (2), 110-122 (2004).
- Ma, Z., Janmey, P. A., Finkel, T. H. The receptor deformation model of TCR triggering. Faseb J. 22 (4), 1002-1008 (2008).
- Ma, Z., Sharp, K. A., Janmey, P. A., Finkel, T. H. Surface-anchored monomeric agonist pMHCs alone trigger TCR with high sensitivity. PLoS Biol. 6 (2), e43 (2008).
- Groves, J. T. Bending mechanics and molecular organization in biological membranes. Annu Rev Phys Chem. 58, 697-717 (2007).
- Xu, C., et al. Regulation of T cell receptor activation by dynamic membrane binding of the CD3epsilon cytoplasmic tyrosine-based motif. Cell. 135 (4), 702-713 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados