Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une cellule endothéliale Planar Modèle for Imaging Dynamics Synapse immunologique
Dans cet article
Résumé
Adaptive immunity is controlled by dynamic 'immunological synapses' formed between T cells and antigen presenting cells. This protocol describes methods for investigating endothelial cells both as understudied physiologic APCs and as a novel type of 'planar cellular APC model'.
Résumé
L'immunité adaptative est régulée par des interactions dynamiques entre les cellules T et les cellules présentatrices d'antigènes ("APC") appelés «synapses immunologiques». Au sein de ces interfaces cellule-cellule intimes grappes de sous-cellulaire discrètes de CMH / Ag-TCR, la F-actine, l'adhérence et des molécules de signalisation forment et remodeler rapidement. Ces dynamiques sont pensés pour être des facteurs déterminants de l'efficacité et la qualité des réponses immunitaires qui développent et donc de protection contre l'immunité pathologique. La compréhension actuelle des synapses immunologiques avec physiologique APC est limitée par l'insuffisance de la résolution obtenue d'imagerie. Bien que les modèles de substrat artificiel (par exemple, des bicouches lipidiques planes) offrent une excellente résolution et ont été des outils extrêmement précieux, ils sont intrinsèquement non-physiologique et simpliste. Les cellules endothéliales vasculaires et lymphatiques ont émergé comme un tissu périphérique importante (ou stroma) compartiment de 'semi-professional APC ». Ces APC (qui expriment la plupart de la machinerie moléculaire des APC professionnelle) ont la particularité de former surface cellulaire pratiquement plane et sont facilement transfectable (par exemple, avec les journalistes de protéines fluorescentes). Ici une approche de base pour mettre en œuvre les cellules endothéliales comme un roman et physiologique »plane modèle APC cellulaire» pour l'amélioration de l'imagerie et de l'interrogatoire des processus fondamentaux de signalisation antigéniques sera décrit.
Introduction
Les lymphocytes T sont une branche du système immunitaire adaptatif caractérisé par la capacité de reconnaître efficacement antigène peptidique (Ag) lié à complexe majeur d'histocompatibilité (CMH) à travers leurs récepteurs de lymphocytes T (TCR) 1. Lymphocytes naïfs migrent constitutivement et Scan 'Ag cellules présentatrices professionnels »(CPA; par exemple, les cellules dendritiques) dans les ganglions lymphatiques, tandis que les cellules mémoire / T effecteurs doivent pouvoir surveiller efficacement une gamme extrêmement large d'APC et les cellules cibles potentielles dans les tissus périphériques.
Dans le min après la comptabilisation initiale de l'apparenté Ag sur une APC, les lymphocytes arrêter leur migration et de commencer à former intime une interface spécialisée cellule-cellule appelée "synapse immunologique" (IS). Soutenue (c.-à 30-60 min) EST contacts sont nécessaires pour amplifier et soutenir signalisation 2-7. Nouvelles études identifient que dans le SI, il est la formation continue et r rapideemodeling de signalisation sous-cellulaire des micro-amas discrets (par exemple, contenant du CMH / Ag-TCR, la F-actine, l'adhérence et des molécules de signalisation) qui déterminent la résistance et la qualité de la réponse immunitaire résultante 2-7. Cependant, les détails dynamiques et mécanisme de régulation de ce processus sont encore mal compris 8,9. Cette situation découle en grande partie des défis techniques liés aux topologies de surfaces irrégulières APC et l'orientation mal contrôlée des plans d'interaction cellule-cellule, les problèmes qui limitent profondément l'imagerie spatio-temporelle requise approches 8-10 (Figure1A).
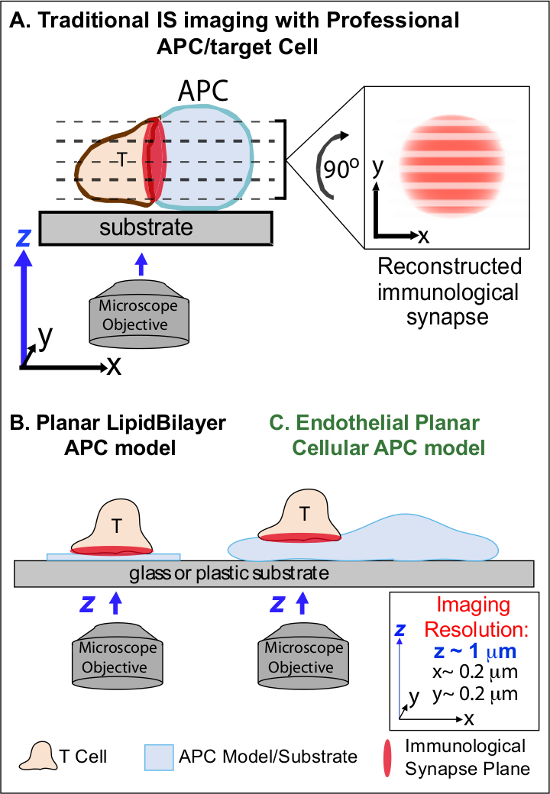
Figure 1. Un physiologique Planar cellulaire APC Modèle pour l'imagerie immunologique Synapse Dynamics. Le schéma illustre l'imagerie traditionnelle de la synapse immunologique entre un lymphocyte T et un professio APC nal (A) et des cellules T et un modèle traditionnel plane lipidique bicouche APC (B) par rapport à ce nouveau modèle plane APC endotheliales (C). APC professionnelles fournissent des synapses immunologiques physiologiques, mais offrent l'interface mal orienté cellule-cellule (par exemple, par rapport au plan optimal de formation d'image xy, la résolution ~ 0,2 pm), ce qui compromet considérablement spatial (z imagerie avion résolution ~ 1 um) et temporelle (par exemple, en raison de la nécessité de numériser plusieurs reprises à travers tous les plans z d'imagerie) résolution de l'imagerie. Modèles bicouches ont une topologie planaire qui fournit une résolution spatio-temporelle optimale d'imagerie, mais sont également très simplifiées, non physiologique et rigide. Ce modèle associe des cellules endotheliales de la topologie du plan de bicouches lipidiques avec le substrat physiologique d'un APC classique pour fournir la résolution de l'imagerie spatiale et temporelle optimale dans un contexte physiologique.m / files / ftp_upload / 53288 / 53288fig1large.jpg "target =" _ blank "> S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Des travaux antérieurs ont partiellement contourné ces obstacles en élaborant des modèles de substrat plan (c.-à-bicouches lipidiques et des surfaces revêtues d'anticorps) qui fournissent la résolution spatio-temporelle optimale (ie, par l'intermédiaire de la fixation de la surface de l'activation des cellules T dans un plan unique qui est parallèle à l'imagerie xy optimale plan) 11 à 15 (figure 1B). Ces modèles ont facilité des informations importantes sur la dynamique sous-cellulaires / moléculaires qui contrôlent la signalisation antigénique des lymphocytes T, y compris la découverte d'actine dynamique / TCR de signalisation micro-clusters 7,11-14. Toutefois, ces modèles sont intrinsèquement trop simplifiées, ainsi que rigide (excluant le développement / étude des caractéristiques topologiques 3 dimensions) (figure 1B). Par conséquent, il reste incertain comment relier ces résultats à PHYcellule-cellule siologic de la surveillance immunitaire.
Bien que toujours sous-étudié, vasculaire et les cellules endothéliales lymphatiques apparaissent comme un grand (ie, plus nombreux que tous les APC professionnelles, par ~ 1000 fois) compartiment périphérique de «semi-professionnel» APC 16-18. Ces cellules expriment MHC-I, MHC-II et une multitude de molécules co-stimulatrices (par exemple, CD40, LFA3, ICOSL, 4-1BB, OX40L, TL1A, PD-L1; mais pas CD80 et CD86) et sont stratégiquement positionné à l'interface sang-tissus où ils remplissent des fonctions sentinelles spécialisées 16-18. Des études antérieures ont démontré que les cellules endothéliales peuvent effectivement re-stimuler effecteur / mémoire, mais pas naïfs, les cellules T 19-25. Ainsi, les cellules endotheliales sont susceptibles de jouer un rôle unique APC en phase effectrice de la réponse immunitaire adaptative dans les tissus périphériques, tels que l'influence locale sur activation des lymphocytes T, la différenciation, de la mémoire et de la tolérance 16,17,26. Critiquement, lorsqu'elles sont cultivées in vitro, les cellules endotheliales forment des surfaces planes pratiquement cellulaires et sont facilement transfectables (par exemple, avec les reporters de protéines fluorescentes). Ces caractéristiques sont idéales pour la résolution spatio-temporelle de haute imagerie de la dynamique topologiques au cours des interactions cellule-cellule 19,27. Ainsi, les cellules endothéliales pourraient servir de «cellulaire plane APC« modèle physiologique distinctement adapté pour l'étude des mécanismes de remodelage sous-cellulaires / moléculaires qui conduisent reconnaissance de l'antigène et régulent les réponses (figure 1C) 19,20.
Auparavant établi des techniques d'imagerie complémentaires (y compris la transfection de cellules endotheliales avec une machine de protéines fluorescentes de la membrane plasmique et cytosol) pour étudier les détails de l'interaction des leucocytes-endothélial au cours de l'adhérence et de la migration transendotheliale 27, ont montré que les leucocytes sonde active la surface de l'endothélium par dynamique l'insertion d'und rétraction de sous-échelle du micron, protubérances cylindriques actine riche (~ 200-1,000 nm de diamètre et de profondeur) appelé invadosome-comme des saillies (ie, "PAI") 27,28. Ces approches d'imagerie ont été encore élargi avec la création de protocoles de profiter de la fonction endothéliale APC pour développer les premières méthodes de résolution spatio-temporelle de l'imagerie haute cellule T-endothélial synapse immunologique comme rapporté 19,20 et décrire plus en détail ici. Une conclusion centrale dérivé de ce roman plane cellulaire modèle APC est que PAI de cellules T fonctionnent à la fois dans la promotion de la détection initiale Ag et dans le maintien de la signalisation subséquente. En effet, les tableaux de multiples PAI (qui ont été stabilisés et comptabilisée en réponse à parapher le flux de calcium) montrent un enrichissement en molécules TCR et suggestives de la signalisation actif tel PKC-Q, ZAP-70, phosphotyrosine et HS1. Par conséquent, PAI semblent représenter une tridimensionnel équivalent physiologique de la micro-TCR de signalisationgrappes observés dans les modèles à deux couches planes. Cette approche, donc, révèle sensible / rapports dynamique moléculaire et architecturaux (et implicites biomécanique) pas autrement détectable.
La méthode décrite ici devrait être utile à combler le fossé entre les APC professionnelle et modèles artificiels de substrat APC afin d'améliorer notre capacité d'interroger les mécanismes de base de réponses immunitaires adaptatives. Alors que l'accent est mis ici sur l'activation de CD4 + de type Th1 effecteur / cellule de mémoire, cette approche de base peut être facilement modifié pour étudier un large éventail de types et de Ags de cellules T, comme on le verra ci-dessous.
Protocole
Toutes les expériences décrites dans ce protocole sont menées avec des cellules T humaines primaires et des cellules primaires humaines disponibles dans le commerce endothéliales (cutanée ou microvasculaire du poumon EC) .Tout protocole de recherche impliquant des sujets humains doit être approuvée par un comité d'examen institutionnel et le consentement éclairé écrit doit être fourni à partir de chaque donneur de sang. Des expériences menées en utilisant ce protocole a été approuvé par la CISR de Beth Israel Deaconess Medical Center.
1. préparation de cellules CD4 + Th1 humain effecteur / T mémoires
- Appliquer garrot au bras du donneur, essuyez veine avec l'alcool, et insérer l'aiguille. Aspirez lentement 15 ml de sang dans vacutainer utilisant de l'EDTA comme anticoagulant. Quand le sang a été établi, dénouer le garrot avant de retirer l'aiguille. Appliquer immédiatement une pression sur la plaie avec de la gaze stérile lorsque l'aiguille est retirée.
- Transférer le sang dans un tube de 50 ml. Ajouter RPMI-1640 à la température ambiante à une dilution 1: 1 (volume final 30 ml). Superposer délicatement le diluéed sang sur deux tubes de 50 ml contenant 15 ml de pré-filtré lymphocytes moyen d'isolation tel que Ficoll-Paque à température ambiante.
- Centrifuger le gradient à température ambiante pendant 30 min à 1200 x g dans un rotor à godets oscillants. Alors que la centrifugation de préparer un milieu de cellules T (500 ml de RPMI-1640, 50 ml de FCS, 5 ml de pénicilline / streptomycine).
- Lors du retrait du tube de 50 ml de la centrifugeuse, d'observer quatre couches: une pastille de globules rouges dans le fond, la paque, une couche de cellules qui contient les globules blancs sanguins (y compris les lymphocytes) et le plasma. Retirez délicatement la couche blanche de cellules de sang avec une pipette pasteur et le transfert dans un tube Falcon de 50 ml.
- Laver la couche de globules blancs en ajoutant du milieu RPMI-1460 RT (jusqu'à 20 ml) et la centrifugation à température ambiante pendant 5 min à 1200 x g. Remettre en suspension les globules blancs dans 1 ml de milieu de cellule T. Ajouter 5 ul de la suspension cellulaire 1 ml de milieu de cellule T de 250 pi dans un tube à centrifuger de 1,5 ml et mélanger doucement vers le haut et vers le bas par pipette.
- Ajouter 25 ul diluentd suspension cellulaire à 25 ul de 0,4% bleu Trypan. Ajouter 10 pi de mélange de chaque côté d'un hémocytomètre standard.
- Placez hémocytomètre sur une faible microscope optique de puissance. L'utilisation d'un objectif 10X compter le nombre de cellules qui ont exclu le colorant bleu Trypan et sont présents dans le carré du milieu des deux côtés de la hémocytomètre vivre.
- Pour calculer la concentration cellulaire, multiplier la moyenne des 2 carrés par 100 (facteur de dilution) et puis multiplier par 10 4 pour donner le nombre de cellules / ml.
- Ajuster la concentration finale de 0,5 x 10 6 cellules / ml dans du milieu de cellules T. Ajouter une concentration finale de 1 pg / ml de chacune des superantigènes bactériens entérotoxine staphylococcique B (SEB) et à toxines de syndrome de choc toxique 1 (TSST) aux cellules. Culture pendant 72 heures (37 ° C et 5% de CO 2) pour élargir la population de cellules T CD4 +.
- Pellet cellules T (1200 xg, 5 min) et les remettre en suspension à 0,5 x 10 6 cellules / ml dans du milieu de cellules T avec laaddition d'IL-15 humaine (20 ng / ml). Transfert des lymphocytes dans un flacon T150. Continuer à développer / fractionnement de cellules en plein moyen-IL-15 tous les 24-48 heures selon les besoins (basées sur la couleur des médias;-à-dire, chaque fois que les médias se détourne de rose à légèrement jaune) par la suite. Maintenir la population de lymphocytes obtenue pendant jusqu'à 15 jours.
NOTE: De par sa conception, ce protocole va activer et de développer spécifiquement sous-ensemble de cellules T CD4 + qui sont réactifs à SEB et TSST puis les conduire vers un phénotype Th1-like effecteur / mémoire. D'autres globules blancs ne parviennent pas à survivre et se développer dans ces conditions, de telle sorte que par l'étape 1.10 les cellules seront au moins 95% CD4 +, des cellules CD45RO + T 19, comme on peut facilement être évaluée par cytométrie en flux. Si l'on désire une purification supplémentaire peut aisément être réalisé par des kits de sélection anticorps disponibles dans le commerce / perle magnétique à base positifs ou négatifs.
2. À partir Primary Cell Culture endothéliales humaines
- Manteau un flacon T25 avec de la fibronectine (FN) 20 pg / ml dans du PBS dans des conditions stériles. Laisser à température ambiante pendant 30-60 min. Retirer le FN et ajoutez 5 ml de milieu complet (endothéliale Basal Medium (EBM-2) supplémenté avec de croissance endothélial moyenne (EGM-2) SingleQuots). Pré-incuber dans 37 ° C incubateur de culture cellulaire pendant au moins 30 min.
- Décongeler une fiole de poumon ou dermique microvasculaires des cellules endothéliales humaines glacés (HLMVECs ou HDMVECs) dans un bain d'eau à 37 ° avec une légère agitation occasionnelle pour ~ 2-3 min. Transférer immédiatement cellules flacon T25 contenant les médias préchauffées. Tourbillonner et le lieu dans un incubateur à 37 ° C en douceur.
- Changer les médias après ~ 4-6 hr. Continuer à changer environ tous les médias 48 h (ou lorsque les médias devient légèrement jaune) jusqu'à ce que la plaque atteint ~ 90-95% de confluence.
3. Fractionnement général et l'expansion des cellules endothéliales
- Cultiver les cellules à une confluence de 90 à 95 ~. Cela peut prendre 2-5 jours. Pour le fractionnement,retirer les supports et rincer avec du PBS. Retirer du PBS et le remplacer par volume minimum de 1x trypsine frais (0,5 ml pour T25 ou T75 pour 1,5 ml). Remuer doucement pour couvrir toute la surface avec de la trypsine. Incuber à 37 ° C pendant ~ 5 min. Surveiller le détachement des cellules de la plaque à l'aide d'un microscope à lumière de faible puissance.
- Lorsque la majorité des cellules apparaissent arrondie ou enlevé, ajouter 5 volume (à savoir, par rapport au volume de la trypsine ajoutée) de milieu complet préchauffé EGM-2 et pipeter doucement sur la surface du flacon pour détacher toutes les cellules.
- Compter les cellules endothéliales avec un hémocytomètre comme décrit dans 1,6-1,7. Pellet les cellules par centrifugation (5 min, 1200 xg). Retirer le surnageant. Ajuster la concentration à 0,5 millions de cellules par ml par addition d'EGM-2 MV milieu complet préchauffé.
- Transférer des aliquotes de cellules pour les plats ou flacons FN revêtues appropriées pour l'entretien. Remuer doucement et placer dans l'incubateur. Changer les médias au sein de 6-12 h de placage. Médias shoULD être changé environ toutes les 48 heures par la suite.
4. La transfection des cellules endothéliales
NOTE: les cellules endothéliales primaires sont réfractaires à la transfection par la plupart des méthodes chimiques et l'électroporation commun. La méthode basée sur la transfection nucléaire décrite ci-dessous permet de l'efficacité de transfection relativement élevé (~ 50-70%). Une autre méthode efficace est l'utilisation de l'infection par des vecteurs viraux appropriés (voir commentaires dans le tableau des matériaux).
- Préparer des flacons T75 T25 ou (si nécessaire) de HLMVECs ou HDMVECs à une densité finale de 90 à 95% confluency.Coat avec de la fibronectine (FN) 20 pg / ml dans du PBS dans des conditions stériles ni microscope des plaques de culture telles que des plaques Delta-T (pour étape 5) ou 12 mm lamelles de verre circulaires placées à l'intérieur d'un puits d'une plaque de culture cellulaire à 24 puits (pour l'étape 6) avec comme décrit ci-dessus (2,1).
- Ajouter 1 ml de complète EGM-2 milieux de culture à la culture de microscope platesor 0,5 ml à chaque 24 puits et équilibrer en plaquesune humidifié 37 ° C / 5% de CO 2 incubateur.
- Récolte et compter les cellules endothéliales comme dans les étapes 3.1-3.3.Centrifuge le volume requis de cellules (0,5 millions de cellules par échantillon) à 1.200 g pendant 5 min à température ambiante. Remettre en suspension le culot cellulaire dans soigneusement pl RT solution de transfection de 100 nucléaire par exemple.
- Moissonneuse 100 pi de suspension de cellules avec 1 à 5 ug d'ADN. Transfert cellules / ADN suspension dans cuvette certifié; échantillon doit couvrir le fond de la cuvette sans bulles d'air.
REMARQUE: Les constructions de ciblage YFP ou la DsRed de la membrane cellulaire (par l'intermédiaire des N-terminaux de 20 acides aminés de neuromodulin qui contient un signal de palmitoylation post-traductionnelle) ont été utilisés seuls (membrane-YFP seul ou membrane-DsRed seul) ou co-transfectées avec un volumétrique marqueur cytoplasmique (par exemple, la membrane-YFP et DsRed soluble). De nombreuses permutations de marqueurs protéiques fluorescents peuvent être utilisés. - Fermez la cuvette avec le couvercle. Insérez la cuvette aveccellules / ADN suspension dans le support de cuvette de l'électroporateur et d'appliquer le programme d'électroporation S-005. Prenez la cuvette sur le support une fois que le programme est terminé.
- Ajouter ~ 500 pi de milieux de culture pré-équilibrée à la cuvette et retirez délicatement la suspension de cellules à partir de la cuvette en utilisant les pipettes de transfert en plastique fournis dans le kit de transfection nucléaire.
- Pour les expériences utilisant des plaques de culture de microscope partitionner la suspension de cellules d'une réaction tout aussi entre deux plats contenant des milieux préchauffé (étapes 4.2-4.3). Pour les expériences utilisant 24 puits / plaque, la partition d'une réaction à parts égales entre 3 puits.
- Incuber les cellules dans un humidifié 37 ° C / 5% de CO 2 incubateur et changer de support 4-6 h, et de nouveau à 12-16 heures post-transfection.
5. direct imagerie cellulaire et de l'analyse
- Préparation de l'endothélium
- Jour 0: Co-transfecter HLMVECs primaires avec membrane-YFP et DsRed soluble via un nucleofection technologie telle que décrite dans l'étape 4 et de la plaque sur cellules vivantes des plaques de culture de formation d'image.
- Jour 1: Remplacer milieu par du milieu frais contenant de l'IFN-γ (100 ng / ml) pour induire l'expression du CMH-II. Le jour 2. Stimuler les cellules transfectées par addition de 20 ng ml de TNF-α / aux médias existants.
- Le Jour 3, incuber l'endothélium avec 1 pg / ml de chacune des superantigènes bactériens entérotoxine staphylococcique B (SEB) et la toxine de syndrome de choc toxique 1 (TSST) à 37 ° C pendant 30 à 60 min juste avant les expériences. Omettre cette étape pour les conditions de contrôle '-Ag'.
- Préparation de Lymphocytes
- En parallèle avec l'étape 5.1.3, préparer le tampon A (rouge de phénol-HBSS libre) supplémenté avec 20 mM de Hepes, pH 7,4 et 0,5% v / v de sérum albumine humaine pré-chauffé à 37 ° C. Prélever un échantillon des cultures de lymphocytes et de déterminer la densité en comptant avec un hémocytomètre (étape 1.12 à 1.17).
- Centrifugeuse 2 millions de cellules par échantillon à 1,200 g pendant 5 min à température ambiante dans un tube conique de 15 ml. Aspirer et multimédia culot cellulaire doucement remettre en suspension dans 2 ml de tampon A de telle sorte qu'aucun amas de cellules demeurent.
- Retirer une nouvelle aliquote de Fura-2 colorant calcium et remettre en suspension dans du DMSO pour obtenir une concentration de l'action de 1 mM.
- Ajouter 2 pi de Fura-2 solution stock à la suspension de cellules T (2 uM concentration finale), plafonner le tube et mélanger immédiatement en inversant le tube pour assurer une dispersion de colorant. Incuber à 37 ° C pendant 30 min.
- Centrifuger comme à l'étape 5.2.2. Aspirer Buffer-A et les lymphocytes doucement mais complètement remettre en suspension dans 20-40 pi de tampon frais-A.
- Configuration de l'imagerie des cellules vivantes et acquisition
REMARQUE: Une grande variété de systèmes peut être utilisé pour l'imagerie de fluorescence en direct-cellulaire sur microscopes optiques droits et inversés. Les exigences de base comprennent une source de lumière de fluorescence et les filtres, d'une caméra CCD, le changement de filtre motorisée et volets, une platine chauffante (ou Microscchambre chauffée ope montage) et logiciel d'acquisition d'image automatisé. Pour ce protocole d'ouverture numérique élevée, fort grossissement (ie, 40X, 63X) lentilles d'immersion d'huile sont nécessaires pour atteindre la résolution spatiale nécessaire. Un soin particulier doit être apporté au choix de la source et de lentilles de fluorescence appropriée pour l'imagerie à base de calcium-Fura-2 ne sont pas tous compatibles avec les longueurs d'onde nécessaires 340/380 nm d'excitation. Une approche alternative (compatible avec les groupes verts et rouges standards filtres de fluorescence) peut être utilisé avec des colorants non-ratiomentic sensibles au calcium (par exemple, Fluo-4, Rhod-3), si ceux-ci ne peuvent pas quantifier avec précision le flux de calcium et seulement fournir un rapport / lecture quantitative.- Tournez sur le système de microscope (PC pour l'opération, microscope, caméra CCD, roue de filtre et la lampe au xénon).
- Ouvrez le logiciel désigné.
- Mettre en place microscope / logiciel multicanaux automatisé imagerie time-lapse. Inclure acquisition séquentielle de l'annonceifferential contraste d'interférence (DIC), la fluorescence verte standard, une fluorescence rouge standard et norme excitation 340 et 380 nm Fura-2 images. Définissez l'intervalle pour l'acquisition de 10-30 secondes et une durée totale de ~ 20-60 min.
- Réglez le temps d'exposition pour Fura-2 imagerie.
- Ajouter l'huile fraîche objectif et monter un plat de microscope contenant seulement 0,5 ml de tampon-A sur l'adaptateur étape de chauffage et tournez immédiatement à équilibrer à 37 ° C (~ prendra 2-3 min).
- Ajouter lymphocytes au repos Fura2-chargés dans la chambre de plat de microscope en utilisant une pipette monté de 20 ul.
- Tournez sur l'imagerie du champ lumineux. Sélectionnez le chemin de lumière pour les oculaires. Utilisez le bouton de mise au point grossière d'apporter objectif en contact avec fond du plat de miscoscope. Utilisez l'oculaire et le bouton de réglage de précision de se concentrer sur les cellules T installés au fond du plat.
- Utilisez les commandes de platine XY pour sélectionner un champ contenant au moins 10 cellules. Évitersurpeuplée champs et amas cellulaires que ceux-ci créent des artefacts d'imagerie.
- Passer du champ lumineux à Fluorescent source de lumière. Passer d'imagerie oculaire à la caméra CCD. Paramètres d'acquisition fixés (par exemple, le temps d'exposition, gain de détecteur et binning). Utilisation de l'acquisition de logiciels reposant Fura2-340 et images Fura2-380 (à commencer par le temps d'exposition identique pour chacun, habituellement dans la gamme de ~ 200-1,000 msec).
- Utilisez les méthodes décrites à l'étape 5.4.1 pour calculer le Fura2-340 / Fura2-380 pour chaque lymphocyte. Effectuer itérations répétées de réglage de la Fura2-340 et des temps d'exposition Fura2-380, l'acquisition d'images et le calcul des ratios jusqu'à ce que les valeurs moyennes sont proches de 1.
- Réglez le temps d'exposition pour les mem-YFP et DsRed.
- Remplacer le plat de microscope utilisé dans l'étape 5.3.3.1 avec un plat de Miscroscope contenant transfectées, activé et SAg traitée (ou non traitée; contrôle) HLMVECs ou HDMVECs de l'incubateur de culture cellulaire (étapes 4.5.1). Utilisez une pipette de transfert jetable pour enlever rapidement les médias, rincer une fois par addition de ~ 1 ml de pré-chauffé Buffer-A. Aspirer et puis ajouter 0,5 ml de tampon-A.
- Identifier les domaines dans lesquels les cellules endothéliales transfectantes positifs brillamment fluorescentes sont présents et semblent en bonne santé avec des jonctions intercellulaires bien formés.
- Réglez les paramètres d'acquisition (par exemple, le temps d'exposition, gain de détecteur et binning) pour mem-YFP et DsRed. Assurez-vous que moyen pour l'intensité de signal de fluorescence dans chaque canal est comprise entre 25% et 75% de la plage dynamique du détecteur.
- Réglez le temps d'exposition pour Fura-2 imagerie.
- Mener expérience d'imagerie des cellules vivantes.
- Utilisez un logiciel automatisé de commencer l'acquisition d'images et de capturer plusieurs intervalles d'images pour établir la ligne de base.
- Lors de l'acquisition appliquer ~ 5 pi de lymphocytes Fura-2-chargés concentrées de l'étape (5.2) au centre du champ de l'imagerie de plat de microscope en insérant la pointe d'un petit volume (P-5ou P-20) pipette dans les médias à proximité du centre de l'objectif et d'éjection lentement.
- Comme les lymphocytes se déposent dans le domaine de l'imagerie, effectuer des réglages fins de la mise au point pour assurer que la cellule-endothélium interface de cellules T (synapses immunologiques) sont maintenues dans le plan focal. Avec 40 et objectif 63X, ~ 10-20 cellules par champ sont optimales. Si le nombre de cellules sont observées dans le domaine de l'imagerie répétez l'étape 5.3.4.2.
- Après l'intervalle d'observation souhaitée de l'expérience est en concurrence, continuer imagerie et pipeter immédiatement ionomycine directement dans le moule en miscoscope (en utilisant la technique comme dans 5.3.4.2) jusqu'à une concentration finale de 2 uM à induire maximale flux de calcium / Fura-2 le signal de signalisation (c.-à- , un moyen d'étalonnage, voir l'analyse 5.4.).
- Comme alternative à 5.3.4.4after l'intervalle d'observation souhaitée de l'expérience est en compétition, immédiatement aspirer le tampon A et le remplacer par 0,5 ml de solution de fixation (3,7% de formaldehyde dans du PBS) pendant 5 min àRT, suivie d'un rinçage trois fois avec du PBS. Puis passez à l'étape 6.
- Analyse de l'imagerie des cellules vivantes
REMARQUE: Après l'enregistrement de fichiers acquis, ils peuvent être analysés directement ou exportés pour analyse par une grande variété d'applications de logiciels d'analyse d'image hors ligne. ImageJ est un package particulièrement précieux, très polyvalent qui est librement disponible et compatible avec presque tous les logiciels d'acquisition. La conception des expériences d'imagerie décrites dans les étapes 5.1-5.3 donnera dynamique spatiale et temporelle haute résolution des interactions entre les lymphocytes et endothéliale APC en l'absence et la présence de apparenté SAG. Presque analyses illimitées sont possibles lorsque l'adressage de données d'imagerie de la dynamique cellulaires morphométrique / de signalisation. Les objectifs spécifiques décrits dans le présent protocole particulier sont de quantifier de manière coordonnée la migration des lymphocytes et de signalisation (à savoir, la cinétique et les niveaux de flux calcique intracellulaire) avec dynamiqueal changements dans l'architecture de la synapse immunologique en mettant l'accent sur des caractéristiques spécifiques (c.-à PAI / podo-prints). Les exemples suivants sont de norme distincte et non-standard (ie, développés spécifiquement pour ces expériences / questions) analyses.- Mesurer lymphocytes calcium flux
- Sélectionnez les Fura2-340 initiale (340 EX-510 nm de EM nm) et Fura2-380 (380 nm-510 nm EX EM) des images qui ont été acquises avant l'addition de lymphocytes. Utilisez-les comme des images de fond et numériquement les soustraire de l'ensemble des images de la série de temps pour chaque canal respectif.
- Pour chaque point de temps de créer une image numérique de rapport de la Fura2-340 de fond soustrait et les images Fura2-380 fond soustrait (soit de 340 nm-510 nm EX EM-fond / 380 nm-510 nm EX EM-fond).
- Sélectionnez l'outil de dessin de logiciel approprié. Cliquez sur l'écran pour dessiner une région circulaire de iNTÉRÊT (ROI) autour de chaque lymphocyte. Ceux-ci vont générer une valeur de pixel moyenne de rapport pour chaque trame de temps et des lymphocytes.
- Tracer le taux calculé en fonction du temps en utilisant un logiciel approprié (par exemple, tableur). Calculer la moyenne de flux de calcium pour l'expérience en additionnant les valeurs des rapports de tous les lymphocytes par champ et en divisant cette valeur par le nombre total de lymphocytes pour chaque point de temps
- Mener lympocyte analyse de suivi de la migration.
- Analyser la migration des cellules T de cellules en utilisant une application appropriée de suivi de cellules de logiciel (par exemple, ImageJ) en utilisant le canal de DIC chaque vidéo. Utiliser ImageJ Cellule de suivi Plugin, d'identifier dans chaque trame du centre de gravité de chaque cellule manuellement en cliquant sur elle dans chaque trame progressive.
- Utilisez les coordonnées xy série résultant (ie, des traces de voies de migration) pour calculer la vitesse moyenne (distance totale migré intervalle / total of imagerie) et la tortuosité (distance totale migrés / la distance linéaire de bout en bout entre l'emplacement de cellule dans la première et la dernière image).
- La meilleure corrélation entre ces paramètres avec le flux de calcium (5.4.1) sur une base dynamique, cellule par cellule.
- Évaluer la densité Podo-print / ILP au sein du SI
- Compter le nombre total de ILP formé dans chaque cellule T intervalles est définie (par exemple, plus de poste à 5 min de cellules T). Ceux-ci peuvent être identifiés échelle du micron discrète fluorescentes anneaux membrane-YFP que co-localisent avec des cernes dans cytoplasmique DsRed sur le APC endothéliale sur un site de l'adhésion des lymphocytes.
- Croix-corréler les indices résultant 'ILP »avec le flux de calcium (5.4.1) dynamique sur une base cellule par cellule.
- Mesurer la podo-print / ILP vies.
- Pour chaque podo-print / ILP dans une donnée est de calculer ses vies comme le dernier point de temps où un PAI était visible - le point de moment où ce podo-print / ILP fipremier est apparu. Moyenne ILP Temps de vie à la fois par EST.
- Croix-corréler ces vies avec le flux de calcium (5.4.1) dynamique sur une base cellule par cellule.
- Évaluer la relation temporelle entre la formation initiale et ILP flux de calcium.
- Pour chaque lymphocyte identifier le point de temps (cadre) à laquelle le calcium se lève le premier dessus de la base 19.
- Pour chaque lymphocyte identifier le point de temps au cours de laquelle le premier podo-print / ILP apparaît.
- Pour chaque lymphocyte calculer un «décalage temporel» en soustrayant le point de temps de flux de calcium initiale de celle de la première podo-print / ILP. La moyenne des valeurs pour toutes les lymphocytes dans un champ. Les valeurs positives indiquent la formation ILP précède le flux de calcium alors que les nombres négatifs indiquent le contraire.
NOTE: Ces expériences peut être facilement effectuée sous flux laminaire de cisaillement physiologiquement pertinente aide disponibles dans le commerce parallèle murales chambres d'imagerie de flux comme décrit 19.
- Mesurer lymphocytes calcium flux
Imagerie et d'analyse 6. cellule fixe
- Critère fixe des cellules T-endothélial est la formation et la coloration
- Préparation des cellules T, comme décrit au point 1. Préparer les cellules endotheliales (- / + SAG) comme dans l'étape 5.1 (transfection, l'étape 5.1.1 est facultative). Prélever un échantillon des cultures de lymphocytes et de déterminer la densité en comptant avec un hémocytomètre (Section 1).
- Transférer un volume culture de lymphocytes équivalente à au moins 3 x 10 5 lymphocytes / échantillon dans un tube conique de 15 ml et centrifuger à 200 g pendant 3 min. Retirer le surnageant et remettre le culot de lymphocytes dans le tampon-A (étape 5.2.1) concentration de 6 x 10 5 lymphocytes / ml.
- Retirez les cellules endothéliales de l'incubateur de culture cellulaire et (poursuivre un échantillon à la fois) des médias successivement aspirer et de le remplacer avec 0,5 ml de suspension de lymphocytes (3 x 10 5 lymphocytes / échantillon) et de le remplacer à 37 ° C incubator.
- Après des temps d'incubation appropriées, mentionné ci-dessus, moyen d'aspiration des puits et ajouter suffisamment de solution de fixateur (3,7% de formaldéhyde dans PBS) pour couvrir complètement l'échantillon (par exemple, en plaque à 24 puits ~ 300-500 pi). Incuber à température ambiante pendant 5-10 min.
- Rincer échantillon 3 fois avec du PBS. Colorer en ajoutant anticorps primaire pendant 60 min à température ambiante (voir la liste). Si l'anticorps primaire est dirigée vers une protéine intracellulaire, une étape supplémentaire de perméabilisation doit être effectuée par addition d'une solution de perméabilisation suffisante (0,01% de Triton X-100 dans du PBS) pour recouvrir complètement l'échantillon pendant 1 minute à température ambiante. Rincer échantillon 3 fois avec du PBS. Ajouter Anticorps secondaires appropriées (dans certains cas, de façon concomitante phalloïdine fluorescente) pendant 60 min à température ambiante.
- Monter la lamelle de verre circulaire sur une lame d'imagerie. Critère fixe des cellules T-endothélial est la formation et la coloration. Pour commencer imagerie lieu la diapositive d'imagerie contenant des échantillons sur la platine du microscope et sélectionnez l'huile 63Xobjectif. Appliquer de l'huile d'immersion fraîche.
- Utilisez le mode d'imagerie champ lumineux et lentilles oculaires pour localiser le plan focal. Passer à épifluorescence et inspecter échantillon via les lentilles oculaires. Sélectionnez un domaine d'intérêt. Passez en mode balayage laser.
- En utilisant le mode balayage rapide et de travail dans le domaine le plus large possible d'ajuster individuellement la puissance laser et le gain de chaque canal afin d'optimiser le signal de telle sorte que, idéalement, l'intensité du signal spécifique atteint au moins environ 25% et pas plus de 75% de la plage dynamique des détecteurs.
- Utilisation de commandes manuelles de discussion, de numériser rapidement à travers l'axe Z et d'identifier les limites supérieures et inférieures de sectionnement (généralement toutes les informations doivent être contenues dans une épaisseur de ~ 15 um). Sélectionnez l'épaisseur de la section de l'axe Z dans la gamme de ~ 0,2-1,0 um.
- Enfin, zoom / recadrer le domaine de l'imagerie de la région d'intérêt spécifique et de la conduite de balayage. En plus d'avoir très lumineux et spécifique fluorescence SIgnal dans l'échantillon, l'acquisition à haute résolution imagerie 3D nécessite itérations de numérisation et de faire des ajustements aux paramètres d'acquisition. L'objectif est d'obtenir xy maximale et la résolution de l'axe z, sans appréciable photo-blanchiment.
- Analyser qualitativement la topologie de la topologie 3D de l'est de mener la reconstruction numérique 3D des sections optiques résultant grâce à un logiciel approprié (par exemple, Image J). Grâce à des applications similaires évaluer quantitativement la distribution de signal de fluorescence (par exemple, la co-localisation et de l'intensité de fluorescence corrélative de l'analyse de la ligne de balayage de Pearson et la visualisation orthogonale).
Résultats
Une nouvelle approche de formation d'image en utilisant des cellules endotheliales et en combinant les avantages de résolution du plan modèle de bicouches lipidiques avec la complexité physiologique et la déformabilité des APC professionnelle a été développé (figure 1). La figure 2 donne des exemples de migration typique, le flux de calcium et de la dynamique topologiques observé avec ce approche. En l'absence d'aff...
Discussion
Dans l'ensemble, ce protocole décrit des méthodes pour étudier les cellules endothéliales que i) APC physiologiques peu étudiés et ii) comme un type de «modèle plane APC cellulaire» de roman. En ce qui concerne le premier, il est devenu de plus en plus apprécié que les APC périphérique non-hématopoïétiques (ou «stromales ') jouent des rôles critiques, non redondants (ie, par rapport à hématopoïétique APC) dans l'élaboration de réponses immunitaires adaptatives 16-18.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
We thank Dr. Peter T. Sage for his assistance in generating some of the representative images. This work was supported by an NIH R01 grant to C.V.C. (HL104006).
matériels
| Name | Company | Catalog Number | Comments |
| BD Vacutainer stretch latex free tourniquet | BD Biosciences | 367203 | |
| BD alcohol swabs | BD Biosciences | 326895 | |
| BD Vacutainer Safety-Lok | BD Biosciences | 367861 | K2 EDTA |
| BD Vacutainer Push Button Blood Collection Set | BD Biosciences | 367335 | |
| RPMI-1640 | Sigma-Aldrich | R8758-1L | |
| Ficoll-Paque | Sigma-Aldrich | GE17-1440-02 | Bring to RT before use |
| FCS-Optima | Atlanta Biologics | s12450 | Heat inactivated |
| Penicillin-Streptomycin | Sigma-Aldrich | P4458-100ML | |
| Trypan blue | Sigma-Aldrich | T8154-20ML | |
| staphylococcal enterotoxin B | Toxin Technology | BT202RED | Stock solution 1mg/ml in PBS |
| toxic shock syndrome toxin 1 | Toxin Technology | TT606RED | Stock solution 1mg/ml in PBS |
| human IL-15 | R&D Systems | 247-IL-025 | Stock solution 50ug/ml in PBS |
| PBS | Life Technologies | 10010-049 | |
| Fibronectin | Life Technologies | 33016-015 | Stock solution 1mg/ml in H20 |
| HMVEC-d Ad-Dermal MV Endo Cells | Lonza | CC-2543 | Other Human Microvascular ECs can be used, i.e. HLMVECs |
| EGM-2 MV bullet kit | Lonza | CC-3202 | |
| Trypsin-EDTA | Sigma-Aldrich | T-4174 | Stock solution 10x, dilute in PBS |
| amaxa-HMVEC-L Nucleofector Kit | Lonza | vpb1003 | Required Kit for step 4 |
| IFN-g | Sigma-Aldrich | I3265 | Stock solution 1mg/ml in H20 |
| TNF-alpha 10ug, human | Life Technologies | PHC3015 | Stock solution 1mg/ml in H20 |
| phenol Red-free HBSS | Life Technologies | 14175-103 | |
| Hepes | Fisher Scientific | BP299-100 | |
| Calcium Chloride | Sigma-Aldrich | C1016-100G | Stock solution 1M in H20 |
| Magnesium chloride | Sigma-Aldrich | 208337 | Stock solution 1M in H20 |
| Human Serum albumin | Sigma-Aldrich | A6909-10ml | |
| Immersol 518 F fluorescence free Immersion oil | Fisher Scientific | 12-624-66A | |
| Fura-2 AM 20x50ug | Life Technologies | F1221 | Stock solution 1mM in DMSO |
| pEYFP-Mem (Mem-YFP) | Clontech | 6917-1 | |
| pDsRed-Monomer (Soluble Cytoplasmic DsRed) | Clontech | 632466 | |
| pDsRed-Monomer Membrane (Mem-DsRed) | Clontech | 632512 | |
| pEGFP-Actin | Clontech | 6116-1 | |
| Alexa Fluor 488 Phalloidin | Life Technologies | A12379 | |
| Formaldehyde solution 37% | Fisher Scientific | BP531-500 | Toxic, use fumehood |
| Triton X-100 | Sigma-Aldrich | X100-5ML | |
| Falcon 15mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-70C | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-49A | |
| Falcon Tissue Culture Treated Flasks T25 | Fisher Scientific | 10-126-9 | |
| Falcon Tissue Culture Treated Flasks T75 | Fisher Scientific | 13-680-65 | |
| Corning Cell Culture Treated T175 | Fisher Scientific | 10-126-61 | |
| Glass coverslips | Fisher Scientific | 12-545-85 | 12 mm diameter |
| Falcon Tissue Culture Plates 24-well | Fisher Scientific | 08-772-1 | |
| Delta-T plates | Bioptechs | 04200415B | |
| Wheaton Disposable Pasteur Pipets | Fisher Scientific | 13-678-8D | |
| 1.5 ml Eppendorf tube | Fisher Scientific | 05-402-25 | |
| ICAM1 mouse anti-human | BD Biosciences | 555509 | |
| HS1 mouse anti-human | BD Biosciences | 610541 | |
| Anti-Human CD11a (LFA-1alpha) Purified | ebioscience | BMS102 | |
| Anti-Human CD3 Alexa Fluor® 488 | ebioscience | 53-0037-41 | |
| Anti-MHC Class II antibody | Abcam | ab55152 | |
| Anti-Talin 1 antibody | Abcam | ab71333 | |
| Anti-PKC theta antibody | Abcam | ab109481 | |
| phosphotyrosine (4G10 Platinum) | Millipore | 50-171-463 | |
| Nucleofector II | Amaxa Biosystems | Required electroporator for step 4 | |
| Zeiss Axiovert | Carl Zeiss MicroImaging | ||
| Zeiss LSM510 | Carl Zeiss MicroImaging | ||
| Zeiss Axiovison Software | Carl Zeiss MicroImaging | ||
| NU-425 (Series 60) Biological Safety Cabinet | NuAIRE | Nu-425-600 | |
| Forma STRCYCLE 37 °C, 5% CO2 Cell culture Incubator | Fisher Scientific | 202370 | |
| Centrifuge 5810 | Eppendorf | EW-02570-02 | |
| Hemocytometer | Sigma-Aldrich | Z359629 | Bright-Line Hemocytometer |
| Isotemp Waterbath model 202 | Fisher Scientific | 15-462-2 |
Références
- von Andrian, U. H., Mackay, C. R. T-cell function and migration. Two sides of the same coin. N. Engl. J. Med. 343 (14), 1020-1034 (2000).
- Springer, T. A. Adhesion receptors of the immune system. Nature. 346 (6283), 425-434 (1990).
- Shaw, A. S., Dustin, M. L. Making the T cell receptor go the distance: a topological view of T cell activation. Immunity. 6 (4), 361-369 (1997).
- Monks, C. R., Freiberg, B. A., Kupfer, H., Sciaky, N., Kupfer, A. Three-dimensional segregation of supramolecular activation clusters in T cells. Nature. 395 (6697), 82-86 (1998).
- Delon, J., Stoll, S., Germain, R. N. Imaging of T-cell interactions with antigen presenting cells in culture and in intact lymphoid tissue. Immunol Rev. 189, 51-63 (2002).
- Brossard, C., et al. Multifocal structure of the T cell - dendritic cell synapse. Eur J Immunol. 35 (6), 1741-1753 (2005).
- Dustin, M. L. The cellular context of T cell signaling. Immunity. 30 (4), 482-492 (2009).
- Balagopalan, L., Sherman, E., Barr, V. A., Samelson, L. E. Imaging techniques for assaying lymphocyte activation in action. Nat Rev Immunol. 11 (1), 21-33 (2011).
- Cebecauer, M., Spitaler, M., Serge, A., Magee, A. I. Signalling complexes and clusters: functional advantages and methodological hurdles. J Cell Sci. 123, 309-320 (2010).
- Oddos, S., et al. High-speed high-resolution imaging of intercellular immune synapses using optical tweezers. Biophys J. 95 (10), L66-L68 (2008).
- Grakoui, A., et al. The immunological synapse: a molecular machine controlling T cell activation. Science. 285 (5425), 221-227 (1999).
- Bunnell, S. C., et al. T cell receptor ligation induces the formation of dynamically regulated signaling assemblies. J Cell Biol. 158 (7), 1263-1275 (2002).
- Yokosuka, T., et al. Newly generated T cell receptor microclusters initiate and sustain T cell activation by recruitment of Zap70 and SLP-76. Nat Immunol. 6 (12), 1253-1262 (2005).
- Seminario, M. C., Bunnell, S. C. Signal initiation in T-cell receptor microclusters. Immunol Rev. 221, 90-106 (2008).
- Dustin, M. L. Supported bilayers at the vanguard of immune cell activation studies. J Struct Biol. 168 (1), 152-160 (2009).
- Martinelli, R., Carman, C. V., Bradshaw, R. A., Stahl, P. Lymphocyte Endothelilal Interactions. Encyclopedia of Cell Biology. , (2015).
- Choi, J., Enis, D. R., Koh, K. P., Shiao, S. L., Pober, J. S. T lymphocyte-endothelial cell interactions. Annu Rev Immunol. 22, 683-709 (2004).
- Marelli-Berg, F. M., Jarmin, S. J. Antigen presentation by the endothelium: a green light for antigen-specific T cell trafficking?. Immunol Lett. 93 (2-3), 109-113 (2004).
- Sage, P. T., et al. Antigen recognition is facilitated by invadosome-like protrusions formed by memory/effector T cells. J Immunol. 188 (8), 3686-3699 (2012).
- Kumari, S., et al. Actin foci facilitate activation of the phospholipase C-gama in primary T lymphocytes via the WASP pathway . eLife. , (2015).
- Marelli-Berg, F. M., et al. Major histocompatibility complex class II-expressing endothelial cells induce allospecific nonresponsiveness in naive T cells. J Exp Med. 183 (4), 1603-1612 (1996).
- Ma, W., Pober, J. S. Human endothelial cells effectively costimulate cytokine production by, but not differentiation of, naive CD4+ T cells. J Immunol. 161 (5), 2158-2167 (1998).
- Perez, V. L., Henault, L., Lichtman, A. H. Endothelial antigen presentation: stimulation of previously activated but not naive TCR-transgenic mouse T cells. Cell Immunol. 189 (1), 31-40 (1998).
- Epperson, D. E., Pober, J. S. Antigen-presenting function of human endothelial cells. Direct activation of resting CD8 T cells. J Immunol. 153 (12), 5402-5412 (1994).
- Shiao, S. L., et al. Human effector memory CD4+ T cells directly recognize allogeneic endothelial cells in vitro and in vivo. J Immunol. 179 (7), 4397-4404 (2007).
- Marelli-Berg, F. M., Okkenhaug, K., Mirenda, V. A two-signal model for T cell trafficking. Trends Immunol. 28 (6), 267-273 (2007).
- Carman, C. V., et al. Transcellular diapedesis is initiated by invasive podosomes. Immunity. 26 (6), 784-797 (2007).
- Carman, C. V. Mechanisms for transcellular diapedesis: probing and pathfinding by 'invadosome-like protrusions'. J Cell Sci. 122 ((Pt 17)), 3025-3035 (2009).
- Dustin, M. L., Tseng, S. Y., Varma, R., Campi, G. T. T cell-dendritic cell immunological synapses. Curr Opin Immunol. 18 (4), 512-516 (2006).
- Saito, T., Yokosuka, T. Immunological synapse and microclusters: the site for recognition and activation of T cells. Curr Opin Immunol. 18 (3), 305-313 (2006).
- Gomez, T. S., et al. HS1 functions as an essential actin-regulatory adaptor protein at the immune synapse. Immunity. 24 (6), 741-752 (2006).
- Burbach, B. J., Medeiros, R. B., Mueller, K. L., Shimizu, Y. T-cell receptor signaling to integrins. Immunol Rev. 218, 65-81 (2007).
- Vicente-Manzanares, M., Sanchez-Madrid, F. Role of the cytoskeleton during leukocyte responses. Nat Rev Immunol. 4 (2), 110-122 (2004).
- Ma, Z., Janmey, P. A., Finkel, T. H. The receptor deformation model of TCR triggering. Faseb J. 22 (4), 1002-1008 (2008).
- Ma, Z., Sharp, K. A., Janmey, P. A., Finkel, T. H. Surface-anchored monomeric agonist pMHCs alone trigger TCR with high sensitivity. PLoS Biol. 6 (2), e43 (2008).
- Groves, J. T. Bending mechanics and molecular organization in biological membranes. Annu Rev Phys Chem. 58, 697-717 (2007).
- Xu, C., et al. Regulation of T cell receptor activation by dynamic membrane binding of the CD3epsilon cytoplasmic tyrosine-based motif. Cell. 135 (4), 702-713 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon