Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aislamiento de células madre mesenquimales del tejido de la pulpa y cocultivo con células de cáncer para estudiar sus interacciones
En este artículo
Resumen
Ofrecemos protocolos para la evaluación de las células madre mesenquimales aisladas de la pulpa dental e interacciones de la célula de cáncer de próstata basadas en métodos directos e indirectos de la cultura Co. Condición media y trans-bien membranas son adecuadas analizar actividad paracrina indirecta. Siembra diferencialmente teñidas células juntos es un modelo apropiado para la interacción directa de la célula.
Resumen
Cáncer como un proceso de varios pasos y enfermedad complicada no sólo reglamentado por el crecimiento y proliferación de células individuales sino también controlado por las interacciones de medio ambiente y la célula de tumor. Identificación del cáncer y de las interacciones de la célula de vástago, incluyendo cambios en el ambiente extracelular, interacciones físicas y factores secretados, podría permitir el descubrimiento de nuevas opciones de tratamiento. Combinamos técnicas de cocultivo conocido para crear un sistema modelo para el cáncer de las células madre mesenquimales (MSCs) y las interacciones de la célula. En el estudio actual, las células madre de pulpa dental (DPSCs) y las interacciones de la célula de cáncer de próstata PC-3 fueron examinadas por técnicas de cocultivo directos e indirectos. Medio (CM) de la condición de DPSCs y 0,4 μm tamaño de poro bien trans membranas fueron utilizadas para estudiar la actividad paracrina. Co-cultivo de diversos tipos celulares juntos fue realizado para estudio de la interacción directa de la célula. Los resultados revelaron que CM aumenta la proliferación celular y disminuye la apoptosis en cultivos de células de cáncer de próstata. CM y sistema de trans-así mayor capacidad de migración de la célula de células PC-3. Células teñidas con tintes de membrana diferentes fueron sembradas en los mismos recipientes de cultivo, y DPSCs participó en una estructura autoorganizada con células PC-3 bajo esta condición de la cultura de cooperación directa. En general, los resultados indicaron que técnicas de cocultivo podrían ser útiles para el cáncer y las interacciones de MSC como sistema modelo.
Introducción
Las células madre mesenquimales (MSCs), con la capacidad de diferenciación y contribución a la regeneración de los tejidos mesenquimales como hueso, cartílago, músculo, ligamento, tendón y adiposo, se han aislado de casi todos los tejidos en el cuerpo adulto1 , 2. aparte proveer homeostasis del tejido produciendo células residentes en caso de inflamación crónica o una lesión, que producen vital citoquinas y factores de crecimiento para orquestar la angiogénesis, sistema inmune y tejido remodelación3. La interacción de MSCs con tejido de cáncer no es bien entendida, pero acumulando evidencias de que MSCs pueden promover la iniciación, progresión y metástasis de tumor4.
La capacidad de recalada de MSCs a la zona lesionada o crónicamente inflamada hace un candidato valioso para terapias basadas en células madre. Sin embargo, los tejidos de cáncer, "nunca curar heridas", también liberación de citoquinas inflamatorias, moléculas de pro-angiogénica y vitales factores de crecimiento, que atraen MSCs cancerígenas zona5. Aunque hay limitada informes que demuestran los efectos inhibitorios de MSCs en cáncer crecimiento6,7la progresión del cáncer y sus metástasis promover efectos han sido ampliamente registrados8. MSCs directa o indirectamente afectan la carcinogénesis de diversas maneras, incluyendo la supresión de las células inmunes, secretando factores de crecimiento/citoquinas que apoyan la proliferación de células de cáncer y la migración, mejorar la actividad angiogénica y regulación transición epitelial-mesenquimal (EMT)9,10. Entorno del tumor consiste en varios tipos celulares, incluyendo fibroblastos asociada al cáncer (CAFs) y miofibroblastos, células endoteliales, adipocitos y células inmunes11. De los cafés son el tipo celular más abundante en la zona de tumor que secretan diferentes quimiocinas promover el crecimiento y metástasis de cáncer8. Se ha demostrado que MSCs de médula ósea pueden diferenciarse en CAF en el estroma tumoral del12.
Células madre de pulpa dental (DPSCs), caracterizadas como el primer MSCs de derivados de tejido dentales por Gronthos et al. 13 en el año 2000 y luego ampliamente investigado por otros14,15, expresar marcadores de pluripotencia tales como Oct4, Sox2y Nanog16 y puede diferenciarse en varios linajes de célula17. Análisis de expresión génica y proteínas demostró que DPSCs producen niveles comparables de factores de crecimiento/citoquinas con otros MSCs como factor de crecimiento endotelial vascular (VEGF), angiogenina, factor de crecimiento fibroblástico 2 (FGF2), interleucina-4 (IL-4), IL-6, IL-10, y factor de células madre (SCF), así como ligando de fms-like tyrosine kinase-3 (Flt - 3L) que puede promover la angiogénesis, modulan las células inmunes y apoyo cáncer célula proliferación y migración de18,19,20 . Mientras que las interacciones de MSCs con entorno de cáncer han sido bien documentadas en la literatura, la relación entre DPSCs y las células de cáncer no ha sido evaluada todavía. En el presente estudio, hemos establecido estrategias de tratamiento medio co-cultivo y condición para una línea celular de cáncer de próstata altamente metastático, PC-3 y DPSCs proponer acción potencial del mecanismo de MSCs dentales en la progresión del cáncer y la metástasis.
Protocolo
Se obtuvo consentimiento informado de los pacientes después de la aprobación del Comité de ética institucional.
1. cultivo y aislamiento de CEDIP
- Transferencia de dientes de la sabiduría de los adultos jóvenes de edades comprendidos entre 17 y 20 a 15 mL tubos que contengan completa de Dulbecco modificado Eagle Medium (DMEM) [bajo medios glucosa DMEM suplementados con 10% suero bovino fetal (FBS) y 1% de penicilina/estreptomicina / solución de anfotericina (PSA)], dentro de las 8 h después de la resección. Mantenga el material tejido frío (4 ° C) durante la transferencia para evitar la potencial muerte de la célula.
- Eliminar cuidadosamente el tejido de la pulpa por pinzas estériles desde el centro del diente, colocar el tejido de la pulpa en el frío medio DMEM completo en platos de cultivo de tejidos de 10 cm y les pique en trozos pequeños (2-3 mm) por bisturí.
Nota: Todos los procedimientos experimentales deben llevarse a cabo en condiciones estériles en campana de flujo laminar. Esta técnica no enzimática ha sido previamente usado21,22,23. - Lugar pequeño pulpa tejidos dentro de la cultura del tejido trataron placas de 6 pocillos y agregar 200 μL de medios DMEM completo para cubrir cada piezas de tejido de la pulpa pequeña.
- Incube los pocillos de cultivo de tejidos a 37 ° C en una atmósfera de aire humidificado (80% de humedad) con 5% CO2 por 2 h para proporcionar fijación del tejido.
Nota: Este paso podría prolongarse a 3-4 h controlando la evaporación de los medios de comunicación. - Añadir el volumen adecuado (2-2.5 mL) de medio DMEM completo a los pozos e incubar a 37 ° C en una atmósfera de aire humidificado con 5% CO2 para las células para la difusión de los tejidos.
Nota: Las células se hacen visibles después de aproximadamente 4 días y alcanzan confluencia después de 8-9 días. - Cuando las células alcanzan el 80% de confluencia, eliminar los medios de comunicación de las placas de 6 pozos, lavar con 2 mL de tampón fosfato salino (PBS) y añadir 2 mL de tripsina. Incúbelos durante 2 minutos en una incubadora a 37 ° C con ambiente de aire humidificado y 5% CO2. Luego agregar 2 mL de medio DMEM completo seguido por la incubación de 2 minutos para inhibir tripsina. Centrifugar las células a 300 x g durante 5 minutos para que sedimenten las células.
- Células de paso a los frascos en total medios DMEM y tienda para otros experimentos. Añadir 15 mL de medio DMEM completo a los frascos de T-75 y las células de transferencia de dos pozos de la placa de 6 pozos para frascos de T-75 una e incubar a 37 ° C con ambiente de aire humidificado y 5% CO2.
2. Caracterización de DPSCs
- Realizar análisis morfológicos.
- Las células de la semilla (criterio 1.6) en cultivo de tejidos recubiertos frascos (o placas de 6 pocillos) en medio DMEM completo para al menos 8 pasos observar la morfología de la célula.
- Visualizar las células por microscopio de luz y definir la morfología de fibroblasto-como de la célula. Las células deben fijar a los platos de cultivo y morfología de la célula del huso-como.
Nota: Alternativamente, las células pueden cultivarse como células individuales por hasta 14 días en placas de pocillos para observar la capacidad de formación de Colonia que es una característica específica de fibroblastos y MSCs.
- Realizar el análisis superficial del marcador.
- Trypsinize las células de paso 1.7. Quitar los medios de comunicación del frasco de cultivo de tejidos T-75, lave con 2 mL de PBS y añadir 2 mL de tripsina. Incúbelos durante 2 minutos en una incubadora a 37 ° C con ambiente de aire humidificado y 5% CO2. Luego agregar 2 mL de medio DMEM completo seguido por la incubación de 2 minutos para inhibir tripsina. Centrifugar las células a 300 x g durante 5 minutos para que sedimenten las células.
- Fijar las células con paraformaldehído al 4% por 20 min a temperatura ambiente en tubos de 1,5 mL y luego lavar con 500 μl de PBS 3 veces para quitar paraformaldehido.
- Incube las células fijas con los anticuerpos contra CD29, CD34, CD14, CD45, CD90, CD105, CD166 y CD73 por 1 h a 4 ° C en 100 μl de PBS.
Nota: La concentración de anticuerpo utilizado es de 0,5 μg/mL. CD34, CD14 y CD45 se utilizan como marcadores negativos, mientras que CD29, CD90, CD105, CD166 y CD73 se utilizan como marcadores de superficie celular positiva para MSCs. - Lavar las células 3 veces con PBS y respectivos anticuerpos secundarios como isotiocianato de fluoresceína (FITC), ficoeritrina (PE), etc. para etiquetado. Incubar las células con anticuerpos secundarios 1: 500 diluido en 100 μl de PBS por 30 min a 4 ° C y lavado 3 veces con PBS.
- Mantener las muestras en la oscuridad para el análisis de citometría de flujo y detección de positivos y negativos de tinción mediante citometría de flujo.
Nota: Utilice las células control sin mancha para organizar la dispersión hacia delante y lateral. Organizar positivamente que bloquean de teñido poblaciones excluyendo las células muertas, residuos y población no manchada. Utilice 100 μm boquilla con 45 psi de presión de vaina y recopilar 10.000 eventos para determinar positivas DPSCs arreglando los canales.
- Realizar la diferenciación de DPSCs.
- Semilla 1 × 104 células en placas de 24 pocillos en completan los medios DMEM e incuban durante 24 h a 37 ° C en una atmósfera de aire humidificado con 5% CO2.
- Formular la diferenciación medios usando compiten medio DMEM como medio base. Preparar medios osteogénicos mezclando 100 nM dexametasona, glicerofosfato-β de 10 mM y 0.2 mM el ácido ascórbico. Preparar los medios condrogénica mezclando 1 × insulina-transferrina-selenio (ITS-G), 100 nM dexametasona, 100 ng/mL transformación factor de crecimiento beta (TGF-β), 14 μg/mL de ácido ascórbico y 1 mg/mL albúmina sérica bovina (BSA). Preparar medios de adipogenic mezclando 100 dexametasona nM, 5 μg/mL insulina, 0.5m m 3-isobutil-1-metilxantina (IBMX) y 60 indometacina μM.
Nota: Los medios de diferenciación pueden conservarse a 4 ° C al menos una semana. - Cambiar crecimiento osteo, chondro o medios de diferenciación adipo-genic y actualizar dos veces a la semana durante dos semanas.
- Confirmar la diferenciación por von Kossa y Alcian coloración azul, actividad de la enzima (actividad de la fosfatasa alcalina), inmunocitoquímica y análisis (qPCR) la reacción en cadena de polimerasa cuantitativa según los protocolos describen previamente21.
- Realizar von Kossa y Azul alcián stainings en células que están fijadas con paraformaldehído al 4% a temperatura ambiente durante 10 min lavado las células fijas con PBS y se manchan con el kit de vonKossa según las recomendaciones del fabricante para observar depósitos de calcio .
- Preparar el Alcian blue tinción solución disolviendo 1,00 g de colorante de azul de Alcian en 100 mL de ácido acético al 3% (v/v) para la tinción más. Lavar las células fijas con PBS y mancha las células por 30 min con Azul alcián en solución. Visualizar las muestras teñidas por un microscopio de luz.
3. preparación de condición media (CM)
- Sustituir los medios de comunicación de las células de paso 1.7 con fresco completo DMEM 24 h antes de CM.
Nota: Paso 2-4 se recomienda. - Recoger media (CM) de la condición de cultivadas DPSCs cuando las células alcanzan el 80% de confluencia. Centrifugue los medios recogidos a 300 x g durante 5 min eliminar los restos de material y de la célula de tejido inútil.
Nota: Alternativamente, utilizar filtros de jeringa de 0,2 μm para eliminar desechos desde el medio de la condición. - Recoger sobrenadante y almacenar a-20 ° C para experimentos adicionales.
Nota: Guarde el sobrenadante a-80 ° C para almacenamiento a largo plazo.
4. tratamiento de las células cancerosas con CM
- Realizar análisis de viabilidad de la célula.
- Semilla PC-3 (células humanas de cáncer de próstata) en placas de 96 pozos a una densidad celular de 5 x 103 células/pozo en completan DMEM e incuban en cámara humidificada a 37 ° C y 5% de CO2 durante 24 h.
- Tratar las células con 10, 20, 30, 40 y 50% de los CM (v/v) mezcladas con DMEM completo durante 24 h.
- Medir la viabilidad celular mediante el uso de 3-(4,5-dimethyl-thiazol-2-yl)-5-(3-carboxymethoxy-phenyl)-2-(4-sulfo-phenyl)-2H-tetrazolium (MTS)-ensayo como se describe previamente24.
- Realizar de terminal deoxynucleotidyl transferase dUTP nick final etiquetado análisis (TUNEL).
- Células PC-3 de semilla en cultivo celular de pozo de 6 placas a una densidad celular de 2 × 105 células/pocillo e incuban en una cámara humidificada a 37 ° C y 5% CO2 durante la noche.
- Mezclar 20% CM (v/v) con completar medio DMEM y aplicar a las células durante 24 h.
- Trypsinize células y suspender en 50 μL de mezcla de reacción de TUNEL (solución + solución de enzima, suministrado con el kit de etiquetado), incubar a 37 ° C durante 60 min en un vapor y 5% CO2 .
Nota: Para tripsinización, eliminar medios de placas celulares 6-pozo de lavado con 1 mL de PBS y agregar 500 μl de la tripsina. Incúbelos durante 2 minutos en una incubadora a 37 ° C con ambiente de aire humidificado y 5% CO2. Añadir 1 mL de medio DMEM completo seguido por la incubación de 2 minutos para inhibir tripsina. Centrifugar las células a 300 x g durante 5 minutos para que sedimenten las células. - Lavar con PBS y analizar las células en PBS mediante citometría de flujo.
- Realizar análisis de qPCR.
- Células PC-3 de semilla en cultivo celular de pozo de 6 placas a una densidad celular de 2 × 105 células/pocillo e incuban en un incubador humedecido a 37 ° C y 5% CO2 durante la noche.
- Mezclar 20% CM (v/v) con completar medio DMEM y aplicar a las células durante 24 h.
- Trypsinize las células y recoger el sedimento celulares para aislamiento y cDNA síntesis de RNA.
Nota: Retire los medios de comunicación de placas celulares 6-pozo de lavado con 1 mL de PBS y agregar 500 μl de la tripsina. Incubar 2 minutos en una incubadora a 37 ° C con ambiente de aire humidificado y 5% CO2. Añadir 1 mL de medio DMEM completo seguido por la incubación de 2 minutos para inhibir tripsina. Centrifugar las células a 300 x g durante 5 minutos para que sedimenten las células. - Realizar experimentos de qPCR según el protocolo anteriormente descrito25.
- Realizar la migración de la célula de las células cancerosas.
- Semilla 1 × 105 PC-3 células en placas de 12 pocillos e incubar en una incubadora humidificada durante la noche a 37 ° C y 5% CO2.
- Rayar las células con una punta de 200 μL estériles y cambiar el medio inmediatamente con medio fresco que contiene diferentes concentraciones de CM [p. ej., 10, 20, 30, 40 y 50% de los CM (v/v) mezclado con DMEM completo].
- Observar rayas bajo un microscopio invertido y toma de fotografías a diferentes intervalos de tiempo (0 y 24 h).
- Medida el cierre scratch mediante software de Image J mediante la fórmula:
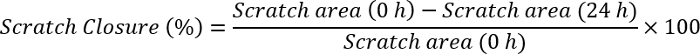
Nota: Abre la imagen cero con el software Image J. Dibujar una línea que tiene la misma magnitud que la barra de escala que ya existe en la imagen. Haga clic en analizar, establecer la escala y observar la distancia en píxeles como la ampliación de la línea dibujada. Escriba el tamaño de la barra de escala a la parte de la distancia conocida, arreglar unidad (píxeles, centímetros, etc.) y haga clic en Aceptar. Volver a la sección de análisis y haga clic en las mediciones. En primer lugar, esto le dará el tamaño de la barra de escala como la unidad seleccionada. Haga clic en un borde de scratch y arrastrar hasta el otro extremo de la raya. Tenga en cuenta el valor de cada punto de tiempo (0 h y 24 h). Llevar estos valores a la fórmula anterior y calcular el cierre scratch.
5. migración por contacto indirecto de las células cancerosas y DPSCs
- Semillas 3 × 104 DPSCs en placa de 24 pozos rellenos con 0,4 μm de poro e incubar en una incubadora humidificada durante la noche a 37 ° C.
- Las células PC-3 de semilla en 24 pocillos platos a una densidad celular de 5 × 104 e incubar en una incubadora humidificada durante la noche a 37 ° C y 5% CO2.
- Rayar las células PC-3 con una punta de 200 μL estériles, cambiar el medio con medio fresco y colocar insertos llevar DPSCs en las células PC-3.
- Observar las células bajo un microscopio invertido y toma de fotografías a diferentes intervalos de tiempo (0 y 24 h) para analizar la migración de la célula.
6. cooperación cultura ensayo y análisis de citometría de flujo
- Etiqueta las células PC-3 y DPSCs mediante PKH67 (verde) y PKH26 (rojo) célula fluorescente vinculador tintes, respectivamente26.
- Trypsinize las células PC-3 y DPSCs, respectivamente. Quitar los medios de comunicación del frasco de cultivo de tejidos T-75, lave con 2 mL de PBS y añadir 2 mL de tripsina. Incúbelos durante 2 minutos en una incubadora a 37 ° C con ambiente de aire humidificado y 5% CO2. Añadir 2 mL de medio DMEM completo seguido por la incubación de 2 minutos para inhibir tripsina.
- Centrifugar las células a 300 x g durante 5 min, descartar el sobrenadante y resuspender el pellet celular en la solución de tinte preparada en tampón diluyente C suministrada por el kit (véase Tabla de materiales).
- Incubar las células en la solución colorante durante 10 minutos y terminar la tinción reacción añadiendo 100 μl de FBS. Centrifugar las células a 300 x g durante 5 min, descartar el sobrenadante y lavar las células con medio de cultivo completo antes de co-cultivo.
- Etiquetado como las células de la placa (5 × 104/well) en placas de 6 pozos en proporción 1:1 (DPSCs: PC3). Mantener las células Co cultivadas en DMEM completo.
- Recoger las células después de períodos de incubación de 24 h o 48 h por centrifugación de las células a 300 x g durante 5 min y lavar con PBS.
- Resuspender las células en 300 μL de células activado por fluorescencia clasificar búfer (FACS) en 5 mL fondo flujo cytometry tubos redondos. Vortex para dispersar a los agregados de la célula justo antes de los análisis de la muestra.
- Use una boquilla de 100 μm con 45 psi de presión de vaina.
Nota: Extremadamente alto flujo puede disminuir la sensibilidad de detección de fluorescencia. - Utilizar células control sin manchas y células color para ajustar el avance apropiado y lado dispersión láser tensión tipos de la célula y la compensación como se mencionó anteriormente27.
Nota: Utilizar sincronización de células vivas para excluir restos celulares, células muertas o agregados. - Recopilar eventos de 10.000 (100.000 es preferible) para determinar el porcentajes positivo DPSCs verdes y células PC-3 rojo arreglando FL-1 (verde) y los canales de FL-2 (rojo).
Resultados
La figura 1 muestra las características generales de la maestría de DPSCs bajo condiciones de cultivo. DPSCs ejercer morfología de fibroblasto-como de la célula después de la galjanoplastia (figura 1B). Antígenos de superficie de MSC (CD29, CD73, CD90, CD105 y CD166) se expresan altamente mientras que los marcadores hematopoyéticos (CD34, CD45 y CD14) son negativos (figura 1). Cambios a nivel ...
Discusión
Contribución de MSCs al entorno del tumor está regulada por varias interacciones incluyendo híbrido celular generación vía celular fusiones, actividades entosis o citoquinas y quimioquinas entre células madre y células de cáncer28. Organización estructural, las interacciones célula-célula y secretadas factores determinan el comportamiento de la célula de cáncer en términos de promoción del tumor, la progresión y la metástasis al tejido circundante. Adecuada ex vivo
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este estudio fue apoyado por la Universidad de Yeditepe. Todos los datos y figuras utilizadas en este artículo fueron previamente publicados34.
Materiales
| Name | Company | Catalog Number | Comments |
| DMEM | Invitrogen | 11885084 | For cell culture |
| FBS | Invitrogen | 16000044 | For cell culture |
| PSA | Lonza | 17-745E | For cell culture |
| Trypsin | Invitrogen | 25200056 | For cell dissociation |
| PBS | Invitrogen | 10010023 | For washes |
| Dexamethasone | Sigma | D4902 | Component of differentiation media |
| β-Glycerophosphate | Sigma | G9422 | Component of osteogenic differentiation medium |
| Ascorbic acid | Sigma | A4544 | Component of osteo- and chondro-genic differentiation medium |
| Insulin-Transferrin-Selenium (ITS −G) | Invitrogen | 41400045 | Component of chondrogenic differentiation medium |
| TGF-β | Sigma | SRP3171 | Component of chondrogenic differentiation medium |
| Insulin | Sigma | I6634 | Component of adipogenic differentiation medium |
| Isobutyl-1-methylxanthine (IBMX) | Sigma | I7018 | Component of adipogenic differentiation medium |
| Indomethacin | Sigma | I7378 | Component of adipogenic differentiation medium |
| MTS Reagent | Promega | G3582 | Cell viability analyses |
| TUNEL Assay | Sigma | 11684795910 | Apoptotic analyses |
| 24-well plate inserts | Corning | 3396 | For trans-well migration assay |
| PKH67 | Sigma | PKH67GL | For co-culture cell staining |
| PKH26 | Sigma | PKH26GL | For co-culture cell staining |
| Paraformaldehyde | Sigma | P6148 | For cell fixation |
| von Kossa Kit | BioOptica | 04-170801.A | For cell staining (differentiation) |
| Alcian blue | Sigma | A2899 | For cell staining (differentiation) |
Referencias
- Camberlain, G., Fox, J., Ashton, B., Middleton, J. Mesenchymal stem cells: their phenotype, differentiation capacity, immunological features, and potential for homing. Stem Cells. 25 (11), 2739-2749 (2007).
- Demirci, S., Doğan, A., Şahin, F. . Dental Stem Cells. , 109-124 (2016).
- Fox, J. M., Chamberlain, G., Ashton, B. A., Middleton, J. Recent advances into the understanding of mesenchymal stem cell trafficking. British journal of haematology. 137 (6), 491-502 (2007).
- Chang, A. I., Schwertschkow, A. H., Nolta, J. A., Wu, J. Involvement of mesenchymal stem cells in cancer progression and metastases. Current cancer drug targets. 15 (2), 88-98 (2015).
- Dvorak, H. F. Tumors: wounds that do not heal. New England Journal of Medicine. 315 (26), 1650-1659 (1986).
- Lu, Y. -. r., et al. The growth inhibitory effect of mesenchymal stem cells on tumor cells in vitro and in vivo. Cancer biology & therapy. 7 (2), 245-251 (2008).
- Secchiero, P., et al. Human bone marrow mesenchymal stem cells display anti-cancer activity in SCID mice bearing disseminated non-Hodgkin's lymphoma xenografts. PloS one. 5 (6), e11140 (2010).
- Hong, I. -. S., Lee, H. -. Y., Kang, K. -. S. Mesenchymal stem cells and cancer: friends or enemies?. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 768, 98-106 (2014).
- Brennen, W. N., Chen, S., Denmeade, S. R., Isaacs, J. T. Quantification of Mesenchymal Stem Cells (MSCs) at sites of human prostate cancer. Oncotarget. 4 (1), 106 (2013).
- Klopp, A. H., Gupta, A., Spaeth, E., Andreeff, M., Marini, F. Concise review: dissecting a discrepancy in the literature: do mesenchymal stem cells support or suppress tumor growth. Stem cells. 29 (1), 11-19 (2011).
- Albini, A., Sporn, M. B. The tumour microenvironment as a target for chemoprevention. Nature Reviews Cancer. 7 (2), 139 (2007).
- Quante, M., et al. Bone marrow-derived myofibroblasts contribute to the mesenchymal stem cell niche and promote tumor growth. Cancer cell. 19 (2), 257-272 (2011).
- Gronthos, S., Mankani, M., Brahim, J., Robey, P. G., Shi, S. Postnatal human dental pulp stem cells (DPSCs) in vitro and in vivo. Proceedings of the National Academy of Sciences. 97 (25), 13625-13630 (2000).
- Mori, G., et al. Dental pulp stem cells: osteogenic differentiation and gene expression. Annals of the new York Academy of Sciences. 1237 (1), 47-52 (2011).
- Mori, G., et al. Osteogenic properties of human dental pulp stem cells. Journal of biological regulators and homeostatic agents. 24 (2), 167-175 (2010).
- Kerkis, I., et al. Isolation and characterization of a population of immature dental pulp stem cells expressing OCT-4 and other embryonic stem cell markers. Cells Tissues Organs. 184 (3-4), 105-116 (2006).
- Potdar, P. D., Jethmalani, Y. D. Human dental pulp stem cells: applications in future regenerative medicine. World journal of stem cells. 7 (5), 839 (2015).
- Ahmed, N. E. -. M. B., Murakami, M., Hirose, Y., Nakashima, M. Therapeutic potential of dental pulp stem cell secretome for Alzheimer's disease treatment: an in vitro study. Stem cells international. 2016, (2016).
- Gorin, C., et al. Priming dental pulp stem cells with fibroblast growth factor-2 increases angiogenesis of implanted tissue-engineered constructs through hepatocyte growth factor and vascular endothelial growth factor secretion. Stem cells translational medicine. 5 (3), 392-404 (2016).
- Wakayama, H., et al. Factors secreted from dental pulp stem cells show multifaceted benefits for treating acute lung injury in mice. Cytotherapy. 17 (8), 1119-1129 (2015).
- Doğan, A., et al. Differentiation of human stem cells is promoted by amphiphilic pluronic block copolymers. International Journal of Nanomedicine. 7, 4849 (2012).
- Taşlı, P. N., Doğan, A., Demirci, S., Şahin, F. Boron enhances odontogenic and osteogenic differentiation of human tooth germ stem cells (hTGSCs) in vitro. Biological trace element research. 153 (1-3), 419-427 (2013).
- Yalvac, M., et al. Isolation and characterization of stem cells derived from human third molar tooth germs of young adults: implications in neo-vascularization, osteo-, adipo-and neurogenesis. The pharmacogenomics journal. 10 (2), 105 (2010).
- Doğan, A., et al. Sodium pentaborate pentahydrate and pluronic containing hydrogel increases cutaneous wound healing in vitro and in vivo. Biological trace element research. 162 (1-3), 72-79 (2014).
- Doğan, A., Yalvaç, M. E., Yılmaz, A., Rizvanov, A., Şahin, F. Effect of F68 on cryopreservation of mesenchymal stem cells derived from human tooth germ. Applied biochemistry and biotechnology. 171 (7), 1819-1831 (2013).
- Rizvanov, A. A., et al. Interaction and self-organization of human mesenchymal stem cells and neuro-blastoma SH-SY5Y cells under co-culture conditions: A novel system for modeling cancer cell micro-environment. European Journal of Pharmaceutics and Biopharmaceutics. 76 (2), 253-259 (2010).
- Troiano, L., et al. Multiparametric analysis of cells with different mitochondrial membrane potential during apoptosis by polychromatic flow cytometry. Nature protocols. 2 (11), 2719 (2007).
- Melzer, C., von der Ohe, J., Lehnert, H., Ungefroren, H., Hass, R. Cancer stem cell niche models and contribution by mesenchymal stroma/stem cells. Molecular cancer. 16 (1), 28 (2017).
- Aguirre, A., Planell, J., Engel, E. Dynamics of bone marrow-derived endothelial progenitor cell/mesenchymal stem cell interaction in co-culture and its implications in angiogenesis. Biochemical and biophysical research communications. 400 (2), 284-291 (2010).
- Bogdanowicz, D. R., Lu, H. H. . Biomimetics and Stem Cells. , 29-36 (2013).
- Plotnikov, E., et al. Cell-to-cell cross-talk between mesenchymal stem cells and cardiomyocytes in co-culture. Journal of cellular and molecular. 12 (5a), 1622-1631 (2008).
- Bogdanowicz, D. R., Lu, H. H. Studying cell-cell communication in co-culture. Biotechnology journal. 8 (4), 395-396 (2013).
- Brunetti, G., et al. High expression of TRAIL by osteoblastic differentiated dental pulp stem cells affects myeloma cell viability. Oncology reports. 39 (4), 2031-2039 (2018).
- Doğan, A., Demirci, S., Apdik, H., Apdik, E. A., Şahin, F. Dental pulp stem cells (DPSCs) increase prostate cancer cell proliferation and migration under in vitro conditions. Tissue and Cell. 49 (6), 711-718 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados