È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento di cellule staminali mesenchimali da tessuto della polpa e co-coltura con cellule tumorali per studiare le loro interazioni
In questo articolo
Riepilogo
Forniamo i protocolli per la valutazione delle cellule staminali mesenchimali isolate da polpa dentale e interazioni delle cellule di carcinoma della prostata basati su metodi diretti e indiretti di co-coltura. Mezzo di condizione e trans-pozzo membrane sono adatte per analizzare attività paracrina indiretta. Seeding differenzialmente macchiato le cellule insieme è un modello appropriato per interazione diretto cellula-cellula.
Abstract
Il cancro come un processo multistep e malattia complicata è non solo regolato da crescita e proliferazione delle cellule individuali ma anche controllato da interazioni cellula-cellula e ambiente del tumore. Identificazione del cancro e cellule staminali interazioni, compresi i cambiamenti nell'ambiente extracellulare, interazioni fisiche e fattori secreti, potrebbe consentire la scoperta di nuove opzioni di terapia. Uniamo le tecniche di co-cultura conosciuta per creare un sistema modello per cancro e cellule staminali mesenchimali (MSCs) interazioni cellula. Nello studio corrente, le cellule staminali della polpa dentale (DPSCs) e interazioni cellula di PC-3 del carcinoma della prostata sono stati esaminati mediante tecniche dirette e indirette di co-coltura. Medio di condizione (CM) ottenuti da DPSCs e 0,4 µm poro dimensioni trans-pozzo membrane sono state usate per studiare l'attività paracrina. Co-coltura di diversi tipi di cellule insieme è stata effettuata per studiare l'interazione diretto cellula-cellula. I risultati hanno rivelato che CM aumentata proliferazione delle cellule ed in diminuzione di apoptosi nelle colture cellulari del carcinoma della prostata. Sia CM e trans-pozzo sistema aumentata capacità di migrazione delle cellule delle cellule PC-3. Le cellule colorate con membrana diversi coloranti erano teste di serie le stesse navi di cultura, e DPSCs ha partecipato a una struttura auto-organizzata con cellule PC-3 in questa condizione di co-coltura diretta. Nel complesso, i risultati hanno indicato che le tecniche di co-coltura potrebbero essere utile per cancro e interazioni di MSC come sistema modello.
Introduzione
Cellule staminali mesenchimali (MSCs), con la capacità di differenziazione e contributo alla rigenerazione dei tessuti mesenchimali quali l'osso, cartilagine, muscolo, legamento, tendine e adiposo, sono state isolate da quasi tutti i tessuti nel corpo adulto1 , 2. diversi dalla fornitura dell'omeostasi tissutale producendo cellule residenti in caso di infiammazione cronica o una ferita, che producono vitale citochine e fattori di crescita per orchestrare l'angiogenesi, sistema immunitario ed il tessuto che ritocca3. L'interazione di MSCs con tessuto del cancro non è ben compreso, ma raccogliendo la prova suggerisce che MSCs potrebbe promuovere di iniziazione, la progressione e la metastasi del tumore4.
La capacità di homing delle MSC nell'area lesa o cronicamente inflamed li rende un valido candidato per terapie basate sulle cellule staminali. Tuttavia, nei tessuti tumorali, "mai guarigione delle ferite", anche rilascio di citochine infiammatorie, molecole pro-angiogenici e fattori di crescita vitale, che attraggono MSCs al cancerogene zona5. Mentre ci sono limitati rapporti che mostrano gli effetti inibitori di MSCs su cancro crescita6,7, loro progressione tumorale e metastasi promuovere effetti sono stati ampiamente riportati8. MSCs direttamente o indirettamente influenzare la carcinogenesi in modi diversi, inclusa l'eliminazione di cellule immunitarie, che secernono le citochine/fattori di crescita che supportano la proliferazione della cellula tumorale e migrazione, migliorando l'attività angiogenica e regolazione transizione epiteliale-mesenchimale (EMT)9,10. Ambiente tumorale è costituito da diversi tipi cellulari fibroblasti cancro-collegati (CAFs) e/o miofibroblasti, cellule endoteliali, adipociti e cellule immuni11. Di quelli, CAFs sono il tipo più abbondante di cellule nella zona del tumore che secernono diverse chemochine, promuovere la crescita e la metastasi di cancro8. È stato dimostrato che MSCs derivate da midollo osseo possono differenziare in CAFs nel tumore dello stroma12.
Cellule staminali della polpa dentale (DPSCs), caratterizzate come il primo MSCs dentale di tessuto-derivato da Gronthos et al. 13 nel 2000 e quindi ampiamente studiato da altri14,15, esprimono marcatori di pluripotenza come Oct4, Sox2, Nanoge16 e possono differenziarsi in varie cellule linages17. Analisi di espressione genica e proteica ha dimostrato che DPSCs produrre livelli comparabili di fattori di crescita/citochine con altri MSCs come fattore di crescita endoteliale vascolare (VEGF), angiogenina, fattore di crescita del fibroblasto 2 (FGF2), interleuchina-4 (IL-4), IL-6, IL-10, e fattore di cellula formativa (SCF), così come ligando di chinasi-3 fms-come tirosina (Flt - 3L) che potrebbe promuovere l'angiogenesi, modulano le cellule immuni e sostenere il cancro delle cellule di proliferazione e migrazione18,19,20 . Mentre le interazioni di MSCs con ambiente di cancro sono state ben documentate nella letteratura, il rapporto tra DPSCs e le cellule tumorali non è stato valutato ancora. Nello studio presente, abbiamo stabilito le strategie di trattamento medio di co-cultura e condizione per una linea cellulare altamente metastatica del carcinoma della prostata, PC-3 e DPSCs a proporre azione potenziale del meccanismo di MSCs dentale nella progressione del cancro e metastasi.
Protocollo
Consenso informato dei pazienti è stata ottenuta dopo l'approvazione del Comitato di etica istituzionale.
1. DPSC isolamento e coltura
- Trasferire i denti del giudizio ottenuti da giovani adulti di età compresa tra 17 e 20 a 15 mL tubi contenenti per volta Eagle Medium (DMEM di Dulbecco completa) [bassa media DMEM glucosio, completato con 10% siero bovino fetale (FBS) e 1% di penicillina/streptomicina / amfotericina (PSA) soluzione], entro 8 h dopo resezione. Mantenere il tessuto materiale freddo (4 ° C) durante il trasferimento per evitare la potenziale delle cellule morte.
- Rimuovere attentamente il tessuto della polpa di pinze dentate sterile dal centro del dente, posizionare il tessuto della polpa a freddo medio DMEM completo in piastre di coltura del tessuto di 10cm e tritateli in piccoli pezzi (2-3 mm) di bisturi.
Nota: Tutte le procedure sperimentali devono essere effettuate in condizioni di sterilità in cappa a flusso laminare. Questa tecnica non-enzimatica è stato utilizzato in precedenza21,22,23. - Tessuti di polpa piccolo posto all'interno della cultura del tessuto trattato piastre da 6 pozzetti e aggiungere 200 µ l di multimediale DMEM completo per coprire ogni pezzi di tessuto di piccola polpa.
- Incubare i pozzetti di coltura del tessuto a 37 ° C in un'atmosfera di aria umidificata (80% di umidità) con 5% CO2 per 2 h fornire il collegamento del tessuto.
Nota: Questo passaggio potrebbe essere prolungato a 3-4 h controllando l'evaporazione dei media. - Aggiungere volume appropriato (2-2,5 mL) di terreno DMEM completo ai pozzetti e incubare a 37 ° C in un'atmosfera di aria umidificata con 5% CO2 per celle a diffondersi dal tessuto.
Nota: Le cellule diventano visibili dopo circa 4 giorni e raggiungono la confluenza dopo 8-9 giorni. - Quando le cellule raggiungono 80% confluency, rimuovere supporti da piastre da 6 pozzetti, lavare con 2 mL di tampone fosfato salino (PBS) e aggiungere 2 mL di tripsina. Incubare per 2 min in un incubatore a 37 ° C con atmosfera di aria umidificata e 5% CO2. Quindi aggiungere 2 mL di terreno DMEM completo seguito da incubazione 2 min per inibire la tripsina. Centrifugare le cellule a 300 x g per 5 min agglomerare le cellule.
- Cellule di passaggio per le beute in completa DMEM media e archivio per ulteriori esperimenti. Aggiungere 15 mL di multimediale completo di DMEM al boccette di T-75 e cellule di trasferimento da due pozzetti della piastra 6 pozzetti per boccette di T-75 uno e incubare a 37 ° C con aria umidificata atmosfera e 5% CO2.
2. caratterizzazione di DPSCs
- Eseguire analisi morfologiche.
- Cellule di seme (passo 1.6) nella coltura del tessuto rivestito boccette (o piastre da 6 pozzetti) in terreno DMEM completo per almeno 8 passaggi osservare la morfologia delle cellule.
- Visualizzare le cellule dal microscopio ottico e definire la morfologia delle cellule del fibroblasto-come. Le cellule dovrebbero allegare per i piatti di cultura e morfologia delle cellule dell'alberino-come.
Nota: In alternativa, le cellule possono essere coltivate come singole cellule fino a 14 giorni in piastre a pozzetti per osservare la capacità di formazione di Colonia che è una caratteristica specifica dei fibroblasti e MSCs.
- Eseguire analisi di superficie dell'indicatore.
- Tripsinizzano le cellule dal punto 1.7. Rimuovere il supporto dal pallone di coltura del tessuto T-75, lavare con 2 mL di PBS e aggiungere 2 mL di tripsina. Incubare per 2 min in un incubatore a 37 ° C con atmosfera di aria umidificata e 5% CO2. Quindi aggiungere 2 mL di terreno DMEM completo seguito da incubazione 2 min per inibire la tripsina. Centrifugare le cellule a 300 x g per 5 min agglomerare le cellule.
- Fissare le cellule con paraformaldeide al 4% per 20 min a temperatura ambiente in provette da 1,5 mL e poi lavarli con 500 µ l di PBS 3 volte per rimuovere paraformaldeide.
- Incubare le cellule fisse con gli anticorpi contro CD29, CD34, CD14, CD45, CD90, CD105, CD166 e CD73 per 1 h a 4 ° C in 100 µ l di PBS.
Nota: La concentrazione di anticorpo utilizzato è 0,5 µ g/mL. CD45, CD14 e CD34 sono usati come indicatori negativi, mentre CD29, CD90, CD105, CD166 e CD73 sono usati come indicatori della superficie delle cellule positive per MSCs. - Lavare le cellule 3 volte con PBS e utilizzare i rispettivi anticorpi secondari quali isotiocianato di fluorescina (FITC), ficoeritrina (PE), ecc per l'etichettatura. Incubare le cellule con anticorpi secondari 1: 500 diluito in 100 µ l di PBS per 30 min a 4 ° C e lavare 3 volte con PBS.
- Mantenere i campioni al buio per l'analisi di citometria a flusso e rilevare positivi e negativi di colorazione tramite flusso cytometry.
Nota: Utilizzare le cellule di controllo non macchiate per disporre dispersione avanti e laterali. Organizzare il gating di positivamente macchiato popolazioni escludendo le cellule morte, detriti e popolazione non-macchiata. Utilizzare 100 µm ugello con 45 psi di pressione di guaina e raccogliere 10.000 eventi per determinare DPSCs positivo organizzando canali.
- Eseguire la differenziazione di DPSCs.
- Cellule di4 seme 1 × 10 sulle piastre a 24 pozzetti in completano media DMEM e incubare per 24 h a 37 ° C in un'atmosfera di aria umidificata con 5% CO2.
- Formulare la differenziazione media utilizzando competere medium DMEM come supporto di base. Preparare i supporti osteogenici mescolando 100 nM desametasone, β-glicerofosfato di 10 mM e acido ascorbico 0.2 mM. Preparare i supporti condrogenica mescolando 1 × insulina-transferrina-selenio (ITS-G), 100 nM desametasone, 100 ng/mL crescita fattore trasformante beta (TGF-β), 14 μg/mL di acido ascorbico e 1 mg/mL albumina di siero bovino (BSA). Preparare i supporti adipogenico mescolando 100 nM desametasone, insulina di 5 μg/mL, 0,5 mM 3-isobutyl-1-methylxanthine (IBMX) e indometacina di 60 μM.
Nota: Media di differenziazione può essere mantenuto a 4 ° C per almeno una settimana. - Cambiare la crescita media a osteo-, condro- o differenziazione adipo-genica supporto e aggiornamento media due volte a settimana per due settimane.
- Confermare la differenziazione di von Kossa e Alcian blu colorazione, attività enzimatica (attività della fosfatasi alcalina), immunocitochimica e analisi (qPCR) reazione a catena della polimerasi quantitativa secondo i protocolli precedentemente descritto21.
- Eseguire colorazioni di Alcian blu e von Kossa sulle cellule che sono fissate con paraformaldeide al 4% a temperatura ambiente per 10 min. lavare le cellule fisse con PBS e li macchia con il kit di vonKossa secondo le raccomandazioni del produttore per osservare i giacimenti del calcio .
- Preparare la soluzione di colorazione sciogliendo 1,00 g di colorante blu di Alcian in 100 mL di acido acetico al 3% (v/v) per ulteriore colorazione blu di Alcian. Lavare le cellule fisse con PBS e macchia le cellule per 30 min con soluzione di Alcian blu. Visualizzare i campioni macchiati da un microscopio ottico.
3. preparazione della condizione medio (CM)
- Sostituire il supporto delle cellule dal passaggio 1.7 con fresca completa DMEM 24 h prima collezione CM.
Nota: Passaggio 2-4 è consigliato. - Raccogliere media condizione (CM) da DPSCs colta quando le cellule di raggiungere confluency di 80%. Centrifugare media raccolti a 300 x g per 5 min rimuovere rifiuti tessuto materiale e detriti cellulari.
Nota: In alternativa, utilizzare filtri per siringa 0,2 µm per rimuovere i detriti dal mezzo di condizione. - Raccogliere il surnatante e conservare a-20 ° C per ulteriori esperimenti.
Nota: Conservare il supernatante a-80 ° C per la conservazione a lungo termine.
4. trattamento delle cellule tumorali con CM
- Eseguire analisi di attuabilità delle cellule.
- Cellule PC-3 semi (cellule di carcinoma della prostata umane) in piastre da 96 pozzetti ad una densità di cella di 5 × 103 cellule/pozzetto in DMEM di completare e incubare in una camera umidificata a 37 ° C e 5% di CO2 per 24 h.
- Curare le cellule con 10, 20, 30, 40 e 50% cm (v/v) mescolate con DMEM completo per 24 h.
- Misurare la vitalità cellulare tramite 3-(4,5-dimethyl-thiazol-2-yl)-5-(3-carboxymethoxy-phenyl)-2-(4-sulfo-phenyl)-2H-tetrazolium (MTS)-dosaggio come descritto in precedenza24.
- Eseguire terminal deoxynucleotidyl transferase dUTP nick fine etichettatura analisi (TUNEL).
- Cellule PC-3 seme su coltura cellulare 6 pozzetti piastre ad una densità di cella di 2 × 105 cellule/pozzetto e incubare per una notte in una camera umidificata a 37 ° C e 5% CO2 .
- Mescolare 20% (v/v) di CM con completare mezzo DMEM e applicare alle celle per 24 h.
- Tripsinizzano cellule e sospendere in 50 μL di miscela di reazione di TUNEL (etichettatura soluzione + soluzione enzimatica, fornito nel kit) e incubare a 37 ° C per 60 min in un'atmosfera di2 umidificata e 5% di CO.
Nota: Per trypsinization, rimuovere i supporti da piastre per colture cellulari a 6 pozzetti, lavare con 1 mL di PBS e aggiungere 500 µ l di tripsina. Incubare per 2 min in un incubatore a 37 ° C con atmosfera di aria umidificata e 5% CO2. Aggiungere 1 mL di terreno DMEM completo seguito da incubazione 2 min per inibire la tripsina. Centrifugare le cellule a 300 x g per 5 min agglomerare le cellule. - Risciacquare con PBS e analizzare le cellule in PBS tramite flusso cytometry.
- Eseguire analisi qPCR.
- Cellule PC-3 seme su coltura cellulare 6 pozzetti piastre ad una densità di cella di 2 × 105 cellule/pozzetto e incubare per una notte in un incubatore a 37 ° C e 5% CO2 .
- Mescolare 20% (v/v) di CM con completare mezzo DMEM e applicare alle celle per 24 h.
- Tripsinizzano cellule e raccogliere il pellet cellulare per la sintesi di RNA isolamento e cDNA.
Nota: Rimuovere i supporti da piastre per colture cellulari a 6 pozzetti, lavare con 1 mL di PBS e aggiungere 500 µ l di tripsina. Incubare 2 min in un incubatore a 37 ° C con atmosfera di aria umidificata e 5% CO2. Aggiungere 1 mL di terreno DMEM completo seguito da incubazione 2 min per inibire la tripsina. Centrifugare le cellule a 300 x g per 5 min agglomerare le cellule. - Esperimenti di qPCR secondo il protocollo descritto in precedenza25.
- Eseguire la migrazione delle cellule delle cellule tumorali.
- Seme 1 × 105 PC-3 cellule sul 12-pozzetti e incubare in un incubatore umidificato durante la notte a 37 ° C e 5% CO2.
- Graffiare le cellule con una punta sterile 200 μL e sostituire il terreno immediatamente con medium fresco contenenti varie concentrazioni di CM [ad es., 10, 20, 30, 40 e 50% cm (v/v) mescolato con DMEM completo].
- Osservare graffi sotto un microscopio invertito e scattare foto a intervalli di tempo diversi (0 e 24 h).
- Misurare la chiusura gratta e Vinci utilizzando il software Image J utilizzando la formula:
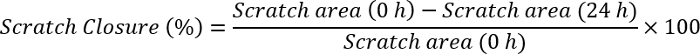
Nota: Aprire l'immagine gratta e Vinci con software Image J. Disegnare una linea che ha la stessa grandezza come la barra della scala che esiste già nell'immagine. Fare clic su analizza, impostare scala e osservare la distanza in pixel come l'ingrandimento della linea disegnata. Scrivere la dimensione della barra della scala alla parte di distanza nota, organizzare unità (pixel, centimetri, ecc.) e fare clic su ok. Vai alla sezione analizza nuovamente e clicca su misure. In primo luogo, questo vi darà la dimensione della barra della scala come unità di misura selezionata. Fare clic su un bordo di gratta e Vinci e trascinare fino a raggiungere l'altra estremità del graffio. Prendere nota del valore di ciascun punto di tempo (0 h e 24 h). Inserire questi valori nella formula sopra riportata e calcolare la chiusura gratta e Vinci.
5. cell migrazione per contatto indiretto delle cellule tumorali e DPSCs
- Seme 3 × 104 DPSCs sulla piastra a 24 pozzetti inserti con 0,4 μm del poro e incubare in un incubatore durante la notte a 37 ° C.
- Le cellule PC-3 di semi sui 24 pozzetti piastre a una densità di cella di 5 × 104 e incubare in un incubatore umidificato durante la notte a 37 ° C e 5% CO2.
- Graffiare le cellule PC-3 con una punta sterile 200 μL, sostituire il terreno con mezzo fresco e posizionare inserti portato DPSCs sulla cellule PC-3.
- Osservare le cellule al microscopio invertito e scattare foto a intervalli di tempo diversi (0 e 24 h) per analizzare la migrazione cellulare.
6. analisi di co-cultura e analisi di citometria a flusso
- Etichetta le cellule PC-3 e DPSCs utilizzando PKH67 (verde) e PKH26 (rosso) delle cellule fluorescenti del linker coloranti, rispettivamente26.
- Tripsinizzano cellule PC-3 e DPSCs, rispettivamente. Rimuovere il supporto dal pallone di coltura del tessuto T-75, lavare con 2 mL di PBS e aggiungere 2 mL di tripsina. Incubare per 2 min in un incubatore a 37 ° C con atmosfera di aria umidificata e 5% CO2. Aggiungere 2 mL di terreno DMEM completo seguito da incubazione 2 min per inibire la tripsina.
- Centrifugare le cellule a 300 x g per 5 min, eliminare il surnatante e risospendere il pellet cellulare nella soluzione colorante preparata in tampone diluente-C fornito dal kit (Vedi Tabella materiali).
- Incubare le cellule nella soluzione colorante per 10 min e terminare la reazione di macchiatura aggiungendo 100 µ l di FBS. Centrifugare le cellule a 300 x g per 5 min, scartare il surnatante e lavare le cellule con il mezzo di crescita completa prima di co-coltura.
- Piastra con l'etichetta cellule (5 × 104/bene) su piastre da 6 pozzetti con rapporto 1:1 (DPSCs: PC3). Mantenere le cellule co-coltivate in DMEM completo.
- Raccogliere le cellule dopo periodi di incubazione di 24 o 48 ore mediante centrifugazione delle cellule a 300 x g per 5 min ed il lavaggio con PBS.
- Risospendere le cellule in 300 µ l di fluorescenza-attivato delle cellule ordinamento buffer (FACS) in 5 mL basso flusso cytometry tubi rotondi. Vortex per disperdere aggregati cellulari in prima analisi del campione.
- Utilizzare un ugello di 100 µm con 45 psi di pressione di guaina.
Nota: Estremamente alta portata potrebbe diminuire la sensibilità di rilevazione di fluorescenza. - Utilizzare le cellule di controllo non macchiate e singole celle colorate per regolare appropriato avanti e tensione di laser a dispersione laterale per tipi cellulari e compensazione come accennato in precedenza il27.
Nota: Utilizzare gating per cellule vive per escludere detriti cellulari, cellule morte o aggregati. - Raccogliere 10.000 eventi (100.000 è preferibile) per determinare la percentuale positiva DPSCs verde e rosse cellule PC-3 organizzando FL-1 (verde) e canali di FL-2 (rosso).
Risultati
Figura 1 illustra le caratteristiche generali di MSC di DPSCs in condizioni di coltura. DPSCs esercitare morfologia fibroblasto-come delle cellule dopo il placcaggio (Figura 1B). Antigeni di superficie MSC (CD29, CD73, CD90, CD105 e CD166) sono altamente espressi mentre marcatori ematopoietici (CD34, CD45 e CD14) sono negativi (Figura 1). Modifiche a livello morfologico e molecolare relativo a osteo-...
Discussione
Contributo di MSCs per ambiente tumorale è regolata da numerose interazioni tra cui ibrido cellulare generazione tramite cella fusioni, Citoplasto o citochine e chemochine attività tra cellule staminali e cancro cellule28. Organizzazione strutturale, interazioni cellula-cellula e fattori secreti determinano il comportamento delle cellule di cancro in termini di promozione del tumore, la progressione e la metastasi al tessuto circostante. Corretto ex vivo sistemi modello per stu...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo studio è stato supportato da Yeditepe University. Tutti i dati e i dati utilizzati in questo articolo sono stati precedentemente pubblicati34.
Materiali
| Name | Company | Catalog Number | Comments |
| DMEM | Invitrogen | 11885084 | For cell culture |
| FBS | Invitrogen | 16000044 | For cell culture |
| PSA | Lonza | 17-745E | For cell culture |
| Trypsin | Invitrogen | 25200056 | For cell dissociation |
| PBS | Invitrogen | 10010023 | For washes |
| Dexamethasone | Sigma | D4902 | Component of differentiation media |
| β-Glycerophosphate | Sigma | G9422 | Component of osteogenic differentiation medium |
| Ascorbic acid | Sigma | A4544 | Component of osteo- and chondro-genic differentiation medium |
| Insulin-Transferrin-Selenium (ITS −G) | Invitrogen | 41400045 | Component of chondrogenic differentiation medium |
| TGF-β | Sigma | SRP3171 | Component of chondrogenic differentiation medium |
| Insulin | Sigma | I6634 | Component of adipogenic differentiation medium |
| Isobutyl-1-methylxanthine (IBMX) | Sigma | I7018 | Component of adipogenic differentiation medium |
| Indomethacin | Sigma | I7378 | Component of adipogenic differentiation medium |
| MTS Reagent | Promega | G3582 | Cell viability analyses |
| TUNEL Assay | Sigma | 11684795910 | Apoptotic analyses |
| 24-well plate inserts | Corning | 3396 | For trans-well migration assay |
| PKH67 | Sigma | PKH67GL | For co-culture cell staining |
| PKH26 | Sigma | PKH26GL | For co-culture cell staining |
| Paraformaldehyde | Sigma | P6148 | For cell fixation |
| von Kossa Kit | BioOptica | 04-170801.A | For cell staining (differentiation) |
| Alcian blue | Sigma | A2899 | For cell staining (differentiation) |
Riferimenti
- Camberlain, G., Fox, J., Ashton, B., Middleton, J. Mesenchymal stem cells: their phenotype, differentiation capacity, immunological features, and potential for homing. Stem Cells. 25 (11), 2739-2749 (2007).
- Demirci, S., Doğan, A., Şahin, F. . Dental Stem Cells. , 109-124 (2016).
- Fox, J. M., Chamberlain, G., Ashton, B. A., Middleton, J. Recent advances into the understanding of mesenchymal stem cell trafficking. British journal of haematology. 137 (6), 491-502 (2007).
- Chang, A. I., Schwertschkow, A. H., Nolta, J. A., Wu, J. Involvement of mesenchymal stem cells in cancer progression and metastases. Current cancer drug targets. 15 (2), 88-98 (2015).
- Dvorak, H. F. Tumors: wounds that do not heal. New England Journal of Medicine. 315 (26), 1650-1659 (1986).
- Lu, Y. -. r., et al. The growth inhibitory effect of mesenchymal stem cells on tumor cells in vitro and in vivo. Cancer biology & therapy. 7 (2), 245-251 (2008).
- Secchiero, P., et al. Human bone marrow mesenchymal stem cells display anti-cancer activity in SCID mice bearing disseminated non-Hodgkin's lymphoma xenografts. PloS one. 5 (6), e11140 (2010).
- Hong, I. -. S., Lee, H. -. Y., Kang, K. -. S. Mesenchymal stem cells and cancer: friends or enemies?. Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 768, 98-106 (2014).
- Brennen, W. N., Chen, S., Denmeade, S. R., Isaacs, J. T. Quantification of Mesenchymal Stem Cells (MSCs) at sites of human prostate cancer. Oncotarget. 4 (1), 106 (2013).
- Klopp, A. H., Gupta, A., Spaeth, E., Andreeff, M., Marini, F. Concise review: dissecting a discrepancy in the literature: do mesenchymal stem cells support or suppress tumor growth. Stem cells. 29 (1), 11-19 (2011).
- Albini, A., Sporn, M. B. The tumour microenvironment as a target for chemoprevention. Nature Reviews Cancer. 7 (2), 139 (2007).
- Quante, M., et al. Bone marrow-derived myofibroblasts contribute to the mesenchymal stem cell niche and promote tumor growth. Cancer cell. 19 (2), 257-272 (2011).
- Gronthos, S., Mankani, M., Brahim, J., Robey, P. G., Shi, S. Postnatal human dental pulp stem cells (DPSCs) in vitro and in vivo. Proceedings of the National Academy of Sciences. 97 (25), 13625-13630 (2000).
- Mori, G., et al. Dental pulp stem cells: osteogenic differentiation and gene expression. Annals of the new York Academy of Sciences. 1237 (1), 47-52 (2011).
- Mori, G., et al. Osteogenic properties of human dental pulp stem cells. Journal of biological regulators and homeostatic agents. 24 (2), 167-175 (2010).
- Kerkis, I., et al. Isolation and characterization of a population of immature dental pulp stem cells expressing OCT-4 and other embryonic stem cell markers. Cells Tissues Organs. 184 (3-4), 105-116 (2006).
- Potdar, P. D., Jethmalani, Y. D. Human dental pulp stem cells: applications in future regenerative medicine. World journal of stem cells. 7 (5), 839 (2015).
- Ahmed, N. E. -. M. B., Murakami, M., Hirose, Y., Nakashima, M. Therapeutic potential of dental pulp stem cell secretome for Alzheimer's disease treatment: an in vitro study. Stem cells international. 2016, (2016).
- Gorin, C., et al. Priming dental pulp stem cells with fibroblast growth factor-2 increases angiogenesis of implanted tissue-engineered constructs through hepatocyte growth factor and vascular endothelial growth factor secretion. Stem cells translational medicine. 5 (3), 392-404 (2016).
- Wakayama, H., et al. Factors secreted from dental pulp stem cells show multifaceted benefits for treating acute lung injury in mice. Cytotherapy. 17 (8), 1119-1129 (2015).
- Doğan, A., et al. Differentiation of human stem cells is promoted by amphiphilic pluronic block copolymers. International Journal of Nanomedicine. 7, 4849 (2012).
- Taşlı, P. N., Doğan, A., Demirci, S., Şahin, F. Boron enhances odontogenic and osteogenic differentiation of human tooth germ stem cells (hTGSCs) in vitro. Biological trace element research. 153 (1-3), 419-427 (2013).
- Yalvac, M., et al. Isolation and characterization of stem cells derived from human third molar tooth germs of young adults: implications in neo-vascularization, osteo-, adipo-and neurogenesis. The pharmacogenomics journal. 10 (2), 105 (2010).
- Doğan, A., et al. Sodium pentaborate pentahydrate and pluronic containing hydrogel increases cutaneous wound healing in vitro and in vivo. Biological trace element research. 162 (1-3), 72-79 (2014).
- Doğan, A., Yalvaç, M. E., Yılmaz, A., Rizvanov, A., Şahin, F. Effect of F68 on cryopreservation of mesenchymal stem cells derived from human tooth germ. Applied biochemistry and biotechnology. 171 (7), 1819-1831 (2013).
- Rizvanov, A. A., et al. Interaction and self-organization of human mesenchymal stem cells and neuro-blastoma SH-SY5Y cells under co-culture conditions: A novel system for modeling cancer cell micro-environment. European Journal of Pharmaceutics and Biopharmaceutics. 76 (2), 253-259 (2010).
- Troiano, L., et al. Multiparametric analysis of cells with different mitochondrial membrane potential during apoptosis by polychromatic flow cytometry. Nature protocols. 2 (11), 2719 (2007).
- Melzer, C., von der Ohe, J., Lehnert, H., Ungefroren, H., Hass, R. Cancer stem cell niche models and contribution by mesenchymal stroma/stem cells. Molecular cancer. 16 (1), 28 (2017).
- Aguirre, A., Planell, J., Engel, E. Dynamics of bone marrow-derived endothelial progenitor cell/mesenchymal stem cell interaction in co-culture and its implications in angiogenesis. Biochemical and biophysical research communications. 400 (2), 284-291 (2010).
- Bogdanowicz, D. R., Lu, H. H. . Biomimetics and Stem Cells. , 29-36 (2013).
- Plotnikov, E., et al. Cell-to-cell cross-talk between mesenchymal stem cells and cardiomyocytes in co-culture. Journal of cellular and molecular. 12 (5a), 1622-1631 (2008).
- Bogdanowicz, D. R., Lu, H. H. Studying cell-cell communication in co-culture. Biotechnology journal. 8 (4), 395-396 (2013).
- Brunetti, G., et al. High expression of TRAIL by osteoblastic differentiated dental pulp stem cells affects myeloma cell viability. Oncology reports. 39 (4), 2031-2039 (2018).
- Doğan, A., Demirci, S., Apdik, H., Apdik, E. A., Şahin, F. Dental pulp stem cells (DPSCs) increase prostate cancer cell proliferation and migration under in vitro conditions. Tissue and Cell. 49 (6), 711-718 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon