Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Técnicas de Espectrometría de Masa de Movilidad de Iones para Determinar la Estructura y Mecanismos de Reconocimiento de Iones Metálicos y Actividad Redox de Oligopéptidos de Unión De Metal
En este artículo
Resumen
Las técnicas de modelado molecular y espectrometría de masas de movilidad ión pueden caracterizar el rendimiento selectivo de quelantes metálicos de los péptidos de unión a metales diseñados y el péptido de unión de cobre methanobactin. El desarrollo de nuevas clases de péptidos quelantes metálicos ayudará a llevar a terapias para enfermedades asociadas con el desequilibrio de iones metálicos.
Resumen
La ionización por electrospray (ESI) puede transferir un péptido de fase acuosa o un complejo de péptidos a la fase gaseosa, al tiempo que conserva su masa, carga general, interacciones de unión de metales y forma de conformación. El acoplamiento ESI con espectrometría de masas de movilidad ión (IM-MS) proporciona una técnica instrumental que permite la medición simultánea de la sección transversal de masa a carga (m/z) y de colisión (CCS) de un péptido que se relacionan con su estequiometría, estado de protonación, y forma conformacional. La carga general de un complejo de péptidos se controla mediante la protonación de 1) los sitios ácidos y básicos del péptido y 2) el estado de oxidación de los iones metálicos. Por lo tanto, el estado de carga general de un complejo es una función del pH de la solución que afecta a los péptidos de la afinidad de unión de iones metálicos. Para los análisis ESI-IM-MS, las soluciones de péptidos e iones metálicos se preparan a partir de soluciones solo acuosas, con el pH ajustado con ácido acético acuoso diluido o hidróxido de amonio. Esto permite determinar la dependencia del pH y la selectividad de iones metálicos para un péptido específico. Además, el m/z y el CCS de un complejo de péptidos se pueden utilizar con el modelado molecular B3LYP/LanL2DZ para discernir los sitios de unión de la coordinación de iones metálicos y la estructura terciaria del complejo. Los resultados muestran cómo ESI-IM-MS puede caracterizar el rendimiento de quelación selectiva de un conjunto de péptidos alternativos de methanobactina y compararlos con el péptido de unión de cobre methanobactin.
Introducción
Los iones de cobre y zinc son esenciales para los organismos vivos y cruciales para procesos como la protección oxidativa, el crecimiento del tejido, la respiración, el colesterol, el metabolismo de la glucosa y la lectura del genoma1. Para habilitar estas funciones, grupos como el tiolato de Cys, el imidazol de Su2,3, (más raramente) tioéter de metionina, y carboxilato de Glu y Asp incorporan selectivamente metales como cofactores en los sitios activos de metaloenzimas. La similitud de estos grupos de coordinación plantea una pregunta intrigante sobre cómo los ligandos Suyos y De Los Cys incorporan selectivamente Cu(I/II) o Zn(II) para asegurar el correcto funcionamiento.
La unión selectiva se realiza a menudo mediante la adquisición y el tráfico de péptidos, que controlan las concentraciones iónquires de Zn(II) o Cu(I/II)4. Cu(I/II) es altamente reactivo y causa daño oxidativo o unión adventicia a las enzimas, por lo que su concentración libre está estrechamente regulada por chaperones de cobre y proteínas reguladoras de cobre que lo transportan de forma segura a varios lugares de la célula y estrechamente controlar su homeostasis5,6. La interrupción del metabolismo del cobre o la homeostasis está directamente implicada en la enfermedad de Menkes y Wilson7, así como en los cánceres7 y trastornos neuronales, como el prión8 y la enfermedad de Alzheimer9.
La enfermedad de Wilson se asocia con el aumento de los niveles de cobre en los ojos, el hígado y las secciones del cerebro, donde las reacciones redox de Cu(I/II) producen especies reactivas de oxígeno, causando degeneración hepatolenticular y neurológica. Las terapias de quelación existentes son el pequeño aminoácido tiol penicilamina y trietiletetramina. Alternativamente, los péptidos metanotropoficos de adquisición de cobre methanobactin (mb)10,11 exhiben potencial terapéutico debido a su alta afinidad de unión para Cu(I)12. Cuando el methanobactin (mb-OB3b) de Methylosinus trichosporium OB3b fue estudiado en un modelo animal de la enfermedad de Wilson, el cobre fue eliminado eficientemente del hígado y excretado a través de la bilis13. Los experimentos in vitro confirmaron que mb-OB3b podría quelar el cobre del metalotionino de cobre contenido en el citosol hepático13. Las técnicas de imágenes de espectrometría de masas plasmáticas acopladas inductivamente de ablación láser han investigado la distribución espacial del cobre en las muestras hepáticas de la enfermedad de Wilson14,15,16 y han demostrado que mb-OB3b elimina el cobre con períodos de tratamiento cortos de sólo 8 días17.
El mb-OB3b también se unirá a otros iones metálicos, incluidos Ag(I), Au(III), Pb(II), Mn(II), Co(II), Fe(II), Ni(II) y Zn(II)18,19. La competencia por el sitio de unión fisiológico Cu(I) es exhibida por Ag(I) porque puede desplazar a Cu(I) del complejo mb-OB3b, con Ag(I) y Ni(II) mostrando también unión irreversible a Mb que no puede ser desplazada por Cu(I)19. Recientemente, se han estudiado una serie de oligopéptidos alternativos de methanobactina (amb) con el motivo de unión 2His-2Cys20,21,y sus propiedades de unión Zn(II) y Cu(I/II) caracterizadas. Sus secuencias de aminoácidos primarios son similares, y todas contienen el motivo 2His-2Cys, Pro y un cesación acetilado N-terminus. Se diferencian principalmente de mb-OB3b porque el motivo 2His-2Cys reemplaza los dos sitios de unión de oxazolona de enethiol de mb-OB3b.
La ionización por electrospray junto con la espectrometría de masas de movilidad ión (ESI-IM-MS) proporciona una poderosa técnica instrumental para determinar las propiedades de unión al metal de los péptidos porque mide su masa a carga (m/z) y la colisión sección transversal (CCS) conservando su masa, carga y forma de conformación de la fase de solución. El m/z y el CCS se relacionan con los péptidos etoquiometría, estado de protonación y forma de conformación. La estequiometría se determina porque la identidad y el número de cada elemento presente en la especie se identifican explícitamente. La carga general del complejo de péptidos se relaciona con el estado de protonación de los sitios ácidos y básicos y el estado de oxidación de los iones metálicos. El CCS proporciona información de la forma conformacional del complejo peptídico porque mide el tamaño medio rotacional que se relaciona con la estructura terciaria del complejo. El estado de carga general del complejo también es una función del pH y afecta a la afinidad de unión de iones metálicos del péptido porque los sitios básicos o ácidos desprotonados como el carboxilo, His, Cys y Tyr son también los sitios de unión potenciales para el ion metálico. Para los análisis, el péptido y el ion metálico se preparan en soluciones acuosas con el pH ajustado por ácido acético acuoso diluido o hidróxido de amonio. Esto permite determinar la dependencia del pH y la selectividad de iones metálicos para el péptido. Además, el m/z y el CCS determinados por ESI-IM-MS se pueden utilizar con el modelado molecular B3LYP/LanL2DZ para descubrir el tipo de coordinación de iones metálicos y la estructura terciaria del complejo. Los resultados mostrados en este artículo revelan cómo ESI-IM-MS puede caracterizar el rendimiento de quelación selectiva de un conjunto de péptidos amb y compararlos con el péptido de unión de cobre mb-OB3b.
Protocolo
1. Preparación de reactivos
- Cultivo Methylosinus trichosporium OB3b, aislar el Cu(I) libre de mb-OB3b18,22,23, congelar-secar la muestra y almacenar a -80 oC hasta su uso.
- Sintetizar los péptidos amb (>98% pureza para amb1, amb2, amb4; >70% pureza para amb7), congelar-secar las muestras, y almacenarlas a -80 oC hasta su uso.
- Compra >98% de cloruro de manganeso de pureza(II), cloruro de cobalto (II), cloruro de nickle(II), cloruro de cobre(II), nitrato de cobre(II), nitrato de plata(I), cloruro de zinc(II), cloruro de hierro (III) y cloruro de plomo(II).
- Comprar los polímeros de poli-DL-alanina utilizados como calibradores para medir las secciones transversales de colisión de la especie amb y el hidróxido de amonio de grado HPLC o superior, ácido acético glacial y acetonitrilo.
2. Preparación de la solución de stock
-
Solución de stock de péptidos
- Pesar con precisión, utilizando al menos tres cifras significativas, la masa de 10,0–20,0 mg del mb-OB3b o amb en un vial de plástico de 1,7 ml.
NOTA: La masa pesada debe producir 12,5 mM o 1,25 mM, dependiendo de la solubilidad del péptido, cuando se añade 1,00 ml de agua desionizada (DI). - Con un pipeta, añada 1,00 ml de agua desionizada (>17,8 M cm) a la muestra de péptido pesado para producir la solución de 12,5 mM o 1,25 mM. Coloque la tapa firmemente y mezcle bien con al menos 20 inversiones.
- Usando un micropipete dispensa alícuotas de 50,0 l a partir de la muestra de péptido en viales de 1,5 ml etiquetados individualmente y guárdelas a -80 oC hasta su uso.
- Pesar con precisión, utilizando al menos tres cifras significativas, la masa de 10,0–20,0 mg del mb-OB3b o amb en un vial de plástico de 1,7 ml.
-
Soluciones de stock de iones metálicos
- Pesar con precisión, utilizando al menos tres cifras significativas, la masa de 10,0–30,0 mg del cloruro metálico o nitrato de plata en un vial de 1,7 ml.
NOTA: La masa pesada debe producir 125 mM cuando se añade 1,00 mL de agua DI. - Añadir los 1,00 ml de agua DI a la muestra de metal pesada en el vial de 1,7 ml para producir la solución de 125 mM. Coloque la tapa firmemente y mezcle bien con al menos 20 inversiones.
- Pesar con precisión, utilizando al menos tres cifras significativas, la masa de 10,0–30,0 mg del cloruro metálico o nitrato de plata en un vial de 1,7 ml.
- Soluciones de stock de hidróxido de amonio:preparar una solución de ácido acético de 1,0 M diluyendo 57 ml de la solución de ácido acético del 99,5% con agua DI a un volumen final de 1,00 ml. Preparar una solución de hidróxido de amonio de 1,0 M diluyendo 90 ml de la solución de hidróxido de amonio del 21% con agua DI a un volumen final de 1,00 ml. Realice dos diluciones sucesivas de cada solución tomando 100 ml de las soluciones de 1,0 M para preparar soluciones de ácido acético y hidróxido de amonio de 0,10 M y 0,010 M.
- Solución de stock de poli-DL-alanina:preparar la poli-DL-alanina (PA) pesando 1,0 mg de PA y disolviendo en 1,0 ml de agua DI para dar 1.000 ppm. Homogeneizar. Con un micropipete, dispensar alís 50,0 l y colocar cada una en un vial de 1,7 ml y almacenar a -80 oC.
3. Análisis de espectrometría de masas de movilidad de iones electrospray
- Limpie a fondo el tubo de entrada ESI y el capilar de la aguja con aproximadamente 500 ml de ácido acético glacial de 0,1 M, hidróxido de amonio de 0,1 M y, finalmente, agua DI.
- Descongelar una alícuota de 50,0 l de la solución de stock PA de 1.000 ppm y diluirla con 450 ml de agua DI para dar un PA de 100 ppm. Pipet 100.0 l de esta solución y diluirla a 1,00 ml con 500 ml de agua DI y 500 ml de acetonitrilo para dar 10 ppm de solución PA.
- Recopile los espectros IM-MS de iones negativos y positivos de la solución PA de 10 ppm durante 10 minutos cada uno utilizando condiciones nativas de ESI-IM-MS como se describe en la sección de discusión.
- Descongelar una alícuota de 50,0 l de la solución de stock de 12,5 mM o 1,25 mM y realizar diluciones sucesivas con agua DI para dar una concentración final de 0,125 mM amb. Mezcle bien cada dilución.
- Pipet 100.0 l de la solución de material de iones metálicos de 125 mM, colocar en un vial de 1,7 ml y diluir a 1,00 ml con agua DI para dar 12,5 mM de iones metálicos. Repita con dos diluciones sucesivas más para dar una concentración final de iones metálicos de 0,125 mM. Mezcle bien cada dilución.
- Pipet 200.0 l de la 0,125 mM amb en un vial de 1,7 ml, diluir con 500 ml de agua DI y mezclar bien la solución.
- Ajuste el pH de la muestra a 3,0 añadiendo 50 ml de solución de ácido acético de 1,0 M.
- Añadir 200,0 l de un ion metálico de 0,125 mM a la muestra ajustada al pH. Añadir agua DI para producir un volumen final de 1,00 ml de la muestra, mezclar a fondo, y permitir que la muestra se equilibre durante 10 minutos en RT.
- Con una jeringa de nariz contundente, tome 500 ml de la muestra y recoja los espectros de iones negativos y positivos ES-IM-MS durante 5 min cada uno. Utilice los 500 ml restantes de la muestra para registrar su pH final utilizando un electrodo de micro pH calibrado.
- Repita los pasos 3.6–3.9, mientras modifica el paso 3.7 para ajustar el pH a 4.0, 5.0, 6.0, 7.0, 8.0, 9.0 o 10.0 agregando nuevos volúmenes de las soluciones de ácido acético o hidróxido de amonio de 0.010 M, 0.10 M o 1.0 M.
- Recoja los espectros ESI-IM-MS de iones negativos y positivos de la solución PA de 10 ppm durante 10 minutos cada uno.
4. Preparación de la valoración de iones metálicos de muestras de amb
- Siga los pasos descritos en los pasos 3.1–3.5.
- Pipet 200.0 l de la amb de 0,125 ml en un vial de 1,7 ml, diluir con 500,0 l de agua DI y mezclar la solución a fondo.
- Ajuste el pH de la muestra a pH a 9,0 agregando 80 ml de la solución de hidróxido de amonio de 0,010 M.
- Añadir 28 ml de la solución de iones metálicos de 0,125 mM para dar 0,14 equivalentes molares del ion metálico, añadir agua DI para hacer el volumen final de la muestra 1,00 ml, mezclar a fondo, y permitir que la muestra se equilibre durante 10 minutos a RT.
- Con una jeringa de nariz contundente, tome 500 ml de la muestra y recoja los espectros esI-IM-MS de iones negativos y positivos durante 5 minutos cada uno. Utilice los 500 ml restantes de la muestra para registrar su pH final utilizando un electrodo de micro pH calibrado.
- Repita los pasos 4.2–4.5, mientras modifica el paso 4.3 para agregar un volumen apropiado de la solución de iones metálicos de 0.125 mM para dar equivalentes molares de 0.28, 0.42, 0.56, 0.70, 0.84, 0.98, 1.12, 1.26 o 1.40.
- Recoja los espectros IM-MS de iones negativos y positivos de la solución PA de 10 ppm durante 10 minutos cada uno.
5. Análisis de los datos de valoración del pH ESI-IM-MS
- A partir de los espectros IM-MS identifican qué especies cargadas de ambs están presentes emparejándolas con sus patrones teóricos de isótopos m/z.
- Abra MassLynx y haga clic en Cromatogram para abrir la ventana de Cromatograma.
- Vaya al menú Archivo y Abrir para localizar y abrir el archivo de datos IM-MS.
- Extraiga el espectro IM-MS haciendo clic con el botón derecho y arrastrando a través del cromatograma y liberando. Se abrirá la ventana de espectro que muestra el espectro IM-MS.
- En la ventana de espectro, haga clic en Herramientas y modelo isótopo. En la ventana de modelado de isótopos, ingrese la fórmula molecular de la especie amb, marque la casilla Mostrar iones cargados e ingrese el estado de carga. Haga clic en Aceptar.
- Repita para identificar todas las especies en el espectro IM-MS y registre su rango de isótopos m/z.
- Para cada especie amb, separe cualquier especie de m/z coincidente y extraiga sus distribuciones de tiempo de llegada (ATD) utilizando sus patrones de isótopos m/z para identificarlas.
- En MassLynx haga clic en DriftScope para abrir el programa. En DriftScope haga clic en Archivo y Abrir para localizar y abrir el archivo de datos IM-MS.
- Utilice el ratón y el botón izquierdo para acercar el patrón de isótopos m/z de la especie amb.
- Utilice la herramienta Selección y el botón izquierdo del ratón para seleccionar el patrón de isótopos. Haga clic en el botón Aceptar selección actual.
- Para separar cualquier especie de m/z coincidente, utilice la herramienta Selección y el botón izquierdo del ratón para seleccionar el patrón de tiempo ATD alineado con el patrón de isótopos de la especie amb. Haga clic en el botón Aceptar selección actual.
- Para exportar el ATD, vaya a Archivo . Exportar a MassLynx, a continuación, seleccione Conservar tiempo de deriva y guardar el archivo en la carpeta adecuada.
- Determinar el centroide del ATD e integrar el área bajo la curva ATD como una medida de la población de especies.
- En la ventana Chromatogram de MassLynx abra el archivo exportado guardado. Haga clic en Proceso de la página de trabajo de la página de trabajo Integre desde el menú. Marque la casilla ApexTrack Peak Integration (Integración de picos de ApexTrack) y haga clic en Aceptar.
- Registre el ATD centroide (tA)y el área integrada como se muestra en la ventana Decrotograma. Repita el proceso para todos los archivos de datos amb y PA IM-MS guardados.
- Utilice el ATD integrado para todas las especies de amb extraídas de los iones positivos o negativos en cada punto de valoración para normalizarla a una escala porcentual relativa.
- Introduzca las identidades de la especie amb y su ATD integrado en cada pH en una hoja de cálculo.
- Para cada pH, utilice la suma de los ATD integrados para normalizar el ATD del amb individual a una escala porcentual.
- Trazar las intensidades porcentuales de cada especie amb frente al pH en un gráfico para mostrar cómo la población de cada especie varía en función del pH.
6. Secciones transversales de colisión
- Usando una hoja de cálculo, convierta los CCS (o) de PA negativo25,26 y27 iones positivos medidos en gas tampón He28 a CCS corregido(c) usando la Ecuación 1 a continuación, donde: z - carga iónda; e c - carga de electrones (1.602-10-19 C); m N 2 - masa de N2 gas (Da); y mion á masa iónda. 29
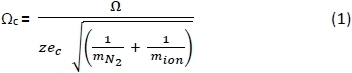
- Convierta los tiempos de llegada promedio (tA) de los calibradores PA y las especies amb en tiempos de deriva (tD) utilizando la Ecuación 2 a continuación, donde: c - el coeficiente de retardo de ciclo de trabajo mejorado (1.41), y m/z es la carga masiva del ion péptido.

- Trazar el tD de los calibradores PA frente a suc. A continuación, utilizando un ajuste de regresión de mínimos cuadrados de la Ecuación 3 que se muestra a continuación, determine los valores A' y B, donde: A' es la corrección para los parámetros de temperatura, presión y campo eléctrico; y B compensa el efecto no lineal del dispositivo IM.

- Usando estos valores A' y B y el valor centroide tD del ATD de los ambs determinan suc usando la Ecuación 3 y su - usando la Ecuación 1. Este método proporciona CCS para las especies de péptidos con errores absolutos estimados de alrededor de 2%25,26,27.
7. Métodos computacionales
- Utilice el nivel de teoría B3LYP/LanL2DZ, que comprende las funciones híbridas Becke de 3 parámetros30 y la base de reclamación establecen31 y los potenciales de núcleo de electrones32,33,34 para localizar conformadores optimizados para geometría para todos los tipos posibles de coordinaciones de la especie m/z amb observada35.
NOTA: Para obtener más información sobre cómo crear y enviar cálculos, consulte el uso de GaussView en Archivo complementario. - Compare la energía libre pronosticada de cada uno de los conformadores y calcule sus CCS teóricos utilizando el método Lennard-Jones (LJ) a escala ióndel programa Sigma36.
- De los conformadores de energía libre más bajos determinan qué conformador exhibe el LJ CCS que está de acuerdo con el IM-MS medido CCS para identificar la estructura terciaria y el tipo de coordinación para los conformadores observados en el experimento.
Resultados
Encuadernación metálica de amb1
El estudio IM-MS20 de amb1 (Figura 1A) mostró que tanto los iones de cobre como los de zinc se unen a amb1 de manera dependiente del pH(Figura 2). Sin embargo, el cobre y el zinc se unen a amb1 a través de diferentes mecanismos de reacción en diferentes sitios de coordinación. Por ejemplo, la adición de Cu(II) a amb...
Discusión
Pasos críticos: conservación de comportamientos en fase de solución para su examen a través de ESI-IM-MS
Se deben utilizar ajustes instrumentales nativos ESI que conserven la estequiometría de los péptidos, el estado de carga y la estructura de conformación. Para condiciones nativas, las condiciones en la fuente ESI tales como los voltajes del cono, las temperaturas y los flujos de gas tienen que ser optimizados. Además, las presiones y voltajes en la fuente, la trampa, la movilidad iónica y ...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este material se basa en el trabajo apoyado por la National Science Foundation bajo 1764436, el apoyo a instrumentos NSF (MRI-0821247), Welch Foundation (T-0014) y recursos informáticos del Departamento de Energía (TX-W-20090427-0004-50) y L3 Communications . Agradecemos al grupo Bower de la Universidad de California - Santa Bárbara por compartir el programa Sigma y Ayobami Ilesanmi por demostrar la técnica en el video.
Materiales
| Name | Company | Catalog Number | Comments |
| acetonitrile HPLC-grade | Fisher Scientific (www.Fishersci.com) | A998SK-4 | |
| ammonium hydroxide (trace metal grade) | Fisher Scientific (www.Fishersci.com) | A512-P500 | |
| cobalt(II) chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 255599-5G | |
| copper(II) chloride 99.999% | Sigma-Aldrich (www.sigmaaldrich.com) | 203149-10G | |
| copper(II) nitrate hydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 229636-5G | |
| designed amb1,2,3,4,5,6,7 peptides | Neo BioLab (neobiolab.com) | designed peptides were synthized by order | |
| designed amb5B,C,D,E,F peptides | PepmicCo (www.pepmic.com) | designed peptides were synthized by order | |
| Driftscope 2.1 software program | Waters (www.waters.com) | software analysis program | |
| Freeze-dried, purified, Cu(I)-free mb-OB3b | cultured and isolated in the lab of Dr. DongWon Choi (Biology Department, Texas A&M-Commerce) | ||
| glacial acetic acid (Optima grade) | Fisher Scientific (www.Fishersci.com) | A465-250 | |
| Iron(III) Chloride Anhydrous 98%+ | Alfa Aesar (www.alfa.com) | 12357-09 | |
| lead(II) nitrate ACS grade | Avantor (www.avantormaterials.com) | 128545-50G | |
| manganese(II) chloride tetrahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203734-5G | |
| MassLynx 4.1 | Waters (www.waters.com) | software analysis program | |
| nickel chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203866-5G | |
| poly-DL-alanine | Sigma-Aldrich (www.sigmaaldrich.com) | P9003-25MG | |
| silver nitrate 99.9%+ | Alfa Aesar (www.alfa.com) | 11414-06 | |
| Waters Synapt G1 HDMS | Waters (www.waters.com) | quadrupole - ion mobility- time-of-flight mass spectrometer | |
| zinc chloride anhydrous | Alfa Aesar (www.alfa.com) | A16281 |
Referencias
- Dudev, T., Lim, C. Competition among Metal Ions for Protein Binding Sites: Determinants of Metal Ion Selectivity in Proteins. Chemical Reviews. 114 (1), 538-556 (2014).
- Sovago, I., Kallay, C., Varnagy, K. Peptides as complexing agents: Factors influencing the structure and thermodynamic stability of peptide complexes. Coordination Chemistry Reviews. 256 (19-20), 2225-2233 (2012).
- Sóvágó, I., Várnagy, K., Lihi, N., Grenács, &. #. 1. 9. 3. ;. Coordinating properties of peptides containing histidyl residues. Coordination Chemistry Reviews. 327, 43-54 (2016).
- Rubino, J. T., Franz, K. J. Coordination chemistry of copper proteins: How nature handles a toxic cargo for essential function. Journal of Inorganic Biochemistry. 107 (1), 129-143 (2012).
- Robinson, N. J., Winge, D. R. Copper Metallochaperones . Annual Review of Biochemistry. 79, 537-562 (2010).
- Scheiber, I. F., Mercer, J. F. B., Dringen, R. Metabolism and functions of copper in brain. Progress in Neurobiology. 116, 33-57 (2014).
- Tisato, F., Marzano, C., Porchia, M., Pellei, M., Santini, C. Copper in Diseases and Treatments, and Copper-Based Anticancer Strategies. Medicinal Research Reviews. 30 (4), 708-749 (2010).
- Millhauser, G. L. Copper and the prion protein: Methods, structures, function, and disease. Annual Review of Physical Chemistry. 58, 299-320 (2007).
- Arena, G., Pappalardo, G., Sovago, I., Rizzarelli, E. Copper(II) interaction with amyloid-beta: Affinity and speciation. Coordination Chemistry Reviews. 256 (1-2), 3-12 (2012).
- Kim, H. J., et al. Methanobactin, a copper-acquisition compound from methane-oxidizing bacteria. Science. 305 (5690), 1612-1615 (2004).
- Di Spirito, A. A., et al. Methanobactin and the link between copper and bacterial methane oxidation. Microbiology Molecular Biology Reviews. 80 (2), 387-409 (2016).
- Kenney, G. E., Rosenzweig, A. C. Chemistry and biology of the copper chelator methanobactin. ACS Chemical Biology. 7 (2), 260-268 (2012).
- Summer, K. H., et al. The biogenic methanobactin is an effective chelator for copper in a rat model for Wilson disease. Journal of Trace Elements in Medicine and Biology. 25 (1), 36-41 (2011).
- Hachmoeller, O., et al. Investigating the influence of standard staining procedures on the copper distribution and concentration in Wilson's disease liver samples by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 44, 71-75 (2017).
- Hachmoeller, O., et al. Spatial investigation of the elemental distribution in Wilson's disease liver after D-penicillamine treatment by LA-ICP-MS. Journal of Trace Elements in Medicine and Biology. 44, 26-31 (2017).
- Hachmoeller, O., et al. Element bioimaging of liver needle biopsy specimens from patients with Wilson's disease by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 35, 97-102 (2016).
- Mueller, J. C., Lichtmannegger, J., Zischka, H., Sperling, M., Karst, U. High spatial resolution LA-ICP-MS demonstrates massive liver copper depletion in Wilson disease rats upon Methanobactin treatment. Journal of Trace Elements in Medicine and Biology. 49, 119-127 (2018).
- Choi, D. W., et al. Spectral and thermodynamic properties of Ag(I), Au(III), Cd(II), Co(II), Fe(III), Hg(II), Mn(II), Ni(II), Pb(II), U(IV), and Zn(II) binding by methanobactin from Methylosinus trichosporium OB3b. Journal of Inorganic Biochemistry. 100, 2150-2161 (2006).
- McCabe, J. W., Vangala, R., Angel, L. A. Binding Selectivity of Methanobactin from Methylosinus trichosporium OB3b for Copper(I), Silver(I), Zinc(II), Nickel(II), Cobalt(II), Manganese(II), Lead(II), and Iron(II). Journal of the American Society of Mass Spectrometry. 28, 2588-2601 (2017).
- Sesham, R., et al. The pH dependent Cu(II) and Zn(II) binding behavior of an analog methanobactin peptide. European Journal of Mass Spectrometry. 19 (6), 463-473 (2013).
- Wagoner, S. M., et al. The multiple conformational charge states of zinc(II) coordination by 2His-2Cys oligopeptide investigated by ion mobility - mass spectrometry, density functional theory and theoretical collision cross sections. Journal of Mass Spectrom. 51 (12), 1120-1129 (2016).
- Bandow, N. L., et al. Isolation of methanobactin from the spent media of methane-oxidizing bacteria. Methods in Enzymology. 495, 259-269 (2011).
- Choi, D. W., et al. Spectral and thermodynamic properties of methanobactin from γ-proteobacterial methane oxidizing bacteria: a case for copper competition on a molecular level. Journal of Inorganic Biochemistry. 104 (12), 1240-1247 (2010).
- Pringle, S. D., et al. An investigation of the mobility separation of some peptide and protein ions using a new hybrid quadrupole/travelling wave IMS/oa-ToF instrument. International Journal of Mass Spectrometry. 261 (1), 1-12 (2007).
- Forsythe, J. G., et al. Collision cross section calibrants for negative ion mode traveling wave ion mobility-mass spectrometry. Analyst. 14 (20), 6853-6861 (2015).
- Allen, S. J., Giles, K., Gilbert, T., Bush, M. F. Ion mobility mass spectrometry of peptide, protein, and protein complex ions using a radio-frequency confining drift cell. Analyst. 141 (3), 884-891 (2016).
- Bush, M. F., Campuzano, I. D. G., Robinson, C. V. Ion Mobility Mass Spectrometry of Peptide Ions: Effects of Drift Gas and Calibration Strategies. Analytical Chemistry. 84 (16), 7124-7130 (2012).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Smith, D. P., et al. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. European Journal of Mass Spectrometry. 15 (2), 113-130 (2009).
- Becke, A. D. Density-functional thermochemistry. III. The role of exact exchange. Journal of Chemical Physics. 98 (7), 5648-5652 (1993).
- Dunning, T. H., Hay, P. J. Gaussian basis sets for molecular calculations. Modern Theoretical Chemistry. 3, 1-27 (1977).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for potassium to gold including the outermost core orbitals. Journal of Chemical Physics. 82 (1), 299-310 (1985).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for the transition metal atoms scandium to mercury. Journal of Chemical Physics. 82 (1), 270-283 (1985).
- Wadt, W. R., Hay, P. J. Ab initio effective core potentials for molecular calculations. Potentials for main group elements sodium to bismuth. Journal of Chemical Physics. 82 (1), 284-298 (1985).
- . Gaussian 09, Revision C.01. Gaussian, Inc. , (2012).
- Wyttenbach, T., von Helden, G., Batka, J. J., Carlat, D., Bowers, M. T. Effect of the long-range potential on ion mobility measurements. Journal of the American Society of Mass Spectrometry. 8 (3), 275-282 (1997).
- Choi, D., et al. Redox activity and multiple copper(I) coordination of 2His-2Cys oligopeptide. Journal of Mass Spectrometry. 50 (2), 316-325 (2015).
- Rigo, A., et al. Interaction of copper with cysteine: stability of cuprous complexes and catalytic role of cupric ions in anaerobic thiol oxidation. Journal of Inorganic Biochemistry. 98 (9), 1495-1501 (2004).
- Vytla, Y., Angel, L. A. Applying Ion Mobility-Mass Spectrometry Techniques for Explicitly Identifying the Products of Cu(II) Reactions of 2His-2Cys Motif Peptides. Analytical Chemistry. 88 (22), 10925-10932 (2016).
- Choi, D., Sesham, R., Kim, Y., Angel, L. A. Analysis of methanobactin from Methylosinus trichosporium OB3b via ion mobility mass spectrometry. European Journal of Mass Spectrometry. 18 (6), 509-520 (2012).
- Martell, A. E., Motekaitis, R. J. NIST Standard Reference Database 46. Institute of Standards and Technology. , (2001).
- Pesch, M. L., Christl, I., Hoffmann, M., Kraemer, S. M., Kretzschmar, R. Copper complexation of methanobactin isolated from Methylosinus trichosporium OB3b: pH-dependent speciation and modeling. Journal of Inorganic Biochemistry. 116, 55-62 (2012).
- Amin, E. A., Truhlar, D. G. Zn Coordination Chemistry: Development of Benchmark Suites for Geometries, Dipole Moments, and Bond Dissociation Energies and Their Use To Test and Validate Density Functionals and Molecular Orbital Theory. Journal of Chemical Theory and Computation. 4 (1), 75-85 (2008).
- Sorkin, A., Truhlar, D. G., Amin, E. A. Energies, Geometries, and Charge Distributions of Zn Molecules, Clusters, and Biocenters from Coupled Cluster, Density Functional, and Neglect of Diatomic Differential Overlap Models. Journal of Chemical Theory and Computation. 5 (5), 1254-1265 (2009).
- Lillo, V., Galan-Mascaros, J. R. Transition metal complexes with oligopeptides: single crystals and crystal structures. Dalton Transactions. 43 (26), 9821-9833 (2014).
- Choutko, A., van Gunsteren, W. F. Conformational Preferences of a beta-Octapeptide as Function of Solvent and Force-Field Parameters. Helvetica Chimica Acta. 96 (2), 189-200 (2013).
- Angel, L. A. Study of metal ion labeling of the conformational and charge states of lysozyme by ion mobility mass spectrometry. European Journal of Mass Spectrometry. 17 (3), 207-215 (2011).
- Kelso, C., Rojas, J. D., Furlan, R. L. A., Padilla, G., Beck, J. L. Characterisation of anthracyclines from a cosmomycin D-producing species of Streptomyces by collisionally-activated dissociation and ion mobility mass spectrometry. European Journal of Mass Spectrometry. 15 (2), 73-81 (2009).
- El Ghazouani, A., et al. Copper-binding properties and structures of methanobactins from Methylosinus trichosporium OB3b. Inorganic Chemistry. 50 (4), 1378-1391 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados