È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Tecniche di spettrometria ione Mobilità-Massa per determinare la struttura e i meccanismi del riconoscimento degli ioni metallici e dell'attività di redox degli oligopeptidi di metal linciazione
In questo articolo
Riepilogo
La spettrometria di massa io-masse ionica e le tecniche di modellazione molecolare possono caratterizzare le prestazioni selettive di chelatura metallica di peptidi progettati e la metanobattina peptide legante al rame. Lo sviluppo di nuove classi di peptidi chelanti metallici contribuirà a portare alle terapie per le malattie associate allo squilibrio degli ioni metallici.
Abstract
La ionizzazione elettrospray (ESI) può trasferire un peptide a queous-fase o complesso di peptidi alla fase gassosa conservando la sua massa, carica complessiva, interazioni di legame metallico, e forma conformante. L'accoppiamento dell'ESI con la spettrometria di massa iosataria (IM-MS) fornisce una tecnica strumentale che consente di misurazione simultanea della massa di carico (m/z) di un peptide e della sezione trasversale di collisione (CCS) che si riferiscono alla sua stoichiometria, allo stato di e forma conformazionale. La carica complessiva di un complesso di peptidi è controllata dalla protonazione di 1) siti acidi e di base del peptide e 2) lo stato di ossidazione degli ioni metallici. Pertanto, lo stato di carica generale di un complesso è una funzione del pH della soluzione che influisce sull'affinità di legame ios metallico peptidi. Per le analisi ESI-IM-MS, le soluzioni per gli ioni di peptidi e metalli sono preparate da soluzioni acquose, con il pH regolato con acido acetico diluito aqueso o idrossido di ammonio. Ciò consente di determinare la dipendenza da pH e la selettività degli ioni metallici per un peptide specifico. Inoltre, l'm/z e il CCS di un complesso di peptidi possono essere utilizzati con la modellazione molecolare B3LYP/LanL2D per discernere i siti di legame del coordinamento degli ioni metallici e della struttura terziaria del complesso. I risultati mostrano come ESI-IM-MS possa caratterizzare le prestazioni di chelating selettivo di una serie di peptidi alternativi di metanobatteriteina e confrontarli con la metanobattina peptide legante in rame.
Introduzione
Gli ioni di rame e zinco sono essenziali per gli organismi viventi e cruciali per i processi tra cui la protezione ossidativa, la crescita dei tessuti, la respirazione, il colesterolo, il metabolismo del glucosio e la lettura del genoma1. Per abilitare queste funzioni, gruppi come il tiolati di Cys, imidazole del Suo2,3, (più raramente) thioether di methionina, e carboxylate di Glu e Asp incorporano selettivamente i metalli come cofattori nei siti attivi di enzimi in metallo. La somiglianza di questi gruppi di coordinamento solleva una domanda intrigante riguardo al modo in cui i ligandi Dei Suoi e dei Cys incorporano selettivamente cu(I/II) o n(II) per garantire il corretto funzionamento.
L'associazione selettiva è spesso realizzata mediante l'acquisizione e la tratta di peptidi, che controllano le concentrazioni di ioni di Cu(I/II)4. Cu(I/II) è altamente reattivo e causa danni ossidativi o legame avventuroso agli enzimi, quindi la sua concentrazione libera è strettamente regolata da chaperones in rame e proteine che regolano il rame che lo trasportano in modo sicuro in varie posizioni nella cellula e strettamente controllare la sua omeostasi5,6. L'interruzione del metabolismo del rame o dell'omeostasi è direttamente implicata nella malattia di Menkes e Wilson7 così come i tumori7 e disturbi neurali, come il prione8 e il morbo di Alzheimer9.
La malattia di Wilson è associata all'aumento dei livelli di rame negli occhi, nel fegato e nelle sezioni del cervello, dove le reazioni di redox di Cu(I/II) producono specie reattive dell'ossigeno, causando degenerazione epatolenticola e neurologica. Le terapie di chelatazione esistenti sono la piccola penicillamina e trietilenetetramina. In alternativa, i peptidi metanoobatteri di rame metatonobatteri (mb)10,11 presentano un potenziale terapeutico a causa della loro elevata affinità legante per Cu(I)12. Quando il metanobatteritina (mb-OB3b) del metilosino trichosporium OB3b è stato studiato in un modello animale della malattia di Wilson, il rame è stato efficacemente rimosso dal fegato ed escreto attraverso la bile13. Gli esperimenti in vitro hanno confermato che mb-OB3b potrebbe masticare il rame dal metallothionein di rame contenuto nel citosol epatico13. Le tecniche di imaging della spettrometria di massa al plasma accoppiato induttivamente hanno studiato la distribuzione spaziale del rame nei campioni epatici della malattia di Wilson14,15,16 e hanno dimostrato che mb-OB3b rimuove il rame con brevi periodi di trattamento di soli 8 giorni17.
L'mb-OB3b si legherà anche con altri ioni metallici, tra cui Ag(I), Au(III), Pb(II), Mn(II), Co(II), Fe(II), Ni(II) e n(II)18,19. Il concorso per il sito di rilegatura fisiologica Cu(I) è esposto da Ag(I) perché può spostando Cu(I) dal complesso mb-OB3b, con Ag(I) e Ni(II) che mostrano anche un legame irreversibile a Mb che non può essere spostato da Cu(I)19. Recentemente, una serie di oligopeptidi alternativi di metanobatterica (amb) con il motivo di rilegatura 2His-2Cys sono stati studiati20,21, e le loro proprietà di rilegatura di N (II) e Cu(I/II) sono state studiate. Le loro sequenze di amminoacidi primarie sono simili, e contengono tutte il motivo 2His-2Cys, Pro e un N-terminus acetilato. Essi differiscono principalmente da mb-OB3b perché il motivo 2His-2Cys sostituisce i due siti di rilegatura enethiol oxazolone di mb-OB3b.
La ionizzazione dell'elettrospray accoppiata con la spettrometria di massa-massa a ioni (ESI-IM-MS) fornisce una potente tecnica strumentale per determinare le proprietà di legame dei metalli dei peptidi perché misura la loro massa a carica (m/z) e la collisione (CCS) conservando la massa, la carica e la forma conformazionale dalla fase della soluzione. I m/z e ccS si riferiscono alla stochichiometria dei peptidi, allo stato di protonazione e alla forma conformazionale. La stoichiometria è determinata perché l'identità e il numero di ogni elemento presente nella specie sono esplicitamente identificati. La carica complessiva del complesso di peptidi si riferisce allo stato di protonazione dei siti acidi e di base e allo stato di ossidazione degli ioni metallici. Il CCS fornisce informazioni sulla forma conformazionale del complesso di peptidi perché misura la dimensione media rotazionale che si riferisce alla struttura terziaria del complesso. Lo stato di carica generale del complesso è anche una funzione di pH e colpisce l'affinità di legame degli ioni metallici del peptide perché i siti di base deprotonizzati o acidi come il carboxyl, His, Cys e Tyr sono anche i potenziali siti di legame per gli ioni metallici. Per le analisi, il peptide e gli ioni metallici vengono preparati in soluzioni acquose con il pH regolato da acido acetico aluto o idrossido di ammonio. Ciò consente di determinare la dipendenza da pH e la selettività degli ioni metallici per il peptide. Inoltre, l'm/z e la CCS determinati da ESI-IM-MS possono essere utilizzati con la modellazione molecolare B3LYP/LanL2D per scoprire il tipo di coordinamento degli ioni metallici e la struttura terziaria del complesso. I risultati mostrati in questo articolo rivelano come ESI-IM-MS può caratterizzare le prestazioni di chelating selettivo di una serie di amb peptidi e confrontarli con il peptide mb-OB3b di legame di rame.
Protocollo
1. Preparazione dei reagenti
- Cultura Methylosinus trichosporium OB3b, isolare il Mb-OB3b senza Cu(I)18,22,23, congelare il campione e conservare a -80 gradi centigradi fino all'uso.
- Sintetizzare i peptidi dell'amb (>98% purezza per amb1, amb2, amb4; >70% purezza per amb7), liofilizzare i campioni, e conservarli a -80 gradi centigradi fino all'uso.
- Acquisto >98% di colcalo di purezza(II), cloruro di cobalto (II), cloruro nickle(II), cloruro di rame(II), nitrato di rame(II), nitrato d'argento(II), cloruro di zinco(II), cloruro di ferro(III) e cloruro di piombo(II).
- Acquistare i polimeri poli-DL-alanina utilizzati come calibronti per misurare le sezioni trasversali di collisione della specie amb e l'idrossido di ammonio superiore o superiore, l'acido acettico glaciale e l'acetonitrile.
2. Preparazione della soluzione di stock
-
Soluzione stock di peptidi
- Pesare con precisione, utilizzando almeno tre figure significative, la massa di 10.0–20.0 mg del mb-OB3b o amb in una fiala di plastica 1.7 mL.
NOTA: La massa pesata dovrebbe produrre 12,5 mM o 1,25 mM, a seconda della solubilità del peptide, quando vengono aggiunti 1,00 mL di acqua deionizzata (DI). - Utilizzando un pipet, aggiungere 1,00 mL di acqua deionizzata (>17,8 cm) al campione di peptide pesato per produrre la soluzione da 12,5 mM o 1,25 mM. Mettere il tappo in modo sicuro e mescolare accuratamente con almeno 20 inversioni.
- L'utilizzo di un micropipet eroga 50,0 aliquote dal campione di peptide in fiale da 1,5 mL etichettate individualmente e le conserva a -80 gradi centigradi fino all'uso.
- Pesare con precisione, utilizzando almeno tre figure significative, la massa di 10.0–20.0 mg del mb-OB3b o amb in una fiala di plastica 1.7 mL.
-
Soluzioni per ioni metallici
- Pesare con precisione, utilizzando almeno tre figure significative, la massa di 10.0–30.0 mg del cloruro metallico o nitrato d'argento in una fiala da 1,7 mL.
NOTA: La massa pesata deve produrre 125 mM quando viene aggiunto 1,00 mL di acqua DI. - Aggiungere 1,00 mL di acqua DI al campione di metallo pesato nella fiala da 1,7 mL per produrre la soluzione da 125 mM. Mettere il tappo in modo sicuro e mescolare accuratamente con almeno 20 inversioni.
- Pesare con precisione, utilizzando almeno tre figure significative, la massa di 10.0–30.0 mg del cloruro metallico o nitrato d'argento in una fiala da 1,7 mL.
- Soluzioni per il serbatoio di idossido di ammonio: preparare una soluzione di acido acetico di 1,0 M diluindo 57 -L della soluzione di acido acetico del 99,5% con acqua DI ad un volume finale di 1,00 mL. Preparare una soluzione di idrossido di ammonio di 1,0 m diluindo 90 -L della soluzione di idrossido di ammonio del 21% con acqua DI ad un volume finale di 1,00 mL. Fare due diluizioni successive di ogni soluzione prendendo 100 L delle soluzioni da 1,0 M per preparare soluzioni di acido acetico e idrossidi di ammonio da 0,10 M e 0,010 M.
- Soluzione di stock poli-DL-alanina: preparare la poli-DL-alanina (PA) pesando 1,0 mg di PA e sciogliendo in 1,0 mL di acqua DI per dare 1.000 ppm. Mescolare accuratamente. Utilizzando un micropipet, erogare aliquote di 50,0 gradi centigradi, e metterle in una fiala da 1,7 mL e conservarle a -80 gradi centigradi.
3. Analisi della spettrometria della mobilità-massa agli ioni elettrospruzzi
- Pulire accuratamente il tubo d'ingresso ESI e l'ago capillare con circa 500 sl di acido acetico glaciale di 0,1 m, idrossido di ammonio da 0,1 m e infine acqua DI.
- Scongelare una 50,0 l'una della soluzione di 1.000 ppm PA e diluirla con 450 l di acqua DI per dare un 100 ppm PA. Pipet 100.0 l di questa soluzione e diluirlo a 1,00 mL con 500 l di acqua DI e 500 L di acetonitrile per dare la soluzione PA 10 ppm.
- Raccogliere gli spettri IM-MS negativi e positivi della soluzione PA da 10 ppm per 10 min ciascuno utilizzando condizioni ESI-IM-MS native, come descritto nella sezione di discussione.
- Scongelare una soluzione di riserva di 12,5 mm o 1,25 m amb m e fare diluizioni successive con acqua DI per dare una concentrazione finale di 0,125 mM amb. Mescolare accuratamente ogni diluizione.
- Tubo 100.0 ll della soluzione di scorta di ioni metallici da 125 mM, mettere in una fiala da 1,7 mL e diluire a 1,00 mL con acqua DI per dare ioni metallici 12,5 mM. Ripetere con altre due diluizioni successive per dare un'ultima concentrazione di 0,125 mm di ioni metallici. Mescolare accuratamente ogni diluizione.
- Pipet 200.0 L dell'amb di 0,125 mM in una fiala da 1,7 ml, diluire con 500 l di acqua DI e mescolare accuratamente la soluzione.
- Regolare il pH del campione a 3,0 aggiungendo 50 , luna di 1,0 M soluzione di acido acetico.
- Aggiungere al campione regolato il pH di 200,0 l ioni metallici. Aggiungere l'acqua DI per produrre un volume finale di 1,00 mL del campione, mescolare accuratamente e lasciare che il campione equilidestina per 10 min a RT.
- Utilizzando una siringa naso smussata prendere 500 L del campione e raccogliere lo spettro ionegativo e positivo ES-IM-MS per 5 min ciascuno. Utilizzare i restanti 500 l del campione per registrare il suo pH finale utilizzando un elettrodo micro pH calibrato.
- Ripetere i passaggi da 3,6 a 3,9, modificando il passaggio 3.7 per regolare il pH a 4.0, 5.0, 6.0, 7.0, 8.0, 9.0 o 10.0 aggiungendo nuovi volumi delle soluzioni 0.010 M, 0.10 M o 1.0 M acetico acido o ammonito hydroxide.
- Raccogliere gli spettri ESI-IM-MS negativi e positivi della soluzione PA da 10 ppm per 10 min ciascuno.
4. Preparazione della tizione degli ioni metallici dei campioni di amb
- Seguire i passaggi descritti nei passaggi da 3.1 a 3.5.
- Pipet 200.0 l of the 0.125 mM amb in a 1.7 mL flebo, diluire con 500,0 l di acqua DI e mescolare accuratamente la soluzione.
- Regolare il pH del campione in modo da ottenere 9,0 ordini di 9,0 m aggiungendo 80 -L della soluzione di idrossido di ammonio da 0,010 m.
- Aggiungere 28 l della soluzione di ioni metallici da 0,125 mm per dare 0,14 equivalenti molare dello ione metallico, aggiungere acqua DI per fare il volume finale del campione 1,00 mL, mescolare accuratamente e lasciare che il campione equiligua 10 min a RT.
- Utilizzando una siringa naso smussata prendere 500 L del campione e raccogliere gli spettri ioni negativi e positivi ESI-IM-MS per 5 min ciascuno. Utilizzare i restanti 500 l del campione per registrare il suo pH finale utilizzando un elettrodo micro pH calibrato.
- Ripetere i passaggi da 4,2 a 4,5, modificando il passaggio 4.3 per aggiungere un volume appropriato della soluzione iosciolataria da 0,125 mM per fornire equivalente a 0,28, 0,42, 0,56, 0,70, 0,84, 0,98, 1,12, 1,26 o 1,40 molari.
- Raccogliere gli spettri IM-MS negativi e positivi della soluzione PA da 10 ppm per 10 min ciascuno.
5. Analisi dei dati di titola di PH ESI-IM-MS
- Dagli spettri IM-MS identificano quali specie cariche di ambs sono presenti abbinandole ai loro modelli teorici di isotopi m/z.
- Aprite MassLynx e fate clic su Cromatogramma per aprire la finestra Chromatogram.
- Vai al menu File e Apri per individuare e aprire il file di dati IM-MS.
- Estrarre lo spettro IM-MS facendo clic con il pulsante destro del mouse e trascinando lo cromatogramma e rilasciandolo. Si aprirà la finestra dello spettro che mostra lo spettro IM-MS.
- Nella finestra spettro, fare clic su Strumenti e modello Isotope. Nella finestra di modellazione degli isotopi, immettere la formula molecolare della specie amb, selezionare la casella Mostra ione caricato e immettere lo stato di carica. Fare clic su OK.
- Ripetere l'operazione per identificare tutte le specie nello spettro IM-MS e registrare la loro gamma di isotopi m/z.
- Per ogni specie di amb, separare tutte le specie casuali m/z ed estrarre le loro distribuzioni del tempo di arrivo (ATD) utilizzando i loro modelli di isotopi m/z per identificarli.
- In MassLynx fare clic su DriftScope per aprire il programma. In DriftScope fare clic su File e Apri per individuare e aprire il file di dati IM-MS.
- Utilizzare il mouse e clic sinistro per ingrandire il modello isotope m/ z della specie amb.
- Usate lo strumento selezione e il pulsante sinistro del mouse per selezionare il motivo isotopo. Fare clic sul pulsante Accetta selezione corrente.
- Per separare qualsiasi specie casuale di m/z, utilizzare lo strumento Selezione e il pulsante sinistro del mouse per selezionare il modello ATD allineato al tempo con il modello isotopo della specie di amb. Fare clic sul pulsante Accetta selezione corrente.
- Per esportare l'ATD, passare a File Esporta in MassLynx, quindi selezionare Mantieni tempo di deriva e salvare il file nella cartella appropriata.
- Determinare il centroide dell'ATD e integrare l'area sotto la curva ATD come misura della popolazione di specie.
- Nella finestra Cromatogramma di MassLynx aprire il file esportato salvato. Fare clic su Processo . Integrare dal menu. Selezionare la casella ApexTrack Peak Integration (Integrazione picco apexTrack) e fare clic su OK.
- Registrare l'ATD centroide (tA) e l'area integrata come mostrato nella finestra del cromatogramma. Ripetere l'operazione per tutti i file di dati amb e PA IM-MS salvati.
- Utilizzare l'ATD integrato per tutte le specie di amb estratti degli ioni positivi o negativi in ogni punto di titore per normalizzare a una scala percentuale relativa.
- Inserisci le identità delle specie di amb e il loro ATD integrato ad ogni pH in un foglio di calcolo.
- Per ogni pH, utilizzare la somma degli ATD integrati per normalizzare l'ATD del singolo amb in una scala percentuale.
- Tracciare le intensità percentuali di ogni specie di amb rispetto al pH in un grafico per mostrare come la popolazione di ogni specie varia in funzione del pH.
6. Sezioni trasversali di collisione
- Utilizzando un foglio di calcolo, convertire il CCSs (z) di PA negativo25,26 e positivo27 ioni misurati in Egli buffer gas28 per correggere CCS(z)utilizzando l'equazione 1 di seguito, dove: z - carica iola; e c - carica elettronica (1.602-10-19 C); m N 2 - massa di Gas N2 (Da); e mione - massa iola. 29 del 22 221
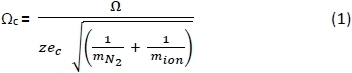
- Convertire gli orari medi di arrivo (tA) dei calibranti PA e delle specie amb in tempi di deriva (tD)utilizzando l'equazione 2 di seguito, dove: c - il coefficiente di ritardo del ciclo di servizio migliorato (1,41), e m/z è la massa a carica dello ione peptide.

- Tracciare i calibranti PA' tD vs. Quindi, utilizzando un adattamento di regressione dei minimi quadrati dell'equazione 3 mostrato di seguito, determinare i valori A' e B, dove: A' è la correzione per i parametri di temperatura, pressione e campo elettrico; e B compensa l'effetto non lineare del dispositivo iM.

- Utilizzando questi valori A' e B e il valore centroide tD dall'ATD degli ambs determinare il loroc utilizzando l'equazione 3 e il loro s utilizzando l'equazione 1. Questo metodo fornisce CCS per le specie di peptidi con errori assoluti stimati di circa 2%25,26,27.
7. Metodi computazionali
- Utilizzare il livello di teoria B3LYP/LanL2D, che comprende il Becke 3-parametro funzioni ibride30 e la base Dunning set31 e potenziali nucleo elettronico32,33,34 per individuare conformatori ottimizzati per la geometria per tutti i possibili tipi di coordinazioni delle specie m/z amb osservate35.
NOTA: per informazioni dettagliate su come creare e inviare calcoli, fare riferimento all'utilizzo di GaussView in File supplementare. - Confrontare l'energia libera prevista di ciascuno dei conformatori e calcolare le loro CS teoriche utilizzando il metodo Lennard-Jones (LJ) in scala iosciata dal programma Sigma36.
- Dai conformatori di energia libera più bassi determinano quale conformatore presenta la LJ CCS che concorda con l'IM-MS misurata CCS per identificare la struttura terziaria e il tipo di coordinamento per i conformatori osservati nell'esperimento.
Risultati
Rilegatura metallica di amb1
Lo studio IM-MS20 di amb1 (Figura 1A) ha mostrato che sia gli ioni di rame che quelli di zinco sono legati all'amb1 in modo dipendente dal pH (Figura 2). Tuttavia, rame e zinco legati all'amb1 attraverso diversi meccanismi di reazione in diversi siti di coordinamento. Ad esempio, l'aggiunta di Cu(II) all'amb1 ha provoca...
Discussione
Passaggi critici: conservazione dei comportamenti della fase della soluzione per l'esame tramite ESI-IM-MS
Devono essere utilizzate impostazioni strumentali ESI native che conservino la stoichiometria dei peptidi, lo stato di carica e la struttura conformazionale. Per le condizioni native, le condizioni nella sorgente ESI, come le tensioni del cono, le temperature e i flussi di gas, devono essere ottimizzate. Inoltre, le pressioni e le tensioni nella sorgente, nella trappola, nella mobilità degli ion...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo materiale si basa sul lavoro supportato dalla National Science Foundation nell'ambito del 1764436, dal supporto dello strumento NSF (MRI-0821247), dalla Welch Foundation (T-0014) e dalle risorse informatiche del Department of Energy (TX-W-20090427-0004-50) e delle comunicazioni l3 . Ringraziamo il gruppo Bower dell'Università della California - Santa Barbara per aver condiviso il programma Sigma e Ayobami Ilesanmi per aver dimostrato la tecnica nel video.
Materiali
| Name | Company | Catalog Number | Comments |
| acetonitrile HPLC-grade | Fisher Scientific (www.Fishersci.com) | A998SK-4 | |
| ammonium hydroxide (trace metal grade) | Fisher Scientific (www.Fishersci.com) | A512-P500 | |
| cobalt(II) chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 255599-5G | |
| copper(II) chloride 99.999% | Sigma-Aldrich (www.sigmaaldrich.com) | 203149-10G | |
| copper(II) nitrate hydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 229636-5G | |
| designed amb1,2,3,4,5,6,7 peptides | Neo BioLab (neobiolab.com) | designed peptides were synthized by order | |
| designed amb5B,C,D,E,F peptides | PepmicCo (www.pepmic.com) | designed peptides were synthized by order | |
| Driftscope 2.1 software program | Waters (www.waters.com) | software analysis program | |
| Freeze-dried, purified, Cu(I)-free mb-OB3b | cultured and isolated in the lab of Dr. DongWon Choi (Biology Department, Texas A&M-Commerce) | ||
| glacial acetic acid (Optima grade) | Fisher Scientific (www.Fishersci.com) | A465-250 | |
| Iron(III) Chloride Anhydrous 98%+ | Alfa Aesar (www.alfa.com) | 12357-09 | |
| lead(II) nitrate ACS grade | Avantor (www.avantormaterials.com) | 128545-50G | |
| manganese(II) chloride tetrahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203734-5G | |
| MassLynx 4.1 | Waters (www.waters.com) | software analysis program | |
| nickel chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203866-5G | |
| poly-DL-alanine | Sigma-Aldrich (www.sigmaaldrich.com) | P9003-25MG | |
| silver nitrate 99.9%+ | Alfa Aesar (www.alfa.com) | 11414-06 | |
| Waters Synapt G1 HDMS | Waters (www.waters.com) | quadrupole - ion mobility- time-of-flight mass spectrometer | |
| zinc chloride anhydrous | Alfa Aesar (www.alfa.com) | A16281 |
Riferimenti
- Dudev, T., Lim, C. Competition among Metal Ions for Protein Binding Sites: Determinants of Metal Ion Selectivity in Proteins. Chemical Reviews. 114 (1), 538-556 (2014).
- Sovago, I., Kallay, C., Varnagy, K. Peptides as complexing agents: Factors influencing the structure and thermodynamic stability of peptide complexes. Coordination Chemistry Reviews. 256 (19-20), 2225-2233 (2012).
- Sóvágó, I., Várnagy, K., Lihi, N., Grenács, &. #. 1. 9. 3. ;. Coordinating properties of peptides containing histidyl residues. Coordination Chemistry Reviews. 327, 43-54 (2016).
- Rubino, J. T., Franz, K. J. Coordination chemistry of copper proteins: How nature handles a toxic cargo for essential function. Journal of Inorganic Biochemistry. 107 (1), 129-143 (2012).
- Robinson, N. J., Winge, D. R. Copper Metallochaperones . Annual Review of Biochemistry. 79, 537-562 (2010).
- Scheiber, I. F., Mercer, J. F. B., Dringen, R. Metabolism and functions of copper in brain. Progress in Neurobiology. 116, 33-57 (2014).
- Tisato, F., Marzano, C., Porchia, M., Pellei, M., Santini, C. Copper in Diseases and Treatments, and Copper-Based Anticancer Strategies. Medicinal Research Reviews. 30 (4), 708-749 (2010).
- Millhauser, G. L. Copper and the prion protein: Methods, structures, function, and disease. Annual Review of Physical Chemistry. 58, 299-320 (2007).
- Arena, G., Pappalardo, G., Sovago, I., Rizzarelli, E. Copper(II) interaction with amyloid-beta: Affinity and speciation. Coordination Chemistry Reviews. 256 (1-2), 3-12 (2012).
- Kim, H. J., et al. Methanobactin, a copper-acquisition compound from methane-oxidizing bacteria. Science. 305 (5690), 1612-1615 (2004).
- Di Spirito, A. A., et al. Methanobactin and the link between copper and bacterial methane oxidation. Microbiology Molecular Biology Reviews. 80 (2), 387-409 (2016).
- Kenney, G. E., Rosenzweig, A. C. Chemistry and biology of the copper chelator methanobactin. ACS Chemical Biology. 7 (2), 260-268 (2012).
- Summer, K. H., et al. The biogenic methanobactin is an effective chelator for copper in a rat model for Wilson disease. Journal of Trace Elements in Medicine and Biology. 25 (1), 36-41 (2011).
- Hachmoeller, O., et al. Investigating the influence of standard staining procedures on the copper distribution and concentration in Wilson's disease liver samples by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 44, 71-75 (2017).
- Hachmoeller, O., et al. Spatial investigation of the elemental distribution in Wilson's disease liver after D-penicillamine treatment by LA-ICP-MS. Journal of Trace Elements in Medicine and Biology. 44, 26-31 (2017).
- Hachmoeller, O., et al. Element bioimaging of liver needle biopsy specimens from patients with Wilson's disease by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 35, 97-102 (2016).
- Mueller, J. C., Lichtmannegger, J., Zischka, H., Sperling, M., Karst, U. High spatial resolution LA-ICP-MS demonstrates massive liver copper depletion in Wilson disease rats upon Methanobactin treatment. Journal of Trace Elements in Medicine and Biology. 49, 119-127 (2018).
- Choi, D. W., et al. Spectral and thermodynamic properties of Ag(I), Au(III), Cd(II), Co(II), Fe(III), Hg(II), Mn(II), Ni(II), Pb(II), U(IV), and Zn(II) binding by methanobactin from Methylosinus trichosporium OB3b. Journal of Inorganic Biochemistry. 100, 2150-2161 (2006).
- McCabe, J. W., Vangala, R., Angel, L. A. Binding Selectivity of Methanobactin from Methylosinus trichosporium OB3b for Copper(I), Silver(I), Zinc(II), Nickel(II), Cobalt(II), Manganese(II), Lead(II), and Iron(II). Journal of the American Society of Mass Spectrometry. 28, 2588-2601 (2017).
- Sesham, R., et al. The pH dependent Cu(II) and Zn(II) binding behavior of an analog methanobactin peptide. European Journal of Mass Spectrometry. 19 (6), 463-473 (2013).
- Wagoner, S. M., et al. The multiple conformational charge states of zinc(II) coordination by 2His-2Cys oligopeptide investigated by ion mobility - mass spectrometry, density functional theory and theoretical collision cross sections. Journal of Mass Spectrom. 51 (12), 1120-1129 (2016).
- Bandow, N. L., et al. Isolation of methanobactin from the spent media of methane-oxidizing bacteria. Methods in Enzymology. 495, 259-269 (2011).
- Choi, D. W., et al. Spectral and thermodynamic properties of methanobactin from γ-proteobacterial methane oxidizing bacteria: a case for copper competition on a molecular level. Journal of Inorganic Biochemistry. 104 (12), 1240-1247 (2010).
- Pringle, S. D., et al. An investigation of the mobility separation of some peptide and protein ions using a new hybrid quadrupole/travelling wave IMS/oa-ToF instrument. International Journal of Mass Spectrometry. 261 (1), 1-12 (2007).
- Forsythe, J. G., et al. Collision cross section calibrants for negative ion mode traveling wave ion mobility-mass spectrometry. Analyst. 14 (20), 6853-6861 (2015).
- Allen, S. J., Giles, K., Gilbert, T., Bush, M. F. Ion mobility mass spectrometry of peptide, protein, and protein complex ions using a radio-frequency confining drift cell. Analyst. 141 (3), 884-891 (2016).
- Bush, M. F., Campuzano, I. D. G., Robinson, C. V. Ion Mobility Mass Spectrometry of Peptide Ions: Effects of Drift Gas and Calibration Strategies. Analytical Chemistry. 84 (16), 7124-7130 (2012).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Smith, D. P., et al. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. European Journal of Mass Spectrometry. 15 (2), 113-130 (2009).
- Becke, A. D. Density-functional thermochemistry. III. The role of exact exchange. Journal of Chemical Physics. 98 (7), 5648-5652 (1993).
- Dunning, T. H., Hay, P. J. Gaussian basis sets for molecular calculations. Modern Theoretical Chemistry. 3, 1-27 (1977).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for potassium to gold including the outermost core orbitals. Journal of Chemical Physics. 82 (1), 299-310 (1985).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for the transition metal atoms scandium to mercury. Journal of Chemical Physics. 82 (1), 270-283 (1985).
- Wadt, W. R., Hay, P. J. Ab initio effective core potentials for molecular calculations. Potentials for main group elements sodium to bismuth. Journal of Chemical Physics. 82 (1), 284-298 (1985).
- . Gaussian 09, Revision C.01. Gaussian, Inc. , (2012).
- Wyttenbach, T., von Helden, G., Batka, J. J., Carlat, D., Bowers, M. T. Effect of the long-range potential on ion mobility measurements. Journal of the American Society of Mass Spectrometry. 8 (3), 275-282 (1997).
- Choi, D., et al. Redox activity and multiple copper(I) coordination of 2His-2Cys oligopeptide. Journal of Mass Spectrometry. 50 (2), 316-325 (2015).
- Rigo, A., et al. Interaction of copper with cysteine: stability of cuprous complexes and catalytic role of cupric ions in anaerobic thiol oxidation. Journal of Inorganic Biochemistry. 98 (9), 1495-1501 (2004).
- Vytla, Y., Angel, L. A. Applying Ion Mobility-Mass Spectrometry Techniques for Explicitly Identifying the Products of Cu(II) Reactions of 2His-2Cys Motif Peptides. Analytical Chemistry. 88 (22), 10925-10932 (2016).
- Choi, D., Sesham, R., Kim, Y., Angel, L. A. Analysis of methanobactin from Methylosinus trichosporium OB3b via ion mobility mass spectrometry. European Journal of Mass Spectrometry. 18 (6), 509-520 (2012).
- Martell, A. E., Motekaitis, R. J. NIST Standard Reference Database 46. Institute of Standards and Technology. , (2001).
- Pesch, M. L., Christl, I., Hoffmann, M., Kraemer, S. M., Kretzschmar, R. Copper complexation of methanobactin isolated from Methylosinus trichosporium OB3b: pH-dependent speciation and modeling. Journal of Inorganic Biochemistry. 116, 55-62 (2012).
- Amin, E. A., Truhlar, D. G. Zn Coordination Chemistry: Development of Benchmark Suites for Geometries, Dipole Moments, and Bond Dissociation Energies and Their Use To Test and Validate Density Functionals and Molecular Orbital Theory. Journal of Chemical Theory and Computation. 4 (1), 75-85 (2008).
- Sorkin, A., Truhlar, D. G., Amin, E. A. Energies, Geometries, and Charge Distributions of Zn Molecules, Clusters, and Biocenters from Coupled Cluster, Density Functional, and Neglect of Diatomic Differential Overlap Models. Journal of Chemical Theory and Computation. 5 (5), 1254-1265 (2009).
- Lillo, V., Galan-Mascaros, J. R. Transition metal complexes with oligopeptides: single crystals and crystal structures. Dalton Transactions. 43 (26), 9821-9833 (2014).
- Choutko, A., van Gunsteren, W. F. Conformational Preferences of a beta-Octapeptide as Function of Solvent and Force-Field Parameters. Helvetica Chimica Acta. 96 (2), 189-200 (2013).
- Angel, L. A. Study of metal ion labeling of the conformational and charge states of lysozyme by ion mobility mass spectrometry. European Journal of Mass Spectrometry. 17 (3), 207-215 (2011).
- Kelso, C., Rojas, J. D., Furlan, R. L. A., Padilla, G., Beck, J. L. Characterisation of anthracyclines from a cosmomycin D-producing species of Streptomyces by collisionally-activated dissociation and ion mobility mass spectrometry. European Journal of Mass Spectrometry. 15 (2), 73-81 (2009).
- El Ghazouani, A., et al. Copper-binding properties and structures of methanobactins from Methylosinus trichosporium OB3b. Inorganic Chemistry. 50 (4), 1378-1391 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon