Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Techniques de spectrométrie de mobilité-masse d'ion pour déterminer la structure et les mécanismes de la reconnaissance d'ion de métal et de l'activité de Redox des Oligopeptides de liaison de métal
Dans cet article
Résumé
Les techniques de spectrométrie et de modélisation moléculaire de la mobilité des ions peuvent caractériser la performance sélective du chélage métallique des peptides de liaison métallique conçus et la méthanoacttine du peptide liant le cuivre. Le développement de nouvelles classes de peptides de chélage de métal aidera à mener aux thérapies pour des maladies liées à l'déséquilibre d'ion métallique.
Résumé
L'ionisation par électrospray (ESI) peut transférer un peptide ou un peptide en phase aqueuse à la phase gazeuse tout en conservant sa masse, sa charge globale, ses interactions métal-liantes et sa forme conformationnelle. L'accouplement de l'ESI avec la spectrométrie de masse de mobilité ionique (IM-MS) fournit une technique instrumentale qui permet de mesurer simultanément la section transversale de masse à charge (m/z) d'un peptide (m/z) qui se rapportent à son état de stoichiométrie, de protonation, et la forme conformationnelle. La charge globale d'un complexe de peptides est contrôlée par la protonation de 1) les sites acides et de base du peptide et 2) l'état d'oxydation de l'ion métallique(s). Par conséquent, l'état de charge global d'un complexe est fonction du pH de la solution qui affecte l'affinité de liaison d'ion métallique de peptides. Pour les analyses ESI-IM-MS, les solutions peptidiques et d'ions métalliques sont préparées à partir de solutions aqueuses seulement, le pH ayant été ajusté avec de l'acide acétique aqueux dilué ou de l'hydroxyde d'ammonium. Cela permet de déterminer la dépendance au pH et la sélectivité des ions métalliques pour un peptide spécifique. En outre, le m/z et le CSC d'un complexe de peptides peuvent être utilisés avec la modélisation moléculaire B3LYP/LanL2DZ pour discerner les sites de liaison de la coordination des ions métalliques et de la structure tertiaire du complexe. Les résultats montrent comment ESI-IM-MS peut caractériser la performance sélective de chélage d'un ensemble de peptides alternatifs de méthanobactine et les comparer à la méthanobacttine de peptide cuivre-contraignante.
Introduction
Les ions de cuivre et de zinc sont essentiels pour les organismes vivants et cruciaux pour les processus comprenant la protection oxydative, la croissance de tissu, la respiration, le cholestérol, le métabolisme de glucose, et la lecture de génome1. Pour activer ces fonctions, des groupes tels que le thiolate de Cys, l'imidazole de ses2,3, (plus rarement) thioether de méthionine, et carboxylate de Glu et Asp incorporer sélectivement des métaux comme cofacteurs dans les sites actifs de metalloenzymes. La similitude de ces groupes de coordination soulève une question intrigante quant à la façon dont les ligands His et Cys intègrent sélectivement Soit Cu(I/II) ou Zn(II) pour assurer un bon fonctionnement.
La liaison sélective est souvent accomplie par l'acquisition et le trafic de peptides, qui contrôlent les concentrations d'ionz(II) ou Cu(I/II)4. Cu(I/II) est très réactif et cause des dommages oxydatifs ou une liaison aventurière aux enzymes, de sorte que sa concentration libre est étroitement réglementée par les chaperons de cuivre et les protéines régulatrices du cuivre qui le transportent en toute sécurité à divers endroits dans la cellule et étroitement contrôler son homéostasie5,6. La perturbation du métabolisme du cuivre ou de l'homéostasie est directement impliquée dans Menkes et la maladie de Wilson7 ainsi que les cancers7 et les troubles neuronaux, tels que le prion8 et la maladie d'Alzheimer9.
La maladie de Wilson est associée à des niveaux accrus de cuivre dans les yeux, le foie et les sections du cerveau, où les réactions redox de Cu(I/II) produit des espèces réactives d'oxygène, causant la dégénérescence hépatolentique et neurologique. Les thérapies existantes de chélation sont la petite pénicillamine d'acide aminé de thiol et la triéthylenetetramine. Alternativement, les peptides de cuivre-acquisition méthanotrophiques methanobactin (mb)10,11 présentent un potentiel thérapeutique en raison de leur affinité de liaison élevée pour Cu(I)12. Lorsque la méthanobacttine (mb-OB3b) de Methylosinus trichosporium OB3b a été étudiée dans un modèle animal de la maladie de Wilson, le cuivre a été efficacement retiré du foie et excrété par la bile13. Des expériences in vitro ont confirmé que mb-OB3b pourrait chélater le cuivre de la métallothionein de cuivre contenue dans le cytosol de foie13. L'ablation au laser a inductivement couplé les techniques d'imagerie par spectrométrie de masse plasmatique ont étudié la distribution spatiale du cuivre dans les échantillons de foie de la maladie de Wilson14,15,16 et ont montré que mb-OB3b enlève le cuivre avec de courtes périodes de traitement de seulement 8 jours17.
Le mb-OB3b se liera également avec d'autres ions métalliques, y compris Ag(I), Au(III), Pb(II), Mn(II), Co(II), Fe(II), Ni(II), et Zn(II)18,19. La concurrence pour le site de liaison physiologique Cu(I) est exposée par Ag(I) parce qu'il peut déplacer Cu(I) du complexe mb-OB3b, avec à la fois Ag(I) et Ni(II) montrant également une liaison irréversible à Mb qui ne peut pas être déplacé par Cu(I)19. Récemment, une série d'oligopeptides alternatifs de méthanobacttine (amb) avec le motif de liaison 2His-2Cys ont été étudiés20,21, et leurs propriétés de liaison Zn(II) et Cu(I/II) caractérisées. Leurs séquences primaires d'acides aminés sont similaires, et elles contiennent toutes le motif 2His-2Cys, Pro et un n-terminus acétylé. Ils diffèrent principalement de mb-OB3b parce que le motif 2His-2Cys remplace les deux sites de liaison enethiol oxazolone de mb-OB3b.
L'ionisation de l'électrospray couplée à la spectrométrie de masse de mobilité ionique (ESI-IM-MS) fournit une technique instrumentale puissante pour déterminer les propriétés métalliques-contraignantes des peptides parce qu'elle mesure leur masse-à-charge (m/z) et collision section transversale (CSC) tout en conservant leur masse, leur charge et leur forme conformationnelle à partir de la phase de solution. Le m/z et le CSC se rapportent à la stoichiométrie des peptides, à l'état de protonation et à la forme conformationnelle. La stoichiométrie est déterminée parce que l'identité et le nombre de chaque élément présent dans l'espèce sont explicitement identifiés. La charge globale du complexe de peptide se rapporte à l'état de protonation des emplacements acides et de base et à l'état d'oxydation de l'ion métal(s). Le CSC donne des informations sur la forme conformationnelle du complexe peptidique parce qu'il mesure la taille moyenne par rotation qui se rapporte à la structure tertiaire du complexe. L'état de charge global du complexe est également fonction du pH et affecte l'affinité de liaison d'ion métallique du peptide parce que les sites de base ou acides déprotonated tels que le carboxyl, his, Cys et Tyr sont également les emplacements de liaison potentiels pour l'ion métallique. Pour les analyses, le peptide et l'ion métallique sont préparés en solutions aqueuses avec le pH ajusté par l'acide acétique aqueous dilué ou l'hydroxyde d'ammonium. Cela permet de déterminer la dépendance au pH et la sélectivité des ions métalliques pour le peptide. En outre, le m/z et le CSC déterminés par ESI-IM-MS peuvent être utilisés avec la modélisation moléculaire B3LYP/LanL2DZ pour découvrir le type de coordination des ions métalliques et la structure tertiaire du complexe. Les résultats montrés dans cet article révèlent comment ESI-IM-MS peut caractériser la performance sélective de chélage d'un ensemble de peptides d'amb et les comparer au mb-OB3b de peptide cuivre-contraignant.
Protocole
1. Préparation des réactifs
- Culture Methylosinus trichosporium OB3b, isoler le Cu(I)-free mb-OB3b18,22,23, lyophiliser l'échantillon et stocker à -80 oC jusqu'à utilisation.
- Synthétiser les peptides amb (98% de pureté pour amb1, amb2, amb4; '70% pureté pour amb7), lyophiliser les échantillons, et les stocker à -80 oC jusqu'à l'utilisation.
- Achat de manganèse de pureté de la pureté de la pureté de 98 % (II) chlorure, cobalt(II) chlorure, nickle(II) chlorure, cuivre(II) chlorure, cuivre(II) nitrate, argent (I) nitrate, zinc(II) chlorure, fer(III) chlorure et plomb(II) chlorure.
- Achetez les polymères poly-DL-alanine utilisés comme calibrants pour mesurer les sections transversales de collision de l'espèce en amb et l'hydroxyde d'ammonium de qualité HPLC ou plus élevé, l'acide acétique glaciaire et l'acétonitrile.
2. Préparation de la solution de stock
-
Solution d'actions Peptide
- Peser avec précision, en utilisant au moins trois chiffres significatifs, la masse de 10,0 à 20,0 mg du mb-OB3b ou amb dans un flacon en plastique de 1,7 ml.
REMARQUE : La masse pesée doit produire soit 12,5 mM, soit 1,25 mM, selon la solubilité du peptide, lorsque 1,00 mL d'eau déionisée (DI) est ajoutée. - À l'aide d'un pipet, ajouter 1,00 ml d'eau déionisée (17,8 cm) à l'échantillon de peptide pesé pour produire soit la solution de 12,5 mM, soit 1,25 mM. Placez le bouchon solidement et mélangez-le bien avec au moins 20 inversions.
- À l'aide d'un micropipet, dispenser 50,0 aliquots de l'échantillon de peptide dans des flacons de 1,5 ml étiquetés individuellement et les stocker à -80 oC jusqu'à leur utilisation.
- Peser avec précision, en utilisant au moins trois chiffres significatifs, la masse de 10,0 à 20,0 mg du mb-OB3b ou amb dans un flacon en plastique de 1,7 ml.
-
Solutions de stock d'ion siens métalliques
- Peser avec précision, à l'aide d'au moins trois chiffres significatifs, la masse de 10,0 à 30,0 mg de chlorure de métal ou de nitrate d'argent dans un flacon de 1,7 ml.
REMARQUE : La masse pesée devrait donner 125 mm lorsque 1,00 ml d'eau DI est ajouté. - Ajouter les 1,00 ml d'eau DI à l'échantillon de métal pesé dans le flacon de 1,7 ml pour donner la solution de 125 mm. Placez le bouchon solidement et mélangez-le bien avec au moins 20 inversions.
- Peser avec précision, à l'aide d'au moins trois chiffres significatifs, la masse de 10,0 à 30,0 mg de chlorure de métal ou de nitrate d'argent dans un flacon de 1,7 ml.
- Solutions de stock d'hydroxyde d'ammonium: préparer une solution d'acide acétique de 1,0 M en diluant 57 L de la solution d'acide acétique de 99,5% avec de l'eau DI à un volume final de 1,00 ml. Préparer une solution d'hydroxyde d'ammonium de 1,0 M en diluant 90 l de la solution d'hydroxyde d'ammonium de 21 % avec de l'eau DI à un volume final de 1,00 ml. Faire deux dilutions successives de chaque solution en prenant 100 L des solutions de 1,0 M pour préparer des solutions d'acide acétique et d'hydroxyde d'ammonium de 0,10 M et 0,010 M.
- Solution de stock poly-DL-alanine: préparer la poly-DL-alanine (PA) en pesant 1,0 mg de PA et en se dissolvant dans 1,0 mL d'eau DI pour donner 1 000 ppm. Mélanger. À l'aide d'un micropipet, dispenser 50,0 aliquots ll et placez-les dans un flacon de 1,7 ml et entreposez-les à -80 oC.
3. Analyse de spectrométrie mobilité-masse Electrospray-ion
- Nettoyez soigneusement le tube d'entrée et le capillaire d'aiguille d'ESI avec environ 500 l d'acide acétique glaciaire de 0,1 M, 0,1 M d'hydroxyde d'ammonium et, enfin, de l'eau DI.
- Décongeler un aliquot de 50,0 L de la solution de stock PA de 1 000 ppm et le diluer avec 450 l'eau DI pour donner une PA de 100 ppm. Pipet 100,0 L de cette solution et le diluer à 1,00 ml avec 500 l d'eau DI et 500 l d'acétonitrile pour donner 10 ppm solution PA.
- Recueillir les spectres ion négatifs et positifs IM-MS de la solution PA de 10 ppm pendant 10 min chacun en utilisant des conditions ESI-IM-MS natives telles que décrites dans la section de discussion.
- Décongeler un aliquot de 50,0 L de la solution de stock de 12,5 mM ou 1,25 mmb et faire des dilutions successives avec de l'eau DI pour donner une concentration finale de 0,125 mm amb. Bien mélanger chaque dilution.
- Pipet 100,0 L de la solution de stock d'ion métallique de 125 mM, placer dans un flacon de 1,7 ml et diluer à 1,00 mL avec de l'eau DI pour donner 12,5 mM d'ion métallique. Répétez l'opération avec deux dilutions successives de plus pour donner une concentration finale d'ion séquestre de 0,125 mM. Bien mélanger chaque dilution.
- Pipet 200,0 L de la 0.125 mm amb dans un flacon de 1,7 ml, diluer avec 500 l d'eau DI, et mélanger la solution à fond.
- Ajustez le pH de l'échantillon à 3,0 en ajoutant 50 L de solution d'acide acétique de 1,0 M.
- Ajouter 200,0 L de l'ion métallique de 0,125 mM à l'échantillon corrigé du pH. Ajouter de l'eau DI pour produire un volume final de 1,00 ml de l'échantillon, bien mélanger et permettre à l'échantillon d'alquer pendant 10 min à RT.
- À l'aide d'une seringue nasale émoussée, prenez 500 l'échantillon et recueillez les spectres ES-IM-MS d'ion négatif et positif pendant 5 min chacun. Utilisez les 500 ll restants de l'échantillon pour enregistrer son pH final à l'aide d'une électrode de micro pH calibrée.
- Répétez les étapes 3.6-3.9, tout en modifiant l'étape 3.7 pour ajuster le pH à 4.0, 5.0, 6.0, 7.0, 8.0, 9.0, ou 10.0 en ajoutant de nouveaux volumes des solutions de 0.010 M, 0.10 M, ou 1.0 M d'acide acétique ou d'hydroxyde d'ammonium.
- Recueillir les spectres eSI-IM-MS ion négatif et positif de la solution PA 10 ppm pour 10 min chacun.
4. Préparation de la titration d'ion métallique des échantillons d'amb
- Suivez les étapes décrites dans les étapes 3.1-3.5.
- Pipet 200,0 L de la 0.125 mM amb dans un flacon de 1,7 ml, diluer avec 500,0 L d'eau DI et mélanger la solution à fond.
- Ajustez le pH de l'échantillon à pH 9,0 en ajoutant 80 L de la solution d'hydroxyde d'ammonium de 0,010 M.
- Ajouter 28 L de la solution d'ions métalliques de 0,125 mM pour donner 0,14 équivalent molaire de l'ion métallique, ajouter de l'eau DI pour faire le volume final de l'échantillon 1,00 ml, bien mélanger, et permettre à l'échantillon d'équilibrer pendant 10 min à RT.
- À l'aide d'une seringue nasale émoussée, prenez 500 l'échantillon et recueillez les spectres ESI-IM-MS négatifs et positifs pendant 5 min chacun. Utilisez les 500 ll restants de l'échantillon pour enregistrer son pH final à l'aide d'une électrode de micro pH calibrée.
- Répéter les étapes 4.2-4.5, tout en modifiant l'étape 4.3 pour ajouter un volume approprié de la solution d'ion métallique de 0,125 mM pour donner soit 0,28, 0,42, 0,56, 0,70, 0,84, 0,98, 1,12, 1,26 ou 1,40 équivalent molaire.
- Recueillir les spectres ion négatif et positif IM-MS de la solution PA 10 ppm pour 10 min chacun.
5. Analyse des données de titration de pH ESI-IM-MS
- À partir des spectres IM-MS, identifier les espèces chargées d'ambs qui sont présentes en les faisant correspondre à leurs modèles théoriques d'isotopes m/z.
- Ouvrez MassLynx et cliquez sur Chromatogram pour ouvrir la fenêtre Chromatogram.
- Rendez-vous dans le menu Fichier et ouvrez pour localiser et ouvrir le fichier de données IM-MS.
- Extraire le spectre IM-MS en cliquant à droite et en faisant glisser à travers le chromatogramme et en libérant. La fenêtre de spectre s'ouvrira montrant le spectre IM-MS.
- Dans la fenêtre du spectre, cliquez sur Le modèle Outils et Isotope. Dans la fenêtre de modélisation des isotopes, entrez la formule moléculaire de l'espèce amb, cochez la boîte d'ions chargées Show et entrez dans l'état de charge. Cliquez OK.
- Répétez l'opération pour identifier toutes les espèces du spectre IM-MS et enregistrer leur aire de répartition des isotopes m/z.
- Pour chaque espèce amb, séparez toutes les espèces m/z fortuites et extrayez leurs distributions d'heures d'arrivée (ATD) en utilisant leurs modèles d'isotopes m/z pour les identifier.
- Dans MassLynx cliquez sur DriftScope pour ouvrir le programme. Dans DriftScope, cliquez sur Fichier et Ouvrez pour localiser et ouvrir le fichier de données IM-MS.
- Utilisez la souris et cliquez à gauche pour zoomer sur le modèle isotopique m/z de l'espèce amb.
- Utilisez l'outil De sélection et le bouton de la souris gauche pour sélectionner le modèle isotopique. Cliquez sur le bouton Acceptez le bouton de sélection actuelle.
- Pour séparer toute espèce m/z fortuite, utilisez l'outil De sélection et le bouton de la souris gauche pour sélectionner l'ATD aligné sur le modèle isotopique de l'espèce en amb. Cliquez sur le bouton Acceptez le bouton de sélection actuelle.
- Pour exporter l'ATD, rendez-vous sur File Exportez vers MassLynx, puis sélectionnez Conserver le temps de dérive et enregistrer le fichier dans le dossier approprié.
- Déterminer le centroïde de l'ATD et intégrer la zone sous la courbe ATD comme mesure de la population d'espèces.
- Dans la fenêtre Chromatogram de MassLynx ouvrez le fichier exporté enregistré. Cliquer sur Le processus Intégrer dans le menu. Vérifiez la case d'intégration ApexTrack Peak et cliquez sur OK.
- Enregistrez le centroïde ATD (tA) et la zone intégrée comme indiqué sur la fenêtre Chromatogram. Répétez l'opération pour tous les fichiers de données amb et PA IM-MS enregistrés.
- Utilisez l'ATD intégré pour toutes les espèces extraites d'amb des ions positifs ou négatifs à chaque point de titration pour normaliser à une échelle de pourcentage relative.
- Entrez l'identité de l'espèce amb et leur ATD intégré à chaque pH dans une feuille de calcul.
- Pour chaque pH, utilisez la somme des DAt intégrés pour normaliser l'ATD de l'amb individuel à une échelle de pourcentage.
- Tracer les intensités pour cent de chaque espèce amb vs pH dans un graphique pour montrer comment la population de chaque espèce varie en fonction du pH.
6. Sections transversales de collision
- À l'aide d'une feuille de calcul, convertir les CSC (MD) de25,26 et27 ions positifs mesurés dans le gaz tamponHe 28 en CPS corrigé(c)en utilisant l'équation 1 ci-dessous, où : z - charge ion; e (en) c charge d'électrons (1,602 à 10-19 C); m (en) N (en) 2 - masse de gaz N2 (Da); et mion - masse ionique. 29 Ans et plus
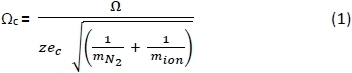
- Convertir les temps d'arrivée moyens (tA) des calibrants PA et des espèces en amb en temps de dérive (tD) en utilisant l'équation 2 ci-dessous, où : c - le coefficient de retard du cycle de service amélioré (1,41), et le m/z est la masse à la charge de l'ion peptidique.

- Terrain de l'AP calibrants' tD vs leurc. Ensuite, à l'aide d'un ajustement de régression de moindre set de l'équation 3 ci-dessous, déterminez les valeurs A' et B, où: A' est la correction pour la température, la pression et les paramètres de champ électrique; et B compense l'effet non linéaire du dispositif de GI.

- L'utilisation de ces valeurs A' et B et la valeur centroïde tD de l'ATD des ambs déterminent leur'c en utilisant l'équation 3 et leur 'en utilisant l'équation 1. Cette méthode fournit des CSC pour les espèces de peptides avec des erreurs absolues estimées d'environ 2%25,26,27.
7. Méthodes de calcul
- Utilisez le niveau de théorie B3LYP/LanL2DZ, composé des fonctions hybrides 3 paramètres Becke30 et de la base Dunning défini31 et des potentiels de noyau d'électrons32,33,34 pour localiser conformateurs optimisés pour tous les types possibles de coordinations de l'espèce m/z amb observée35.
REMARQUE: Pour plus de détails sur la façon de construire et de soumettre des calculs se référer à l'utilisation GaussView dans le fichier supplémentaire. - Comparez l'énergie libre prévue de chacun des conformistes et calculez leurs CSC théoriques à l'aide de la méthode Lennard-Jones (LJ) à l'échelle ionique du programme Sigma36.
- À partir de la plus faible énergie libre, les conformateurs déterminent quel consécration présente le LJ CCS qui est d'accord avec le CSC mesuré par IM-MS pour identifier la structure tertiaire et le type de coordination pour les conformateurs observés dans l'expérience.
Résultats
Fixation métallique d'amb1
L'étude IM-MS20 de l'amb1 (Figure 1A) a montré que les ions de cuivre et de zinc liés à amb1 d'une manière pH-dépendante (figure 2). Cependant, le cuivre et le zinc liés à amb1 par différents mécanismes de réaction à différents sites de coordination. Par exemple, l'ajout de Cu(II) à l'amb1 a entraîné l'ox...
Discussion
Étapes critiques : conservation des comportements en phase de solution pour examen via ESI-IM-MS
Les paramètres instrumentaux eSI indigènes doivent être utilisés pour conserver la stoichiométrie des peptides, l'état de charge et la structure conformationnelle. Pour les conditions indigènes, les conditions dans la source ESI telles que les tensions du cône, les températures et les flux de gaz doivent être optimisées. En outre, les pressions et les tensions dans la source, le piège, la mobi...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce matériel est basé sur les travaux soutenus par la National Science Foundation en vertu de 1764436, le soutien des instruments NSF (MRI-0821247), la Fondation Welch (T-0014), et les ressources informatiques du ministère de l'Énergie (TX-W-20090427-0004-50) et L3 Communications . Nous remercions le groupe Bower de l'Université de Californie - Santa Barbara pour le partage du programme Sigma et Ayobami Ilesanmi pour la démonstration de la technique dans la vidéo.
matériels
| Name | Company | Catalog Number | Comments |
| acetonitrile HPLC-grade | Fisher Scientific (www.Fishersci.com) | A998SK-4 | |
| ammonium hydroxide (trace metal grade) | Fisher Scientific (www.Fishersci.com) | A512-P500 | |
| cobalt(II) chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 255599-5G | |
| copper(II) chloride 99.999% | Sigma-Aldrich (www.sigmaaldrich.com) | 203149-10G | |
| copper(II) nitrate hydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 229636-5G | |
| designed amb1,2,3,4,5,6,7 peptides | Neo BioLab (neobiolab.com) | designed peptides were synthized by order | |
| designed amb5B,C,D,E,F peptides | PepmicCo (www.pepmic.com) | designed peptides were synthized by order | |
| Driftscope 2.1 software program | Waters (www.waters.com) | software analysis program | |
| Freeze-dried, purified, Cu(I)-free mb-OB3b | cultured and isolated in the lab of Dr. DongWon Choi (Biology Department, Texas A&M-Commerce) | ||
| glacial acetic acid (Optima grade) | Fisher Scientific (www.Fishersci.com) | A465-250 | |
| Iron(III) Chloride Anhydrous 98%+ | Alfa Aesar (www.alfa.com) | 12357-09 | |
| lead(II) nitrate ACS grade | Avantor (www.avantormaterials.com) | 128545-50G | |
| manganese(II) chloride tetrahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203734-5G | |
| MassLynx 4.1 | Waters (www.waters.com) | software analysis program | |
| nickel chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203866-5G | |
| poly-DL-alanine | Sigma-Aldrich (www.sigmaaldrich.com) | P9003-25MG | |
| silver nitrate 99.9%+ | Alfa Aesar (www.alfa.com) | 11414-06 | |
| Waters Synapt G1 HDMS | Waters (www.waters.com) | quadrupole - ion mobility- time-of-flight mass spectrometer | |
| zinc chloride anhydrous | Alfa Aesar (www.alfa.com) | A16281 |
Références
- Dudev, T., Lim, C. Competition among Metal Ions for Protein Binding Sites: Determinants of Metal Ion Selectivity in Proteins. Chemical Reviews. 114 (1), 538-556 (2014).
- Sovago, I., Kallay, C., Varnagy, K. Peptides as complexing agents: Factors influencing the structure and thermodynamic stability of peptide complexes. Coordination Chemistry Reviews. 256 (19-20), 2225-2233 (2012).
- Sóvágó, I., Várnagy, K., Lihi, N., Grenács, &. #. 1. 9. 3. ;. Coordinating properties of peptides containing histidyl residues. Coordination Chemistry Reviews. 327, 43-54 (2016).
- Rubino, J. T., Franz, K. J. Coordination chemistry of copper proteins: How nature handles a toxic cargo for essential function. Journal of Inorganic Biochemistry. 107 (1), 129-143 (2012).
- Robinson, N. J., Winge, D. R. Copper Metallochaperones . Annual Review of Biochemistry. 79, 537-562 (2010).
- Scheiber, I. F., Mercer, J. F. B., Dringen, R. Metabolism and functions of copper in brain. Progress in Neurobiology. 116, 33-57 (2014).
- Tisato, F., Marzano, C., Porchia, M., Pellei, M., Santini, C. Copper in Diseases and Treatments, and Copper-Based Anticancer Strategies. Medicinal Research Reviews. 30 (4), 708-749 (2010).
- Millhauser, G. L. Copper and the prion protein: Methods, structures, function, and disease. Annual Review of Physical Chemistry. 58, 299-320 (2007).
- Arena, G., Pappalardo, G., Sovago, I., Rizzarelli, E. Copper(II) interaction with amyloid-beta: Affinity and speciation. Coordination Chemistry Reviews. 256 (1-2), 3-12 (2012).
- Kim, H. J., et al. Methanobactin, a copper-acquisition compound from methane-oxidizing bacteria. Science. 305 (5690), 1612-1615 (2004).
- Di Spirito, A. A., et al. Methanobactin and the link between copper and bacterial methane oxidation. Microbiology Molecular Biology Reviews. 80 (2), 387-409 (2016).
- Kenney, G. E., Rosenzweig, A. C. Chemistry and biology of the copper chelator methanobactin. ACS Chemical Biology. 7 (2), 260-268 (2012).
- Summer, K. H., et al. The biogenic methanobactin is an effective chelator for copper in a rat model for Wilson disease. Journal of Trace Elements in Medicine and Biology. 25 (1), 36-41 (2011).
- Hachmoeller, O., et al. Investigating the influence of standard staining procedures on the copper distribution and concentration in Wilson's disease liver samples by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 44, 71-75 (2017).
- Hachmoeller, O., et al. Spatial investigation of the elemental distribution in Wilson's disease liver after D-penicillamine treatment by LA-ICP-MS. Journal of Trace Elements in Medicine and Biology. 44, 26-31 (2017).
- Hachmoeller, O., et al. Element bioimaging of liver needle biopsy specimens from patients with Wilson's disease by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 35, 97-102 (2016).
- Mueller, J. C., Lichtmannegger, J., Zischka, H., Sperling, M., Karst, U. High spatial resolution LA-ICP-MS demonstrates massive liver copper depletion in Wilson disease rats upon Methanobactin treatment. Journal of Trace Elements in Medicine and Biology. 49, 119-127 (2018).
- Choi, D. W., et al. Spectral and thermodynamic properties of Ag(I), Au(III), Cd(II), Co(II), Fe(III), Hg(II), Mn(II), Ni(II), Pb(II), U(IV), and Zn(II) binding by methanobactin from Methylosinus trichosporium OB3b. Journal of Inorganic Biochemistry. 100, 2150-2161 (2006).
- McCabe, J. W., Vangala, R., Angel, L. A. Binding Selectivity of Methanobactin from Methylosinus trichosporium OB3b for Copper(I), Silver(I), Zinc(II), Nickel(II), Cobalt(II), Manganese(II), Lead(II), and Iron(II). Journal of the American Society of Mass Spectrometry. 28, 2588-2601 (2017).
- Sesham, R., et al. The pH dependent Cu(II) and Zn(II) binding behavior of an analog methanobactin peptide. European Journal of Mass Spectrometry. 19 (6), 463-473 (2013).
- Wagoner, S. M., et al. The multiple conformational charge states of zinc(II) coordination by 2His-2Cys oligopeptide investigated by ion mobility - mass spectrometry, density functional theory and theoretical collision cross sections. Journal of Mass Spectrom. 51 (12), 1120-1129 (2016).
- Bandow, N. L., et al. Isolation of methanobactin from the spent media of methane-oxidizing bacteria. Methods in Enzymology. 495, 259-269 (2011).
- Choi, D. W., et al. Spectral and thermodynamic properties of methanobactin from γ-proteobacterial methane oxidizing bacteria: a case for copper competition on a molecular level. Journal of Inorganic Biochemistry. 104 (12), 1240-1247 (2010).
- Pringle, S. D., et al. An investigation of the mobility separation of some peptide and protein ions using a new hybrid quadrupole/travelling wave IMS/oa-ToF instrument. International Journal of Mass Spectrometry. 261 (1), 1-12 (2007).
- Forsythe, J. G., et al. Collision cross section calibrants for negative ion mode traveling wave ion mobility-mass spectrometry. Analyst. 14 (20), 6853-6861 (2015).
- Allen, S. J., Giles, K., Gilbert, T., Bush, M. F. Ion mobility mass spectrometry of peptide, protein, and protein complex ions using a radio-frequency confining drift cell. Analyst. 141 (3), 884-891 (2016).
- Bush, M. F., Campuzano, I. D. G., Robinson, C. V. Ion Mobility Mass Spectrometry of Peptide Ions: Effects of Drift Gas and Calibration Strategies. Analytical Chemistry. 84 (16), 7124-7130 (2012).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Smith, D. P., et al. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. European Journal of Mass Spectrometry. 15 (2), 113-130 (2009).
- Becke, A. D. Density-functional thermochemistry. III. The role of exact exchange. Journal of Chemical Physics. 98 (7), 5648-5652 (1993).
- Dunning, T. H., Hay, P. J. Gaussian basis sets for molecular calculations. Modern Theoretical Chemistry. 3, 1-27 (1977).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for potassium to gold including the outermost core orbitals. Journal of Chemical Physics. 82 (1), 299-310 (1985).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for the transition metal atoms scandium to mercury. Journal of Chemical Physics. 82 (1), 270-283 (1985).
- Wadt, W. R., Hay, P. J. Ab initio effective core potentials for molecular calculations. Potentials for main group elements sodium to bismuth. Journal of Chemical Physics. 82 (1), 284-298 (1985).
- . Gaussian 09, Revision C.01. Gaussian, Inc. , (2012).
- Wyttenbach, T., von Helden, G., Batka, J. J., Carlat, D., Bowers, M. T. Effect of the long-range potential on ion mobility measurements. Journal of the American Society of Mass Spectrometry. 8 (3), 275-282 (1997).
- Choi, D., et al. Redox activity and multiple copper(I) coordination of 2His-2Cys oligopeptide. Journal of Mass Spectrometry. 50 (2), 316-325 (2015).
- Rigo, A., et al. Interaction of copper with cysteine: stability of cuprous complexes and catalytic role of cupric ions in anaerobic thiol oxidation. Journal of Inorganic Biochemistry. 98 (9), 1495-1501 (2004).
- Vytla, Y., Angel, L. A. Applying Ion Mobility-Mass Spectrometry Techniques for Explicitly Identifying the Products of Cu(II) Reactions of 2His-2Cys Motif Peptides. Analytical Chemistry. 88 (22), 10925-10932 (2016).
- Choi, D., Sesham, R., Kim, Y., Angel, L. A. Analysis of methanobactin from Methylosinus trichosporium OB3b via ion mobility mass spectrometry. European Journal of Mass Spectrometry. 18 (6), 509-520 (2012).
- Martell, A. E., Motekaitis, R. J. NIST Standard Reference Database 46. Institute of Standards and Technology. , (2001).
- Pesch, M. L., Christl, I., Hoffmann, M., Kraemer, S. M., Kretzschmar, R. Copper complexation of methanobactin isolated from Methylosinus trichosporium OB3b: pH-dependent speciation and modeling. Journal of Inorganic Biochemistry. 116, 55-62 (2012).
- Amin, E. A., Truhlar, D. G. Zn Coordination Chemistry: Development of Benchmark Suites for Geometries, Dipole Moments, and Bond Dissociation Energies and Their Use To Test and Validate Density Functionals and Molecular Orbital Theory. Journal of Chemical Theory and Computation. 4 (1), 75-85 (2008).
- Sorkin, A., Truhlar, D. G., Amin, E. A. Energies, Geometries, and Charge Distributions of Zn Molecules, Clusters, and Biocenters from Coupled Cluster, Density Functional, and Neglect of Diatomic Differential Overlap Models. Journal of Chemical Theory and Computation. 5 (5), 1254-1265 (2009).
- Lillo, V., Galan-Mascaros, J. R. Transition metal complexes with oligopeptides: single crystals and crystal structures. Dalton Transactions. 43 (26), 9821-9833 (2014).
- Choutko, A., van Gunsteren, W. F. Conformational Preferences of a beta-Octapeptide as Function of Solvent and Force-Field Parameters. Helvetica Chimica Acta. 96 (2), 189-200 (2013).
- Angel, L. A. Study of metal ion labeling of the conformational and charge states of lysozyme by ion mobility mass spectrometry. European Journal of Mass Spectrometry. 17 (3), 207-215 (2011).
- Kelso, C., Rojas, J. D., Furlan, R. L. A., Padilla, G., Beck, J. L. Characterisation of anthracyclines from a cosmomycin D-producing species of Streptomyces by collisionally-activated dissociation and ion mobility mass spectrometry. European Journal of Mass Spectrometry. 15 (2), 73-81 (2009).
- El Ghazouani, A., et al. Copper-binding properties and structures of methanobactins from Methylosinus trichosporium OB3b. Inorganic Chemistry. 50 (4), 1378-1391 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon