Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ionen-Mobilitäts-Massenspektrometrie-Techniken zur Bestimmung der Struktur und Mechanismen der Metallionenerkennung und Redox-Aktivität von Metallbindungsoligopeptiden
In diesem Artikel
Zusammenfassung
Ionen-Mobilitäts-Massenspektrometrie und molekulare Modellierungstechniken können die selektive Metallchelating-Performance von entworfenen metallbindenden Peptiden und dem kupferbindenden Peptid-Methanobactin charakterisieren. Die Entwicklung neuer Klassen von Metallchelat-Peptiden wird dazu beitragen, Therapeutika für Krankheiten im Zusammenhang mit Metallionen-Misbalance.
Zusammenfassung
Die Elektrospray-Ionisation (ESI) kann einen wässrigen Peptid- oder Peptidkomplex in die Gasphase übertragen und gleichzeitig seine Masse, Gesamtladung, Metallbindungswechselwirkungen und Konformationsform erhalten. Die Kopplung von ESI mit der Ionenmobilitäts-Massenspektrometrie (IM-MS) bietet eine instrumentelle Technik, die die gleichzeitige Messung des Masse-zu-Lade- (m/z) und des Kollisionsquerschnitts (CCS) eines Peptids ermöglicht, die sich auf seine Stoiximetrie, Protonationszustand, und konforme Form. Die Gesamtladung eines Peptidkomplexes wird durch die Protonierung von 1) den sauren und grundlegenden Stellen des Peptids und 2) dem Oxidationszustand des Metallions(en) gesteuert. Daher ist der Gesamtladungszustand eines Komplexes eine Funktion des pH-Werts der Lösung, die die Peptide MetallionenbindungAffinität beeinflusst. Für ESI-IM-MS-Analysen werden Peptid- und Metallionenlösungen aus wässrigen Lösungen hergestellt, wobei der pH-Wert mit verdünnter wässriger Essigsäure oder Ammoniumhydroxid eingestellt wird. Dadurch können pH-Abhängigkeit und Metallionenselektivität für ein bestimmtes Peptid bestimmt werden. Darüber hinaus können die m/z und CCS eines Peptidkomplexes mit B3LYP/LanL2DZ molekularer Modellierung verwendet werden, um Bindungsstellen der Metallionenkoordination und tertiären Struktur des Komplexes zu erkennen. Die Ergebnisse zeigen, wie ESI-IM-MS die selektive Chelaterfüllung einer Reihe alternativer Methanobactin-Peptide charakterisieren und mit dem kupferbindenden Peptid-Methanobactin vergleichen kann.
Einleitung
Kupfer- und Zinkionen sind für lebende Organismen unerlässlich und entscheidend für Prozesse wie oxidativen Schutz, Gewebewachstum, Atmung, Cholesterin, Glukosestoffwechsel und Genom-Lesung1. Um diese Funktionen zu ermöglichen, integrieren Gruppen wie das Thiolat von Cys, Imidazol seiner2,3, (seltener) Thioether von Methionin und Carboxylat von Glu und Asp selektiv Metalle als Kofaktoren in die aktiven Metalloenzyme. Die Ähnlichkeit dieser Koordinierungsgruppen wirft eine faszinierende Frage auf, wie die Seine und Cys-Liganden selektiv entweder Cu(I/II) oder Zn(II) integrieren, um ein korrektes Funktionieren zu gewährleisten.
Selektive Bindung erfolgt häufig durch Erwerb und Handel Peptide, die Zn(II) oder Cu(I/II) Ionenkonzentrationen steuern4. Cu(I/II) ist sehr reaktiv und verursacht oxidative Schäden oder zufällige Bindung an Enzyme, so dass seine freie Konzentration durch Kupferchaperones und kupferregulierende Proteine streng reguliert wird, die es sicher an verschiedene Stellen in der Zelle und fest transportieren. seine Homöostasekontrollieren 5,6. Störungen des Kupferstoffwechsels oder der Homöostase sind direkt in Menkes und Wilsons Krankheit7 sowieKrebserkrankungen 7 und neuronale Erkrankungen wie Prion8 undAlzheimer-Krankheit 9involviert.
Wilson-Krankheit ist verbunden mit erhöhten Kupferspiegeln in den Augen, Leber und Abschnitten des Gehirns, wo die Redox-Reaktionen von Cu(I/II) reaktive Sauerstoffspezies produzieren, was hepatolenticularundäre und neurologische Degeneration verursacht. Bestehende Chelationstherapien sind die kleine Thiol-Aminosäure Penicillamin und Triethylenetetramine. Alternativ weisen die methanotrophen Kupfer-Akquise-Peptide Methanobactin (mb)10,11 aufgrund ihrer hohen Bindungsaffinität zu Cu(I)12therapeutisches Potenzial auf. Als das Methanobactin (mb-OB3b) von Methylosinus trichosporium OB3b in einem Tiermodell der Wilson-Krankheit untersucht wurde, wurde Kupfer effizient aus der Leber entfernt und über die Galleausgeschieden 13. In-vitro-Experimente bestätigten, dass mb-OB3b das Kupfer aus dem kupfernen Metallothionein, das im Leberzytosolenthalten ist, chelatierenkonnte 13 . Laserablation induktiv gekoppelte Plasma-Massenspektrometrie-Bildgebungstechniken haben die räumliche Verteilung von Kupfer in Wilsons Krankheitsleberproben14,15,16 untersucht und gezeigt, dass mb-OB3b entfernt das Kupfer mit kurzen Behandlungsperioden von nur 8 Tagen17.
Die mb-OB3b bindet auch mit anderen Metallionen, einschließlich Ag(I), Au(III), Pb(II), Mn(II), Co(II), Fe(II), Ni(II) und Zn(II)18,19. Der Wettbewerb um die physiologische Cu(I)-Bindungsstelle wird von Ag(I) ausgestellt, weil er Cu(I) aus dem mb-OB3b-Komplex verdrängen kann, wobei sowohl Ag(I) als auch Ni(II) eine irreversible Bindung an Mb aufweisen, die nicht durch Cu(I)19verdrängt werden kann. Kürzlich wurden eine Reihe alternativer Methanobactin (amb) Oligopeptide mit dem Bindungsmotiv 2His-2Cys20,21, und ihre Zn(II) und Cu(I/II) Bindungseigenschaften charakterisiert. Ihre primären Aminosäuresequenzen sind ähnlich, und sie alle enthalten das 2His-2Cys Motiv, Pro und einen acetylierten N-Terminus. Sie unterscheiden sich hauptsächlich von mb-OB3b, da das 2His-2Cys-Motiv die beiden Enethiol-Oxazolon-Bindungsstellen von mb-OB3b ersetzt.
Die Elektrospray-Ionisierung in Verbindung mit der Ionen-Mobilitäts-Massenspektrometrie (ESI-IM-MS) sorgt für eine leistungsstarke Instrumentaltechnik zur Bestimmung der Metallbindungseigenschaften von Peptiden, da sie deren Massen-Zu-Ladung (m/z) und Kollisionmisst Querschnitt (CCS) unter Beibehaltung ihrer Masse, Ladung und Konformationsform aus der Lösungsphase. Die m/z und CCS beziehen sich auf die Peptide Stoichiometrie, Protonationszustand und Konformationsform. Die Stoichiometrie wird bestimmt, da die Identität und Anzahl jedes elements, das in der Art vorhanden ist, explizit identifiziert wird. Die Gesamtladung des Peptidkomplexes bezieht sich auf den Protonationszustand der sauren und grundlegenden Stellen und den Oxidationszustand der Metallionen. Das CCS gibt Auskunft über die konforme Form des Peptidkomplexes, da es die rotationsgemittelte Größe misst, die sich auf die tertiäre Struktur des Komplexes bezieht. Der Gesamtladungszustand des Komplexes ist auch eine Funktion des pH-Werts und wirkt sich auf die Metallionenbindungsaffinität des Peptids aus, da die deprotonierten Basis- oder Saurierstellen wie Carboxyl, His, Cys und Tyr auch die potenziellen Bindungsstellen für das Metallion sind. Für die Analysen werden Peptid und Metallion in wässrigen Lösungen mit dem pH-Wert durch verdünnte wässrige Essigsäure oder Ammoniumhydroxid eingestellt. Dadurch können die pH-Abhängigkeit und die Metallionenselektivität für das Peptid bestimmt werden. Darüber hinaus können die von ESI-IM-MS ermittelten m/z und CCS mit B3LYP/LanL2DZ molekularer Modellierung verwendet werden, um die Art der Metallionenkoordination und die tertiäre Struktur des Komplexes zu ermitteln. Die in diesem Artikel gezeigten Ergebnisse zeigen, wie ESI-IM-MS die selektive Chelaterfüllung eines Satzes von Amb-Peptiden charakterisieren und mit dem Kupferbindenden Peptid mb-OB3b vergleichen kann.
Protokoll
1. Herstellung von Reagenzien
- Kultur Methylosinus trichosporium OB3b, isolieren Sie die Cu(I)-freie mb-OB3b18,22,23, gefrieren-trocknen Sie die Probe und lagern Sie bei -80 °C bis zum Gebrauch.
- Synthetisieren Sie die Amb-Peptide (>98% Reinheit für amb1, amb2, amb4; >70% Reinheit für amb7), gefrieren-trocknen Sie die Proben, und lagern Sie sie bei -80 °C bis zur Verwendung.
- Kauf >98% Reinheitsmangan(II)chlorid, Kobalt(II)chlorid, Nickle(II)chlorid, Kupfer(II)chlorid, Kupfer(II) Nitrat, Silber(I) Nitrat, Zink(II)chlorid, Eisen(III)chlorid und Blei(II)chlorid.
- Kaufen Sie die Poly-DL-Alaninpolymere, die als Kalibrame zur Messung der Kollisionsquerschnitte der Amb-Arten und HPLC-Grade oder höhere ammoniumhydroxid, Eisessigsäure und Acetonitril verwendet werden.
2. Vorbereitung der Lagerlösung
-
Peptid-Lagerlösung
- Wiegen Sie genau, mit mindestens drei signifikanten Zahlen, die Masse von 10,0–20,0 mg der mb-OB3b oder amb in einer 1,7 ml Kunststoff-Durchstechflasche.
HINWEIS: Die gewogene Masse sollte je nach Löslichkeit des Peptids entweder 12,5 mM oder 1,25 mM ergeben, wenn 1,00 ml deionisiertes (DI) Wasser zugegeben wird. - Mit einer Pipette 1,00 ml entionisiertes Wasser (>17,8 Mio.) in die gewogene Peptidprobe geben, um entweder die 12,5 mM- oder 1,25 mM-Lösung zu erhalten. Kappe sicher aufstellen und mit mindestens 20 Inversionen gründlich vermischen.
- Mit einem Mikropipet 50,0 L Aliquots aus der Peptidprobe in einzeln markierte 1,5 ml Durchstechflaschen geben und bis zur Verwendung bei -80 °C lagern.
- Wiegen Sie genau, mit mindestens drei signifikanten Zahlen, die Masse von 10,0–20,0 mg der mb-OB3b oder amb in einer 1,7 ml Kunststoff-Durchstechflasche.
-
Metallionen-Lagerlösungen
- Mit mindestens drei signifikanten Zahlen die Masse von 10,0–30,0 mg des Metallchlorids oder Silbernitrats in einer 1,7 ml Durchstechflasche genau wiegen.
HINWEIS: Die gewogene Masse sollte 125 mM ergeben, wenn 1,00 ml DI-Wasser zugegeben wird. - Fügen Sie die 1,00 ml DI-Wasser in die gewogene Metallprobe in die 1,7 ml-Durchstechflasche ein, um die 125 ml Lösung zu erhalten. Kappe sicher aufstellen und mit mindestens 20 Inversionen gründlich vermischen.
- Mit mindestens drei signifikanten Zahlen die Masse von 10,0–30,0 mg des Metallchlorids oder Silbernitrats in einer 1,7 ml Durchstechflasche genau wiegen.
- Ammoniumhydroxid-Stammlösungen:Bereiten Sie eine 1,0 M Essigsäurelösung vor, indem Sie 57 l der 99,5%igen Essigsäurelösung mit DI-Wasser auf ein Endvolumen von 1,00 ml verdünnen. Bereiten Sie eine 1,0 M Ammoniumhydroxid-Lösung vor, indem Sie 90 l der 21%igen Ammoniumhydroxidlösung mit DI-Wasser auf ein Endvolumen von 1,00 ml verdünnen. Machen Sie zwei aufeinanderfolgende Verdünnungen jeder Lösung, indem Sie 100 l der 1,0 M Lösungen zur Herstellung von 0,10 M und 0,010 M Essigsäure- und Ammoniumhydroxidlösungen einnehmen.
- Poly-DL-Alanin-Stammlösung:Bereiten Sie das Poly-DL-Alanin (PA) mit einem Gewicht von 1,0 mg PA vor und lösen Sie es in 1,0 ml DI-Wasser auf, um 1.000 ppm zu geben. Mischen Sie gründlich. Mit hilfe einer Mikropipet, geben Sie 50,0 L Aliquots, und legen Sie jeweils in eine 1,7 ml Durchstechflasche und lagern Sie bei -80 °C.
3. Elektrospray-Ionen-Mobilitäts-Massenspektrometrie-Analyse
- Reinigen Sie die ESI-Eingangsschläuche und Nadelkapillare gründlich mit ca. 500 l 0,1 M Eisessigsäure, 0,1 M Ammoniumhydroxid und schließlich DI-Wasser.
- Einen 50,0 L-Aliquot der 1.000 ppm PA-Lagerlösung auftauen und mit 450 l DI-Wasser verdünnen, um eine 100 ppm PA zu erhalten. Pipetten Sie 100,0 l dieser Lösung und verdünnen Sie sie auf 1,00 ml mit 500 l DI-Wasser und 500 l Acetonitril, um 10 ppm PA-Lösung zu geben.
- Sammeln Sie die negativen und positiven Ionen-IM-MS-Spektren der 10 ppm PA-Lösung für jeweils 10 min unter Verwendung nativer ESI-IM-MS-Bedingungen, wie im Diskussionsabschnitt beschrieben.
- Eine 50,0 L Aliquot der 12,5 mM oder 1,25 mM amb-Stammlösung auftauen und aufeinanderfolgende Verdünnungen mit DI-Wasser vornehmen, um eine Endkonzentration von 0,125 mM amb zu ergeben. Jede Verdünnung gründlich vermischen.
- Pipetten Sie 100,0 l der 125 mM Metallionen-Stammlösung, in eine 1,7 ml Durchstechflasche geben und mit DI-Wasser auf 1,00 ml verdünnen, um 12,5 mM Metallionen zu geben. Wiederholen Sie dies mit zwei weiteren aufeinanderfolgenden Verdünnungen, um eine endgültige Metallionenkonzentration von 0,125 mM zu ergeben. Jede Verdünnung gründlich vermischen.
- Pipetten Sie 200,0 l der 0,125 mM amb in eine 1,7 ml Durchstechflasche, verdünnen Sie sie mit 500 l DI-Wasser und mischen Sie die Lösung gründlich.
- Stellen Sie den pH-Wert der Probe auf 3,0 ein, indem Sie 50 l 1,0 M Essigsäurelösung hinzufügen.
- Fügen Sie der pH-angepassten Probe 200,0 l des 0,125 mM-Metallionens hinzu. Di-Wasser hinzufügen, um ein Endvolumen von 1,00 ml der Probe zu ergeben, gründlich mischen und die Probe 10 min bei RT ausdemieren lassen.
- Mit einer stumpfen Nasenspritze nehmen Sie 500 l der Probe und sammeln Sie die negativen und positiven Ionen-ES-IM-MS-Spektren für jeweils 5 min. Verwenden Sie die restlichen 500 l der Probe, um den endgültigen pH-Wert mit einer kalibrierten Mikro-pH-Elektrode aufzuzeichnen.
- Wiederholen Sie die Schritte 3.6–3.9, während Sie Schritt 3.7 ändern, um den pH-Wert auf 4,0, 5,0, 6,0, 7,0, 8,0, 9,0 oder 10,0 anzupassen, indem Sie neue Volumina der Lösungen 0,010 M, 0,10 M oder 1,0 M Essigsäure oder Ammoniumhydroxid hinzufügen.
- Sammeln Sie die negativen und positiven Ionen-ESI-IM-MS-Spektren der 10 ppm PA-Lösung für jeweils 10 min.
4. Vorbereitung der Metallionentitration von Ambproben
- Führen Sie die in den Schritten 3.1–3.5 beschriebenen Schritte aus.
- Pipetten Sie 200,0 l der 0,125 mM amb in eine 1,7 ml Durchstechflasche, verdünnen Sie sie mit 500,0 l DI-Wasser und mischen Sie die Lösung gründlich.
- Stellen Sie den pH-Wert der Probe auf pH = 9,0 ein, indem Sie 80 l der 0,010 M Ammoniumhydroxidlösung hinzufügen.
- Fügen Sie 28 l der 0,125 mM Metallionenlösung hinzu, um 0,14 Moläquivalente des Metallionens zu geben, DI-Wasser hinzuzufügen, um das Endvolumen der Probe 1,00 ml zu machen, gründlich zu mischen und die Probe 10 min bei RT ausgleichen zu lassen.
- Mit einer stumpfen Nasenspritze nehmen Sie 500 l der Probe und sammeln Sie die negativen und positiven Ionen-ESI-IM-MS-Spektren für jeweils 5 min. Verwenden Sie die restlichen 500 l der Probe, um den endgültigen pH-Wert mit einer kalibrierten Mikro-pH-Elektrode aufzuzeichnen.
- Wiederholen Sie die Schritte 4.2–4.5, während Sie Schritt 4.3 ändern, um ein entsprechendes Volumen der 0,125 mM Metallionenlösung hinzuzufügen, um entweder 0,28, 0,42, 0,56, 0,70, 0,84, 0,98, 1,12, 1,26 oder 1,40 Moläquivalente zu erhalten.
- Sammeln Sie die negativen und positiven Ionen-IM-MS-Spektren der 10 ppm PA-Lösung für jeweils 10 min.
5. Analyse der ESI-IM-MS pH-Titrationsdaten
- Aus den IM-MS-Spektren wird ermittelt, welche geladenen Arten von Ambs vorhanden sind, indem sie sie mit ihren theoretischen m/z-Isotopenmustern abgleichen.
- Öffnen Sie MassLynx und klicken Sie auf Chromatogramm, um das Chromatogramm-Fenster zu öffnen.
- Gehen Sie zum Menü Datei und Öffnen, um die IM-MS-Datendatei zu suchen und zu öffnen.
- Extrahieren Sie das IM-MS-Spektrum, indem Sie mit der rechten Maustaste auf das Chromatogramm klicken und es loslassen. Das Spektrumfenster wird geöffnet, das das IM-MS-Spektrum anzeigt.
- Klicken Sie im Spektrumfenster auf Werkzeuge und Isotope Modell. Geben Sie im Isotopenmodellierungsfenster die Molekularformel der amb-Arten ein, aktivieren Sie das Feld Geladene Ionen anzeigen, und geben Sie den Ladezustand ein. Klicken Sie auf OK.
- Wiederholen Sie dies, um alle Arten im IM-MS-Spektrum zu identifizieren und ihren m/z-Isotopenbereich aufzuzeichnen.
- Trennen Sie für jede amb-Art alle zufälligen m/z-Arten und extrahieren Sie ihre Ankunftszeitverteilungen (ATD) unter Verwendung ihrer m/z-Isotopenmuster, um sie zu identifizieren.
- Klicken Sie in MassLynx auf DriftScope, um das Programm zu öffnen. Klicken Sie in DriftScope auf Datei und Öffnen, um die IM-MS-Datendatei zu suchen und zu öffnen.
- Verwenden Sie die Maus und klicken Sie mit der linken Maustaste, um das m/z Isotopenmuster der amb-Arten zu vergrößern.
- Verwenden Sie das Auswahlwerkzeug und die linke Maustaste, um das Isotopenmuster auszuwählen. Klicken Sie auf die Schaltfläche Aktuelle Auswahl akzeptieren.
- Um alle zufälligen m/z-Arten zu trennen, verwenden Sie das Auswahlwerkzeug und die linke Maustaste, um das ATD mit dem Isotopmuster der amb-Arten auszurichten. Klicken Sie auf die Schaltfläche Aktuelle Auswahl akzeptieren.
- Um die ATD zu exportieren, gehen Sie zu Datei | Exportieren Sie nach MassLynx, dann wählen Sie Drift Time beibehalten und speichern Sie die Datei in einem entsprechenden Ordner.
- Bestimmen Sie den Schwerpunkt der ATD und integrieren Sie das Gebiet unter der ATD-Kurve als Maß für die Artenpopulation.
- Im Chromatogramm-Fenster von MassLynx öffnen Sie die gespeicherte exportierte Datei. Klicken Sie auf Prozess | Integrieren Sie aus dem Menü. Aktivieren Sie das Kontrollkästchen ApexTrack Peak Integration, und klicken Sie auf OK.
- Zeichnen Sie den Schwerpunkt ATD (tA) und den integrierten Bereich auf, wie im Chromatogrammfenster gezeigt. Wiederholen Sie dies für alle gespeicherten amb- und PA IM-MS-Datendateien.
- Verwenden Sie die integrierte ATD für alle extrahierten Amb-Arten der positiven oder negativen Ionen an jedem Titrationspunkt, um sich auf eine relative prozentuale Skala zu normalisieren.
- Geben Sie die Identitäten der amb-Art und ihre integrierte ATD bei jedem pH-Wert in eine Tabelle ein.
- Verwenden Sie für jeden pH-Wert die Summe der integrierten ATDs, um die ATD der einzelnen amb auf eine prozentuale Skala zu normalisieren.
- Plotten Sie die prozentualen Intensitäten jeder Amb-Art im Vergleich zum pH-Wert in einer Grafik, um zu zeigen, wie die Population jeder Art je nach pH-Wert variiert.
6. Kollisionsquerschnitte
- Konvertieren Sie mithilfe einer Kalkulationstabelle die CCSs ()) von PA-Negativ25,26 und positiven27 Ionen, gemessen in He Puffergas28, in korrigiertes CCS(c) mit Gleichung 1 unten, wobei: z = Ionenladung; e c = Elektronenladung (1,602 x 10-19 C); m N 2 = Masse vonN2 Gas (Da); und mion = Ionenmasse. 29
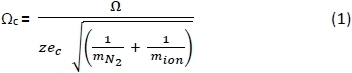
- Konvertieren Sie die durchschnittlichen Ankunftszeiten (tA) der PA-Kalibrazianten und Amb-Arten in Driftzeiten (tD) unter Verwendung von Gleichung 2 unten, wobei: c = der erhöhte Ztastzyklus-Delay-Koeffizient (1,41) und m/z die Masse-zu-Ladung des Peptid-Ions ist.

- Zeichnen Sie die PA-Kalibratoren' tD vs. ihrec. Bestimmen Sie dann anhand einer Regressionsanpassung der nachstehenden Gleichung 3 die A'- und B-Werte, wobei: A' die Korrektur der Temperatur-, Druck- und elektrischen Feldparameter ist; und B kompensiert die nichtlineare Wirkung des IM-Geräts.

- Unter Verwendung dieser A' und B-Werte und des Zentroiden t D-Werts aus dem ATD der ambs bestimmen sie ihrec mit Gleichung 3 und ihre mit Gleichung 1. Diese Methode bietet CCS für die Peptidarten mit geschätzten absoluten Fehlern von etwa 2%25,26,27.
7. Berechnungsmethoden
- Verwenden Sie die Theorieebene B3LYP/LanL2DZ, bestehend aus den Becke 3-Parameter-Hybridfunktionen30 und dem Dunning-Basissatz31 und den Elektronenkernpotentialen32,33,34, um geometrieoptimierte Konformer für alle möglichen Koordinationsarten der beobachteten m/z amb Art35.
HINWEIS: Details zum Erstellen und Übermitteln von Berechnungen finden Sie in der GaussView-Verwendung in Der Zusatzdatei. - Vergleichen Sie die vorhergesagte freie Energie der einzelnen Konformer und berechnen Sie ihre theoretischen CCS mit der ionenskalierten Lennard-Jones (LJ)-Methode aus dem Sigma-Programm36.
- Aus den niedrigsten freien Energiekonformern bestimmen, welche Konformere den LJ CCS aufweisen, der mit dem im-MS gemessenen CCS übereinstimmt, um die tertiäre Struktur und Art der Koordination für die im Experiment beobachteten Konformer zu identifizieren.
Ergebnisse
Metallbindung von amb1
Die IM-MS-Studie20 von amb1 (Abbildung 1A) zeigte, dass sowohl Kupfer- als auch Zinkionen, die auf pH-abhängige Weise an amb1 gebunden sind (Abbildung 2). Kupfer und Zink sind jedoch durch unterschiedliche Reaktionsmechanismen an verschiedenen Koordinationsstellen an amb1 gebunden. Das Hinzufügen von Cu(II) zu amb1 führte ...
Diskussion
Kritische Schritte: Erhaltung von Lösungsphasenverhalten zur Untersuchung über ESI-IM-MS
Native ESI-Instrumenteinstellungen müssen verwendet werden, um die Peptide Stoichiometrie, Ladezustand und Konformationsstruktur zu erhalten. Für native Bedingungen müssen die Bedingungen in der ESI-Quelle wie Kegelspannungen, Temperaturen und Gasströme optimiert werden. Außerdem müssen die Drücke und Spannungen in der Quelle, Falle, Ionenmobilität und Übertragungs-Reisewellen (insbesondere die DC-Falle...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Dieses Material basiert auf Arbeiten, die von der National Science Foundation unter 1764436, NSF-Instrumentenunterstützung (MRT-0821247), Welch Foundation (T-0014) und Rechenressourcen des Department of Energy (TX-W-20090427-0004-50) und L3 Communications unterstützt werden. . Wir danken der Bower-Gruppe der University of California - Santa Barbara für die gemeinsame Nutzung des Sigma-Programms und Ayobami Ilesanmi für die Demonstration der Technik im Video.
Materialien
| Name | Company | Catalog Number | Comments |
| acetonitrile HPLC-grade | Fisher Scientific (www.Fishersci.com) | A998SK-4 | |
| ammonium hydroxide (trace metal grade) | Fisher Scientific (www.Fishersci.com) | A512-P500 | |
| cobalt(II) chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 255599-5G | |
| copper(II) chloride 99.999% | Sigma-Aldrich (www.sigmaaldrich.com) | 203149-10G | |
| copper(II) nitrate hydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 229636-5G | |
| designed amb1,2,3,4,5,6,7 peptides | Neo BioLab (neobiolab.com) | designed peptides were synthized by order | |
| designed amb5B,C,D,E,F peptides | PepmicCo (www.pepmic.com) | designed peptides were synthized by order | |
| Driftscope 2.1 software program | Waters (www.waters.com) | software analysis program | |
| Freeze-dried, purified, Cu(I)-free mb-OB3b | cultured and isolated in the lab of Dr. DongWon Choi (Biology Department, Texas A&M-Commerce) | ||
| glacial acetic acid (Optima grade) | Fisher Scientific (www.Fishersci.com) | A465-250 | |
| Iron(III) Chloride Anhydrous 98%+ | Alfa Aesar (www.alfa.com) | 12357-09 | |
| lead(II) nitrate ACS grade | Avantor (www.avantormaterials.com) | 128545-50G | |
| manganese(II) chloride tetrahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203734-5G | |
| MassLynx 4.1 | Waters (www.waters.com) | software analysis program | |
| nickel chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203866-5G | |
| poly-DL-alanine | Sigma-Aldrich (www.sigmaaldrich.com) | P9003-25MG | |
| silver nitrate 99.9%+ | Alfa Aesar (www.alfa.com) | 11414-06 | |
| Waters Synapt G1 HDMS | Waters (www.waters.com) | quadrupole - ion mobility- time-of-flight mass spectrometer | |
| zinc chloride anhydrous | Alfa Aesar (www.alfa.com) | A16281 |
Referenzen
- Dudev, T., Lim, C. Competition among Metal Ions for Protein Binding Sites: Determinants of Metal Ion Selectivity in Proteins. Chemical Reviews. 114 (1), 538-556 (2014).
- Sovago, I., Kallay, C., Varnagy, K. Peptides as complexing agents: Factors influencing the structure and thermodynamic stability of peptide complexes. Coordination Chemistry Reviews. 256 (19-20), 2225-2233 (2012).
- Sóvágó, I., Várnagy, K., Lihi, N., Grenács, &. #. 1. 9. 3. ;. Coordinating properties of peptides containing histidyl residues. Coordination Chemistry Reviews. 327, 43-54 (2016).
- Rubino, J. T., Franz, K. J. Coordination chemistry of copper proteins: How nature handles a toxic cargo for essential function. Journal of Inorganic Biochemistry. 107 (1), 129-143 (2012).
- Robinson, N. J., Winge, D. R. Copper Metallochaperones . Annual Review of Biochemistry. 79, 537-562 (2010).
- Scheiber, I. F., Mercer, J. F. B., Dringen, R. Metabolism and functions of copper in brain. Progress in Neurobiology. 116, 33-57 (2014).
- Tisato, F., Marzano, C., Porchia, M., Pellei, M., Santini, C. Copper in Diseases and Treatments, and Copper-Based Anticancer Strategies. Medicinal Research Reviews. 30 (4), 708-749 (2010).
- Millhauser, G. L. Copper and the prion protein: Methods, structures, function, and disease. Annual Review of Physical Chemistry. 58, 299-320 (2007).
- Arena, G., Pappalardo, G., Sovago, I., Rizzarelli, E. Copper(II) interaction with amyloid-beta: Affinity and speciation. Coordination Chemistry Reviews. 256 (1-2), 3-12 (2012).
- Kim, H. J., et al. Methanobactin, a copper-acquisition compound from methane-oxidizing bacteria. Science. 305 (5690), 1612-1615 (2004).
- Di Spirito, A. A., et al. Methanobactin and the link between copper and bacterial methane oxidation. Microbiology Molecular Biology Reviews. 80 (2), 387-409 (2016).
- Kenney, G. E., Rosenzweig, A. C. Chemistry and biology of the copper chelator methanobactin. ACS Chemical Biology. 7 (2), 260-268 (2012).
- Summer, K. H., et al. The biogenic methanobactin is an effective chelator for copper in a rat model for Wilson disease. Journal of Trace Elements in Medicine and Biology. 25 (1), 36-41 (2011).
- Hachmoeller, O., et al. Investigating the influence of standard staining procedures on the copper distribution and concentration in Wilson's disease liver samples by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 44, 71-75 (2017).
- Hachmoeller, O., et al. Spatial investigation of the elemental distribution in Wilson's disease liver after D-penicillamine treatment by LA-ICP-MS. Journal of Trace Elements in Medicine and Biology. 44, 26-31 (2017).
- Hachmoeller, O., et al. Element bioimaging of liver needle biopsy specimens from patients with Wilson's disease by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 35, 97-102 (2016).
- Mueller, J. C., Lichtmannegger, J., Zischka, H., Sperling, M., Karst, U. High spatial resolution LA-ICP-MS demonstrates massive liver copper depletion in Wilson disease rats upon Methanobactin treatment. Journal of Trace Elements in Medicine and Biology. 49, 119-127 (2018).
- Choi, D. W., et al. Spectral and thermodynamic properties of Ag(I), Au(III), Cd(II), Co(II), Fe(III), Hg(II), Mn(II), Ni(II), Pb(II), U(IV), and Zn(II) binding by methanobactin from Methylosinus trichosporium OB3b. Journal of Inorganic Biochemistry. 100, 2150-2161 (2006).
- McCabe, J. W., Vangala, R., Angel, L. A. Binding Selectivity of Methanobactin from Methylosinus trichosporium OB3b for Copper(I), Silver(I), Zinc(II), Nickel(II), Cobalt(II), Manganese(II), Lead(II), and Iron(II). Journal of the American Society of Mass Spectrometry. 28, 2588-2601 (2017).
- Sesham, R., et al. The pH dependent Cu(II) and Zn(II) binding behavior of an analog methanobactin peptide. European Journal of Mass Spectrometry. 19 (6), 463-473 (2013).
- Wagoner, S. M., et al. The multiple conformational charge states of zinc(II) coordination by 2His-2Cys oligopeptide investigated by ion mobility - mass spectrometry, density functional theory and theoretical collision cross sections. Journal of Mass Spectrom. 51 (12), 1120-1129 (2016).
- Bandow, N. L., et al. Isolation of methanobactin from the spent media of methane-oxidizing bacteria. Methods in Enzymology. 495, 259-269 (2011).
- Choi, D. W., et al. Spectral and thermodynamic properties of methanobactin from γ-proteobacterial methane oxidizing bacteria: a case for copper competition on a molecular level. Journal of Inorganic Biochemistry. 104 (12), 1240-1247 (2010).
- Pringle, S. D., et al. An investigation of the mobility separation of some peptide and protein ions using a new hybrid quadrupole/travelling wave IMS/oa-ToF instrument. International Journal of Mass Spectrometry. 261 (1), 1-12 (2007).
- Forsythe, J. G., et al. Collision cross section calibrants for negative ion mode traveling wave ion mobility-mass spectrometry. Analyst. 14 (20), 6853-6861 (2015).
- Allen, S. J., Giles, K., Gilbert, T., Bush, M. F. Ion mobility mass spectrometry of peptide, protein, and protein complex ions using a radio-frequency confining drift cell. Analyst. 141 (3), 884-891 (2016).
- Bush, M. F., Campuzano, I. D. G., Robinson, C. V. Ion Mobility Mass Spectrometry of Peptide Ions: Effects of Drift Gas and Calibration Strategies. Analytical Chemistry. 84 (16), 7124-7130 (2012).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Smith, D. P., et al. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. European Journal of Mass Spectrometry. 15 (2), 113-130 (2009).
- Becke, A. D. Density-functional thermochemistry. III. The role of exact exchange. Journal of Chemical Physics. 98 (7), 5648-5652 (1993).
- Dunning, T. H., Hay, P. J. Gaussian basis sets for molecular calculations. Modern Theoretical Chemistry. 3, 1-27 (1977).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for potassium to gold including the outermost core orbitals. Journal of Chemical Physics. 82 (1), 299-310 (1985).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for the transition metal atoms scandium to mercury. Journal of Chemical Physics. 82 (1), 270-283 (1985).
- Wadt, W. R., Hay, P. J. Ab initio effective core potentials for molecular calculations. Potentials for main group elements sodium to bismuth. Journal of Chemical Physics. 82 (1), 284-298 (1985).
- . Gaussian 09, Revision C.01. Gaussian, Inc. , (2012).
- Wyttenbach, T., von Helden, G., Batka, J. J., Carlat, D., Bowers, M. T. Effect of the long-range potential on ion mobility measurements. Journal of the American Society of Mass Spectrometry. 8 (3), 275-282 (1997).
- Choi, D., et al. Redox activity and multiple copper(I) coordination of 2His-2Cys oligopeptide. Journal of Mass Spectrometry. 50 (2), 316-325 (2015).
- Rigo, A., et al. Interaction of copper with cysteine: stability of cuprous complexes and catalytic role of cupric ions in anaerobic thiol oxidation. Journal of Inorganic Biochemistry. 98 (9), 1495-1501 (2004).
- Vytla, Y., Angel, L. A. Applying Ion Mobility-Mass Spectrometry Techniques for Explicitly Identifying the Products of Cu(II) Reactions of 2His-2Cys Motif Peptides. Analytical Chemistry. 88 (22), 10925-10932 (2016).
- Choi, D., Sesham, R., Kim, Y., Angel, L. A. Analysis of methanobactin from Methylosinus trichosporium OB3b via ion mobility mass spectrometry. European Journal of Mass Spectrometry. 18 (6), 509-520 (2012).
- Martell, A. E., Motekaitis, R. J. NIST Standard Reference Database 46. Institute of Standards and Technology. , (2001).
- Pesch, M. L., Christl, I., Hoffmann, M., Kraemer, S. M., Kretzschmar, R. Copper complexation of methanobactin isolated from Methylosinus trichosporium OB3b: pH-dependent speciation and modeling. Journal of Inorganic Biochemistry. 116, 55-62 (2012).
- Amin, E. A., Truhlar, D. G. Zn Coordination Chemistry: Development of Benchmark Suites for Geometries, Dipole Moments, and Bond Dissociation Energies and Their Use To Test and Validate Density Functionals and Molecular Orbital Theory. Journal of Chemical Theory and Computation. 4 (1), 75-85 (2008).
- Sorkin, A., Truhlar, D. G., Amin, E. A. Energies, Geometries, and Charge Distributions of Zn Molecules, Clusters, and Biocenters from Coupled Cluster, Density Functional, and Neglect of Diatomic Differential Overlap Models. Journal of Chemical Theory and Computation. 5 (5), 1254-1265 (2009).
- Lillo, V., Galan-Mascaros, J. R. Transition metal complexes with oligopeptides: single crystals and crystal structures. Dalton Transactions. 43 (26), 9821-9833 (2014).
- Choutko, A., van Gunsteren, W. F. Conformational Preferences of a beta-Octapeptide as Function of Solvent and Force-Field Parameters. Helvetica Chimica Acta. 96 (2), 189-200 (2013).
- Angel, L. A. Study of metal ion labeling of the conformational and charge states of lysozyme by ion mobility mass spectrometry. European Journal of Mass Spectrometry. 17 (3), 207-215 (2011).
- Kelso, C., Rojas, J. D., Furlan, R. L. A., Padilla, G., Beck, J. L. Characterisation of anthracyclines from a cosmomycin D-producing species of Streptomyces by collisionally-activated dissociation and ion mobility mass spectrometry. European Journal of Mass Spectrometry. 15 (2), 73-81 (2009).
- El Ghazouani, A., et al. Copper-binding properties and structures of methanobactins from Methylosinus trichosporium OB3b. Inorganic Chemistry. 50 (4), 1378-1391 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten