É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Técnicas de espectrometria de massa de mobilidade de íons para determinação da estrutura e mecanismos de reconhecimento de íons metálicos e atividade redox de oligopeptídeos de ligação metálica
Neste Artigo
Resumo
A mobilidade do íon-espectrometria maciça e as técnicas de modelagem molecular podem caracterizar o desempenho quelante do metal seletivo de peptídeos metal-obrigatórios projetados e do methanobactin Copper-Binding do peptide. O desenvolvimento de novas classes de peptídeos quelantes metálicos ajudará a levar a terapêutica para doenças associadas ao desbalanceamento de íons metálicos.
Resumo
A ionização por electrospray (ESI) pode transferir um peptídeo de fase aquosa ou peptídeo complexo para a fase gasosa, conservando sua massa, carga global, interações de ligação metálica e forma conformacional. O acoplamento ESI com a espectrometria de massa da mobilidade do íon (im-MS) fornece uma técnica instrumental que permita a medida simultânea da massa-à-carga de um peptide (m/z) e da seção transversal da colisão (CCS) que se relacionam a sua estequiometria, estado do protonação, e forma conformacional. A carga geral de um complexo peptídeo é controlada pela protonação de 1) os sítios ácidos e básicos do peptídeo e 2) o estado de oxidação do íon metálico (s). Portanto, o estado de carga geral de um complexo é uma função do pH da solução que afeta a afinidade de ligação de íons metálicos de peptídeos. Para as análises de ESI-IM-MS, as soluções de peptídeos e íons metálicos são preparadas a partir de soluções somente aquosas, com o pH ajustado com ácido acético aquoso diluído ou hidróxido de amônio. Isso permite que a dependência de pH e a seletividade de íons metálicos sejam determinadas para um peptídeo específico. Além disso, o m/z e o CCS de um complexo do peptide podem ser usados com modelagem molecular de B3LYP/LanL2DZ para discernir locais obrigatórios da coordenação do íon do metal e da estrutura terciária do complexo. Os resultados mostram como o ESI-IM-MS pode caracterizar o desempenho quelante seletivo de um conjunto de peptídeos de metanobacttina alternativos e compará-los com o peptídeo de ligação de cobre methanobactin.
Introdução
Os íons de cobre e zinco são essenciais para os organismos vivos e cruciais para os processos, incluindo proteção oxidativa, crescimento tecidual, respiração, colesterol, metabolismo da glicose e leitura do genoma1. Para habilitar essas funções, grupos como o tiolato de Cys, imidazol de his2,3, (mais raramente) thioether de metionina, e carboxilato de Glu e ASP seletivamente incorporar metais como cofatores para os locais ativos de metaloenzimas. A similaridade destes grupos de coordenação levanta uma pergunta intrigante a respeito de como os ligantes his e de Cys incorporam seletivamente tanto o UC (I/II) ou o Zn (II) para assegurar o funcionamento correto.
A ligação seletiva é muitas vezes realizada por peptídeos de aquisição e tráfico, que controlam as concentrações de íons Zn (II) ou UC (I/II)4. O UC (I/II) é altamente reactivo e causa dano oxidativo ou ligação adventícia às enzimas, assim que sua concentração livre é regulada firmemente por chaperonas de cobre e por proteínas de regulação de cobre que o transportam com segurança aos vários locais na pilha e firmemente controle sua homeostase5,6. O rompimento do metabolismo ou da homeostase de cobre é implicado diretamente em Menkes e Wilson ' doença de s7 assim como cancros7 e desordens neural, tais como do prião8 e doença de Alzheimer9.
A doença de Wilson está associada ao aumento dos níveis de cobre nos olhos, fígado e seções do cérebro, onde as reações redox do UC (I/II) produzem espécies reativas de oxigênio, causando degeneração hepatolenticular e neurológica. As terapias existentes da quelação são o aminoácido penicilamina pequeno do tiol e o triethylenetetramine. Alternativamente, os peptídeos de aquisição de cobre metanotróficos methanobactin (MB)10,11exibem potencial terapêutico por causa de sua alta afinidade de ligação para o UC (I)12. Quando o methanobactin (MB-OB3b) do Methylosinus trichosporium OB3b foi estudado em um modelo animal de Wilson ' a doença de s, cobre foi removida eficientemente do fígado e excretada através da bilis13. Experimentos in vitro confirmaram que o MB-OB3b poderia quelato o cobre da metalotionina de cobre contida no citosol do fígado13. A ablação por laser indutivamente acoplada a espectrometria de massas de massa plasmática investigou a distribuição espacial do cobre nas amostras de fígado da doença de Wilson14,15,16e demonstrou que a MB-OB3b Remove o cobre com curtos períodos de tratamento de apenas 8 dias17.
O MB-OB3b também se associará a outros íons metálicos, incluindo AG (I), au (III), PB (II), MN (II), co (II), FE (II), Ni (II) e Zn (II)18,19. O concurso para o sítio de ligação de UC (I) fisiológico é exibido pela AG (I) porque pode deslocar o UC (I) do complexo MB-OB3b, com AG (I) e Ni (II) também mostrando ligação irreversível a MB que não pode ser deslocada pelo UC (I)19. Recentemente, uma série de oligopeptídeos alternativos de methanobactin (AMB) com o motivo de ligação 2his-2cys foram estudados20,21, e suas propriedades de ligação Zn (II) e UC (I/II) caracterizadas. Suas seqüências de aminoácidos primários são semelhantes, e todos eles contêm o motivo 2His-2Cys, pro e um N-Terminus acetilado. Eles diferem principalmente de MB-OB3b porque o motivo 2His-2Cys substitui os dois locais de ligação de oxazolona enethiol de MB-OB3b.
A ionização de electrospray acoplada com a espectrometria da mobilidade-massa do íon (ESI-IM-MS) fornece para uma técnica instrumental poderosa para determinar as propriedades metal-obrigatórias dos peptídeos porque mede sua massa-à-carga (m/z) e colisão seção transversal (CCS) ao conservar sua massa, carga, e forma conformacional da solução-fase. O m/z e CCS referem-se à estequiometria de peptídeos, estado de protonação e forma conformacional. A estequiometria é determinada porque a identidade e o número de cada elemento presente na espécie são explicitamente identificados. A carga geral do complexo peptídeo relaciona-se com o estado de protonação dos sítios ácidos e básicos e o estado de oxidação do íon metálico (s). O CCS dá a informação da forma conformacional do complexo do peptide porque mede o tamanho médio rotatório que se relaciona à estrutura terciária do complexo. O estado de carga geral do complexo também é uma função do pH e afeta a afinidade de ligação de íons metálicos do peptídeo porque os locais de base ou ácidos desprotonados como o carboxilo, his, Cys e Tyr também são os locais de ligação potenciais para o íon metálico. Para as análises, o peptídeo e o íon metálico são preparados em soluções aquosas com o pH ajustado por diluir o ácido acético aquoso ou hidróxido de amônio. Isso permite que a dependência de pH e a seletividade de íons metálicos sejam determinadas para o peptídeo. Além disso, o m/z e o CCS determinados por ESI-im-MS podem ser usados com modelagem molecular de B3LYP/LanL2DZ para descobrir o tipo de coordenação do íon do metal e a estrutura terciária do complexo. Os resultados apresentados neste artigo revelam como o ESI-IM-MS pode caracterizar o desempenho quelante seletivo de um conjunto de peptídeos AMB e compará-los com o peptídeo de ligação de cobre MB-OB3b.
Protocolo
1. preparação dos reagentes
- Cultura methylosinus trichosporium OB3b, isolar a UC (I)-livre MB-OB3b18,22,23, congelar-secar a amostra e armazenar em-80 ° c até o uso.
- Sintetizar os peptídeos AMB (> 98% de pureza para AMB1, AMB2, AMB4; > 70% de pureza para AMB7), congelar-secar as amostras, e armazená-los em-80 ° c até o uso.
- Comprar > 98% pureza manganês (II) cloreto, cobalto (II) cloreto, niquelar (II) cloreto, cobre (II) cloreto, cobre (II) nitrato, prata (I) nitrato, zinco (II) cloreto, ferro (III) cloreto e chumbo (II) cloreto.
- Adquira os polímeros poli-DL-alanina utilizados como calibrantes para medir as secções transversais de colisão das espécies AMB e HPLC ou hidróxido de amónio superior, ácido acético glacial e acetonitrila.
2. preparação da solução de ações
-
Solução de peptídeos
- Pesar com precisão, usando pelo menos três figuras significativas, a massa de 10,0 – 20,0 mg do MB-OB3b ou AMB em um frasco de 1,7 mL de plástico.
Nota: a massa pesada deve produzir 12,5 mM ou 1,25 mM, dependendo da solubilidade do peptídeo, quando se acrescenta 1, 0 mL de água desionizada (DI). - Usando um Pipet, adicione 1, 0 mL de água deionizada (> 17,8 MΩ cm) à amostra de peptídeo pesado para produzir a solução de 12,5 mM ou 1,25 mM. Coloc firmemente a tampa e misture-a completamente com pelo menos 20 Inversions.
- A utilização de um micropipetas dispensa 50,0 μL de alíquotas da amostra de peptídeos em frascos de 1,5 mL rotulados individualmente e armazená-los a-80 ° c até o uso.
- Pesar com precisão, usando pelo menos três figuras significativas, a massa de 10,0 – 20,0 mg do MB-OB3b ou AMB em um frasco de 1,7 mL de plástico.
-
Soluções de estoque de íons metálicos
- Pesar com precisão, usando pelo menos três figuras significativas, a massa de 10,0 – 30,0 mg do cloreto de metal ou nitrato de prata em um frasco de 1,7 mL.
Nota: a massa pesada deve produzir 125 mM quando 1, 0 mL de água DI é adicionado. - Adicione o 1, 0 mL de água DI à amostra de metal pesado no frasco para injetáveis de 1,7 mL para produzir a solução de 125 mM. Coloc firmemente a tampa e misture-a completamente com pelo menos 20 Inversions.
- Pesar com precisão, usando pelo menos três figuras significativas, a massa de 10,0 – 30,0 mg do cloreto de metal ou nitrato de prata em um frasco de 1,7 mL.
- Soluções de hidróxido de amónio: preparar uma solução de ácido acético a 1,0 M diluindo 57 μl da solução de ácido acético a 99,5% com água di para um volume final de 1, 0 ml. Prepare uma solução de hidróxido de amónio de 1,0 M diluindo 90 μL da solução de hidróxido de amónio a 21% com água DI para um volume final de 1, 0 mL. Faça duas diluições sucessivas de cada solução, tomando 100 μL das soluções 1,0 M para preparar as soluções de ácido acético 0,10 M e 0, 10 M e hidróxido de amônio.
- Solução de estoque de poli-DL-alanina: Prepare a poli-DL-alanina (PA) pesando 1,0 mg de PA e dissolvendo em 1,0 ml de água di para dar 1.000 ppm. Homogeneizar. Usando um micropipet, dispense 50,0 μL de alíquotas e coloque cada um em um frasco para injetáveis de 1,7 mL e armazene a-80 ° c.
3. a mobilidade do electrospray-íon-análise da espectrometria maciça
- Limpe o tubo de entrada ESI e a agulha capilar cuidadosamente com cerca de 500 μL de ácido acético glacial de 0,1 M, hidróxido de amônio de 0,1 M e, finalmente, água DI.
- Descongelar uma alíquota de 50,0 μL da solução de estoque de PA 1.000 ppm e dilui-la com 450 μL de água DI para dar um PA de 100 ppm. Pipet 100,0 μL desta solução e dilui-lo para 1, 0 mL com 500 μL de água DI e 500 μL de acetonitrila para dar solução de PA de 10 ppm.
- Colete os espectros de IM-MS de íons negativos e positivos da solução de PA de 10 ppm por 10 min cada um usando as condições de ESI-IM-MS nativas, conforme descrito na seção de discussão.
- Descongelar uma alíquota de 50,0 μL da solução de estoque de 12,5 mM ou 1,25 mM AMB e fazer diluições sucessivas com água DI para dar uma concentração final de 0,125 mM amb. Misture cuidadosamente cada diluição.
- Pipet 100,0 μL da solução de iões de metal de 125 mM, coloque num frasco para injetáveis de 1,7 mL e diluir para 1, 0 mL com água DI para dar um íon metálico de 12,5 mM. Repita com duas diluições sucessivas para dar uma concentração final de íons metálicos de 0,125 mM. Misture cuidadosamente cada diluição.
- Pipet 200,0 μL de 0,125 mM AMB num frasco para injetáveis de 1,7 mL, diluir com 500 μL de água DI e misturar a solução completamente.
- Ajuste o pH da amostra para 3,0 adicionando 50 μL de solução de ácido acético a 1,0 M.
- Adicionar 200,0 μL do íon metálico de 0,125 mM à amostra ajustada por pH. Adicione a água de DI para produzir um volume final de 1, 0 mL da amostra, misture completamente, e permita que a amostra equilibe por 10 minutos em RT.
- Usando uma seringa sem corte do nariz Tome 500 μL da amostra e colete os espectros negativos e positivos do íon ES-IM-MS por 5 minutos cada um. Use o restante 500 μL da amostra para registrar seu pH final usando um micro eletrodo calibrado de pH.
- Repita as etapas 3.6 – 3.9, enquanto modifica a etapa 3,7 para ajustar o pH para 4,0, 5,0, 6,0, 7,0, 8,0, 9,0 ou 10,0 adicionando novos volumes das soluções de hidróxido de amónio ou de ácido acético 0, 10 m, 0,10 M ou 1,0 M.
- Colete os espectros negativos e positivos do íon ESI-IM-MS da solução de PA de 10 ppm por 10 min cada.
4. preparação da titulação de íons metálicos de amostras AMB
- Siga as etapas descritas nas etapas 3.1 – 3.5.
- Pipet 200,0 μL de 0,125 mM AMB num frasco para injetáveis de 1,7 mL, diluir com 500,0 μL de água DI e misturar a solução completamente.
- Ajuste o pH da amostra para pH = 9,0 adicionando 80 μL da solução de hidróxido de amônio de 0, 10 M.
- Adicione 28 μL da solução do íon do metal de 0,125 milímetros para dar 0,14 equivalentes do molar do íon do metal, adicionam a água do DI para fazer o volume final da amostra 1, 0 mL, misturam-se completamente, e permitem que a amostra equilibe por 10 minutos em RT.
- Usando uma seringa sem corte do nariz Tome 500 μL da amostra e colete os espectros negativos e positivos do íon ESI-IM-MS por 5 minutos cada um. Use o restante 500 μL da amostra para registrar seu pH final usando um micro eletrodo calibrado de pH.
- Repita as etapas 4.2 – 4.5, enquanto modifica a etapa 4,3 para adicionar um volume apropriado da solução de íons metálicos de 0,125 mM para dar 0,28, 0,42, 0,56, 0,70, 0,84, 0,98, 1,12, 1,26 ou 1,40 equivalentes molar.
- Colete os espectros negativos e positivos do íon IM-MS da solução do PA de 10 ppm por 10 minutos cada um.
5. análise dos dados de titulação de pH ESI-IM-MS
- A partir dos espectros IM-MS identificam quais espécies carregadas de ambs estão presentes combinando-as com seus padrões teóricos de isótopos m/z.
- Abra o MassLynx e clique em cromatograma para abrir a janela cromatograma.
- Vá para o menu arquivo e abra para localizar e abra o arquivo de dados im-MS.
- Extraia o espectro de IM-MS clicando com o botão direito do mouse e arrastando através do cromatograma e liberando. A janela do espectro abrirá mostrando o espectro IM-MS.
- Na janela de espectro, clique em ferramentas e modelo isotópica. Na janela de modelagem de isótopos, insira a fórmula molecular da espécie AMB, marque a caixa de íon carregada show e insira o estado da carga. Clique em OK.
- Repita para identificar todas as espécies no espectro IM-MS e gravar a sua gama de isótopos m/z.
- Para cada espécie AMB, separe qualquer espécie de m/z coincidente e extraia suas distribuições de tempo de chegada (ATD) usando seus padrões de isótopos m/z para identificá-los.
- Em MassLynx clique em Driftscope para abrir o programa. Em DriftScope clique em arquivo e abrir para localizar e abrir o arquivo de dados im-MS.
- Use o mouse e clique com o botão esquerdo para ampliar o padrão de isótopos m/z da espécie amb.
- Use a ferramenta de seleção e o botão esquerdo do mouse para selecionar o padrão de isótopos. Clique no botão aceitar seleção atual .
- Para separar qualquer espécie de m/z coincidente use a ferramenta de seleção e o botão esquerdo do mouse para selecionar o tempo de ATD alinhado com o padrão isotópico da espécie amb. Clique no botão aceitar seleção atual .
- Para exportar o ATD, vá para arquivo | Exportar para MassLynx, em seguida, selecione reter tempo de deriva e salvar o arquivo na pasta apropriada.
- Determine o centroide do ATD e integre a área a curva ATD como uma medida da população de espécies.
- Na janela cromatograma do MassLynx, abra o arquivo exportado salvo. Clique em processo | Integre a partir do menu. Verifique a caixa de integração de pico de ApexTrack e clique em OK.
- Registre o centroide ATD(t a) e a área integrada, conforme mostrado na janela do cromatograma . Repita para todos os arquivos de dados salvos AMB e PA IM-MS.
- Use o ATD integrado para todas as espécies de AMB extraídas de íons positivos ou negativos em cada ponto de titulação para normalizar a uma escala de porcentagem relativa.
- Insira as identidades das espécies AMB e seu ATD integrado em cada pH em uma planilha.
- Para cada pH, use a soma dos ATDs integrados para normalizar o ATD do AMB individual para uma escala percentual.
- Plotar as intensidades percentuais de cada espécie AMB versus pH em um gráfico para mostrar como a população de cada espécie varia em função do pH.
6. secções transversais de colisão
- Usando uma planilha, converta o CCSS (ω) de PA negativa25,26 e positivo27 íons medidos em ele gás tampão28 para corrigido CCS (ωc) usando a equação 1 abaixo, onde: z = carga de íons; e carga de c = Electron (1.602 × 10-19 c); m N 2 = massa de N2 gás (da); e mIon = Ion massa. 29. º
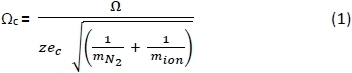
- Converta os tempos médios de chegada (ta) das calibrantes PA e das espécies AMB em tempos de deriva (tD) usando a equação 2 abaixo, onde: c = o coeficiente de retardo do ciclo de trabalho aprimorado (1,41) e m/z é a massa-a-carga do íon peptídeo.

- Plotar os calibrantes PA ' tD vs. seus Ωc. Em seguida, usando um ajuste de regressão de mínimos quadrados da equação 3 mostrada abaixo, determine os valores a ' e B, onde: a ' é a correção para os parâmetros de temperatura, pressão e campo elétrico; e B compensa o efeito não linear do dispositivo IM.

- Usando esses valores A ' e B eo valor de t de centroide do ATD dos ambs determinam seus ωc usando a equação 3 e seus ω usando a equação 1. Este método fornece CCSS para as espécies de peptídeos com erros absolutos estimados de cerca de 2%25,26,27.
7. métodos computacionais
- Use o nível B3LYP/LanL2DZ da teoria, compreendendo dos funcionais híbridos do Becke 3-parâmetro30 e a base de advertência ajustou31 e os potenciais do núcleo de elétron32,33,34 para encontrar conformadores otimizados em geometria para todos os tipos possíveis de coordenações da espécie m/z amb observada35.
Observação: para obter detalhes sobre como compilar e enviar cálculos, consulte o uso de GaussView no arquivo suplementar. - Compare a energia livre prevista de cada um dos conformadores e calcule seus CCSs teóricos usando o método Lennard-Jones (LJ) em escala de íons do programa Sigma36.
- A partir dos mais baixos conformadores de energia livre determinar qual conformer exibe o LJ CCS que concorda com o IM-MS medido CCS para identificar a estrutura terciária e tipo de coordenação para os conformadores observados no experimento.
Resultados
Emperramento do metal de AMB1
O estudo IM-MS20 da AMB1 (Figura 1a) mostrou que os íons cobre e zinco vinculados à AMB1 de forma dependente do pH (Figura 2). No entanto, cobre e zinco vinculados à AMB1 através de diferentes mecanismos de reação em diferentes locais de coordenação. Por exemplo, a adição de UC (II) a AMB1 resultou na oxidaç...
Discussão
Etapas críticas: conservando os comportamentos da solução-fase para o exame através de ESI-IM-MS
As configurações instrumentais do ESI nativo devem ser usadas para conservar os peptídeos estequiometria, estado de carga e estrutura conformacional. Para condições nativas, as condições da fonte ESI, como as tensões do cone, as temperaturas e os fluxos de gás, devem ser otimizadas. Além disso, as pressões e tensões na fonte, armadilha, mobilidade de íons e ondas de viagem de transferênci...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este material é baseado no trabalho apoiado pela Fundação Nacional da ciência 1764436, sustentação do instrumento do NSF (MRI-0821247), Fundação de Welch (T-0014), e recursos computando do departamento de energia (TX-W-20090427-0004-50) e comunicações L3 . Agradecemos ao grupo Bower da Universidade da Califórnia-Santa Barbara por compartilhar o programa Sigma e Ayobami Ilesanmi para demonstrar a técnica no vídeo.
Materiais
| Name | Company | Catalog Number | Comments |
| acetonitrile HPLC-grade | Fisher Scientific (www.Fishersci.com) | A998SK-4 | |
| ammonium hydroxide (trace metal grade) | Fisher Scientific (www.Fishersci.com) | A512-P500 | |
| cobalt(II) chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 255599-5G | |
| copper(II) chloride 99.999% | Sigma-Aldrich (www.sigmaaldrich.com) | 203149-10G | |
| copper(II) nitrate hydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 229636-5G | |
| designed amb1,2,3,4,5,6,7 peptides | Neo BioLab (neobiolab.com) | designed peptides were synthized by order | |
| designed amb5B,C,D,E,F peptides | PepmicCo (www.pepmic.com) | designed peptides were synthized by order | |
| Driftscope 2.1 software program | Waters (www.waters.com) | software analysis program | |
| Freeze-dried, purified, Cu(I)-free mb-OB3b | cultured and isolated in the lab of Dr. DongWon Choi (Biology Department, Texas A&M-Commerce) | ||
| glacial acetic acid (Optima grade) | Fisher Scientific (www.Fishersci.com) | A465-250 | |
| Iron(III) Chloride Anhydrous 98%+ | Alfa Aesar (www.alfa.com) | 12357-09 | |
| lead(II) nitrate ACS grade | Avantor (www.avantormaterials.com) | 128545-50G | |
| manganese(II) chloride tetrahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203734-5G | |
| MassLynx 4.1 | Waters (www.waters.com) | software analysis program | |
| nickel chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203866-5G | |
| poly-DL-alanine | Sigma-Aldrich (www.sigmaaldrich.com) | P9003-25MG | |
| silver nitrate 99.9%+ | Alfa Aesar (www.alfa.com) | 11414-06 | |
| Waters Synapt G1 HDMS | Waters (www.waters.com) | quadrupole - ion mobility- time-of-flight mass spectrometer | |
| zinc chloride anhydrous | Alfa Aesar (www.alfa.com) | A16281 |
Referências
- Dudev, T., Lim, C. Competition among Metal Ions for Protein Binding Sites: Determinants of Metal Ion Selectivity in Proteins. Chemical Reviews. 114 (1), 538-556 (2014).
- Sovago, I., Kallay, C., Varnagy, K. Peptides as complexing agents: Factors influencing the structure and thermodynamic stability of peptide complexes. Coordination Chemistry Reviews. 256 (19-20), 2225-2233 (2012).
- Sóvágó, I., Várnagy, K., Lihi, N., Grenács, &. #. 1. 9. 3. ;. Coordinating properties of peptides containing histidyl residues. Coordination Chemistry Reviews. 327, 43-54 (2016).
- Rubino, J. T., Franz, K. J. Coordination chemistry of copper proteins: How nature handles a toxic cargo for essential function. Journal of Inorganic Biochemistry. 107 (1), 129-143 (2012).
- Robinson, N. J., Winge, D. R. Copper Metallochaperones . Annual Review of Biochemistry. 79, 537-562 (2010).
- Scheiber, I. F., Mercer, J. F. B., Dringen, R. Metabolism and functions of copper in brain. Progress in Neurobiology. 116, 33-57 (2014).
- Tisato, F., Marzano, C., Porchia, M., Pellei, M., Santini, C. Copper in Diseases and Treatments, and Copper-Based Anticancer Strategies. Medicinal Research Reviews. 30 (4), 708-749 (2010).
- Millhauser, G. L. Copper and the prion protein: Methods, structures, function, and disease. Annual Review of Physical Chemistry. 58, 299-320 (2007).
- Arena, G., Pappalardo, G., Sovago, I., Rizzarelli, E. Copper(II) interaction with amyloid-beta: Affinity and speciation. Coordination Chemistry Reviews. 256 (1-2), 3-12 (2012).
- Kim, H. J., et al. Methanobactin, a copper-acquisition compound from methane-oxidizing bacteria. Science. 305 (5690), 1612-1615 (2004).
- Di Spirito, A. A., et al. Methanobactin and the link between copper and bacterial methane oxidation. Microbiology Molecular Biology Reviews. 80 (2), 387-409 (2016).
- Kenney, G. E., Rosenzweig, A. C. Chemistry and biology of the copper chelator methanobactin. ACS Chemical Biology. 7 (2), 260-268 (2012).
- Summer, K. H., et al. The biogenic methanobactin is an effective chelator for copper in a rat model for Wilson disease. Journal of Trace Elements in Medicine and Biology. 25 (1), 36-41 (2011).
- Hachmoeller, O., et al. Investigating the influence of standard staining procedures on the copper distribution and concentration in Wilson's disease liver samples by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 44, 71-75 (2017).
- Hachmoeller, O., et al. Spatial investigation of the elemental distribution in Wilson's disease liver after D-penicillamine treatment by LA-ICP-MS. Journal of Trace Elements in Medicine and Biology. 44, 26-31 (2017).
- Hachmoeller, O., et al. Element bioimaging of liver needle biopsy specimens from patients with Wilson's disease by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 35, 97-102 (2016).
- Mueller, J. C., Lichtmannegger, J., Zischka, H., Sperling, M., Karst, U. High spatial resolution LA-ICP-MS demonstrates massive liver copper depletion in Wilson disease rats upon Methanobactin treatment. Journal of Trace Elements in Medicine and Biology. 49, 119-127 (2018).
- Choi, D. W., et al. Spectral and thermodynamic properties of Ag(I), Au(III), Cd(II), Co(II), Fe(III), Hg(II), Mn(II), Ni(II), Pb(II), U(IV), and Zn(II) binding by methanobactin from Methylosinus trichosporium OB3b. Journal of Inorganic Biochemistry. 100, 2150-2161 (2006).
- McCabe, J. W., Vangala, R., Angel, L. A. Binding Selectivity of Methanobactin from Methylosinus trichosporium OB3b for Copper(I), Silver(I), Zinc(II), Nickel(II), Cobalt(II), Manganese(II), Lead(II), and Iron(II). Journal of the American Society of Mass Spectrometry. 28, 2588-2601 (2017).
- Sesham, R., et al. The pH dependent Cu(II) and Zn(II) binding behavior of an analog methanobactin peptide. European Journal of Mass Spectrometry. 19 (6), 463-473 (2013).
- Wagoner, S. M., et al. The multiple conformational charge states of zinc(II) coordination by 2His-2Cys oligopeptide investigated by ion mobility - mass spectrometry, density functional theory and theoretical collision cross sections. Journal of Mass Spectrom. 51 (12), 1120-1129 (2016).
- Bandow, N. L., et al. Isolation of methanobactin from the spent media of methane-oxidizing bacteria. Methods in Enzymology. 495, 259-269 (2011).
- Choi, D. W., et al. Spectral and thermodynamic properties of methanobactin from γ-proteobacterial methane oxidizing bacteria: a case for copper competition on a molecular level. Journal of Inorganic Biochemistry. 104 (12), 1240-1247 (2010).
- Pringle, S. D., et al. An investigation of the mobility separation of some peptide and protein ions using a new hybrid quadrupole/travelling wave IMS/oa-ToF instrument. International Journal of Mass Spectrometry. 261 (1), 1-12 (2007).
- Forsythe, J. G., et al. Collision cross section calibrants for negative ion mode traveling wave ion mobility-mass spectrometry. Analyst. 14 (20), 6853-6861 (2015).
- Allen, S. J., Giles, K., Gilbert, T., Bush, M. F. Ion mobility mass spectrometry of peptide, protein, and protein complex ions using a radio-frequency confining drift cell. Analyst. 141 (3), 884-891 (2016).
- Bush, M. F., Campuzano, I. D. G., Robinson, C. V. Ion Mobility Mass Spectrometry of Peptide Ions: Effects of Drift Gas and Calibration Strategies. Analytical Chemistry. 84 (16), 7124-7130 (2012).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Smith, D. P., et al. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. European Journal of Mass Spectrometry. 15 (2), 113-130 (2009).
- Becke, A. D. Density-functional thermochemistry. III. The role of exact exchange. Journal of Chemical Physics. 98 (7), 5648-5652 (1993).
- Dunning, T. H., Hay, P. J. Gaussian basis sets for molecular calculations. Modern Theoretical Chemistry. 3, 1-27 (1977).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for potassium to gold including the outermost core orbitals. Journal of Chemical Physics. 82 (1), 299-310 (1985).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for the transition metal atoms scandium to mercury. Journal of Chemical Physics. 82 (1), 270-283 (1985).
- Wadt, W. R., Hay, P. J. Ab initio effective core potentials for molecular calculations. Potentials for main group elements sodium to bismuth. Journal of Chemical Physics. 82 (1), 284-298 (1985).
- . Gaussian 09, Revision C.01. Gaussian, Inc. , (2012).
- Wyttenbach, T., von Helden, G., Batka, J. J., Carlat, D., Bowers, M. T. Effect of the long-range potential on ion mobility measurements. Journal of the American Society of Mass Spectrometry. 8 (3), 275-282 (1997).
- Choi, D., et al. Redox activity and multiple copper(I) coordination of 2His-2Cys oligopeptide. Journal of Mass Spectrometry. 50 (2), 316-325 (2015).
- Rigo, A., et al. Interaction of copper with cysteine: stability of cuprous complexes and catalytic role of cupric ions in anaerobic thiol oxidation. Journal of Inorganic Biochemistry. 98 (9), 1495-1501 (2004).
- Vytla, Y., Angel, L. A. Applying Ion Mobility-Mass Spectrometry Techniques for Explicitly Identifying the Products of Cu(II) Reactions of 2His-2Cys Motif Peptides. Analytical Chemistry. 88 (22), 10925-10932 (2016).
- Choi, D., Sesham, R., Kim, Y., Angel, L. A. Analysis of methanobactin from Methylosinus trichosporium OB3b via ion mobility mass spectrometry. European Journal of Mass Spectrometry. 18 (6), 509-520 (2012).
- Martell, A. E., Motekaitis, R. J. NIST Standard Reference Database 46. Institute of Standards and Technology. , (2001).
- Pesch, M. L., Christl, I., Hoffmann, M., Kraemer, S. M., Kretzschmar, R. Copper complexation of methanobactin isolated from Methylosinus trichosporium OB3b: pH-dependent speciation and modeling. Journal of Inorganic Biochemistry. 116, 55-62 (2012).
- Amin, E. A., Truhlar, D. G. Zn Coordination Chemistry: Development of Benchmark Suites for Geometries, Dipole Moments, and Bond Dissociation Energies and Their Use To Test and Validate Density Functionals and Molecular Orbital Theory. Journal of Chemical Theory and Computation. 4 (1), 75-85 (2008).
- Sorkin, A., Truhlar, D. G., Amin, E. A. Energies, Geometries, and Charge Distributions of Zn Molecules, Clusters, and Biocenters from Coupled Cluster, Density Functional, and Neglect of Diatomic Differential Overlap Models. Journal of Chemical Theory and Computation. 5 (5), 1254-1265 (2009).
- Lillo, V., Galan-Mascaros, J. R. Transition metal complexes with oligopeptides: single crystals and crystal structures. Dalton Transactions. 43 (26), 9821-9833 (2014).
- Choutko, A., van Gunsteren, W. F. Conformational Preferences of a beta-Octapeptide as Function of Solvent and Force-Field Parameters. Helvetica Chimica Acta. 96 (2), 189-200 (2013).
- Angel, L. A. Study of metal ion labeling of the conformational and charge states of lysozyme by ion mobility mass spectrometry. European Journal of Mass Spectrometry. 17 (3), 207-215 (2011).
- Kelso, C., Rojas, J. D., Furlan, R. L. A., Padilla, G., Beck, J. L. Characterisation of anthracyclines from a cosmomycin D-producing species of Streptomyces by collisionally-activated dissociation and ion mobility mass spectrometry. European Journal of Mass Spectrometry. 15 (2), 73-81 (2009).
- El Ghazouani, A., et al. Copper-binding properties and structures of methanobactins from Methylosinus trichosporium OB3b. Inorganic Chemistry. 50 (4), 1378-1391 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados