JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
금속 결합 올리고펩타이드의 금속 이온 인식 및 Redox 활성의 구조 및 메커니즘을 결정하기 위한 이온 이동성 질량 분광법 기술
요약
이온 이동성 질량 분광법 및 분자 모델링 기술은 설계된 금속 결합 펩타이드 및 구리 결합 펩티드 메탄오박틴의 선택적 금속 킬레이트화 성능을 특성화할 수 있다. 금속 킬레이트 펩티드의 새로운 클래스를 개발하는 것은 금속 이온 과다 균형과 관련된 질병에 대한 치료제로 이어질 도움이 될 것입니다.
초록
전기 분무 이온화 (ESI)는 질량, 전체 전하, 금속 결합 상호 작용 및 형태 모양을 보존하면서 수성 상 펩타이드 또는 펩타이드 복합체를 가스 상으로 전달할 수 있습니다. ESI와 이온 이동성 질량 분석법(IM-MS)을 결합하면 화학조량계, 프로토네이션 상태와 관련된 펩타이드의 질량 대 충전(m/z) 및 충돌 단면(CCS)을 동시에 측정할 수 있는 기법 기술을 제공합니다. 및 형태 모양. 펩티드 복합체의 전체 전하는 1) 펩티드의 산성 및 기본 부위및 2) 금속 이온의 산화 상태의 프로토네이션에 의해 조절된다. 따라서, 복합체의 전체 전하 상태는 펩티드 금속 이온 결합 친화도에 영향을 미치는 용액의 pH의 함수이다. ESI-IM-MS 분석의 경우, 펩타이드 및 금속 이온 용액은 수성 전용 솔루션에서 제조되며, pH는 희석 된 수성 아세트산 또는 수산화 암모늄으로 조정됩니다. 이는 pH 의존성 및 금속 이온 선택성을 특정 펩티드에 대해 결정하도록 허용한다. 또한 펩타이드 복합체의 m/z 및 CCS는 B3LYP/LanL2DZ 분자 모델링과 함께 사용하여 복합체의 금속 이온 조정 및 삼차 구조의 결합 부위를 식별할 수 있습니다. 결과는 ESI-IM-MS가 대체 메탄백틴 펩타이드 세트의 선택적 킬레이트화 성능을 특성화하고 이를 구리 결합 펩티드 메탄노박틴과 비교할 수 있는 방법을 보여준다.
서문
구리 와 아연 이온은 살아있는 유기체에 필수적이며 산화 보호, 조직 성장, 호흡, 콜레스테롤, 포도당 대사 및 게놈 판독을 포함한 과정에 매우 중요합니다1. 이러한 기능을 활성화하기 위해, Cys의 티오레이트, 그의2,3의imidazole , (더 드물게) 메티오닌의 티오에테르, 글루와 Asp의 카르복실레이트와 같은 그룹은 금속을 활성 부위에 보조 인자로 선택적으로 통합합니다. 금속 효소. 이러한 조정 그룹의 유사성은 그와 Cys 리간드가 올바른 기능을 보장하기 위해 Cu (I / II) 또는 Zn (II)을 선택적으로 통합하는 방법에 관한 흥미로운 질문을 제기합니다.
선택적 결합은 종종 Zn (II) 또는 Cu (I / II) 이온 농도를 제어하는 펩티드를 획득하고 인신 매매하여 수행됩니다4. Cu (I/II)는 반응성이 높고 효소에 산화 손상 또는 모험적 결합을 일으키므로 자유 농도는 구리 보호막과 구리 조절 단백질에 의해 단단히 조절되어 세포의 다양한 위치로 안전하게 운반하고 단단히 운반합니다. 그것의 항상성제어 5,6. 구리 대사 또는 항상성의 중단은 Menkes 및 Wilson의 질병7뿐만 아니라 암7 및 신경 질환, 예컨대 프리온8 및 알츠하이머 병9에직접적으로 연루된다.
윌슨의 질병은 눈의 증가 구리 수준과 관련, 간 및 뇌의 섹션, Cu의 레독스 반응 (I/II) 반응성 산소 종을 생산, 간 장 성 및 신경 변성을 일으키는. 기존 킬레이트 요법은 작은 티올 아미노산 페니실라민과 트리에틸네테트라민입니다. 대안적으로, 메탄영양구리-펩타이드(mb)10,11은 Cu(I)에 대한 그들의 높은 결합 친화도 때문에 치료적 잠재력을나타낸다.12. 메틸로시누스 트리코스포륨 OB3b로부터의 메탄백박틴(mb-OB3b)이 윌슨병의 동물 모델에서 연구되었을 때, 구리는 간에서 효율적으로 제거되고 담즙13을통해 배설되었다. 시험관내 실험에서 mb-OB3b는 간시토졸(13)에함유된 구리 메탈로티오닌으로부터 구리를 킬레이트할 수 있음을 확인하였다. 레이저 절제 유도 결합 플라즈마 질량 분광법 이미징 기술은 윌슨 병 간 샘플14,15,16에서 구리의 공간 분포를 조사하고 MB-OB3b를 나타내고 있다. 단 8 일17의짧은 처리 기간으로 구리를 제거합니다.
mb-OB3b는 또한 Ag(I), Au(III), Pb(II), Mn(II), Co(II), Fe(II), Ni(II) 및 Zn(II)18,19를포함하는 다른 금속 이온과 결합한다. 생리학적 Cu(I) 결합 부위에 대한 경쟁은 Mb-OB3b 복합체로부터 Cu(I)를 대체할 수 있기 때문에 Ag(I)에 의해 전시되며, Ag(I) 및 Ni(II)는 또한 Cu(I)19에의해 변위될 수 없는 MB에 돌이킬 수 없는 결합을 나타낸다. 최근, 2His-2Cys 결합 모티프를 가진 일련의 대체 메탄백액틴(amb) 올리고펩티드가20,21,및 그들의 Zn(II) 및 Cu(I/II) 결합 특성을 특징으로 하는 것으로 연구되었다. 그들의 1 차적인 아미노산 서열은 유사하고, 그들은 모두 2His-2Cys 모티프, 프로 및 아세틸화 된 N-종단을 함유하고 있습니다. 그들은 주로 2His-2Cys 모티프가 mb-OB3b의 두 enethiol 옥사졸론 결합 부위를 대체하기 때문에 mb-OB3b와 주로 다릅니다.
이온 이동성 질량 분석법(ESI-IM-MS)과 결합된 전기 분무 이온화는 질량 대충전(m/z)및 충돌을 측정하기 때문에 펩타이드의 금속 결합 특성을 결정하는 강력한 기법을 제공합니다. 용액 상에서 질량, 전하 및 형태 형상을 보존하면서 단면(CCS)을 m/z 및 CCS는 펩티드 stoichiometry, protonation 상태 및 형태 모양과 관련이 있습니다. Stoichiometry는 종에 존재하는 각 요소의 정체성 그리고 수가 명시적으로 식별되기 때문에 결정됩니다. 펩티드 복합체의 전체 전하는 산성 및 기본 부위의 프로토네이션 상태 및 금속 이온의 산화 상태에 관한 것이다. CCS는 복합체의 삼차 구조와 관련된 회전 평균 크기를 측정하기 때문에 펩타이드 복합체의 형태 형성에 대한 정보를 제공한다. 복합체의 전체 전하 상태는 또한 pH의 기능이며, 카르복실, 그의, Cys 및 Tyr와 같은 탈프로톤화된 기본 또는 산성 부위가 또한 금속 이온에 대한 잠재적 결합 부위이기 때문에 펩티드의 금속 이온 결합 친화도에 영향을 미친다. 분석을 위해 펩타이드 및 금속 이온은 희석 된 아세트산 또는 수산화 암모늄에 의해 조정 된 pH로 수성 용액으로 제조됩니다. 이는 펩티드에 대해 pH 의존성 및 금속 이온 선택성을 결정할 수 있게 한다. 또한 ESI-IM-MS에 의해 결정된 m/z 및 CCS는 B3LYP/LanL2DZ 분자 모델링과 함께 사용하여 복합체의 금속 이온 조정 및 삼차 구조의 유형을 발견할 수 있습니다. 이 문서에 나타난 결과는 ESI-IM-MS가 암펩티드 세트의 선택적 킬레이트화 성능을 특성화하고 이를 구리 결합 펩티드 mb-OB3b와 비교할 수 있는 방법을 보여줍니다.
프로토콜
1. 시약의 준비
- 배양 메틸로시누스 트리코스포륨 OB3b는 Cu(I)-무음 MB-OB3b18,22,23,동결건조시 시료를 동결건조시키고 사용전까지 -80°C에서 보관한다.
- 앰브 펩티드 (amb;98 % 순도 amb1,amb2,amb4;>70% 순도 amb7)샘플을 동결 건조하고 사용할 때까지 -80 °C에 보관하십시오.
- 구매 >98% 순도 망간 (II) 염화물, 코발트(II) 염화물, 니클(II) 염화물, 구리(II) 염화물, 구리(II) 질산염, 은(I) 질산염, 아연(II) 염화물, 철(III) 염화물 및 납(II) 염화물.
- 암종 및 HPLC 등급 이상의 수산화암모늄, 빙하 아세트산 및 아세토니트릴의 충돌 단면을 측정하기 위한 캘리브란트로 사용되는 폴리-DL-알라닌 폴리머를 구입한다.
2. 재고 솔루션 준비
-
펩타이드 스톡 솔루션
- 1.7 mL 플라스틱 바이알에서 MB-OB3b 또는 앰브의 질량10.0-20.0 mg의 질량을 사용하여 적어도 3개의 유의한 수치를 사용하여 정확하게 계량한다.
참고: 계량된 질량은 1.00 mL의 탈이온화(DI) 물이 첨가될 때 펩티드의 용해도에 따라 12.5 mM 또는 1.25 mM을 산출해야 한다. - 파이펫을 사용하여 1.00 mL의 탈이온수 (>17.8 MΩ cm)를 계량 된 펩티드 샘플에 추가하여 12.5 mM 또는 1.25 mM 용액을 산출합니다. 캡을 단단히 놓고 적어도 20 개의 반전과 철저히 섞습니다.
- 마이크로파이펫 디스펜스 50.0 μL aliquots를 펩티드 샘플로부터 개별적으로 라벨이 부착된 1.5 mL 바이알로 추출하고 사용 전까지 -80°C에 보관하였다.
- 1.7 mL 플라스틱 바이알에서 MB-OB3b 또는 앰브의 질량10.0-20.0 mg의 질량을 사용하여 적어도 3개의 유의한 수치를 사용하여 정확하게 계량한다.
-
금속 이온 스톡 솔루션
- 1.7 mL 바이알에서 적어도 3개의 유의한 수치를 사용하여, 금속 염화물 또는 질산은 10.0-30.0 mg의 질량을 사용하여 정확하게 계량한다.
참고: 1.00 mL의 DI 물을 추가하면 계량 질량은 125 mM을 산출해야합니다. - 1.7 mL 바이알의 계량 된 금속 샘플에 1.00 mL의 DI 물을 추가하여 125 mM 용액을 산출하십시오. 캡을 단단히 놓고 적어도 20 개의 반전과 철저히 섞습니다.
- 1.7 mL 바이알에서 적어도 3개의 유의한 수치를 사용하여, 금속 염화물 또는 질산은 10.0-30.0 mg의 질량을 사용하여 정확하게 계량한다.
- 수산화 암모늄 스톡 솔루션: DI 물로 99.5 % 아세트산 용액의 57 μL을 최종 부피 1.00 mL로 희석하여 1.0 M 아세트산 용액을 준비합니다. DI 물로 21% 수산화암모늄 용액의 90 μL을 1.00 mL의 최종 부피로 희석하여 1.0 M 수산화 암모늄 용액을 준비합니다. 1.0 M 용액의 100 μL을 복용하여 0.10 M 및 0.010 M 아세트산 및 수산화 암모늄 용액을 사용하여 각 용액을 연속적으로 희석시다.
- 폴리-DL-알라닌 스톡 솔루션: PA 1.0 mg의 무게를 측정하고 1.0 mL의 DI 물에 용해하여 폴리 DL 알라닌 (PA)을 준비하여 1,000 ppm을 제공합니다. 철저히 섞으세요. 마이크로파이펫을 사용하여 50.0 μL 의 별칭을 분배하고 각각 1.7 mL 바이알에 넣고 -80°C에 보관합니다.
3. 전기 분무 이온 이동성 질량 분석
- ESI 입구 튜브와 바늘 모세관을 0.1 M 빙하 아세트산, 0.1 M 암모늄 수산화물, 그리고 마지막으로 DI 물로 약 500 μL로 철저히 청소합니다.
- 1,000 ppm PA 스톡 용액의 50.0 μL aliquot를 해동하고 450 μL의 DI 물로 희석하여 100 ppm PA를 제공합니다. 이 용액의 파이펫 100.0 μL을 1.00 mL로 희석하고 DI 물 500 μL과 아세토니트릴 500 μL로 10 ppm PA 용액을 제공합니다.
- 토론 섹션에 기재된 바와 같이 네이티브 ESI-IM-MS 조건을 사용하여 각각 10 ppm PA 용액의 음수 및 양성 이온 IM-MS 스펙트럼을 수집한다.
- 12.5 mM 또는 1.25 mM 의 50.0 μL aliquot를 해동하고 DI 물로 연속희석하여 0.125 mM 의 최종 농도를 부여합니다. 희석할 때마다 완전히 섞으세요.
- 125 mM 금속 이온 스톡 용액의 파이펫 100.0 μL, 1.7 mL 바이알에 놓고 DI 물로 1.00 mL로 희석하여 12.5 mM 금속 이온을 제공합니다. 최종 0.125 mM 금속 이온 농도를 제공하기 위해 두 개의 연속 희석을 반복합니다. 희석할 때마다 완전히 섞으세요.
- 0.125 mM amb의 파이펫 200.0 μL을 1.7 mL 바이알에 넣고 500 μL의 DI 물로 희석하고 용액을 철저히 혼합합니다.
- 1.0 M 아세트산 용액의 50 μL을 추가하여 시료의 pH를 3.0으로 조정합니다.
- 0.125 mM 금속 이온의 200.0 μL을 pH 조정 샘플에 추가합니다. DI 물을 추가하여 시료의 최종 부피를 1.00 mL산출하고, 철저히 혼합하고, RT에서 10분 동안 시료가 평형화되도록 하십시오.
- 무딘 코 주사기를 사용하여 샘플의 500 μL을 취하고 각각 5 분 동안 음의 양성 이온 ES-IM-MS 스펙트럼을 수집합니다. 시료의 나머지 500 μL을 사용하여 교정된 마이크로 pH 전극을 사용하여 최종 pH를 기록합니다.
- 3.6-3.9단계를 반복하면서 pH를 4.0, 5.0, 6.0, 7.0, 8.0, 9.0 또는 10.0으로 조정하면서 0.010 M, 0.10 M, 또는 1.0 M 아세트산 또는 암모늄 수산화액의 새로운 부피를 추가한다.
- 각각 10 ppm PA 용액의 음수 및 양성 이온 ESI-IM-MS 스펙트럼을 수집합니다.
4. 암두 시료의 금속 이온 적정 준비
- 3.1-3.5 단계에 설명된 단계를 따릅니다.
- 0.125 mM amb의 파이펫 200.0 μL을 1.7 mL 바이알에 넣고 500.0 μL의 DI 물로 희석하고 용액을 철저히 혼합합니다.
- 0.010 M 수산화 암모늄 용액의 80 μL을 첨가하여 시료의 pH를 pH = 9.0으로 조정한다.
- 0.125 mM 금속 이온 용액의 28 μL을 추가하여 금속 이온의 0.14 어금니와 동등한 것을 주고, DI 물을 추가하여 샘플의 최종 부피를 1.00 mL로 만들고, 철저히 혼합하고, RT에서 10 분 동안 평형을 처리합니다.
- 무딘 코 주사기를 사용하여 샘플의 500 μL을 취하고 각각 5 분 동안 음의 양성 이온 ESI-IM-MS 스펙트럼을 수집합니다. 시료의 나머지 500 μL을 사용하여 교정된 마이크로 pH 전극을 사용하여 최종 pH를 기록합니다.
- 4.2-4.5단계를 반복하면서, 0.125 mM 금속 이온 용액의 적절한 부피를 추가하여 0.28, 0.42, 0.56, 0.70, 0.84, 0.98, 1.12, 1.26, 또는 1.40 모금 등가물을 부여한다.
- 각각 10 ppm PA 용액의 음수 및 양성 이온 IM-MS 스펙트럼을 수집한다.
5. ESI-IM-MS pH 적정 데이터 분석
- IM-MS 스펙트럼에서 암스의 충전 된 종은 이론적 인 m / z 동위 원소 패턴과 일치하여 존재하는 것을 식별합니다.
- MassLynx를 열고 크로마토그램창을 클릭하여 크로마토그램 창을 엽니다.
- 파일 메뉴로 이동하여 열기로 이동하여 IM-MS 데이터 파일을 찾아 엽니다.
- 오른쪽 단추로 클릭하고 크로마토그램을 가로질러 드래그하여 IM-MS 스펙트럼을 추출하고 해제합니다. IM-MS 스펙트럼을 보여주는 스펙트럼 창이 열립니다.
- 스펙트럼 창에서 도구 및 동위원소 모델을클릭합니다. 동위원소 모델링 창에서 앰브 종의 분자 공식을 입력하고 충전 된 이온 표시 상자를 확인하고 충전 상태를 입력합니다. 확인을 클릭합니다.
- 반복하여 IM-MS 스펙트럼의 모든 종을 식별하고 m/z 동위원소 범위를 기록합니다.
- 각 암종종에 대해 우연한 m/z 종을 분리하고 m/z 동위원소 패턴을 사용하여 도착 시간 분포(ATD)를 추출하여 식별합니다.
- MassLynx에서 프로그램을 엽니 다 드리프트 스코프를 클릭합니다. 드리프트 스코프에서 파일을 클릭하고 열기를 클릭하여 IM-MS 데이터 파일을 찾아 엽니다.
- 마우스를 사용하고 왼쪽 클릭을 사용하여 amb 종의 m/z 동위원소 패턴을 확대합니다.
- 선택 도구와 왼쪽 마우스 단추를 사용하여 동위원소 패턴을 선택합니다. 현재 선택 수락 단추를 클릭합니다.
- 우연히 m/z 종을 분리하려면 선택 도구와 왼쪽 마우스 버튼을 사용하여 AMB 종의 동위원소 패턴과 정렬된 ATD 시간을 선택합니다. 현재 선택 수락 단추를 클릭합니다.
- ATD를 내보내려면 FILE | 으로 이동 MassLynx로 내보낸다음 드리프트 시간 유지를 선택하고 파일을 적절한 폴더에 저장합니다.
- ATD의 중심을 결정하고 종 인구의 척도로 ATD 곡선 아래의 영역을 통합합니다.
- MassLynx의 크로마토그램 창에서 저장된 내보낸 파일을 엽니다. 프로세스 | 클릭 메뉴에서 통합합니다. ApexTrack 피크 통합 확인란을 선택하고 확인을클릭합니다.
- 크로마토그램 창에 표시된 대로 중심 ATD(tA)와통합 영역을 기록합니다. 저장된 모든 앰브 및 PA IM-MS 데이터 파일에 대해 반복합니다.
- 각 적정 점에서 양수 또는 음이온의 추출된 모든 암브 종에 대해 통합 ATD를 사용하여 상대백분율 척도로 정규화합니다.
- 각 pH에서 앰브 종의 ID와 통합 ATD를 스프레드시트에 입력합니다.
- 각 pH에 대해 통합 ATD의 합계를 사용하여 개별 앰브의 ATD를 백분율 척도로 정규화합니다.
- 그래프에서 각 암종 과 pH의 백분율 강도를 플롯하여 각 종의 개체수가 pH 의 함수로 어떻게 변하는지 보여줍니다.
6. 충돌 단면
- 스프레드시트를 사용하여, HE 버퍼 가스28에서 측정된 PA 음수25,26 및 양수27 이온의 CCS(Ω)를 아래 방정식 1을 사용하여 수정된 CCS(Ωc)로변환합니다. 전자 (것)이 c = 전자 전하 (1.602 × 10-19 C); m (것)m N 2 = N2 가스 (Da)의 질량; m 이온 = 이온 질량. 29세
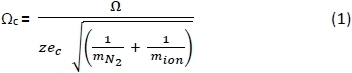
- PA 캘리브란트 및 앰브 종의 평균 도착 시간(tA)을아래 방정식 2를 사용하여 드리프트 시간(tD)으로변환합니다: c= 향상된 듀티 사이클 지연 계수(1.41) 및 m/z는 펩티드 이온의 질량-전하이다.

- PA 교정란트의 tD 대 Ωc를플로팅합니다. 그런 다음 아래 그림과 같은 방정식 3의 최소 제곱 회귀 적합을 사용하여 A' 및 B 값을 결정합니다. 및 B는 IM 장치의 비선형 효과를 보정합니다.

- 이러한 A' 및 B 값과 앰브의 ATD에서 중심 tD 값을 사용하면 방정식 3을 사용하여 Ω c와 방정식 1을사용하여Ω을 결정합니다. 이 방법은 약 2%25,26,27의추정 절대 오차를 가진 펩티드 종에 대한 CCS를 제공한다.
7. 전산 방법
- 베케 3 파라미터 하이브리드기능(30)과 Dunning 기초 세트31 및 전자 코어 전위32,33,34로 구성된 B3LYP/LanL2DZ 이론 레벨을 사용하여 찾을 수 있습니다. 관측 된 m / z amb 종35의모든 가능한 조정 유형에 대한 지오메트리 최적화 된 응수 .
참고: 계산을 빌드하고 제출하는 방법에 대한 자세한 내용은 보충 파일의GaussView 사용을 참조하십시오. - 각각의 응셈자의 예측된 자유 에너지를 비교하고 시그마프로그램(36)으로부터이온 스케일렌나드 존스(LJ) 방법을 사용하여 그들의 이론적 CCS를 계산한다.
- 가장 낮은 자유 에너지 순응자로부터 실험에서 관찰된 응수자에 대한 삼차 구조 및 조정 유형을 식별하기 위해 IM-MS 측정 CCS에 동의하는 LJ CCS를 나타내는 컨포머가 결정됩니다.
결과
Amb1의 금속 결합
IM-MS 연구20 amb1 (그림 1A)은구리 와 아연 이온 이온이 모두 pH 의존적 방식으로 amb1에 결합되는 것으로 나타났다(그림2). 그러나, 구리와 아연은 상이한 조정 부위에서 상이한 반응 메커니즘을 통해 amb1에 결합한다. 예를 들어, Cu(II)를 amb1에 첨가하면 이...
토론
중요한 단계: ESI-IM-MS를 통한 검사를 위한 솔루션 단계 거동 보존
네이티브 ESI 기악 설정은 펩티드 stoichiometry, 전하 상태 및 형태 구조를 보존하는 데 사용되어야 합니다. 기본 조건의 경우 원뿔 전압, 온도 및 가스 흐름과 같은 ESI 소스의 조건을 최적화해야 합니다. 또한 소스, 트랩, 이온 이동성 및 전송 이동 파(특히 IM 셀로의 사출 전압을 제어하는 DC 트랩 바이어스)의 압력과 전압?...
공개
저자는 공개 할 것이 없다.
감사의 말
이 자료는 1764436, NSF 기기 지원(MRI-0821247), 웰치 재단(T-0014), 에너지부(TX-W-20090427-0004-50) 및 L3 통신의 컴퓨팅 리소스에 따라 국립 과학 재단이 지원하는 작업을 기반으로 합니다. . 우리는 시그마 프로그램과 아요바미 Ilesanmi비디오에서 기술을 시연에 대한 공유 산타 바바라 캘리포니아 대학의 바우어의 그룹에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| acetonitrile HPLC-grade | Fisher Scientific (www.Fishersci.com) | A998SK-4 | |
| ammonium hydroxide (trace metal grade) | Fisher Scientific (www.Fishersci.com) | A512-P500 | |
| cobalt(II) chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 255599-5G | |
| copper(II) chloride 99.999% | Sigma-Aldrich (www.sigmaaldrich.com) | 203149-10G | |
| copper(II) nitrate hydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 229636-5G | |
| designed amb1,2,3,4,5,6,7 peptides | Neo BioLab (neobiolab.com) | designed peptides were synthized by order | |
| designed amb5B,C,D,E,F peptides | PepmicCo (www.pepmic.com) | designed peptides were synthized by order | |
| Driftscope 2.1 software program | Waters (www.waters.com) | software analysis program | |
| Freeze-dried, purified, Cu(I)-free mb-OB3b | cultured and isolated in the lab of Dr. DongWon Choi (Biology Department, Texas A&M-Commerce) | ||
| glacial acetic acid (Optima grade) | Fisher Scientific (www.Fishersci.com) | A465-250 | |
| Iron(III) Chloride Anhydrous 98%+ | Alfa Aesar (www.alfa.com) | 12357-09 | |
| lead(II) nitrate ACS grade | Avantor (www.avantormaterials.com) | 128545-50G | |
| manganese(II) chloride tetrahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203734-5G | |
| MassLynx 4.1 | Waters (www.waters.com) | software analysis program | |
| nickel chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203866-5G | |
| poly-DL-alanine | Sigma-Aldrich (www.sigmaaldrich.com) | P9003-25MG | |
| silver nitrate 99.9%+ | Alfa Aesar (www.alfa.com) | 11414-06 | |
| Waters Synapt G1 HDMS | Waters (www.waters.com) | quadrupole - ion mobility- time-of-flight mass spectrometer | |
| zinc chloride anhydrous | Alfa Aesar (www.alfa.com) | A16281 |
참고문헌
- Dudev, T., Lim, C. Competition among Metal Ions for Protein Binding Sites: Determinants of Metal Ion Selectivity in Proteins. Chemical Reviews. 114 (1), 538-556 (2014).
- Sovago, I., Kallay, C., Varnagy, K. Peptides as complexing agents: Factors influencing the structure and thermodynamic stability of peptide complexes. Coordination Chemistry Reviews. 256 (19-20), 2225-2233 (2012).
- Sóvágó, I., Várnagy, K., Lihi, N., Grenács, &. #. 1. 9. 3. ;. Coordinating properties of peptides containing histidyl residues. Coordination Chemistry Reviews. 327, 43-54 (2016).
- Rubino, J. T., Franz, K. J. Coordination chemistry of copper proteins: How nature handles a toxic cargo for essential function. Journal of Inorganic Biochemistry. 107 (1), 129-143 (2012).
- Robinson, N. J., Winge, D. R. Copper Metallochaperones . Annual Review of Biochemistry. 79, 537-562 (2010).
- Scheiber, I. F., Mercer, J. F. B., Dringen, R. Metabolism and functions of copper in brain. Progress in Neurobiology. 116, 33-57 (2014).
- Tisato, F., Marzano, C., Porchia, M., Pellei, M., Santini, C. Copper in Diseases and Treatments, and Copper-Based Anticancer Strategies. Medicinal Research Reviews. 30 (4), 708-749 (2010).
- Millhauser, G. L. Copper and the prion protein: Methods, structures, function, and disease. Annual Review of Physical Chemistry. 58, 299-320 (2007).
- Arena, G., Pappalardo, G., Sovago, I., Rizzarelli, E. Copper(II) interaction with amyloid-beta: Affinity and speciation. Coordination Chemistry Reviews. 256 (1-2), 3-12 (2012).
- Kim, H. J., et al. Methanobactin, a copper-acquisition compound from methane-oxidizing bacteria. Science. 305 (5690), 1612-1615 (2004).
- Di Spirito, A. A., et al. Methanobactin and the link between copper and bacterial methane oxidation. Microbiology Molecular Biology Reviews. 80 (2), 387-409 (2016).
- Kenney, G. E., Rosenzweig, A. C. Chemistry and biology of the copper chelator methanobactin. ACS Chemical Biology. 7 (2), 260-268 (2012).
- Summer, K. H., et al. The biogenic methanobactin is an effective chelator for copper in a rat model for Wilson disease. Journal of Trace Elements in Medicine and Biology. 25 (1), 36-41 (2011).
- Hachmoeller, O., et al. Investigating the influence of standard staining procedures on the copper distribution and concentration in Wilson's disease liver samples by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 44, 71-75 (2017).
- Hachmoeller, O., et al. Spatial investigation of the elemental distribution in Wilson's disease liver after D-penicillamine treatment by LA-ICP-MS. Journal of Trace Elements in Medicine and Biology. 44, 26-31 (2017).
- Hachmoeller, O., et al. Element bioimaging of liver needle biopsy specimens from patients with Wilson's disease by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 35, 97-102 (2016).
- Mueller, J. C., Lichtmannegger, J., Zischka, H., Sperling, M., Karst, U. High spatial resolution LA-ICP-MS demonstrates massive liver copper depletion in Wilson disease rats upon Methanobactin treatment. Journal of Trace Elements in Medicine and Biology. 49, 119-127 (2018).
- Choi, D. W., et al. Spectral and thermodynamic properties of Ag(I), Au(III), Cd(II), Co(II), Fe(III), Hg(II), Mn(II), Ni(II), Pb(II), U(IV), and Zn(II) binding by methanobactin from Methylosinus trichosporium OB3b. Journal of Inorganic Biochemistry. 100, 2150-2161 (2006).
- McCabe, J. W., Vangala, R., Angel, L. A. Binding Selectivity of Methanobactin from Methylosinus trichosporium OB3b for Copper(I), Silver(I), Zinc(II), Nickel(II), Cobalt(II), Manganese(II), Lead(II), and Iron(II). Journal of the American Society of Mass Spectrometry. 28, 2588-2601 (2017).
- Sesham, R., et al. The pH dependent Cu(II) and Zn(II) binding behavior of an analog methanobactin peptide. European Journal of Mass Spectrometry. 19 (6), 463-473 (2013).
- Wagoner, S. M., et al. The multiple conformational charge states of zinc(II) coordination by 2His-2Cys oligopeptide investigated by ion mobility - mass spectrometry, density functional theory and theoretical collision cross sections. Journal of Mass Spectrom. 51 (12), 1120-1129 (2016).
- Bandow, N. L., et al. Isolation of methanobactin from the spent media of methane-oxidizing bacteria. Methods in Enzymology. 495, 259-269 (2011).
- Choi, D. W., et al. Spectral and thermodynamic properties of methanobactin from γ-proteobacterial methane oxidizing bacteria: a case for copper competition on a molecular level. Journal of Inorganic Biochemistry. 104 (12), 1240-1247 (2010).
- Pringle, S. D., et al. An investigation of the mobility separation of some peptide and protein ions using a new hybrid quadrupole/travelling wave IMS/oa-ToF instrument. International Journal of Mass Spectrometry. 261 (1), 1-12 (2007).
- Forsythe, J. G., et al. Collision cross section calibrants for negative ion mode traveling wave ion mobility-mass spectrometry. Analyst. 14 (20), 6853-6861 (2015).
- Allen, S. J., Giles, K., Gilbert, T., Bush, M. F. Ion mobility mass spectrometry of peptide, protein, and protein complex ions using a radio-frequency confining drift cell. Analyst. 141 (3), 884-891 (2016).
- Bush, M. F., Campuzano, I. D. G., Robinson, C. V. Ion Mobility Mass Spectrometry of Peptide Ions: Effects of Drift Gas and Calibration Strategies. Analytical Chemistry. 84 (16), 7124-7130 (2012).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Smith, D. P., et al. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. European Journal of Mass Spectrometry. 15 (2), 113-130 (2009).
- Becke, A. D. Density-functional thermochemistry. III. The role of exact exchange. Journal of Chemical Physics. 98 (7), 5648-5652 (1993).
- Dunning, T. H., Hay, P. J. Gaussian basis sets for molecular calculations. Modern Theoretical Chemistry. 3, 1-27 (1977).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for potassium to gold including the outermost core orbitals. Journal of Chemical Physics. 82 (1), 299-310 (1985).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for the transition metal atoms scandium to mercury. Journal of Chemical Physics. 82 (1), 270-283 (1985).
- Wadt, W. R., Hay, P. J. Ab initio effective core potentials for molecular calculations. Potentials for main group elements sodium to bismuth. Journal of Chemical Physics. 82 (1), 284-298 (1985).
- . Gaussian 09, Revision C.01. Gaussian, Inc. , (2012).
- Wyttenbach, T., von Helden, G., Batka, J. J., Carlat, D., Bowers, M. T. Effect of the long-range potential on ion mobility measurements. Journal of the American Society of Mass Spectrometry. 8 (3), 275-282 (1997).
- Choi, D., et al. Redox activity and multiple copper(I) coordination of 2His-2Cys oligopeptide. Journal of Mass Spectrometry. 50 (2), 316-325 (2015).
- Rigo, A., et al. Interaction of copper with cysteine: stability of cuprous complexes and catalytic role of cupric ions in anaerobic thiol oxidation. Journal of Inorganic Biochemistry. 98 (9), 1495-1501 (2004).
- Vytla, Y., Angel, L. A. Applying Ion Mobility-Mass Spectrometry Techniques for Explicitly Identifying the Products of Cu(II) Reactions of 2His-2Cys Motif Peptides. Analytical Chemistry. 88 (22), 10925-10932 (2016).
- Choi, D., Sesham, R., Kim, Y., Angel, L. A. Analysis of methanobactin from Methylosinus trichosporium OB3b via ion mobility mass spectrometry. European Journal of Mass Spectrometry. 18 (6), 509-520 (2012).
- Martell, A. E., Motekaitis, R. J. NIST Standard Reference Database 46. Institute of Standards and Technology. , (2001).
- Pesch, M. L., Christl, I., Hoffmann, M., Kraemer, S. M., Kretzschmar, R. Copper complexation of methanobactin isolated from Methylosinus trichosporium OB3b: pH-dependent speciation and modeling. Journal of Inorganic Biochemistry. 116, 55-62 (2012).
- Amin, E. A., Truhlar, D. G. Zn Coordination Chemistry: Development of Benchmark Suites for Geometries, Dipole Moments, and Bond Dissociation Energies and Their Use To Test and Validate Density Functionals and Molecular Orbital Theory. Journal of Chemical Theory and Computation. 4 (1), 75-85 (2008).
- Sorkin, A., Truhlar, D. G., Amin, E. A. Energies, Geometries, and Charge Distributions of Zn Molecules, Clusters, and Biocenters from Coupled Cluster, Density Functional, and Neglect of Diatomic Differential Overlap Models. Journal of Chemical Theory and Computation. 5 (5), 1254-1265 (2009).
- Lillo, V., Galan-Mascaros, J. R. Transition metal complexes with oligopeptides: single crystals and crystal structures. Dalton Transactions. 43 (26), 9821-9833 (2014).
- Choutko, A., van Gunsteren, W. F. Conformational Preferences of a beta-Octapeptide as Function of Solvent and Force-Field Parameters. Helvetica Chimica Acta. 96 (2), 189-200 (2013).
- Angel, L. A. Study of metal ion labeling of the conformational and charge states of lysozyme by ion mobility mass spectrometry. European Journal of Mass Spectrometry. 17 (3), 207-215 (2011).
- Kelso, C., Rojas, J. D., Furlan, R. L. A., Padilla, G., Beck, J. L. Characterisation of anthracyclines from a cosmomycin D-producing species of Streptomyces by collisionally-activated dissociation and ion mobility mass spectrometry. European Journal of Mass Spectrometry. 15 (2), 73-81 (2009).
- El Ghazouani, A., et al. Copper-binding properties and structures of methanobactins from Methylosinus trichosporium OB3b. Inorganic Chemistry. 50 (4), 1378-1391 (2011).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유