Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Inducción y análisis del estrés oxidativo en células epiteliales pigmentarias de la retina humana transfectadas por transposones de la bella durmiente
En este artículo
Resumen
Presentamos un protocolo para el desarrollo y uso de un modelo de estrés oxidativo mediante el tratamiento de células epiteliales pigmentarias de la retina con H2O2,analizando la morfología celular, la viabilidad, la densidad, el glutatión y el nivel de UCP-2. Es un modelo útil para investigar el efecto antioxidante de las proteínas secretadas por las células transfectadas por transposones para tratar la degeneración neurorretiniana.
Resumen
El estrés oxidativo juega un papel crítico en varias enfermedades degenerativas, incluida la degeneración macular relacionada con la edad (DMAE), una patología que afecta a ~ 30 millones de pacientes en todo el mundo. Conduce a una disminución de los factores neuroprotectores sintetizados por el epitelio pigmentario de la retina (EPR), por ejemplo, el factor derivado del epitelio pigmentario (PEDF) y el factor estimulante de colonias de granulocitos y macrófagos (GM-CSF), seguido de la pérdida de células RPE y, finalmente, la muerte de células fotorreceptoras y ganglionares retinianas (RGC). Planteamos la hipótesis de que la reconstitución del ambiente retiniano neuroprotector y neurogénico mediante el trasplante subretiniano de células RPE transfectadas que sobreexpresan PEDF y GM-CSF tiene el potencial de prevenir la degeneración retiniana al mitigar los efectos del estrés oxidativo, inhibir la inflamación y apoyar la supervivencia celular. Utilizando el sistema de transposones de la Bella Durmiente (SB100X),las células RPE humanas se han transfectado con los genes PEDF y GM-CSF y han demostrado una integración génica estable, expresión génica a largo plazo y secreción de proteínas utilizando qPCR, western blot, ELISA e inmunofluorescencia. Para confirmar la funcionalidad y la potencia del PEDF y GM-CSF secretados por las células RPE transfectadas, hemos desarrollado un ensayo in vitro para cuantificar la reducción del estrés oxidativo inducido por H2O2en células RPE en cultivo. La protección celular se evaluó mediante el análisis de la morfología celular, la densidad, el nivel intracelular de glutatión, la expresión del gen UCP2 y la viabilidad celular. Tanto las células RPE transfectadas que sobreexpresan PEDF y/o GM-CSF como las células no transfectadas pero pretratadas con PEDF y/o GM-CSF (disponibles comercialmente o purificadas a partir de células transfectadas) mostraron una protección celular antioxidante significativa en comparación con los controles no tratados. El presente modelo H2O2es un enfoque simple y efectivo para evaluar el efecto antioxidante de factores que pueden ser efectivos para tratar la DMAE o enfermedades neurodegenerativas similares.
Introducción
El modelo descrito aquí, ofrece un enfoque útil para evaluar la eficiencia de los agentes biofarmacéuticos para reducir el estrés oxidativo en las células. Hemos utilizado el modelo para investigar los efectos protectores del PEDF y GM-CSF sobre el estrés oxidativo mediado por H2O2sobre las células epiteliales pigmentarias de la retina, que están expuestas a altos niveles de O2y luz visible, y la fagocitosis de las membranas del segmento externo fotorreceptor, generando niveles significativos de especies reactivas de oxígeno (ROS)1, 2. Se consideran un importante contribuyente a la patogénesis de la degeneración macular avascular relacionada con la edad (aAMD)3,4,5,6,7,8. Además, hay una disminución en los factores neuroprotectores sintetizados por RPE, específicamente el factor derivado del epitelio pigmentario (PEDF), los factores de crecimiento similares a la insulina (IGF) y el factor estimulante de colonias de macrófagos de granulocitos (GM-CSF) que conducen a la disfunción y pérdida de células RPE, seguidos de la muerte de células fotorreceptoras y ganglionares de la retina (RGC)3,4,5 . La DMAE es una enfermedad compleja que resulta de la interacción entre factores metabólicos, funcionales, genéticos y ambientales4. La falta de tratamientos para la AAMD es la principal causa de ceguera en pacientes mayores de 60 años en países industrializados9,10. La reconstitución del ambiente retiniano neuroprotector y neurogénico mediante el trasplante subretiniano de células RPE modificadas genéticamente que sobreexpresan PEDF y GM-CSF tiene el potencial de prevenir la degeneración retiniana al mitigar los efectos del estrés oxidativo, inhibir la inflamación y apoyar la supervivencia celular11,12,13,14,15,16 . A pesar de que existen varias metodologías para entregar genes a las células, hemos elegido el sistema de transposones de la Bella Durmiente hiperactiva no viral para entregar los genes PEDF y GM-CSF a las células RPE debido a su perfil de seguridad, la integración de los genes en el genoma de las células huésped y su propensión a integrar los genes entregados en sitios no transcripcionalmente activos como hemos demostrado anteriormente17, 18,19.
El estrés oxidativo celular puede ser inducido en células cultivadas in vitro por varios agentes oxidativos, incluyendo peróxido de hidrógeno (H2O2),4-hidroinonenal (HNE), tertbututilhidroperóxido (tBH), altas tensiones de oxígeno y luz visible (espectro completo o irradiación UV)20,21. Las altas tensiones de oxígeno y la luz requieren equipos y condiciones especiales, lo que limita la transferibilidad a otros sistemas. Agentes como H2O2,HNE y tBH inducen cambios moleculares y celulares superpuestos de estrés oxidativo. Elegimos H2O2 para probar la actividad antioxidante de PEDF y GM-CSF porque es conveniente y biológicamente relevante ya que es producido por las células RPE como un intermedio reactivo de oxígeno durante la fagocitosis del segmento externo fotorreceptor22 y se encuentra en los tejidos oculares in vivo23. Dado que la oxidación del glutatión puede ser parcialmente responsable de la producción de H2O2 en el ojo, hemos analizado los niveles de GSH/glutatión en nuestros estudios, que están relacionados con el estrés oxidativo inducido por H2O2y la capacidad regenerativa de las células21,22. El análisis de los niveles de glutatión es especialmente relevante ya que participa en los mecanismos de protección antioxidante en el ojo24. La exposición a H2O2 se utiliza con frecuencia como modelo para examinar la susceptibilidad al estrés oxidativo y la actividad antioxidante de las células RPE1,25,26,27,28,29,30, y, además, muestra similitudes con el daño por estrés oxidativo inducido por la luz, una fuente "fisiológica" de estrés oxidativo21.
Para evaluar la funcionalidad y la eficacia de los factores neuroprotectores, hemos desarrollado un modelo in vitro que permite cuantificar el efecto antioxidante de los factores de crecimiento expresados por células modificadas genéticamente para sobreexpresar PEDF y GM-CSF. Aquí, mostramos que las células RPE transfectadas con los genes para PEDF y GM-CSF son más resistentes a los efectos nocivos de H2O2 que las células de control no transfectadas, como lo demuestra la morfología celular, la densidad, la viabilidad, el nivel intracelular de glutatión y la expresión del gen UCP2, que codifica para la proteína de desacoplamiento mitocondrial 2 que se ha demostrado que reduce las especies reactivas de oxígeno (ROS)31.
Protocolo
Los procedimientos para la recolección y el uso de los ojos humanos fueron aprobados por la Comisión Ética Cantonal para la Investigación (no. 2016-01726).
1. Aislamiento celular y condiciones de cultivo
- Línea celular arpe-19 humana
- Cultivo 5 x 105 células ARPE-19, una línea celular de EPR humano, en la mezcla de medio/nutriente F-12 ham (DMEM/Ham's F-12) de Dulbecco suplementada con suero bovino fetal (FBS) al 10%, penicilina U/mL, estreptomicina de 80 μg/mL y 2,5 μg/ml de anfotericina B (medio completo) a 37 °C en una atmósfera humidificada de 5% de CO2 y 95% de aire en un matraz T75 (para otras densidades celulares ver Tabla 1).
- Cambie el medio tres veces por semana.
- Una vez que las células crecen a aproximadamente el 90% de confluencia (evaluada cualitativamente), aspire el medio y lave las células con 1x PBS estéril.
- Incubar las células con una solución de TRIPSINA-2% de EDTA al 5% durante 7-10 min a 37 °C (para volúmenes ver Tabla 1). Monitoree el desprendimiento visualmente.
- Detenga la tripsinización agregando un medio completo que contenga un 10% de FBS (para volúmenes, consulte la Tabla 1).
- Transfectar las células (ver paso 2. del protocolo), subcultivar las células en una proporción de 1:10 (una vez por semana), o sembrar en una placa de 96 pocillos como se detalla a continuación (ver pasos 3.3 y 3.4 del protocolo).
| Medio (ml) | ||||||
| Superficie (cm²) | Densidad de siembra para células ARPE-19 (células/pozo) | Aplicación | Para cultivo celular | Para detener la tripsina | Volumen de tripsina (ml) | |
| Matraz T75 | 75 | 5,00,000 | Crecimiento celular de ARPE-19 | 10 | 7 | 3 |
| Placa de 6 pozos | 9.6 | 1,00,000 | Siembra de células ARPE-19 transfectadas | 3 | 1 | 0.5 |
| Placa de 24 pocillos | 2 | 50,000 | Siembra de células hRPE transfectadas | 1 | 0.8 | 0.2 |
| Placa de 96 pozos | 0.32 | 5.000 para experimentos de estrés oxidativo con células transfectadas (Fig. 1) | Experimentos de estrés oxidativo | 0.2 | ||
| 3.000 para experimentos de estrés oxidativo con células no transfectadas más proteínas (Fig. 1) | ||||||
Tabla 1: Volúmenes de cultivo celular. Volúmenes de medios recomendados para placas de cultivo celular y matraces para el cultivo de ARPE-19 y células RPE humanas primarias.
- Células RPE humanas primarias
- Aislar células RPE humanas primarias según lo descrito por Thumann et al.17, y células de cultivo en medio completo suplementado con 20% FBS.
- Cambie el medio dos veces por semana. Una vez que las células alcanzan la confluencia (monitoreadas visualmente), reduzca el FBS al 1% para evitar el crecimiento excesivo.
- Transfectar las células (ver paso 2 del protocolo), o sembrar en una placa de 96 pocillos como se detalla a continuación (ver pasos 3.3 y 3.4 del protocolo).
NOTA: Los datos aquí presentados fueron recolectados del cultivo de células RPE obtenidas de los ojos de cuatro donantes humanos. La Tabla 2 detalla la demografía de los donantes del Lions Gift of Sight Eye Bank (Saint Paul, MN). Los ojos se enuclearon 12,7 ± 5,7 h (media ± SD) post mortem después de obtener el consentimiento informado de acuerdo con la Declaración de Helsinki.
| No | edad | género | muerte a preservación (horas) | muerte al aislamiento | cultivo | cultivo | Símbolo en el gráfico | |
| (días) | antes de la transfección (días) | después de la transfección (días) | ||||||
| 2 | 80 | M | 20.7 | 8 | 140 | 36 |  | |
| 3 | 86 | F | 12.8 | 8 | 85 | 45 |  | |
| 4 | 86 | F | 8.5 | 5 | 26 | 133 |  | |
| 8 | 83 | F | 8.9 | 6 | 18 | 27 |  | |
| significar | 83.8 | 12.7 | 6.8 | 67.3 | 60.3 | |||
| SD | 2.9 | 5.7 | 1.5 | 57.0 | 49.1 |
Tabla 2: Demografía de donantes humanos para células epiteliales pigmentarias de la retina.
2. Electroporación de ARPE-19 y células primarias de EPR humano
- Tripsinizar células ARPE-19 o células RPE humanas primarias como se describe en los pasos 1.1.3-1.1.5 del protocolo.
- Realice la electroporación con el kit de transfección disponible en el mercado (consulte la Tabla de materiales).
- Para la transfección de células ARPE-19 consulte Johnen et al.32 y para hRPE primario a Thumann et al.17. Brevemente, resuspenda 1 x 105 células ARPE-19 o 5 x 104 células primarias de hRPE en 11 μL de tampón R y añadir 2 μL de mezcla de plásmidos que contenga 0,03 μg pSB100X transposasa33 y 0,47 μg pT2-CMV-PEDF-His o pT2-CMV-GMCSF-Su transposón (relación transposasa:transposón 1:16). Para las células dobles transfectadas de PEDF y GM-CSF, utilice una relación de 1:16:16 (0,03 μg pSB100X, 0,47 μg pT2-CMV-PEDF-His,y 0,47 μg pT2-CMV-GMCSF-His). Utilice los siguientes parámetros de electroporación: dos pulsos de 1.350 V para 20 ms (ancho de pulso) para celdas ARPE-19; dos pulsos de 1.100 V para 20 ms para células primarias.
- Semilla 1 x 105 ARPE-19 transfectado o 5 x 104 células primarias de hRPE transfectadas en placas de 6 pocillos y 24 pocillos, respectivamente, en medio suplementado con 10% fbS sin antibióticos ni antimicóticos. Agregue penicilina (80 U/mL), estreptomicina (80 μg/mL) y anfotericina B (2,5 μg/mL) con el primer intercambio medio 3 días después de la transfección.
- Determinar el crecimiento celular mediante el monitoreo microscópico semanal de las células. La eficacia de la transfección se controla mediante el análisis de la expresión génica mediante RT-PCR, y la secreción de proteínas mediante ELISA y WB (métodos explicados en Material complementario).
NOTA: La eficiencia de la transfección se puede evaluar por primera vez una vez que las células alcanzan la confluencia, es decir, a ~ 7 días y 4 semanas después de la transfección para las células ARPE-19 y las células primarias de hRPE, respectivamente. - Células de semilla en una placa de 96 pocillos como se detalla a continuación (ver paso 3.5 del protocolo).
3. Inducción del estrés oxidativo (tratamiento H2O2) y neuroprotección (tratamiento PEDF y/o GM-CSF)
- Preparación del medio acondicionado de células ARPE-19 transfectadas
- Utilizar células ARPE-19 transfectadas con los genes PEDF, GM-CSF, o ambos (ver paso 2 del protocolo); células de cultivo durante 28 días como se describe en el paso 1.1 del protocolo.
- A los 28 días después de la transfección, tripsinizar las células (ver pasos 1.1.3-1.1.5 del protocolo), contar las células utilizando una cámara de Neubauer34,35y sembrar 5 x 105 células en matraces T75 en medio completo como se describe en el paso 1.1.1 del protocolo. Intercambiar el medio cuando el cultivo celular sea aproximadamente 80% confluente (aproximadamente después de 1 semana; verificado cualitativamente). Recoger el medio después de 24 h.
- Conservar el medio a -20 °C hasta su uso.
NOTA: La concentración suficiente del PEDF recombinante y gm-CSF en el medio acondicionado fue verificada por WB y cuantificada por ELISA como se describe en Material suplementario.
- Purificación de PEDF y GM-CSF a partir de medio acondicionado de células ARPE-19 transfectadas
- Centrifugar el medio recogido del paso 3.1.2 a 10.000 x g durante 15 min a 4 °C.
- Utilice el superflujo Ni-NTA (consulte la Tabla de materiales) deacuerdo con los protocolos del fabricante para purificar las proteínas marcadas con His como se describe a continuación.
- Pipetear 30 μL de mezcla de Ni-NTA en un tubo de 1,5 ml y centrifugar a 2.600 x g durante 30 s y desechar el flujo a través. Lave el pellet dos veces con 200 μL de tampón de incubación 1x.
- Centrifugar a 2.600 x g durante 30 s y desechar el flujo. Añadir 40 μL de tampón de incubación 4x y resuspend.
- Añadir 900 μL de medio acondicionado centrifugado e incubar a 70 rpm (agitador orbital) durante 1 h a RT. Centrifugar a 2.600 x g durante 1 min y el flujo de descarte.
- Lave el pellet dos veces con 175 μL de tampón de incubación 1x. Centrifugar a 2.600 x g durante 30 s y desechar el flujo.
- Para eludir las proteínas PEDF y GM-CSF marcadas con His, agregue 20 μL de tampón de elución e incube a 70 rpm (agitador orbital) durante 20 min en RT. Centrífuga a 2.600 x g durante 30 s. Mantenga el sobrenadante que contenga PEDF recombinante o GM-CSF.
- Cuantificar la proteína total utilizando el kit de ensayo de proteína BCA disponible comercialmente (ver Tabla de Materiales) deacuerdo con las instrucciones del fabricante.
- Guarde la solución proteica a -20 °C hasta su uso.
NOTA: El tampón de incubación (4x) contiene 200 mM NaH2PO4,1.2 M NaCl y 40 mM Imidazol; El tampón de elución contiene 50 mM NaH2PO4, 300 mM NaCl y 250 mM Imidazol.
- Tratamiento de células ARPE-19/hRPE primarias no transfectadas con medio acondicionado más H2O2 (Figura 1A)
- Sembrar 3.000 células ARPE-19 no transfectadas (a partir de la etapa 1.1.6 del protocolo) o hRPE primario (a partir de la etapa 1.2.3 del protocolo) por pozo en placa de 96 pocillos y cultivo en 200 μL de medio acondicionado a partir de células ARPE-19 transfectadas.
- Cultive las células durante 10 días a 37 °C en una atmósfera humidificada de 5% de CO2 y 95% de aire. Cambie el medio acondicionado todos los días. Exponer las células a 350 μM H2O2 durante 24 h.
- Evaluar el daño por estrés oxidativo y determinar el efecto antioxidante de PEDF y GM-CSF mediante la cuantificación de los niveles de glutatión (ver paso 4.1 del protocolo), microscopía (ver paso 4.2 del protocolo) y ensayo de citotoxicidad (ver paso 4.2 del protocolo).
NOTA: La duración del experimento es de 12 días. Las placas de micropocillos de fondo plano transparente se utilizan para evaluar la luminiscencia y la morfología celular. Para realizar simultáneamente el ensayo de citotoxicidad y glutatión, se deben sembrar dos placas con células el mismo día.
- Tratamiento de células ARPE-19/hRPE primarias no transfectadas con factores de crecimiento PEDF y GM-CSF más H2O2 (Figura 1B)
- Sembrar 3.000 células ARPE-19 no transfectadas (a partir de la etapa 1.1.6 del protocolo) o hRPE primario (a partir de la etapa 1.2.3 del protocolo) por pocillo (placas de 96 pocillos con un fondo plano transparente) en 200 μL de medio de cultivo completo que contenga 500 ng/mL de PEDF recombinante y/o 50 ng/mL de GM-CSF recombinante, purificado a partir del medio de células de ARPE-19 transfectadas o disponible comercialmente. Células de cultivo durante 48 h a 37 °C en una atmósfera humidificada de 5% de CO2 y 95% de aire. Renovar diariamente el medio, incluidos los factores de crecimiento PEDF y GM-CSF.
NOTA: Agregue los factores de crecimiento frescos al medio. - Después de 48 h de tratar las células con los factores de crecimiento, retire el medio y agregue el medio completo que contenga 350 μM H2O2 más 500 ng / ml PEDF y / o 50 ng / mL GM-CSF.
- Evaluar el daño por estrés oxidativo y determinar el efecto antioxidante de PEDF y GM-CSF mediante la cuantificación de los niveles de glutatión (ver paso 4.1 del protocolo), microscopía (ver paso 4.2 del protocolo) y ensayo de citotoxicidad (ver paso 4.2 del protocolo).
NOTA: La duración del experimento es de 3 días.
- Sembrar 3.000 células ARPE-19 no transfectadas (a partir de la etapa 1.1.6 del protocolo) o hRPE primario (a partir de la etapa 1.2.3 del protocolo) por pocillo (placas de 96 pocillos con un fondo plano transparente) en 200 μL de medio de cultivo completo que contenga 500 ng/mL de PEDF recombinante y/o 50 ng/mL de GM-CSF recombinante, purificado a partir del medio de células de ARPE-19 transfectadas o disponible comercialmente. Células de cultivo durante 48 h a 37 °C en una atmósfera humidificada de 5% de CO2 y 95% de aire. Renovar diariamente el medio, incluidos los factores de crecimiento PEDF y GM-CSF.
- Tratamiento de células ARPE-19/hRPE primarias transfectadas con H2O2 (Figura 1C)
- Verificar la suficiente expresión génica y la secreción de proteínas de las células transfectadas por WB y ELISA como se describe en el Material Suplementario.
- Retire el medio de los pocillos que contienen las células transfectadas (consulte el paso 2 del protocolo).
- Tripsinizar las células como se describe en los pasos 1.1.3-1.1.5 del protocolo. Contar las células usando una cámara de Neubauer34,35.
- Sembrar 5.000 células transfectadas/pozo en placa de 96 pocillos en 200 μL de medio completo. Células de cultivo durante 24 h a 37 °C en una atmósfera humidificada de 5% de CO2 y 95% de aire. Después de 24 h, exponer las células a 350 μM H2O2 durante 24 h.
- Evaluar el daño por estrés oxidativo y determinar el efecto antioxidante del PEDF y el GM-CSF mediante la cuantificación de los niveles de glutatión (ver paso 4.1 del protocolo), microscopía (ver paso 4.2 del protocolo), ensayo de citotoxicidad (ver paso 4.2 del protocolo) y determinación de la expresión génica de UCP2 (ver paso 4.3 del protocolo).
NOTA: La duración del experimento es de 2 días.
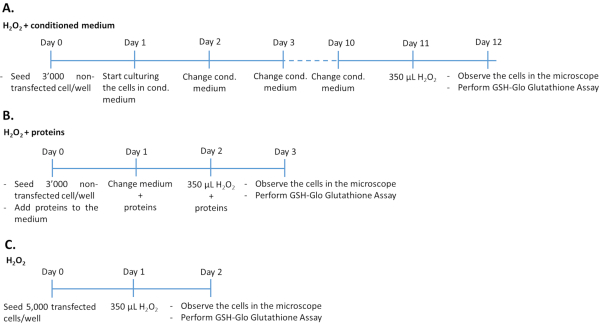
Figura 1: Líneas de tiempo del ensayo H2O2 en los tres enfoques experimentales diferentes. Se sembraron 3.000 células no transfectadas tratadas con el medio acondicionado/proteínas recombinantes o 5.000 células transfectadas en placas de 96 pocillos para su tratamiento con H2O2. Para determinar el efecto del medio condicionado, las células se cultivaron en medio 100% cultivado durante 10 días consecutivos, cambiando de medio todos los días. Para determinar el efecto de los factores de crecimiento recombinantes, las células se cultivaron agregando la cantidad adecuada de factores de crecimiento cada día durante 3 días consecutivos. Tenga en cuenta que las células no transfectadas se sembraron a 3.000 células por pozo para evitar el crecimiento excesivo durante la mayor duración del cultivo en comparación con las células transfectadas. Haga clic aquí para ver una versión más grande de esta figura.
4. Análisis del nivel de estrés oxidativo y capacidad antioxidante
- Ensayo de glutatión
- Mida los niveles de glutatión (GSH) utilizando el kit disponible comercialmente (consulte la Tabla de materiales)siguiendo las instrucciones del fabricante. Brevemente, prepare y asigne el volumen de 1x mezcla de reactivos (reactivo de 100 μL/pozo): sustrato de luciferina-NT y glutatión S-transferasa diluido 1:100 en Reaction Buffer.
NOTA: Una placa de 96 pocillos requiere 10 ml de mezcla de reactivo 1x, que se prepara agregando 100 μL de sustrato de Luciferin-NT y 100 μL de glutatión S-transferasa a 10 mL de tampón de reacción. Prepare la mezcla de reactivos 1x inmediatamente antes de usarla. No almacene la mezcla de reactivos preparada para su uso futuro. - Prepare el reactivo de detección de luciferina transfiriendo una botella de tampón de reconstitución al reactivo de detección de luciferina liofilizado.
- Preparar una curva estándar utilizando una solución estándar de glutatión (GSH) (5 mM). Diluir 5 mM de solución de GSH 1:100 con dH2O (añadir 10 μL de solución de GSH de 5 mM a 990 μL de dH2O). Realizar 7 diluciones seriadas 1:1 en 500 μL de dH2O. Transferir 10 μL de cada estándar diluido a un pozo apropiado por duplicado.
NOTA: La concentración final de glutatión oscilará entre 0,039 μM y 5 μM. - Prepare el espacio en blanco (1x mezcla de reactivos) y transfiera 10 μL (duplicados) a los pozos apropiados.
- Retire las células tratadas con H2O2de la incubadora.
NOTA: Documentar la morfología de las células tratadas con H2O2mediante microscopía de campo brillante (40x).
Cuando las células se oxidan, se ven más redondeadas y menos dispersas. - Aspirar cuidadosamente el medio de cultivo. Agregue 100 μL de mezcla de reactivos 1x preparada a cada pocillo. Mezcle las celdas con el reactivo durante 15 s a 500 rpm en un agitador orbital.
- Incubar la placa en RT durante 30 min. Agregue 100 μL de reactivo de detección de luciferina reconstituido a cada pozo.
- Mezcle la solución durante 15 s a 500 rpm en un agitador orbital. Incubar la placa durante 15 min en RT.
- Determine la luminiscencia utilizando un lector de placas utilizando un programa preinstalado ADP-Glo.
NOTA: Coloque la placa dentro del lector de placas sin la tapa.- Haga clic en Cambiar diseño y elija la siguiente configuración en Parámetros básicos:Placa Costar de 96 pocillos; óptica superior; retraso de posicionamiento: 0.1; hora de inicio de la medición: 0.0; tiempo de intervalo de medición: 1.0; tiempo para normalizar los resultados: 0.0; la ganancia es ajustada automáticamente por el dispositivo. Defina espacios en blanco, estándares y ejemplos. Haga clic en Iniciar medición.
- Exporte los datos como un archivo de Excel. Calcular la concentración de GSH en cada muestra mediante interpolación de la curva estándar.
- Mida los niveles de glutatión (GSH) utilizando el kit disponible comercialmente (consulte la Tabla de materiales)siguiendo las instrucciones del fabricante. Brevemente, prepare y asigne el volumen de 1x mezcla de reactivos (reactivo de 100 μL/pozo): sustrato de luciferina-NT y glutatión S-transferasa diluido 1:100 en Reaction Buffer.
- Ensayo de citotoxicidad y análisis microscópico
- Aspire el medio de las células y agregue 100 μL de medio completo que contenga 1% de FBS a cada pozo. Devuelva las células a la incubadora.
NOTA: Se utiliza un 1% de FBS porque los porcentajes más altos de FBS pueden interferir con la medición de la luminiscencia, por lo tanto, se utiliza un 1% de FBS en este caso. - Mida la viabilidad celular utilizando el kit de ensayo de citotoxicidad disponible en el mercado (consulte la Tabla de materiales)siguiendo las instrucciones del fabricante. Brevemente, prepare la mezcla de reactivos agregando el tampón de ensayo al sustrato liofilizado. Prepare el reactivo de lisis agregando 33 μL de digitonina a un tampón de ensayo de 5 ml (para una placa de 96 pocillos). Mezclar bien mediante pipeteo hacia arriba y hacia abajo para garantizar la homogeneidad.
NOTA: Para obtener resultados óptimos, use una mezcla de reactivos recién preparada. Úselo dentro de las 12 h si se almacena en RT. La mezcla de reactivos se puede almacenar a 4 °C durante un máximo de 7 días y puede almacenarse en alícuotas de un solo uso durante un máximo de 4 meses a -70 °C. Debe evitarse la congelación y la descongelación. El reactivo de lisis se puede almacenar a 4 °C durante un máximo de 7 días. - Prepare una curva estándar con células ARPE-19 no tratadas.
- Tripsinizar las células como se describe en los pasos 1.1.3-1.1.5 del protocolo y contar las células utilizando una cámara de Neubauer34,35. Centrifugar las células a 120 g durante 10 min en RT. Aspirar el sobrenadante y resuspendir el pellet celular en el medio F12 de DMEM/Ham que contiene 1% de FBS a una concentración final de 1 x 105 células/ml.
- Preparar 7 diluciones seriales 1:1 en medio de 200 μL que contengan 1% de FBS. Transfiera 100 μL de cada estándar a los pozos apropiados (duplicados). Añadir 50 μL de mezcla de reactivos a todos los pocillos.
- Mezcle las celdas con el reactivo durante 15 s a 500 rpm en un agitador orbital. Incubar la placa durante 15 min en RT. Mida la luminiscencia utilizando el lector de placas como se describe en el paso 4.1.9 del protocolo. Añadir 50 μL del reactivo de lisis e incubar durante 15 min. Mida la luminiscencia utilizando el lector de placas como se describe en el paso 4.1.9 del protocolo.
- Calcule el porcentaje de células viables: (100 - % de células muertas) y el porcentaje de células muertas = [1ª medición de luminiscencia ((células muertas en la muestra))/ 2ª medición de luminiscencia (todas las células muertas después del tratamiento con digitoninas)] x 100.
- Aspire el medio de las células y agregue 100 μL de medio completo que contenga 1% de FBS a cada pozo. Devuelva las células a la incubadora.
- Análisis de expresión de UCP2 mediante RT-qPCR
- Tripsinizar las células transfectadas como se describió anteriormente (pasos 1.1.3-1.1.5 del protocolo).
- Contar las células usando una cámara de Neubauer34,35.
- Sembra 5.000 células/pozo ARPE-19 transfectados en placas de 96 pocillos.
- Después de 24 h de cultivo, tratar las células con 350 μM H2O2 durante 24 h.
- Aislar el ARN total utilizando un kit comercial para el aislamiento del ARN de un bajo número de células (ver Tabla de Materiales)siguiendo las instrucciones del fabricante.
- Realizar PCR cuantitativa en tiempo real (RT-qPCR) como se describe en Material suplementario. Brevemente, genere ADNc por retrotranscripción utilizando una mezcla disponible comercialmente que contenga una transcriptasa inversa M-MLV optimizada (ver Tabla de materiales).
- Para qPCR emplear un cóctel de reacción listo para usar que contenga todos los componentes (incluido SYBR Green) excepto los cebadores (ver Tabla S1 de Material Suplementario)y la plantilla de ADN. Utilice las siguientes condiciones de termociclado: desnaturalización inicial a 95 °C durante 10 min, 40 ciclos con desnaturalización a 95 °C durante 15 s, recocido a 60 °C durante 30 s y alargamiento a 72 °C durante 32 s.
- Utilice el método 2^(-ΔΔCT) para el análisis36.
- Preparación de lisado celular para análisis SDS-PAGE y WB de pAkt (Ser473)
- Sembrar 3 x 105 células ARPE-19 transfectadas GM-CSF/pozo en placas de 6 pocillos (≥21 días después de la transfección) para determinar si GM-CSF protege a las células RPE del daño por H2O2 a través de la activación de la vía de supervivencia Akt15.
- Después de 24 h de cultivo las células se exponen a 350 μM H2O2 durante 24 h.
- Mezclar 1 ml de tampón RIPA con 10 μL de cóctel inhibidor de la proteasa fosfatasa, 10 μL de 0,5 M de EDTA y 25 μL de 8 M de urea (volúmenes utilizados para un pocillo).
- Aspire cuidadosamente el medio y lave las células con 1x PBS.
- Agregue todo el volumen de la mezcla de búfer RIPA a las celdas.
- Pipeta arriba y abajo.
- Recoger el lisado en tubos de 1,5 ml.
- Centrifugadora a 20.000 x g durante 30 min a 4 °C.
- Transfiera el sobrenadante a un nuevo tubo de 1,5 ml.
- Determinar los niveles de pAkt en 15 μL de lisado celular sin diluir por WB como se describe en Material suplementario.
Resultados
Inducción de estrés oxidativo en células epiteliales pigmentarias retinianas humanas
Las células ARPE-19 y hRPE primarias fueron tratadas con concentraciones variables de H2O2 durante 24 h y se cuantificó el nivel intracelular del antioxidante glutatión(Figura 2A,B). H2O2 a 50 μM y 100 μM no afectaron la producción de glutatión, mientras que a 350 μM hubo una disminuci...
Discusión
El protocolo presentado aquí ofrece un enfoque para analizar la función antioxidante y protectora de PEDF y GM-CSF producidos por células transfectadas, que se puede aplicar a células transfectadas con cualquier gen beneficioso putativo. En las estrategias terapéuticas génicas que tienen el objetivo de entregar proteínas al tejido mediante el trasplante de células modificadas genéticamente, es fundamental obtener información sobre el nivel de expresión de proteínas, la longevidad de la expresión y la efectiv...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores desean agradecer a Gregg Sealy y Alain Conti por su excelente asistencia técnica y a la Prof. Zsuzsanna Izsvák del Centro Max-Delbrück en Berlín por proporcionar amablemente los plásmidos pSB100X y pT2-CAGGS-Venus. Este trabajo fue apoyado por la Fundación Nacional Suiza de Ciencias y la Comisión Europea en el contexto del Séptimo Programa Marco. Z.I fue financiado por el Consejo Europeo de Investigación, ERC Advanced [ERC-2011-ADG 294742].
Materiales
| Name | Company | Catalog Number | Comments |
| 24-well plates | Corning | 353047 | |
| 6-well plates | Greiner | 7657160 | |
| 96-well culture plate white with clear flat bottom | Costar | 3610 | Allows to check the cells before measuring the luminescence (GSH-Glo Assay) |
| 96-well plates | Corning | 353072 | |
| Acrylamid 40% | Biorad | 161-0144 | |
| Amphotericin B | AMIMED | 4-05F00-H | |
| Antibody anti-GMCSF | ThermoFisher Scientific | PA5-24184 | |
| Antibody anti-mouse IgG/IgA/IgM | Agilent | P0260 | |
| Antibody anti-PEDF | Santa Cruz Biotechnology Inc | sc-390172 | |
| Antibody anti-penta-His | Qiagen | 34660 | |
| Antibody anti-phospho-Akt | Cell Signaling Technology | 9271 | |
| Antibody anti-rabbit IgG H&L-HRP | Abcam | ab6721 | |
| Antibody donkey anti-rabbit Alexa Fluor 594 | ThermoFisher Scientific | A11034 | |
| Antibody goat anti-mouse Alexa 488 | ThermoFisher Scientific | A-11029 | |
| ARPE-19 cell line | ATCC | CRL-2302 | |
| BSA | Sigma-Aldrich | A9418-500G | |
| chamber culture glass slides | Corning | 354118 | |
| CytoTox-Glo Cytotoxicity Assay | Promega | G9291 | |
| DAPI | Sigma-Aldrich | D9542-5MG | |
| DMEM/Ham`s F12 | Sigma-Aldrich | D8062 | |
| Duo Set ELISA kit | R&D Systems | DY215-05 | |
| EDTA | ThermoFisher Scientific | 78440 | |
| ELISAquant kit | BioProducts MD | PED613-10-Human | |
| Eyes (human) | Lions Gift of Sight Eye Bank (Saint Paul, MN) | ||
| FBS | Brunschwig | P40-37500 | |
| Fluoromount Aqueous Mounting Medium | Sigma-Aldrich | F4680-25ML | |
| FLUOstar Omega plate reader | BMG Labtech | ||
| GraphPad Prism software (version 8.0) | GraphPad Software, Inc. | ||
| GSH-Glo Glutathione Assay | Promega | V6912 | |
| hydrogen peroxide (H2O2) | Merck | 107209 | |
| ImageJ software (image processing program) | W.S. Rasband, NIH, Bethesda, MD, USA; https://imagej.nih.gov/ij/; 1997–2014 | ||
| Imidazol | Axonlab | A1378.0010 | |
| Leica DMI4000B microscope | Leica Microsystems | ||
| LightCycler 480 Instrument II | Roche Molecular Systems | ||
| LightCycler 480 SW1.5.1 software | Roche Molecular Systems | ||
| NaCl | Sigma-Aldrich | 71376-1000 | |
| NaH2PO4 | Axonlab | 3468.1000 | |
| Neon Transfection System | ThermoFisher Scientific | MPK5000 | |
| Neon Transfection System 10 µL Kit | ThermoFisher Scientific | MPK1096 | |
| Neubauer chamber | Marienfeld-superior | 640010 | |
| Ni-NTA superflow | Qiagen | 30410 | |
| Nitrocellulose | VWR | 732-3197 | |
| Omega Lum G Gel Imaging System | Aplegen Life Science | ||
| PBS 1X | Sigma-Aldrich | D8537 | |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781-100 | |
| PerfeCTa SYBR Green FastMix | Quantabio | 95072-012 | |
| PFA | Sigma-Aldrich | 158127-100G | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23227 | |
| Primers | Invitrogen | See Table 1 in Supplementary Materials | |
| pSB100X (250 ng/µL) | Mátés et al., 2009. Provide by Prof. Zsuzsanna Izsvak | ||
| pT2-CMV-GMCSF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| pT2-CMV-PEDF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| QIAamp DNA Mini Kit | QIAGEN | 51304 | |
| recombinant hGM-CSF | Peprotech | 100-11 | |
| recombinant hPEDF | BioProductsMD | 004-096 | |
| ReliaPrep RNA Cell Miniprep System | Promega | Z6011 | |
| RIPA buffer | ThermoFisher Scientific | 89901 | |
| RNase-free DNase Set | QIAGEN | 79254 | |
| RNeasy Mini Kit | QIAGEN | 74204 | |
| SDS | Applichem | A2572 | |
| Semi-dry transfer system for WB | Bio-Rad | ||
| SuperMix qScript | Quantabio | 95048-025 | |
| Tris-buffered saline (TBS) | ThermoFisher Scientific | 15504020 | |
| Triton X-100 | AppliChem | A4975 | |
| Trypsin/EDTA | Sigma-Aldrich | T4174 | |
| Tween | AppliChem | A1390 | |
| Urea | ThermoFisher Scientific | 29700 | |
| WesternBright ECL HRP substrate | Advansta | K-12045-D50 | |
| Whatman nitrocellulose membrane | Chemie Brunschwig | MNSC04530301 |
Referencias
- Zareba, M., Raciti, M. W., Henry, M. M., Sarna, T., Burke, J. M. Oxidative stress in ARPE-19 cultures: Do melanosomes confer cytoprotection. Free Radical Biology and Medicine. 40 (1), 87-100 (2006).
- Gong, X., Draper, C. S., Allison, G. S., Marisiddaiah, R., Rubin, L. P. Effects of the macular carotenoid lutein in human retinal pigment epithelial cells. Antioxidants. 6 (4), (2017).
- Sacconi, R., Corbelli, E., Querques, L., Bandello, F., Querques, G. A Review of current and future management of geographic atrophy. Ophthalmology and Therapy. 6, 69-77 (2017).
- Al-Zamil, W. M., Yassin, S. A. Recent developments in age-related macular degeneration: a review. Clinical Interventions in Aging. 12, 1313-1330 (2017).
- Kumar-Singh, R. The role of complement membrane attack complex in dry and wet AMD - From hypothesis to clinical trials. Experimental Eye Research. 184, 266-277 (2019).
- Ung, L., Pattamatta, U., Carnt, N., Wilkinson-Berka, J. L., Liew, G., White, A. J. R. Oxidative stress and reactive oxygen species. Clinical Science. 131, 2865-2883 (2017).
- Beatty, S., Koh, H. H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Alhasani, R. H., et al. Gypenosides protect retinal pigment epithelium cells from oxidative stress. Food and Chemical Toxicology. 112, 76-85 (2018).
- . National Institute of Health Available from: https://nei.nih.gov/learn-about-eye-health/resources-for-health-educators/eye-health-data-and-statistics/age-related-macular-degeneration-amd-data-and-statistics (2020)
- Mitchell, P., Liew, G., Gopinath, B., Wong, T. Y. Age-related macular degeneration. The Lancet. 392, 1147-1159 (2018).
- He, Y., Leung, K. W., Ren, Y., Jinzhi, P., Jian, G., Tombran-Tink, J. PEDF improves mitochondrial function in RPE cells during oxidative stress. Investigative Ophthalmology & Visual Science. 55, 6742-6755 (2014).
- Cao, S., Walker, G. B., Wang, X., Cui, J. Z., Matsubara, J. A. Altered cytokine profiles of human retinal pigment epithelium: Oxidant injury and replicative senescence. Molecular Vision. 19, 718-728 (2013).
- Farnoodian, M., Sorenson, C. M., Sheibani, N. PEDF expression affects the oxidative and inflammatory state of choroidal endothelial cells. Amercian Journal of Physiology and Cell Physiology. 314 (4), 456-472 (2018).
- Polato, F., Becerra, S. P. Retinal Degenerative Diseases: Mechanisms and Experimental Therapies. Retinal Degenerative Diseases. , 699-706 (2016).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF regulates the ERK1/2 pathways and protects injured retinal ganglion cells from induced death. Experimental Eye Research. 89, 665-677 (2009).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF protects rat photoreceptors from death by activating the SRC-dependent signalling and elevating anti-apoptotic factors and neurotrophins. Graefes Archives for Clinical and Experimental Ophthalmology. 250, 699-712 (2012).
- Thumann, G., et al. Engineering of PEDF-expressing primary pigment epithelial cells by the SB transposon system delivered by pFAR4 plasmids. Molecular Therapy - Nucleic Acids. 6, 302-314 (2017).
- Garcia-Garcia, L., et al. Long-term PEDF release in rat iris and retinal epithelial cells after Sleeping Beauty transposon-mediated gene delivery. Molecular Therapy - Nucleic Acids. 9, 1-11 (2017).
- Johnen, S., et al. Antiangiogenic and neurogenic activities of Sleeping Beauty-mediated PEDF-transfected RPE cells in vitro and in vivo. BioMed Research International. 2015, (2015).
- Weigel, A. L., Handa, J. T., Hjelmeland, M. L. Microarray analysis of H2O2-, HNE-, or tBH-treated ARPE-19 cells. Free Radical Biology & Medicine. 33 (10), 1419-1432 (2002).
- Allen, R. G., Tresini, M. Oxidative stress and gene regulation. Free Radical Biology and Medicine. 28 (3), 463-499 (2000).
- Tate, D. J., Miceli, M. V., Newsome, D. A. Phagocytosis and H2O2 induce catalase and metallothionein gene expression in human retinal pigment epithelial cells. Investigative Ophthalmology and Visual Science. 36 (7), 1271-1279 (1995).
- Halliwell, B., Clement, M. V., Long, L. H. Hydrogen peroxide in the human body. FEBS Letters. 486 (1), 10-13 (2000).
- Giblin, F. J., McCready, J. P., Kodama, T., Reddy, V. N. A direct correlation between the levels of ascorbic acid and H2O2 in aqueous humor. Experimental Eye Research. 38, 87-93 (1984).
- Geiger, R. C., Waters, C. M., Kamp, D. W., Glucksberg, M. R. KGF prevents oxygen-mediated damage in ARPE-19 cells. Investigative Ophthalmology and Visual Science. 46, 3435-3442 (2005).
- Campochiaro, P. A., et al. Lentiviral vector gene transfer of endostatin/angiostatin for macular degeneration (GEM) study. Human Gene Therapy. 28, 99-111 (2017).
- Chen, X. -. D., Su, M. -. Y., Chen, T. -. T., Hong, H. -. Y., Han, A. -. D., Li, W. -. S. Oxidative stress affects retinal pigment epithelial cell survival through epidermal growth factor receptor/AKT signaling pathway. International Journal of Ophthalmology. 10 (4), 507-514 (2017).
- Tu, G., et al. Allicin attenuates H2O2 - induced cytotoxicity in retinal pigmented epithelial cells by regulating the levels of reactive oxygen species. Molecular Medicine Reports. 13, 2320-2326 (2016).
- Hao, Y., Liu, J., Wang, Z., Yu, L., Wang, J. Piceatannol protects human retinal pigment epithelial cells against hydrogen peroxide induced oxidative stress and apoptosis through modulating. Nutrients. 11, 1-13 (2019).
- Ballinger, S. W., Van Houten, B., Conklin, C. A., Jin, G. F., Godley, B. F. Hydrogen peroxide causes significant mitochondrial DNA damage in human RPE cells. Experimental Eye Research. 68 (6), 765-772 (1999).
- Ma, S., et al. Transgenic overexpression of uncoupling protein 2 attenuates salt-induced vascular dysfunction by inhibition of oxidative stress. American Journal of Hypertension. 27 (3), 345-354 (2014).
- Johnen, S., et al. Sleeping Beauty transposon-mediated transfection of retinal and iris pigment epithelial cells. Investigative Ophthalmology and Visual Science. 53 (8), 4787-4796 (2012).
- Mátés, L., et al. Molecular evolution of a novel hyperactive Sleeping Beauty transposase enables robust stable gene transfer in vertebrates. Nature Genetics. 41 (6), 753-761 (2009).
- . Marienfeld Technical information Neubauer-improved Available from: https://www.marienfeld-superior.com/information-about-our-counting-chambers.html (2020)
- . Electron Microscopy Sciences. Neubauer Haemocytometry Available from: https://www.emsdiasum.com/microscopy/technical/datasheet/68052-14.aspx (2020)
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2^(-ΔΔCT) method. Methods. 25 (4), 402-408 (2001).
- Bascuas, T., et al. Non-virally transfected primary human pigment epithelium cells overexpressing the oxidative stress reduction factors PEDF and GM-CSF to treat retinal neurodegeneration neurodegenerationl. Human Gene Therapy. 30 (11), (2019).
- Zhuge, C. C., et al. Fullerenol protects retinal pigment epithelial cells from oxidative stress-induced premature senescence via activating SIRT1. Investigative Ophthalmology & Visual Science. 55 (7), 4628-4638 (2014).
- Kaczara, P., Sarna, T., Burke, M. Dynamics of H2O2 Availability to ARPE-19 cultures in models of oxidative stress. Free Radical Biology and Medicine. 48 (8), 1068-1070 (2010).
- Gorrini, C., Harris, I. S., Mak, T. W. Modulation of oxidative stress as an anticancer strategy. Nature Reviews Drug Discovery. 12, 931-947 (2013).
- Wang, X., et al. PEDF protects human retinal pigment epithelial cells against oxidative stress via upregulation of UCP2 expression. Molecular Medicine Reports. 19 (1), 59-74 (2019).
- Donadelli, M., Dando, I., Fiorini, C., Palmieri, M. UCP2, a mitochondrial protein regulated at multiple levels. Cellular and Molecular Life Sciences. 71, 1171-1190 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados