É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Indução e Análise do Estresse Oxidativo em Células Epiteliais de Pigmento Epitelial Humano Transposon-Transfected Da Bela Adormecida
Neste Artigo
Resumo
Apresentamos um protocolo para o desenvolvimento e uso de um modelo de estresse oxidativo, tratando células epiteliais de pigmento da retina com H2O2, analisando morfologia celular, viabilidade, densidade, glutationa e nível UCP-2. É um modelo útil para investigar o efeito antioxidante de proteínas secretadas por células transfeminadas transposon para tratar a degeneração neuroretinal.
Resumo
O estresse oxidativo desempenha um papel crítico em várias doenças degenerativas, incluindo a degeneração macular relacionada à idade (AMD), uma patologia que afeta ~30 milhões de pacientes em todo o mundo. Leva a uma diminuição no epitélio do pigmento da retina (RPE)-fatores neuroprotetores sintetizados, por exemplo, fator derivado do epitélio pigmento (PEDF) e fator estimulante da colônia granutócito-macrófago (GM-CSF), seguido pela perda de células RPE, e eventualmente fotorreceptor e morte de células gânglios de retina (RGC). Temos a hipótese de que a reconstituição do ambiente neuroprotetor e neurogênico da retina pelo transplante subretinal de células RPE transfecidas que superexpressam PEDF e GM-CSF tem o potencial de prevenir a degeneração da retina, mitigando os efeitos do estresse oxidativo, inibindo inflamação e apoiando a sobrevivência celular. Usando o sistema transposon da Bela Adormecida (SB100X)as células RPE humanas foram transfectadas com os genes PEDF e GM-CSF e mostraram integração genética estável, expressão genética de longo prazo e secreção de proteínas usando qPCR, mancha ocidental, ELISA e imunofluorescência. Para confirmar a funcionalidade e a potência do PEDF e gm-CSF secretados pelas células RPE transfeinadas, desenvolvemos um ensaio in vitro para quantificar a redução do estresse oxidativo induzido por H2O2em células RPE na cultura. A proteção celular foi avaliada pela análise da morfologia celular, densidade, nível intracelular de glutationa, expressão genética UCP2 e viabilidade celular. Ambas as células RPE transfectadas que expressam o FPE e/ou GM-CSF e as células não transfetizadas, mas pré-tratadas com PEDF e/ou GM-CSF (comercialmente disponíveis ou purificadas de células transfetizadas) apresentaram proteção celular antioxidante significativa em comparação com controles não tratados. O atual modelo H2O2é uma abordagem simples e eficaz para avaliar o efeito antioxidante de fatores que podem ser eficazes para tratar a AMD ou doenças neurodegenerativas similares.
Introdução
O modelo descrito aqui, oferece uma abordagem útil para avaliar a eficiência dos agentes biofarmacêuticos para reduzir o estresse oxidativo nas células. Utilizamos o modelo para investigar os efeitos protetores do PEDF e do GM-CSF no estresse oxidativo mediado H2O2nas células epiteliais do pigmento retinal, que estão expostas a altos níveis de O2, luz visível, e a fagocitose das membranas do segmento externo fotoreceptor, gerando níveis significativos de espécies reativas de oxigênio (ROS)1, 2. São considerados um dos principais contribuintes para a patogênese da degeneração macular avascular relacionada à idade (aAMD)3,4,5,6,7,8. Além disso, há uma diminuição nos fatores neuroprotetores sintetizados por RPE, especificamente o fator derivado do epitélio pigmento (PEDF), fatores de crescimento semelhantes à insulina (IGFs) e fator estimulante da colônia-colônia granucyte (GM-CSF) levando à disfunção e perda de células RPE, seguidos pela morte de células fotorreceptoras e gânglios da retina (RGC)3,4,5 . A AMD é uma doença complexa que resulta da interação entre fatores metabólicos, funcionais, genéticos e ambientais4. A falta de tratamentos para a AAMD é a principal causa de cegueira em pacientes com mais de 60 anos de idade em países industrializados9,10. A reconstituição do ambiente neuroprotetor e neurogênico da retina pelo transplante subretinal de células RPE geneticamente modificadas que superexpressam PEDF e GM-CSF tem o potencial de prevenir a degeneração da retina, mitigando os efeitos do estresse oxidativo, inibindo inflamação e apoiando a sobrevivência celular11,12,13,14,15,16 . Embora existam várias metodologias para entregar genes às células, escolhemos o sistema transposon da Bela Adormecida hiperativo não viral para entregar os genes PEDF e GM-CSF às células RPE por causa de seu perfil de segurança, a integração dos genes no genoma das células hospedeiras e sua propensão a integrar os genes entregues em locais não transcricionalmente, como mostramos anteriormente17, 18,19.
O estresse oxidativo celular pode ser induzido em células cultivadas in vitro por vários agentes oxidativos, incluindo peróxido de hidrogênio (H2O2),4-hidroynonenal (HNE), tertbutylhydroperoxide (tBH), altas tensões de oxigênio e luz visível (espectro completo ou irradiação UV)20,21. Altas tensões de oxigênio e luz requerem equipamentos e condições especiais, o que limita a transferência para outros sistemas. Agentes como H2O2,HNE e tBH induzem alterações moleculares e celulares por estresse oxidativo. Escolhemos H2O2 para testar a atividade antioxidante do PEDF e GM-CSF porque é conveniente e biologicamente relevante, pois é produzido por células RPE como um intermediário de oxigênio reativo durante a fagocitose do segmento externo fotoreceptor22 e é encontrado em tecidos oculares in vivo23. Como a oxidação da glutationa pode ser parcialmente responsável pela produção de H2O2 no olho, analisamos os níveis de GSH/glutationa em nossos estudos, que estão ligados ao estresse oxidativo induzido por H2O2e à capacidade regenerativa das células21,22. A análise dos níveis de glutationa é especialmente relevante, pois participa dos mecanismos de proteção antioxidantes no olho24. A exposição a H2O2 é utilizada com frequência como modelo para examinar a suscetibilidade ao estresse oxidativo e a atividade antioxidante das células RPE1,25,26,27,28,29,30, e, além disso, apresenta semelhanças com danos oxidativos induzidos pela luz, fonte "fisiológica" de estresse oxidativo21.
Para avaliar a funcionalidade e a eficácia dos fatores neuroprotetores, desenvolvemos um modelo in vitro que permite que a análise quantifique o efeito antioxidante dos fatores de crescimento expressos por células geneticamente modificadas para superexpressar o PEDF e o GM-CSF. Aqui, mostramos que as células RPE transfeminadas com os genes para PEDF e GM-CSF são mais resistentes aos efeitos nocivos de H2O2 do que são células de controle não transfectadas, como evidenciado pela morfologia celular, densidade, viabilidade, nível intracelular de glutationa e expressão do gene UCP2, que codifica a proteína mitocondrial de desacoplamento 2 que tem sido demonstrada para reduzir espécies reativas de oxigênio (ROS)31.
Protocolo
Os procedimentos de coleta e uso de olhos humanos foram aprovados pela Comissão de Ética Cantonnal de Pesquisa (nº 2016-01726).
1. Condições de isolamento celular e cultura
- Linha celular HUMANA ARPE-19
- Cultura 5 x 105 células ARPE-19, uma linha celular RPE humana, na mistura de médio/nutrientes da Águia Modificada de Dulbecco F-12 Ham (DMEM/Ham's F-12) suplementada com 10% de soro bovino fetal (FBS), penicilina U/mL de 80 U/mL, Estreptomicina de 80 μg/mL, e 2,5 μg/mL de anfotericina B (meio completo) a 37 °C em uma atmosfera umidificada de 5% de CO2 e 95% de ar em um frasco T75 (para outras densidades ver tabela 1).
- Troque o meio três vezes por semana.
- Uma vez que as células são cultivadas para aproximadamente 90% de confluência (avaliada qualitativamente), aspirar o meio e lavar as células com 1x PBS estérei.
- Incubar as células com uma solução EDTA de 5% de Trypsin-2% para 7-10 min a 37 °C (para volumes ver Tabela 1). Monitore o desprendimento visualmente.
- Pare de tentarpsinização adicionando meio completo contendo 10% de FBS (para volumes ver Tabela 1).
- Transfeito as células (ver etapa 2. do protocolo), subsulo as células a uma razão de 1:10 (uma vez por semana), ou sementes em uma placa de 96 poços como detalhado abaixo (ver etapas 3.3 e 3.4 do protocolo).
| Médio (mL) | ||||||
| Área (cm²) | Densidade de semeadura para células ARPE-19 (células/bem) | Aplicação | Para a cultura celular | Para parar a trippsina | Volume de trippsina (mL) | |
| Frasco T75 | 75 | 5,00,000 | Crescimento celular ARPE-19 | 10 | 7 | 3 |
| 6 Placa de poço | 9.6 | 1,00,000 | Semeadura de células ARPE-19 transfeinadas | 3 | 1 | 0.5 |
| 24 Placa de poço | 2 | 50,000 | Semeadura de células hRPE transfectadas | 1 | 0.8 | 0.2 |
| 96 Placa de poço | 0.32 | 5.000 para experimentos de estresse oxidativo com células transfeinadas (Fig. 1) | Experimentos de estresse oxidativo | 0.2 | ||
| 3.000 para experimentos de estresse oxidativo com células não transfectadas mais proteínas (Fig. 1) | ||||||
Tabela 1: Volumes de cultura celular. Volumes de mídia recomendados para placas de cultura celular e frascos para a cultura de células ARPE-19 e RPE humanos primários.
- Células RPE humanas primárias
- Isole as células RPE humanas primárias, como descrito por Thumann et al.17, e as células de cultura em meio completo suplementadas com 20% de FBS.
- Troque o meio duas vezes por semana. Uma vez que as células atinjam a confluência (monitorada visualmente), reduza a FBS para 1% para evitar o crescimento excessivo.
- Transfeito as células (ver passo 2 do protocolo), ou semente em uma placa de 96 poços como detalhado abaixo (ver etapas 3.3 e 3.4 do protocolo).
NOTA: Os dados aqui apresentados foram coletados a partir da cultura das células RPE obtidas a partir dos olhos de quatro doadores humanos. A Tabela 2 detalha a demografia dos doadores do Lions Gift of Sight Eye Bank (Saint Paul, MN). Os olhos foram enucleados 12,7 ± 5,7 h (média ± SD) após a obtenção de consentimento informado de acordo com a Declaração de Helsinque.
| Não | idade | Gênero | morte à preservação (horas) | morte ao isolamento | cultivo | cultivo | Símbolo em gráfico | |
| (dias) | antes da transfecção (dias) | após a transfecção (dias) | ||||||
| 2 | 80 | M | 20.7 | 8 | 140 | 36 |  | |
| 3 | 86 | F | 12.8 | 8 | 85 | 45 |  | |
| 4 | 86 | F | 8.5 | 5 | 26 | 133 |  | |
| 8 | 83 | F | 8.9 | 6 | 18 | 27 |  | |
| significar | 83.8 | 12.7 | 6.8 | 67.3 | 60.3 | |||
| SD | 2.9 | 5.7 | 1.5 | 57.0 | 49.1 |
Tabela 2: Demografia dos doadores humanos para células epiteliais de pigmento retiniano.
2. Eletroporação de células ARPE-19 e RPE humanas primárias
- Trippsinize células ARPE-19 ou células RPE humanas primárias, conforme descrito nas etapas 1.1.3-1.1.5 do protocolo.
- Realizar eletroporação com o kit de transfecção comercialmente disponível (ver Tabela de Materiais).
- Para transfecção de células ARPE-19 consulte Johnen et al.32 e para hRPE primário para Thumann et al.17. Resumidamente, resuspend 1 x 105 células ARPE-19 ou 5 x 104 células primárias hRPE em 11 μL de tampão R e adicionar 2 μL de mistura plasmida contendo 0,03 0μg pSB100X transposase33 e 0,47 μg pT2-CMV-PEDF-His ou pT2-CMV-GMCSF-Seu transposon (razão transposase:transposon 1:16). Para células transfetícias duplas PEDF e GM-CSF, utilize uma razão de 1:16:16 (0,03 μg pSB100X, 0,47 μg pT2-CMV-PEDF-His, e 0,47 μg pT2-CMV-GMCSF-His). Utilize os seguintes parâmetros de eletroporação: dois pulsos de 1.350 V para 20 ms (largura de pulso) para células ARPE-19; dois pulsos de 1.100 V para 20 ms para células primárias.
- Seed 1 x 105 ARPE-19 ou 5 x 104 células hRPE transfectadas em placas de 6-well e 24-well, respectivamente, em médio suplementado com 10% de FBS sem antibióticos ou antimióticos. Adicione penicilina (80 U/mL), estreptomicina (80 μg/mL) e anfotericina B (2,5 μg/mL) com a primeira troca média 3 dias após a transfecção.
- Determinar o crescimento celular por monitoramento microscópico semanal das células. A eficiência da transfecção é monitorada pela análise da expressão genética por RT-PCR, e secreção de proteínas por ELISA e WB (métodos explicados em Material Suplementar).
NOTA: A eficiência da transfecção pode ser avaliada pela primeira vez quando as células atingirem a confluência, ou seja, em ~7 dias e 4 semanas de pós-transfecção para células ARPE-19 e células hRPE primárias, respectivamente. - Células de sementes em uma placa de 96 poços como detalhadas abaixo (ver o passo 3.5 do protocolo).
3. Indução de estresse oxidativo (tratamento H2O2) e neuroproteção (tratamento PEDF e/ou GM-CSF)
- Preparação do meio condicionado de células ARPE-19 transfectadas
- Use células ARPE-19 transfeinadas com os genes PEDF, GM-CSF ou ambas (ver o passo 2 do protocolo); células de cultura por 28 dias como descrito na etapa 1.1 do protocolo.
- Aos 28 dias após a transfecção, células de trypsinize (ver passos 1.1.3-1.1.5 do protocolo), contam células usando uma câmara neubauer34,35, e sementes 5 x 105 células em frascos T75 em meio completo, conforme descrito na etapa 1.1.1 do protocolo. Troque o meio quando a cultura celular estiver aproximadamente 80% confluente (aproximadamente após 1 semana; verificada qualitativamente). Colete o meio depois das 24h.
- Armazene o meio a -20 °C até usar.
NOTA: A concentração suficiente do PEDF recombinante e do GM-CSF no meio condicionado foi verificada pela WB e quantificada por ELISA conforme descrito em Material Suplementar.
- Purificação de PEDF e GM-CSF a partir de meio condicionado de células ARPE-19 transfectadas
- Centrifugar o meio coletado da etapa 3.1.2 a 10.000 x g para 15 min a 4 °C.
- Use o superfluxo Ni-NTA (ver Tabela de Materiais) de acordo com os protocolos do fabricante para purificar proteínas marcadas por Sua, conforme descrito abaixo.
- Pipeta 30 μL de mistura Ni-NTA em um tubo de 1,5 mL e centrífuga a 2.600 x g para 30 s e descartar o fluxo-through. Lave a pelota duas vezes com 200 μL de tampão de incubação de 1x.
- Centrifugar a 2.600 x g para 30 s e descartar o fluxo. Adicione 40 μL de tampão de incubação 4x e resuspend.
- Adicione 900 μL de meio condicionado centrifusado e incubar a 70 rpm (agitador orbital) por 1 h na RT. Centrífuga a 2.600 x g por 1 min e o fluxo de descarte.
- Lave a pelota duas vezes com 175 μL de tampão de incubação de 1x. Centrifugar a 2.600 x g para 30 s e descartar o fluxo.
- Para eluto Suas proteínas PEDF e GM-CSF, adicione 20 μL de tampão de elução e incubar a 70 rpm (agitador orbital) por 20 min na RT. Centrífuga a 2.600 x g por 30 s. Mantenha o supernatante contendo PEDF recombinante ou GM-CSF.
- Quantifique a proteína total utilizando o kit de ensaio de proteína BCA comercialmente disponível (ver Tabela de Materiais) de acordo com as instruções do fabricante.
- Armazene a solução de proteína a -20 °C até usar.
NOTA: O buffer de incubação (4x) contém 200 mM NaH2PO4, 1,2 M NaCl e 40 mM Imidazol; O buffer de elução contém 50 mM NaH2PO4, 300 mM NaCl e 250 mM Imidazol.
- Tratamento de células ARPE-19/hRPE não transfectadas com meio condicionado mais H2O2 (Figura1A)
- Semente 3.000 células ARPE-19 não transfectadas (a partir da etapa 1.1.6 do protocolo) ou hRPE primário (a partir da etapa 1.2.3 do protocolo) células por poço em placa e cultura de 96 poços em 200 μL de meio condicionado de células ARPE-19 transfectadas.
- Cultura as células por 10 dias a 37 °C em uma atmosfera umidificada de 5% de CO2 e 95% de ar. Mude o meio condicionado todos os dias. Exponha as células a 350 μM H2O2 por 24 h.
- Avalie os danos oxidativos do estresse e determine o efeito antioxidante do PEDF e do GM-CSF por quantificação dos níveis de glutationa (ver etapa 4.1 do protocolo), microscopia (ver etapa 4.2 do protocolo) e ensaio de citotoxicidade (ver etapa 4.2 do protocolo).
NOTA: A duração do experimento é de 12 dias. Placas de microwell de fundo plano claro são usadas para avaliar a luminescência, bem como a morfologia celular. Para realizar simultaneamente o ensaio de citotoxicidade e glutationa, duas placas devem ser semeadas com células no mesmo dia.
- Tratamento de células ARPE-19/hRPE não transfectadas com fatores de crescimento PEDF e GM-CSF mais H2O2 (Figura 1B)
- Semente 3.000 células ARPE-19 não transfectadas (a partir da etapa 1.1.6 do protocolo) ou hRPE primário (a partir da etapa 1.2.3 do protocolo) células por poço (placas de 96 poços com fundo plano claro) em 200 μL de cultura completa contendo PEDF recombinante de 500 ng/mL e/ou 50 ng/mL recombinantes GM-CSF, purificados a partir do meio de células ARPE-19 transfectadas ou comercialmente disponíveis. Células de cultura para 48 h a 37 °C em uma atmosfera umidificada de 5% de CO2 e 95% de ar. Renove diariamente os fatores de crescimento do PEDF e do GM-CSF.
NOTA: Adicione os fatores de crescimento frescos ao meio. - Após 48h de tratamento das células com os fatores de crescimento, remova o meio médio e adicione o meio completo contendo 350 μM H2O2 mais 500 ng/mL PEDF e/ou 50 ng/mL GM-CSF.
- Avalie os danos oxidativos do estresse e determine o efeito antioxidante do PEDF e do GM-CSF por quantificação dos níveis de glutationa (ver etapa 4.1 do protocolo), microscopia (ver etapa 4.2 do protocolo) e ensaio de citotoxicidade (ver etapa 4.2 do protocolo).
NOTA: A duração do experimento é de 3 dias.
- Semente 3.000 células ARPE-19 não transfectadas (a partir da etapa 1.1.6 do protocolo) ou hRPE primário (a partir da etapa 1.2.3 do protocolo) células por poço (placas de 96 poços com fundo plano claro) em 200 μL de cultura completa contendo PEDF recombinante de 500 ng/mL e/ou 50 ng/mL recombinantes GM-CSF, purificados a partir do meio de células ARPE-19 transfectadas ou comercialmente disponíveis. Células de cultura para 48 h a 37 °C em uma atmosfera umidificada de 5% de CO2 e 95% de ar. Renove diariamente os fatores de crescimento do PEDF e do GM-CSF.
- Tratamento de células ARPE-19/hRPE primárias transfeinadas com H2O2 (Figura 1C)
- Verifique a expressão genética suficiente e a secreção proteica das células transfeinadas por WB e ELISA, conforme descrito no Material Suplementar.
- Remova o meio dos poços que contêm as células transfeinadas (ver o passo 2 do protocolo).
- Células de trypsinize, conforme descrito nas etapas 1.1.3-1.1.5 do protocolo. Conte as células usando uma câmara de Neubauer34,35.
- Semente 5.000 células transfectadas/bem em placa de 96 poços em 200 μL de meio completo. Células de cultura por 24 h a 37 °C em uma atmosfera umidificada de 5% de CO2 e 95% de ar. Após 24 horas, exponha as células a 350 μM H2O2 por 24 h.
- Avalie os danos oxidativos do estresse e determine o efeito antioxidante do PEDF e gm-CSF por quantificação dos níveis de glutationa (ver etapa 4.1 do protocolo), microscopia (ver etapa 4.2 do protocolo), ensaio de citotoxicidade (ver etapa 4.2 do protocolo) e determinação da expressão genética UCP2 (ver etapa 4.3 do protocolo).
NOTA: A duração do experimento é de 2 dias.
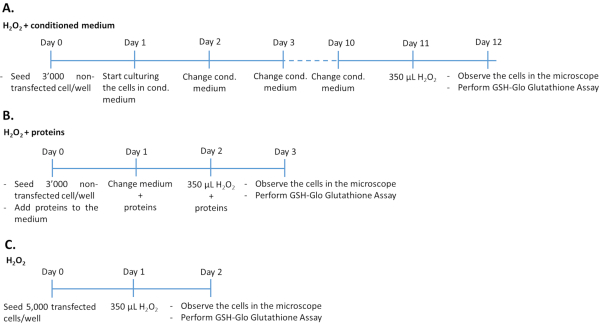
Figura 1: Cronogramas do ensaio H2O2 nas três diferentes abordagens experimentais. 3.000 células não transfectadas tratadas com as proteínas médias/recombinantes condicionadas ou 5.000 células transfeinadas foram semeadas em 96 placas de poço para tratamento com H2O2. Para determinar o efeito do meio condicionado, as células foram cultivadas em meio 100% cultivado por 10 dias consecutivos, mudando de meio a cada dia. Para determinar o efeito dos fatores de crescimento recombinantes, as células foram cultivadas adicionando a quantidade adequada de fatores de crescimento todos os dias por 3 dias consecutivos. Observe que as células não transfectadas foram semeadas a 3.000 células por poço para evitar o crescimento excessivo durante a maior duração da cultura em comparação com as células transfeinadas. Clique aqui para ver uma versão maior desta figura.
4. Análise do nível de estresse oxidativo e capacidade antioxidante
- Ensaio de glutationa
- Meça os níveis de Glutationa (GSH) utilizando o kit comercialmente disponível (ver Tabela de Materiais) seguindo as instruções do fabricante. Brevemente, prepare e desemuente o volume da mistura de reagente 1x (100 reagente/bem): substrato de luciferina-NT e Glutathione S-Transferase diluídos 1:100 no Buffer de Reação.
NOTA: Uma placa de 96 poços requer 10 mL de mistura de reagente 1x, que é preparada adicionando 100 μL de substrato luciferina-NT e 100 μL de Glutathione S-Transferase a 10 mL de tampão de reação. Prepare a mistura de reagente 1x imediatamente antes de usar. Não armazene o mix de Reagente preparado para uso futuro. - Prepare o Reagente de Detecção de Luciferina transferindo uma garrafa de tampão de reconstituição para o Reagente de Detecção de Lúciferina liofilizada.
- Prepare uma curva padrão usando uma solução padrão glutathione (GSH) (5 mM). Diluir solução GSH de 5 mM 1:100 com dH2O (adicione 10 μL de solução GSH de 5 mM a 990 μL de dH2O). Realize 7 diluição serial 1:1 em 500 μL de dH2O. Transfira 10 μL de cada padrão diluído para um poço apropriado em duplicata.
NOTA: A concentração final de glutationa varia de 0,039 μM a 5 μM. - Prepare o branco (mistura de reagente 1x) e transfira 10 μL (duplicatas) para os poços apropriados.
- Remova as células tratadas H2O2da incubadora.
NOTA: Documente a morfologia das células tratadas H2O2por microscopia de campo brilhante (40x).
Quando as células são oxidadas, elas parecem mais arredondadas e menos espalhadas. - Aspire cuidadosamente o meio de cultura. Adicione 100 μL de mistura de reagente 1x preparada a cada poço. Misture as células com o reagente por 15 s a 500 rpm em um agitador orbital.
- Incubar a placa em RT por 30 minutos. Adicione 100 μL de reagente de detecção de lúciferina reconstituído a cada poço.
- Misture a solução por 15 s a 500 rpm em um agitador orbital. Incubar a placa por 15 min na RT.
- Determine a luminescência usando um leitor de placas usando um programa pré-instalado ADP-Glo.
NOTA: Coloque a placa dentro do leitor de placas sem a tampa.- Clique em Alterar layout e escolha as seguintes configurações em Parâmetros Básicos: Placa costar de 96 poços; alta óptica; atraso de posicionamento: 0.1; tempo de início da medição: 0.0; tempo de intervalo de medição: 1.0; tempo para normalizar os resultados: 0.0; o ganho é ajustado automaticamente pelo dispositivo. Defina espaços em branco, padrões e amostras. Clique em Iniciar a medição.
- Exporte os dados como um arquivo Excel. Calcule a concentração de GSH em cada amostra por interpolação da curva padrão.
- Meça os níveis de Glutationa (GSH) utilizando o kit comercialmente disponível (ver Tabela de Materiais) seguindo as instruções do fabricante. Brevemente, prepare e desemuente o volume da mistura de reagente 1x (100 reagente/bem): substrato de luciferina-NT e Glutathione S-Transferase diluídos 1:100 no Buffer de Reação.
- Ensaio de citotoxicidade e análise microscópica
- Aspire o meio das células e adicione 100 μL de meio completo contendo 1% de FBS a cada poço. Devolva as células para a incubadora.
NOTA: 1% de FBS é utilizado porque percentuais mais elevados de FBS podem interferir na medição da luminescência, portanto 1% de FBS é utilizado neste caso. - Medir a viabilidade celular usando o kit de ensaio de citotoxicidade disponível comercialmente (ver Tabela de Materiais) seguindo as instruções do fabricante. Brevemente, prepare a mistura reagente adicionando o tampão de ensaio ao substrato liofilizado. Prepare o Reagente de Lysis adicionando 33 μL Digitonin a 5 mL Assay buffer (para uma placa de 96 poços). Misture bem por pipetting para cima e para baixo para garantir a homogeneidade.
NOTA: Para obter resultados ótimos, use a mistura de reagente recém-preparada. Use em até 12 horas se armazenado no RT. A mistura de reagente pode ser armazenada a 4 °C por até 7 dias e pode ser armazenada em alíquotas de uso único por até 4 meses a -70 °C. O congelamento e o descongelamento devem ser evitados. O Reagente de Lise pode ser armazenado a 4 °C por até 7 dias. - Prepare uma curva padrão com células ARPE-19 não tratadas.
- Trypsinize as células conforme descrito nas etapas 1.1.3-1.1.5 do protocolo e conte as células usando uma câmara de Neubauer34,35. Centrifugar as células a 120 g por 10 min na RT. Aspire o supernasce e resuspenha a pelota celular no meio F12 do DMEM/Ham contendo 1% de FBS a uma concentração final de 1 x 105 células/mL.
- Prepare 7 diluições de série 1:1 em 200 μL médio contendo 1% de FBS. Transfira 100 μL de cada padrão para os poços apropriados (duplicatas). Adicione 50 μL de mistura de reagente a todos os poços.
- Misture as células com o reagente por 15 s a 500 rpm em um agitador orbital. Incubar a placa por 15 min na RT. Medir a luminescência usando o leitor de placas conforme descrito na etapa 4.1.9 do protocolo. Adicione 50 μL do reagente de lise e incubar por 15 min. Medir a luminescência usando o leitor de placas conforme descrito na etapa 4.1.9 do protocolo.
- Calcule a porcentagem de células viáveis: (100 - % células mortas) e a porcentagem de células mortas = [1ª medição de luminescência ((células mortas na amostra)) / 2ª medida de luminescência (todas as células mortas após o tratamento de digitonina)] x 100.
- Aspire o meio das células e adicione 100 μL de meio completo contendo 1% de FBS a cada poço. Devolva as células para a incubadora.
- Análise de expressão UCP2 por RT-qPCR
- Tripular células transfetí infectadas conforme descrito acima (etapas 1.1.3-1.1.5 do protocolo).
- Conte as células usando uma câmara de Neubauer34,35.
- Sementes 5.000 células ARPE-19 transfectadas/bem em placas de 96 poços.
- Após 24 horas de cultura, trate as células com 350 μM H2O2 por 24 h.
- Isole o RNA total usando um kit comercial para isolamento do RNA de baixo número de células (ver Tabela de Materiais) seguindo a instrução do fabricante.
- Executar PCR quantitativo em tempo real (RT-qPCR) conforme descrito em Material Suplementar. Brevemente, gere cDNA por retrotrancriação usando um mix comercialmente disponível contendo uma Transcriptase Reversa M-MLV otimizada (ver Tabela de Materiais).
- Para qPCR, utilize um coquetel de reação pronto para uso contendo todos os componentes (incluindo O Verde SYBR), exceto primers (ver Tabela S1 de Material Suplementar) e modelo de DNA. Use as seguintes condições de termociclismo: denaturação inicial a 95 °C para 10 min, 40 ciclos com denaturação a 95 °C para 15 s, ressarcimento a 60 °C para 30 s e alongamento a 72 °C para 32 s.
- Use o método 2^(-ΔΔCT) para análise36.
- Preparação de lise celular para análise de SDS-PAGE e WB de pAkt (Ser473)
- Seed 3 x 105 GM-CSF-transfectdam arpe-19 células/poço em placas de 6 poços (≥21 dias após transfecção) para determinar se o GM-CSF protege as células RPE contra danos por H2O2 através da ativação da via de sobrevivência Akt15.
- Após 24 h de células culturais são expostas a 350 μM H2O2 por 24 h.
- Misture 1 mL de tampão RIPA com 10 μL de coquetel inibidor de fosfatase protease, 10 μL de 0,5 M EDTA e 25 μL de 8 M de ureia (volumes usados para um poço).
- Aspire cuidadosamente o meio e lave as células com 1x PBS.
- Adicione todo o volume da mistura de buffer RIPA às células.
- Pipeta para cima e para baixo.
- Colete o lysate em tubos de 1,5 mL.
- Centrifugar a 20.000 x g por 30 min a 4 °C.
- Transfira o supernatante para um novo tubo de 1,5 mL.
- Determine os níveis de pAkt em 15 μL de lise celular não diluída por WB, conforme descrito em Material Suplementar.
Resultados
Indução do estresse oxidativo nas células epiteliais do pigmento da retina humana
As células ARPE-19 e hRPE primária foram tratadas com concentrações variadas de H2O2 por 24 h e o nível intracelular da glutationa antioxidante foi quantificado(Figura 2A,B). H2O2 a 50 μM e 100 μM não afetaram a produção de glutationa, enquanto em 350 μM houve uma diminuição signific...
Discussão
O protocolo aqui apresentado oferece uma abordagem para analisar a função antioxidante e protetora do PEDF e GM-CSF produzido por células transfeinadas, que podem ser aplicadas a células transfeinadas com qualquer gene benéfico putativo. Em estratégias terapêuticas genéticas que têm o objetivo de fornecer proteínas ao tecido por meio do transplante de células geneticamente modificadas, é fundamental obter informações quanto ao nível de expressão proteica, à longevidade da expressão e à eficácia da pro...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores gostariam de agradecer a Gregg Sealy e Alain Conti pela excelente assistência técnica e prof. Zsuzsanna Izsvák do Centro Max-Delbrück em Berlim por fornecer gentilmente os plasmídeos pSB100X e pT2-CAGGS-Venus. Este trabalho foi apoiado pela Fundação Nacional de Ciências suíça e pela Comissão Europeia no contexto do Sétimo Programa-Quadro. Z.I foi financiado pelo European Research Council, ERC Advanced [ERC-2011-ADG 294742].
Materiais
| Name | Company | Catalog Number | Comments |
| 24-well plates | Corning | 353047 | |
| 6-well plates | Greiner | 7657160 | |
| 96-well culture plate white with clear flat bottom | Costar | 3610 | Allows to check the cells before measuring the luminescence (GSH-Glo Assay) |
| 96-well plates | Corning | 353072 | |
| Acrylamid 40% | Biorad | 161-0144 | |
| Amphotericin B | AMIMED | 4-05F00-H | |
| Antibody anti-GMCSF | ThermoFisher Scientific | PA5-24184 | |
| Antibody anti-mouse IgG/IgA/IgM | Agilent | P0260 | |
| Antibody anti-PEDF | Santa Cruz Biotechnology Inc | sc-390172 | |
| Antibody anti-penta-His | Qiagen | 34660 | |
| Antibody anti-phospho-Akt | Cell Signaling Technology | 9271 | |
| Antibody anti-rabbit IgG H&L-HRP | Abcam | ab6721 | |
| Antibody donkey anti-rabbit Alexa Fluor 594 | ThermoFisher Scientific | A11034 | |
| Antibody goat anti-mouse Alexa 488 | ThermoFisher Scientific | A-11029 | |
| ARPE-19 cell line | ATCC | CRL-2302 | |
| BSA | Sigma-Aldrich | A9418-500G | |
| chamber culture glass slides | Corning | 354118 | |
| CytoTox-Glo Cytotoxicity Assay | Promega | G9291 | |
| DAPI | Sigma-Aldrich | D9542-5MG | |
| DMEM/Ham`s F12 | Sigma-Aldrich | D8062 | |
| Duo Set ELISA kit | R&D Systems | DY215-05 | |
| EDTA | ThermoFisher Scientific | 78440 | |
| ELISAquant kit | BioProducts MD | PED613-10-Human | |
| Eyes (human) | Lions Gift of Sight Eye Bank (Saint Paul, MN) | ||
| FBS | Brunschwig | P40-37500 | |
| Fluoromount Aqueous Mounting Medium | Sigma-Aldrich | F4680-25ML | |
| FLUOstar Omega plate reader | BMG Labtech | ||
| GraphPad Prism software (version 8.0) | GraphPad Software, Inc. | ||
| GSH-Glo Glutathione Assay | Promega | V6912 | |
| hydrogen peroxide (H2O2) | Merck | 107209 | |
| ImageJ software (image processing program) | W.S. Rasband, NIH, Bethesda, MD, USA; https://imagej.nih.gov/ij/; 1997–2014 | ||
| Imidazol | Axonlab | A1378.0010 | |
| Leica DMI4000B microscope | Leica Microsystems | ||
| LightCycler 480 Instrument II | Roche Molecular Systems | ||
| LightCycler 480 SW1.5.1 software | Roche Molecular Systems | ||
| NaCl | Sigma-Aldrich | 71376-1000 | |
| NaH2PO4 | Axonlab | 3468.1000 | |
| Neon Transfection System | ThermoFisher Scientific | MPK5000 | |
| Neon Transfection System 10 µL Kit | ThermoFisher Scientific | MPK1096 | |
| Neubauer chamber | Marienfeld-superior | 640010 | |
| Ni-NTA superflow | Qiagen | 30410 | |
| Nitrocellulose | VWR | 732-3197 | |
| Omega Lum G Gel Imaging System | Aplegen Life Science | ||
| PBS 1X | Sigma-Aldrich | D8537 | |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781-100 | |
| PerfeCTa SYBR Green FastMix | Quantabio | 95072-012 | |
| PFA | Sigma-Aldrich | 158127-100G | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23227 | |
| Primers | Invitrogen | See Table 1 in Supplementary Materials | |
| pSB100X (250 ng/µL) | Mátés et al., 2009. Provide by Prof. Zsuzsanna Izsvak | ||
| pT2-CMV-GMCSF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| pT2-CMV-PEDF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| QIAamp DNA Mini Kit | QIAGEN | 51304 | |
| recombinant hGM-CSF | Peprotech | 100-11 | |
| recombinant hPEDF | BioProductsMD | 004-096 | |
| ReliaPrep RNA Cell Miniprep System | Promega | Z6011 | |
| RIPA buffer | ThermoFisher Scientific | 89901 | |
| RNase-free DNase Set | QIAGEN | 79254 | |
| RNeasy Mini Kit | QIAGEN | 74204 | |
| SDS | Applichem | A2572 | |
| Semi-dry transfer system for WB | Bio-Rad | ||
| SuperMix qScript | Quantabio | 95048-025 | |
| Tris-buffered saline (TBS) | ThermoFisher Scientific | 15504020 | |
| Triton X-100 | AppliChem | A4975 | |
| Trypsin/EDTA | Sigma-Aldrich | T4174 | |
| Tween | AppliChem | A1390 | |
| Urea | ThermoFisher Scientific | 29700 | |
| WesternBright ECL HRP substrate | Advansta | K-12045-D50 | |
| Whatman nitrocellulose membrane | Chemie Brunschwig | MNSC04530301 |
Referências
- Zareba, M., Raciti, M. W., Henry, M. M., Sarna, T., Burke, J. M. Oxidative stress in ARPE-19 cultures: Do melanosomes confer cytoprotection. Free Radical Biology and Medicine. 40 (1), 87-100 (2006).
- Gong, X., Draper, C. S., Allison, G. S., Marisiddaiah, R., Rubin, L. P. Effects of the macular carotenoid lutein in human retinal pigment epithelial cells. Antioxidants. 6 (4), (2017).
- Sacconi, R., Corbelli, E., Querques, L., Bandello, F., Querques, G. A Review of current and future management of geographic atrophy. Ophthalmology and Therapy. 6, 69-77 (2017).
- Al-Zamil, W. M., Yassin, S. A. Recent developments in age-related macular degeneration: a review. Clinical Interventions in Aging. 12, 1313-1330 (2017).
- Kumar-Singh, R. The role of complement membrane attack complex in dry and wet AMD - From hypothesis to clinical trials. Experimental Eye Research. 184, 266-277 (2019).
- Ung, L., Pattamatta, U., Carnt, N., Wilkinson-Berka, J. L., Liew, G., White, A. J. R. Oxidative stress and reactive oxygen species. Clinical Science. 131, 2865-2883 (2017).
- Beatty, S., Koh, H. H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Alhasani, R. H., et al. Gypenosides protect retinal pigment epithelium cells from oxidative stress. Food and Chemical Toxicology. 112, 76-85 (2018).
- . National Institute of Health Available from: https://nei.nih.gov/learn-about-eye-health/resources-for-health-educators/eye-health-data-and-statistics/age-related-macular-degeneration-amd-data-and-statistics (2020)
- Mitchell, P., Liew, G., Gopinath, B., Wong, T. Y. Age-related macular degeneration. The Lancet. 392, 1147-1159 (2018).
- He, Y., Leung, K. W., Ren, Y., Jinzhi, P., Jian, G., Tombran-Tink, J. PEDF improves mitochondrial function in RPE cells during oxidative stress. Investigative Ophthalmology & Visual Science. 55, 6742-6755 (2014).
- Cao, S., Walker, G. B., Wang, X., Cui, J. Z., Matsubara, J. A. Altered cytokine profiles of human retinal pigment epithelium: Oxidant injury and replicative senescence. Molecular Vision. 19, 718-728 (2013).
- Farnoodian, M., Sorenson, C. M., Sheibani, N. PEDF expression affects the oxidative and inflammatory state of choroidal endothelial cells. Amercian Journal of Physiology and Cell Physiology. 314 (4), 456-472 (2018).
- Polato, F., Becerra, S. P. Retinal Degenerative Diseases: Mechanisms and Experimental Therapies. Retinal Degenerative Diseases. , 699-706 (2016).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF regulates the ERK1/2 pathways and protects injured retinal ganglion cells from induced death. Experimental Eye Research. 89, 665-677 (2009).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF protects rat photoreceptors from death by activating the SRC-dependent signalling and elevating anti-apoptotic factors and neurotrophins. Graefes Archives for Clinical and Experimental Ophthalmology. 250, 699-712 (2012).
- Thumann, G., et al. Engineering of PEDF-expressing primary pigment epithelial cells by the SB transposon system delivered by pFAR4 plasmids. Molecular Therapy - Nucleic Acids. 6, 302-314 (2017).
- Garcia-Garcia, L., et al. Long-term PEDF release in rat iris and retinal epithelial cells after Sleeping Beauty transposon-mediated gene delivery. Molecular Therapy - Nucleic Acids. 9, 1-11 (2017).
- Johnen, S., et al. Antiangiogenic and neurogenic activities of Sleeping Beauty-mediated PEDF-transfected RPE cells in vitro and in vivo. BioMed Research International. 2015, (2015).
- Weigel, A. L., Handa, J. T., Hjelmeland, M. L. Microarray analysis of H2O2-, HNE-, or tBH-treated ARPE-19 cells. Free Radical Biology & Medicine. 33 (10), 1419-1432 (2002).
- Allen, R. G., Tresini, M. Oxidative stress and gene regulation. Free Radical Biology and Medicine. 28 (3), 463-499 (2000).
- Tate, D. J., Miceli, M. V., Newsome, D. A. Phagocytosis and H2O2 induce catalase and metallothionein gene expression in human retinal pigment epithelial cells. Investigative Ophthalmology and Visual Science. 36 (7), 1271-1279 (1995).
- Halliwell, B., Clement, M. V., Long, L. H. Hydrogen peroxide in the human body. FEBS Letters. 486 (1), 10-13 (2000).
- Giblin, F. J., McCready, J. P., Kodama, T., Reddy, V. N. A direct correlation between the levels of ascorbic acid and H2O2 in aqueous humor. Experimental Eye Research. 38, 87-93 (1984).
- Geiger, R. C., Waters, C. M., Kamp, D. W., Glucksberg, M. R. KGF prevents oxygen-mediated damage in ARPE-19 cells. Investigative Ophthalmology and Visual Science. 46, 3435-3442 (2005).
- Campochiaro, P. A., et al. Lentiviral vector gene transfer of endostatin/angiostatin for macular degeneration (GEM) study. Human Gene Therapy. 28, 99-111 (2017).
- Chen, X. -. D., Su, M. -. Y., Chen, T. -. T., Hong, H. -. Y., Han, A. -. D., Li, W. -. S. Oxidative stress affects retinal pigment epithelial cell survival through epidermal growth factor receptor/AKT signaling pathway. International Journal of Ophthalmology. 10 (4), 507-514 (2017).
- Tu, G., et al. Allicin attenuates H2O2 - induced cytotoxicity in retinal pigmented epithelial cells by regulating the levels of reactive oxygen species. Molecular Medicine Reports. 13, 2320-2326 (2016).
- Hao, Y., Liu, J., Wang, Z., Yu, L., Wang, J. Piceatannol protects human retinal pigment epithelial cells against hydrogen peroxide induced oxidative stress and apoptosis through modulating. Nutrients. 11, 1-13 (2019).
- Ballinger, S. W., Van Houten, B., Conklin, C. A., Jin, G. F., Godley, B. F. Hydrogen peroxide causes significant mitochondrial DNA damage in human RPE cells. Experimental Eye Research. 68 (6), 765-772 (1999).
- Ma, S., et al. Transgenic overexpression of uncoupling protein 2 attenuates salt-induced vascular dysfunction by inhibition of oxidative stress. American Journal of Hypertension. 27 (3), 345-354 (2014).
- Johnen, S., et al. Sleeping Beauty transposon-mediated transfection of retinal and iris pigment epithelial cells. Investigative Ophthalmology and Visual Science. 53 (8), 4787-4796 (2012).
- Mátés, L., et al. Molecular evolution of a novel hyperactive Sleeping Beauty transposase enables robust stable gene transfer in vertebrates. Nature Genetics. 41 (6), 753-761 (2009).
- . Marienfeld Technical information Neubauer-improved Available from: https://www.marienfeld-superior.com/information-about-our-counting-chambers.html (2020)
- . Electron Microscopy Sciences. Neubauer Haemocytometry Available from: https://www.emsdiasum.com/microscopy/technical/datasheet/68052-14.aspx (2020)
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2^(-ΔΔCT) method. Methods. 25 (4), 402-408 (2001).
- Bascuas, T., et al. Non-virally transfected primary human pigment epithelium cells overexpressing the oxidative stress reduction factors PEDF and GM-CSF to treat retinal neurodegeneration neurodegenerationl. Human Gene Therapy. 30 (11), (2019).
- Zhuge, C. C., et al. Fullerenol protects retinal pigment epithelial cells from oxidative stress-induced premature senescence via activating SIRT1. Investigative Ophthalmology & Visual Science. 55 (7), 4628-4638 (2014).
- Kaczara, P., Sarna, T., Burke, M. Dynamics of H2O2 Availability to ARPE-19 cultures in models of oxidative stress. Free Radical Biology and Medicine. 48 (8), 1068-1070 (2010).
- Gorrini, C., Harris, I. S., Mak, T. W. Modulation of oxidative stress as an anticancer strategy. Nature Reviews Drug Discovery. 12, 931-947 (2013).
- Wang, X., et al. PEDF protects human retinal pigment epithelial cells against oxidative stress via upregulation of UCP2 expression. Molecular Medicine Reports. 19 (1), 59-74 (2019).
- Donadelli, M., Dando, I., Fiorini, C., Palmieri, M. UCP2, a mitochondrial protein regulated at multiple levels. Cellular and Molecular Life Sciences. 71, 1171-1190 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados