Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Induktion und Analyse von oxidativem Stress in Dornröschen-Transposon-transfizierten menschlichen retinalen Pigmentepithelzellen
In diesem Artikel
Zusammenfassung
Wir stellen ein Protokoll für die Entwicklung und Verwendung eines oxidativen Stressmodells vor, indemwir retinale Pigmentepithelzellen mit H2O2behandeln und Zellmorphologie, Lebensfähigkeit, Dichte, Glutathion und UCP-2-Spiegel analysieren. Es ist ein nützliches Modell, um die antioxidative Wirkung von Proteinen zu untersuchen, die von Transposon-transfizierten Zellen sezerniert werden, um neuroretinale Degeneration zu behandeln.
Zusammenfassung
Oxidativer Stress spielt eine entscheidende Rolle bei mehreren degenerativen Erkrankungen, einschließlich der altersbedingten Makuladegeneration (AMD), einer Pathologie, von der weltweit etwa 30 Millionen Patienten betroffen sind. Es führt zu einer Abnahme der retinalen Pigmentepithel (RPE) -synthetisierten neuroprotektiven Faktoren, z. B. Pigmentepithel-abgeleiteter Faktor (PEDF) und Granulozyten-Makrophagen-Kolonie-stimulierender Faktor (GM-CSF), gefolgt vom Verlust von RPE-Zellen und schließlich dem Tod von Photorezeptoren und retinalen Ganglienzellen (RGC). Wir stellen die Hypothese auf, dass die Rekonstitution der neuroprotektiven und neurogenen netztinalen Umgebung durch die subretinale Transplantation von transfizierten RPE-Zellen, die PEDF und GM-CSF überexprimieren, das Potenzial hat, die Degeneration der Netzhaut zu verhindern, indem sie die Auswirkungen von oxidativem Stress mildert, Entzündungen hemmt und das Überleben der Zellen unterstützt. Mit dem Sleeping Beauty Transposon-System (SB100X) wurden menschliche RPE-Zellen mit den PEDF- und GM-CSF-Genen transfiziert und zeigten eine stabile Genintegration, langfristige Genexpression und Proteinsekretion mittels qPCR, Western Blot, ELISA und Immunfluoreszenz. Um die Funktionalität und Die Wirksamkeit der von den transfizierten RPE-Zellen sezernierten PEDF und GM-CSF zu bestätigen, haben wir einen In-vitro-Assay entwickelt, um die Reduktion vonH2O2-induziertemoxidativem Stress auf RPE-Zellen in Kultur zu quantifizieren. Der Zellschutz wurde durch die Analyse der Zellmorphologie, der Dichte, des intrazellulären Glutathionspiegels, der UCP2-Genexpression und der Zelllebensfähigkeit bewertet. Sowohl transfizierte RPE-Zellen, die PEDF und/oder GM-CSF überexprimieren, als auch Zellen, die nicht transfiziert, aber mit PEDF und/oder GM-CSF (kommerziell erhältlich oder aus transfizierten Zellen gereinigt) vorbehandelt wurden, zeigten im Vergleich zu nicht behandelten Kontrollen einen signifikanten antioxidativen Zellschutz. Das vorliegendeH2O2-Modellist ein einfacher und effektiver Ansatz zur Bewertung der antioxidativen Wirkung von Faktoren, die zur Behandlung von AMD oder ähnlichen neurodegenerativen Erkrankungen wirksam sein können.
Einleitung
Das hier beschriebene Modell bietet einen nützlichen Ansatz, um die Wirksamkeit biopharmazeutischer Wirkstoffe zur Reduzierung von oxidativem Stress in Zellen zu bewerten. Wir haben das Modell verwendet, um die schützenden Wirkungen von PEDF und GM-CSF auf denH2O2-vermitteltenoxidativen Stress auf retinale Pigmentepithelzellen, die hohen Konzentrationen vonO2und sichtbarem Licht ausgesetzt sind, und die Phagozytose von Photorezeptor-Außensegmentmembranen zu untersuchen, wodurch signifikante Mengen an reaktiven Sauerstoffspezies (ROS) erzeugt werden1. (2)Die Mitgliedstaaten sind der Ansicht, Sie gelten als hauptverursacher der Pathogenese der avaskulären altersbedingten Makuladegeneration (aAMD)3,4,5,6,7,8. Außerdem gibt es eine Abnahme der RPE-synthetisierten neuroprotektiven Faktoren, insbesondere des Pigmentepithel-abgeleiteten Faktors (PEDF), der insulinähnlichen Wachstumsfaktoren (IGFs) und des Granulozyten-Makrophagen-Kolonie-stimulierenden Faktors (GM-CSF), was zur Dysfunktion und zum Verlust von RPE-Zellen führt, gefolgt vom Tod durch Photorezeptoren und retinale Ganglienzellen (RGC)3,4,5 . AMD ist eine komplexe Erkrankung, die aus der Wechselwirkung zwischen metabolischen, funktionellen, genetischen und Umweltfaktorenresultiert 4. Der Mangel an Behandlungen für aAMD ist die Hauptursache für Blindheit bei Patienten älter als 60 Jahre in Industrieländern9,10. Die Rekonstitution der neuroprotektiven und neurogenen Netzhautumgebung durch die subretinale Transplantation genetisch veränderter RPE-Zellen, die PEDF und GM-CSF überexprimieren, hat das Potenzial, eine Netzhautdegeneration zu verhindern, indem die Auswirkungen von oxidativem Stress gemildert, Entzündungen gehemmt und das Überleben der Zellen unterstütztwerden 11,12,13,14,15,16 . Obwohl es mehrere Methoden gibt, um Gene an Zellen zu liefern, haben wir das nicht-virale hyperaktive Dornröschen-Transposon-System gewählt, um die PEDF- und GM-CSF-Gene an RPE-Zellen zu liefern, aufgrund seines Sicherheitsprofils, der Integration der Gene in das Genom der Wirtszellen und seiner Neigung, die gelieferten Gene in nicht-transkriptionell aktive Stellen zu integrieren, wie wir zuvor gezeigt haben17. 18,19.
Zellulärer oxidativer Stress kann in Zellen induziert werden, die in vitrodurch mehrere oxidative Wirkstoffe kultiviert werden, einschließlich Wasserstoffperoxid(H2O2),4-Hydroynonenal (HNE), Tertbutylhydroperoxid (tBH), hohe Sauerstoffspannungen und sichtbares Licht (Vollspektrum- oder UV-Bestrahlung)20,21. Hohe Sauerstoffspannungen und Licht erfordern spezielle Geräte und Bedingungen, die die Übertragbarkeit auf andere Systeme einschränken. Wirkstoffe wieH2O2,HNE und tBH induzieren überlappende molekulare und zelluläre Veränderungen des oxidativen Stresses. Wir wählten H2O2,um die antioxidative Aktivität von PEDF und GM-CSF zu testen, weil es praktisch und biologisch relevant ist, da es von RPE-Zellen als reaktives Sauerstoffzwischenprodukt während der Photorezeptor-Phagozytose des äußeren Segments22 produziert wird und in Augengewebe in vivo gefunden wird 23. Da die Oxidation von Glutathion teilweise für die Produktion vonH2O2im Auge verantwortlich sein kann, haben wir in unseren Studien die GSH/Glutathion-Spiegel analysiert, die mitH2O2-induziertemoxidativem Stress und der Regenerationsfähigkeit der Zellenzusammenhängen 21,22. Die Analyse des Glutathionspiegels ist besonders relevant, da es an den antioxidativen Schutzmechanismen im Augebeteiligt ist 24. Die Exposition gegenüber H2O2wird häufig als Modell verwendet, um die Anfälligkeit für oxidativen Stress und die antioxidative Aktivität der RPE-Zellen1,25,26,27,28,29,30zuuntersuchen, und zeigt zusätzlich Ähnlichkeiten mit lichtinduzierten oxidativen Stressschäden, einer "physiologischen" Quelle von oxidativem Stress21.
Um die Funktionalität und Wirksamkeit neuroprotektiver Faktoren zu bewerten, haben wir ein In-vitro-Modell entwickelt, das die Analyse ermöglicht, um die antioxidative Wirkung von Wachstumsfaktoren zu quantifizieren, die von Zellen exprimiert werden, die genetisch verändert wurden, um PEDF und GM-CSF zu überexprimieren. Hier zeigen wir, dass RPE-Zellen, die mit den Genen für PEDF und GM-CSF transfiziert wurden, resistenter gegen die schädlichen Wirkungen von H2O2 sind als nicht-transfizierte Kontrollzellen, wie Zellmorphologie, Dichte, Lebensfähigkeit, intrazellulärer Glutathionspiegel und Expression des UCP2-Gens zeigen, das für das mitochondriale Entkopplungsprotein 2 kodiert, das nachweislich reaktive Sauerstoffspezies (ROS)reduziert 31.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Verfahren zur Entnahme und Verwendung menschlicher Augen wurden von der kantonalen Ethikkommission für Forschung (Nr. 2016-01726) genehmigt.
1. Zellisolierung und Kulturbedingungen
- Humane ARPE-19 Zelllinie
- Kultur 5 x 105 ARPE-19-Zellen, eine humane RPE-Zelllinie, in Dulbecco's Modified Eagle's Medium/Nutrient Mixture F-12 Ham (DMEM/Ham's F-12), ergänzt mit 10% fetalem Rinderserum (FBS), 80 U/ml Penicillin, 80 μg/ml Streptomycin und 2,5 μg/ml Amphotericin B (komplettes Medium) bei 37 °C in einer befeuchteten Atmosphäre von 5 %CO2 und 95 % Luft in einem T75-Kolben (für andere Zelldichten siehe Tabelle 1).
- Wechseln Sie das Medium dreimal pro Woche.
- Sobald die Zellen zu etwa 90% Konfluenz gewachsen sind (qualitativ bewertet), aspirieren Sie das Medium und waschen Sie die Zellen mit sterilem 1x PBS.
- Inkubieren Sie die Zellen mit einer 5% igen Trypsin-2% EDTA-Lösung für 7-10 min bei 37 °C (Volumina siehe Tabelle 1). Überwachen Sie die Ablösung visuell.
- Stoppen Sie die Trypsinisierung, indem Sie ein komplettes Medium mit 10% FBS hinzufügen (für Volumes siehe Tabelle 1).
- Transfizieren Sie die Zellen (siehe Schritt 2 des Protokolls), subkultivieren Sie die Zellen in einem Verhältnis von 1:10 (einmal pro Woche) oder säen Sie in eine 96-Well-Platte, wie unten beschrieben (siehe Schritte 3.3 und 3.4 des Protokolls).
| Mittel (ml) | ||||||
| Fläche (cm²) | Seeding-Dichte für ARPE-19 Zellen (Zellen/Well) | Anwendung | Für die Zellkultur | So stoppen Sie Trypsin | Volumen von Trypsin (ml) | |
| Kolben T75 | 75 | 5,00,000 | ARPE-19 Zellwachstum | 10 | 7 | 3 |
| 6 Brunnenplatte | 9.6 | 1,00,000 | Seeding von transfizierten ARPE-19 Zellen | 3 | 1 | 0.5 |
| 24 Brunnenplatte | 2 | 50,000 | Seeding von transfizierten hRPE-Zellen | 1 | 0.8 | 0.2 |
| 96 Brunnenplatte | 0.32 | 5.000 für oxidative Stressexperimente mit transfizierten Zellen (Abb. 1) | Experimente mit oxidativem Stress | 0.2 | ||
| 3.000 für oxidative Stressexperimente mit nicht-transfizierten Zellen plus Proteinen (Abb. 1) | ||||||
Tabelle 1: Zellkulturvolumen. Empfohlene Medienvolumina für Zellkulturplatten und Kolben für die Kultur von ARPE-19 und primären menschlichen RPE-Zellen.
- Primäre menschliche RPE-Zellen
- Isolieren Sie primäre humane RPE-Zellen, wie von Thumann et al.17beschrieben, und kultivieren Sie Zellen in einem vollständigen Medium, das mit 20% FBS ergänzt wird.
- Wechseln Sie das Medium zweimal pro Woche. Sobald die Zellen die Konfluenz erreicht haben (visuell überwacht), reduzieren Sie FBS auf 1%, um ein Überwachsen zu vermeiden.
- Transfizieren Sie die Zellen (siehe Schritt 2 des Protokolls) oder setzen Sie sie in eine 96-Well-Platte, wie unten beschrieben (siehe Schritte 3.3 und 3.4 des Protokolls).
HINWEIS: Die hier vorgestellten Daten stammen aus der Kultur von RPE-Zellen, die aus den Augen von vier menschlichen Spendern gewonnen wurden. Tabelle 2 zeigt die demographischen Daten der Spender der Lions Gift of Sight Eye Bank (Saint Paul, MN). Die Augen wurden 12,7 ± 5,7 h (mittlere ± SD) post mortem enukleiert, nachdem die Einverständniserklärung gemäß der Erklärung von Helsinki eingeholt worden war.
| Nein | Alter | Geschlecht | Tod bis zur Erhaltung (Stunden) | Tod durch Isolation | Anbau | Anbau | Symbol in Der Grafik | |
| (Tage) | vor der Transfektion (Tage) | nach Transfektion (Tage) | ||||||
| 2 | 80 | M | 20.7 | 8 | 140 | 36 |  | |
| 3 | 86 | F | 12.8 | 8 | 85 | 45 |  | |
| 4 | 86 | F | 8.5 | 5 | 26 | 133 |  | |
| 8 | 83 | F | 8.9 | 6 | 18 | 27 |  | |
| bedeuten | 83.8 | 12.7 | 6.8 | 67.3 | 60.3 | |||
| SD | 2.9 | 5.7 | 1.5 | 57.0 | 49.1 |
Tabelle 2: Demographie menschlicher Spender für retinale Pigmentepithelzellen.
2. Elektroporation von ARPE-19 und primären menschlichen RPE-Zellen
- Trypsinisieren Sie ARPE-19-Zellen oder primäre menschliche RPE-Zellen, wie in den Schritten 1.1.3-1.1.5 des Protokolls beschrieben.
- Führen Sie die Elektroporation mit dem handelsüblichen Transfektionskit durch (siehe Materialtabelle).
- Zur Transfektion von ARPE-19-Zellen siehe Johnen et al.32 und für primäre hRPE Thumann et al.17. Kurz gesagt, resuspend 1 x 105 ARPE-19 Zellen oder 5 x 104 primäre hRPE-Zellen in 11 μL R-Puffer und fügen Sie 2 μL Plasmidmischung mit 0,03 μg pSB100X Transposase33 und 0,47 μg pT2-CMV-PEDF-His oder pT2-CMV-GMCSF-His Transposon hinzu (Verhältnis Transposase:Transposon 1:16). Verwenden Sie für PEDF- und GM-CSF-Doppeltransfizierte Zellen ein Verhältnis von 1:16:16 (0,03 μg pSB100X, 0,47 μg pT2-CMV-PEDF-His und 0,47 μg pT2-CMV-GMCSF-His). Verwenden Sie die folgenden Elektroporationsparameter: zwei Impulse von 1.350 V für 20 ms (Pulsbreite) für ARPE-19-Zellen; zwei Impulse von 1.100 V für 20 ms für Primärzellen.
- Samen 1 x 105 transfizierte ARPE-19 oder 5 x10 4 transfizierte primäre hRPE-Zellen in 6-Well- bzw. 24-Well-Platten, in Medium, ergänzt mit 10% FBS ohne Antibiotika oder Antimykotika. Fügen Sie Penicillin (80 U / ml), Streptomycin (80 μg / ml) und Amphotericin B (2,5 μg / ml) mit dem ersten Medienaustausch 3 Tage nach der Transfektion hinzu.
- Bestimmen Sie das Zellwachstum durch wöchentliche mikroskopische Überwachung der Zellen. Die Transfektionseffizienz wird durch die Analyse der Genexpression mittels RT-PCR und der Proteinsekretion durch ELISA und WB (Methoden, die in Supplementary Materialerläutert werden) überwacht.
HINWEIS: Die Transfektionseffizienz kann zum ersten Mal bewertet werden, sobald die Zellen die Konfluenz erreicht haben, d.h. nach ~ 7 Tagen bzw. 4 Wochen nach der Transfektion für ARPE-19-Zellen bzw. primäre hRPE-Zellen. - Samenzellen in einer 96-Well-Platte, wie unten beschrieben (siehe Schritt 3.5 des Protokolls).
3. Induktion vonoxidativem Stress (H2O2-Behandlung) und Neuroprotektion (PEDF- und/oder GM-CSF-Behandlung)
- Herstellung eines konditionierten Mediums aus transfizierten ARPE-19-Zellen
- Verwenden Sie ARPE-19-Zellen, die mit den Genen PEDF, GM-CSF oder beidem transfiziert sind (siehe Schritt 2 des Protokolls); Kulturzellen für 28 Tage, wie in Schritt 1.1 des Protokolls beschrieben.
- Nach 28 Tagen nach der Transfektion trypsinisieren sie die Zellen (siehe Schritte 1.1.3-1.1.5 des Protokolls), zählen die Zellen unter Verwendung einer Neubauer-Kammer34,35und seeden 5 x 10 5 Zellen in T75-Kolben in vollständigem Medium, wie in Schritt 1.1.1 des Protokolls beschrieben. Tauschen Sie das Medium aus, wenn die Zellkultur zu ca. 80% konfluent ist (ca. nach 1 Woche; qualitativ verifiziert). Sammeln Sie das Medium nach 24 h.
- Lagern Sie das Medium bis zum Gebrauch bei -20 °C.
ANMERKUNG: Die ausreichende Konzentration des rekombinanten PEDF und DES GM-CSF im konditionierten Medium wurde durch WB nachgewiesen und mittels ELISA quantifiziert, wie unter Ergänzendes Materialbeschrieben.
- Reinigung von PEDF und GM-CSF aus konditioniertem Medium transfizierter ARPE-19-Zellen
- Zentrifugieren Sie das gesammelte Medium aus Schritt 3.1.2 bei 10.000 x g für 15 min bei 4 °C.
- Verwenden Sie den Ni-NTA-Superflow (siehe Materialtabelle)gemäß den Protokollen des Herstellers, um His-markierte Proteine wie unten beschrieben zu reinigen.
- 30 μL Ni-NTA-Gemisch in ein 1,5-ml-Röhrchen pipettieren und bei 2.600 x g für 30 s zentrifugieren und den Durchfluss verwerfen. Waschen Sie das Pellet zweimal mit 200 μL 1x Inkubationspuffer.
- Zentrifugiere bei 2.600 x g für 30 s und entsorge den Durchfluss. Fügen Sie 40 μL 4x Inkubationspuffer hinzu und resuspendieren Sie.
- 900 μL zentrifugiertes konditioniertes Medium zugeben und bei 70 U/min (Orbitalschüttler) für 1 h RT inkubieren. Zentrifuge bei 2.600 x g für 1 min und den Discard-Durchfluss.
- Waschen Sie das Pellet zweimal mit 175 μL 1x Inkubationspuffer. Zentrifugiere bei 2.600 x g für 30 s und entsorge den Durchfluss.
- Um His-tagged PEDF- und GM-CSF-Proteine zu eluieren, fügen Sie 20 μL Elutionspuffer hinzu und inkubieren Sie bei 70 U / min (Orbital-Shaker) für 20 min bei RT. Zentrifuge bei 2.600 x g für 30 s. Bewahren Sie den Überstand auf, der rekombinantes PEDF oder GM-CSF enthält.
- Quantifizieren Sie das Gesamtprotein mit dem handelsüblichen BCA-Protein-Assay-Kit (siehe Materialtabelle) gemäß den Anweisungen des Herstellers.
- Die Proteinlösung wird bis zur Anwendung bei -20 °C gelagert.
ANMERKUNG: Inkubationspuffer (4x) enthält 200 mMNaH2PO4,1,2 M NaCl und 40 mM Imidazol; Elutionspuffer enthält 50 mMNaH2PO4,300 mM NaCl und 250 mM Imidazol.
- Behandlung von nicht-transfizierten ARPE-19/primären hRPE-Zellen mit konditioniertem Medium plusH2O2(Abbildung 1A)
- Säen Sie 3.000 nicht transfizierte ARPE-19 (aus Schritt 1.1.6 des Protokolls) oder primäre hRPE-Zellen (aus Schritt 1.2.3 des Protokolls) pro Vertiefung in 96-Well-Platte und Kultur in 200 μL konditioniertem Medium aus transfizierten ARPE-19-Zellen.
- Kultivieren Sie die Zellen für 10 Tage bei 37 °C in einer befeuchteten Atmosphäre von 5%CO2 und 95% Luft. Wechseln Sie das konditionierte Medium jeden Tag. Setzen Sie die Zellen 24 h lang 350μMH2O2 aus.
- Bewerten Sie oxidative Stressschäden und bestimmen Sie die antioxidative Wirkung von PEDF und GM-CSF durch Quantifizierung der Glutathionspiegel (siehe Schritt 4.1 des Protokolls), der Mikroskopie (siehe Schritt 4.2 des Protokolls) und des Zytotoxizitätstests (siehe Schritt 4.2 des Protokolls).
HINWEIS: Die Dauer des Experiments beträgt 12 Tage. Klare Mikrotiterplatten mit flachem Boden werden verwendet, um die Lumineszenz sowie die Zellmorphologie zu bewerten. Um den Zytotoxizitäts- und Glutathion-Assay gleichzeitig durchzuführen, müssen zwei Platten am selben Tag mit Zellen ausgesät werden.
- Behandlung von nicht-transfizierten ARPE-19/primären hRPE-Zellen mit PEDF- und GM-CSF-Wachstumsfaktoren plusH2O2(Abbildung 1B)
- Säen Sie 3.000 nicht transfizierte ARPE-19 (aus Schritt 1.1.6 des Protokolls) oder primäre hRPE-Zellen (aus Schritt 1.2.3 des Protokolls) pro Vertiefung (96-Well-Platten mit einem klaren flachen Boden) in 200 μL vollständigem Kulturmedium mit 500 ng/ml rekombinantem PEDF und/oder 50 ng/ml rekombinantem GM-CSF, gereinigt aus dem Medium transfizierter ARPE-19-Zellen oder kommerziell erhältlich. Kulturzellen für 48 h bei 37 °C in einer befeuchteten Atmosphäre von 5%CO2 und 95% Luft. Erneuern Sie das Medium einschließlich PEDF- und GM-CSF-Wachstumsfaktoren täglich.
HINWEIS: Fügen Sie die Wachstumsfaktoren frisch zum Medium hinzu. - Nach 48 h Behandlung der Zellen mit den Wachstumsfaktoren wird das Medium entfernt und ein komplettes Medium mit 350 μMH2O2plus 500 ng/mL PEDF und/oder 50 ng/mL GM-CSF zugegeben.
- Bewerten Sie oxidative Stressschäden und bestimmen Sie die antioxidative Wirkung von PEDF und GM-CSF durch Quantifizierung der Glutathionspiegel (siehe Schritt 4.1 des Protokolls), der Mikroskopie (siehe Schritt 4.2 des Protokolls) und des Zytotoxizitätstests (siehe Schritt 4.2 des Protokolls).
HINWEIS: Die Dauer des Experiments beträgt 3 Tage.
- Säen Sie 3.000 nicht transfizierte ARPE-19 (aus Schritt 1.1.6 des Protokolls) oder primäre hRPE-Zellen (aus Schritt 1.2.3 des Protokolls) pro Vertiefung (96-Well-Platten mit einem klaren flachen Boden) in 200 μL vollständigem Kulturmedium mit 500 ng/ml rekombinantem PEDF und/oder 50 ng/ml rekombinantem GM-CSF, gereinigt aus dem Medium transfizierter ARPE-19-Zellen oder kommerziell erhältlich. Kulturzellen für 48 h bei 37 °C in einer befeuchteten Atmosphäre von 5%CO2 und 95% Luft. Erneuern Sie das Medium einschließlich PEDF- und GM-CSF-Wachstumsfaktoren täglich.
- Behandlung von transfizierten ARPE-19/primären hRPE-Zellen mitH2O2( Abbildung 1C)
- Überprüfung einer ausreichenden Genexpression und Proteinsekretion transfizierter Zellen mittels WB und ELISA, wie im Ergänzungsmaterialbeschrieben.
- Entfernen Sie das Medium aus den Vertiefungen, die die transfizierten Zellen enthalten (siehe Schritt 2 des Protokolls).
- Trypsinisieren Sie Zellen wie in den Schritten 1.1.3-1.1.5 des Protokolls beschrieben. Zählen Sie die Zellen mit einer Neubauer-Kammer34,35.
- Säen Sie 5.000 transfizierte Zellen / Well in 96-Well-Platte in 200 μL komplettem Medium. Kulturzellen für 24 h bei 37 °C in einer befeuchteten Atmosphäre von 5%CO2 und 95% Luft. Nach 24 h werden die Zellen 24h lang 350 μMH2O2 ausgesetzt.
- Bewerten Sie oxidative Stressschäden und bestimmen Sie die antioxidative Wirkung von PEDF und GM-CSF durch Quantifizierung der Glutathionspiegel (siehe Schritt 4.1 des Protokolls), Mikroskopie (siehe Schritt 4.2 des Protokolls), Zytotoxizitätstest (siehe Schritt 4.2 des Protokolls) und Bestimmung der UCP2-Genexpression (siehe Schritt 4.3 des Protokolls).
HINWEIS: Die Dauer des Experiments beträgt 2 Tage.
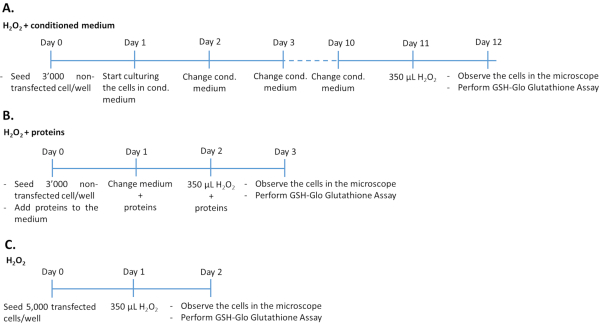
Abbildung 1: Zeitlinien des H2O2-Assays in den drei verschiedenen experimentellen Ansätzen. 3.000 nicht transfizierte Zellen, die mit den konditionierten mediums/rekombinanten Proteinen behandelt wurden, oder 5.000 transfizierte Zellen wurden zur Behandlung mitH2O2in 96-Well-Platten ausgesät. Um die Wirkung des konditionierten Mediums zu bestimmen, wurden die Zellen an 10 aufeinanderfolgenden Tagen in 100% kultiviertem Medium kultiviert, wobei das Medium jeden Tag gewechselt wurde. Um die Wirkung rekombinanter Wachstumsfaktoren zu bestimmen, wurden die Zellen kultiviert, indem an 3 aufeinanderfolgenden Tagen täglich die entsprechende Menge an Wachstumsfaktoren hinzugefügt wurde. Beachten Sie, dass nicht transfizierte Zellen mit 3.000 Zellen pro Vertiefung ausgesät wurden, um ein Überwachsen während der längeren Kulturdauer im Vergleich zu transfizierten Zellen zu vermeiden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Analyse des oxidativen Stressniveaus und der antioxidativen Kapazität
- Glutathion-Assay
- Messen Sie den Glutathionspiegel (GSH) mit dem handelsüblichen Kit (siehe Materialtabelle)gemäß den Anweisungen des Herstellers. Kurz vorbereiten und entsprechendes Volumen von 1x Reagenzmischung (100 μL Reagenz/Well) anfertigen: Luciferin-NT-Substrat und Glutathion-S-Transferase verdünnt 1:100 im Reaktionspuffer.
HINWEIS: Eine 96-Well-Platte benötigt 10 ml 1x Reagenzmischung, die durch Zugabe von 100 μL Luciferin-NT-Substrat und 100 μL Glutathion-S-Transferase zu 10 ml Reaktionspuffer hergestellt wird. Bereiten Sie die 1x Reagenzmischung unmittelbar vor Gebrauch vor. Lagern Sie die vorbereitete Reagenzmischung nicht für die zukünftige Verwendung. - Bereiten Sie das Luciferin-Detektionsreagenz vor, indem Sie eine Flasche Rekonstitutionspuffer auf das lyophilisierte Luciferin-Detektionsreagenz geben.
- Bereiten Sie eine Standardkurve mit einer Glutathion (GSH) Standardlösung (5 mM) vor. Verdünnen Sie 5 mM GSH-Lösung 1:100 mit dH2O (fügen Sie 10 μL 5 mM GSH-Lösung zu 990 μLdH2O hinzu). 7 serielle 1:1-Verdünnung in 500 μLdH2O durchführen. 10 μL jedes verdünnten Standards in doppelter Duplikation in eine geeignete Vertiefung überführen.
HINWEIS: Die Endkonzentration von Glutathion liegt zwischen 0,039 μM und 5 μM. - Bereiten Sie den Rohling (1x Reagenzienmischung) vor und geben Sie 10 μL (Duplikate) in die entsprechenden Vertiefungen.
- Entfernen Sie diemitH2O2behandelten Zellen aus dem Inkubator.
HINWEIS: Dokumentieren Sie die Morphologie der H2O2-behandelten Zellen durch Hellfeldmikroskopie (40x).
Wenn die Zellen oxidiert sind, sehen sie abgerundeter und weniger ausgebreitet aus. - Das Kulturmedium vorsichtig absaugen. Fügen Sie 100 μL vorbereitete 1x Reagenzmischung zu jeder Vertiefung hinzu. Mischen Sie die Zellen mit dem Reagenz für 15 s bei 500 U / min auf einem Orbitalschüttler.
- Die Platte bei RT für 30 Min. inkubieren. Zu jeder Vertiefung werden 100 μL rekonstituiertes Luciferin-Detektionsreagenz gegeben.
- Mischen Sie die Lösung für 15 s bei 500 U / min auf einem Orbitalschüttler. Inkubieren Sie die Platte für 15 min bei RT.
- Bestimmen Sie die Lumineszenz mit einem Plattenleser mit einem vorinstallierten Programm ADP-Glo.
HINWEIS: Legen Sie die Platte ohne Deckel in den Plattenleser.- Klicken Sie auf Layout ändern und wählen Sie die folgenden Einstellungen unter Grundparameter:Costar 96-well Platte; Top-Optik; Positionierungsverzögerung: 0,1; Startzeit der Messung: 0,0; Messintervallzeit: 1,0; Zeit zum Normalisieren der Ergebnisse: 0,0; die Verstärkung wird vom Gerät automatisch angepasst. Definieren Sie Rohlinge, Standards und Muster. Klicken Sie auf Messung starten.
- Exportieren Sie die Daten als Excel-Datei. Berechnen Sie die Konzentration von GSH in jeder Probe durch Interpolation der Standardkurve.
- Messen Sie den Glutathionspiegel (GSH) mit dem handelsüblichen Kit (siehe Materialtabelle)gemäß den Anweisungen des Herstellers. Kurz vorbereiten und entsprechendes Volumen von 1x Reagenzmischung (100 μL Reagenz/Well) anfertigen: Luciferin-NT-Substrat und Glutathion-S-Transferase verdünnt 1:100 im Reaktionspuffer.
- Zytotoxizitätstest und mikroskopische Analyse
- Aspirieren Sie das Medium aus den Zellen und geben Sie 100 μL vollständiges Medium, das 1% FBS enthält, zu jeder Vertiefung hinzu. Bringen Sie die Zellen in den Inkubator zurück.
HINWEIS: 1% FBS wird verwendet, da höhere Prozentsätze von FBS die Messung der Lumineszenz stören können, daher wird in diesem Fall 1% FBS verwendet. - Messen Sie die Zelllebensfähigkeit mit dem handelsüblichen Zytotoxizitäts-Assay-Kit (siehe Materialtabelle)gemäß den Anweisungen des Herstellers. Bereiten Sie kurz die Reagenzmischung vor und fügen Sie den Assay-Puffer zum lyophilisierten Substrat hinzu. Bereiten Sie das Lysereagenz vor, indem Sie 33 μL Digitonin zu 5 ml Assay-Puffer (für eine 96-Well-Platte) hinzufügen. Mischen Sie gut, indem Sie nach oben und unten pipettieren, um Homogenität zu gewährleisten.
HINWEIS: Für optimale Ergebnisse verwenden Sie frisch zubereitete Reagenzienmischung. Reagenzienmischung kann bei 4 °C bis zu 7 Tage gelagert werden und kann bis zu 7 Tage in Einweg-Aliquots bis zu 4 Monate bei -70 °C gelagert werden. Einfrieren und Auftauen müssen vermieden werden. Das Lysis Reagenz kann bei 4 °C bis zu 7 Tage gelagert werden. - Bereiten Sie eine Standardkurve mit unbehandelten ARPE-19-Zellen vor.
- Trypsinisieren Sie die Zellen wie in den Schritten 1.1.3-1.1.5 des Protokolls beschrieben und zählen Sie die Zellen mit einer Neubauer-Kammer34,35. Zentrifugieren Sie die Zellen bei 120 g für 10 min bei RT. Aspirieren Sie den Überstand und resuspenieren Sie das Zellpellet in DMEM/Hams F12-Medium, das 1% FBS enthält, auf eine Endkonzentration von 1 x 105 Zellen/ml.
- 7 serielle 1:1-Verdünnungen in 200 μL Medium mit 1% FBS herstellen. Übertragen Sie 100 μL jedes Standards in die entsprechenden Vertiefungen (Duplikate). Fügen Sie 50 μL Reagenzmischung zu allen Vertiefungen hinzu.
- Mischen Sie die Zellen mit dem Reagenz für 15 s bei 500 U / min auf einem Orbitalschüttler. Inkubieren Sie die Platte für 15 min bei RT. Messen Sie die Lumineszenz mit dem Plattenleser, wie in Schritt 4.1.9 des Protokolls beschrieben. 50 μL des Lysereagenzes zugeben und 15 min inkubieren. Messen Sie die Lumineszenz mit dem Plattenleser wie in Schritt 4.1.9 des Protokolls beschrieben.
- Berechnen Sie den Prozentsatz der lebensfähigen Zellen: (100 - % tote Zellen) und den Prozentsatz der toten Zellen = [1. Lumineszenzmessung ((tote Zellen in der Probe))/ 2. Lumineszenzmessung (alle Zellen tot nach Digitoninbehandlung)] x 100.
- Aspirieren Sie das Medium aus den Zellen und geben Sie 100 μL vollständiges Medium, das 1% FBS enthält, zu jeder Vertiefung hinzu. Bringen Sie die Zellen in den Inkubator zurück.
- UCP2-Expressionsanalyse mittels RT-qPCR
- Trypsinisieren Sie infizierte Zellen wie oben beschrieben (Schritte 1.1.3-1.1.5 des Protokolls).
- Zählen Sie die Zellen mit einer Neubauer-Kammer34,35.
- Säen Sie 5.000 transfizierte ARPE-19-Zellen / Well in 96-Well-Platten.
- Nach 24 h Kultur werden die Zellen mit 350 μMH2O2für24 h behandelt.
- Isolieren Sie die Gesamt-RNA mit einem kommerziellen Kit zur Isolierung von RNA aus einer geringen Anzahl von Zellen (siehe Materialtabelle)gemäß den Anweisungen des Herstellers.
- Führen Sie eine quantitative Real-Time-PCR (RT-qPCR) durch, wie unter Ergänzendes Materialbeschrieben. Kurz gesagt, erzeugen Sie cDNA durch Retrotranskription unter Verwendung einer kommerziell erhältlichen Mischung, die eine optimierte M-MLV Reverse Transkriptase enthält (siehe Tabelle der Materialien).
- Für die qPCR ist ein gebrauchsfertiger Reaktionscocktail zu verwenden, der alle Komponenten (einschließlich SYBR Green) mit Ausnahme der Primer (siehe Tabelle S1 des Ergänzungsmaterials)und der DNA-Vorlage enthält. Verwenden Sie die folgenden Thermocycling-Bedingungen: Anfängliche Denaturierung bei 95 °C für 10 min, 40 Zyklen mit Denaturierung bei 95 °C für 15 s, Glühen bei 60 °C für 30 s und Dehnung bei 72 °C für 32 s.
- Verwenden Sie die 2^(-ΔΔCT)-Methode für die Analyse36.
- Herstellung von Zelllysat für die SDS-PAGE- und WB-Analyse von pAkt (Ser473)
- Säen Sie 3 x 105 GM-CSF-transfizierte ARPE-19-Zellen/Well in 6-Well-Platten (≥21 Tage nach der Transfektion), um festzustellen, ob GM-CSF RPE-Zellen durch die Aktivierung des Akt-Überlebensweges vor Schäden durchH2O2 schützt15.
- Nach 24 h Kultur werden die Zellen 24h lang 350 μMH2O2 ausgesetzt.
- Mischen Sie 1 ml RIPA-Puffer mit 10 μL Proteasephosphatase-Inhibitor-Cocktail, 10 μL 0,5 M EDTA und 25 μL 8 M Harnstoff (Volumen, die für eine Vertiefung verwendet werden).
- Medium vorsichtig absaugen und die Zellen mit 1x PBS waschen.
- Fügen Sie das gesamte Volumen der RIPA-Puffermischung zu den Zellen hinzu.
- Pipetten Sie auf und ab.
- Sammeln Sie das Lysat in 1,5 ml Röhrchen.
- Zentrifuge bei 20.000 x g für 30 min bei 4 °C.
- Übertragen Sie den Überstand auf ein neues 1,5-ml-Rohr.
- Bestimmen Sie die pAkt-Spiegel in 15 μL unverdünntem Zelllysat durch WB, wie in Ergänzungsmaterialbeschrieben.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Induktion von oxidativem Stress in menschlichen retinalen Pigmentepithelzellen
ARPE-19 und primäre hRPE-Zellen wurden mit unterschiedlichen Konzentrationen vonH2O2für 24 h behandelt und der intrazelluläre Spiegel des Antioxidans Glutathion quantifiziert (Abbildung 2A,B). H2O2 bei 50 μM und 100 μM hatte keinen Einfluss auf die Glutathionproduktion, während bei 350 μM eine signifikante...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Das hier vorgestellte Protokoll bietet einen Ansatz zur Analyse der antioxidativen und schützenden Funktion von PEDF und GM-CSF, die von transfizierten Zellen produziert werden, die auf Zellen angewendet werden können, die mit jedem vermeintlich vorteilhaften Gen transfiziert sind. Bei gentherapeutischen Strategien, die das Ziel haben, Proteine durch Transplantation genetisch veränderter Zellen an Gewebe abzugeben, ist es wichtig, Informationen über das Niveau der Proteinexpression, die Langlebigkeit der Expression u...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Die Autoren danken Gregg Sealy und Alain Conti für die hervorragende technische Unterstützung sowie Prof. Zsuzsanna Izsvák vom Max-Delbrück-Centrum in Berlin für die freundliche Bereitstellung der pSB100X- und pT2-CAGGS- Venus-Plasmide. Diese Arbeit wurde vom Schweizerischen Nationalfonds und der Europäischen Kommission im Rahmen des Siebten Rahmenprogramms unterstützt. Z.I wurde gefördert durch den Europäischen Forschungsrat, ERC Advanced [ERC-2011-ADG 294742].
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 24-well plates | Corning | 353047 | |
| 6-well plates | Greiner | 7657160 | |
| 96-well culture plate white with clear flat bottom | Costar | 3610 | Allows to check the cells before measuring the luminescence (GSH-Glo Assay) |
| 96-well plates | Corning | 353072 | |
| Acrylamid 40% | Biorad | 161-0144 | |
| Amphotericin B | AMIMED | 4-05F00-H | |
| Antibody anti-GMCSF | ThermoFisher Scientific | PA5-24184 | |
| Antibody anti-mouse IgG/IgA/IgM | Agilent | P0260 | |
| Antibody anti-PEDF | Santa Cruz Biotechnology Inc | sc-390172 | |
| Antibody anti-penta-His | Qiagen | 34660 | |
| Antibody anti-phospho-Akt | Cell Signaling Technology | 9271 | |
| Antibody anti-rabbit IgG H&L-HRP | Abcam | ab6721 | |
| Antibody donkey anti-rabbit Alexa Fluor 594 | ThermoFisher Scientific | A11034 | |
| Antibody goat anti-mouse Alexa 488 | ThermoFisher Scientific | A-11029 | |
| ARPE-19 cell line | ATCC | CRL-2302 | |
| BSA | Sigma-Aldrich | A9418-500G | |
| chamber culture glass slides | Corning | 354118 | |
| CytoTox-Glo Cytotoxicity Assay | Promega | G9291 | |
| DAPI | Sigma-Aldrich | D9542-5MG | |
| DMEM/Ham`s F12 | Sigma-Aldrich | D8062 | |
| Duo Set ELISA kit | R&D Systems | DY215-05 | |
| EDTA | ThermoFisher Scientific | 78440 | |
| ELISAquant kit | BioProducts MD | PED613-10-Human | |
| Eyes (human) | Lions Gift of Sight Eye Bank (Saint Paul, MN) | ||
| FBS | Brunschwig | P40-37500 | |
| Fluoromount Aqueous Mounting Medium | Sigma-Aldrich | F4680-25ML | |
| FLUOstar Omega plate reader | BMG Labtech | ||
| GraphPad Prism software (version 8.0) | GraphPad Software, Inc. | ||
| GSH-Glo Glutathione Assay | Promega | V6912 | |
| hydrogen peroxide (H2O2) | Merck | 107209 | |
| ImageJ software (image processing program) | W.S. Rasband, NIH, Bethesda, MD, USA; https://imagej.nih.gov/ij/; 1997–2014 | ||
| Imidazol | Axonlab | A1378.0010 | |
| Leica DMI4000B microscope | Leica Microsystems | ||
| LightCycler 480 Instrument II | Roche Molecular Systems | ||
| LightCycler 480 SW1.5.1 software | Roche Molecular Systems | ||
| NaCl | Sigma-Aldrich | 71376-1000 | |
| NaH2PO4 | Axonlab | 3468.1000 | |
| Neon Transfection System | ThermoFisher Scientific | MPK5000 | |
| Neon Transfection System 10 µL Kit | ThermoFisher Scientific | MPK1096 | |
| Neubauer chamber | Marienfeld-superior | 640010 | |
| Ni-NTA superflow | Qiagen | 30410 | |
| Nitrocellulose | VWR | 732-3197 | |
| Omega Lum G Gel Imaging System | Aplegen Life Science | ||
| PBS 1X | Sigma-Aldrich | D8537 | |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781-100 | |
| PerfeCTa SYBR Green FastMix | Quantabio | 95072-012 | |
| PFA | Sigma-Aldrich | 158127-100G | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23227 | |
| Primers | Invitrogen | See Table 1 in Supplementary Materials | |
| pSB100X (250 ng/µL) | Mátés et al., 2009. Provide by Prof. Zsuzsanna Izsvak | ||
| pT2-CMV-GMCSF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| pT2-CMV-PEDF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| QIAamp DNA Mini Kit | QIAGEN | 51304 | |
| recombinant hGM-CSF | Peprotech | 100-11 | |
| recombinant hPEDF | BioProductsMD | 004-096 | |
| ReliaPrep RNA Cell Miniprep System | Promega | Z6011 | |
| RIPA buffer | ThermoFisher Scientific | 89901 | |
| RNase-free DNase Set | QIAGEN | 79254 | |
| RNeasy Mini Kit | QIAGEN | 74204 | |
| SDS | Applichem | A2572 | |
| Semi-dry transfer system for WB | Bio-Rad | ||
| SuperMix qScript | Quantabio | 95048-025 | |
| Tris-buffered saline (TBS) | ThermoFisher Scientific | 15504020 | |
| Triton X-100 | AppliChem | A4975 | |
| Trypsin/EDTA | Sigma-Aldrich | T4174 | |
| Tween | AppliChem | A1390 | |
| Urea | ThermoFisher Scientific | 29700 | |
| WesternBright ECL HRP substrate | Advansta | K-12045-D50 | |
| Whatman nitrocellulose membrane | Chemie Brunschwig | MNSC04530301 |
Referenzen
- Zareba, M., Raciti, M. W., Henry, M. M., Sarna, T., Burke, J. M. Oxidative stress in ARPE-19 cultures: Do melanosomes confer cytoprotection. Free Radical Biology and Medicine. 40 (1), 87-100 (2006).
- Gong, X., Draper, C. S., Allison, G. S., Marisiddaiah, R., Rubin, L. P. Effects of the macular carotenoid lutein in human retinal pigment epithelial cells. Antioxidants. 6 (4), (2017).
- Sacconi, R., Corbelli, E., Querques, L., Bandello, F., Querques, G. A Review of current and future management of geographic atrophy. Ophthalmology and Therapy. 6, 69-77 (2017).
- Al-Zamil, W. M., Yassin, S. A. Recent developments in age-related macular degeneration: a review. Clinical Interventions in Aging. 12, 1313-1330 (2017).
- Kumar-Singh, R. The role of complement membrane attack complex in dry and wet AMD - From hypothesis to clinical trials. Experimental Eye Research. 184, 266-277 (2019).
- Ung, L., Pattamatta, U., Carnt, N., Wilkinson-Berka, J. L., Liew, G., White, A. J. R. Oxidative stress and reactive oxygen species. Clinical Science. 131, 2865-2883 (2017).
- Beatty, S., Koh, H. H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Alhasani, R. H., et al. Gypenosides protect retinal pigment epithelium cells from oxidative stress. Food and Chemical Toxicology. 112, 76-85 (2018).
- National Institute of Health. , Available from: https://nei.nih.gov/learn-about-eye-health/resources-for-health-educators/eye-health-data-and-statistics/age-related-macular-degeneration-amd-data-and-statistics (2020).
- Mitchell, P., Liew, G., Gopinath, B., Wong, T. Y. Age-related macular degeneration. The Lancet. 392, 1147-1159 (2018).
- He, Y., Leung, K. W., Ren, Y., Jinzhi, P., Jian, G., Tombran-Tink, J. PEDF improves mitochondrial function in RPE cells during oxidative stress. Investigative Ophthalmology & Visual Science. 55, 6742-6755 (2014).
- Cao, S., Walker, G. B., Wang, X., Cui, J. Z., Matsubara, J. A. Altered cytokine profiles of human retinal pigment epithelium: Oxidant injury and replicative senescence. Molecular Vision. 19, 718-728 (2013).
- Farnoodian, M., Sorenson, C. M., Sheibani, N. PEDF expression affects the oxidative and inflammatory state of choroidal endothelial cells. Amercian Journal of Physiology and Cell Physiology. 314 (4), 456-472 (2018).
- Polato, F., Becerra, S. P. Retinal Degenerative Diseases: Mechanisms and Experimental Therapies. Retinal Degenerative Diseases. , Springer. 699-706 (2016).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF regulates the ERK1/2 pathways and protects injured retinal ganglion cells from induced death. Experimental Eye Research. 89, 665-677 (2009).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF protects rat photoreceptors from death by activating the SRC-dependent signalling and elevating anti-apoptotic factors and neurotrophins. Graefes Archives for Clinical and Experimental Ophthalmology. 250, 699-712 (2012).
- Thumann, G., et al. Engineering of PEDF-expressing primary pigment epithelial cells by the SB transposon system delivered by pFAR4 plasmids. Molecular Therapy - Nucleic Acids. 6, 302-314 (2017).
- Garcia-Garcia, L., et al. Long-term PEDF release in rat iris and retinal epithelial cells after Sleeping Beauty transposon-mediated gene delivery. Molecular Therapy - Nucleic Acids. 9, 1-11 (2017).
- Johnen, S., et al. Antiangiogenic and neurogenic activities of Sleeping Beauty-mediated PEDF-transfected RPE cells in vitro and in vivo. BioMed Research International. 2015, (2015).
- Weigel, A. L., Handa, J. T., Hjelmeland, M. L. Microarray analysis of H2O2-, HNE-, or tBH-treated ARPE-19 cells. Free Radical Biology & Medicine. 33 (10), 1419-1432 (2002).
- Allen, R. G., Tresini, M. Oxidative stress and gene regulation. Free Radical Biology and Medicine. 28 (3), 463-499 (2000).
- Tate, D. J., Miceli, M. V., Newsome, D. A. Phagocytosis and H2O2 induce catalase and metallothionein gene expression in human retinal pigment epithelial cells. Investigative Ophthalmology and Visual Science. 36 (7), 1271-1279 (1995).
- Halliwell, B., Clement, M. V., Long, L. H. Hydrogen peroxide in the human body. FEBS Letters. 486 (1), 10-13 (2000).
- Giblin, F. J., McCready, J. P., Kodama, T., Reddy, V. N. A direct correlation between the levels of ascorbic acid and H2O2 in aqueous humor. Experimental Eye Research. 38, 87-93 (1984).
- Geiger, R. C., Waters, C. M., Kamp, D. W., Glucksberg, M. R. KGF prevents oxygen-mediated damage in ARPE-19 cells. Investigative Ophthalmology and Visual Science. 46, 3435-3442 (2005).
- Campochiaro, P. A., et al. Lentiviral vector gene transfer of endostatin/angiostatin for macular degeneration (GEM) study. Human Gene Therapy. 28, 99-111 (2017).
- Chen, X. -D., Su, M. -Y., Chen, T. -T., Hong, H. -Y., Han, A. -D., Li, W. -S. Oxidative stress affects retinal pigment epithelial cell survival through epidermal growth factor receptor/AKT signaling pathway. International Journal of Ophthalmology. 10 (4), 507-514 (2017).
- Tu, G., et al. Allicin attenuates H2O2 - induced cytotoxicity in retinal pigmented epithelial cells by regulating the levels of reactive oxygen species. Molecular Medicine Reports. 13, 2320-2326 (2016).
- Hao, Y., Liu, J., Wang, Z., Yu, L., Wang, J. Piceatannol protects human retinal pigment epithelial cells against hydrogen peroxide induced oxidative stress and apoptosis through modulating. Nutrients. 11, 1-13 (2019).
- Ballinger, S. W., Van Houten, B., Conklin, C. A., Jin, G. F., Godley, B. F. Hydrogen peroxide causes significant mitochondrial DNA damage in human RPE cells. Experimental Eye Research. 68 (6), 765-772 (1999).
- Ma, S., et al. Transgenic overexpression of uncoupling protein 2 attenuates salt-induced vascular dysfunction by inhibition of oxidative stress. American Journal of Hypertension. 27 (3), 345-354 (2014).
- Johnen, S., et al. Sleeping Beauty transposon-mediated transfection of retinal and iris pigment epithelial cells. Investigative Ophthalmology and Visual Science. 53 (8), 4787-4796 (2012).
- Mátés, L., et al. Molecular evolution of a novel hyperactive Sleeping Beauty transposase enables robust stable gene transfer in vertebrates. Nature Genetics. 41 (6), 753-761 (2009).
- Marienfeld Technical information Neubauer-improved. , Available from: https://www.marienfeld-superior.com/information-about-our-counting-chambers.html (2020).
- Electron Microscopy Sciences. Neubauer Haemocytometry. , Available from: https://www.emsdiasum.com/microscopy/technical/datasheet/68052-14.aspx (2020).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2^(-ΔΔCT) method. Methods. 25 (4), San Diego, California. 402-408 (2001).
- Bascuas, T., et al. Non-virally transfected primary human pigment epithelium cells overexpressing the oxidative stress reduction factors PEDF and GM-CSF to treat retinal neurodegeneration neurodegenerationl. Human Gene Therapy. 30 (11), (2019).
- Zhuge, C. C., et al. Fullerenol protects retinal pigment epithelial cells from oxidative stress-induced premature senescence via activating SIRT1. Investigative Ophthalmology & Visual Science. 55 (7), 4628-4638 (2014).
- Kaczara, P., Sarna, T., Burke, M. Dynamics of H2O2 Availability to ARPE-19 cultures in models of oxidative stress. Free Radical Biology and Medicine. 48 (8), 1068-1070 (2010).
- Gorrini, C., Harris, I. S., Mak, T. W. Modulation of oxidative stress as an anticancer strategy. Nature Reviews Drug Discovery. 12, 931-947 (2013).
- Wang, X., et al. PEDF protects human retinal pigment epithelial cells against oxidative stress via upregulation of UCP2 expression. Molecular Medicine Reports. 19 (1), 59-74 (2019).
- Donadelli, M., Dando, I., Fiorini, C., Palmieri, M. UCP2, a mitochondrial protein regulated at multiple levels. Cellular and Molecular Life Sciences. 71, 1171-1190 (2014).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten