Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Induction et analyse du stress oxydatif dans les cellules épithéliales pigmentaires rétiniennes transfectées par transposon de la belle endormie
Dans cet article
Résumé
Nous présentons un protocole pour le développement et l’utilisation d’un modèle de stress oxydatif en traitant les cellules épithéliales pigmentaires rétiniennes avec H2O2, en analysant la morphologie cellulaire, la viabilité, la densité, le glutathion et le niveau UCP-2. C’est un modèle utile pour étudier l’effet antioxydant des protéines sécrétées par les cellules transfectées par transposon pour traiter la dégénérescence neurorétinienne.
Résumé
Le stress oxydatif joue un rôle essentiel dans plusieurs maladies dégénératives, y compris la dégénérescence maculaire liée à l’âge (DMLA), une pathologie qui touche environ 30 millions de patients dans le monde. Il entraîne une diminution des facteurs neuroprotecteurs synthétisés par l’épithélium pigmentaire rétinien (EPR), par exemple le facteur dérivé de l’épithélium pigmentaire (PEDF) et le facteur de stimulation des colonies de granulocytes-macrophages (GM-CSF), suivis de la perte de cellules RPE et éventuellement de la mort des photorécepteurs et des cellules ganglionnaires de la rétine (RGC). Nous émettons l’hypothèse que la reconstitution de l’environnement rétinien neuroprotecteur et neurogène par la transplantation sous-rétinienne de cellules EPR transfectées surexprimant le PEDF et le GM-CSF a le potentiel de prévenir la dégénérescence rétinienne en atténuant les effets du stress oxydatif, en inhibant l’inflammation et en soutenant la survie cellulaire. En utilisant le système de transposon de la Belle au bois dormant (SB100X),les cellules RPE humaines ont été transfectées avec les gènes PEDF et GM-CSF et ont montré une intégration génétique stable, une expression génique à long terme et une sécrétion de protéines à l’aide de la qPCR, du transfert western, de l’ELISA et de l’immunofluorescence. Pour confirmer la fonctionnalité et la puissance du PEDF et du GM-CSF sécrétés par les cellules RPE transfectées, nous avons développé un test in vitro pour quantifier la réduction du stress oxydatif induit parH2O2sur les cellules RPE en culture. La protection cellulaire a été évaluée en analysant la morphologie cellulaire, la densité, le niveau intracellulaire de glutathion, l’expression du gène UCP2 et la viabilité cellulaire. Les cellules EPR transfectées surexprimant le PEDF et/ou le GM-CSF et les cellules non transfectées mais prétraitées avec du PEDF et/ou du GM-CSF (disponibles dans le commerce ou purifiées à partir de cellules transfectées) ont montré une protection cellulaire antioxydante significative par rapport aux témoins non traités. Le présent modèleH2O2est une approche simple et efficace pour évaluer l’effet antioxydant de facteurs qui peuvent être efficaces pour traiter la DMLA ou des maladies neurodégénératives similaires.
Introduction
Le modèle décrit ici offre une approche utile pour évaluer l’efficacité des agents biopharmaceutiques pour réduire le stress oxydatif dans les cellules. Nous avons utilisé le modèle pour étudier les effets protecteurs du PEDF et du GM-CSF sur le stress oxydatif médié parH2O2sur les cellules épithéliales pigmentaires rétiniennes, qui sont exposées à des niveaux élevés d’O2et de lumière visible, et la phagocytose des membranes du segment externe des photorécepteurs, générant des niveaux significatifs d’espèces réactives de l’oxygène (ROS)1, 2. Ils sont considérés comme un contributeur majeur à la pathogenèse de la dégénérescence maculaire avasculaire liée à l’âge (AMMA)3,4,5,6,7,8. En outre, il y a une diminution des facteurs neuroprotecteurs synthétisés par l’EPR, en particulier le facteur dérivé de l’épithélium pigmentaire (PEDF), les facteurs de croissance analogues à l’insuline (IGF) et le facteur de stimulation des colonies de macrophages granulocytaires (GM-CSF) conduisant au dysfonctionnement et à la perte de cellules RPE, suivis de la mort des photorécepteurs et des cellules ganglionnaires rétiniennes (RGC)3,4,5 . La DMLA est une maladie complexe qui résulte de l’interaction entre des facteurs métaboliques, fonctionnels, génétiques et environnementaux4. Le manque de traitements pour la MAA est la principale cause de cécité chez les patients âgés de plus de 60 ans dans les pays industrialisés9,10. La reconstitution de l’environnement rétinien neuroprotecteur et neurogène par la transplantation sous-rétinienne de cellules RPE génétiquement modifiées surexprimant le PEDF et le GM-CSF a le potentiel de prévenir la dégénérescence rétinienne en atténuant les effets du stress oxydatif, en inhibant l’inflammation et en soutenant la survie cellulaire11,12,13,14,15,16 . Même s’il existe plusieurs méthodologies pour délivrer des gènes aux cellules, nous avons choisi le système de transposon hyperactif non viral de la Belle au bois dormant pour délivrer les gènes PEDF et GM-CSF aux cellules RPE en raison de son profil de sécurité, de l’intégration des gènes dans le génome des cellules hôtes et de sa propension à intégrer les gènes délivrés dans des sites non transcriptionnellement actifs comme nous l’avons montré précédemment17, 18,19.
Le stress oxydatif cellulaire peut être induit dans les cellules cultivées in vitro par plusieurs agents oxydatifs, dont le peroxyde d’hydrogène(H2O2),le 4-hydroynonenal (HNE), le tertbutylhydroperoxyde (tBH), les tensions élevées en oxygène et la lumière visible (spectre complet ou irradiation UV)20,21. Les tensions élevées d’oxygène et la lumière nécessitent un équipement et des conditions spéciales, ce qui limite la transférabilité à d’autres systèmes. Des agents tels queH2O2,HNE et tBH induisent des changements moléculaires et cellulaires de stress oxydatif qui se chevauchent. Nous avons choisi H2O2 pour tester l’activité antioxydante du PEDF et du GM-CSF parce qu’il est pratique et biologiquement pertinent puisqu’il est produit par les cellules RPE comme intermédiaire réactif de l’oxygène lors de la phagocytose du segment externe des photorécepteurs22 et qu’il se trouve dans les tissus oculaires in vivo23. Étant donné que l’oxydation du glutathion peut être partiellement responsable de la production deH2O2dans l’œil, nous avons analysé les niveaux de GSH / glutathion dans nos études, qui sont liés au stress oxydatif induit par H2O2et à la capacité de régénération des cellules21,22. L’analyse des niveaux de glutathion est particulièrement pertinente car elle participe aux mécanismes de protection anti-oxydatifs dans l’œil24. L’exposition àH2O2 est fréquemment utilisée comme modèle pour examiner la susceptibilité au stress oxydatif et l’activité antioxydante des cellules RPE1,25,26,27,28,29,30, et, en outre, elle montre des similitudes avec les dommages causés par le stress oxydatif induit par la lumière, une source « physiologique » de stress oxydatif21.
Pour évaluer la fonctionnalité et l’efficacité des facteurs neuroprotecteurs, nous avons développé un modèle in vitro qui permet à l’analyse de quantifier l’effet antioxydant des facteurs de croissance exprimés par les cellules génétiquement modifiées pour surexprimer le PEDF et le GM-CSF. Ici, nous montrons que les cellules RPE transfectées avec les gènes du PEDF et du GM-CSF sont plus résistantes aux effets nocifs deH2O2 que ne le sont les cellules témoins non transfectées, comme en témoignent la morphologie cellulaire, la densité, la viabilité, le niveau intracellulaire de glutathion et l’expression du gène UCP2, qui code pour la protéine de découplage mitochondrial 2 dont il a été démontré qu’elle réduit les espèces réactives de l’oxygène (ROS)31.
Protocole
Les procédures de collecte et d’utilisation des yeux humains ont été approuvées par la Commission éthique cantonale pour la recherche (n° 2016-01726).
1. Isolement cellulaire et conditions de culture
- Lignée cellulaire humaine ARPE-19
- Culture 5 x 105 cellules ARPE-19, une lignée cellulaire RPE humaine, dans le mélange medium/nutritif F-12 (DMEM/Ham’s F-12) de Dulbecco complété par 10 % de sérum fœtal bovin (FBS), 80 U/mL de pénicilline, 80 μg/mL de streptomycine et 2,5 μg/mL d’amphotéricine B (milieu complet) à 37 °C dans une atmosphère humide de 5 % deCO2 et 95 % d’air dans une fiole T75 (pour les autres densités cellulaires, voir le tableau 1).
- Changez le support trois fois par semaine.
- Une fois que les cellules sont cultivées à environ 90% de confluence (évalué qualitativement), aspirez le milieu et lavez les cellules avec 1x PBS stérile.
- Incuber les cellules avec une solution d’EDTA à 5 % de trypsine-2 % pendant 7 à 10 min à 37 °C (pour les volumes, voir tableau 1). Surveillez visuellement le détachement.
- Arrêtez la trypsinisation en ajoutant un milieu complet contenant 10% de FBS (pour les volumes, voir tableau 1).
- Transfecter les cellules (voir l’étape 2. du protocole), sous-cultiver les cellules dans un rapport de 1:10 (une fois par semaine) ou semer dans une plaque de 96 puits comme détaillé ci-dessous (voir les étapes 3.3 et 3.4 du protocole).
| Moyen (mL) | ||||||
| Superficie (cm²) | Densité d’ensemencement pour les cellules ARPE-19 (cellules/puits) | Application | Pour la culture cellulaire | Pour arrêter la trypsine | Volume de trypsine (mL) | |
| Flacon T75 | 75 | 5,00,000 | Croissance cellulaire ARPE-19 | 10 | 7 | 3 |
| 6 Plaque de puits | 9.6 | 1,00,000 | Ensemencement de cellules ARPE-19 transfectées | 3 | 1 | 0.5 |
| Plaque de puits 24 | 2 | 50,000 | Ensemencement de cellules hRPE transfectées | 1 | 0.8 | 0.2 |
| 96 Plaque de puits | 0.32 | 5 000 pour des expériences de stress oxydatif avec des cellules transfectées (Fig. 1) | Expériences sur le stress oxydatif | 0.2 | ||
| 3 000 pour les expériences de stress oxydatif avec des cellules non transfectées plus des protéines (Fig. 1) | ||||||
Tableau 1 : Volumes de culture cellulaire. Volumes de milieux recommandés pour les plaques de culture cellulaire et les flacons pour la culture d’ARPE-19 et de cellules RPE humaines primaires.
- Cellules RPE humaines primaires
- Isolez les cellules RPE humaines primaires telles que décrites par Thumann et al.17, et les cellules de culture dans un milieu complet complété par 20% fbS.
- Changez le support deux fois par semaine. Une fois que les cellules atteignent la confluence (surveillée visuellement), réduisez le FBS à 1% pour éviter la prolifération.
- Transfecter les cellules (voir l’étape 2 du protocole) ou semer dans une plaque de 96 puits comme détaillé ci-dessous (voir les étapes 3.3 et 3.4 du protocole).
NOTE : Les données présentées ici ont été recueillies à partir de la culture de cellules EPR obtenues à partir des yeux de quatre donneurs humains. Le tableau 2 détaille les données démographiques des donateurs de la Lions Gift of Sight Eye Bank (Saint Paul, MN). Les yeux ont été énucléés 12,7 ± 5,7 h (moyenne ± SD) après avoir obtenu le consentement éclairé conformément à la Déclaration d’Helsinki.
| Non | âge | genre | mort à la conservation (heures) | décès à l’isolement | culture | culture | Symbole dans le graphique | |
| (jours) | avant la transfection (jours) | après transfection (jours) | ||||||
| 2 | 80 | M | 20.7 | 8 | 140 | 36 |  | |
| 3 | 86 | F | 12.8 | 8 | 85 | 45 |  | |
| 4 | 86 | F | 8.5 | 5 | 26 | 133 |  | |
| 8 | 83 | F | 8.9 | 6 | 18 | 27 |  | |
| méchant | 83.8 | 12.7 | 6.8 | 67.3 | 60.3 | |||
| SD | 2.9 | 5.7 | 1.5 | 57.0 | 49.1 |
Tableau 2 : Données démographiques des donneurs humains de cellules épithéliales pigmentaires rétiniennes.
2. Électroporation de l’ARPE-19 et des cellules RPE humaines primaires
- Trypsiniser les cellules ARPE-19 ou les cellules RPE humaines primaires comme décrit aux étapes 1.1.3-1.1.5 du protocole.
- Effectuer l’électroporation avec le kit de transfection disponible dans le commerce (voir tableau des matériaux).
- Pour la transfection des cellules ARPE-19, se référer à Johnen et al.32 et pour l’hRPE primaire à Thumann et al.17. Brièvement, remettez en suspension 1 x 105 cellules ARPE-19 ou 5 x 104 cellules primaires hRPE dans 11 μL de tampon R et ajoutez 2 μL de mélange plasmidique contenant 0,03 μg pSB100X transposase33 et 0,47 μg pT2-CMV-PEDF-His ou pT2-CMV-GMCSF-His transposon (ratio transposase:transposon 1:16). Pour les cellules à double transfectation PEDF et GM-CSF, utilisez un rapport de 1:16:16 (0,03 μg pSB100X,0,47 μg pT2-CMV-PEDF-His et 0,47 μg pT2-CMV-GMCSF-His). Utilisez les paramètres d’électroporation suivants : deux impulsions de 1 350 V pendant 20 ms (largeur d’impulsion) pour les cellules ARPE-19 ; deux impulsions de 1 100 V pendant 20 ms pour les cellules primaires.
- Semence 1 x 105 ARPE-19 transfecté ou 5 x 104 cellules primaires hRPE transfectées dans des plaques de 6 et 24 puits, respectivement, dans un milieu complété par 10% FBS sans antibiotiques ni antimycotiques. Ajouter la pénicilline (80 U/mL), la streptomycine (80 μg/mL) et l’amphotéricine B (2,5 μg/mL) avec le premier échange moyen 3 jours après la transfection.
- Déterminer la croissance cellulaire par une surveillance microscopique hebdomadaire des cellules. L’efficacité de la transfection est surveillée par l’analyse de l’expression des gènes par RT-PCR, et la sécrétion de protéines par ELISA et WB (méthodes expliquées dans Supplementary Material).
REMARQUE: L’efficacité de la transfection peut être évaluée pour la première fois une fois que les cellules atteignent la confluence, c’est-à-dire à environ 7 jours et 4 semaines après la transfection pour les cellules ARPE-19 et les cellules hRPE primaires, respectivement. - Ensemencez des cellules dans une plaque de 96 puits comme détaillé ci-dessous (voir l’étape 3.5 du protocole).
3. Induction du stress oxydatif (traitement H2O2) et neuroprotection (traitement PEDF et/ou GM-CSF)
- Préparation du milieu conditionné de cellules ARPE-19 transfectées
- Utiliser des cellules ARPE-19 transfectées avec les gènes PEDF, GM-CSF, ou les deux (voir l’étape 2 du protocole); cellules de culture pendant 28 jours, comme décrit à l’étape 1.1 du protocole.
- 28 jours après la transfection, trypsiniser les cellules (voir les étapes 1.1.3-1.1.5 du protocole), compter les cellules à l’aide d’une chambre de Neubauer34,35et ensemencer 5 x10 5 cellules dans des flacons T75 en milieu complet comme décrit à l’étape 1.1.1 du protocole. Échangez le milieu lorsque la culture cellulaire est confluente à environ 80% (environ après 1 semaine; vérifié qualitativement). Recueillir le milieu après 24 h.
- Conserver le milieu à -20 °C jusqu’à utilisation.
NOTE: Une concentration suffisante de PEDF recombinant et de GM-CSF dans le milieu conditionné a été vérifiée par WB et quantifiée par ELISA comme décrit dans Matériel supplémentaire.
- Purification du PEDF et du GM-CSF à partir d’un milieu conditionné de cellules ARPE-19 transfectées
- Centrifuger le milieu collecté à partir de l’étape 3.1.2 à 10 000 x g pendant 15 min à 4 °C.
- Utilisez le superflow Ni-NTA (voir tableau des matériaux)selon les protocoles du fabricant pour purifier les protéines marquées His comme décrit ci-dessous.
- Pipette 30 μL de mélange Ni-NTA dans un tube de 1,5 mL et centrifuger à 2 600 x g pendant 30 s et jeter le flux. Lavez la pastille deux fois avec 200 μL de tampon d’incubation 1x.
- Centrifuger à 2 600 x g pendant 30 s et jeter le débit. Ajouter 40 μL de tampon d’incubation 4x et remettre en suspension.
- Ajouter 900 μL de milieu conditionné centrifugé et incuber à 70 tr/min (agitateur orbital) pendant 1 h à RT. Centrifuger à 2 600 x g pendant 1 min et jeter l’écoulement.
- Lavez la pastille deux fois avec 175 μL de tampon d’incubation 1x. Centrifuger à 2 600 x g pendant 30 s et jeter le débit.
- Pour éluer les protéines PEDF et GM-CSF marquées par His, ajoutez 20 μL de tampon elution et incubez à 70 tr/min (agitateur orbital) pendant 20 min à RT. Centrifuger à 2 600 x g pendant 30 s. Conservez le surnageant contenant du PEDF recombinant ou du GM-CSF.
- Quantifier la protéine totale à l’aide du kit d’analyse des protéines BCA disponible dans le commerce (voir la table des matériaux)conformément aux instructions du fabricant.
- Conserver la solution protéique à -20 °C jusqu’à utilisation.
REMARQUE: Le tampon d’incubation (4x) contient 200 mM de NaH2PO4,1,2 M de NaCl et 40 mM d’imidazol; Le tampon d’élution contient 50 mM de NaH2PO4,300 mM de NaCl et 250 mM d’imidazol.
- Traitement des cellules ARPE-19/hRPE primaires non transfectées avec un milieu conditionné plusH2O2 (Figure 1A)
- Ensemencez 3 000 cellules ARPE-19 non transfectées (à partir de l’étape 1.1.6 du protocole) ou de hRPE primaire (à partir de l’étape 1.2.3 du protocole) par puits dans une plaque de 96 puits et une culture dans 200 μL de milieu conditionné à partir de cellules ARPE-19 transfectées.
- Cultiver les cellules pendant 10 jours à 37 °C dans une atmosphère humidifiée de 5 % de CO2 et de 95 % d’air. Changez le milieu conditionné tous les jours. Exposer les cellules à 350 μMH2O2 pendant 24 h.
- Évaluer les dommages causés par le stress oxydatif et déterminer l’effet antioxydant du PEDF et du GM-CSF par quantification des niveaux de glutathion (voir l’étape 4.1 du protocole), la microscopie (voir l’étape 4.2 du protocole) et le test de cytotoxicité (voir l’étape 4.2 du protocole).
REMARQUE: La durée de l’expérience est de 12 jours. Des plaques de micropuits à fond plat transparent sont utilisées pour évaluer la luminescence ainsi que la morphologie cellulaire. Pour effectuer simultanément le test de cytotoxicité et de glutathion, deux plaques doivent être ensemencées avec des cellules le même jour.
- Traitement des cellules ARPE-19/hRPE primaires non transfectées avec des facteurs de croissance PEDF et GM-CSF plus H2O2 (Figure 1B)
- Semer 3 000 cellules ARPE-19 non transfectées (à partir de l’étape 1.1.6 du protocole) ou hRPE primaires (à partir de l’étape 1.2.3 du protocole) par puits (plaques de 96 puits avec un fond plat clair) dans 200 μL de milieu de culture complet contenant 500 ng/mL de PEDF recombinant et/ou 50 ng/mL de GMcombinant-CSF, purifiés à partir du milieu de cellules ARPE-19 transfectées ou disponibles dans le commerce. Cellules de culture pendant 48 h à 37 °C dans une atmosphère humidifiée de 5 % de CO2 et de 95 % d’air. Renouvelez quotidiennement le milieu, y compris les facteurs de croissance PEDF et GM-CSF.
REMARQUE: Ajoutez les facteurs de croissance frais au milieu. - Après 48 h de traitement des cellules avec les facteurs de croissance, prélever le milieu et ajouter le milieu complet contenant 350 μMH2O2 plus 500 ng/mL PEDF et/ou 50 ng/mL GM-CSF.
- Évaluer les dommages causés par le stress oxydatif et déterminer l’effet antioxydant du PEDF et du GM-CSF par quantification des niveaux de glutathion (voir l’étape 4.1 du protocole), la microscopie (voir l’étape 4.2 du protocole) et le test de cytotoxicité (voir l’étape 4.2 du protocole).
REMARQUE: La durée de l’expérience est de 3 jours.
- Semer 3 000 cellules ARPE-19 non transfectées (à partir de l’étape 1.1.6 du protocole) ou hRPE primaires (à partir de l’étape 1.2.3 du protocole) par puits (plaques de 96 puits avec un fond plat clair) dans 200 μL de milieu de culture complet contenant 500 ng/mL de PEDF recombinant et/ou 50 ng/mL de GMcombinant-CSF, purifiés à partir du milieu de cellules ARPE-19 transfectées ou disponibles dans le commerce. Cellules de culture pendant 48 h à 37 °C dans une atmosphère humidifiée de 5 % de CO2 et de 95 % d’air. Renouvelez quotidiennement le milieu, y compris les facteurs de croissance PEDF et GM-CSF.
- Traitement des cellules ARPE-19/hRPE primaire transfectées avec H2O2 (Figure 1C)
- Vérifier l’expression génique suffisante et la sécrétion protéique des cellules transfectées par WB et ELISA comme décrit dans le matériel supplémentaire.
- Retirer le milieu des puits contenant les cellules transfectées (voir l’étape 2 du protocole).
- Trypsiniser les cellules comme décrit aux étapes 1.1.3-1.1.5 du protocole. Comptez les cellules à l’aide d’une chambre de Neubauer34,35.
- Ensemencez 5 000 cellules/puits transfectés dans une plaque de 96 puits dans 200 μL de milieu complet. Cellules de culture pendant 24 h à 37 °C dans une atmosphère humidifiée de 5 % de CO2 et de 95 % d’air. Après 24 h, exposer les cellules à 350 μMH2O2 pendant 24 h.
- Évaluer les dommages causés par le stress oxydatif et déterminer l’effet antioxydant du PEDF et du GM-CSF par quantification des niveaux de glutathion (voir l’étape 4.1 du protocole), la microscopie (voir l’étape 4.2 du protocole), le test de cytotoxicité (voir l’étape 4.2 du protocole) et la détermination de l’expression du gène UCP2 (voir l’étape 4.3 du protocole).
REMARQUE: La durée de l’expérience est de 2 jours.
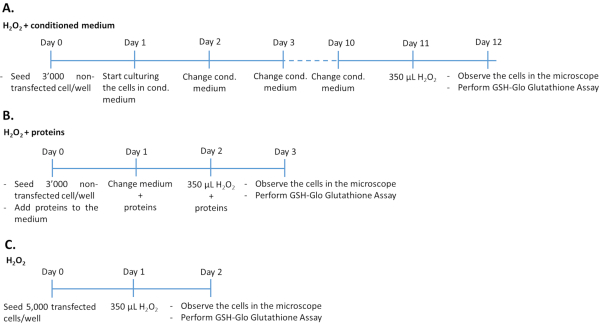
Figure 1: Chronologie du test H2O2 dans les trois différentes approches expérimentales. 3 000 cellules non transfectées traitées avec le milieu conditionné/protéines recombinantes ou 5 000 cellules transfectées ont été ensemencées dans des plaques de 96 puits pour un traitement avecH2O2. Pour déterminer l’effet du milieu conditionné, les cellules ont été cultivées dans un milieu de culture à 100% pendant 10 jours consécutifs, changeant de milieu tous les jours. Pour déterminer l’effet des facteurs de croissance recombinants, les cellules ont été cultivées en ajoutant la quantité appropriée de facteurs de croissance chaque jour pendant 3 jours consécutifs. Notez que les cellules non transfectées ont été ensemencées à 3 000 cellules par puits pour éviter la prolifération pendant la durée de culture plus longue par rapport aux cellules transfectées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Analyse du niveau de stress oxydatif et de la capacité antioxydante
- Dosage du glutathion
- Mesurez les niveaux de glutathion (GSH) à l’aide de la trousse disponible dans le commerce (voir la table des matériaux)en suivant les instructions du fabricant. En bref, préparer et le volume approprié de 1x mélange de réactif (réactif de 100 μL / puits): substrat de luciférine-NT et glutathion S-transférase dilué 1:100 dans le tampon de réaction.
REMARQUE: Une plaque de 96 puits nécessite 10 mL de mélange de réactif 1x, qui est préparé en ajoutant 100 μL de substrat de luciférine-NT et 100 μL de glutathion S-transférase à 10 mL de tampon de réaction. Préparez le mélange de réactif 1x immédiatement avant utilisation. Ne conservez pas le mélange de réactifs préparé pour une utilisation ultérieure. - Préparez le réactif de détection de la luciférine en transférant une bouteille de tampon de reconstitution au réactif de détection de la luciférine lyophilisé.
- Préparer une courbe étalon à l’aide d’une solution étalon de glutathion (GSH) (5 mM). Diluer 5 mM de solution de GSH 1:100 avec dH2O (ajouter 10 μL de solution de GSH de 5 mM à 990 μL de dH2O). Effectuer 7 dilutions série 1:1 dans 500 μL de dH2O. Transférer 10 μL de chaque étalon dilué vers un puits approprié en double exemplaire.
REMARQUE: La concentration finale de glutathion variera de 0,039 μM à 5 μM. - Préparer le blanc (1x mélange de réactifs) et transférer 10 μL (doublons) dans les puits appropriés.
- Retirer les cellules traitéesenH2 O2de l’incubateur.
NOTE: Documenter la morphologie des cellules traitées par H2O2par microscopie à fond clair (40x).
Lorsque les cellules sont oxydées, elles semblent plus arrondies et moins répandues. - Aspirez soigneusement le milieu de culture. Ajouter 100 μL de mélange préparé de 1x réactif à chaque puits. Mélanger les cellules avec le réactif pendant 15 s à 500 tr/min sur un agitateur orbital.
- Incuber la plaque à RT pendant 30 min. Ajouter 100 μL de réactif de détection de la luciférine reconstitué à chaque puits.
- Mélanger la solution pendant 15 s à 500 tr/min sur un agitateur orbital. Incuber la plaque pendant 15 min à RT.
- Déterminez la luminescence à l’aide d’un lecteur de plaques à l’aide d’un programme préinstallé ADP-Glo.
REMARQUE: Placez la plaque à l’intérieur du lecteur de plaque sans le couvercle.- Cliquez sur Modifier la mise en page et choisissez les paramètres suivants dans Paramètres de base: Plaque Costar 96 puits; optique supérieure; délai de positionnement: 0,1; heure de début de la mesure: 0,0; temps d’intervalle de mesure: 1,0; temps pour normaliser les résultats: 0,0; le gain est ajusté automatiquement par l’appareil. Définissez des blancs, des normes et des échantillons. Cliquez sur Démarrer la mesure.
- Exportez les données sous forme de fichier Excel. Calculer la concentration de GSH dans chaque échantillon par interpolation de la courbe standard.
- Mesurez les niveaux de glutathion (GSH) à l’aide de la trousse disponible dans le commerce (voir la table des matériaux)en suivant les instructions du fabricant. En bref, préparer et le volume approprié de 1x mélange de réactif (réactif de 100 μL / puits): substrat de luciférine-NT et glutathion S-transférase dilué 1:100 dans le tampon de réaction.
- Test de cytotoxicité et analyse microscopique
- Aspirer le milieu des cellules et ajouter 100 μL de milieu complet contenant 1% de FBS à chaque puits. Renvoyer les cellules à l’incubateur.
REMARQUE: 1% FBS est utilisé parce que des pourcentages plus élevés de FBS peuvent interférer avec la mesure de la luminescence, donc 1% FBS est utilisé dans ce cas. - Mesurez la viabilité des cellules à l’aide de la trousse d’analyse de cytotoxicité disponible dans le commerce (voir tableau des matériaux)en suivant les instructions du fabricant. En bref, préparez le mélange de réactifs en ajoutant le tampon de dosage au substrat lyophilisé. Préparez le réactif de lyse en ajoutant 33 μL de digitonine à 5 mL de tampon d’essai (pour une plaque de 96 puits). Bien mélanger en pipetant de haut en bas pour assurer l’homogénéité.
REMARQUE: Pour des résultats optimaux, utilisez un mélange de réactif fraîchement préparé. Utiliser dans les 12 h s’il est conservé à TA. Le mélange de réactifs peut être conservé à 4 °C jusqu’à 7 jours et peut être conservé dans des aliquotes à usage unique jusqu’à 4 mois à -70 °C. La congélation et la décongélation doivent être évitées. Le réactif de lyse peut être conservé à 4 °C jusqu’à 7 jours. - Préparez une courbe standard avec des cellules ARPE-19 non traitées.
- Trypsiniser les cellules comme décrit aux étapes 1.1.3-1.1.5 du protocole et compter les cellules à l’aide d’une chambre de Neubauer34,35. Centrifuger les cellules à 120 g pendant 10 min à RT. Aspirer le surnageant et remettre la pastille de cellule dans le milieu F12 de DMEM/Ham contenant 1 % de FBS jusqu’à une concentration finale de 1 x 105 cellules/mL.
- Préparer 7 dilutions série 1:1 dans un milieu de 200 μL contenant 1 % de FBS. Transférer 100 μL de chaque étalon vers les puits appropriés (doublons). Ajouter 50 μL de mélange de réactifs à tous les puits.
- Mélanger les cellules avec le réactif pendant 15 s à 500 tr/min sur un agitateur orbital. Incuber la plaque pendant 15 min à RT. Mesurer la luminescence à l’aide du lecteur de plaque comme décrit à l’étape 4.1.9 du protocole. Ajouter 50 μL du réactif de lyse et incuber pendant 15 min. Mesurer la luminescence à l’aide du lecteur de plaques comme décrit à l’étape 4.1.9 du protocole.
- Calculer le pourcentage de cellules viables: (100 - % de cellules mortes) et le pourcentage de cellules mortes = [1ère mesure de luminescence ((cellules mortes dans l’échantillon))/ 2ème mesure de luminescence (toutes les cellules mortes après traitement à la digitonine)] x 100.
- Aspirer le milieu des cellules et ajouter 100 μL de milieu complet contenant 1% de FBS à chaque puits. Renvoyer les cellules à l’incubateur.
- Analyse d’expression UCP2 par RT-qPCR
- Trypsiniser les cellules transfectées comme décrit ci-dessus (étapes 1.1.3-1.1.5 du protocole).
- Comptez les cellules à l’aide d’une chambre de Neubauer34,35.
- Ensemencez 5 000 cellules/puits ARPE-19 transfectés dans des plaques de 96 puits.
- Après 24 h de culture, traiter les cellules avec 350 μMH2O2 pendant 24 h.
- Isoler l’ARN total à l’aide d’un kit commercial pour isoler l’ARN d’un faible nombre de cellules (voir tableau des matériaux)en suivant les instructions du fabricant.
- Effectuer une PCR quantitative en temps réel (RT-qPCR) comme décrit dans Matériel supplémentaire. En bref, générez de l’ADNc par rétrotranscription à l’aide d’un mélange disponible dans le commerce contenant une transcriptase inverse M-MLV optimisée (voir Table des matériaux).
- Pour la qPCR, utilisez un cocktail de réaction prêt à l’emploi contenant tous les composants (y compris SYBR Green) à l’exception des amorces (voir le tableau S1 du matériel supplémentaire)et du modèle d’ADN. Utilisez les conditions de thermocyclage suivantes : dénaturation initiale à 95 °C pendant 10 min, 40 cycles avec dénaturation à 95 °C pendant 15 s, recuit à 60 °C pendant 30 s et allongement à 72 °C pendant 32 s.
- Utilisez la méthode 2^(-ΔΔCT) pour l’analyse36.
- Préparation du lysat cellulaire pour l’analyse SDS-PAGE et WB de pAkt (Ser473)
- Semence 3 x 105 cellules/puits ARPE-19 transfectés par GM-CSF dans des plaques de 6 puits (≥21 jours après la transfection) pour déterminer si gm-CSF protège les cellules RPE contre les dommages causés parH2O2 par l’activation de la voie de survie Akt15.
- Après 24 h de culture, les cellules sont exposées à 350 μM H2O2 pendant 24 h.
- Mélanger 1 mL de tampon RIPA avec 10 μL de cocktail d’inhibiteurs de la protéase phosphatase, 10 μL de 0,5 M d’EDTA et 25 μL d’urée de 8 M (volumes utilisés pour un puits).
- Aspirer soigneusement le milieu et laver les cellules avec 1x PBS.
- Ajoutez tout le volume de mélange tampon RIPA aux cellules.
- Pipette de haut en bas.
- Recueillir le lysat dans des tubes de 1,5 mL.
- Centrifuger à 20 000 x g pendant 30 min à 4 °C.
- Transférer le surnageant dans un nouveau tube de 1,5 mL.
- Déterminer les niveaux de pAkt dans 15 μL de lysat cellulaire non dilué par WB comme décrit dans Matériel supplémentaire.
Résultats
Induction du stress oxydatif dans les cellules épithéliales pigmentaires rétiniennes humaines
L’ARPE-19 et les cellules primaires hRPE ont été traitées avec des concentrations variables deH2O2 pendant 24 h et le niveau intracellulaire du glutathion antioxydant a été quantifié (Figure 2A,B). H2O2 à 50 μM et 100 μM n’a pas affecté la production de glutathion, alors...
Discussion
Le protocole présenté ici offre une approche pour analyser la fonction antioxydante et protectrice du PEDF et du GM-CSF produits par les cellules transfectées, qui peuvent être appliquées aux cellules transfectées avec n’importe quel gène bénéfique putatif. Dans les stratégies thérapeutiques géniques qui ont pour objectif de délivrer des protéines aux tissus en transplantant des cellules génétiquement modifiées, il est essentiel d’obtenir des informations sur le niveau d’expression des protéines, ...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs tiennent à remercier Gregg Sealy et Alain Conti pour l’excellente assistance technique et le professeur Zsuzsanna Izsvák du Centre Max-Delbrück à Berlin pour avoir aimablement fourni les plasmides pSB100X et pT2-CAGGS-Venus. Ces travaux ont été soutenus par le Fonds national suisse de la recherche scientifique et la Commission européenne dans le cadre du septième programme-cadre. Z.I a été financé par le Conseil européen de la recherche, ERC Advanced [ERC-2011-ADG 294742].
matériels
| Name | Company | Catalog Number | Comments |
| 24-well plates | Corning | 353047 | |
| 6-well plates | Greiner | 7657160 | |
| 96-well culture plate white with clear flat bottom | Costar | 3610 | Allows to check the cells before measuring the luminescence (GSH-Glo Assay) |
| 96-well plates | Corning | 353072 | |
| Acrylamid 40% | Biorad | 161-0144 | |
| Amphotericin B | AMIMED | 4-05F00-H | |
| Antibody anti-GMCSF | ThermoFisher Scientific | PA5-24184 | |
| Antibody anti-mouse IgG/IgA/IgM | Agilent | P0260 | |
| Antibody anti-PEDF | Santa Cruz Biotechnology Inc | sc-390172 | |
| Antibody anti-penta-His | Qiagen | 34660 | |
| Antibody anti-phospho-Akt | Cell Signaling Technology | 9271 | |
| Antibody anti-rabbit IgG H&L-HRP | Abcam | ab6721 | |
| Antibody donkey anti-rabbit Alexa Fluor 594 | ThermoFisher Scientific | A11034 | |
| Antibody goat anti-mouse Alexa 488 | ThermoFisher Scientific | A-11029 | |
| ARPE-19 cell line | ATCC | CRL-2302 | |
| BSA | Sigma-Aldrich | A9418-500G | |
| chamber culture glass slides | Corning | 354118 | |
| CytoTox-Glo Cytotoxicity Assay | Promega | G9291 | |
| DAPI | Sigma-Aldrich | D9542-5MG | |
| DMEM/Ham`s F12 | Sigma-Aldrich | D8062 | |
| Duo Set ELISA kit | R&D Systems | DY215-05 | |
| EDTA | ThermoFisher Scientific | 78440 | |
| ELISAquant kit | BioProducts MD | PED613-10-Human | |
| Eyes (human) | Lions Gift of Sight Eye Bank (Saint Paul, MN) | ||
| FBS | Brunschwig | P40-37500 | |
| Fluoromount Aqueous Mounting Medium | Sigma-Aldrich | F4680-25ML | |
| FLUOstar Omega plate reader | BMG Labtech | ||
| GraphPad Prism software (version 8.0) | GraphPad Software, Inc. | ||
| GSH-Glo Glutathione Assay | Promega | V6912 | |
| hydrogen peroxide (H2O2) | Merck | 107209 | |
| ImageJ software (image processing program) | W.S. Rasband, NIH, Bethesda, MD, USA; https://imagej.nih.gov/ij/; 1997–2014 | ||
| Imidazol | Axonlab | A1378.0010 | |
| Leica DMI4000B microscope | Leica Microsystems | ||
| LightCycler 480 Instrument II | Roche Molecular Systems | ||
| LightCycler 480 SW1.5.1 software | Roche Molecular Systems | ||
| NaCl | Sigma-Aldrich | 71376-1000 | |
| NaH2PO4 | Axonlab | 3468.1000 | |
| Neon Transfection System | ThermoFisher Scientific | MPK5000 | |
| Neon Transfection System 10 µL Kit | ThermoFisher Scientific | MPK1096 | |
| Neubauer chamber | Marienfeld-superior | 640010 | |
| Ni-NTA superflow | Qiagen | 30410 | |
| Nitrocellulose | VWR | 732-3197 | |
| Omega Lum G Gel Imaging System | Aplegen Life Science | ||
| PBS 1X | Sigma-Aldrich | D8537 | |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781-100 | |
| PerfeCTa SYBR Green FastMix | Quantabio | 95072-012 | |
| PFA | Sigma-Aldrich | 158127-100G | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23227 | |
| Primers | Invitrogen | See Table 1 in Supplementary Materials | |
| pSB100X (250 ng/µL) | Mátés et al., 2009. Provide by Prof. Zsuzsanna Izsvak | ||
| pT2-CMV-GMCSF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| pT2-CMV-PEDF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| QIAamp DNA Mini Kit | QIAGEN | 51304 | |
| recombinant hGM-CSF | Peprotech | 100-11 | |
| recombinant hPEDF | BioProductsMD | 004-096 | |
| ReliaPrep RNA Cell Miniprep System | Promega | Z6011 | |
| RIPA buffer | ThermoFisher Scientific | 89901 | |
| RNase-free DNase Set | QIAGEN | 79254 | |
| RNeasy Mini Kit | QIAGEN | 74204 | |
| SDS | Applichem | A2572 | |
| Semi-dry transfer system for WB | Bio-Rad | ||
| SuperMix qScript | Quantabio | 95048-025 | |
| Tris-buffered saline (TBS) | ThermoFisher Scientific | 15504020 | |
| Triton X-100 | AppliChem | A4975 | |
| Trypsin/EDTA | Sigma-Aldrich | T4174 | |
| Tween | AppliChem | A1390 | |
| Urea | ThermoFisher Scientific | 29700 | |
| WesternBright ECL HRP substrate | Advansta | K-12045-D50 | |
| Whatman nitrocellulose membrane | Chemie Brunschwig | MNSC04530301 |
Références
- Zareba, M., Raciti, M. W., Henry, M. M., Sarna, T., Burke, J. M. Oxidative stress in ARPE-19 cultures: Do melanosomes confer cytoprotection. Free Radical Biology and Medicine. 40 (1), 87-100 (2006).
- Gong, X., Draper, C. S., Allison, G. S., Marisiddaiah, R., Rubin, L. P. Effects of the macular carotenoid lutein in human retinal pigment epithelial cells. Antioxidants. 6 (4), (2017).
- Sacconi, R., Corbelli, E., Querques, L., Bandello, F., Querques, G. A Review of current and future management of geographic atrophy. Ophthalmology and Therapy. 6, 69-77 (2017).
- Al-Zamil, W. M., Yassin, S. A. Recent developments in age-related macular degeneration: a review. Clinical Interventions in Aging. 12, 1313-1330 (2017).
- Kumar-Singh, R. The role of complement membrane attack complex in dry and wet AMD - From hypothesis to clinical trials. Experimental Eye Research. 184, 266-277 (2019).
- Ung, L., Pattamatta, U., Carnt, N., Wilkinson-Berka, J. L., Liew, G., White, A. J. R. Oxidative stress and reactive oxygen species. Clinical Science. 131, 2865-2883 (2017).
- Beatty, S., Koh, H. H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Alhasani, R. H., et al. Gypenosides protect retinal pigment epithelium cells from oxidative stress. Food and Chemical Toxicology. 112, 76-85 (2018).
- . National Institute of Health Available from: https://nei.nih.gov/learn-about-eye-health/resources-for-health-educators/eye-health-data-and-statistics/age-related-macular-degeneration-amd-data-and-statistics (2020)
- Mitchell, P., Liew, G., Gopinath, B., Wong, T. Y. Age-related macular degeneration. The Lancet. 392, 1147-1159 (2018).
- He, Y., Leung, K. W., Ren, Y., Jinzhi, P., Jian, G., Tombran-Tink, J. PEDF improves mitochondrial function in RPE cells during oxidative stress. Investigative Ophthalmology & Visual Science. 55, 6742-6755 (2014).
- Cao, S., Walker, G. B., Wang, X., Cui, J. Z., Matsubara, J. A. Altered cytokine profiles of human retinal pigment epithelium: Oxidant injury and replicative senescence. Molecular Vision. 19, 718-728 (2013).
- Farnoodian, M., Sorenson, C. M., Sheibani, N. PEDF expression affects the oxidative and inflammatory state of choroidal endothelial cells. Amercian Journal of Physiology and Cell Physiology. 314 (4), 456-472 (2018).
- Polato, F., Becerra, S. P. Retinal Degenerative Diseases: Mechanisms and Experimental Therapies. Retinal Degenerative Diseases. , 699-706 (2016).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF regulates the ERK1/2 pathways and protects injured retinal ganglion cells from induced death. Experimental Eye Research. 89, 665-677 (2009).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF protects rat photoreceptors from death by activating the SRC-dependent signalling and elevating anti-apoptotic factors and neurotrophins. Graefes Archives for Clinical and Experimental Ophthalmology. 250, 699-712 (2012).
- Thumann, G., et al. Engineering of PEDF-expressing primary pigment epithelial cells by the SB transposon system delivered by pFAR4 plasmids. Molecular Therapy - Nucleic Acids. 6, 302-314 (2017).
- Garcia-Garcia, L., et al. Long-term PEDF release in rat iris and retinal epithelial cells after Sleeping Beauty transposon-mediated gene delivery. Molecular Therapy - Nucleic Acids. 9, 1-11 (2017).
- Johnen, S., et al. Antiangiogenic and neurogenic activities of Sleeping Beauty-mediated PEDF-transfected RPE cells in vitro and in vivo. BioMed Research International. 2015, (2015).
- Weigel, A. L., Handa, J. T., Hjelmeland, M. L. Microarray analysis of H2O2-, HNE-, or tBH-treated ARPE-19 cells. Free Radical Biology & Medicine. 33 (10), 1419-1432 (2002).
- Allen, R. G., Tresini, M. Oxidative stress and gene regulation. Free Radical Biology and Medicine. 28 (3), 463-499 (2000).
- Tate, D. J., Miceli, M. V., Newsome, D. A. Phagocytosis and H2O2 induce catalase and metallothionein gene expression in human retinal pigment epithelial cells. Investigative Ophthalmology and Visual Science. 36 (7), 1271-1279 (1995).
- Halliwell, B., Clement, M. V., Long, L. H. Hydrogen peroxide in the human body. FEBS Letters. 486 (1), 10-13 (2000).
- Giblin, F. J., McCready, J. P., Kodama, T., Reddy, V. N. A direct correlation between the levels of ascorbic acid and H2O2 in aqueous humor. Experimental Eye Research. 38, 87-93 (1984).
- Geiger, R. C., Waters, C. M., Kamp, D. W., Glucksberg, M. R. KGF prevents oxygen-mediated damage in ARPE-19 cells. Investigative Ophthalmology and Visual Science. 46, 3435-3442 (2005).
- Campochiaro, P. A., et al. Lentiviral vector gene transfer of endostatin/angiostatin for macular degeneration (GEM) study. Human Gene Therapy. 28, 99-111 (2017).
- Chen, X. -. D., Su, M. -. Y., Chen, T. -. T., Hong, H. -. Y., Han, A. -. D., Li, W. -. S. Oxidative stress affects retinal pigment epithelial cell survival through epidermal growth factor receptor/AKT signaling pathway. International Journal of Ophthalmology. 10 (4), 507-514 (2017).
- Tu, G., et al. Allicin attenuates H2O2 - induced cytotoxicity in retinal pigmented epithelial cells by regulating the levels of reactive oxygen species. Molecular Medicine Reports. 13, 2320-2326 (2016).
- Hao, Y., Liu, J., Wang, Z., Yu, L., Wang, J. Piceatannol protects human retinal pigment epithelial cells against hydrogen peroxide induced oxidative stress and apoptosis through modulating. Nutrients. 11, 1-13 (2019).
- Ballinger, S. W., Van Houten, B., Conklin, C. A., Jin, G. F., Godley, B. F. Hydrogen peroxide causes significant mitochondrial DNA damage in human RPE cells. Experimental Eye Research. 68 (6), 765-772 (1999).
- Ma, S., et al. Transgenic overexpression of uncoupling protein 2 attenuates salt-induced vascular dysfunction by inhibition of oxidative stress. American Journal of Hypertension. 27 (3), 345-354 (2014).
- Johnen, S., et al. Sleeping Beauty transposon-mediated transfection of retinal and iris pigment epithelial cells. Investigative Ophthalmology and Visual Science. 53 (8), 4787-4796 (2012).
- Mátés, L., et al. Molecular evolution of a novel hyperactive Sleeping Beauty transposase enables robust stable gene transfer in vertebrates. Nature Genetics. 41 (6), 753-761 (2009).
- . Marienfeld Technical information Neubauer-improved Available from: https://www.marienfeld-superior.com/information-about-our-counting-chambers.html (2020)
- . Electron Microscopy Sciences. Neubauer Haemocytometry Available from: https://www.emsdiasum.com/microscopy/technical/datasheet/68052-14.aspx (2020)
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2^(-ΔΔCT) method. Methods. 25 (4), 402-408 (2001).
- Bascuas, T., et al. Non-virally transfected primary human pigment epithelium cells overexpressing the oxidative stress reduction factors PEDF and GM-CSF to treat retinal neurodegeneration neurodegenerationl. Human Gene Therapy. 30 (11), (2019).
- Zhuge, C. C., et al. Fullerenol protects retinal pigment epithelial cells from oxidative stress-induced premature senescence via activating SIRT1. Investigative Ophthalmology & Visual Science. 55 (7), 4628-4638 (2014).
- Kaczara, P., Sarna, T., Burke, M. Dynamics of H2O2 Availability to ARPE-19 cultures in models of oxidative stress. Free Radical Biology and Medicine. 48 (8), 1068-1070 (2010).
- Gorrini, C., Harris, I. S., Mak, T. W. Modulation of oxidative stress as an anticancer strategy. Nature Reviews Drug Discovery. 12, 931-947 (2013).
- Wang, X., et al. PEDF protects human retinal pigment epithelial cells against oxidative stress via upregulation of UCP2 expression. Molecular Medicine Reports. 19 (1), 59-74 (2019).
- Donadelli, M., Dando, I., Fiorini, C., Palmieri, M. UCP2, a mitochondrial protein regulated at multiple levels. Cellular and Molecular Life Sciences. 71, 1171-1190 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon