このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
睡眠美容トランスポゾントランスポゾントランスフェクトヒト網膜色素上皮細胞における酸化ストレスの誘導と分析
要約
網膜色素上皮細胞をH2O2で処理し、細胞の形態、生存率、密度、グルタチオン、UCP-2レベルを解析することにより、酸化ストレスモデルの開発と使用に関するプロトコルを提示します。これは、トランスポゾントランスフェクション細胞によって分泌されるタンパク質の抗酸化効果を調べて神経レチンの変性を治療するのに有用なモデルです。
要約
酸化ストレスは、加齢黄斑変性症(AMD)を含むいくつかの変性疾患において重要な役割を果たします。網膜色素上皮(RPE)合成神経保護因子の減少、例えば、色素上皮由来因子(PEDF)および顆粒球マクロファージコロニー刺激因子(GM-CSF)、RPE細胞の喪失、そして最終的には光受容体および網膜神経節細胞(RGC)死につながる。我々は、PEDFおよびGM-CSFを過剰発現するトランスフェクションRPE細胞の遺伝子導入による神経保護および神経原性のレチナル環境の再構成は、酸化ストレスの影響を緩和し、炎症を抑制し、細胞生存を支持することによって、レチナル変性を防ぐ可能性があると仮定する。眠れる森の美しさトランスポゾンシステム(SB100X)を用いて、ヒトRPE細胞はPEDFおよびGM-CSF遺伝子にトランスフェクトされ、qPCR、ウェスタンブロット、ELISA、および免疫蛍光を用いた安定した遺伝子集積、長期遺伝子発現、タンパク質分泌を示した。トランスフェクトされたRPE細胞によって分泌されるPEDFおよびGM-CSFの機能性と効力を確認するために、培養中のRPE細胞に対するH2O2誘導酸化ストレスの低減を定量化するインビトロアッセイを開発しました。細胞保護は、細胞の形態、密度、グルタチオンの細胞内レベル、UCP2遺伝子発現、および細胞生存率を分析することによって評価した。いずれも、トランスフェクトされたRPE細胞は、非トランスフェクトされたPEDFおよびGM-CSFおよび未導入の細胞および未導入の細胞(市販または遺伝子組み換え細胞から精製された)で前処理された細胞は、非治療対照と比較して有意な抗酸化細胞保護を示した。本H2O2-modelは、AMDまたは類似の神経変性疾患を治療するのに有効であり得る因子の抗酸化効果を評価するための簡便かつ有効なアプローチである。
概要
ここで説明するモデルは、細胞内の酸化ストレスを低減するためのバイオ医薬品の効率を評価するのに有用なアプローチを提供する。このモデルを用いて、高レベルのO2に曝露される網膜色素上皮細胞に対するH2O2-媒介酸化ストレス、可視光、光受容体外皮膜の貪食作用、活性酸素種(ROS)1に対する保護効果を調べた。 2.彼らは血管の加齢黄斑変性症(aAMD)3、4、5、6、7、8の病因に大きく貢献していると考えられている。また、RPE合成神経保護因子、特に色素上皮由来因子(PEDF)、インスリン様成長因子(IIF)、顆粒球マクロファージ-コロニー刺激因子(GM-CSF)がRPE細胞の機能不全および喪失をもたらし、続いて光受容体および網膜神経節細胞(RGC)死亡が減少している。.AMDは、代謝、機能、遺伝的、および環境因子4間の相互作用から生じる複雑な疾患である。aAMDの治療の欠如は、先進国9,10の60歳以上の患者における失明の主な原因である。PEDFおよびGM-CSFを過剰発現する遺伝子組み換えRPE細胞の遺伝子組み換え後移植による神経保護および神経原性のレチナル環境の再構成は、酸化ストレスの影響を軽減し、炎症を抑制し、細胞生存を支持することによって、レチナル変性を防ぐ可能性を秘めている11、12、13、14、15、16.細胞に遺伝子を送達するいくつかの方法論があるにもかかわらず、我々は、その安全性プロファイル、宿主細胞のゲノムへの遺伝子の統合、および以前に示したように非転写活性部位に提供された遺伝子を統合する傾向のために、PEDFおよびGM-CSF遺伝子をRPE細胞に送達する非ウイルス性多動性睡眠美容トランスポゾンシステムを選択した。 18,19.
細胞酸化ストレスは、過酸化水素(H2O2)、4-ヒドロノネナー(HNE)、テルブチルヒドロペルオキシド(tBH)、高酸素張力、可視光(全スペクトルまたはUV照射)20、21を含む、いくつかの酸化剤によってインビトロで培養された細胞において誘導され得る。高い酸素の緊張およびライトは他のシステムへの移動性を制限する特別な装置および条件を要求する。H2O2、HNE、およびtBHなどの薬剤は、分子および細胞変化と重なり合う酸化ストレスを誘発する。我々は、RPE細胞が感光体外セグメント食細胞化22中に活性酸素中間体としてRPE細胞によって産生され、生体23の眼組織に見られるため、PEDFおよびGM-CSFの抗酸化活性を試験するためにH2O2を選択した。グルタチオンの酸化は、目の中のH2O2の産生に部分的に関与する可能性があるため、我々は、H2O2誘導酸化ストレスおよび細胞21,22の再生能力に関連するGSH/グルタチオンのレベルを分析した。グルタチオンレベルの分析は、眼24における抗酸化保護機構に関与するため、特に関連性が高い。H2O2への曝露は、RPE細胞1、25、26、27、28、29、30、および加えて、酸化ストレスの「生理学的」な刺激源である、酸化ストレスの「生理学的」源であるRPE細胞の酸化ストレス感受性および抗酸化活性を調べるモデルとして頻繁に使用される。
神経保護因子の機能性と有効性を評価するために、PEDFおよびGM-CSFを過剰発現するように遺伝子組み換え細胞によって発現される成長因子の抗酸化効果を定量化する分析を可能にするインビトロモデルを開発した。ここでは、PEDFおよびGM-CSFの遺伝子にトランスフェクトされたRPE細胞は、細胞の形態、密度、生存率、グルタチオンの細胞内レベル、およびミトコンドリア非結合タンパク質2の発現によって証明されるように、非トランスフェクション対照細胞よりもH2O2の有害な影響に対してより耐性があることを示す(ROS1酸素種)。
プロトコル
人間の目の収集と使用のための手順は、研究のための教育倫理委員会によって承認されました (no. 2016-01726).
1. 細胞の分離と培養条件
- ヒトARPE-19細胞株
- ダルベッコの修飾ワシのミディアム/栄養素混合物F-12ハム(DMEM/ハムのF-12)に10%のウシ胎児血清(FBS)、80 U/mLペニシリンを加えた培養5 x 105 ARPE-19細胞、 T75フラスコの加湿雰囲気で37°Cで80μg/mLレンサプレプトマイシン、2.5μg/mLアンホテリシンB(完全培地)を37°Cで、T75フラスコの5%CO2および95%の空気の空気(他の細胞密度については表1参照)
- 週に 3 回、メディアを変更します。
- 細胞が約90%合流(定性評価)に成長したら、培地を吸引し、滅菌1x PBSで細胞を洗浄する。
- 細胞を37°Cで7〜10分間の5%トリプシン-2%EDTA溶液でインキュベートする(容積については 表1参照)。デタッチメントを視覚的に監視します。
- 10% FBS を含む完全なメディアを追加してトリプシン化を停止します(ボリュームについては 表 1を参照)。
- 細胞をトランスフェクト(プロトコルのステップ2.参照)、細胞を1:10(週に1回)の割合でサブ培養するか、96ウェルプレートに播種します(プロトコルのステップ3.3と3.4を参照)。
| ミディアム(mL) | ||||||
| エリア (cm²) | ARPE-19細胞(細胞/ウェル)の播種密度 | アプリケーション | 細胞培養用 | トリプシンを停止するには | トリプシンの体積(mL) | |
| フラスコ T75 | 75 | 5,00,000 | ARPE-19細胞増殖 | 10 | 7 | 3 |
| 6 ウェルプレート | 9.6 | 1,00,000 | トランスフェクトARPE-19細胞の播種 | 3 | 1 | 0.5 |
| 24 ウェルプレート | 2 | 50,000 | トランスフェクトされたhRPE細胞の播種 | 1 | 0.8 | 0.2 |
| 96 ウェルプレート | 0.32 | トランスフェクト細胞による酸化ストレス実験用5,000個(図1) | 酸化ストレス実験 | 0.2 | ||
| 非トランスフェクト細胞とタンパク質を用いた酸化ストレス実験用3,000個(図1) | ||||||
表1:細胞培養量。ARPE-19およびヒト初等型RPE細胞の培養に適した細胞培養プレートおよびフラスコに推奨される培地量。
- ヒト初等RPE細胞
- Thumann et al.17で説明した一次ヒト RPE 細胞を分離し、培養細胞を 20% FBS を添加した完全培地中に含む。
- 週に 2 回メディアを変更します。細胞が合流に達したら(視覚的に監視)、過成長を避けるためにFBSを1%に減らします。
- 細胞をトランスフェクト(プロトコルのステップ2を参照)、または96ウェルプレートに播種します(プロトコルのステップ3.3と3.4を参照)。
注:ここに提示されたデータは、4人のヒトドナーの目から得られたRPE細胞の培養物から収集された。 表2 は、ライオンズ・ギフト・オブ・サイト・アイバンク(セントポール、MN)からの寄付者の人口統計を詳述しています。ヘルシンキ宣言に従ってインフォームド・コンセントが得られた後、目は12.7±5.7時間(平均±SD)死後に核化された。
| いいえ | 年齢 | ジェンダー | 死を保存する(時間) | 死から孤立へ | 耕作 | 耕作 | グラフ内の記号 | |
| (日) | トランスフェクション前(日) | トランスフェクション後(日) | ||||||
| 2 | 80 | M | 20.7 | 8 | 140 | 36 |  | |
| 3 | 86 | F | 12.8 | 8 | 85 | 45 |  | |
| 4 | 86 | F | 8.5 | 5 | 26 | 133 |  | |
| 8 | 83 | F | 8.9 | 6 | 18 | 27 |  | |
| 意味する | 83.8 | 12.7 | 6.8 | 67.3 | 60.3 | |||
| SD | 2.9 | 5.7 | 1.5 | 57.0 | 49.1 |
表2:網膜色素上皮細胞に対するヒトドナーの人口統計
2. ARPE-19およびヒト初原生RPE細胞のエレクトロポレーション
- プロトコルのステップ 1.1.3-1.1.5 で説明されているように、ARPE-19 細胞またはヒトのプライマリ RPE 細胞をトリプシン化します。
- 市販のトランスフェクションキットでエレクトロポレーションを行います( 材料表参照)。
- ARPE-19細胞のトランスフェクションについては、Johnenら32と、Thumannらへの一次hRPEを参照してください。簡単に言えば、 1 x 10 5 ARPE-19細胞または5 x 104の一次hRPEセルを11 μLのRバッファーに再サスペンドし、0.03 μg pSB100Xトランスを含むプラスミド混合物を2 μL加える33および0.47 μg pT2-CMV-PEDF-彼またはpT2-CMV-GMCSF-トランスポゾン(トランスポザーゼ比:トランスポゾン1:16)。PEDFおよびGM-CSF二重トランスフェクション細胞の場合、1:16:16(0.03 μg pSB100X、0.47μg pT2-CMV-PEDF-His、および0.47 μg pT2-CMV-GMCSF-His)の比率を使用してください。ARPE-19細胞の場合は、20 ms(パルス幅)の1,350 Vの2パルスを使用します。1 次細胞の場合は 20 ミリ秒の場合、1,100 V の 2 つのパルスが使用されます。
- 種子1 x 105 トランスフェクションARPE-19または5 x 104 トランスフェクトされた原発hRPE細胞を6ウェルおよび24ウェルプレートで、抗生物質または抗ミコティックなしで10%FBSを添加した培地で。トランスフェクションの3日後にペニシリン(80 U/mL)、ストレプトマイシン(80μg/mL)、アンホテリシンB(2.5 μg/mL)を加えます。
- 細胞の顕微鏡モニタリングを週1回で行い細胞増殖を決定する。トランスフェクション効率は、RT-PCRによる遺伝子発現の解析、およびELISAおよびWBによるタンパク質分泌( 補足材料で説明した方法)によって監視される。
注:トランスフェクション効率は、細胞が合流度に達すると初めて、すなわち、ARPE-19細胞および原発性hRPE細胞のトランスフェクション後〜7日および4週間で初めて評価することができる。 - 96 ウェルプレートのシードセル (プロトコルのステップ 3.5 を参照)。
酸化ストレス誘導(H2O2治療)と神経保護(PEDFおよび/またはGM-CSF治療)
- トランスフェクトARPE-19細胞のコンディション培地の調製
- PEDF、GM-CSF、またはその両方の遺伝子にトランスフェクトされたARPE-19細胞を使用してください(プロトコルのステップ2を参照)。培養細胞は、プロトコルのステップ1.1に記載されているように28日間培養する。
- トランスフェクション後28日で、トリプシン化細胞(プロトコルのステップ1.1.3-1.1.5を参照)、ニューバウアーチャンバー34、35、およびT75フラスコの種子5 x105細胞を完全な媒体に数えます。細胞培養が約80%コンフルエントである場合に培地を交換する(約1週間後、定性的に検証される)。24時間後に培地を収集します。
- 使用するまで-20°Cで保存してください。
注: コンディショナリ培地中の組換え PEDF および GM-CSF の十分な濃度を WB で検証し、 補足材料に記載されているように ELISA で定量しました。
- トランスフェクトARPE-19細胞のコンディション培地からのPEDFおよびGM-CSFの精製
- 4°Cで15分間10,000 x g でステップ3.1.2から回収された培地を遠心分離する。
- 以下に説明するように、メーカーのプロトコルに従ってNi-NTAスーパーフロー( 材料表を参照)を使用して、Hisタグ付きタンパク質を精製してください。
- 30sの2,600 x g で1.5 mLチューブと遠心分離機にNi-NTA混合物のピペット30 μLを、フロースルーを廃棄します。ペレットを200 μLの1xインキュベーションバッファーで2回洗浄します。
- 30 sの2,600 x g で遠心分離機を使用し、フロースルーを廃棄します。4xインキュベーションバッファーの40 μLを追加し、再中断します。
- 900 μLの遠心分離調整された培地を加え、RT.遠心分離機で1分間、70rpm(軌道シェーカー)でインキュベートします。
- 1xインキュベーションバッファーの175 μLでペレットを2回洗浄します。30 sの2,600 x g で遠心分離機を使用し、フロースルーを廃棄します。
- His タグ付き PEDF および GM-CSF タンパク質を溶出するには、溶出バッファーを 20 μL 追加し、RT. 30 s の 2,600 x g で 20 分間、70 rpm (軌道シェーカー) でインキュベートします。組み換えPEDFまたはGM-CSFを含む上清を保管してください。
- メーカーの指示に従って、市販のBCAタンパク質アッセイキット( 材料表を参照)を使用して、全タンパク質を定量化します。
- 使用するまで-20°Cでタンパク質溶液を保管してください。
注:インキュベーションバッファ(4x)は200 mM NaH2PO 4、1.2 M NaCl、および40 mMイミダゾールを含みます。溶出の緩衝は50 mM NaH2PO 4、300 mM NaCl、および250 mMイミダゾールを含んでいる。
- コンディションされた培地とH2O2を用いた非トランスフェクトARPE-19/プライマリhRPE細胞の治療(図1A)
- シード 3,000 非トランスフェクション ARPE-19 (プロトコルのステップ 1.1.6 から) またはプライマリ hRPE (プロトコルのステップ 1.2.3 から) 細胞 96 ウェルプレートおよび培養 200 μL のコンディショニングされた培地のトランスフェクション ARPE-19 細胞から。
- 5%CO2及び95%空気の加湿雰囲気で37°Cで10日間培養した。毎日コンディショムを入れ替えます。細胞を350μMH2O2に24時間曝します。
- 酸化ストレス損傷を評価し、グルタチオンレベル(プロトコルのステップ4.1を参照)、顕微鏡(プロトコルのステップ4.2を参照)、および細胞毒性アッセイ(プロトコルのステップ4.2を参照)の定量化により、PEDFおよびGM-CSFの抗酸化効果を決定します。
注: 実験の期間は 12 日間です。透明な平底マイクロウェルプレートは、細胞形態と同様に発光を評価するために使用されます。細胞毒性とグルタチオンアッセイを同時に行うには、2枚のプレートを同じ日に細胞で播種する必要があります。
- PEDFおよびGM-CSF増殖因子とH2O2を加えた非トランスフェクトARPE-19/一次hRPE細胞の治療(図1B)
- シード 3,000 非トランスフェクション ARPE-19 (プロトコルのステップ 1.1.6 から) またはプライマリ hRPE (プロトコルのステップ 1.2.3 から) ウェルあたりのセル (平坦な底を持つ 96 ウェルプレート) 500 ng/mL 組み換え PEDF および/または 50 ng/mL 組換え組換え CSF,CSF,500 ng/mL 組換え体 PEDF および/または 50 ng/mL 組換え CSF,500 ng/mL 組換え組換え CSF を含む完全な培地の 200 μL の完全な培地 トランスフェクションARPE-19細胞または市販の培地から精製。培養細胞は、5%CO2及び95%空気の加湿雰囲気下で37°Cで48時間培養した。PEDF および GM-CSF の成長要因を含む培地を毎日更新します。
注: 新しい成長因子を培地に追加します。 - 増殖因子で細胞を処理した48時間後、培地を取り除き、350 μM H2O2 プラス500 ng/mL PEDFおよび/または50 ng/mL GM-CSFを含む完全な培地を加えます。
- 酸化ストレス損傷を評価し、グルタチオンレベル(プロトコルのステップ4.1を参照)、顕微鏡(プロトコルのステップ4.2を参照)、および細胞毒性アッセイ(プロトコルのステップ4.2を参照)の定量化により、PEDFおよびGM-CSFの抗酸化効果を決定します。
注: 実験の所要時間は 3 日間です。
- シード 3,000 非トランスフェクション ARPE-19 (プロトコルのステップ 1.1.6 から) またはプライマリ hRPE (プロトコルのステップ 1.2.3 から) ウェルあたりのセル (平坦な底を持つ 96 ウェルプレート) 500 ng/mL 組み換え PEDF および/または 50 ng/mL 組換え組換え CSF,CSF,500 ng/mL 組換え体 PEDF および/または 50 ng/mL 組換え CSF,500 ng/mL 組換え組換え CSF を含む完全な培地の 200 μL の完全な培地 トランスフェクションARPE-19細胞または市販の培地から精製。培養細胞は、5%CO2及び95%空気の加湿雰囲気下で37°Cで48時間培養した。PEDF および GM-CSF の成長要因を含む培地を毎日更新します。
- H2O2を用いてトランスフェクトARPE-19/プライマリhRPE細胞の治療(図1C)
- 補足材料に記載されているようにWBおよびELISAによるトランスフェクト細胞の十分な遺伝子発現およびタンパク質分泌を検証する。
- トランスフェクトされた細胞を含むウェルから培地を取り出します(プロトコルのステップ2を参照)。
- プロトコルのステップ 1.1.3-1.1.5 で説明されているようにセルをトリプシン化します。ノイバウアーチャンバー34、35を使用して細胞を数えます。
- 完全な媒体の200 μLの96-wellプレートに5,000のトランスフェクト細胞/ウェルを入れ。培養細胞は、5%CO2及び95%空気の加湿雰囲気下で37°Cで24時間培養した。24時間後、細胞を24時間350μMH2O2に曝します。
- 酸化ストレス損傷を評価し、グルタチオンレベル(プロトコルのステップ4.1を参照)、顕微鏡(プロトコルのステップ4.2を参照)、細胞毒性アッセイ(プロトコルのステップ4.2を参照)、 およびUCP2 遺伝子発現の測定(プロトコルのステップ4.3を参照)の定量によって、PEDFおよびGM-CSFの抗酸化効果を決定する。
注: 実験の期間は 2 日間です。
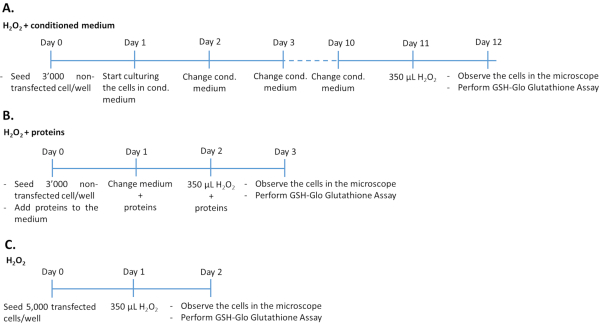
図1:3つの異なる実験手法におけるH2O2アッセイのタイムライン3,000個の非トランスフェクト細胞を、条件培地/組換えタンパク質または5,000個のトランスフェクト細胞で処理し、H2O2で処理するために96ウェルプレートに播種した。コンディショネ培地の効果を決定するために、細胞を100%連続培養培地で10日間培養し、毎日培地を変化させる。組換え増殖因子の効果を決定するために、細胞は3日間連続して毎日適切な量の増殖因子を加えて培養した。非トランスフェクト細胞は、トランスフェクト細胞と比較して培養期間が長い間の過剰増殖を避けるために、ウェルあたり3,000個の細胞で播種することに注意してください。この図の大きなバージョンを表示するには、ここをクリックしてください。
4. 酸化ストレスレベルと抗酸化力の解析
- グルタチオンアッセイ
- メーカーの指示に従って、市販キット( 資料表を参照)を使用してグルタチオン(GSH)レベルを測定します。簡単に、調製し、適切な量の1x試薬ミックス(100 μL試薬/ウェル):ルシフェリン-NT基質とグルタチオンS-トランストランスナーゼ希釈1:100反応バッファー。
注:96ウェルプレートは、10mLの1x試薬ミックスを必要とし、ルシフェリン-NT基質100μLと100μLのグルタチオンS-トランスパーゼを10mLの反応バッファーに加えることで調製されます。使用直前に1x試薬ミックスを準備してください。今後の使用のために準備された試薬ミックスを保管しないでください。 - リコンフィションバッファーのボトル 1 本を凍結乾燥したルシフェリン検出試薬に移して、ルシフェリン検出試薬を調製します。
- グルタチオン(GSH)標準溶液(5mM)を使用して標準曲線を調製します。希釈5 mM GSH溶液 1:100 dH2O(dH2Oの990 μLに5 mM GSH溶液の10 μLを加える)。dH2 O.各希釈規格の 10 μL を適切なウェルに転送して、dH 2 O の 500 μL で 7 シリアル 1:1 希釈を実行します。
注意:グルタチオンの最終濃度は0.039 μMから5 μMの範囲です。 - ブランク(1x試薬ミックス)を準備し、適切なウェルに10 μL(重複)を移します。
- H2O2-処理細胞をインキュベーターから取り出す。
注:明視野顕微鏡(40x)によってH2O2-処理細胞の形態を文書化します。
細胞が酸化されると、彼らはより丸みを帯びた、より少ない広がりを見ます。 - 慎重に培養液を吸引する。調製した1x試薬ミックスを100μLずつ各ウェルに加えます。細胞を500rpmで15sの試薬と共にオービタルシェーカーで混ぜます。
- RTでプレートを30分間インキュベートします。100 μLの再構成されたルシフェリン検出試薬を各ウェルに加えます。
- 軌道シェーカーで500rpmで15 sの溶液を混ぜます。RTでプレートを15分間インキュベートします。
- プレインストールされたプログラムADP-Gloを使用してプレートリーダーを使用して発光を決定します。
メモ:プレートを蓋なしでプレートリーダーの中に入れてください。- レイアウトを 変更 をクリックし、 基本パラメータ: Costar 96-wellプレートで次の設定を選択します。トップオプティカル;位置決め遅延: 0.1;測定開始時間: 0.0;測定間隔時間:1.0;結果を正規化する時間: 0.0;ゲインはデバイスによって自動的に調整されます。ブランク、標準、およびサンプルを定義します。[ 計測の開始] をクリックします。
- データを Excel ファイルとしてエクスポートします。標準曲線の補間によって各サンプル中のGSH濃度を計算します。
- メーカーの指示に従って、市販キット( 資料表を参照)を使用してグルタチオン(GSH)レベルを測定します。簡単に、調製し、適切な量の1x試薬ミックス(100 μL試薬/ウェル):ルシフェリン-NT基質とグルタチオンS-トランストランスナーゼ希釈1:100反応バッファー。
- 細胞毒性アッセイと顕微鏡解析
- 細胞から培地を吸引し、各ウェルに1%FBSを含む完全な培地の100 μLを加えます。細胞をインキュベーターに戻します。
注: 1% FBS は、FBS の高い割合が発光の測定を妨げる可能性があるため、この場合は 1% FBS が使用されます。 - 市販の細胞毒性アッセイキット( 材料表参照)を使用して、製造者の指示に従って細胞の生存率を測定します。簡単に言えば、試薬ミックスを調製し、凍結乾燥した基質にアッセイバッファーを加える。5 mLアッセイバッファーに33 μLジトニンを加えて、リシス試薬を調製します(96ウェルプレート1個)。均質性を確保するために上下にピペットでよく混ぜます。
注:最適な結果を得るには、準備した新しい試薬ミックスを使用してください。RTで保存した場合は12時間以内に使用してください。 凍結と解凍は避ける必要があります。リシス試薬は、最大7日間4°Cで保存できます。 - 未処理の ARPE-19 セルを使用して標準曲線を作成します。
- プロトコルのステップ1.1.3-1.1.5に記載されているように細胞をトリプシン化し、ノイバウアーチャンバー34、35を使用して細胞を数える。RTで120gで細胞を遠心分離し、上澄み液を吸引し、1%FBSを含むDMEM/HamのF12培地中の細胞ペレットを1 x 105細胞/mLの最終濃度に再懸濁する。
- 1%FBSを含む200 μL培地に7個のシリアル1:1希釈液を用意します。各規格の100 μLを適切なウェル(重複)に移します。すべてのウェルに試薬ミックスの50 μLを追加します。
- 細胞を500rpmで15sの試薬と共にオービタルシェーカーで混ぜます。RTでプレートを15分間インキュベートし、プロトコルのステップ4.1.9に記載されているようにプレートリーダーを使用して発光を測定します。50 μLのリシス試薬を加え、15分間インキュベートします。プロトコルのステップ4.1.9に記載されているように、プレートリーダーを用いて発光を測定する。
- 生存細胞の割合を計算します: (100 - % 死んだ細胞) および死んだ細胞の割合 = [第1発光測定 ((標本中の死細胞))/第2の発光測定 (デジトニン処理後全細胞死) x 100.
- 細胞から培地を吸引し、各ウェルに1%FBSを含む完全な培地の100 μLを加えます。細胞をインキュベーターに戻します。
- RT-qPCR によるUCP2発現解析
- 上記のようにトランスフェクトされた細胞をトリプシン化する(プロトコルのステップ1.1.3-1.1.5)。
- ノイバウアーチャンバー34、35を使用して細胞を数えます。
- シード 5,000 トランスフェクション ARPE-19 細胞/ウェル 96 ウェルプレート.
- 24時間培養した後、350μMH2O2で24時間細胞を処理します。
- メーカーの指示に従って、低い細胞からRNAを単離するための商業キットを使用して、トータルRNAを分離します( 材料表を参照)。
- 補足材料に記載されているように、リアルタイム定量 PCR (RT-qPCR) を実行します。簡単に説明すると、最適化されたM-MLV逆転写酵素を含む市販のミックスを用いて転写によってcDNAを生成する(資料表を参照)。
- qPCRの場合は、プライマー(補足材料の表S1を参照)およびDNAテンプレートを除くすべての成分(SYBRグリーンを含む)を含むすぐに使用できる反応カクテルを使用します。次のサーモサイクリング条件を使用してください:10分間95°Cでの最初の変性、15のsの95°Cで変性を伴う40サイクル、30のsのための60°Cでのアニール、および32のsのための72°Cで伸び。
- 分析36に 2^(ΔΔCT) 法を使用します。
- pAktのSDS-PAGEおよびWB分析のための細胞ライセートの調製(Ser473)
- Seed 3 x 105 GM-CSF トランスフェクト ARPE-19 細胞/ウェル 6 ウェルプレート (トランスフェクション後 ≥21 日) を使用して、AKT 生存経路15の活性化を通じて H2O2による損傷から RPE 細胞を保護するかどうかを決定する。
- 24時間培養細胞の後、350 μMH2O2に24時間曝露される。
- 1 mL の RIPA バッファーにプロテアーゼホスファターゼインヒビターカクテル 10 μL、0.5 M EDTA の 10 μL、8 M 尿素 25 μL (1 ウェルに使用されるボリューム) を混合します。
- 慎重に培地を吸引し、1x PBSで細胞を洗浄します。
- RIPA バッファー ミックスのボリューム全体をセルに追加します。
- ピペットの上下。
- 1.5 mLチューブでライセートを収集します。
- 4°Cで30分間20,000xgで遠心分離機。
- 上清を新しい1.5 mLチューブに移します。
- 補充材料に記載されているようにWBによって未希釈細胞のリセートの15 μLのpAktのレベルを決定する。
結果
ヒト網膜色素上皮細胞における酸化ストレスの誘導
ARPE-19および一次hRPE細胞を24時間H2O2の様々な濃度で処理し、抗酸化グルタチオンの細胞内レベルを定量化した(図2A、B)。H2O2は50μMおよび100μMでグルタチオンの産生に影響を与えなかったのに対し、350μMではARPE-19および一次hRPE細胞においてグ?...
ディスカッション
ここで紹介するプロトコルは、トランスフェクトされた細胞によって産生されるPEDFおよびGM-CSFの抗酸化および保護機能を分析するアプローチを提供し、これは任意の推定有益な遺伝子をトランスフェクトした細胞に適用することができる。遺伝子組換え細胞を移植してタンパク質を組織に送達することを目的とする遺伝子治療戦略では、タンパク質発現のレベル、発現の長さ、および発現タ...
開示事項
著者らは開示するものは何もない。
謝辞
著者らは、グレッグ・シーリーとアラン・コンティに優れた技術支援と、ベルリンのマックス・デルブリュック・センターのツズザンナ・イズスヴァーク教授に感謝したいと考えています。この研究は、第7枠組みプログラムの文脈でスイス国立科学財団と欧州委員会によって支援されました。Z.Iは欧州研究評議会、ERCアドバンス[ERC-2011-ADG 294742]によって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 24-well plates | Corning | 353047 | |
| 6-well plates | Greiner | 7657160 | |
| 96-well culture plate white with clear flat bottom | Costar | 3610 | Allows to check the cells before measuring the luminescence (GSH-Glo Assay) |
| 96-well plates | Corning | 353072 | |
| Acrylamid 40% | Biorad | 161-0144 | |
| Amphotericin B | AMIMED | 4-05F00-H | |
| Antibody anti-GMCSF | ThermoFisher Scientific | PA5-24184 | |
| Antibody anti-mouse IgG/IgA/IgM | Agilent | P0260 | |
| Antibody anti-PEDF | Santa Cruz Biotechnology Inc | sc-390172 | |
| Antibody anti-penta-His | Qiagen | 34660 | |
| Antibody anti-phospho-Akt | Cell Signaling Technology | 9271 | |
| Antibody anti-rabbit IgG H&L-HRP | Abcam | ab6721 | |
| Antibody donkey anti-rabbit Alexa Fluor 594 | ThermoFisher Scientific | A11034 | |
| Antibody goat anti-mouse Alexa 488 | ThermoFisher Scientific | A-11029 | |
| ARPE-19 cell line | ATCC | CRL-2302 | |
| BSA | Sigma-Aldrich | A9418-500G | |
| chamber culture glass slides | Corning | 354118 | |
| CytoTox-Glo Cytotoxicity Assay | Promega | G9291 | |
| DAPI | Sigma-Aldrich | D9542-5MG | |
| DMEM/Ham`s F12 | Sigma-Aldrich | D8062 | |
| Duo Set ELISA kit | R&D Systems | DY215-05 | |
| EDTA | ThermoFisher Scientific | 78440 | |
| ELISAquant kit | BioProducts MD | PED613-10-Human | |
| Eyes (human) | Lions Gift of Sight Eye Bank (Saint Paul, MN) | ||
| FBS | Brunschwig | P40-37500 | |
| Fluoromount Aqueous Mounting Medium | Sigma-Aldrich | F4680-25ML | |
| FLUOstar Omega plate reader | BMG Labtech | ||
| GraphPad Prism software (version 8.0) | GraphPad Software, Inc. | ||
| GSH-Glo Glutathione Assay | Promega | V6912 | |
| hydrogen peroxide (H2O2) | Merck | 107209 | |
| ImageJ software (image processing program) | W.S. Rasband, NIH, Bethesda, MD, USA; https://imagej.nih.gov/ij/; 1997–2014 | ||
| Imidazol | Axonlab | A1378.0010 | |
| Leica DMI4000B microscope | Leica Microsystems | ||
| LightCycler 480 Instrument II | Roche Molecular Systems | ||
| LightCycler 480 SW1.5.1 software | Roche Molecular Systems | ||
| NaCl | Sigma-Aldrich | 71376-1000 | |
| NaH2PO4 | Axonlab | 3468.1000 | |
| Neon Transfection System | ThermoFisher Scientific | MPK5000 | |
| Neon Transfection System 10 µL Kit | ThermoFisher Scientific | MPK1096 | |
| Neubauer chamber | Marienfeld-superior | 640010 | |
| Ni-NTA superflow | Qiagen | 30410 | |
| Nitrocellulose | VWR | 732-3197 | |
| Omega Lum G Gel Imaging System | Aplegen Life Science | ||
| PBS 1X | Sigma-Aldrich | D8537 | |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781-100 | |
| PerfeCTa SYBR Green FastMix | Quantabio | 95072-012 | |
| PFA | Sigma-Aldrich | 158127-100G | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23227 | |
| Primers | Invitrogen | See Table 1 in Supplementary Materials | |
| pSB100X (250 ng/µL) | Mátés et al., 2009. Provide by Prof. Zsuzsanna Izsvak | ||
| pT2-CMV-GMCSF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| pT2-CMV-PEDF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| QIAamp DNA Mini Kit | QIAGEN | 51304 | |
| recombinant hGM-CSF | Peprotech | 100-11 | |
| recombinant hPEDF | BioProductsMD | 004-096 | |
| ReliaPrep RNA Cell Miniprep System | Promega | Z6011 | |
| RIPA buffer | ThermoFisher Scientific | 89901 | |
| RNase-free DNase Set | QIAGEN | 79254 | |
| RNeasy Mini Kit | QIAGEN | 74204 | |
| SDS | Applichem | A2572 | |
| Semi-dry transfer system for WB | Bio-Rad | ||
| SuperMix qScript | Quantabio | 95048-025 | |
| Tris-buffered saline (TBS) | ThermoFisher Scientific | 15504020 | |
| Triton X-100 | AppliChem | A4975 | |
| Trypsin/EDTA | Sigma-Aldrich | T4174 | |
| Tween | AppliChem | A1390 | |
| Urea | ThermoFisher Scientific | 29700 | |
| WesternBright ECL HRP substrate | Advansta | K-12045-D50 | |
| Whatman nitrocellulose membrane | Chemie Brunschwig | MNSC04530301 |
参考文献
- Zareba, M., Raciti, M. W., Henry, M. M., Sarna, T., Burke, J. M. Oxidative stress in ARPE-19 cultures: Do melanosomes confer cytoprotection. Free Radical Biology and Medicine. 40 (1), 87-100 (2006).
- Gong, X., Draper, C. S., Allison, G. S., Marisiddaiah, R., Rubin, L. P. Effects of the macular carotenoid lutein in human retinal pigment epithelial cells. Antioxidants. 6 (4), (2017).
- Sacconi, R., Corbelli, E., Querques, L., Bandello, F., Querques, G. A Review of current and future management of geographic atrophy. Ophthalmology and Therapy. 6, 69-77 (2017).
- Al-Zamil, W. M., Yassin, S. A. Recent developments in age-related macular degeneration: a review. Clinical Interventions in Aging. 12, 1313-1330 (2017).
- Kumar-Singh, R. The role of complement membrane attack complex in dry and wet AMD - From hypothesis to clinical trials. Experimental Eye Research. 184, 266-277 (2019).
- Ung, L., Pattamatta, U., Carnt, N., Wilkinson-Berka, J. L., Liew, G., White, A. J. R. Oxidative stress and reactive oxygen species. Clinical Science. 131, 2865-2883 (2017).
- Beatty, S., Koh, H. H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Alhasani, R. H., et al. Gypenosides protect retinal pigment epithelium cells from oxidative stress. Food and Chemical Toxicology. 112, 76-85 (2018).
- . National Institute of Health Available from: https://nei.nih.gov/learn-about-eye-health/resources-for-health-educators/eye-health-data-and-statistics/age-related-macular-degeneration-amd-data-and-statistics (2020)
- Mitchell, P., Liew, G., Gopinath, B., Wong, T. Y. Age-related macular degeneration. The Lancet. 392, 1147-1159 (2018).
- He, Y., Leung, K. W., Ren, Y., Jinzhi, P., Jian, G., Tombran-Tink, J. PEDF improves mitochondrial function in RPE cells during oxidative stress. Investigative Ophthalmology & Visual Science. 55, 6742-6755 (2014).
- Cao, S., Walker, G. B., Wang, X., Cui, J. Z., Matsubara, J. A. Altered cytokine profiles of human retinal pigment epithelium: Oxidant injury and replicative senescence. Molecular Vision. 19, 718-728 (2013).
- Farnoodian, M., Sorenson, C. M., Sheibani, N. PEDF expression affects the oxidative and inflammatory state of choroidal endothelial cells. Amercian Journal of Physiology and Cell Physiology. 314 (4), 456-472 (2018).
- Polato, F., Becerra, S. P. Retinal Degenerative Diseases: Mechanisms and Experimental Therapies. Retinal Degenerative Diseases. , 699-706 (2016).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF regulates the ERK1/2 pathways and protects injured retinal ganglion cells from induced death. Experimental Eye Research. 89, 665-677 (2009).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF protects rat photoreceptors from death by activating the SRC-dependent signalling and elevating anti-apoptotic factors and neurotrophins. Graefes Archives for Clinical and Experimental Ophthalmology. 250, 699-712 (2012).
- Thumann, G., et al. Engineering of PEDF-expressing primary pigment epithelial cells by the SB transposon system delivered by pFAR4 plasmids. Molecular Therapy - Nucleic Acids. 6, 302-314 (2017).
- Garcia-Garcia, L., et al. Long-term PEDF release in rat iris and retinal epithelial cells after Sleeping Beauty transposon-mediated gene delivery. Molecular Therapy - Nucleic Acids. 9, 1-11 (2017).
- Johnen, S., et al. Antiangiogenic and neurogenic activities of Sleeping Beauty-mediated PEDF-transfected RPE cells in vitro and in vivo. BioMed Research International. 2015, (2015).
- Weigel, A. L., Handa, J. T., Hjelmeland, M. L. Microarray analysis of H2O2-, HNE-, or tBH-treated ARPE-19 cells. Free Radical Biology & Medicine. 33 (10), 1419-1432 (2002).
- Allen, R. G., Tresini, M. Oxidative stress and gene regulation. Free Radical Biology and Medicine. 28 (3), 463-499 (2000).
- Tate, D. J., Miceli, M. V., Newsome, D. A. Phagocytosis and H2O2 induce catalase and metallothionein gene expression in human retinal pigment epithelial cells. Investigative Ophthalmology and Visual Science. 36 (7), 1271-1279 (1995).
- Halliwell, B., Clement, M. V., Long, L. H. Hydrogen peroxide in the human body. FEBS Letters. 486 (1), 10-13 (2000).
- Giblin, F. J., McCready, J. P., Kodama, T., Reddy, V. N. A direct correlation between the levels of ascorbic acid and H2O2 in aqueous humor. Experimental Eye Research. 38, 87-93 (1984).
- Geiger, R. C., Waters, C. M., Kamp, D. W., Glucksberg, M. R. KGF prevents oxygen-mediated damage in ARPE-19 cells. Investigative Ophthalmology and Visual Science. 46, 3435-3442 (2005).
- Campochiaro, P. A., et al. Lentiviral vector gene transfer of endostatin/angiostatin for macular degeneration (GEM) study. Human Gene Therapy. 28, 99-111 (2017).
- Chen, X. -. D., Su, M. -. Y., Chen, T. -. T., Hong, H. -. Y., Han, A. -. D., Li, W. -. S. Oxidative stress affects retinal pigment epithelial cell survival through epidermal growth factor receptor/AKT signaling pathway. International Journal of Ophthalmology. 10 (4), 507-514 (2017).
- Tu, G., et al. Allicin attenuates H2O2 - induced cytotoxicity in retinal pigmented epithelial cells by regulating the levels of reactive oxygen species. Molecular Medicine Reports. 13, 2320-2326 (2016).
- Hao, Y., Liu, J., Wang, Z., Yu, L., Wang, J. Piceatannol protects human retinal pigment epithelial cells against hydrogen peroxide induced oxidative stress and apoptosis through modulating. Nutrients. 11, 1-13 (2019).
- Ballinger, S. W., Van Houten, B., Conklin, C. A., Jin, G. F., Godley, B. F. Hydrogen peroxide causes significant mitochondrial DNA damage in human RPE cells. Experimental Eye Research. 68 (6), 765-772 (1999).
- Ma, S., et al. Transgenic overexpression of uncoupling protein 2 attenuates salt-induced vascular dysfunction by inhibition of oxidative stress. American Journal of Hypertension. 27 (3), 345-354 (2014).
- Johnen, S., et al. Sleeping Beauty transposon-mediated transfection of retinal and iris pigment epithelial cells. Investigative Ophthalmology and Visual Science. 53 (8), 4787-4796 (2012).
- Mátés, L., et al. Molecular evolution of a novel hyperactive Sleeping Beauty transposase enables robust stable gene transfer in vertebrates. Nature Genetics. 41 (6), 753-761 (2009).
- . Marienfeld Technical information Neubauer-improved Available from: https://www.marienfeld-superior.com/information-about-our-counting-chambers.html (2020)
- . Electron Microscopy Sciences. Neubauer Haemocytometry Available from: https://www.emsdiasum.com/microscopy/technical/datasheet/68052-14.aspx (2020)
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2^(-ΔΔCT) method. Methods. 25 (4), 402-408 (2001).
- Bascuas, T., et al. Non-virally transfected primary human pigment epithelium cells overexpressing the oxidative stress reduction factors PEDF and GM-CSF to treat retinal neurodegeneration neurodegenerationl. Human Gene Therapy. 30 (11), (2019).
- Zhuge, C. C., et al. Fullerenol protects retinal pigment epithelial cells from oxidative stress-induced premature senescence via activating SIRT1. Investigative Ophthalmology & Visual Science. 55 (7), 4628-4638 (2014).
- Kaczara, P., Sarna, T., Burke, M. Dynamics of H2O2 Availability to ARPE-19 cultures in models of oxidative stress. Free Radical Biology and Medicine. 48 (8), 1068-1070 (2010).
- Gorrini, C., Harris, I. S., Mak, T. W. Modulation of oxidative stress as an anticancer strategy. Nature Reviews Drug Discovery. 12, 931-947 (2013).
- Wang, X., et al. PEDF protects human retinal pigment epithelial cells against oxidative stress via upregulation of UCP2 expression. Molecular Medicine Reports. 19 (1), 59-74 (2019).
- Donadelli, M., Dando, I., Fiorini, C., Palmieri, M. UCP2, a mitochondrial protein regulated at multiple levels. Cellular and Molecular Life Sciences. 71, 1171-1190 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved