È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Induzione e analisi dello stress ossidativo nelle cellule epiteliali del pigmento retinico umano trasforato dalla bella addormentata nel bosco
In questo articolo
Riepilogo
Presentiamo un protocollo per lo sviluppo e l'uso di un modello di stress ossidativo trattando le cellule epiteliali pigmentate retiniche con H2O2, analizzando la morfologia cellulare, la vitalità, la densità, il glutatione e il livello di UCP-2. È un modello utile per studiare l'effetto antiossidante delle proteine secrete dalle cellule trasfettate con trasposone per trattare la degenerazione neuroretinica.
Abstract
Lo stress ossidativo svolge un ruolo fondamentale in diverse malattie degenerative, tra cui la degenerazione maculare legata all'età (AMD), una patologia che colpisce circa 30 milioni di pazienti in tutto il mondo. Porta ad una diminuzione dei fattori neuroprotettivi sintetizzati dall'epitelio pigmentato retinico (RPE), ad esempio il fattore derivato dall'epitelio pigmentato (PEDF) e il fattore stimolante le colonie di granulociti-macrofagi (GM-CSF), seguiti dalla perdita di cellule RPE e infine dalla morte dei fotorecettori e delle cellule gangliari retiniche (RGC). Ipotizziamo che la ricostituzione dell'ambiente retinico neuroprotettivo e neurogenico mediante trapianto subretinico di cellule RPE trasfettate sovraesprimendo PEDF e GM-CSF abbia il potenziale per prevenire la degenerazione retinica mitigando gli effetti dello stress ossidativo, inibendo l'infiammazione e sostenendo la sopravvivenza cellulare. Utilizzando il sistema di trasposone della Bella Addormentata nel Bosco(SB100X)le cellule RPE umane sono state trasfettate con i geni PEDF e GM-CSF e hanno mostrato un'integrazione genica stabile, un'espressione genica a lungo termine e una secrezione proteica utilizzando qPCR, western blot, ELISA e immunofluorescenza. Per confermare la funzionalità e la potenza del PEDF e del GM-CSF secreti dalle cellule RPE trasfettate, abbiamo sviluppato un test in vitro per quantificare la riduzione dello stress ossidativo indotto da H2O2sulle cellule RPE in coltura. La protezione cellulare è stata valutata analizzando la morfologia cellulare, la densità, il livello intracellulare di glutatione, l'espressione genica UCP2 e la vitalità cellulare. Sia le cellule RPE trasfettate che sovraesprimono PEDF e/o GM-CSF che le cellule non trasfettate ma pretrattate con PEDF e/o GM-CSF (disponibili in commercio o purificate da cellule trasfettate) hanno mostrato una significativa protezione delle cellule antiossidanti rispetto ai controlli non trattati. L'attuale modello H2O2è un approccio semplice ed efficace per valutare l'effetto antiossidante di fattori che possono essere efficaci per il trattamento di AMD o malattie neurodegenerative simili.
Introduzione
Il modello qui descritto, offre un approccio utile per valutare l'efficienza degli agenti biofarmaceutici per ridurre lo stress ossidativo nelle cellule. Abbiamo utilizzato il modello per studiare gli effetti protettivi di PEDF e GM-CSF sullo stress ossidativo mediato da H2O2sulle cellule epiteliali pigmentate retiniche, che sono esposte ad alti livelli di O2e luce visibile, e la fagocitosi delle membrane del segmento esterno dei fotorecettori, generando livelli significativi di specie reattive dell'ossigeno (ROS)1, 2. Sono considerati un importante contributo alla patogenesi della degenerazione maculare avascolare legata all'età (aAMD)3,4,5,6,7,8. Inoltre, vi è una diminuzione dei fattori neuroprotettivi sintetizzati RPE, in particolare il fattore derivato dall'epitelio pigmentato (PEDF), i fattori di crescita insulino-simili (IGF) e il fattore stimolante le colonie di macrofagi dei granulociti (GM-CSF) che porta alla disfunzione e alla perdita delle cellule RPE, seguito dalla morte dei fotorecettori e delle cellule gangliari retiniche (RGC)3,4,5 . L'AMD è una malattia complessa che deriva dall'interazione tra fattori metabolici, funzionali, genetici e ambientali4. La mancanza di trattamenti per l'aAMD è la principale causa di cecità nei pazienti di età superiore ai 60 anni nei paesiindustrializzati9,10. La ricostituzione dell'ambiente retinico neuroprotettivo e neurogeno mediante trapianto subretinico di cellule RPE geneticamente modificate sovraesprimendo PEDF e GM-CSF ha il potenziale per prevenire la degenerazione retinica mitigando gli effetti dello stress ossidativo, inibendo l'infiammazione e sostenendo la sopravvivenza cellulare11,12,13,14,15,16 . Anche se ci sono diverse metodologie per fornire geni alle cellule, abbiamo scelto il sistema di trasposoni iperattivi non virali della Bella Addormentata per fornire i geni PEDF e GM-CSF alle cellule RPE a causa del suo profilo di sicurezza, dell'integrazione dei geni nel genoma delle cellule ospiti e della sua propensione a integrare i geni consegnati in siti non trascrizionalmente attivi come abbiamo mostrato in precedenza17, 18,19.
Lo stress ossidativo cellulare può essere indotto in cellule coltivate in vitro da diversi agenti ossidativi, tra cui perossido di idrogeno (H2O2), 4-idronetonale (HNE), terzbutilidroperossido (tBH), alte tensioni di ossigeno e luce visibile (spettro completo o irradiazione UV)20,21. Le elevate tensioni di ossigeno e la luce richiedono attrezzature e condizioni speciali, che limitano la trasferibilità ad altri sistemi. Agenti come H2O2, HNE e tBH inducono cambiamenti molecolari e cellulari da stress ossidativo sovrapposti. Abbiamo scelto H2O2 per testare l'attività antiossidante di PEDF e GM-CSF perché è conveniente e biologicamente rilevante poiché è prodotto dalle cellule RPE come intermedio reattivo dell'ossigeno durante la fagocitosi del segmento esterno dei fotorecettori22 e si trova nei tessuti oculari in vivo23. Poiché l'ossidazione del glutatione può essere parzialmente responsabile della produzione di H2O2 nell'occhio, abbiamo analizzato i livelli di GSH / glutatione nei nostri studi, che sono legati allo stress ossidativo indotto da H2O2e alla capacità rigenerativa delle cellule21,22. L'analisi dei livelli di glutatione è particolarmente rilevante poiché partecipa ai meccanismi protettivi antiossidanti nell'occhio24. L'esposizione a H2O2 è usata frequentemente come modello per esaminare la suscettibilità allo stress ossidativo e l'attività antiossidante dellecellule RPE1,25,26, 27,28, 29,30e, inoltre, mostra somiglianze con il danno da stress ossidativo indotto dalla luce, una fonte "fisiologica" di stress ossidativo21.
Per valutare la funzionalità e l'efficacia dei fattori neuroprotettivi, abbiamo sviluppato un modello in vitro che consente l'analisi per quantificare l'effetto antiossidante dei fattori di crescita espressi da cellule geneticamente modificate per sovraesprimere PEDF e GM-CSF. Qui, mostriamo che le cellule RPE trasfettate con i geni per PEDF e GM-CSF sono più resistenti agli effetti dannosi di H2O2 rispetto alle cellule di controllo non trasfettate, come evidenziato dalla morfologia cellulare, densità, vitalità, livello intracellulare di glutatione ed espressione del gene UCP2, che codifica per la proteina di disaccoppiamento mitocondriale 2 che ha dimostrato di ridurre le specie reattive dell'ossigeno (ROS)31.
Protocollo
Le procedure per la raccolta e l'uso degli occhi umani sono state approvate dalla Commissione etica cantonale per la ricerca (n. 2016-01726).
1. Isolamento cellulare e condizioni di coltura
- Linea cellulare umana ARPE-19
- Coltura 5 x 105 cellule ARPE-19, una linea cellulare RPE umana, nella miscela media/nutritiva F-12 di Dulbecco Modified Eagle (DMEM/Ham's F-12) integrata con il 10% di siero bovino fetale (FBS), 80 U/mL di penicillina, 80 μg/mL di streptomicina e 2,5 μg/mL di amfotericina B (mezzo completo) a 37 °C in un'atmosfera umidificata di 5% DI CO2 e 95% di aria in un matraccio T75 (per altre densità cellulari vedere Tabella 1).
- Cambia il mezzo tre volte a settimana.
- Una volta che le cellule sono cresciute a circa il 90% di confluenza (valutata qualitativamente), aspirare il mezzo e lavare le cellule con 1x PBS sterile.
- Incubare le cellule con una soluzione di TRIPSINA-2% EDTA al 5% per 7-10 minuti a 37 °C (per i volumi vedere Tabella 1). Monitora visivamente il distacco.
- Interrompere la tripsinizzazione aggiungendo un supporto completo contenente il 10% di FBS (per i volumi vedere Tabella 1).
- Trasfettate le cellule (vedi fase 2. del protocollo), sottocoltivate le cellule con un rapporto di 1:10 (una volta alla settimana) o seminate in una piastra a 96 pozzetti come descritto di seguito (vedere i passaggi 3.3 e 3.4 del protocollo).
| Medio (mL) | ||||||
| Superficie (cm²) | Densità di semina per cellule ARPE-19 (cellule/pozzetto) | Applicazione | Per colture cellulari | Per fermare la tripsina | Volume di tripsina (mL) | |
| Pallone T75 | 75 | 5,00,000 | Crescita cellulare ARPE-19 | 10 | 7 | 3 |
| 6 Piastra del pozzo | 9.6 | 1,00,000 | Semina di cellule ARPE-19 trasfettate | 3 | 1 | 0.5 |
| Piatto da 24 pozzetti | 2 | 50,000 | Semina di cellule hRPE trasfettate | 1 | 0.8 | 0.2 |
| 96 Piastra pozzo | 0.32 | 5.000 per esperimenti di stress ossidativo con cellule trasfettate (Fig. 1) | Esperimenti di stress ossidativo | 0.2 | ||
| 3.000 per esperimenti di stress ossidativo con cellule non trasfettate più proteine (Fig. 1) | ||||||
Tabella 1: Volumi di coltura cellulare. Volumi di media raccomandati per piastre di coltura cellulare e palloni per la coltura di ARPE-19 e cellule RPE umane primarie.
- Cellule RPE umane primarie
- Isolare le cellule RPE umane primarie come descritto da Thumann et al.17e le cellule di coltura in mezzo completo integrate con il 20% di FBS.
- Cambia il mezzo due volte a settimana. Una volta che le cellule raggiungono la confluenza (monitorate visivamente), ridurre l'FBS all'1% per evitare una crescita eccessiva.
- Trasfettare le cellule (vedere il passaggio 2 del protocollo) o seminare in una piastra a 96 pozzetti come descritto di seguito (vedere i passaggi 3.3 e 3.4 del protocollo).
NOTA: I dati qui presentati sono stati raccolti dalla coltura di cellule RPE ottenute dagli occhi di quattro donatori umani. La Tabella 2 descrive in dettaglio i dati demografici dei donatori della Lions Gift of Sight Eye Bank (Saint Paul, MN). Gli occhi sono stati enucleati 12,7 ± 5,7 ore (media ± SD) post-mortem dopo che il consenso informato è stato ottenuto in conformità con la Dichiarazione di Helsinki.
| No | età | genere | morte alla conservazione (ore) | morte all'isolamento | coltivazione | coltivazione | Simbolo nel grafico | |
| (giorni) | prima della trasfezione (giorni) | dopo la trasfezione (giorni) | ||||||
| 2 | 80 | M | 20.7 | 8 | 140 | 36 |  | |
| 3 | 86 | F | 12.8 | 8 | 85 | 45 |  | |
| 4 | 86 | F | 8.5 | 5 | 26 | 133 |  | |
| 8 | 83 | F | 8.9 | 6 | 18 | 27 |  | |
| Significare | 83.8 | 12.7 | 6.8 | 67.3 | 60.3 | |||
| SD | 2.9 | 5.7 | 1.5 | 57.0 | 49.1 |
Tabella 2: Dati demografici dei donatori umani per le cellule epiteliali pigmentate retiniche.
2. Elettroporazione di ARPE-19 e cellule RPE umane primarie
- Tripsinizzare le cellule ARPE-19 o le cellule RPE umane primarie come descritto nei passaggi 1.1.3-1.1.5 del protocollo.
- Eseguire l'elettroporazione con il kit di trasfezione disponibile in commercio (vedi Tabella dei materiali).
- Per la trasfezione delle cellule ARPE-19 fare riferimento a Johnen et al.32 e per hRPE primario a Thumann et al.17. In breve, sospendere 1 x 105 cellule ARPE-19 o 5 x 104 cellule hRPE primarie in 11 μL di tampone R e aggiungere 2 μL di miscela di plasmidi contenenti 0,03 μg pSB100X trasposasi33 e 0,47 μg pT2-CMV-PEDF-His o pT2-CMV-GMCSF-Il suo trasposone (rapporto trasposasi:trasposone 1:16). Per le cellule a doppia trasfezione PEDF e GM-CSF, utilizzare un rapporto di 1:16:16 (0,03 μg pSB100X,0,47 μg pT2-CMV-PEDF-His e 0,47 μg pT2-CMV-GMCSF-His). Utilizzare i seguenti parametri di elettroporazione: due impulsi di 1.350 V per 20 ms (larghezza di impulso) per celle ARPE-19; due impulsi di 1.100 V per 20 ms per le cellule primarie.
- Seme 1 x 105 ARPE-19 trasfettato o 5 x 104 cellule hRPE primarie trasfettate in piastre a 6 pozzetti e 24 pozzetti, rispettivamente, in mezzo integrato con FBS al 10% senza antibiotici o antimicotici. Aggiungere penicillina (80 U/mL), streptomicina (80 μg/mL) e amfotericina B (2,5 μg/mL) con il primo scambio medio 3 giorni dopo la trasfezione.
- Determinare la crescita cellulare mediante monitoraggio microscopico settimanale delle cellule. L'efficienza della trasfezione è monitorata dall'analisi dell'espressione genica mediante RT-PCR e della secrezione proteica mediante ELISA e WB (metodi spiegati in Materiale supplementare).
NOTA: L'efficienza della trasfezione può essere valutata per la prima volta che le cellule raggiungono la confluenza, cioè a ~ 7 giorni e 4 settimane dopo la trasfezione rispettivamente per le cellule ARPE-19 e le cellule hRPE primarie. - Cellule seme in una piastra a 96 pozzetti come descritto di seguito (vedere il passaggio 3.5 del protocollo).
3. Induzione da stress ossidativo (trattamento H2O2) e neuroprotezione (trattamento PEDF e/o GM-CSF)
- Preparazione del mezzo condizionato di cellule ARPE-19 trasfettate
- Utilizzare cellule ARPE-19 trasfettate con i geni PEDF, GM-CSF, o entrambi (vedere fase 2 del protocollo); cellule di coltura per 28 giorni come descritto nel passaggio 1.1 del protocollo.
- A 28 giorni dopo la trasfezione, tripsinare le cellule (vedere i passaggi 1.1.3-1.1.5 del protocollo), contare le cellule usando una camera Neubauer34,35e seminare 5 x 105 cellule in palloni T75 in mezzo completo come descritto nel passaggio 1.1.1 del protocollo. Scambiare il mezzo quando la coltura cellulare è confluente all'80% circa (circa dopo 1 settimana; verificata qualitativamente). Raccogliere il mezzo dopo 24 ore.
- Conservare il mezzo a -20 °C fino all'uso.
NOTA: La concentrazione sufficiente del PEDF ricombinante e del liquido GM-CSF nel mezzo condizionato è stata verificata da WB e quantificata da ELISA come descritto in Materiale supplementare.
- Purificazione di PEDF e GM-CSF da mezzo condizionato di cellule ARPE-19 trasfettate
- Centrifugare il mezzo raccolto dal punto 3.1.2 a 10.000 x g per 15 minuti a 4 °C.
- Utilizzare il superflusso Ni-NTA (vedi Tabella dei materiali)secondo i protocolli del produttore per purificare le proteine his-tagged come descritto di seguito.
- Pipettare 30 μL di miscela Ni-NTA in un tubo da 1,5 mL e centrifugare a 2.600 x g per 30 s ed eliminare il flusso attraverso. Lavare il pellet due volte con 200 μL di 1x tampone di incubazione.
- Centrifugare a 2.600 x g per 30 s ed eliminare il flusso attraverso. Aggiungere 40 μL di 4x tampone di incubazione e risospendare.
- Aggiungere 900 μL di mezzo condizionato centrifugato e incubare a 70 rpm (agitatore orbitale) per 1 ora a RT. Centrifugare a 2.600 x g per 1 minuto e il flusso di scarto.
- Lavare il pellet due volte con 175 μL di 1x tampone di incubazione. Centrifugare a 2.600 x g per 30 s ed eliminare il flusso attraverso.
- Per eluire le proteine PEDF e GM-CSF marcate con His, aggiungere 20 μL di tampone di eluizione e incubare a 70 rpm (agitatore orbitale) per 20 minuti a RT. Centrifuga a 2.600 x g per 30 s. Conservare il surnatante contenente PEDF ricombinante o GM-CSF.
- Quantificare la proteina totale utilizzando il kit di analisi delle proteine BCA disponibile in commercio (vedere Tabella dei materiali)secondo le istruzioni del produttore.
- Conservare la soluzione proteica a -20 °C fino all'uso.
NOTA: tampone di incubazione (4x) contiene 200 mM NaH2PO4, 1,2 M NaCl e 40 mM Imidazol; Il tampone di eluizione contiene 50 mM NaH2PO4,300 mM NaCl e 250 mM Imidazol.
- Trattamento di cellule ARPE-19/hRPE primarie non trasfettate con mezzo condizionato più H2O2 (Figura 1A)
- Seminare 3.000 cellule ARPE-19 non trasfettate (dal passo 1.1.6 del protocollo) o hRPE primarie (dal passo 1.2.3 del protocollo) cellule per pozzetto in piastra a 96 pozzetti e coltura in 200 μL di mezzo condizionato da cellule ARPE-19 trasfettate.
- Coltivare le cellule per 10 giorni a 37 °C in un'atmosfera umidificata del 5% di CO2 e del 95% di aria. Cambia il mezzo condizionato ogni giorno. Esporre le celle a 350 μM H2O2 per 24 ore.
- Valutare il danno da stress ossidativo e determinare l'effetto antiossidante di PEDF e GM-CSF mediante quantificazione dei livelli di glutatione (vedere il punto 4.1 del protocollo), la microscopia (vedere il punto 4.2 del protocollo) e il test di citotossicità (vedere il punto 4.2 del protocollo).
NOTA: la durata dell'esperimento è di 12 giorni. Le piastre microwell a fondo piatto trasparente vengono utilizzate per valutare la luminescenza e la morfologia cellulare. Per eseguire contemporaneamente il test di citotossicità e glutatione, due piastre devono essere seminate con cellule nello stesso giorno.
- Trattamento di cellule ARPE-19/hRPE primarie non trasfettate con fattori di crescita PEDF e GM-CSF più H2O2 (Figura 1B)
- Seminare 3.000 cellule ARPE-19 non trasfettate (dal punto 1.1.6 del protocollo) o hRPE primarie (dal punto 1.2.3 del protocollo) per pozzetto (piastre a 96 pozzetti con fondo piatto chiaro) in 200 μL di terreno di coltura completo contenente 500 ng/mL PEDF ricombinante e/o 50 ng/mL di GM-CSF ricombinante, purificato dal mezzo delle cellule ARPE-19 trasfettate o disponibile in commercio. Celle di coltura per 48 ore a 37 °C in atmosfera umidificata al 5% CO2 e al 95% aria. Rinnovare quotidianamente il mezzo, compresi i fattori di crescita PEDF e GM-CSF.
NOTA: aggiungere i fattori di crescita freschi al mezzo. - Dopo 48 ore di trattamento delle cellule con i fattori di crescita, rimuovere il mezzo e aggiungere un mezzo completo contenente 350 μM H2O2 più 500 ng / mL PEDF e / o 50 ng / mL GM-CSF.
- Valutare il danno da stress ossidativo e determinare l'effetto antiossidante di PEDF e GM-CSF mediante quantificazione dei livelli di glutatione (vedere il punto 4.1 del protocollo), la microscopia (vedere il punto 4.2 del protocollo) e il test di citotossicità (vedere il punto 4.2 del protocollo).
NOTA: la durata dell'esperimento è di 3 giorni.
- Seminare 3.000 cellule ARPE-19 non trasfettate (dal punto 1.1.6 del protocollo) o hRPE primarie (dal punto 1.2.3 del protocollo) per pozzetto (piastre a 96 pozzetti con fondo piatto chiaro) in 200 μL di terreno di coltura completo contenente 500 ng/mL PEDF ricombinante e/o 50 ng/mL di GM-CSF ricombinante, purificato dal mezzo delle cellule ARPE-19 trasfettate o disponibile in commercio. Celle di coltura per 48 ore a 37 °C in atmosfera umidificata al 5% CO2 e al 95% aria. Rinnovare quotidianamente il mezzo, compresi i fattori di crescita PEDF e GM-CSF.
- Trattamento delle cellule ARPE-19/hRPE primarie trasfettate con H2O2 (Figura 1C)
- Verificare una sufficiente espressione genica e secrezione proteica delle cellule trasfettate da WB ed ELISA come descritto nel Materiale supplementare.
- Rimuovere il mezzo dai pozzetti contenenti le cellule trasfettate (vedere il passaggio 2 del protocollo).
- Tripsinizzare le cellule come descritto nei passaggi 1.1.3-1.1.5 del protocollo. Contare le cellule usando una camera neubauer34,35.
- Semina 5.000 cellule trasfettate/pozzetto in piastra a 96 pozzetti in 200 μL di mezzo completo. Celle di coltura per 24 ore a 37 °C in atmosfera umidificata del 5% di CO2 e del 95% di aria. Dopo 24 ore, esporre le cellule a 350 μM H2O2 per 24 ore.
- Valutare il danno da stress ossidativo e determinare l'effetto antiossidante di PEDF e GM-CSF mediante quantificazione dei livelli di glutatione (vedere fase 4.1 del protocollo), microscopia (vedere fase 4.2 del protocollo), test di citotossicità (vedere passo 4.2 del protocollo) e determinazione dell'espressione genica UCP2 (vedere passo 4.3 del protocollo).
NOTA: la durata dell'esperimento è di 2 giorni.
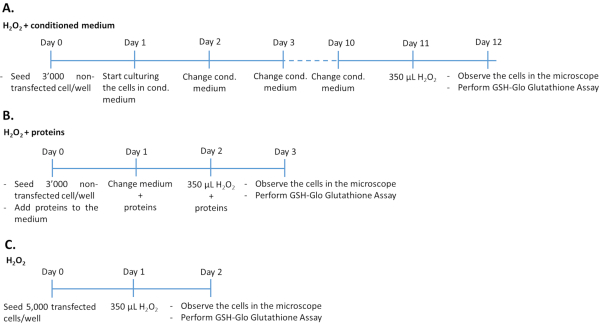
Figura 1: Tempistiche del saggio H2O2 nei tre diversi approcci sperimentali. 3.000 cellule non trasfettate trattate con proteine medie/ricombinanti condizionate o 5.000 cellule trasfettate sono state seminate in piastre a 96 pozzetti per il trattamento con H2O2. Per determinare l'effetto del mezzo condizionato, le cellule sono state coltivate in terreno coltivato al 100% per 10 giorni consecutivi, cambiando mezzo ogni giorno. Per determinare l'effetto dei fattori di crescita ricombinanti, le cellule sono state coltivate aggiungendo la quantità appropriata di fattori di crescita ogni giorno per 3 giorni consecutivi. Si noti che le cellule non trasfettate sono state seminate a 3.000 cellule per pozzetto per evitare la crescita eccessiva durante la durata della coltura più lunga rispetto alle cellule trasfettate. Fare clic qui per visualizzare una versione più grande di questa figura.
4. Analisi del livello di stress ossidativo e della capacità antiossidante
- Saggio del glutatione
- Misurare i livelli di glutatione (GSH) utilizzando il kit disponibile in commercio (vedere Tabella dei materiali)seguendo le istruzioni del produttore. In breve, preparare e volume appropriato di 1x miscela di reagenti (100 μL reagente/ pozzetto): substrato di luciferina-NT e glutatione S-transferasi diluita 1:100 nel tampone di reazione.
NOTA: una piastra a 96 pozzetti richiede 10 mL di 1x miscela di reagenti, che viene preparata aggiungendo 100 μL di substrato di luciferina-NT e 100 μL di glutatione S-transferasi a 10 mL di tampone di reazione. Preparare la miscela di reagenti 1x immediatamente prima dell'uso. Non conservare la miscela di reagenti preparata per un uso futuro. - Preparare il reagente di rilevamento della luciferina trasferendo una bottiglia di tampone di ricostituzione al reagente di rilevamento della luciferina liofilizzato.
- Preparare una curva standard utilizzando una soluzione standard di glutatione (GSH) (5 mM). Diluire 5 mM GSH soluzione 1:100 con dH2O (aggiungere 10 μL di 5 mM GSH soluzione a 990 μL di dH2O). Eseguire 7 diluizioni seriali 1:1 in 500 μL di dH2O. Trasferire 10 μL di ogni standard diluito in un pozzo appropriato in duplice copia.
NOTA: La concentrazione finale di glutatione sarà compresa tra 0,039 μM e 5 μM. - Preparare il bianco (1x miscela di reagenti) e trasferire 10 μL (duplicati) nei pozzetti appropriati.
- Rimuovere le cellule trattate H2O2dall'incubatore.
NOTA: Documentare la morfologia delle cellule trattate con H2O2mediante microscopia a campo luminoso (40x).
Quando le cellule sono ossidate, sembrano più arrotondate e meno diffuse. - Aspirare accuratamente il terreno di coltura. Aggiungere 100 μL di 1x miscela di reagenti preparata a ciascun pozzetto. Mescolare le celle con il reagente per 15 s a 500 giri / min su uno shaker orbitale.
- Incubare la piastra a RT per 30 min. Aggiungere 100 μL di reagente ricostituito per il rilevamento della luciferina a ciascun pozzetto.
- Mescolare la soluzione per 15 s a 500 giri / min su uno shaker orbitale. Incubare la piastra per 15 minuti a RT.
- Determinare la luminescenza utilizzando un lettore di piastre utilizzando un programma preinstallato ADP-Glo.
NOTA: Inserire la piastra all'interno del lettore di piastre senza coperchio.- Fare clic su Cambia layout e scegliere le seguenti impostazioni in Parametri di base:Piastra Costar da 96 pozzetti; ottica superiore; ritardo di posizionamento: 0,1; tempo di inizio della misurazione: 0,0; tempo intervallo di misurazione: 1.0; tempo per normalizzare i risultati: 0.0; il guadagno viene regolato automaticamente dal dispositivo. Definire spazi vuoti, standard ed esempi. Fare clic su Avvia misurazione.
- Esportare i dati come file Excel. Calcolare la concentrazione di GSH in ciascun campione mediante interpolazione della curva standard.
- Misurare i livelli di glutatione (GSH) utilizzando il kit disponibile in commercio (vedere Tabella dei materiali)seguendo le istruzioni del produttore. In breve, preparare e volume appropriato di 1x miscela di reagenti (100 μL reagente/ pozzetto): substrato di luciferina-NT e glutatione S-transferasi diluita 1:100 nel tampone di reazione.
- Saggio di citotossicità e analisi microscopica
- Aspirare il mezzo dalle celle e aggiungere 100 μL di mezzo completo contenente l'1% di FBS a ciascun pozzetto. Restituire le cellule all'incubatore.
NOTA: l'1% FBS viene utilizzato perché percentuali più elevate di FBS possono interferire con la misurazione della luminescenza, quindi in questo caso viene utilizzato l'1% FBS. - Misurare la vitalità cellulare utilizzando il kit per il dosaggio della citotossicità disponibile in commercio (vedere Tabella dei materiali)seguendo le istruzioni del produttore. In breve, preparare la miscela di reagenti aggiungendo il tampone di analisi al substrato liofilizzato. Preparare il reagente di lisi aggiungendo 33 μL di digitonina a 5 mL di tampone di saggio (per una piastra da 96 pozzetti). Mescolare bene pipettando su e giù per garantire omogeneità.
NOTA: per risultati ottimali, utilizzare una miscela di reagenti preparata al momento. Utilizzare entro 12 ore se conservato a RT. La miscela di reagenti può essere conservata a 4 °C per un massimo di 7 giorni e può essere conservata in aliquote monouso per un massimo di 4 mesi a -70 °C. Il congelamento e lo scongelamento devono essere evitati. Il reagente di lisi può essere conservato a 4 °C per un massimo di 7 giorni. - Preparare una curva standard con cellule ARPE-19 non trattate.
- Tripsinizzare le cellule come descritto nei passaggi 1.1.3-1.1.5 del protocollo e contare le cellule usando una camera neubauer34,35. Centrifugare le cellule a 120 g per 10 minuti a RT. Aspirare il surnatante e risospesere il pellet cellulare nel mezzo F12 di DMEM/Ham contenente 1% FBS fino ad una concentrazione finale di 1 x 105 celle/ml.
- Preparare 7 diluizioni seriali 1:1 in mezzo da 200 μL contenente l'1% di FBS. Trasferire 100 μL di ogni standard ai pozzetti appropriati (duplicati). Aggiungere 50 μL di miscela di reagenti a tutti i pozzetti.
- Mescolare le celle con il reagente per 15 s a 500 giri / min su uno shaker orbitale. Incubare la piastra per 15 minuti a RT. Misurare la luminescenza utilizzando il lettore di piastre come descritto nel punto 4.1.9 del protocollo. Aggiungere 50 μL del reagente di lisi e incubare per 15 min. Misurare la luminescenza utilizzando il lettore di piastre come descritto al punto 4.1.9 del protocollo.
- Calcolare la percentuale di cellule vitali: (100 - % cellule morte) e la percentuale di cellule morte = [1a misurazione della luminescenza ((cellule morte nel campione))/ 2a misurazione della luminescenza (tutte le cellule morte dopo il trattamento con digitonina)] x 100.
- Aspirare il mezzo dalle celle e aggiungere 100 μL di mezzo completo contenente l'1% di FBS a ciascun pozzetto. Restituire le cellule all'incubatore.
- Analisi dell'espressione UCP2 mediante RT-qPCR
- Tripsinizzare le cellule trasfettate come descritto sopra (passi 1.1.3-1.1.5 del protocollo).
- Contare le cellule usando una camera neubauer34,35.
- Seme 5.000 cellule ARPE-19 trasfettate / pozzetto in piastre da 96 pozzetti.
- Dopo 24 ore di coltura, trattare le cellule con 350 μM H2O2 per 24 ore.
- Isolare l'RNA totale utilizzando un kit commerciale per l'isolamento dell'RNA da un basso numero di cellule (vedere Tabella dei materiali)seguendo le istruzioni del produttore.
- Eseguire la PCR quantitativa in tempo reale (RT-qPCR) come descritto in Materiale supplementare. In breve, generare cDNA per retrotrascrizione utilizzando un mix disponibile in commercio contenente una trascrittasi inversa M-MLV ottimizzata (vedere Tabella dei materiali).
- Per qPCR utilizzare un cocktail di reazione pronto all'uso contenente tutti i componenti (incluso SYBR Green) ad eccezione dei primer (vedere Tabella S1 del materiale supplementare)e del modello di DNA. Utilizzare le seguenti condizioni di termociclizzazione: denaturazione iniziale a 95 °C per 10 min, 40 cicli con denaturazione a 95 °C per 15 s, ricottura a 60 °C per 30 s e allungamento a 72 °C per 32 s.
- Utilizzare il metodo 2^(-ΔΔCT) per l'analisi36.
- Preparazione del lisato cellulare per l'analisi SDS-PAGE e WB di pAkt (Ser473)
- Seme 3 x 105 cellule ARPE-19 trasfettate con GM-CSF/pozzetto in piastre a 6 pozzetti (≥21 giorni dopo la trasfezione) per determinare se GM-CSF protegge le cellule RPE dal danno da H2O2 attraverso l'attivazione della via di sopravvivenza Akt15.
- Dopo 24 ore di coltura le cellule sono esposte a 350 μM H2O2 per 24 ore.
- Mescolare 1 mL di tampone RIPA con 10 μL di cocktail inibitore della proteasi fosfatasi, 10 μL di 0,5 M EDTA e 25 μL di 8 M di urea (volumi utilizzati per un pozzo).
- Aspirare accuratamente il mezzo e lavare le celle con 1x PBS.
- Aggiungere l'intero volume della miscela tampone RIPA alle celle.
- Pipetta su e giù.
- Raccogliere il lisato in tubi da 1,5 ml.
- Centrifugare a 20.000 x g per 30 min a 4 °C.
- Trasferire il surnatante in un nuovo tubo da 1,5 ml.
- Determinare i livelli di pAkt in 15 μL di lisato cellulare non diluito da WB come descritto in Materiale supplementare.
Risultati
Induzione dello stress ossidativo nelle cellule epiteliali pigmentate retiniche umane
ARPE-19 e cellule hRPE primarie sono state trattate con concentrazioni variabili di H2O2 per 24 ore ed è stato quantificato il livello intracellulare del glutatione antiossidante (Figura 2A,B). H2O2 a 50 μM e 100 μM non ha influenzato la produzione di glutatione, mentre a 350 μM c'è stata u...
Discussione
Il protocollo qui presentato offre un approccio per analizzare la funzione antiossidante e protettiva di PEDF e GM-CSF prodotti da cellule trasfettate, che possono essere applicate a cellule trasfettate con qualsiasi gene benefico putativo. Nelle strategie terapeutiche geniche che hanno l'obiettivo di fornire proteine ai tessuti trapiantando cellule geneticamente modificate, è fondamentale ottenere informazioni sul livello di espressione proteica, sulla longevità dell'espressione e sull'efficacia della proteina espress...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare Gregg Sealy e Alain Conti per l'eccellente assistenza tecnica e il Prof. Zsuzsanna Izsvák del Max-Delbrück Center di Berlino per aver gentilmente fornito i plasmidi pSB100X e pT2-CAGGS-Venus. Questo lavoro è stato sostenuto dalla Fondazione nazionale svizzera per le scienze e dalla Commissione europea nell'ambito del Settimo programma quadro. Z.I è stato finanziato dal Consiglio europeo della ricerca, ERC Advanced [ERC-2011-ADG 294742].
Materiali
| Name | Company | Catalog Number | Comments |
| 24-well plates | Corning | 353047 | |
| 6-well plates | Greiner | 7657160 | |
| 96-well culture plate white with clear flat bottom | Costar | 3610 | Allows to check the cells before measuring the luminescence (GSH-Glo Assay) |
| 96-well plates | Corning | 353072 | |
| Acrylamid 40% | Biorad | 161-0144 | |
| Amphotericin B | AMIMED | 4-05F00-H | |
| Antibody anti-GMCSF | ThermoFisher Scientific | PA5-24184 | |
| Antibody anti-mouse IgG/IgA/IgM | Agilent | P0260 | |
| Antibody anti-PEDF | Santa Cruz Biotechnology Inc | sc-390172 | |
| Antibody anti-penta-His | Qiagen | 34660 | |
| Antibody anti-phospho-Akt | Cell Signaling Technology | 9271 | |
| Antibody anti-rabbit IgG H&L-HRP | Abcam | ab6721 | |
| Antibody donkey anti-rabbit Alexa Fluor 594 | ThermoFisher Scientific | A11034 | |
| Antibody goat anti-mouse Alexa 488 | ThermoFisher Scientific | A-11029 | |
| ARPE-19 cell line | ATCC | CRL-2302 | |
| BSA | Sigma-Aldrich | A9418-500G | |
| chamber culture glass slides | Corning | 354118 | |
| CytoTox-Glo Cytotoxicity Assay | Promega | G9291 | |
| DAPI | Sigma-Aldrich | D9542-5MG | |
| DMEM/Ham`s F12 | Sigma-Aldrich | D8062 | |
| Duo Set ELISA kit | R&D Systems | DY215-05 | |
| EDTA | ThermoFisher Scientific | 78440 | |
| ELISAquant kit | BioProducts MD | PED613-10-Human | |
| Eyes (human) | Lions Gift of Sight Eye Bank (Saint Paul, MN) | ||
| FBS | Brunschwig | P40-37500 | |
| Fluoromount Aqueous Mounting Medium | Sigma-Aldrich | F4680-25ML | |
| FLUOstar Omega plate reader | BMG Labtech | ||
| GraphPad Prism software (version 8.0) | GraphPad Software, Inc. | ||
| GSH-Glo Glutathione Assay | Promega | V6912 | |
| hydrogen peroxide (H2O2) | Merck | 107209 | |
| ImageJ software (image processing program) | W.S. Rasband, NIH, Bethesda, MD, USA; https://imagej.nih.gov/ij/; 1997–2014 | ||
| Imidazol | Axonlab | A1378.0010 | |
| Leica DMI4000B microscope | Leica Microsystems | ||
| LightCycler 480 Instrument II | Roche Molecular Systems | ||
| LightCycler 480 SW1.5.1 software | Roche Molecular Systems | ||
| NaCl | Sigma-Aldrich | 71376-1000 | |
| NaH2PO4 | Axonlab | 3468.1000 | |
| Neon Transfection System | ThermoFisher Scientific | MPK5000 | |
| Neon Transfection System 10 µL Kit | ThermoFisher Scientific | MPK1096 | |
| Neubauer chamber | Marienfeld-superior | 640010 | |
| Ni-NTA superflow | Qiagen | 30410 | |
| Nitrocellulose | VWR | 732-3197 | |
| Omega Lum G Gel Imaging System | Aplegen Life Science | ||
| PBS 1X | Sigma-Aldrich | D8537 | |
| Penicillin/Streptomycin | Sigma-Aldrich | P0781-100 | |
| PerfeCTa SYBR Green FastMix | Quantabio | 95072-012 | |
| PFA | Sigma-Aldrich | 158127-100G | |
| Pierce BCA Protein Assay Kit | ThermoFisher Scientific | 23227 | |
| Primers | Invitrogen | See Table 1 in Supplementary Materials | |
| pSB100X (250 ng/µL) | Mátés et al., 2009. Provide by Prof. Zsuzsanna Izsvak | ||
| pT2-CMV-GMCSF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| pT2-CMV-PEDF-His plasmid DNA (250 ng/µL) | Constructed using the existing pT2-CMV-PEDF-EGFP plasmid reported in Johnen, S. et al. (2012) IOVS, 53 (8), 4787-4796. | ||
| QIAamp DNA Mini Kit | QIAGEN | 51304 | |
| recombinant hGM-CSF | Peprotech | 100-11 | |
| recombinant hPEDF | BioProductsMD | 004-096 | |
| ReliaPrep RNA Cell Miniprep System | Promega | Z6011 | |
| RIPA buffer | ThermoFisher Scientific | 89901 | |
| RNase-free DNase Set | QIAGEN | 79254 | |
| RNeasy Mini Kit | QIAGEN | 74204 | |
| SDS | Applichem | A2572 | |
| Semi-dry transfer system for WB | Bio-Rad | ||
| SuperMix qScript | Quantabio | 95048-025 | |
| Tris-buffered saline (TBS) | ThermoFisher Scientific | 15504020 | |
| Triton X-100 | AppliChem | A4975 | |
| Trypsin/EDTA | Sigma-Aldrich | T4174 | |
| Tween | AppliChem | A1390 | |
| Urea | ThermoFisher Scientific | 29700 | |
| WesternBright ECL HRP substrate | Advansta | K-12045-D50 | |
| Whatman nitrocellulose membrane | Chemie Brunschwig | MNSC04530301 |
Riferimenti
- Zareba, M., Raciti, M. W., Henry, M. M., Sarna, T., Burke, J. M. Oxidative stress in ARPE-19 cultures: Do melanosomes confer cytoprotection. Free Radical Biology and Medicine. 40 (1), 87-100 (2006).
- Gong, X., Draper, C. S., Allison, G. S., Marisiddaiah, R., Rubin, L. P. Effects of the macular carotenoid lutein in human retinal pigment epithelial cells. Antioxidants. 6 (4), (2017).
- Sacconi, R., Corbelli, E., Querques, L., Bandello, F., Querques, G. A Review of current and future management of geographic atrophy. Ophthalmology and Therapy. 6, 69-77 (2017).
- Al-Zamil, W. M., Yassin, S. A. Recent developments in age-related macular degeneration: a review. Clinical Interventions in Aging. 12, 1313-1330 (2017).
- Kumar-Singh, R. The role of complement membrane attack complex in dry and wet AMD - From hypothesis to clinical trials. Experimental Eye Research. 184, 266-277 (2019).
- Ung, L., Pattamatta, U., Carnt, N., Wilkinson-Berka, J. L., Liew, G., White, A. J. R. Oxidative stress and reactive oxygen species. Clinical Science. 131, 2865-2883 (2017).
- Beatty, S., Koh, H. H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Alhasani, R. H., et al. Gypenosides protect retinal pigment epithelium cells from oxidative stress. Food and Chemical Toxicology. 112, 76-85 (2018).
- . National Institute of Health Available from: https://nei.nih.gov/learn-about-eye-health/resources-for-health-educators/eye-health-data-and-statistics/age-related-macular-degeneration-amd-data-and-statistics (2020)
- Mitchell, P., Liew, G., Gopinath, B., Wong, T. Y. Age-related macular degeneration. The Lancet. 392, 1147-1159 (2018).
- He, Y., Leung, K. W., Ren, Y., Jinzhi, P., Jian, G., Tombran-Tink, J. PEDF improves mitochondrial function in RPE cells during oxidative stress. Investigative Ophthalmology & Visual Science. 55, 6742-6755 (2014).
- Cao, S., Walker, G. B., Wang, X., Cui, J. Z., Matsubara, J. A. Altered cytokine profiles of human retinal pigment epithelium: Oxidant injury and replicative senescence. Molecular Vision. 19, 718-728 (2013).
- Farnoodian, M., Sorenson, C. M., Sheibani, N. PEDF expression affects the oxidative and inflammatory state of choroidal endothelial cells. Amercian Journal of Physiology and Cell Physiology. 314 (4), 456-472 (2018).
- Polato, F., Becerra, S. P. Retinal Degenerative Diseases: Mechanisms and Experimental Therapies. Retinal Degenerative Diseases. , 699-706 (2016).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF regulates the ERK1/2 pathways and protects injured retinal ganglion cells from induced death. Experimental Eye Research. 89, 665-677 (2009).
- Schallenberg, M., Charalambous, P., Thanos, S. GM-CSF protects rat photoreceptors from death by activating the SRC-dependent signalling and elevating anti-apoptotic factors and neurotrophins. Graefes Archives for Clinical and Experimental Ophthalmology. 250, 699-712 (2012).
- Thumann, G., et al. Engineering of PEDF-expressing primary pigment epithelial cells by the SB transposon system delivered by pFAR4 plasmids. Molecular Therapy - Nucleic Acids. 6, 302-314 (2017).
- Garcia-Garcia, L., et al. Long-term PEDF release in rat iris and retinal epithelial cells after Sleeping Beauty transposon-mediated gene delivery. Molecular Therapy - Nucleic Acids. 9, 1-11 (2017).
- Johnen, S., et al. Antiangiogenic and neurogenic activities of Sleeping Beauty-mediated PEDF-transfected RPE cells in vitro and in vivo. BioMed Research International. 2015, (2015).
- Weigel, A. L., Handa, J. T., Hjelmeland, M. L. Microarray analysis of H2O2-, HNE-, or tBH-treated ARPE-19 cells. Free Radical Biology & Medicine. 33 (10), 1419-1432 (2002).
- Allen, R. G., Tresini, M. Oxidative stress and gene regulation. Free Radical Biology and Medicine. 28 (3), 463-499 (2000).
- Tate, D. J., Miceli, M. V., Newsome, D. A. Phagocytosis and H2O2 induce catalase and metallothionein gene expression in human retinal pigment epithelial cells. Investigative Ophthalmology and Visual Science. 36 (7), 1271-1279 (1995).
- Halliwell, B., Clement, M. V., Long, L. H. Hydrogen peroxide in the human body. FEBS Letters. 486 (1), 10-13 (2000).
- Giblin, F. J., McCready, J. P., Kodama, T., Reddy, V. N. A direct correlation between the levels of ascorbic acid and H2O2 in aqueous humor. Experimental Eye Research. 38, 87-93 (1984).
- Geiger, R. C., Waters, C. M., Kamp, D. W., Glucksberg, M. R. KGF prevents oxygen-mediated damage in ARPE-19 cells. Investigative Ophthalmology and Visual Science. 46, 3435-3442 (2005).
- Campochiaro, P. A., et al. Lentiviral vector gene transfer of endostatin/angiostatin for macular degeneration (GEM) study. Human Gene Therapy. 28, 99-111 (2017).
- Chen, X. -. D., Su, M. -. Y., Chen, T. -. T., Hong, H. -. Y., Han, A. -. D., Li, W. -. S. Oxidative stress affects retinal pigment epithelial cell survival through epidermal growth factor receptor/AKT signaling pathway. International Journal of Ophthalmology. 10 (4), 507-514 (2017).
- Tu, G., et al. Allicin attenuates H2O2 - induced cytotoxicity in retinal pigmented epithelial cells by regulating the levels of reactive oxygen species. Molecular Medicine Reports. 13, 2320-2326 (2016).
- Hao, Y., Liu, J., Wang, Z., Yu, L., Wang, J. Piceatannol protects human retinal pigment epithelial cells against hydrogen peroxide induced oxidative stress and apoptosis through modulating. Nutrients. 11, 1-13 (2019).
- Ballinger, S. W., Van Houten, B., Conklin, C. A., Jin, G. F., Godley, B. F. Hydrogen peroxide causes significant mitochondrial DNA damage in human RPE cells. Experimental Eye Research. 68 (6), 765-772 (1999).
- Ma, S., et al. Transgenic overexpression of uncoupling protein 2 attenuates salt-induced vascular dysfunction by inhibition of oxidative stress. American Journal of Hypertension. 27 (3), 345-354 (2014).
- Johnen, S., et al. Sleeping Beauty transposon-mediated transfection of retinal and iris pigment epithelial cells. Investigative Ophthalmology and Visual Science. 53 (8), 4787-4796 (2012).
- Mátés, L., et al. Molecular evolution of a novel hyperactive Sleeping Beauty transposase enables robust stable gene transfer in vertebrates. Nature Genetics. 41 (6), 753-761 (2009).
- . Marienfeld Technical information Neubauer-improved Available from: https://www.marienfeld-superior.com/information-about-our-counting-chambers.html (2020)
- . Electron Microscopy Sciences. Neubauer Haemocytometry Available from: https://www.emsdiasum.com/microscopy/technical/datasheet/68052-14.aspx (2020)
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2^(-ΔΔCT) method. Methods. 25 (4), 402-408 (2001).
- Bascuas, T., et al. Non-virally transfected primary human pigment epithelium cells overexpressing the oxidative stress reduction factors PEDF and GM-CSF to treat retinal neurodegeneration neurodegenerationl. Human Gene Therapy. 30 (11), (2019).
- Zhuge, C. C., et al. Fullerenol protects retinal pigment epithelial cells from oxidative stress-induced premature senescence via activating SIRT1. Investigative Ophthalmology & Visual Science. 55 (7), 4628-4638 (2014).
- Kaczara, P., Sarna, T., Burke, M. Dynamics of H2O2 Availability to ARPE-19 cultures in models of oxidative stress. Free Radical Biology and Medicine. 48 (8), 1068-1070 (2010).
- Gorrini, C., Harris, I. S., Mak, T. W. Modulation of oxidative stress as an anticancer strategy. Nature Reviews Drug Discovery. 12, 931-947 (2013).
- Wang, X., et al. PEDF protects human retinal pigment epithelial cells against oxidative stress via upregulation of UCP2 expression. Molecular Medicine Reports. 19 (1), 59-74 (2019).
- Donadelli, M., Dando, I., Fiorini, C., Palmieri, M. UCP2, a mitochondrial protein regulated at multiple levels. Cellular and Molecular Life Sciences. 71, 1171-1190 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon