Method Article
Incubadora de plataformas con etapa XY móvil: una nueva plataforma para implementar la oxidación fotoquímica rápida en células de proteínas
En este artículo
Resumen
Una nueva plataforma estática se utiliza para caracterizar la estructura proteica y los sitios de interacción en el entorno celular nativo utilizando una técnica de huella proteica llamada oxidación fotoquímica rápida en células de proteínas (IC-FPOP).
Resumen
La oxidación fotoquímica rápida de proteínas (FPOP) junto con la espectrometría de masas (EM) se ha convertido en una herramienta invaluable en la proteómica estructural para interrogar interacciones proteicas, estructura y dinámicas de conformación de proteínas en función de la accesibilidad a los disolventes. En los últimos años, el alcance de FPOP, una técnica de impresión de pies de proteína de radical hidroxilo (HRPF), se ha ampliado al etiquetado de proteínas en cultivos celulares vivos, proporcionando los medios para estudiar las interacciones proteicas en el entorno celular enrevesado. Las modificaciones de proteínas en las células pueden proporcionar información sobre los cambios estructurales inducidos por ligando o los cambios de conformación que acompañan la formación compleja de proteínas, todo dentro del contexto celular. Se ha logrado la huella proteica empleando un sistema basado en flujo habitual y un láser de excitador KrF de 248 nm para producir radicales hidroxilos mediante fotolisis de peróxido de hidrógeno, requiriendo 20 minutos de análisis para una muestra celular. Para facilitar experimentos FPOP resueltos en el tiempo, se fue pionero en el uso de una nueva plataforma IC-FPOP basada en placas de 6 pozos. En el sistema actual, un solo pulso láser irradia un pozo entero, lo que trunca el marco de tiempo experimental FPOP, lo que resulta en 20 segundos de tiempo de análisis, una disminución de 60 veces. Este tiempo de análisis muy reducido permite investigar mecanismos celulares como cascadas de señalización bioquímica, plegado de proteínas y experimentos diferenciales (es decir, libres de drogas frente a fármacos) de una manera dependiente del tiempo. Esta nueva instrumentación, titulada Incubadora de plataforma con etapa XY móvil (PIXY), permite al usuario realizar cultivo celular e IC-FPOP directamente en el banco óptico utilizando una incubadora de plataforma con temperatura, CO2 y control de humedad. La plataforma también incluye una etapa de posicionamiento, bombas peristálticas y óptica de espejo para la orientación del rayo láser. Las condiciones IC-FPOP tales como la configuración óptica, los caudales, las transcciones transitorias, y la concentración H2O2 en PIXY se han optimizado y revisado por pares. La automatización de todos los componentes del sistema reducirá la manipulación humana y aumentará el rendimiento.
Introducción
Las técnicas de huella proteica pueden revelar información profunda sobre la organización de proteínas. Estas técnicas esenciales basadas en EM de biología estructural son un componente de la caja de herramientas de espectrometría de masas. Estos métodos sondean la estructura de orden superior de proteínas (HOS) y la sinergia a través del etiquetado covalente1,2,3,4. La oxidación fotoquímica rápida de las proteínas (FPOP) emplea radicales hidroxilos para modificar oxidativamente las cadenas laterales accesibles al disolvente de los aminoácidos5,6 (Tabla 1). El método utiliza un láser de excitación a 248 nm para la fotolisis de peróxido de hidrógeno (H2O2)para generar radicales hidroxilos. Teóricamente, 19 de los 20 aminoácidos pueden ser modificados oxidativamente con Gly siendo la única excepción. Sin embargo, debido a las diferentes tasas de reactividad de los aminoácidos con radicales hidroxilos, modificación de sólo un subconjunto de estos se ha observado experimentalmente. Aún así, el método tiene el potencial de análisis a lo largo de la longitud de una secuencia deproteínas 5. FPOP modifica las proteínas en la escala de tiempo del microsegundo, por lo que es útil para estudiar interacciones débiles con velocidades de apagado rápido. La accesibilidad al disolvente cambia en la unión de ligando o un cambio en la conformación de proteínas, por lo tanto, el poder del método radica en la comparación del patrón de etiquetado de una proteína en múltiples estados (es decir, libre de ligando en comparación con el ligando). Como resultado, FPOP ha tenido éxito en la identificación de sitios de interacción proteína-proteína y ligando proteínas y regiones de cambio conformacional7,8,9,10. El método FPOP se ha extendido desde el estudio de sistemas de proteínas purificadas hasta el análisis en células. FPOP en células (IC-FPOP) puede modificar oxidativamente más de mil proteínas en las células para proporcionar información estructural a través del proteoma11,12. La plataforma IC-FPOP convencional utiliza un sistema de flujo para fluir las celdas un solo archivo más allá del rayo láser. El desarrollo de este sistema permitió que las células individuales tuvieran la misma exposición a la irradiación láser. Esto condujo a un aumento de 13 veces en el número de proteínas etiquetadas oxidativamente12. Sin embargo, una limitación del sistema de flujo es la longitud de un único experimento de muestra que consiste en un intervalo de irradiación de 10 minutos durante el cual se produce una modificación y un ciclo de lavado adicional de 10 minutos. Las limitaciones de tiempo de IC-FPOP hacen que no sea adecuado para estudiar intermedios plegables de proteínas de corta duración o cambios que existen entre las redes de interacción en cascadas de señalización bioquímica. Esta limitación temporal inspiró el diseño de una nueva plataforma IC-FPOP equipada con mayor rendimiento.
Para medir con precisión la estructura de orden superior de proteínas en el entorno celular nativo, el nuevo diseño permite que el cultivo celular se realice directamente en la plataforma láser, lo que permite que IC-FPOP tenga un alto rendimiento. Esta configuración también permite minimizar las perturbaciones al entorno celular, a diferencia de IC-FPOP utilizando el flujo donde las células adherentes deben ser eliminadas del sustrato. La nueva plataforma permite que IC-FPOP se produzca en un sistema de incubación estéril utilizando una cámara superior de etapa controlada por temperatura y CO2 mientras utiliza óptica de espejo configurada para la guía de haz láser, un sistema de posicionamiento para movimiento XY y bombas peristálticas para intercambio químico. La nueva plataforma para la realización de IC-FPOP se titula Incubadora de Plataformas con Etapa XY Móvil (PIXY) (Figura 1). En PIXY, IC-FPOP se lleva a cabo en células humanas cultivadas en placas de seis pozos dentro de la cámara de incubadora de plataforma. Para esta configuración, el rayo láser se refleja hacia abajo en la placa utilizando espejos compatibles con vigas como una etapa de posicionamiento que sostiene la incubadora se mueve, en el plano XY, por lo que el rayo láser está estratégicamente alineado para irradiar sólo un pozo a la vez. Los estudios de validación demuestran que IC-FPOP se puede realizar más rápido en PIXY que en el sistema de flujo y conduce a mayores modificaciones de aminoácidos por proteína. El desarrollo de esta nueva plataforma IC-FPOP expondrá el conocimiento que se puede obtener de los experimentos celulares13.
Protocolo
1. Montaje de la Incubadora de Plataformas con etapa XY móvil
NOTA: La nueva plataforma incluye el sistema de incubación, la etapa de posicionamiento y los controladores, las bombas peristálticas, el láser de excitador KrF de 248 nm y los espejos ópticos ensamblados en una placa de pan óptico imperial.
- Montar el sistema de incubación: la unidad de temperatura, la unidad de dióxido de carbono, el humidificador, la bomba de aire y el sistema de monitoreo táctil (Figura 2A-E).
NOTA: El fabricante proporciona instrucciones detalladas de montaje. El sistema de incubación debe estabilizarse a 5% CO2,37 °C y 85% de humedad antes de crecer células dentro de la incubadora. Estos parámetros pueden depender de la línea celular. - Monte la incubadora de placas personalizada de seis pozos, la etapa de accionamiento de nanoposicionamiento y la etapa de accionamiento XY. El primero se mete en el segundo, respectivamente.
NOTA: Instrucciones detalladas de montaje proporcionadas por el fabricante. - Conecte las cuatro bombas peristálticas en una secuencia de "cadena margarita" a través de cables RS-232, con un cable RS-232 a USB conectado al ordenador controlador. Conecte el tubo de polímero id de 3,18 mm (por ejemplo, Tygon) a cada canal de cada rodillo del canal de la bomba.
NOTA: Cada bomba tiene cuatro rodillos. La dirección y el caudal de los rodillos se manipulan manualmente. - Inserte conectores de 1/16" x 1/8" al final de cada tubo de polímero de identificación de 3,18 mm. Inserte el extremo de 1/16'' del conector en tubos de polímero de identificación 1.59 y, a continuación, inserte el tubo de polímero en la incubadora a través de puertos personalizados. Coloque el otro extremo del tubo en la solución que se infundirá durante la experimentación IC-FPOP.
NOTA: Para las líneas de perfusión, la incubadora tiene 36 puertos personalizados para tuberías alrededor de la periferia para dar cabida a todos los reactivos utilizados. - Atornille un espejo de línea láser de 50 mm, 248 nm y 45° dentro de un soporte de espejo cinemático para óptica de Ø2" en la placa de pan a 10-11 puntos de rejilla de la abertura láser de 248 nm. Coloque el segundo espejo en un ángulo de 90° al primero del otro lado de la incubadora. En ángulo el segundo espejo hacia abajo a aproximadamente 45° para la guía del haz láser a la incubadora (Figura 2F-J).
2. Sincronización y automatización inicial del sistema a través de software de integración
- Instale la última versión del software de integración necesario para controlar los controladores y bombas.
- Refiriéndose al manual de la bomba, cambie el nombre de las bombas comenzando por '5' y aumentando su valor. Los comandos se pueden enviar al sistema de bombas utilizando el subprograma Control manual en el software de integración.
NOTA: Al ajustar una bomba al modo canal, se cambia automáticamente la convención de nomenclatura de la bomba en el conjunto, 1, 2, 3 y 4, correspondientes a los cuatro canales de bomba. - Abra el programa de software de integración para la automatización de la incubadora de plataformas.
- Elija el nombre apropiado del dispositivo de comport USB (por ejemplo, COM4) correspondiente al cable USB del sistema de bombeo en el menú desplegable de conexión etiquetado Comport para bombas.
- Haga clic en el botón Generador de scripts para editar o crear un script para la plataforma de incubadora de plataforma automatizada. Haga clic en Guardar script para guardar la secuencia como un archivo de texto (Figura 3A).
NOTA: El generador de scripts tiene una interfaz de usuario para definir el script con definiciones de número de bomba, dirección, velocidad, volumen, diámetro de tubería, modo de canal y retardo temporzado. - Si se desea el modo de canal en la fabricación del script, un paso en el script debe dedicarse a cambiar la bomba dentro y fuera del modo de canal, y los siguientes pasos correspondientes a los canales de bomba deben etiquetarse como bombas 1-4.
NOTA: Esto garantiza que no haya dos bombas simultáneamente en modo canal durante un script de incubadora de plataforma y que cada bomba se vuelva a pasar al modo heredado después de que los comandos se hayan enviado a los canales necesarios. - Haga clic en el botón Leer script y elija el archivo de script adecuado deseado para la operación de incubadora de plataforma.
- Cambie el botón Ejecutar secuencia a la posición ON (verde) para ejecutar el script y, a continuación, haga clic en el botón INICIO (Figura 3B).
3. Cultivar células en la incubadora de plataformas
NOTA: Las células deben colocarse en la incubadora de plataformas en condiciones estériles en una campana de cultivo celular el día antes de la experimentación.
- Desenrosque la incubadora de plataformas de la etapa de nanoposicionamiento y desconecte las líneas de temperatura, gas y humidificador. Después de rociar la incubadora con 70% etanol, colóquela en una capucha de cultivo celular.
- Cultivar células en un T-175 a aproximadamente 80-90% de confluencia antes de la transferencia.
NOTA: El término confluencia se utiliza como medida del número/cobertura de las células en un plato de cultivo celular o un matraz. - Retire los medios y enjuague con el búfer.
- Desasociar las células mediante trypsin-EDTA mediante el protocolo del fabricante.
- Resuspend en 8 ml de medios y contar las celdas.
- Semillas fibronectina/colágeno14 placas recubiertas de seis pozos con aproximadamente 90-95 μL de células resuspended en cada pozo. Este volumen será apropiado para lograr un 80-90% de confluencia (~1,2 millones de celdas) en cada pozo al día siguiente.
NOTA: Las placas de seis pozos deben estar recubiertas con colágeno antes de la siembra. Los reactivos utilizados durante IC-FPOP están infundidos con velocidades de flujo rápidas. Las placas recubiertas garantizan que las células no se desprendin prematuramente debido a la perfusión de reactivos. - Coloque la placa de semillas en la incubadora de plataformas y cubra con la tapa de cuarzo.
NOTA: Durante el diseño y desarrollo, se cambió una tapa de incubadora de vidrio a cuarzo, por lo que es compatible con la luz láser ultravioleta. - Vuelva a colocar y asegurar la incubadora de plataformas en la etapa de accionamiento de nanoposicionamiento. Vuelva a conectar las líneas de temperatura, gas y humidificador.
- Deje que las células crezcan a la confluencia de la noche a la mañana.
NOTA: Los siguientes pasos de cultivo celular son opcionales y están destinados a experimentos en los que las células humanas se transfectan transitoriamente. Estas medidas evalúan la eficiencia de la transfección en condiciones de cultivo celular. - Transfectar el plásmido que contiene el gen de la proteína quimérica GCaMP2 en células HEK 293 utilizando un kit comercial de transfección catiónica-lipídica.
- Realice transfectaciones transitorias de GCaMP2 en células HEK293T en dos placas de seis pozos.
- Incubar una placa de seis pozos en la incubadora de plataformas, y la segunda placa de seis pozos en una incubadora estándar de CO2.
- Compare la eficiencia de transfección dentro de cada placa mediante imágenes de fluorescencia utilizando un microscopio de fluorescencia.
4. Haga el búfer de saciado y H2O2
- Hacer 100 ml de búfer de enfriamiento que contiene 125 mM N-tert-Butyl-α-fenilnitrone (PBN) y 125 mM N, N′-Dimetilthiourea (DMTU).
NOTA: DMTU y PBN son carroñeros de radicales libres y son permeables a las células. - Diluir H2O2 a 200 mM. Cada muestra requiere 6 ml de H2O2 para sumergir completamente la capa de células en la parte inferior de cada pozo.
NOTA: El búfer de enfriamiento se realiza el día anterior y se almacena a 4 °C durante la noche, protegido de la luz. Diluir el H2O2 el día del experimento.
5. Configure la incubadora de plataformas para IC-FPOP
NOTA: La incubadora de plataforma debe montarse en condiciones estériles. Montar la incubadora en una capucha de cultivo celular estéril.
- Desenrosque la incubadora de plataforma de la etapa de posicionamiento y desconecte las líneas de temperatura, gas y humidificador.
- Después de rociar la incubadora con 70% etanol, colóquela en la campana del cultivo celular.
- Retire la placa de seis pozos de la incubadora de plataforma, asegure la tapa original de la placa y confirme la confluencia celular utilizando un microscopio.
- Una vez confirmada la confluencia celular, reemplace la placa de seis pozos por células confluentes en la incubadora de plataformas dentro de la campana de cultivo celular.
- Inserte tres tubos poliméricos de precortado (15 cm) 1,59 ID en cada pozo a través de los puertos incrustados. Enjuague los tubos a las paredes del pozo y sostenga con anillos impresos 3D personalizados.
NOTA: Seis anillos impresos pla filamento 3D de 33 mm fueron diseñados a medida para asegurar el tubo a las paredes de la placa sin perturbar las células ni interfirarse en el camino del pulso láser. - Cubra la incubadora de plataforma con su tapa de cuarzo. Reemplace y asegure la incubadora superior del escenario en el sistema de posicionamiento. Vuelva a conectar las líneas de temperatura, gas y humidificador.
- Conecte el tubo de polímero de 1,59 ID al extremo de 1/16" de los conectores de 1/16x1/8".
6. Realización de IC-FPOP en la incubadora de plataformas
- Prepare un script de software de integración para la retirada de la bomba. Utilice una bomba peristáltica (Bomba 8) para eliminar completamente los medios celulares de los seis pozos.
- Disolventes prime en cada canal de las otras tres bombas peristálticas (Bombas 5-7). Infunda H2O2 y enfríe el búfer en sus respectivos tubos alternos hasta que los reactivos lleguen a los puertos de incubadora.
- Confirme que el rayo láser ha sido angulado correctamente por los espejos y llega a la incubadora desinhibido.
NOTA: Las gafas de seguridad láser deben usarse siempre que el láser esté en uso. No irradia prematuramente las células durante el proceso de pesca/alineación. Utilice cartón para cubrir completamente la tapa del cuarzo al alinear la viga. Además, utilice un contorno impreso en el papel blanco de una placa de seis pozos para confirmar aún más que el rayo láser está golpeando el centro de cada pozo. Utilice el ajuste Pulso continuo con la frecuencia y la energía más bajas para la alineación. - Compruebe la energía láser utilizando un sensor externo. Utilice un solo pulso láser de 160 mJ a 50 Hz y 27 kV.
NOTA: Se necesita un temporizador para los siguientes pasos. - Prepare el script de software de integración para la perfusión de la bomba después de confirmar la alineación del haz.
- Comience el temporizador y pulse el botón Inicio en el script de la bomba de software de integración al mismo tiempo.
- Infundir 200 mM H2O2 a 35 mL/min en el primer pozo (marca de 6-10 segundos en el temporizador).
NOTA: Hay un retraso de cinco segundos antes de que una bomba comience a infundir. También toma el láser siete segundos antes de que se active el pulso. El pulso láser debe venir inmediatamente después de la perfusión H2O2. - Pulse el botón Inicio del software láser en la marca de 5 segundos para activar el pulso a la marca de 11 segundos en el temporizador.
- Infunda solución de enfriamiento de 125 mM a 35 ml/min en el primer pozo inmediatamente después del pulso láser. Mueva manualmente la etapa de posicionamiento para alinear el siguiente pozo con el rayo láser.
- Repita los pasos anteriores 6.5-6.9 hasta que se haya procesado cada pozo.
NOTA: IC-POP se realiza en triplicato técnico de tres láser y tres muestras no láser. Una placa procesada de seis pozos sirve como una réplica biológica. - En una campana de cultivo celular, utilice un raspador de células para transferir las células de cada pozo a tubos cónicos individuales de 15 ml. Células centrífugas a 1.200 x g durante 5 minutos.
- Deseche las células de sobrenadante y resuspend en 100 μL de búfer de lisis RIPA.
- Transfiera las células a tubos individuales de polipropileno de 1,2 ml.
- Flash congelar todas las muestras en nitrógeno líquido y colocar en un congelador de -80 °C hasta su uso.
NOTA: El protocolo se puede pausar aquí.
7. Extracción de proteínas, purificación y proteolisis
- Descongele las muestras y caliente a 95 °C en un bloque de calor durante 5 minutos.
- Después de calentar, enfríe en hielo durante 5 minutos.
- Agregue 25 unidades de nucleasa al lisato celular para degradar el ADN y arneses lineales y circulares de una sola hebra, doble hebra e incubar en la habitación templada durante 15 minutos.
- Muestras de centrífuga a 16.000 x g durante 10 minutos a 4 °C.
- Recoger el sobrenadante y transferir a un tubo de polipropileno limpio.
- Compruebe la concentración de proteínas utilizando un ensayo de proteína colorimétrica.
- Transfiera 100 μg de muestra a un tubo de polipropileno limpio y lleve a 100 μL con el búfer de lisis celular.
- Reduzca las muestras con ditiotiotioreitol (TDT) de 10 mM a 50 °C durante 45 minutos.
- Muestras frías a temperatura ambiente durante 10 minutos.
- Alquilado con iodoacetamida de 50 mM (IAA) a temperatura ambiente durante 20 minutos.
NOTA: Proteger el IAA de la luz - Añadir 460 μL de acetona pre-refrigerada (-20 °C). Muestras de vórtice y colocar a -20 °C durante la noche.
NOTA: El protocolo se puede pausar aquí. - Al día siguiente, muestras de centrifugadoras a 16.000 x g durante 10 minutos a 4 °C.
- Retire la acetona sin interrumpir el pellet proteico.
- Añadir 50 μL de acetona pre-refrigerada al 90% (-20 °C). Muestras de vórtice para mezclar y centrífuga a 16.000 x g durante 5 minutos adicionales a 4 °C.
- Retire la acetona y deje que las muestras se sequen durante 2-3 minutos.
- Pellet de proteína resuspend con tampón Tris de 10 mM pH 8.
- Trypsina resuspend grado MS (20 μg de stock) en 40 μL de 10 mM Tris buffer pH 8 y añadir 2,5 μg de trypsin a cada muestra.
- Incuba muestras a 37 °C durante la noche.
NOTA: El protocolo se puede pausar aquí. - Evalúe la concentración de péptidos utilizando un ensayo de péptido colorimétrico.
- Saciar las muestras con un 5% de ácido fórmico.
- Transfiera 10 μg de muestra a un tubo de polipropileno limpio.
- Seque la muestra con una centrífuga de vacío y vuelva a consumir con 20 μL de ácido fórmico (FA) grado MS 0,1% en agua.
- Transfiera cada muestra a viales de autoamplificador limpios con tapas pre-cortadas.
8. Cromatografía líquida de alto rendimiento-Espectrometría de masa tándem (LC-MS/MS)
- Para localizar las modificaciones de FPOP, analice el lysate de celda digerido mediante el análisis LC-MS/MS.
- Utilice fases móviles de 0,1% FA en agua (A) y 0,1% FA en acetonitrilo (ACN) (B).
- Cargue 0,5 μg de muestra en una columna de captura de 180 μm x 20 mm C18 (5 μm y 100 Å) y lave la columna con el 99% (A) y el 1% (B) durante 15 minutos.
- Utilizando una columna analítica de 75 μm x 30 cm C18 (5 μm y 125 Å), péptidos digeridos eluto y separados con un caudal de 0,300 μL/min durante 120 minutos.
- Ejecute el gradiente LC de la siguiente manera: 0-1 min, 3% solvente B; 2-100 min, 10-45% B;100−110 min, 45-100% B; 110-115 min, 100% B.
- Vuelva a acondicionar la columna al 3% (B) de 115 a 116 minutos y mantenga pulsado el 3% (B) de 116 a 120 minutos.
- Analice péptidos elutados en modo iones positivos con ionización nano electrospray.
- Adquiera espectros MS1 sobre un rango de escaneo m/z de 375-1500 a una resolución de 60.000.
- Establezca el objetivo de control automático de ganancia (AGC) en 5,0e5 con un tiempo máximo de inyección de 50 ms y 5,0e4 umbral de intensidad.
- Aísle los iones precursores con los estados de carga 2-6 a través de la adquisición dependiente de datos (DDA) con una ventana de aislamiento de 1,2 m/z y un tiempo de ciclo de 4 segundos.
- Someta los iones MS2 a la disociación colisional de alta energía (HCD) (32% de energía normalizada).
- Excluya péptidos después de 1 adquisición de MS/MS durante 60 segundos.
- Establezca la resolución MS/MS en 15.000 con un objetivo AGC de 5,0e4 y un tiempo máximo de inyección de 35 ms.
9. Descubridor de proteome/procesamiento de datos
- Busque archivos de datos sin procesar en tándem en el software de análisis proteómico de abajo hacia arriba disponible en una base de datos de proteínas Homo sapiens relevante y enzima digerida.
- Establezca los parámetros de búsqueda de análisis de proteínas.
- Establezca la tolerancia de fragmento en Tolerancia de iones 0,02 Da y padre en 10 ppm.
- Establezca la especificidad de la enzima para probar la triptina y permita un escote perdido.
- Ajuste el rango de masa a 375-1500 m/z.
- Establecer la confianza de péptidos en el 95% (medio) y la confianza en los residuos en el 99% (alto).
- Acepte proteínas si se identifican al menos dos péptidos distintos con el filtro de tasa de descubrimiento (FDR) del 5%.
- Establezca la carbamidometilación como una modificación estática y todas las modificaciones conocidas de la cadena lateral del radical hidroxilo15,16 como modificaciones dinámicas.
- Una vez que los archivos hayan terminado de buscar, exporte secuencia, ubicaciones de modificación, adhesión a proteínas, archivo de espectro, abundancia de precursores e información de tiempo de retención a una base de datos electrónica.
- Calcule el alcance de la modificación por péptido o residuo a partir de esta ecuación:
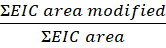
NOTA: El área EIC modificada es el área cromatográfica iónica extraída (EIC) del péptido o residuos con una modificación oxidativa, y el área EIC es el área total del mismo péptido o residuo con y sin la modificación oxidativa. Con el tiempo, la proteína en presencia de peróxido de hidrógeno se oxidará, lo que resulta en oxidaciones de fondo. Para calcular el alcance de la modificación, el área del péptido modificado se divide por el área total. Una muestra de control no irradiada explica la oxidación de fondo. La oxidación de fondo de la muestra de control se resta de la muestra tratada con láser para identificar una modificación de FPOP.
Resultados
Para confirmar que las condiciones de la incubadora de plataformas son suficientes para el cultivo celular en la plataforma láser, GCaMP2 se transfectó transitoriamente a HEK293T y la eficiencia de transfección para ambas placas se evaluó a través de imágenes de fluorescencia(Figura 4A). GCaMP2 es una proteína fluorescente de detección de calcio utilizada como un indicador de calcio intracelular codificado genéticamente. Es una fusión de proteína fluorescente verde (GFP) y la proteína que une calcio, la calmodulina. Se realizó un ensayo luciferasa en células HEK 293 transfectadas con prl-TK plásmido con el fin de cuantificar la eficiencia de la transfección (Figura 4B). Estos resultados muestran que la incubadora de plataformas superó el rendimiento de la incubadora estándar, con un aumento de 1,13 veces en la eficiencia de transfección, proporcionando un punto de referencia cuantitativo para un entorno óptimo de cultivo celular.
Las modificaciones de FPOP en las células HEK293T etiquetadas en el sistema de flujo se compararon con las etiquetadas en la incubadora de plataformas y mostraron que la incubadora de plataformas supera al sistema de flujo tanto en el número de proteínas modificadas(Figura 5)como en la cobertura total de FPOP en esas proteínas. El número de proteínas modificadas FPOP adquiridas en la incubadora de plataformas fue aproximadamente 1051, 2,2 veces más que las adquiridas en un experimento típico. Las modificaciones se combinaron entre dos réplicas biológicas para cada experimento. Además, PIXY proporciona un mayor rendimiento.
Para demostrar la ventaja de una mayor cobertura de modificación a través de una proteína, las modificaciones ic-fpop se localizaron en el nivel de péptido y el grado de modificación se cuantificó para distinguir las diferencias en los resultados entre los sistemas de actina, una proteína de 375 aminoácidos. En el sistema de flujo, se detectaron dos péptidos modificados, proporcionando información estructural limitada (Figura 6A). Sin embargo, se detectaron cinco péptidos modificados que abarcaban la secuencia actin en la incubadora de plataforma. Los espectros de masa tándem indican que los residuos Pro322 fueron modificados y detectados en cada experimento (Figura 6B). Los cinco péptidos modificados en las muestras de incubadora de plataforma contenían doce residuos modificados, mientras que sólo cuatro residuos fueron modificados con el sistema de flujo(Figura 6C). El aumento en la cobertura de oxidación proporciona más información estructural a través de la proteína.
Espino et al. demostraron la capacidad de FPOP para ser realizado in vivo (IV-FPOP) dentro de C. elegans,un modelo de gusano para los estados de enfermedad humana17. Mientras que iv-FPOP también se realiza a través de un sistema de flujo, el sistema PIXY fue probado para la compatibilidad con los gusanos. Aproximadamente 10.000 gusanos fueron incubados en cada pozo de la incubadora de plataformas a 20 °C. El análisis LC-MS/MS reveló que 792 proteínas fueron modificadas por IV-FPOP en la incubadora de plataformas en comparación con las 545 proteínas modificadas con el sistema de flujo (Figura 7). Estos resultados demuestran que además del cultivo celular 2D, esta nueva metodología también es compatible con el estudio de otros sistemas biológicos como C. elegans.
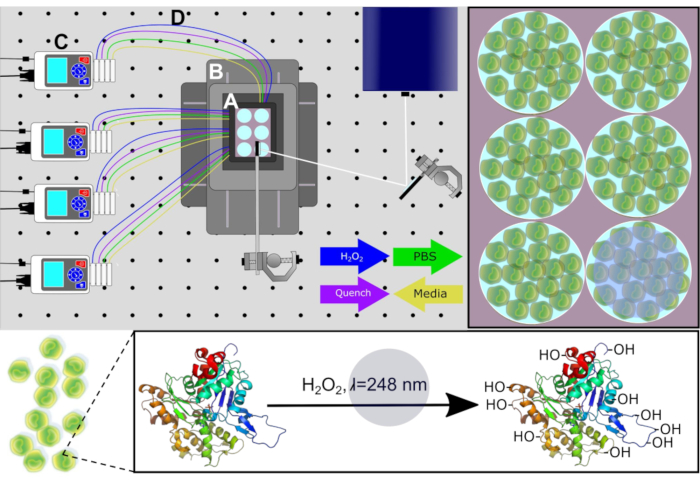
Figura 1. Esquema del sistema PIXY. Componentes del sistema: (A) incubadora de etapa superior, (B) sistema de posicionamiento , (C) bombas peristálticas, y (D) líneas de perfusión. Los medios de cultivo celular se eliminan de cada pozo a través de bombas antes de H2O2 y las soluciones de enfriamiento se infunden en puntos de tiempo calculados. Trayectoria láser para irradiación exhibida en blanco. Reimpreso con permiso de Johnson, D. T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidación fotoquímica rápida en células de proteínas en una incubadora de plataforma con una etapa XY móvil. Química Analítica, 92(2), 1691-1696 2019. Derechos de autor 2020 Sociedad Química Americana. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2. Sistema PIXY totalmente ensamblado. (A) Sistema de monitoreo táctil, (B) unidad de dióxido de carbono, (C) unidad de temperatura, (D) bomba de aire, (E) humidificador, (F) espejos ópticos, (G) incubadora de plataforma, (H) etapa de posicionamiento, (I) láser de excitador KrF de 248nm, y (J) bombas peristálticas. Haga clic aquí para ver una versión más grande de esta figura.
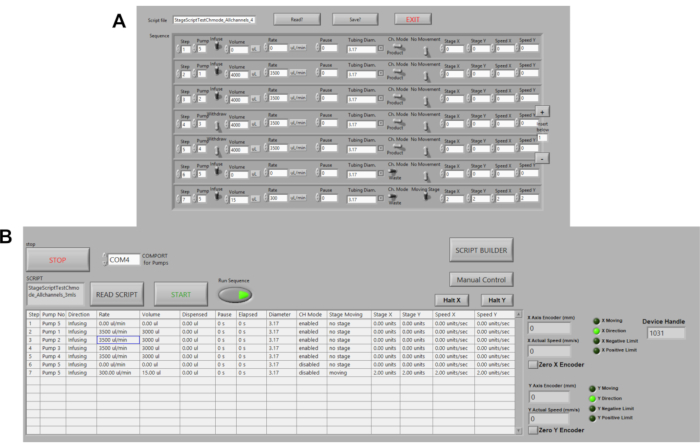
Figura 3. Automatización de bombas peristálticas. (A) Ejemplo de script de comandos en LABVIEW. Las opciones de comando incluyen volumen, caudal, pausas, dirección de flujo. La velocidad, la distancia de etapa y la ubicación se están automatizando actualmente. (B) Lector de scripts en LABVIEW. Aquí, los scripts de comandos se cargan y luego se presiona Ejecutar secuencia y START para iniciar las bombas. Haga clic aquí para ver una versión más grande de esta figura.
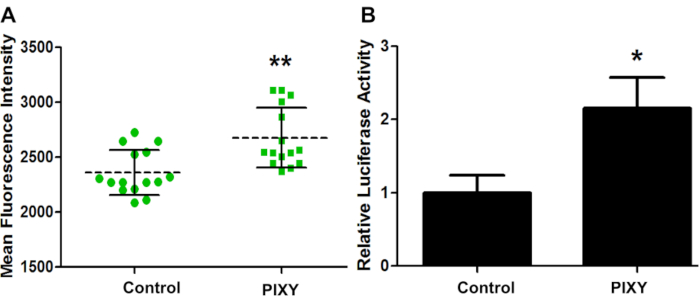
Figura 4. Eficiencia de transfección de células HEK. (A) Intensidad fluorescente media de la comparación de transfección GCaMP2 entre incubadora estándar (Control) y incubadora de etapa superior (PIXY). Puntos y cuadrados representan cada punto en un pozo donde se tomó una medida. B) Eficiencia de transfección cuantificada y validada con un plásmido vectorial diferente, pRL-TK. Valor P< 0,005. Reimpreso con permiso de Johnson, D. T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidación fotoquímica rápida en células de proteínas en una incubadora de plataforma con una etapa XY móvil. Química Analítica, 92(2), 1691-1696 2019. Derechos de autor 2020 Sociedad Química Americana. Haga clic aquí para ver una versión más grande de esta figura.
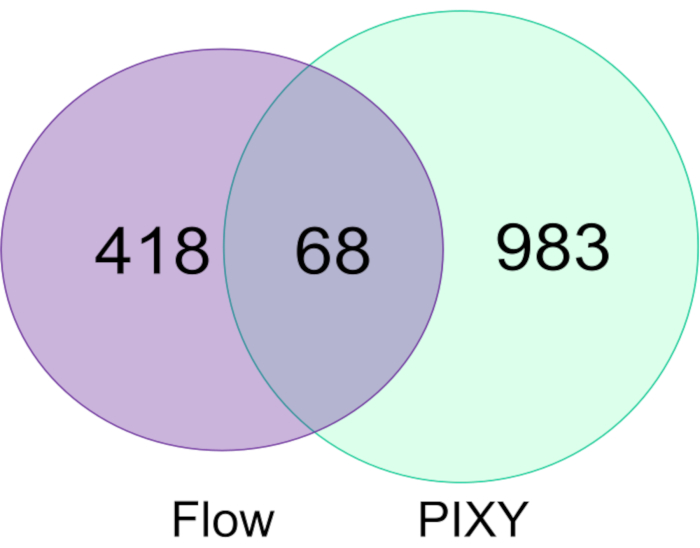
Figura 5. Comparación de proteínas modificadas en el sistema de flujo celular único y PIXY. Diagrama de venina de proteínas modificadas utilizando en el sistema de flujo (púrpura) y en PIXY (verde). Reimpreso con permiso de Johnson, D. T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidación fotoquímica rápida en células de proteínas en una incubadora de plataforma con una etapa XY móvil. Química Analítica, 92(2), 1691-1696 2019. Derechos de autor 2020 Sociedad Química Americana. Haga clic aquí para ver una versión más grande de esta figura.
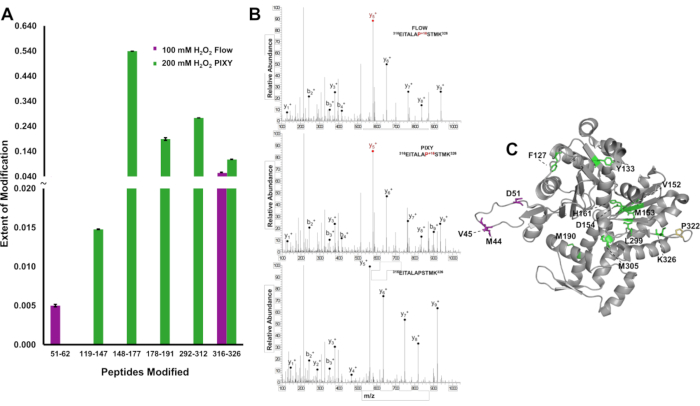
Figura 6. Localización de modificaciones IC-FPOP. Comparación de modificaciones IC-FPOP entre sistemas. (A) Gráfico de barras de péptidos modificados oxidativamente dentro de actin desde el sistema de flujo (púrpura) vs incubadora de plataforma (verde). (B) Espectros de EM tándem de actina (péptido 316-326) con prolínea modificada en ambos sistemas y péptido actin no modificado (C) Residuos modificados FPOP de actina (PDB: 6ZXJ, cadena A 11 residuos modificados en incubadora de plataforma (verde), 3 residuos modificados en el sistema de flujo (púrpura), 1 residuos modificados superpuestos (amarillo). Reimpreso con permiso de Johnson, D. T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidación fotoquímica rápida en células de proteínas en una incubadora de plataforma con una etapa XY móvil. Química Analítica, 92(2), 1691-1696 2019. Derechos de autor 2020 Sociedad Química Americana. Haga clic aquí para ver una versión más grande de esta figura.
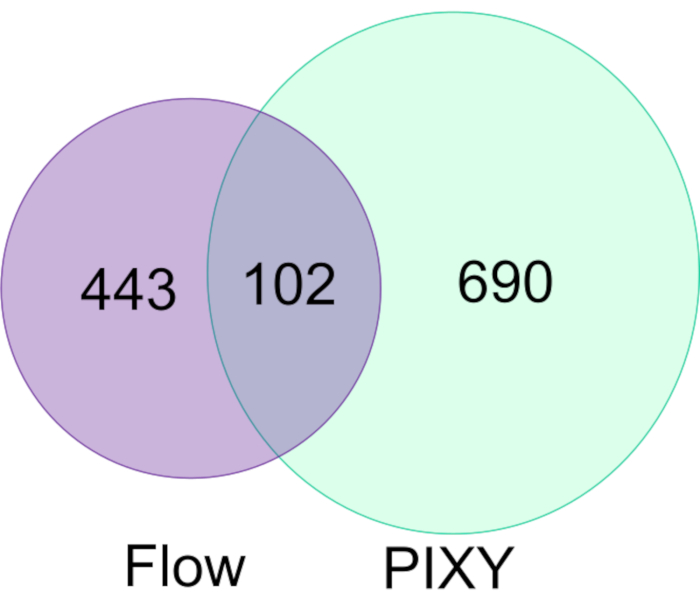
Figura 7. Comparación de proteínas modificadas FPOP en C. elegans por flujo vs PIXY. Hay un aumento de 1,5 veces en las proteínas modificadas oxidativamente usando PIXY en comparación con el sistema de flujo. Reimpreso con permiso de Johnson, D. T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidación fotoquímica rápida en células de proteínas en una incubadora de plataforma con una etapa XY móvil. Química Analítica, 92(2), 1691-1696 2019. Derechos de autor 2020 Sociedad Química Americana. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1. Distribución de modificación de flujo de trabajo y desplazamientos masivos (Da). Haga clic aquí para descargar esta tabla.
Discusión
Las proteínas realizan gran parte del trabajo en células vivas. Dada esta importancia, se necesitan datos detallados sobre la función proteica y la estructura de orden superior (HOS) en el entorno celular para profundizar la comprensión de los entresijos en complejos más grandes y reacciones enzimáticas en las células en lugar de sistemas purificados. Para ello, se adoptó un método de impresión de pies de proteína de radical hidroxilo (HRFP) titulado Oxidación fotoquímica rápida en células de proteínas (IC-FPOP). La mayoría de los estudios de FPOP se han realizado in vitro en sistemas proteicos relativamente puros, lo que contrasta notablemente con el ambiente molecular lleno de gente que afecta a las interacciones de unión y la dinámica de conformación de proteínas. Como resultado, hay un abismo entre los hallazgos de los experimentos in vitro 18 y los que se obtendrían en un entorno celular real. Para cerrar la brecha entre las condiciones idealizadas de un experimento FPOP in vitro y la naturaleza compleja de la célula, se ha desarrollado una nueva plataforma automatizada de seis pozos basada en placas en células-FPOP. Esta novedosa tecnología FPOP es capaz de identificar y caracterizar estas especies moleculares y rastrear sus interacciones moleculares dinámicas tanto en estados sanos como enfermos. Esta nueva plataforma se llama Incubadora de Plataforma con etapa XY Móvil (PIXY).
FPOP se ha utilizado con éxito para caracterizar la información estructural dentro del proteoma. Sin embargo, cada técnica biológica tiene ciertas limitaciones que requieren una mejora adicional. Se requieren reactivos específicos durante la fotolisis láser y para saciar eficientemente los radicales hidroxilos no reactivos. La separación de péptidos digeridos puede requerir grandes cantidades de tiempo para maximizar la información estructural. Esta gran cantidad de información también puede requerir una cuantificación extensa durante el análisis de datos posteriores a la EM1. La incubadora de plataformas, incluida la maquinaria periférica necesaria para el cultivo celular y IC-FPOP en la plataforma láser, tiene un gran costo que puede no ser factible para algunos laboratorios. A medida que se sigan avanzando, el software robusto y las herramientas de análisis deben seguir avanzando en la técnica; algunos de los cuales se muestran en este estudio. Los estudios actuales en esta incubadora de plataforma se han realizado en células HEK293T y en C. elegans. Se ha demostrado que el método IC-FPOP es compatible con una amplia variedad de líneas celulares, incluyendo ovario de hámster chino (CHO), vero, MCF-7 y células MCF10-A19. Puesto que el método general IC-FPOP es traducible a esta plataforma estática, estas líneas celulares deben ser amenables para el estudio usando PIXY también.
IC-FPOP utiliza H2O2 para modificar oxidativamente cadenas laterales accesibles de disolvente de aminoácidos, para luego discernir aún más las interacciones proteicas, estructura, y efectos metabólicos dentro de las células viables que es significativo en proporcionar contexto biológico. Es esencial antes de un experimento IC-FPOP para confirmar que las células son viables después de la adición H2O2. Los estudios de viabilidad celular demostraron que las células eran viables en presencia de concentraciones H2O2 de hasta 200 mM 13. También es importante asegurarse de que H2O2 está infundido a una concentración final de 200 mM directamente en las células después de que se elimina el medio. Si no se eliminan completamente los medios de cultivo celular, se realizarán concentraciones variables de H2O2. En comparación con las condiciones estándar, aumentar el tiempo de incubación a 10 segundos junto con el aumento de la concentración H2O2 condujo a un mayor número de proteínas modificadas por IC-FPOP en la incubadora de plataformas. Es imperativo preparar bombas peristálticas antes de su uso para garantizar que las bombas funcionen correctamente y que el líquido se disperse. Si no lo hace, puede provocar burbujas de aire en el tubo, un volumen insuficiente de H2O2 para sumergir las células y/o un volumen insuficiente de solución de enfriamiento.
Otro problema que puede surgir son los retrasos no deseados en el sistema. Un ejemplo de esto es el proceso de verificar los comandos recibidos para los sistemas de bomba que agrega retrasos significativos en el orden de 1000 o más milisegundos utilizando el software de integración. Este problema se puede solucionar minimizando la comunicación con las bombas durante el experimento y utilizando comandos preestables con anticipación tanto como sea posible.
En el futuro, el objetivo de PIXY es producir un sistema totalmente automatizado e integrado. Además de las bombas peristálticas, se automatizará la activación del pulso láser. También se utilizará un nuevo sistema de posicionamiento para el rápido movimiento de la incubadora de plataformas para mejorar la velocidad y la precisión. Todos los componentes del sistema seguirán estadándose utilizando el software de integración para aumentar aún más el rendimiento.
Divulgaciones
Los autores no declaran ningún interés financiero competidor.
La investigación presentada en este documento está asociada a la solicitud de patente a la que se hace referencia a continuación:
Número de solicitud de patente no provisional de ee.UU.: 17/042.565
Título: "Dispositivo y método para determinar el plegado de proteínas"
Número de expediente UMB: LJ-2018-104 UMass Ref: UMA 18-059.
Agradecimientos
Este trabajo fue apoyado por una subvención del NIH R01 GM128983-01.
Materiales
| Name | Company | Catalog Number | Comments |
| Ismatec Reglo ICC Peristaltic Pumps | Cole-Palmer | 122270050 | |
| Kinematic Mirror Mount for Ø2" Optics | Thor Labs | ||
| 10X trypsin−EDTA | Corning | AB00490-00005 | |
| 50.0mm 248nm 45°, Excimer Laser Line Mirror | Edmond Optics | ||
| Acetone, HPLC Grade | Fisher Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | ||
| ACQUITY UPLC M-Class Symmetry C18 Trap Column, 100Å, 5 µm, 180 µm x 20 mm, 2G, V/M, 1/pkg | Waters | 23275 | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| ACQUITY UPLC M-Class System | Waters | A53225 | |
| Afinia H480 3D Printer | (Innovation Space University of Maryland Baltimore Campus Library) | ||
| air pump | OKOlabs | D12345 | |
| Aluminum Foil | Fisher Scientific | Stock #63-120 | |
| Aqua 5 µm C18 125 Å packing material | Phenomenex | ||
| carbon dioxide unit | OKOlabs | MadMotor®-UHV | |
| Centrifuge | Eppendorf | ||
| connectors (Y PP 1/16” and 1/16x1/8”) | Cole-Palmer | 116891000 | |
| Delicate Task Wipers | Fisher Scientific | 022625501 | |
| Dithiothreiotol (DTT) | AmericanBio | ||
| DMSO, Anhydrous | Invitrogen | LS118-500 | |
| Dulbecco’s phosphate-buffered saline (DPBS) | Corning | SK-78001-82 | |
| EX350 excimer laser | GAM Laser | PI89900 | |
| Fetal bovine serum (FBS) | Corning | SK-12023-78 | |
| Formic Acid, LC/MS Grade | Fisher Scientific | A929-4 | 4 L quantity is not necessary |
| HEK293T cells | Paul Shapiro Lab (University of Maryland Baltimore) | ||
| humidifier | OKOlabs | Nano-LPMW stage | |
| HV3-2 VALVE | Hamilton | BP152-500 | |
| Hydrogen Peroxide | Fisher Scientific | LS120-500 | |
| Iodoacetamide (IAA) | ACROS Organics | ||
| LABVIEW Professional 2018 | National Instruments | 86728 | |
| MadMotor Positioning system and controllers | Mad City Labs | W6-4 | |
| Methanol, LC/MS Grade | Fisher Scientific | 01-213-100 | any brand is sufficient |
| Microcentrifuge | Thermo Scientific | H301-T Unit-BL-PLUS | |
| N,N′-Dimethylthiourea (DMTU) | ACROS Organics | ||
| Nanopositioinging stage | Mad City Labs | ||
| N-tert-Butyl-α-phenylnitrone (PBN) | ACROS Organics | ||
| OKO Touch Monitoring System | OKOlabs | ||
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | 7Z02936 | |
| PE50-C pyroelectric energy meter | Ophir Optronics | ||
| Penicillin-streptomycin | Corning | ||
| Pierce Quantitative Colorimetric Peptide Assay | Thermo Scientific | ||
| Pierce Rapid Gold BCA Protein Assay Kit | Thermo Scientific | ||
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 75002436 | |
| Pierce Universal Nuclease for Cell Lysis | Fisher Scientific | 06-666A | |
| pressure gauge | OKOlabs | OKO-AIR-PUMP-BL | |
| PTFE filter | OKOlabs | CO2-UNIT-BL | |
| Six 33 mm PLA filament rings | (Innovation Space University of Maryland Baltimore Campus Library) | ||
| sterile incubator | OKOlabs | ||
| temperature unit | OKOlabs | OKO-TOUCH | |
| Thermo Scientific Pierce RIPA Buffer | Fisher Scientific | A117-50 | |
| TiNKERcad | (Innovation Space University of Maryland Baltimore Campus Library) | ||
| Tris Base | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| tubing (Tygon 3.18 and 1.59 ID) | Cole-Palmer | 177350250 | |
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | A454SK-4 | 4 L quantity is not necessary |
| Water, LC/MS Grade | Fisher Scientific | 88702 | |
| 90058 | |||
| KM200 | |||
| 186007496 |
Referencias
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins (FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. 294 (32), 11969-11979 (2019).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355-4454 (2019).
- Li, J., Chen, G. The use of fast photochemical oxidation of proteins coupled with mass spectrometry in protein therapeutics discovery and development. Drug Discovery Today. 24 (3), 829-883 (2019).
- Zhao, B., et al. Covalent Labeling with an α,β-Unsaturated Carbonyl Scaffold for Studying Protein Structure and Interactions by Mass Spectrometry. Analytical Chemistry. 92 (9), 6637-6644 (2020).
- Hambly, D. M., Gross, M. L. Laser Flash Photolysis of Hydrogen Peroxide to Oxidize Protein Solvent-Accessible Residues on the Microsecond Timescale. Journal of the American Society for Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Chea, E. E., Jones, L. M. Modifications generated by fast photochemical oxidation of proteins reflect the native conformations of proteins. Protein Science. 27 (6), 1047-1056 (2018).
- Hambly, D., Gross, M. Laser flash photochemical oxidation to locate heme binding and conformational changes in myoglobin. International Journal of Mass Spectrometry. 259 (1), 124-129 (2007).
- Yan, Y. Fast photochemical oxidation of proteins (FPOP) maps the epitope of EGFR binding to adnectin. Journal of the American Society for Mass Spectrometry. 25 (12), 2084-2092 (2014).
- Zhang, Y., Wecksler, A. T., Molina, P., Deperalta, G., Gross, M. L. Mapping the Binding Interface of VEGF and a Monoclonal Antibody Fab-1 Fragment with Fast Photochemical Oxidation of Proteins (FPOP) and Mass Spectrometry. Journal of the American Society for Mass Spectrometry. 28 (5), 850-858 (2017).
- Espino, J. A., Mali, V. S., Jones, L. M. In Cell Footprinting Coupled with Mass Spectrometry for the Structural Analysis of Proteins in Live Cells. Analytical Chemistry. 87 (15), 7971-7978 (2015).
- Rinas, A., Mali, V. S., Espino, J. A., Jones, L. M. Development of a Microflow System for In-Cell Footprinting Coupled with Mass Spectrometry. Analytical Chemistry. 88 (20), 10052-10058 (2016).
- Johnson, D. T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L. M. Implementing In-Cell Fast Photochemical Oxidation of Proteins in a Platform Incubator with a Movable XY Stage. Analytical Chemistry. 92 (2), 1691-1696 (2019).
- Bangel-Ruland, N., et al. Cystic fibrosis transmembrane conductance regulator-mRNA delivery: a novel alternative for cystic fibrosis gene therapy. The Journal of Gene Medicine. 15 (11-12), 414-426 (2013).
- Gau, B. C., Chen, H., Zhang, Y., Gross, M. L. Sulfate Radical Anion as a New Reagent for Fast Photochemical Oxidation of Proteins. Analytical Chemistry. 82 (18), 7821-7827 (2010).
- Xu, G. C., et al. Hydroxyl Radical-Mediated Modification of Proteins as Probes for StructuralProteomics. Chemical Reviews. American Chemical Society. 107 (8), 3514-3543 (2007).
- Espino, J. A., Jones, L. M. Illuminating Biological Interactions with In Vivo Protein Footprinting. Analytical Chemistry. 9 (10), 6577-6584 (2019).
- Gau, B. C., Sharp, J. S., Rempel, D. L., Gross, M. L. Fast Photochemical Oxidation of Protein Footprints Faster than Protein Unfolding. Analytical Chemistry. 81 (16), 6563-6571 (2009).
- Kaur, U., Johnson, D. T., Jones, L. M. Validation of the Applicability of In-Cell Fast Photochemical Oxidation of Proteins across Multiple Eukaryotic Cell Lines. Journal of the American Society for Mass Spectrometry. 31 (7), 1372-1379 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados