Method Article
Incubadora de plataformas com estágio XY móvel: uma nova plataforma para implementar oxidação fotoquímica rápida em células de proteínas
Neste Artigo
Resumo
Uma nova plataforma estática é usada para caracterizar a estrutura proteica e locais de interação no ambiente celular nativo utilizando uma técnica de pegada de proteína chamada oxidação fotoquímica rápida em células de proteínas (IC-FPOP).
Resumo
A oxidação fotoquímica rápida de proteínas (FPOP) juntamente com espectrometria de massa (MS) tornou-se uma ferramenta inestimável na proteômica estrutural para interrogar interações proteicas, estrutura e dinâmicas conformais proteicas em função da acessibilidade de solventes. Nos últimos anos, o escopo do FPOP, uma técnica de impressão de pé de proteína radical hidroxíl (HRPF), foi expandido para rotulagem de proteínas em culturas de células vivas, fornecendo os meios para estudar interações proteicas no ambiente celular complicado. Modificações de proteínas em células podem fornecer insights sobre ligantes e mudanças estruturais induzidas ou alterações conformais que acompanham a formação do complexo proteico, tudo dentro do contexto celular. A pegada proteica foi realizada empregando um sistema habitual baseado em fluxo e um laser excimer KrF de 248 nm para produzir radicais hidroxil através da fotolise do peróxido de hidrogênio, exigindo 20 minutos de análise para uma amostra celular. Para facilitar os experimentos FPOP resolvidos pelo tempo, o uso de uma nova plataforma IC-FPOP baseada em placas de 6 poços foi pioneiro. No sistema atual, um único pulso laser irradia um poço inteiro, que trunca o período experimental FPOP resultando em 20 segundos de tempo de análise, uma redução de 60 vezes. Esse tempo de análise muito reduzido torna possível pesquisar mecanismos celulares como cascatas de sinalização bioquímica, dobramento de proteínas e experimentos diferenciais (ou seja, livre de drogas versus drogas) de forma dependente do tempo. Esta nova instrumentação, intitulada Incubadora de Plataforma com Estágio Móvel XY (PIXY), permite que o usuário realize a cultura celular e o IC-FPOP diretamente no banco óptico usando uma incubadora de plataforma com controle de temperatura, CO2 e umidade. A plataforma também inclui um estágio de posicionamento, bombas peristálticas e óptica espelhada para orientação de raios laser. As condições de IC-FPOP, como configuração óptica, taxas de fluxo, transfesões transitórias e concentração de H2O2 em PIXY foram otimizadas e revisadas por pares. A automação de todos os componentes do sistema reduzirá a manipulação humana e aumentará o throughput.
Introdução
Técnicas de pegada de proteínas podem revelar informações profundas sobre a organização de proteínas. Essas técnicas essenciais baseadas em biologia estrutural são um componente da caixa de ferramentas de espectrometria de massa. Estes métodos sondam a estrutura de ordem superior proteica (HOS) e a sinergia através da rotulagem covalente1,2,3,4. A oxidação fotoquímica rápida das proteínas (FPOP) emprega radicais hidroxil para modificar oxidativamente cadeias laterais acessíveis de solventes dos aminoácidos5,6 (Tabela 1). O método utiliza um laser excimer a 248 nm para fotolise de peróxido de hidrogênio (H2O2) para gerar radicais hidroxil. Teoricamente, 19 dos 20 aminoácidos podem ser oxidativamente modificados, sendo Gly a única exceção. No entanto, devido às diferentes taxas de reatividade de aminoácidos com radicais hidroxílicos, a modificação de apenas um subconjunto destes tem sido observada experimentalmente. Ainda assim, o método tem potencial para análise sobre o comprimento de uma sequência proteica5. O FPOP modifica proteínas na escala de tempo microssegundo, tornando-a útil no estudo de interações fracas com taxas de alta velocidade. A acessibilidade dos solventes muda sobre a ligação de ligas ou uma mudança na conformação proteica, assim, o poder do método reside na comparação do padrão de rotulagem de uma proteína em vários estados (ou seja, livre de ligantes em comparação com ligantes). Como resultado, o FPOP tem sido bem sucedido na identificação de locais de interação proteína-proteína e proteína-ligantes e regiões de mudança conformacional7,8,9,10. O método FPOP foi estendido desde o estudo de sistemas proteicos purificados até a análise em células. O FPOP (IC-FPOP) pode modificar oxidativamente mais de mil proteínas nas células para fornecer informações estruturais em todo o proteome11,12. A plataforma IC-FPOP convencional utiliza um sistema de fluxo para fluir células um único arquivo após o raio laser. O desenvolvimento deste sistema permitiu que células individuais tivessem exposição igual à irradiação a laser. Isso levou a um aumento de 13 vezes no número de proteínas oxidativamente rotuladas12. No entanto, uma limitação do sistema de fluxo é o comprimento de um único experimento amostral que consiste em um intervalo de irradiação de 10 minutos durante o qual ocorre a modificação e um ciclo adicional de lavagem de 10 minutos. As restrições de tempo do IC-FPOP tornam-no inadequado para estudar intermediários dobráveis de proteínas de curta duração ou mudanças que existem entre as redes de interação em cascatas de sinalização bioquímica. Essa limitação temporal inspirou o design de uma nova plataforma IC-FPOP equipada com maior rendimento.
Para medir com precisão a estrutura de ordem proteica no ambiente celular nativo, o novo design permite que a cultura celular seja realizada diretamente na plataforma laser, o que permite que o IC-FPOP seja de alta produtividade. Esta configuração também permite perturbações minimizadas ao ambiente celular, em contraste com o IC-FPOP usando fluxo onde as células aderentes devem ser removidas do substrato. A nova plataforma permite que o IC-FPOP ocorra em um sistema de incubação estéril usando uma câmara superior de estágio controlada por CO2 e temperatura, utilizando óptica espelhada configurada para orientação de feixe de laser, um sistema de posicionamento para movimento XY e bombas peristálticas para troca química. A nova plataforma de realização do IC-FPOP é intitulada Incubadora de Plataformas com Estágio Móvel XY (PIXY) (Figura 1). Em PIXY, o IC-FPOP é realizado em células humanas cultivadas em placas de seis poços dentro da câmara de incubadora de plataforma. Para esta configuração, o raio laser é refletido para baixo na placa usando espelhos compatíveis com feixe como um estágio de posicionamento que mantém a incubadora é movido, no plano XY, de modo que o raio laser está estrategicamente alinhado para irradiar apenas um poço de cada vez. Estudos de validação mostram que o IC-FPOP pode ser realizado mais rápido em PIXY do que no sistema de fluxo e leva ao aumento de modificações de aminoácidos por proteína. O desenvolvimento desta nova plataforma IC-FPOP exporá sobre o conhecimento que pode ser adquirido a partir de experimentos celulares13.
Protocolo
1. Montagem da Incubadora de Plataformas com estágio XY móvel
NOTA: A nova plataforma inclui o sistema de incubação, o estágio de posicionamento e os controladores, as bombas peristálticas, o laser excimer KrF de 248 nm e os espelhos ópticos montados em uma placa óptica imperial.
- Montar o sistema de incubação: a unidade de temperatura, a unidade de dióxido de carbono, umidificador, a bomba de ar e o sistema de monitoramento do toque(Figura 2A-E).
NOTA: Instruções detalhadas de montagem são fornecidas pelo fabricante. O sistema de incubação deve estabilizar para 5% de CO2, 37 °C e 85% de umidade antes de cultivar células dentro da incubadora. Esses parâmetros podem depender da linha celular. - Monte a incubadora personalizada de placas de seis poços, o estágio de unidade de nanoposicionamento e o estágio de unidade XY. O primeiro parafusos para o segundo, respectivamente.
NOTA: Instruções detalhadas de montagem fornecidas pelo fabricante. - Conecte as quatro bombas peristálticas em uma sequência de "corrente de margarida" através de cabos RS-232, com um cabo RS-232 a USB conectado ao computador controlador. Conecte tubos de polímero de 3,18 mm de ID (por exemplo, Tygon) a cada canal em cada rolo de canal de bomba.
NOTA: Cada bomba tem quatro rolos. A direção e a taxa de fluxo dos rolos são manipuladas manualmente. - Insira conectores de 1/16" x 1/8" no final de cada tubo de polímero de 3,18 mm de ID. Insira a extremidade de 1/16'' do conector em tubos de polímero de 1,59 80m e, em seguida, insira a tubulação de polímero na incubadora através de portas personalizadas. Coloque a outra extremidade do tubo na solução que será infundida durante a experimentação IC-FPOP.
NOTA: Para as linhas de perfusão, a incubadora possui 36 portas personalizadas para linhas de tubulação ao redor da periferia para acomodar todos os reagentes utilizados. - Parafuso um espelho de 50 mm, 248 nm, 45° excimer laser espelho dentro de um suporte de espelho cinemático para ø2 " óptica na prancha 10-11 pontos de grade a partir da abertura laser de 248 nm. Coloque o segundo espelho em um ângulo de 90° para o primeiro do outro lado da incubadora. Anguore o segundo espelho para baixo em aproximadamente 45° para orientação do feixe de laser para a incubadora (Figura 2F-J).
2. Sincronização e automação inicial do sistema via software de integração
- Instale a versão mais recente do software de integração necessário para controlar os drivers e bombas.
- Referindo-se ao manual da bomba, renomeie as bombas começando com '5' e aumentando de valor. Os comandos podem ser enviados para o sistema de bomba usando o subse programa controle manual no software de integração.
NOTA: A configuração de uma bomba para o modo de canal altera automaticamente a convenção de nomeação da bomba para o conjunto, 1, 2, 3 e 4, correspondente aos quatro canais de bomba. - Abra o programa de software de integração para a automação da incubadora da plataforma.
- Escolha o nome apropriado do dispositivo de comport USB (por exemplo, COM4) correspondente ao cabo USB do sistema de bomba a partir do menu suspenso de conexão rotulado Comport para bombas.
- Clique no botão Script Builder para editar/criar um script para a plataforma automatizada de incubadora de plataformas. Clique em Salvar script para salvar a sequência como um arquivo de texto ( Figura3A).
NOTA: O construtor de scripts tem uma interface de usuário para definir o script com definições de número de bomba, direção, taxa, volume, diâmetro do tubo, modo de canal e atraso cronometrado. - Se o modo de canal for desejado na confecção do script, um passo no script deve ser dedicado à alteração da bomba dentro e fora do modo de canal, e as seguintes etapas correspondentes aos canais de bomba devem ser rotuladas como bombas 1-4.
NOTA: Isso garante que nenhuma bomba pode estar simultaneamente no modo de canal durante um script de incubadora de plataforma e que cada bomba seja trocada de volta para o modo legado depois que os comandos foram enviados para os canais necessários. - Clique no botão Read Script e escolha o arquivo de script apropriado desejado para a operação da incubadora da plataforma.
- Mude o botão Executar sequência para a posição ON (verde) para executar o script e, em seguida, clique no botão START (Figura 3B) .
3. Cultivar células na incubadora da plataforma
NOTA: As células devem ser colocadas na incubadora da plataforma em condições estéreis em um capô de cultura celular no dia anterior à experimentação.
- Desaparafusar a incubadora da plataforma do estágio de nanoposicionamento e desconectar as linhas de temperatura, gás e umidificador. Depois de pulverizar a incubadora com 70% de etanol, coloque-a em uma capa de cultura celular.
- Cultivar células em um T-175 para cerca de 80-90% de confluência antes da transferência.
NOTA: O termo confluência é usado como medida do número/cobertura das células em um prato de cultura celular ou um frasco. - Remova a mídia e enxágue com tampão.
- Desapegar células usando trypsin-EDTA usando o protocolo do fabricante.
- Resuspend em 8 mL de mídia e conte as células.
- Fibronectina de sementes/colágeno14 placas revestidas de seis poços com aproximadamente 90-95 μL de células resuspended em cada poço. Este volume será apropriado para atingir 80-90% de confluência (~1,2 milhões de células) em cada poço no dia seguinte.
NOTA: As placas de seis poços devem ser revestidas com colágeno antes da semeadura. Os reagentes utilizados durante o IC-FPOP são infundidos com taxas de fluxo rápido. As placas revestidas garantem que as células não sejam prematuramente separadas devido à infusão de reagentes. - Coloque a placa semeada na incubadora da plataforma e cubra com a tampa de quartzo.
NOTA: Durante o projeto e desenvolvimento, uma tampa de incubadora de vidro foi alterada para quartzo, por isso é compatível com a luz laser ultravioleta. - Substitua e proteja a incubadora da plataforma de volta no estágio de unidade de nanoposicionamento. Reconecte as linhas de temperatura, gás e umidificador.
- Deixe as células crescerem até a confluência da noite para o dia.
NOTA: As seguintes etapas de cultura celular são opcionais e destinam-se a experimentos nos quais as células humanas são transitoriamente transfetadas. Essas etapas avaliam a eficiência da transfecção em condições de cultura celular. - Transfeito o plasmídeo contendo o gene para a proteína quimrica GCaMP2 em células HEK 293 usando um kit comercial de transfecção cataônica-lipídica.
- Realizar transfecções transitórias de GCaMP2 em células HEK293T em duas placas de seis poços.
- Incubar uma placa de seis poços na incubadora da plataforma, e a segunda placa de seis poços em uma incubadora padrão de CO2.
- Compare a eficiência de transfecção dentro de cada placa por imagens de fluorescência usando um microscópio de fluorescência.
4. Faça tampão de saciecia e H2O2
- Faça 100 mL de tampão de sufoco contendo 125 mM N-tert-Butyl-α-fenilnitrone (PBN) e 125 mM N, N'-Dimethylthiourea (DMTU).
NOTA: DMTU e PBN são carniceiros radicais livres e são permeáveis por células. - Diluir H2O2 a 200 mM. Cada amostra requer 6 mL de H2O2 para imergir totalmente a camada de células na parte inferior de cada poço.
NOTA: O tampão de sumosidade é feito no dia anterior e armazenado a 4 °C durante a noite, protegido contra a luz. Diluir o H2O2 no dia do experimento.
5. Configure a incubadora de plataforma para IC-FPOP
NOTA: A incubadora da plataforma deve ser montada em condições estéreis. Monte a incubadora em uma capa de cultura celular estéril.
- Desaparafusar a incubadora da plataforma do estágio de posicionamento e desconectar as linhas de temperatura, gás e umidificador.
- Depois de pulverizar a incubadora com 70% de etanol, coloque-a na capa de cultura celular.
- Remova a placa de seis poços da incubadora da plataforma, proteja a tampa original da placa e confirme a confluência celular usando um microscópio.
- Depois que a confluência celular tiver sido confirmada, substitua a placa de seis poços por células confluentes de volta na incubadora da plataforma dentro da capa de cultura celular.
- Insira três tubos de polímero de 1,59 80m em cada poço através das portas embarcadas. Lave os tubos nas paredes do poço e segure com anéis impressos 3D personalizados.
NOTA: Seis anéis impressos pla 3D de 33 mm foram projetados sob medida para fixar o tubo nas paredes da placa sem perturbar as células ou ficar no caminho do pulso laser. - Cubra a incubadora da plataforma com sua tampa de quartzo. Substitua e proteja a incubadora superior do estágio no sistema de posicionamento. Reconecte as linhas de temperatura, gás e umidificador.
- Conecte tubos de polímero de 1,59 ID ao final de 1/16" dos conectores de 1/16x1/8".
6. Realizando IC-FPOP na incubadora da plataforma
- Prepare um script de software de integração para retirada da bomba. Use uma bomba peristáltica (Bomba 8) para remover completamente a mídia celular de todos os seis poços.
- Solventes prime em cada canal das outras três bombas peristálticas (Bombas 5-7). Infundir H2O2 e saciar o tampão em seus respectivos tubos alternados até que os reagentes cheguem às portas da incubadora.
- Confirme se o raio laser foi angulado corretamente pelos espelhos e atinge a incubadora desinibida.
NOTA: Os óculos de segurança a laser devem ser usados sempre que o laser estiver em uso. Não irradie prematuramente as células durante o processo de angling/alinhamento. Use papelão para cobrir completamente a tampa de quartzo ao alinhar o feixe. Além disso, use um contorno impresso em papel branco de uma placa de seis poços para confirmar ainda mais que o raio laser está atingindo o centro de cada poço. Use a configuração de pulso contínuo na menor frequência e energia para alinhamento. - Verifique a energia do laser usando um sensor externo. Use um único pulso laser de 160 mJ a 50 Hz e 27 kV.
NOTA: É necessário um temporizador para as seguintes etapas. - Prepare o script do software de integração para infusão da bomba após confirmar o alinhamento do feixe.
- Inicie o temporizador e pressione o botão Iniciar no script da bomba de software de integração ao mesmo tempo.
- Infundir 200 mM H2O2 a 35 mL/min no primeiro poço (marca de 6-10 segundos no temporizador).
NOTA: Há um atraso de cinco segundos antes que uma bomba comece a infundir. Também leva o laser sete segundos antes do pulso ser acionado. O pulso laser deve vir imediatamente após a infusão H2O2. - Pressione o botão Iniciar no software laser na marca de 5 segundos para acionar o pulso na marca de 11 segundos no temporizador.
- Infundir a solução de saciar 125 mM a 35 mL/min no primeiro poço imediatamente após o pulso do laser. Mova manualmente o estágio de posicionamento para alinhar o próximo poço com o raio laser.
- Repita as etapas acima 6.5-6.9 até que cada poço tenha sido processado.
NOTA: O IC-POP é realizado em triplicado técnico de três amostras de laser e três não-laser. Uma placa de seis poços processada serve como uma réplica biológica. - Em uma capa de cultura celular, use um raspador de células para transferir as células de cada poço para tubos cônicos individuais de 15 mL. Centrífugas a 1.200 x g por 5 minutos.
- Descarte as células supernascidas e resuspendas em 100 μL de tampão de lise RIPA.
- Transfira células para tubos individuais de polipropileno de 1,2 mL.
- Flash congele todas as amostras em nitrogênio líquido e coloque em um congelador de -80 °C até usar.
NOTA: O protocolo pode ser pausado aqui.
7. Extração de proteínas, purificação e proteólise
- Descongele as amostras e aqueça a 95 °C em um bloco de calor por 5 minutos.
- Depois do aquecimento, esfrie no gelo por 5 minutos.
- Adicione 25 unidades de nuclease ao lisetê celular para degradar o DNA e o RNA de um único fio, de dois fios, lineares e circulares e RNA e incubar na sala temperado por 15 minutos.
- Centrífuga amostras a 16.000 x g por 10 minutos a 4 °C.
- Pegue o supernatante e transfira para um tubo de polipropileno limpo.
- Verifique a concentração de proteínas usando um ensaio de proteína colorimétrica.
- Transfira 100 μg de amostra para um tubo de polipropileno limpo e leve a 100 μL com tampão de lise celular.
- Reduza as amostras com dithiothreitol de 10 mM (DTT) a 50 °C por 45 minutos.
- Resfrie amostras em temperatura ambiente por 10 minutos.
- Alquila com iodoacetamida de 50 mM (IAA) em temperatura ambiente por 20 minutos.
NOTA: Proteja o IAA contra a luz - Adicione 460 μL de acetona pré-refrigerada (-20 °C). Amostras de vórtice e coloque a -20 °C durante a noite.
NOTA: O protocolo pode ser pausado aqui. - No dia seguinte, amostras de centrífugas a 16.000 x g durante 10 minutos a 4 °C.
- Remova a acetona sem interromper a pelota de proteína.
- Adicione 50 μL de acetona pré-refrigerada (-20 °C). Amostras de vórtice para misturar e centrífugas a 16.000 x g por mais 5 minutos a 4 °C.
- Remova a acetona e deixe as amostras secarem por 2-3 minutos.
- Pelota de proteína resuspend com 10 mM Tris tampão pH 8.
- Resuspend MS grau trypsin (20 μg estoque) em 40 μL de 10 mM Tris tampão pH 8 e adicionar 2,5 μg de trippsina a cada amostra.
- Incubar amostras a 37 °C durante a noite.
NOTA: O protocolo pode ser pausado aqui. - Avalie a concentração de peptídeos usando um ensaio de peptídeo colorimétrico.
- Sacie as amostras com 5% de ácido fórmico.
- Transfira 10 μg de amostra para um tubo limpo de polipropileno.
- Seque a amostra usando uma centrífuga de vácuo e resuspend com 20 μL de MS grau 0,1% ácido fórmico (FA) na água.
- Transfira cada amostra para limpar frascos de autosampler com tampas pré-fenda.
8. Espectrometria de Massa Líquida de alto desempenho -Tandem (LC-MS/MS)
- Para localizar as modificações do FPOP, analise o lisesato celular digerido utilizando a análise de LC-MS/MS.
- Use fases móveis de 0,1% FA na água (A) e 0,1% FA em acetonitrilo (ACN) (B).
- Carregue 0,5 μg de amostra em uma coluna de trapping de 180 μm x 20 mm C18 (5 μm e 100 Å) e lave a coluna com 99% (A) e 1% (B) por 15 minutos.
- Utilizando uma coluna analítica de 75 μm x 30 cm C18 (5 μm e 125 Å), elute e peptídeos digeridos separados com uma taxa de fluxo de 0,300 μL/min por 120 minutos.
- Executar gradiente LC da seguinte forma: 0-1 min, 3% solvente B; 2-100 min, 10-45% B;100-110 min, 45-100% B; 110-115 min, 100% B.
- Recondicionar a coluna a 3% (B) de 115-116 minutos e manter em 3% (B) de 116-120 minutos.
- Analise peptídeos elucidos no modo íon positivo com ionização de nano eletrospray.
- Adquira espectros MS1 ao longo de uma faixa de varredura m/z de 375-1500 com uma resolução de 60.000.
- Defina a meta de controle automático de ganho (AGC) para 5,0e5 com um tempo máximo de injeção de 50 ms e 5,0e4 limiar de intensidade.
- Isolar íons precursores com carga afirma 2-6 através de aquisição dependente de dados (DDA) com uma janela de isolamento de 1,2 m/z e um tempo de ciclo de 4 segundos.
- Sujeito ms2 íons à dissociação collisional de alta energia (HCD) (32% energia normalizada).
- Exclua peptídeos após 1 aquisição de MS/MS por 60 segundos.
- Defina a resolução MS/MS para 15.000 com uma meta AGC de 5,0e4 e um tempo máximo de injeção de 35 ms.
9. Descobridor/processamento de dados proteome
- Pesquise arquivos de dados brutos em tandem em software de análise de proteômica de baixo para cima disponível contra um banco de dados de proteínas Homo sapiens relevante e enzima digerir.
- Defina os parâmetros de busca de análise de proteínas.
- Defina tolerância de fragmentos a 0,02 Da e tolerância ao íon-pai a 10 ppm.
- Defina a especificidade da enzima para trypsin e permita um decote perdido.
- Definir faixa de massa para 375-1500 m/z.
- Estabeleça a confiança do peptídeo em 95% (média) e a confiança dos resíduos em 99% (alta).
- Aceite proteínas se pelo menos dois peptídeos distintos forem identificados com o filtro de taxa de descoberta de 5%.
- Defina a carbamidometilação como uma modificação estática e todas as modificações de cadeia lateral radical hidroxil conhecidas15,16 como modificações dinâmicas.
- Uma vez que os arquivos são terminados de pesquisa, sequência de exportação, locais de modificação, adesão de proteínas, arquivo de espectro, abundância precursora e informações de tempo de retenção para um banco de dados eletrônico.
- Calcule a extensão da modificação por peptídeo ou resíduo desta equação:
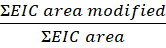
NOTA: A área EIC modificada é a área cromatográfica de íon extraída (EIC) do peptídeo ou resíduo com modificação oxidativa, e a área EIC é a área total do mesmo peptídeo ou resíduo com e sem a modificação oxidativa. Com o tempo, a proteína na presença de peróxido de hidrogênio oxidará, resultando em oxidações de fundo. Para calcular a extensão da modificação, a área do peptídeo modificado é dividida pela área total. Uma amostra de controle não irradiada explica a oxidação de fundo. A oxidação de fundo da amostra de controle é subtraída da amostra tratada a laser para identificar uma modificação FPOP.
Resultados
Para confirmar que as condições da incubadora da plataforma são suficientes para a cultura celular na plataforma laser, o GCaMP2 foi transitoriamente transfectado em HEK293T e a eficiência de transfecção para ambas as placas foi avaliada via imagem de fluorescência(Figura 4A). GCaMP2 é uma proteína fluorescente de detecção de cálcio usada como um indicador de cálcio intracelular geneticamente codificado. É uma fusão de proteína fluorescente verde (GFP) e a proteína de ligação de cálcio, calmodulin. Um ensaio de luciferase foi realizado em células HEK 293 transfectadas com prl-TK plasmid, a fim de quantificar a eficiência da transfecção(Figura 4B). Esses resultados mostram que a incubadora da plataforma excedeu o desempenho da incubadora padrão, com um aumento de 1,13 vezes na eficiência da transfecção, fornecendo um benchmark quantitativo para o ambiente ideal de cultura celular.
As modificações do FPOP nas células HEK293T rotuladas no sistema de fluxo foram comparadas às rotuladas na incubadora de plataformas e mostraram que a incubadora de plataforma supera o sistema de fluxo tanto no número de proteínas modificadas (Figura 5) quanto na cobertura total de FPOP nessas proteínas. O número de proteínas modificadas FPOP adquiridas na incubadora da plataforma foi de aproximadamente 1051, 2,2 vezes mais do que as adquiridas em um experimento típico. Modificações foram combinadas entre duas réplicas biológicas para cada experimento. Além disso, o PIXY fornece maior rendimento.
Para demonstrar a vantagem de uma maior cobertura de modificação em uma proteína, as modificações ic-FPOP foram localizadas no nível do peptídeo e a extensão da modificação foi quantificada para distinguir diferenças nos desfechos entre os sistemas de actina, uma proteína de 375 aminoácidos. No sistema de fluxo, foram detectados dois peptídeos modificados, fornecendo informações estruturais limitadas(Figura 6A). No entanto, cinco peptídeos modificados que abrangem a sequência de actina foram detectados na incubadora da plataforma. Espectros de massa tandem indicam que o resíduo Pro322 foi modificado e detectado em cada experimento (Figura 6B). Os cinco peptídeos modificados nas amostras da incubadora da plataforma continham doze resíduos modificados, enquanto apenas quatro resíduos foram modificados com o sistema de fluxo(Figura 6C). O aumento da cobertura de oxidação fornece mais informações estruturais em toda a proteína.
Espino et al. demonstraram a capacidade do FPOP a ser realizado in vivo (IV-FPOP) dentro de C. elegans, um modelo de verme para estados de doença humana17. Enquanto o IV-FPOP também é realizado através de um sistema de fluxo, o sistema PIXY foi testado para compatibilidade com os worms. Aproximadamente 10.000 vermes foram incubados em cada poço na incubadora da plataforma a 20 °C. A análise LC-MS/MS revelou que 792 proteínas foram modificadas pelo IV-FPOP na incubadora da plataforma em comparação com as 545 proteínas modificadas com o sistema de fluxo(Figura 7). Esses resultados demonstram que, além da cultura celular 2D, essa nova metodologia também é compatível com o estudo de outros sistemas biológicos, como o C. elegans.
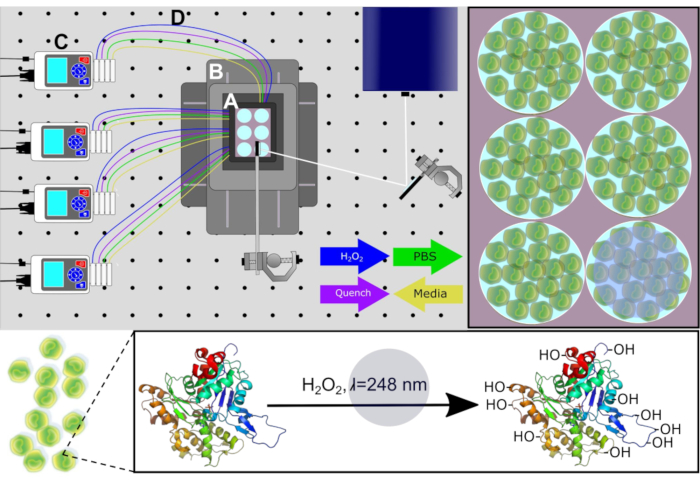
Figura 1. Esquema do Sistema PIXY. Componentes do sistema: (A) incubadora de topo de palco, (B) sistema de posicionamento ,(C) bombas peristálticas e(D) linhas de perfusão. A mídia de cultura celular é removida de cada poço através de bombas antes de H2O2 e as soluções de saciamento são infundidas em pontos de tempo calculados. Caminho laser para irradiação exibido em branco. Reimpresso com permissão de Johnson, D.T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidação fotoquímica rápida em células de proteínas em uma Incubadora de Plataforma com um Estágio XY Móvel. Química Analítica, 92(2), 1691-1696 2019. Copyright 2020 American Chemical Society. Clique aqui para ver uma versão maior desta figura.

Figura 2. Sistema PIXY totalmente montado. (A) Sistema de monitoramento de toque,(B) unidade de dióxido de carbono,(C) unidade detemperatura, (D) bomba de ar, (E) umidificador óptico,(F) incubadora de plataforma,(H) estágio de posicionamento, (I) 248nm KrF excimer laser, e (J) bombas peristálticas. Clique aqui para ver uma versão maior desta figura.
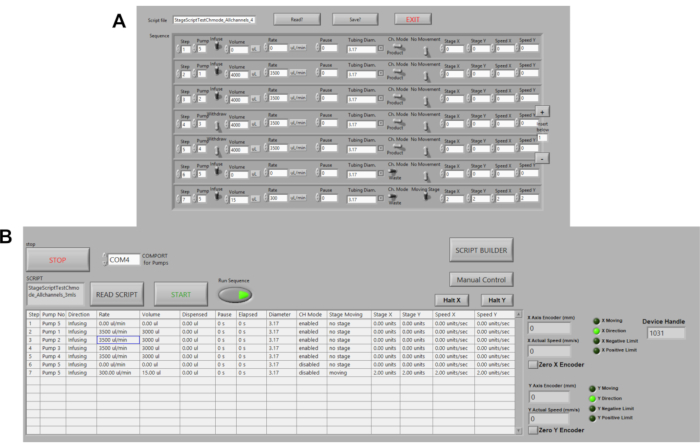
Figura 3. Automação de Bombas Peristáticas. (A) Exemplo de script de comando no LABVIEW. As opções de comando incluem volume, taxa de fluxo, pausas, direção de fluxo. Velocidade, distância do estágio e localização estão sendo automatizados. (B) Leitor de script no LABVIEW. Aqui, os scripts de comando são carregados, em seguida, executar sequência e START são pressionados para iniciar bombas. Clique aqui para ver uma versão maior desta figura.
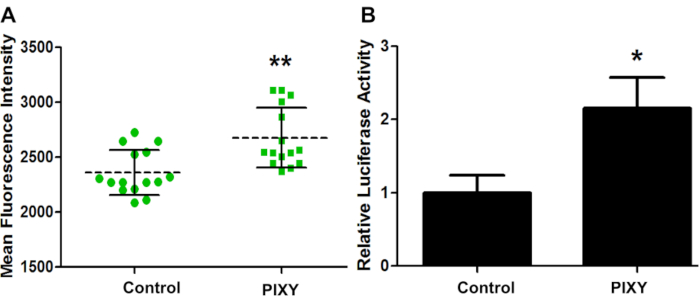
Figura 4. Eficiência de transfecção celular HEK. (A) Intensidade fluorescente média da comparação de transfecção GCaMP2 entre a incubadora padrão (Controle) e a incubadora de topo de palco (PIXY). Pontos e quadrados representam cada ponto em um poço onde uma medida foi tomada. (B) Eficiência de transfecção quantitada e validada com um plasmídeo vetorial diferente, pRL-TK. Valor P< 0,005. Reimpresso com permissão de Johnson, D.T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidação fotoquímica rápida em células de proteínas em uma Incubadora de Plataforma com um Estágio XY Móvel. Química Analítica, 92(2), 1691-1696 2019. Copyright 2020 American Chemical Society. Clique aqui para ver uma versão maior desta figura.
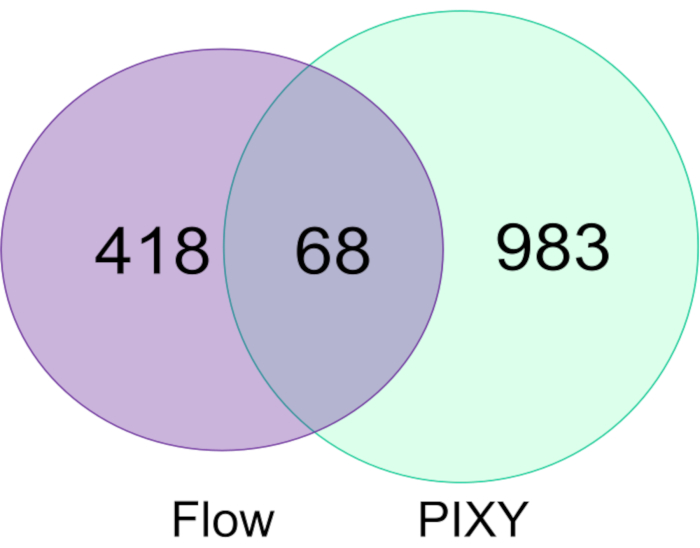
Figura 5. Comparação de proteínas modificadas no sistema de fluxo celular único e PIXY. Diagrama de venn de proteínas modificadas usando no sistema de fluxo (roxo) e em PIXY (verde). Reimpresso com permissão de Johnson, D.T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidação fotoquímica rápida em células de proteínas em uma Incubadora de Plataforma com um Estágio XY Móvel. Química Analítica, 92(2), 1691-1696 2019. Copyright 2020 American Chemical Society. Clique aqui para ver uma versão maior desta figura.
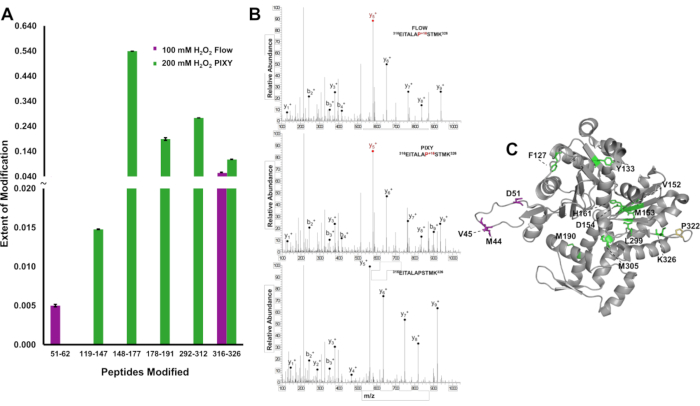
Figura 6. Localização das modificações ic-FPOP. Comparação das modificações IC-FPOP entre os sistemas. (A) Gráfico de barras de peptídeos oxidativamente modificados dentro de actin do sistema de fluxo (roxo) vs incubadora de plataforma (verde). (B) Tandem MS espectro de actina (peptídeo 316-326) com prolina modificada em ambos os sistemas e peptídeo de actina não modificado (C) FPOP resíduos modificados de actina (PDB: 6ZXJ, cadeia A 11 resíduos modificados na incubadora de plataforma (verde), 3 resíduos modificados no sistema de fluxo (roxo), 1 resíduos modificados sobrepostos (amarelo). Reimpresso com permissão de Johnson, D.T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidação fotoquímica rápida em células de proteínas em uma Incubadora de Plataforma com um Estágio XY Móvel. Química Analítica, 92(2), 1691-1696 2019. Copyright 2020 American Chemical Society. Clique aqui para ver uma versão maior desta figura.
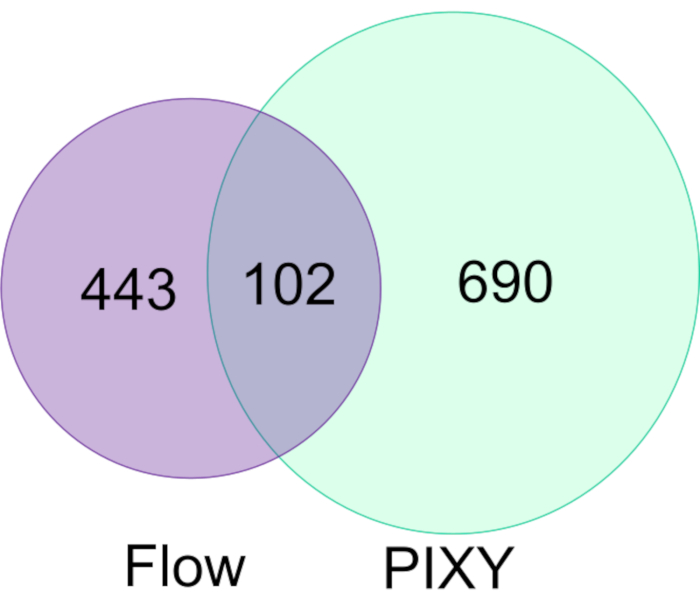
Figura 7. Comparação de proteínas modificadas FPOP em C. elegans por flow vs PIXY. Há um aumento de 1,5 vezes nas proteínas oxidativamente modificadas usando PIXY quando comparado com o sistema de fluxo. Reimpresso com permissão de Johnson, D.T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L.M., Implementando oxidação fotoquímica rápida em células de proteínas em uma Incubadora de Plataforma com um Estágio XY Móvel. Química Analítica, 92(2), 1691-1696 2019. Copyright 2020 American Chemical Society. Clique aqui para ver uma versão maior desta figura.
Mesa 1. Distribuição de modificação do fluxo de trabalho e mudanças em massa(Da) . Por favorclique aqui para baixar esta tabela.
Discussão
As proteínas realizam grande parte do trabalho em células vivas. Dada essa importância, dados detalhados sobre a função proteica e a estrutura de ordem superior (HOS) no ambiente celular são necessários para aprofundar a compreensão das complexidades em complexos maiores e reações enzimáticas nas células em oposição aos sistemas purificados. Para isso, foi adotado um método de impressão de pé de proteína radical hidroxíl (HRFP) intitulado Oxidação Fotoquímica Rápida de Proteínas (IC-FPOP). A maioria dos estudos de FPOP tem sido feita in vitro em sistemas de proteínas relativamente puras, o que contrasta significativamente com o ambiente molecular lotado que afeta interações vinculativas e dinâmicas conformais proteicas. Como resultado, há um abismo entre os achados dos experimentos in vitro 18 e aqueles que seriam obtidos em um ambiente celular real. Para preencher a lacuna entre as condições idealizadas de um experimento FPOP in vitro e a natureza complexa da célula, foi desenvolvida uma nova placa automatizada de seis poços baseada em células-FPOP. Esta nova tecnologia FPOP é capaz de identificar e caracterizar essas espécies moleculares e traçar suas interações moleculares dinâmicas em estados saudáveis e doentes. Esta nova plataforma se chama Incubadora de Plataformas com estágio Móvel XY (PIXY).
O FPOP tem sido usado com sucesso para caracterizar as informações estruturais dentro do proteome. No entanto, toda técnica biológica tem certas limitações que requerem melhorias adicionais. Reagentes específicos são necessários durante a fotolise a laser e para saciar eficientemente radicais hidroxil não redigidos. A separação de peptídeos digeridos pode exigir grandes quantidades de tempo para maximizar informações estruturais. Essa riqueza de informações também pode exigir uma extensa quantitação durante a análise de dados pós-MS1. A incubadora da plataforma, incluindo as máquinas periféricas necessárias para a cultura celular e IC-FPOP na plataforma laser, vem com um grande custo que pode não ser viável para alguns laboratórios. À medida que os progressos continuam a ser feitos, ferramentas robustas de software e análise devem avançar ainda mais a técnica; alguns dos quais são mostrados neste estudo. Estudos atuais nesta incubadora de plataformas têm sido realizados em células HEK293T e em C. elegans. O método IC-FPOP mostrou-se compatível com uma grande variedade de linhas celulares, incluindo o ovário de hamster chinês (CHO), Vero, MCF-7 e células MCF10-A19. Uma vez que o método geral IC-FPOP é traduzível para esta plataforma estática, essas linhas celulares devem ser favoráveis para estudo usando PIXY também.
O IC-FPOP utiliza h2O2 para modificar oxidativamente cadeias laterais acessíveis de aminoácidos, para então discernir ainda mais as interações proteicas, estrutura e efeitos metabólicos dentro de células viáveis que é significativa no fornecimento de contexto biológico. É essencial antes de um experimento IC-FPOP confirmar que as células são viáveis após a adição de H2O2. Estudos de viabilidade celular demonstraram que as células eram viáveis na presença de concentrações H2O2 até 200 mM 13. Também é importante garantir que h2O2 seja infundido em uma concentração final de 200 mM diretamente nas células após a mídia ser removida. A falha na remoção completa da mídia de cultura celular causará concentrações variadas de H2O2. Em comparação com as condições padrão, o aumento do tempo de incubação para 10 segundos, juntamente com o aumento da concentração de H2O2 levou a um maior número de proteínas modificadas pelo IC-FPOP na incubadora da plataforma. É imperativo prime bombas peristálticas antes de usar para garantir que as bombas estejam funcionando corretamente e o líquido esteja sendo dispersado. A não orealização pode causar bolhas de ar na tubulação, volume insuficiente de H2O2 para imergir células e/ou volume insuficiente de solução de saciamento.
Outra questão que pode surgir são atrasos indesejados no sistema. Um exemplo disso é o processo de verificação de comandos recebidos para os sistemas de bomba que adiciona atrasos significativos na ordem de 1000 ou mais milissegundos usando o software de integração. Esse problema pode ser corrigido minimizando a comunicação com as bombas durante o experimento e usando comandos pré-definidos antes do tempo, tanto quanto possível.
No futuro, o objetivo da PIXY é produzir um sistema totalmente automatizado e integrado. Além das bombas peristálticas, o acionamento do pulso laser será automatizado. Um novo sistema de posicionamento também será utilizado para o rápido movimento da incubadora da plataforma para melhorar a velocidade e a precisão. Todos os componentes do sistema continuarão a ser programados usando o software de integração para aumentar ainda mais o throughput.
Divulgações
Os autores declaram não haver interesse financeiro concorrente.
A pesquisa aqui apresentada está associada ao pedido de patente abaixo mencionado:
Número de pedido de patente não provisório dos EUA: 17/042.565
Título: "Dispositivo e Método para determinar o dobrável de proteínas"
Número da Lista UMB: LJ-2018-104 UMass Ref: UMA 18-059.
Agradecimentos
Este trabalho foi apoiado por uma subvenção do NIH R01 GM128983-01.
Materiais
| Name | Company | Catalog Number | Comments |
| Ismatec Reglo ICC Peristaltic Pumps | Cole-Palmer | 122270050 | |
| Kinematic Mirror Mount for Ø2" Optics | Thor Labs | ||
| 10X trypsin−EDTA | Corning | AB00490-00005 | |
| 50.0mm 248nm 45°, Excimer Laser Line Mirror | Edmond Optics | ||
| Acetone, HPLC Grade | Fisher Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | ||
| ACQUITY UPLC M-Class Symmetry C18 Trap Column, 100Å, 5 µm, 180 µm x 20 mm, 2G, V/M, 1/pkg | Waters | 23275 | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| ACQUITY UPLC M-Class System | Waters | A53225 | |
| Afinia H480 3D Printer | (Innovation Space University of Maryland Baltimore Campus Library) | ||
| air pump | OKOlabs | D12345 | |
| Aluminum Foil | Fisher Scientific | Stock #63-120 | |
| Aqua 5 µm C18 125 Å packing material | Phenomenex | ||
| carbon dioxide unit | OKOlabs | MadMotor®-UHV | |
| Centrifuge | Eppendorf | ||
| connectors (Y PP 1/16” and 1/16x1/8”) | Cole-Palmer | 116891000 | |
| Delicate Task Wipers | Fisher Scientific | 022625501 | |
| Dithiothreiotol (DTT) | AmericanBio | ||
| DMSO, Anhydrous | Invitrogen | LS118-500 | |
| Dulbecco’s phosphate-buffered saline (DPBS) | Corning | SK-78001-82 | |
| EX350 excimer laser | GAM Laser | PI89900 | |
| Fetal bovine serum (FBS) | Corning | SK-12023-78 | |
| Formic Acid, LC/MS Grade | Fisher Scientific | A929-4 | 4 L quantity is not necessary |
| HEK293T cells | Paul Shapiro Lab (University of Maryland Baltimore) | ||
| humidifier | OKOlabs | Nano-LPMW stage | |
| HV3-2 VALVE | Hamilton | BP152-500 | |
| Hydrogen Peroxide | Fisher Scientific | LS120-500 | |
| Iodoacetamide (IAA) | ACROS Organics | ||
| LABVIEW Professional 2018 | National Instruments | 86728 | |
| MadMotor Positioning system and controllers | Mad City Labs | W6-4 | |
| Methanol, LC/MS Grade | Fisher Scientific | 01-213-100 | any brand is sufficient |
| Microcentrifuge | Thermo Scientific | H301-T Unit-BL-PLUS | |
| N,N′-Dimethylthiourea (DMTU) | ACROS Organics | ||
| Nanopositioinging stage | Mad City Labs | ||
| N-tert-Butyl-α-phenylnitrone (PBN) | ACROS Organics | ||
| OKO Touch Monitoring System | OKOlabs | ||
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | 7Z02936 | |
| PE50-C pyroelectric energy meter | Ophir Optronics | ||
| Penicillin-streptomycin | Corning | ||
| Pierce Quantitative Colorimetric Peptide Assay | Thermo Scientific | ||
| Pierce Rapid Gold BCA Protein Assay Kit | Thermo Scientific | ||
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 75002436 | |
| Pierce Universal Nuclease for Cell Lysis | Fisher Scientific | 06-666A | |
| pressure gauge | OKOlabs | OKO-AIR-PUMP-BL | |
| PTFE filter | OKOlabs | CO2-UNIT-BL | |
| Six 33 mm PLA filament rings | (Innovation Space University of Maryland Baltimore Campus Library) | ||
| sterile incubator | OKOlabs | ||
| temperature unit | OKOlabs | OKO-TOUCH | |
| Thermo Scientific Pierce RIPA Buffer | Fisher Scientific | A117-50 | |
| TiNKERcad | (Innovation Space University of Maryland Baltimore Campus Library) | ||
| Tris Base | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| tubing (Tygon 3.18 and 1.59 ID) | Cole-Palmer | 177350250 | |
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | A454SK-4 | 4 L quantity is not necessary |
| Water, LC/MS Grade | Fisher Scientific | 88702 | |
| 90058 | |||
| KM200 | |||
| 186007496 |
Referências
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins (FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. 294 (32), 11969-11979 (2019).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355-4454 (2019).
- Li, J., Chen, G. The use of fast photochemical oxidation of proteins coupled with mass spectrometry in protein therapeutics discovery and development. Drug Discovery Today. 24 (3), 829-883 (2019).
- Zhao, B., et al. Covalent Labeling with an α,β-Unsaturated Carbonyl Scaffold for Studying Protein Structure and Interactions by Mass Spectrometry. Analytical Chemistry. 92 (9), 6637-6644 (2020).
- Hambly, D. M., Gross, M. L. Laser Flash Photolysis of Hydrogen Peroxide to Oxidize Protein Solvent-Accessible Residues on the Microsecond Timescale. Journal of the American Society for Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Chea, E. E., Jones, L. M. Modifications generated by fast photochemical oxidation of proteins reflect the native conformations of proteins. Protein Science. 27 (6), 1047-1056 (2018).
- Hambly, D., Gross, M. Laser flash photochemical oxidation to locate heme binding and conformational changes in myoglobin. International Journal of Mass Spectrometry. 259 (1), 124-129 (2007).
- Yan, Y. Fast photochemical oxidation of proteins (FPOP) maps the epitope of EGFR binding to adnectin. Journal of the American Society for Mass Spectrometry. 25 (12), 2084-2092 (2014).
- Zhang, Y., Wecksler, A. T., Molina, P., Deperalta, G., Gross, M. L. Mapping the Binding Interface of VEGF and a Monoclonal Antibody Fab-1 Fragment with Fast Photochemical Oxidation of Proteins (FPOP) and Mass Spectrometry. Journal of the American Society for Mass Spectrometry. 28 (5), 850-858 (2017).
- Espino, J. A., Mali, V. S., Jones, L. M. In Cell Footprinting Coupled with Mass Spectrometry for the Structural Analysis of Proteins in Live Cells. Analytical Chemistry. 87 (15), 7971-7978 (2015).
- Rinas, A., Mali, V. S., Espino, J. A., Jones, L. M. Development of a Microflow System for In-Cell Footprinting Coupled with Mass Spectrometry. Analytical Chemistry. 88 (20), 10052-10058 (2016).
- Johnson, D. T., Punshon-Smith, B., Espino, J. A., Gershenson, A., Jones, L. M. Implementing In-Cell Fast Photochemical Oxidation of Proteins in a Platform Incubator with a Movable XY Stage. Analytical Chemistry. 92 (2), 1691-1696 (2019).
- Bangel-Ruland, N., et al. Cystic fibrosis transmembrane conductance regulator-mRNA delivery: a novel alternative for cystic fibrosis gene therapy. The Journal of Gene Medicine. 15 (11-12), 414-426 (2013).
- Gau, B. C., Chen, H., Zhang, Y., Gross, M. L. Sulfate Radical Anion as a New Reagent for Fast Photochemical Oxidation of Proteins. Analytical Chemistry. 82 (18), 7821-7827 (2010).
- Xu, G. C., et al. Hydroxyl Radical-Mediated Modification of Proteins as Probes for StructuralProteomics. Chemical Reviews. American Chemical Society. 107 (8), 3514-3543 (2007).
- Espino, J. A., Jones, L. M. Illuminating Biological Interactions with In Vivo Protein Footprinting. Analytical Chemistry. 9 (10), 6577-6584 (2019).
- Gau, B. C., Sharp, J. S., Rempel, D. L., Gross, M. L. Fast Photochemical Oxidation of Protein Footprints Faster than Protein Unfolding. Analytical Chemistry. 81 (16), 6563-6571 (2009).
- Kaur, U., Johnson, D. T., Jones, L. M. Validation of the Applicability of In-Cell Fast Photochemical Oxidation of Proteins across Multiple Eukaryotic Cell Lines. Journal of the American Society for Mass Spectrometry. 31 (7), 1372-1379 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados