Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis cuantitativo de la dinámica del borde celular durante la propagación celular
* Estos autores han contribuido por igual
En este artículo
Resumen
En este protocolo, presentamos los procedimientos experimentales de un ensayo de propagación celular que se basa en microscopía de células vivas. Proporcionamos una herramienta computacional de código abierto para la segmentación imparcial de células etiquetadas fluorescentemente y análisis cuantitativos de dinámicas de lamellipodia durante la propagación celular.
Resumen
La propagación celular es un proceso dinámico en el que una celda suspendida en medios se adhiere a un sustrato y se aplana de una forma redondeada a una forma delgada y dispersa. Después del apego de sustrato celular, la célula forma una fina lámina de lamellipodia que emana del cuerpo celular. En la lamellipodia, los monómeros de actina globular (G-actin) polimerizan en una densa malla de actina filamentosa (F-actin) que empuja contra la membrana plasmática, proporcionando así las fuerzas mecánicas necesarias para que la célula se propague. En particular, los actores moleculares que controlan la polimerización de actina en lamellipodia son esenciales para muchos otros procesos celulares, como la migración celular y la endocitosis.
Dado que las células de dispersión forman lamellipodia continua que abarca toda la periferia celular y se expanden persistentemente hacia afuera, los ensayos de propagación celular se han convertido en una herramienta eficiente para evaluar la cinética de las protuberancias lamellipodiales. Aunque se han desarrollado varias implementaciones técnicas del ensayo de propagación celular, actualmente falta una descripción detallada del flujo de trabajo, que incluiría tanto un protocolo paso a paso como herramientas computacionales para el análisis de datos. Aquí, describimos los procedimientos experimentales del ensayo de propagación celular y presentamos una herramienta de código abierto para el análisis cuantitativo e imparcial de la dinámica del borde celular durante la propagación. Cuando se combina con manipulaciones farmacológicas y/o técnicas de silenciamiento de genes, este protocolo es apto para una pantalla a gran escala de jugadores moleculares que regulan las protuberancias lamellipodiales.
Introducción
Las protuberancias lamellipodiales son estructuras citoesqueléticos prominentes formadas en la parte delantera de una célula migratoria. En lamellipodia, la polimerización de actina con la ayuda del complejo Arp2/3 y las fímínas crea una malla actin ramificada de rápido crecimiento que empuja contra la membrana plasmática1,2. La fuerza de empuje generada por el trabajo de malla actin impulsa físicamente la celda hacia adelante1,3,4,5. El agotamiento del complejo Arp2/3 o la interrupción de las vías de señalización esenciales para las protuberancias lamellipodiales a menudo afectan la migración celular6, 7. Aunque la migración de células deficientes en lamellipodia también se ha reportado8,9, la importancia de la lamellipodia en la migración celular es evidente ya que el agotamiento de esta estructura sobretrusiva perturba la capacidad de la célula para moverse a través de microambientes biológicos complejos6,10.
Un obstáculo importante para entender la regulación de la lamellipodia en las células migratorias es la variabilidad natural en lamellipodial protrusión cinética, tamaño, y forma11,12,13,14. Además, estudios recientes han demostrado que la lamellipodia exhibe comportamientos protrusivos complejos, incluyendo protuberancias fluctuantes, periódicas y aceleradas14,15. En comparación con la lamellipodia altamente variable de las células migratorias6,16,lamellipodia formada durante la propagación celular son más uniformes12. Dado que la actividad protrusiva de las células de propagación y migración está impulsada por conjuntos macromoleculares idénticos, que incluyen una red actin ramificada, paquetes de actomiosina contractile y adherencias de matriz celular basadas en integrina17,18,las células de propagación han sido ampliamente utilizadas como modelo para investigar la regulación de la dinámica de lamellipodia.
La propagación celular es un proceso mecanoquímico dinámico en el que una célula en suspensión se adhiere primero a un sustrato a través de adherencias basadas en integrina17,19,20 y luego se propaga extendiendo protuberancias basadas en actin21,22,23. Durante la fase de propagación, lamellipodia que emana del cuerpo celular sobresale isotrópica y persistentemente con poca o ninguna retracción o semental12. Los protocolos de propagación celular más utilizados son los ensayos de puntos finales, donde las células de dispersión se fijan en varias ocasiones después de chapar19,24. Estos ensayos, aunque rápidos y sencillos, son limitados en su poder de diagnóstico para detectar cambios en las características dinámicas de la lamellipodia. Para determinar los mecanismos moleculares que controlan la dinámica de la lamellipodia, el grupo Sheetz fue pionero en el uso del análisis cuantitativo de células de propagación en vivo y descubrió muchas propiedades fundamentales de las protuberancias de borde celular11,12,22. Estos estudios han demostrado que el ensayo de propagación de células vivas es una técnica robusta y poderosa en la caja de herramientas de un laboratorio de biología celular. A pesar de eso, un protocolo detallado y una herramienta computacional de código abierto para un ensayo de propagación de células vivas actualmente no están disponibles para la comunidad de biología celular. Con este fin, nuestro protocolo describe los procedimientos de imágenes que propagan en vivo las células y proporciona una herramienta automatizada de análisis de imágenes. Para validar este método, utilizamos la inhibición de Arp2/3 como tratamiento experimental y demostramos que la inhibición de la función del complejo Arp2/3 no detuvo la propagación celular, sino que causó una reducción significativa en la velocidad de protuberancia celular, así como la estabilidad de las protuberancias del borde celular, dando lugar a bordes celulares irregulares. Estos datos demuestran que la combinación de imágenes de células vivas y análisis automatizado de imágenes es una herramienta útil para analizar la dinámica del borde celular e identificar componentes moleculares que regulan la lamellipodia.
Protocolo
1. Siembra celular
NOTA: El protocolo de propagación celular descrito se realizó utilizando fibroblastos embrionarios de ratón (MEF) que expresan PH-Akt-GFP (un marcador fluorescente para PIP3/PI(3,4)P2). Esta línea celular fue generada mediante la integración genómica de una construcción de expresión para PH-Akt-GFP (Addgene #21218) mediante la edición de genes mediados por CRISPR. Sin embargo, otros marcadores fluorescentes que se expresan de forma transitoria o integrado en el genoma también se pueden utilizar en este ensayo. Para una segmentación óptima de la imagen, recomendamos utilizar marcadores fluorescentes que se distribuyan uniformemente en el citoplasma, por ejemplo, GFP citosósmico.
- Cultiva un plato de 10 cm de células al 90% de confluencia.
- Una vez que las células hayan alcanzado la confluencia adecuada, coloque un cubrecoche de 22 mm x 22 mm (#1,5; 0,17 mm de espesor) en un plato de cultivo celular de 35 mm. Cubra el cubrebosque con 400 μL de fibronectina que se ha diluido en PBS a una concentración final de 2,5 μg/ml.
NOTA: El número de puntos de cubierta necesarios para el ensayo viene determinado por el número de condiciones experimentales y réplica técnica. - Coloque el plato de 35 mm con el cubrecubres recubierto de fibronectina en una incubadora de 37 °C, 5% CO2 durante 1 hora.
- Retire el plato con el clip de la incubadora. Aspirar la fibronectina y lavar el tubo de cubierta con PBS pipendo suavemente alrededor de la cubierta de dos a tres veces.
- Aspirar los medios de cultivo celular de la antena de 10 cm de celdas y lavar el plato con PBS.
- Añadir 650 μL de 0.05% trypsin-EDTA al plato 90% confluente de las células, inclinando el plato para distribuir uniformemente la enzima. Coloque el plato con la trypsin en la incubadora durante 1 minuto.
- Retire el plato con las células de la incubadora. Agregue 10 ml de medios de cultivo celular en un tubo centrífugo de 15 ml. Agregue rápidamente otros 10 ml de medios en el plato para apagar la trypsin.
- Pipeta 1 mL de las células trippsinizadas en el tubo centrífuga de 15 ml con el fin de diluir las células. Pipetear el contenido del tubo hacia arriba y hacia abajo para asegurar una distribución uniforme de las células dentro del medio. Para los tipos de células con propensión a la agregación alta, se recomienda filtrar las células a través de un colador de células (tamaño de malla de 100 μm) para minimizar la aparición de aglutinamiento celular.
- Desde el tubo, pipeta 500 - 1000 μL de células diluidas en el plato de 35 mm que contiene el tubo de cubierta.
- Agitar suavemente el plato para extender uniformemente las células. Asegúrese de que el deslizamiento de cubierta esté a una confluencia ~10% (~50,000 células/ml) y ajuste el volumen de las células diluidas según sea necesario.
NOTA: El propósito de tener celdas con una confluencia tan baja es asegurarse de que haya 1-2 celdas polarizadas en cada campo de visión que se utilizarán para enfocar el objetivo a lo largo de la adquisición de propagación celular. - Paso 1/5 de las células restantes en el plato de 10 cm en un plato de 6 cm por condición de tratamiento. Coloque los platos de paso y el plato de 35 mm con el cubrecubrolas en la incubadora durante la noche.
NOTA: Estas serán las células que se analizarán para la dinámica de propagación.
2. Incubación de fármacos y recuperación celular
- Agregue 5 ml de medios de cultivo celular en cada uno de dos tubos centrífugos de 15 ml, y 20 ml de DMEM libre de fenol rojo en cada uno de dos tubos centrífugos de 50 ml.
NOTA: El número de pares de tubos (15 ml + 50 ml) debe corresponder al número de condiciones experimentales. - Para probar la importancia de Arp2/3 para la propagación celular, pipetear ya sea el inhibidor farmacológico de Arp2/3, CK-666, o el tratamiento de control, como dmso, en cada par de tubos centrífugas hasta la concentración deseada.
- Retire los platos de 6 cm de paso (véase el paso 1.11) de la incubadora y aspire a los medios de comunicación. Lave los platos con PBS caliente.
- Añade el contenido de los tubos centrífugas CK-666- o DMSO-complementados con 15 ml en cada uno de los platos de paso. Etiquete cada plato con el tratamiento farmacológico correcto y coloque los platos en la incubadora durante una hora.
- Retire los platos de la incubadora y aspire a los medios de comunicación. Lave los platos con PBS caliente con el fin de eliminar a fondo todos los medios rojos de fenol restantes.
- Añadir 230 μL de 0.05% trypsin-EDTA a cada plato de 6 cm e incubar células durante 1 minuto.
NOTA: Si corresponde, la triptina se puede reemplazar por un bloqueador de adherencia celular no proteótica. - Retire los platos de la incubadora. Para cada tratamiento, añadir 5 ml de fenol sin fármacos DMEM libre de fenol en un tubo centrífuga de 15 ml designado como "Tubo B". Añade 5 ml adicionales de los mismos medios en el plato correspondiente para apagar la trypsin. Transfiera el contenido del plato a un tubo centrífuga de 15 ml designado como "Tubo A".
- Transferir 1 ml de células del tubo A al tubo B. Repita para cada tratamiento.
- Coloque los tubos A y B en la incubadora durante 45 minutos para permitir que las células se recuperen de la trippsinización. Afloje ligeramente la tapa de los tubos centrífugas antes de colocarlos en la incubadora para permitir la penetrancia de CO2.
NOTA: La duración del tiempo de recuperación puede variar para diferentes tipos de celdas. Aunque en nuestros experimentos la recuperación de 45 minutos de duración tuvo un efecto insignificante en la viabilidad celular, algunos tipos de células pueden someterse a anoikis cuando se mantienen en suspensión durante demasiado tiempo. Por lo tanto, recomendamos determinar el tiempo óptimo de recuperación empíricamente. El tiempo óptimo de recuperación permite una propagación rápida y sincrónica de células sin células muertas o apoptóticas en la muestra.
3. Preparación de la cámara magnética
- Asegúrese de que todas las partes de una cámara magnética de células de chamluro de 1 pozo que pueden acomodar un control de cubierta cuadrada de 22 mm x 22 mm se hayan limpiado antes de su uso.
- Retire el plato de 35 mm con el clip (ver Paso 1.11) de la incubadora. Aspirar los medios de cultivo celular y lavar el control de cubierta con PBS caliente.
- Retire el clip de cubierta del plato de 35 mm con un par de fórceps y coloque suavemente el clip en la placa inferior de la cámara magnética.
- Coloque la junta de silicona encima de la cubierta.
NOTA: Una junta de silicona colocada incorrectamente es la causa más común de una cámara magnética con fugas. Asegúrese de que la junta descansa en la sangría de la placa inferior y no se eleva más allá de la sangría. - Coloque el cuerpo principal en la placa inferior.
NOTA: Haga esta parte muy lentamente. Una buena punta es mantener la placa inferior con una mano mientras se coloca el cuerpo principal en la parte superior. Esto asegura que los imanes del cuerpo principal no levanten la placa inferior hacia arriba, lo que potencialmente podría desplazar y romper el deslizamiento de cubierta. - Añadir 1 ml de fenol sin rojo DMEM con suplemento de drogas a la cámara magnética. Tome un tejido libre de pelusas y coloque cuidadosamente la carcasa entre el cuerpo principal y la placa inferior para comprobar si hay fugas.
NOTA: Si hay fugas, aspire rápidamente el medio y proceda de nuevo desde el paso 3.4. - Baje la cubierta transparente sobre el cuerpo principal para encerrar la cámara magnética.
- Rocíe primero un tejido de laboratorio con agua y limpie la parte inferior de la cámara magnética (el cubrecabes, no la parte metálica). Después, rocía un segundo tejido de laboratorio con una pequeña cantidad de 70% de etanol y límpielo, teniendo cuidado de no romper el deslizamiento de las cubiertas.
4. Adquisición de imágenes
- Precaliente la incubadora superior de la etapa y el calentador objetivo a 37 °C y establezca el nivel de CO2 en la incubadora superior de la etapa en un 5%.
NOTA: Si la incubadora superior de etapa no está conectada a un suministro de CO2, los medios de cultivo celular deben complementarse con HEPES de 25 mM para mantener constante pH 7.4. - Aplique una cantidad suficiente de aceite de inmersión al objetivo de aceite 60X, 1.4 N.A. precalentado.
NOTA: Utilizamos un objetivo de inmersión de aceite 60X, 1.4 N.A. en este protocolo debido a su campo de visión razonablemente grande y su excelente eficiencia en la recolección de luz. Si se requiere un campo de visión más grande, se puede utilizar un objetivo de ampliación inferior(por ejemplo,20x) siempre y cuando la relación señal-ruido de las imágenes sea mayor que 2,5. - Lleve tanto la cámara magnética completada como el tubo B (paso 2.9) al microscopio confocal. Coloque la cámara magnética en la incubadora superior del escenario.
NOTA: Coloque la cámara magnética suavemente en el escenario para evitar crear burbujas en el aceite de inmersión. - Establezca el foco en las células fluorescentes utilizando el canal GFP. Asegúrese de que el borde de la celda esté afilado y bien definido.
- Retire la cubierta transparente de la cámara magnética y la pipeta 500 μL del tubo B en la cámara magnética. Coloque la cubierta transparente en la parte superior de la cámara magnética.
- Para identificar las células ideales para el análisis de propagación celular, busque "halos" de células que aún no se han adjunte a la mancha pero que ya no están rodando alrededor. Las células que se encuentran en las primeras etapas de la conexión de coverslip también son grandes candidatos, pero la adquisición de imágenes debe ser rápida para capturar la propagación.
- Configure la adquisición de imágenes de lapso de tiempo para que el canal verde incluya cuatro campos de vista, con imágenes a intervalos de 6 segundos.
NOTA: Debido a la alta variabilidad de la velocidad de protuberancia de lamellipodia entre diferentes tipos de células, la velocidad de fotogramas óptima debe determinarse empíricamente. El intervalo de imágenes de 6 segundos utilizado en nuestros experimentos es un buen punto de partida para el análisis de muchas células mesenquimales y epiteliales. Sin embargo, las células que se propagan muy rápidamente(por ejemplo,células inmunes) pueden requerir una velocidad de fotogramas mucho mayor (intervalo de imágenes más corto). La velocidad de fotogramas óptima para las películas de propagación de celdas garantiza un desplazamiento de 2-5 píxeles del borde de la celda saliente entre fotogramas posteriores. Teniendo en cuenta la precisión del accesorio de curva utilizado para identificar la meseta de la propagación celular, la velocidad de fotogramas óptima también debe garantizar 50-100 mediciones del desplazamiento del borde de la célula durante la fase de expansión rápida de la propagación celular. El número de campos de visión debe ajustarse en función del tiempo de exposición, la distancia entre los puntos de adquisición y la velocidad de movimiento de la etapa. Se recomienda a los usuarios determinar el número máximo de campos de vista que se pueden adquirir con la velocidad de fotogramas deseada. - Después de identificar un campo de visión adecuado, guarde las coordenadas X e Y de la etapa del microscopio. Proceda a identificar otros tres campos de visión que están relativamente cerca unos de otros en el panel de cubiertas. Guarde las coordenadas de la etapa del microscopio para cada campo de visión deseado.
NOTA: Se recomienda encarecidamente optimizar la trayectoria de movimiento de la etapa entre campos de visión para minimizar cualquier movimiento de muestra innecesario. Dicha optimización se puede realizar manual o automáticamente. El movimiento excesivo de la muestra ralentiza la adquisición y puede hacer que las células se desplieguen fuera de la vista a medida que descienden. - Adquiera imágenes durante 15 minutos a una velocidad de fotogramas de 6 segundos y guarde los archivos. Si se requieren más adquisiciones, repita a partir del paso 4.6.
5. Análisis del área celular, la circularidad y la dinámica de protuberancia durante la propagación celular
- Preparar imágenes para el procesamiento y análisis de datos
NOTA: El software requiere una imagen en formato .tiff y un tamaño de píxel como parámetros de entrada. Ambos requisitos se pueden cumplir utilizando el software de adquisición o Fiji (en este protocolo). Si se cumplen estos requisitos, continúe con el paso 5.2.- Instale la última versión de la aplicación Fiji (https://imagej.net/Fiji/Downloads).
- Abra una imagen de lapso de tiempo utilizando Fiji.
- Copie el tamaño de píxel de la imagen seleccionando Propiedades de image >. Copie y pegue el tamaño del píxel en μm hasta el Bloc de notas/Word.
- Para el análisis del área de dispersión celular y la circularidad, guarde la imagen de lapso de tiempo como una pila de imágenes tiff. El software de análisis de compilación personalizada no admite formatos de archivo propietarios. Guarde la pila de imágenes tiff de celda individual seleccionando Archivo > Guardar como > Tiff.
- Instale python IDE (Spyder) y los paquetes necesarios (PySimpleGUI y tifffile) para el procesamiento y análisis de datos.
NOTA: La instalación de Python y paquetes solo es necesaria para la configuración inicial.- Las películas de lapso de tiempo se analizarán en el IDE de Spyder mediante una secuencia de comandos de Python de compilación personalizada. Para descargar el Spyder IDE, descargue el distribuidor Anaconda (https://www.anaconda.com/products/individual) que incluye Spyder IDE y la mayoría de las bibliotecas y paquetes necesarios para este análisis.
- Instale Anaconda y inicie Spyder a través de Anaconda Navigator.
- En la pestaña consola de IPython (ubicada en la sección inferior derecha de Spyder), copie y pegue el siguiente comando: pip instalar PySimpleGUI y pulse la tecla enter. La ejecución de este comando instalará el paquete necesario para iniciar la interfaz gráfica de usuario (GUI).
- En la misma consola, copie y pegue el siguiente comando: pip install tifffile y pulse la tecla Intro. La ejecución de este comando instalará el paquete necesario para guardar imágenes como archivos tiff.
- Descargue todos los scripts de Python de los archivos complementarios o los scripts más actualizados de GitHub en: https://github.com/ernestiu/Cell-spreading-analysis.git
- Cuantificar el área celular y los factores de forma celular durante la propagación celular
- Abra el script de análisis principal "cell_spreading_GUI.py" seleccionando la opción abrir archivo en el panel superior de Spyder o utilizando el acceso directo Ctrl + O.
- Abra la GUI de análisis de dispersión de celdas seleccionando "Ejecutar archivo" en el panel superior o utilizando el acceso directo F5.
- Haga clic en la pestaña Área de dispersión de celda ( Figura3A).
- Seleccione la imagen tiff que desea analizar con el botón Examinar.
NOTA: El archivo seleccionado debe ser un archivo tiff. - Especifique el directorio de destino donde se guardarán las salidas de datos (por ejemplo, máscaras de celda, valores).
- Especifique la configuración de salida de datos:
- Guardar máscaras: guarde las máscaras de celda generadas durante el proceso de segmentación.
- Exportar datos: exporte una hoja de cálculo de Excel (.xlsx) que contenga todos los datos de análisis a la carpeta de destino.
El área celular, la circularidad y la relación de aspecto de todas las celdas de dispersión se guardarán como una hoja de cálculo de Excel en la carpeta de destino. El área de la celda se calcula como: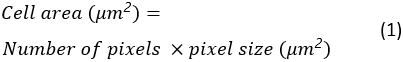
La circularidad celular es una medida de lo cerca que está una célula de una célula perfectamente redonda. Se calcula como: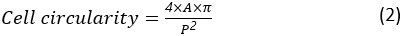
donde A y P son el área celular y el perímetro de la célula, respectivamente. La relación de aspecto de la celda representa lo alargada que está la celda. Una célula de dispersión debe tener una relación de aspecto cercana a 1. La relación de aspecto se calcula como: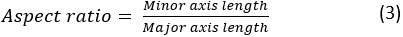
- Guardar contornos: guarde las imágenes de superposición de contorno de contorno de celda en la carpeta de destino.
- Especifique la configuración de segmentación
- Mostrar segmentación: muestra el resultado de segmentación en la consola de Spyder durante el proceso de análisis.
- Área celular más pequeña (μm2):Introduzca el valor mínimo para el área celular, incluidos los valores de área de las células en las etapas iniciales del apego. Los objetos con un área menor que este umbral no se considerarán células de propagación. Este número afectará al proceso de segmentación.
- Especifique los parámetros de imagen.
- Intervalo de adquisición(s): Introduzca la frecuencia de la adquisición de la imagen en segundos.
- Tamaño del píxel (μm): introduzca el tamaño de píxel que se grabó al preparar imágenes para su análisis.
- Profundidad de bits de imagen: Introduzca la profundidad de bits de la cámara/detector.
- Haga clic en Ejecutar. Si surge un error, aparecerá un mensaje de error en la consola de Spyder. De lo contrario, el proceso de análisis de imágenes se mostrará en la consola.
NOTA: La primera imagen que aparece en la sección consola/Trazados (dependiendo de la configuración de Spyder) muestra todas las celdas identificadas en el campo de visión. Las cajas verdes colocadas alrededor de las celdas indican células que se extienden que son adecuadas para la segmentación y el análisis. Las cajas grises indican celdas que no son adecuadas para el análisis. El número total de celdas de propagación identificadas también aparecerá en la pestaña Consola. El software traza el área celular (en azul) y la circularidad celular (en rojo) en función del tiempo. Estos gráficos permiten a los usuarios evaluar la precisión de la segmentación celular. Una segmentación exitosa produce una curva que aumenta monótonamente para el área de celda. Para obtener una curva representativa del área de dispersión de la celda, la fase de retraso debe eliminarse del gráfico manualmente. La fase de retraso incluye mediciones del área celular antes de que la célula comience a propagarse. La fase de retraso se indica mediante fluctuaciones rápidas, como se representa en la gráfica del área de celda(figura 3C derecha).
6. Cuantificar la dinámica del borde celular durante la propagación celular usando kymographs
- Antes de ejecutar el análisis, recorte las películas crudas de las células de propagación para crear series temporales de células de propagación individuales.
- Utilice la herramienta Rectángulo en la barra de herramientas de Fiji para seleccionar manualmente una región de interés (ROI) que encapsula una sola celda. (Para asegurarse de que el ROI encapsula completamente la célula de dispersión, utilice la función de desplazamiento para inspeccionar el ROI en todos los puntos de tiempo.)
- Haga clic con el botón derecho en el ROI y seleccione Duplicar.
- Compruebe Duplicar pila y haga clic en Aceptar.
- Abra el script de análisis principal "cell_spreading_GUI.py" seleccionando el botón Abrir archivo en la barra de herramientas de Spyder o utilizando el acceso directo Ctrl + O. Si el GUI ya se ha abierto, vaya directamente al paso 6.3.
- Abra la GUI de análisis de dispersión de celdas seleccionando Ejecutar archivo en el panel superior o utilizando el acceso directo F5 (Figura 3B).
- Haga clic en la pestaña Generador de kymógrafos y análisis.
- Utilice el botón Examinar para seleccionar la imagen tiff para el análisis.
NOTA: Los formatos de archivo propietarios, por ejemplo, nd2, lif, zen, no son compatibles con el script. - Especifique la carpeta de destino para guardar los datos de salida (máscaras de celda y valores).
- Especifique la configuración de salida.
- Exportar datos: exporte una hoja de cálculo de Excel (.xlsx) a la carpeta de destino que contiene posiciones relativas de borde de celda y eventos de retracción de los kymographs.
- Especifique los parámetros de imagen:
- Intervalo de adquisición (s): Introduzca la frecuencia de adquisición de imágenes en segundos.
- Tamaño del píxel (μm): introduzca el tamaño de píxel que se grabó al preparar imágenes para el análisis en el paso 5.1.3.
- Área celular más pequeña (μm2):Introduzca el valor mínimo para el área celular, incluidos los valores de área de las células en las etapas iniciales del apego. Los objetos con un área menor que este umbral no se considerarán células de propagación. Este número afectará al proceso de segmentación.
- Profundidad de bits de imagen: Introduzca la profundidad de bits de la cámara/detector.
- Haga clic en Ejecutar. Si surge un error, aparecerá un mensaje de error en la consola de Spyder. De lo contrario, se mostrará un resumen de las cuantificaciones de dinámicas de protuberancia en la consola. Habrá 4 pares de mediciones de frecuencia de retracción y velocidad de protuberancia, que se extraen de 4 kymographs generados de las partes superior, inferior, izquierda y derecha de la célula. La frecuencia de retracción se calcula como:
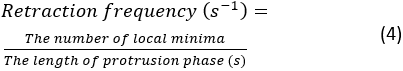
NOTA: Este número demuestra la frecuencia con la que el lamellipodium se retrae en el transcurso de la propagación. La velocidad media de protuberancia se mide por la pendiente entre el comienzo de la protuberancia y el punto de meseta en el kymograph. Una figura de kymograph de resumen se mostrará en la consola después de la segmentación. Para guardar la figura de resumen, haga clic con el botón derecho en la figura y guarde la imagen.
Resultados
El protocolo anterior describe los procedimientos experimentales para la toma de imágenes de células vivas de células de dispersión y una herramienta computacional para el análisis cuantitativo de la dinámica de propagación celular. La herramienta computacional se puede utilizar en un formato de bajo o alto rendimiento para identificar los reproductores moleculares que regulan la maquinaria de polimerización actin en el borde de ataque celular.
La representación esquemática de los pr...
Discusión
El ensayo de propagación celular descrito permite el seguimiento continuo de los cambios morfológicos(por ejemplo, el tamaño y la forma de la célula) y los movimientos del borde celular(es decir, la velocidad de protuberancia y la frecuencia de retracción), que son características que faltan en la mayoría de los protocolos de propagación celular19,24. Si bien los ensayos de propagación de células de punto final utilizados comúnmente p...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por el Premio Connaught Fund New Investigator a S.P., Canada Foundation for Innovation, NSERC Discovery Grant Program (subvenciones RGPIN-2015-05114 y RGPIN-2020-05881), University of Manchester y University of Toronto Joint Research Fund, y University of Toronto XSeed Program.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
Referencias
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados