JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
셀 확산 시 셀 엣지 역학의 정량적 분석
요약
이 프로토콜에서, 우리는 살아있는 세포 현미경 검사법을 기반으로 하는 세포 퍼지는 분석의 실험 절차를 제시합니다. 당사는 형광으로 표지된 세포의 편견없는 세분화와 세포 확산 중 라멜리포디아 역학의 정량적 분석을 위한 오픈 소스 계산 도구를 제공합니다.
초록
세포 확산은 매체에 매달려 있는 셀이 기판에 부착되어 둥근 모양에서 얇고 확산된 모양으로 자신을 평평하게 하는 동적 프로세스입니다. 세포 기판 부착에 따라, 세포는 세포 체에서 방출되는 라멜리포디아의 얇은 시트를 형성한다. 라멜리포디아에서, 구형 액틴(G-actin) 단량체는 플라즈마 멤브레인에 밀어 넣는 조밀한 필라멘트 액틴(F-actin) 메쉬워크로 중합하여 세포가 퍼지는 데 필요한 기계적 힘을 제공한다. 특히, 라멜리포디아에서 액틴 중합화를 제어하는 분자 플레이어는 세포 이동 및 내분비증과 같은 많은 다른 세포 공정에 필수적입니다.
확산 세포는 전체 세포 주변을 가로 지르고 지속적으로 바깥쪽으로 확장 연속 라멜리포디아를 형성하기 때문에 세포 확산 분석은 lamellipodial 돌출의 운동을 평가하는 효율적인 도구가되었습니다. 셀 확산 분석의 여러 기술적 구현이 개발되었지만 단계별 프로토콜과 데이터 분석을 위한 계산 도구가 모두 포함된 워크플로우에 대한 자세한 설명은 현재 부족합니다. 여기서, 우리는 세포 확산 분석의 실험 절차를 설명하고 확산 하는 동안 세포 가장자리 역학의 정량적이고 편견 없는 분석을 위한 오픈 소스 도구를 제시. 약리학적 조작 및/또는 유전자 침묵 기술과 결합될 때, 이 프로토콜은 적각돌출을 조절하는 분자 플레이어의 대규모 스크린에 순종적입니다.
서문
Lamellipodial 돌출은 마이그레이션 셀의 전면에 형성된 눈에 띄는 사이토켈레탈 구조입니다. 라멜리포디아에서는 Arp2/3 복합체 및 포르민의 도움으로 액틴의 중합화가 플라즈마 멤브레인1,2에밀어내는 빠르게 성장하는 분기 액틴 메쉬워크를 생성한다. 액틴 메쉬워크에 의해 생성된 푸시 력은셀을1,3,4,5로물리적으로추진한다. Arp2/3 복잡하고 라멜리포성 돌출에 필수적인 신호 경로의 중단은 종종 세포 마이그레이션6, 7을손상시합니다. 라멜레포디아 결핍 세포의 이동도8,9,세포 이동에서 라멜리포디아의 중요성은 이러한 돌출 구조의 고갈로 명백하다 는 것은 세포의 복잡한 생물학적 미세환경6,10을통해 이동하는 세포의 능력을 퍼튜브한다.
이동 세포에서 라멜리포디아의 조절을 이해하는 주요 장애물은 라미포리 포성 돌출 운동, 크기 및 모양11,12,13,14의자연적인 가변성이다. 더욱이, 최근 연구는 라멜리포디아가 변동, 주기적, 가속돌출14,15를포함하여 복잡한 돌출행동을 나타낸다는 것을 보여주었습니다. 이동 세포의 고가변 라멜리포디아에 비해6,16,세포 확산 시 형성된 라멜리포디아는 더 균일한12이다. 세포 확산 및 이동세포의 돌출활성은 분지액망, 수축성 아토묘신 번들, 및 통합계 세포매트릭스접착(17,18)을포함하는 동일한 거시분자 어셈블리에 의해 구동되기 때문에, 확산세포는 라멜리포디아역학의 조절을 조사하기 위한 모델로 널리 사용되고 있다.
세포확산은 현탁액내 세포가 먼저 정립기반 접착착(17,19,20)을 통해 기판에 부착한 다음 액틴기반돌출21,22,23을연장하여 확산되는 동적 메카노케미컬 공정이다. 확산 단계에서, 세포 체에서 방출되는 라멜리포디아는 거의 또는 전혀 후퇴또는 실속없이 이소열대및 지속적으로 돌출된다12. 가장 일반적으로 사용되는 세포 확산 프로토콜은19,24를도금한 후 확산 세포가 여러 번에 고정되는 엔드포인트 분석입니다. 이 assays는 빠르고 간단하지만 라멜리포디아의 동적 기능의 변화를 감지하기 위해 진단 능력이 제한됩니다. 라멜리포디아 역학을 제어하는 분자 메커니즘을 결정하기 위해, 시트츠 그룹은 살아있는 확산 세포의 정량적 분석의 사용을 개척하고 세포 가장자리 돌출의 많은 기본 특성을 발견11,12,22. 이러한 연구는 살아있는 세포 확산 분석이 세포 생물학 실험실의 도구 상자에 강력하고 강력한 기술임을 입증했다. 그럼에도 불구하고, 살아있는 세포 확산 분석에 대한 상세한 프로토콜 및 오픈 소스 계산 도구는 현재 세포 생물학 커뮤니티에 사용할 수 없습니다. 이를 위해 당사의 프로토콜은 라이브 확산 세포를 이미징하는 절차를 설명하고 자동화된 이미지 분석 도구를 제공합니다. 이러한 방법을 검증하기 위해 Arp2/3 억제를 실험적 치료법으로 사용했으며, Arp2/3 복합체의 기능을 억제하는 것은 세포 확산을 억제하지 는 않았지만 세포 돌출 속도의 안정성뿐만 아니라 세포 가장자리 돌출의 안정성뿐만 아니라 들쭉날쭉한 세포 가장자리를 야기하는 것으로 나타났다. 이러한 데이터는 라이브 셀 이미징과 자동화된 이미지 분석의 조합이 세포 가장자리 역학을 분석하고 lamellipodia를 조절하는 분자 구성 요소를 식별하는 데 유용한 도구임을 보여줍니다.
프로토콜
1. 셀 시드
참고: 기술된 세포 확산 프로토콜은 PH-Akt-GFP를 발현하는 마우스 배아 섬유아세포(MEFs)를 사용하여 수행되었다(PIP3/PI(3,4)P2용형광 마커). 이러한 세포주들은 CRISPR 중재 유전자 편집에 의해 PH-Akt-GFP(Addgene #21218)에 대한 발현 구조를 genomically 통합하여 생성하였다. 그러나, 게놈에서 일시적으로 표현되거나 통합되는 그밖 형광 마커는 또한 이 분석에서 이용될 수 있다. 최적의 이미지 세분화를 위해 세포질(예: 세포질 GFP)에 균등하게 분포되는 형광 마커를 사용하는 것이 좋습니다.
- 90%의 수렴성으로 10cm의 세포 요리를 배양합니다.
- 세포가 적절한 인플루엔지를 달성하면 22mm x 22mm 커버슬립(#1.5; 0.17mm 두께)을 35mm 세포 배양 접시에 넣습니다. PBS에서 희석된 400μl의 커버슬립을 최종 농도인 2.5 μg/mL로 코팅합니다.
참고: 분석에 필요한 커버립 수는 실험 조건 및 기술 복제본 수에 따라 결정됩니다. - 35mm 접시에 섬유네틴 코팅 커버슬립을 37°C, 5% CO2 인큐베이터에 1시간 동안 넣습니다.
- 인큐베이터에서 커버슬립으로 접시를 제거합니다. 섬유네틴을 흡인하고 커버슬립 을 2~3번 부드럽게 피펫하여 PBS로 커버슬립을 씻습니다.
- 세포 배양 매체를 셀 10cm 접시에서 흡인시키고 PBS로 접시를 씻는다.
- 650 μL0 0.05% 트립신-EDTA를 90% 컨물수 있는 세포 접시에 넣고, 접시를 기울여 효소를 고르게 분배합니다. 트립신과 함께 접시를 인큐베이터에 1분간 넣습니다.
- 인큐베이터에서 셀로 접시를 제거합니다. 15mL 원심분리기 튜브에 세포 배양 배지 10mL를 추가합니다. 신속하게 트립신을 담금질 접시에 미디어의 또 다른 10 mL을 추가합니다.
- 피펫 1 mL의 트립시화 된 세포는 세포를 희석시키기 위해 15 mL 원심분리기 튜브로 한다. 튜브의 내용이 위아래로 피펫되어 미디어 내에서 세포의 균등한 분포를 보장합니다. 응집 성향이 높은 세포 유형의 경우 세포 응집성(100 μm 메쉬 크기)을 통해 세포를 필터링하여 세포 응집발생을 최소화하는 것이 좋습니다.
- 튜브에서, 파이펫 500 - 1000 μL 희석 된 세포의 커버 슬립을 포함하는 35mm 접시에.
- 부드럽게 셀을 퍼지기 위해 접시를 흔들어 줍니다. 커버슬립이 ~10%의 인플루엔자(~50,000셀/mL)에 있는지 확인하고 필요에 따라 희석된 세포의 부피를 조정합니다.
참고: 이러한 낮은 수렴성에서 세포를 갖는 목적은 각 시야에 1-2 개의 편광 세포가 있는지 확인하는 것입니다. - 나머지 세포의 1/5를 10cm 접시에 넣고 치료 조건당 6cm 접시 1개씩 통과합니다. 통로 요리와 35mm 접시에 커버슬립을 넣고 하룻밤 동안 인큐베이터에 넣습니다.
참고: 이러한 역학 확산을 위해 분석될 세포입니다.
2. 약물 인큐베이션 및 세포 회수
- 각각 2개의 15mL 원심분리기 튜브에 5mL의 세포 배양 배지를 추가하고, 20mL의 페놀 레드 프리 DMEM을 각각 2개의 50mL 원심분리기 튜브에 넣습니다.
참고: 튜브 쌍(15mL + 50 mL)의 수는 실험 조건수에 해당한다. - 세포 확산을 위한 Arp2/3의 중요성을 테스트하기 위해, 파이펫은 Arp2/3, CK-666 또는 DMSO와 같은 제어 처리의 약리학적 억제제 중 하나를 원하는 농도까지 원심분리기 튜브의 각 쌍으로 한다.
- 인큐베이터에서 구절6cm 요리(1.11단계 참조)를 제거하고 미디어를 흡인합니다. 따뜻한 PBS로 설거지합니다.
- CK-666 또는 DMSO 보충 15mL 원심분리기 튜브의 내용을 각 구절 요리에 추가합니다. 각 요리에 올바른 약물 치료라벨을 지정하고 요리를 인큐베이터에 1시간 동안 넣습니다.
- 인큐베이터에서 요리를 제거하고 미디어를 흡인합니다. 남은 페놀 레드 미디어를 철저히 제거하기 위해 따뜻한 PBS로 설거지하십시오.
- 각 6cm 접시에 0.05% 트립신-EDTA의 230 μL을 추가하고 1 분 동안 세포를 배양합니다.
참고: 해당하는 경우 트립신은 비성촉매 세포 접착 차단제로 대체될 수 있습니다. - 인큐베이터에서 요리를 제거합니다. 각 치료에 대해 5mL의 약물 보충 페놀 레드 프리 DMEM을 "튜브 B"로 지정된 15mL 원심분리기 튜브에 추가하십시오. 트립신을 담금질하기 위해 관련 접시에 동일한 미디어의 5mL를 추가합니다. 접시의 내용을 "튜브 A"로 지정된 15mL 원심분리기 튜브로 옮기습니다.
- 튜브 A에서 튜브 B. 각 치료에 대해 반복으로 세포의 1 mL을 전송합니다.
- 튜브 A와 B를 45분 동안 인큐베이터에 넣고 세포가 트립시화에서 회복할 수 있도록 합니다. CO2 침투를 허용하기 위해 인큐베이터에 넣기 전에 원심분리기 튜브의 캡을 약간 느슨하게 합니다.
참고: 회복 시간의 기간은 다른 세포 유형에 따라 다를 수 있습니다. 비록 우리의 실험에서 45 분 긴 복구 세포 생존에 무시할 수 있는 영향을 했다, 일부 세포 유형 너무 오래 현 탁 트에서 유지 될 때 anoikis를 겪을 수 있습니다. 따라서 최적의 복구 시간을 경험적으로 결정하는 것이 좋습니다. 최적의 회복 시간을 통해 샘플에서 죽거나 세포가 없는 빠르고 동기적인 세포가 확산될 수 있습니다.
3. 마그네틱 챔버 준비
- 22mm x 22mm 정사각형 커버슬립을 수용할 수 있는 1웰 차멜라이드 셀 자기 챔버의 모든 부분이 사용하기 전에 청소되었는지 확인합니다.
- 인큐베이터에서 커버슬립(1.11단계 참조)으로 35mm 접시를 제거합니다. 세포 배양 매체를 흡인하고 따뜻한 PBS로 커버슬립을 씻으세요.
- 35mm 접시에서 커버슬립을 제거하여 한 쌍의 포셉을 사용하여 커버슬립을 부드럽게 마그네틱 챔버의 하단 플레이트에 놓습니다.
- 실리콘 개스킷을 커버슬립 위에 놓습니다.
참고: 부적절하게 배치된 실리콘 개스킷은 새는 자기 챔버의 가장 흔한 원인입니다. 개스킷이 바닥 판의 들여쓰기에 놓여 있고 들여쓰기 를 넘어 상승하지 않도록 하십시오. - 본체를 하단 플레이트에 부착합니다.
참고: 이 부분을 매우 느리게 수행합니다. 좋은 팁은 본체를 위에 놓으면서 한 손으로 하단 플레이트를 누르는 것입니다. 이것은 본체의 자석이 바닥 판을 들어 올리지 않도록 보장하며, 이는 잠재적으로 변위시키고 커버슬립을 해독할 수 있습니다. - 자기 챔버에 약물 보충 페놀 레드 프리 DMEM의 1 mL을 추가합니다. 보풀이 없는 조직을 가지고 조심스럽게 누출을 확인하기 위해 본체와 하단 플레이트 사이의 인클로저를 두드려.
참고: 누출이 있는 경우 미디어를 신속하게 흡인하고 3.4단계에서 다시 진행합니다. - 투명 커버를 본체에 내리고 자기 챔버를 둘러싸습니다.
- 실험실 조직을 먼저 물로 분무하고 자기 챔버의 바닥을 닦아냅니다 (금속 부분이 아닌 덮개 슬립). 그 후, 소량의 70 %의 에탄올로 두 번째 실험실 조직을 분사하고 닦아, 커버 슬립을 균열하지 않도록주의.
4. 이미지 수집
- 스테이지 탑 인큐베이터와 목표 히터를 37°C로 예열하고 스테이지 탑 인큐베이터에서 CO2 레벨을 5%로 설정합니다.
참고: 스테이지 탑 인큐베이터가CO2 공급에 연결되지 않으면 세포 배양 매체를 25m HEPES로 보충하여 일정한 pH 7.4를 유지해야 합니다. - 미리 데워진 60X, 1.4 N.A. 오일 목표에 충분한 양의 침수 오일을 적용합니다.
참고: 당사는 합리적으로 넓은 시야와 뛰어난 광 수집 효율성으로 인해 이 프로토콜에서 60X, 1.4 N.A. 오일 침수 목표를 사용합니다. 더 큰 시야가 필요한 경우, 이미지의 신호 대 잡음 비율이 2.5보다 큰 한 배율목표(예:20x)를 사용할 수 있다. - 완성된 자기 챔버와 튜브 B(2.9단계)를 모두 공초점 현미경으로 가져옵니다. 자기 챔버를 스테이지 상단 인큐베이터에 놓습니다.
참고: 자성챔버를 무대에 부드럽게 배치하여 침수 오일에 거품을 일으키지 않도록 합니다. - GFP 채널을 사용하여 형광 세포에 초점을 설정합니다. 셀 가장자리가 날카롭고 잘 정의된지 확인합니다.
- 자기 챔버와 파이펫 500 μL의 투명 커버를 튜브 B에서 자기 챔버로 제거합니다. 투명 커버를 자기 챔버 위에 다시 놓습니다.
- 세포 확산 분석에 이상적인 세포를 식별하려면 아직 커버슬립에 부착하지 않았지만 더 이상 굴러다니지 않는 세포의 "후광"을 검색합니다. 커버슬립 부착의 초기 단계에 있는 세포도 훌륭한 후보이지만 확산을 포착하려면 이미지 수집이 신속해야 합니다.
- 녹색 채널에 대한 시간 경과 이미지 수집을 구성하여 6초 간격으로 이미지된 4개의 뷰 필드를 포함합니다.
참고: 다른 세포 유형 중 lamellipodia 돌출 속도의 높은 가변성으로 인해 최적의 프레임 속도를 경험적으로 결정해야 합니다. 우리의 실험에 사용된 6 초의 화상 진찰 간격은 많은 중간엽 및 상피 세포의 분석을 위한 좋은 출발점입니다. 그러나, 매우 빠르게 퍼지는세포(예를 들어,면역 세포)는 훨씬 더 높은 프레임 속도(짧은 이미징 간격)를 요구할 수 있다. 셀 퍼짐 동영상에 대한 최적의 프레임 속도는 후속 프레임 사이의 돌출 셀 가장자리의 2-5픽셀 변위를 보장합니다. 세포 확산의 고원을 식별하는 데 사용되는 곡선 피팅의 정확도를 고려하여 최적의 프레임 속도는 세포 확산의 급속한 팽창 단계에서 셀 에지 변위의 50-100 측정을 보장해야 합니다. 뷰 필드 수는 노출 시간, 획득 지점 사이의 거리 및 스테이지 이동 속도에 따라 조정되어야 합니다. 사용자는 원하는 프레임 속도로 획득할 수 있는 최대 보기 필드 수를 결정하는 것이 좋습니다. - 적당한 시야를 확인한 후 현미경 단계의 X 및 Y 좌표를 저장하십시오. 커버슬립에서 서로 비교적 가까운 세 가지 다른 시야 필드를 확인합니다. 원하는 모든 시야에 대한 현미경 단계의 좌표를 저장합니다.
참고: 불필요한 샘플 움직임을 최소화하기 위해 시야 간 스테이지 이동 경로를 최적화하는 것이 좋습니다. 이러한 최적화는 수동으로 또는 자동으로 수행할 수 있습니다. 과도한 샘플 이동은 획득 속도를 늦추고 셀이 내림차순으로 시야에서 굴러 나올 수 있습니다. - 6초 프레임 속도로 15분 동안 이미지를 획득하고 파일을 저장합니다. 더 많은 인수가 필요한 경우 4.6 단계에서 반복하십시오.
5. 세포 영역, 세포 확산 중 세포 영역, 원형 및 돌출 역학 분석
- 데이터 처리 및 분석을 위한 이미지 준비
참고: 소프트웨어는 .tiff 형식의 이미지와 입력 매개 변수로 픽셀 크기가 필요합니다. 두 요구 사항은 인수 소프트웨어 또는 피지(이 프로토콜)를 사용하여 충족될 수 있습니다. 이러한 요구 사항이 충족되면 5.2 단계로 진행하십시오.- 피지 응용 프로그램의 최신 버전을 설치 (https://imagej.net/Fiji/Downloads).
- 피지를 사용하여 시간 경과 이미지를 엽니 다.
- 이미지 > 속성을 선택하여 이미지의 픽셀 크기를 복사합니다. 픽셀 크기를 피치패드/Word에 복사하여 붙여넣습니다.
- 셀 스프레드 영역 및 원형 분석을 위해 시간 경과 이미지를 티프 이미지 스택으로 저장합니다. 사용자 지정 빌드 분석 소프트웨어는 독점 파일 형식을 지원하지 않습니다. 파일 > > Tiff로 저장을 선택하여 개별 셀 티프 이미지 스택을 저장합니다.
- 데이터 처리 및 분석을 위해 파이썬 IDE(스파이더)와 필요한패키지(PySimpleGUI 및 tifffile)를 설치합니다.
참고: 초기 설정에 대해서만 파이썬 및 패키지를 설치해야 합니다.- 시간 경과 영화는 사용자 정의 빌드 파이썬 스크립트를 사용하여 스파이더 IDE에서 분석됩니다. Spyder IDE를 다운로드하려면 스파이더 IDE와 이 분석에 필요한 대부분의 라이브러리 및 패키지를 포함하는 아나콘다 배포자(https://www.anaconda.com/products/individual)를 다운로드하십시오.
- 아나콘다를 설치하고 아나콘다 네비게이터를 통해 스파이더를 시작합니다.
- IPython 콘솔 탭(Spyder의 오른쪽 아래 부분에 위치)에서 다음 명령을 복사하여 붙여 넣습니다: 핍 설치 PySimpleGUI및 enter 키를 누릅니다. 이 명령을 실행하면 그래픽 사용자 인터페이스(GUI)를 시작하는 데 필요한 패키지가 설치됩니다.
- 동일한 콘솔에서 다음 명령을 복사하여 붙여 넣습니다: 핍 설치 티프파일및 Enter 키를 누릅니다. 이 명령을 실행하면 이미지를 티프 파일로 저장하는 데 필요한 패키지가 설치됩니다.
- 보충 파일또는 GitHub에서 가장 업데이트 된 스크립트에서 모든 파이썬 스크립트를 다운로드 : https://github.com/ernestiu/Cell-spreading-analysis.git
- 세포 확산 중 세포 영역 및 세포 모양 인자를 정량화
- Spyder의 상단 패널에서 열린 파일 옵션을 선택하거나 바로 가기 Ctrl + O를사용하여 기본 분석 스크립트 "cell_spreading_GUI.py"를 엽니 다.
- 상단 패널에서 "파일 실행"을 선택하거나 바로 가기 F5를사용하여 셀 확산 분석 GUI를 엽니다.
- 셀 스프레드 영역 탭(그림3A)을클릭합니다.
- 찾아보기 버튼을 사용하여 분석할 티프 이미지를 선택합니다.
참고: 선택한 파일은 티프 파일이어야 합니다. - 데이터 출력(예: 셀 마스크, 값)이 저장되는 대상 디렉터리지정을 지정합니다.
- 데이터 출력 설정을 지정합니다.
- 마스크 저장: 세분화 과정에서 생성된 셀 마스크를 저장합니다.
- 데이터 내보내기: 모든 분석 데이터를 대상 폴더에 포함하는 엑셀 스프레드시트(.xlsx)를 내보냅니다.
모든 확산 셀의 셀 영역, 원형 및 종횡비는 대상 폴더의 Excel 스프레드시트로 저장됩니다. 셀 영역은 다음과 같이 계산됩니다.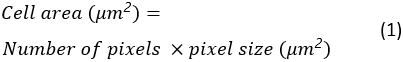
세포 순환은 셀이 완벽하게 둥근 셀에 얼마나 가까운지 측정합니다. 다음과 같이 계산됩니다.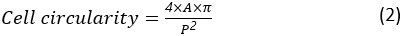
여기서 A와 P는 각각 세포 영역과 세포 둘레이다. 셀의 종횡비는 셀이 얼마나 길었는지 나타냅니다. 확산 셀은 종횡비비율이 1에 가깝어야 합니다. 종횡비는 다음과 같이 계산됩니다.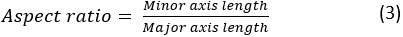
- 윤곽 저장: 대상 폴더에 셀 경계 등고선 오버레이 이미지를 저장합니다.
- 세분화 설정 지정
- 세분화 표시: 분석 프로세스 중에 Spyder 콘솔에서 세분화 결과를 표시합니다.
- 가장 작은 세포 영역(μm2):부착의 시작 단계에서 세포의 영역 값을 포함하여 셀 영역에 대한 최소 값을 입력합니다. 이 임계값보다 작은 영역을 가진 객체는 확산 셀로 간주되지 않습니다. 이 숫자는 세분화 프로세스에 영향을 미칩니다.
- 이미지 매개 변수를 지정합니다.
- 획득 간격(들): 이미지 수집 빈도를 초 단위로 입력합니다.
- 픽셀 크기(μm): 분석을 위해 이미지를 준비할 때 기록된 픽셀 크기를 입력합니다.
- 이미지 비트 깊이: 카메라/감지기의 비트 깊이를 입력합니다.
- 실행 실행을클릭합니다. 오류가 발생하면 스파이더의 콘솔에 오류 메시지가 나타납니다. 그렇지 않으면 이미지 분석 프로세스가 콘솔에 표시됩니다.
참고: 콘솔/플롯 섹션에 표시되는 첫 번째 이미지는 스파이더 설정에 따라 보기 필드에 식별된 모든 셀을 표시합니다. 셀 주위에 배치된 녹색 상자는 세분화 및 분석에 적합한 확산 셀을 나타냅니다. 회색 상자는 분석에 적합하지 않은 셀을 나타냅니다. 확인된 확산 셀의 총 수는 콘솔 탭에도 표시됩니다. 소프트웨어는 셀 영역(파란색) 및 셀 순환(빨간색)을 시간의 함수로 플롯합니다. 이러한 그래프를 통해 사용자는 셀 세분화의 정확도를 평가할 수 있습니다. 성공적인 세분화는 세포 영역에 대해 모노톤 증가 곡선을 생성합니다. 셀 스프레드 영역의 대표적인 곡선을 얻으려면 지연 위상을 그래프에서 수동으로 제거해야 합니다. 지연 단계는 세포가 퍼지기 시작하기 전에 세포 영역의 측정을 포함합니다. 지연 단계는 세포 영역플롯(그림 3C 오른쪽)에서 나타내는 바와 같이 빠른 변동에 의해 표시됩니다.
6. kymographs를 사용하여 세포 확산 시 셀 에지 역학을 정량화
- 분석을 실행하기 전에 세포를 확산하는 원시 영화를 자르면 개별 확산 셀의 시간 시리즈를 만듭니다.
- 피지 도구 모음의 사각형 도구를 사용하여 단일 셀을캡슐화하는 관심 영역(ROI)을 수동으로 선택합니다. ROI가 확산 셀을 완전히 캡슐화하도록 하려면 스크롤 기능을 사용하여 항상 ROI를 검사합니다.)
- ROI를 마우스 오른쪽 버튼으로 클릭하고 중복을선택합니다.
- 중복 스택을 확인하고 확인을 클릭합니다.
- Spyder의 도구 막대에서 열린 파일 버튼을 선택하거나 바로 가기 Ctrl + O를사용하여 기본 분석 스크립트 "cell_spreading_GUI.py"을 엽니 다. GUI가 이미 열려 있는 경우 6.3단계로 직접 이동합니다.
- 상단 패널에서 실행 파일을 선택하거나 바로 가기 F5(그림 3B)를사용하여 셀 확산 분석 GUI를 엽니다.
- Kymograph 생성기 및 분석 탭을 클릭합니다.
- 찾아보기 버튼을 사용하여 분석을 위한 티프 이미지를 선택합니다.
참고: 독점 파일 형식(예: nd2, lif, zen)은 스크립트에서 지원되지 않습니다. - 대상 폴더를 지정하여 출력 데이터(셀 마스크 및 값)를 저장합니다.
- 출력 설정을 지정합니다.
- 데이터 내보내기: 키모그래프의 상대 셀 에지 위치 및 철회 이벤트가 포함된 대상 폴더에 엑셀 스프레드시트(.xlsx)를 내보냅니다.
- 이미지 매개 변수지정:
- 획득 간격(들): 이미지 수집 빈도를 몇 초 만에 입력합니다.
- 픽셀 크기(μm): 5.1.3 단계에서 분석을 위해 이미지를 준비할 때 기록된 픽셀 크기를 입력합니다.
- 가장 작은 세포 영역(μm2):부착의 시작 단계에서 세포의 영역 값을 포함하여 셀 영역에 대한 최소 값을 입력합니다. 이 임계값보다 작은 영역을 가진 객체는 확산 셀로 간주되지 않습니다. 이 숫자는 세분화 프로세스에 영향을 미칩니다.
- 이미지 비트 깊이: 카메라/감지기의 비트 깊이를 입력합니다.
- 실행 실행을클릭합니다. 오류가 발생하면 스파이더의 콘솔에 오류 메시지가 나타납니다. 그렇지 않으면 돌출 역학 정량화에 대한 요약이 콘솔에 표시됩니다. 제거 주파수 및 돌출 속도 측정의 4 쌍이있을 것입니다, 이는 셀의 상단, 하단, 왼쪽 및 오른쪽 부분에서 생성 된 4 kymographs에서 추출된다. 철회 빈도는 다음과 같이 계산됩니다.
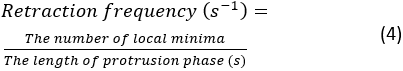
참고: 이 숫자는 라멜리포디포디움이 확산되는 과정에서 얼마나 자주 후퇴하는지 보여줍니다. 평균 돌출 속도는 돌출의 시작과 kymograph의 고원 점 사이의 경사로 측정됩니다. 요약 kymograph 그림은 분할 후 콘솔에 표시됩니다. 요약 그림을 저장하려면 그림을 마우스 오른쪽 단추로 클릭하고 이미지를 저장합니다.
결과
위의 프로토콜은 확산 세포의 라이브 셀 이미징에 대한 실험 절차와 세포 확산 역학의 정량적 분석을위한 계산 도구를 설명합니다. 상기 계산 도구는 낮은 또는 고처리량 형식으로 사용될 수 있으며, 세포 앞가장자리에서 액틴 중합 기계류를 조절하는 분자 플레이어를 식별할 수 있다.
실험 절차의 회로도 표현은 도 1에묘사된다. 세포 확산 분석은
토론
설명된 세포 확산 분석법은 대부분의 세포 확산프로토콜(19,24)에서누락된 특징인 형태학적 변화(예를들어, 세포 크기 및 형상) 및 세포 에지운동(즉, 돌출 속도 및 회수 주파수)의 지속적인 추적을 허용한다. 일반적으로 사용되는 종점 세포 확산 은 세포 확산 속도의 측정을 허용하지만, 이러한 아사약은 셀 에지 운동의 시간 역학을 해결하?...
공개
저자는 공개 할 것이 없습니다.
감사의 말
이 작품은 S.P.에 Connaught 기금 새로운 조사상에 의해 지원되었다, 혁신을위한 캐나다 재단, NSERC 디스커버리 보조금 프로그램 (보조금 RGPIN-2015-05114 및 RGPIN-2020-05881), 맨체스터 대학과 토론토 대학 공동 연구 기금, 토론토 XSeed 프로그램 대학.
자료
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
참고문헌
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유