A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ניתוח כמותי של דינמיקת קצה התא במהלך הפצת תאים
* These authors contributed equally
In This Article
Summary
בפרוטוקול זה, אנו מציגים את ההליכים הניסיוניים של בדיקה מתפשטת של תא המבוססת על מיקרוסקופיה של תאים חיים. אנו מספקים כלי חישובי קוד פתוח עבור פילוח משוחד של תאים בעלי תווית פלואורסצנטית וניתוח כמותי של דינמיקת lamellipodia במהלך התפשטות התא.
Abstract
הפצת תאים היא תהליך דינמי שבו תא המושעה במדיה מתחבר למצע ומשטח את עצמו מצורה מעוגלת לצורה דקה ומתפשטת. בעקבות הקובץ המצורף למצע התא, התא יוצר גיליון דק של lamellipodia הנובע מגוף התא. ב lamellipodia, אקטין כדורי (G-actin) מונומרים polymerize לתוך אקטין סיבי צפוף (F-actin) רשת שדוחף נגד קרום הפלזמה, ובכך לספק את הכוחות המכניים הדרושים עבור התא להתפשט. יש לציין כי השחקנים המולקולריים השולטים בפולמורליזציה של האקטין ב- lamellipodia חיוניים לתהליכים תאיים רבים אחרים, כגון נדידת תאים ואנדוציטוזיס.
מאז הפצת תאים טופס lamellipodia רציפה המשתרעת על פני הפריפריה התא כולו בהתמדה להתרחב כלפי חוץ, תא מתפשט מבחנים הפכו כלי יעיל כדי להעריך את הקינטיקה של בליטות lamellipodial. למרות מספר יישומים טכניים של התא מתפשט assay פותחו, תיאור מפורט של זרימת העבודה, אשר יכלול הן צעד אחר צעד פרוטוקול וכלים חישוביים לניתוח נתונים, חסר כעת. כאן אנו מתארים את ההליכים הניסיוניים של התא המתפשט ומציגים כלי קוד פתוח לניתוח כמותי ובלתי משוחד של דינמיקת קצה התא במהלך ההתפשטות. בשילוב עם מניפולציות תרופתיות ו/או טכניקות להשתקת גנים, פרוטוקול זה נוח למסך בקנה מידה גדול של שחקנים מולקולריים המסדירים בליטות lamellipodial.
Introduction
בליטות Lamellipodial הם מבנים cytoskeletal בולטים שנוצרו בחזית תא נודד. ב lamellipodia, פולמריזציה של actin בעזרת קומפלקס Arp2/3 ו formins יוצר רשת actin מסועפת הגדלה במהירות שדוחפת נגד קרום הפלזמה1,2. הכוח הדוחף שנוצר על ידי רשת actin מניע פיזית את התא קדימה1,3,4,5. דלדול של Arp2/3 מורכב או שיבוש של נתיבי איתות חיוניים עבור בליטות lamellipodial לעתים קרובות לפגוע נדידת תאים6, 7. למרות ההגירה של תאים לקויים lamellipodia דווחה גם8,9, החשיבות של lamellipodia בנדידת תאים ניכרת כמו דלדול של מבנה זה protrusive מפריע ליכולתו של התא לנוע דרך microenvironments ביולוגי מורכב6,10.
מכשול מרכזי להבנת הרגולציה של lamellipodia בתאים נודדים הוא השונות הטבעית בקינטיקה בליטה lamellipodial, גודל, וצורה11,12,13,14. יתר על כן, מחקרים שנעשו לאחרונה הראו כי lamellipodia להפגין התנהגויות בולטות מורכבות, כולל תנודות, תקופתיות, והאצת בליטות14,15. בהשוואה lamellipodia משתנה מאוד של תאים נודדים6,16, lamellipodia נוצר במהלך התפשטות התא הם אחידים יותר12. מאז הפעילות הבולטת של הפצה והעברת תאים מונעת על ידי הרכבות מקרומולקולריות זהות, הכוללות רשת actin מסועפת, חבילות actomyosin התכווצות, הידבקויות מטריצת תאים מבוססי integrin17,18, תאים מתפשטים שימשו נרחב כמודל לחקירת הרגולציה של דינמיקה lamellipodia.
הפצת תאים היא תהליך מכני דינמי שבו תא בהשעיה תחילה דבק מצע באמצעות הידבקויות מבוססות אינגרין17,19,20 ולאחר מכן מתפשט על ידי הרחבת בליטות מבוססות actin21,22,23. במהלך שלב ההתפשטות, lamellipodia הנובעים מגוף התא בולטים isotropically בהתמדה עם מעט עד ללא נסיגה או השתהות12. הפרוטוקולים הנפוצים ביותר להפצת תאים הם מבחני קצה, שבהם תאים מתפשטים קבועים בזמנים שונים לאחר ציפוי19,24. מבחנים אלה, אם כי מהירים ופשוטים, מוגבלים בכוח האבחון שלהם כדי לזהות שינויים בתכונות הדינמיות של lamellipodia. כדי לקבוע את המנגנונים המולקולריים השולטים בדינמיקה של לאמליפודיה, קבוצת Sheetz הייתה חלוצה בשימוש בניתוח כמותי של תאים מתפשטים חיים וחשפה תכונות בסיסיות רבות של בליטות קצה התא11,12,22. מחקרים אלה הראו כי התסעפות המתפשטת של תאים חיים היא טכניקה חזקה ורבת עוצמה בארגז הכלים של מעבדה לביולוגיה של התא. למרות זאת, פרוטוקול מפורט וכלי חישובי בקוד פתוח לבדיקה מתפשטת של תאים חיים אינם זמינים כיום עבור הקהילה לביולוגיה של התא. למטרה זו, הפרוטוקול שלנו מתאר את ההליכים של הדמיה תאים מתפשטים חיים ומספק כלי ניתוח תמונה אוטומטי. כדי לאמת שיטה זו, השתמשנו בעיכוב Arp2/3 כטיפול ניסיוני והראינו כי עיכוב הפונקציה של קומפלקס Arp2/3 לא עצר את התפשטות התאים אלא גרם לירידה משמעותית במהירות בליטות התא, כמו גם ליציבות של בליטות קצה התא, מה שהוליד קצוות תא משוננים. נתונים אלה מראים כי השילוב של הדמיה של תאים חיים וניתוח תמונה אוטומטי הוא כלי שימושי לניתוח דינמיקה של קצה התא ולזיהוי רכיבים מולקולריים המווסתים lamellipodia.
Protocol
1. זריעת תאים
הערה: פרוטוקול התפשטות התא המתואר בוצע באמצעות פיברובלסטים עובריים של העכבר (MEFs) המבטאים PH-Akt-GFP (סמן פלואורסצנטי עבור PIP3/PI(3,4)P2). קו תא זה נוצר על ידי שילוב גנומי של מבנה ביטוי עבור PH-Akt-GFP (addgene #21218) על ידי עריכת גנים בתיווך CRISPR. עם זאת, סמנים פלואורסצנטיים אחרים המתבטאים באופן ארעי או משולב בגנום יכולים לשמש גם במדורה זו. לפילוח תמונה אופטימלי, אנו ממליצים להשתמש בסמנים פלואורסצנטיים המופצים באופן שווה בציטופלסמה, למשל, GFP ציטוזולית.
- תרבות צלחת 10 ס"מ של תאים ל 90% מפגש.
- לאחר שהתאים השיגו את המפגש הנכון, הנח כיסוי של 22 מ"מ x 22 מ"מ (עובי #1.5; 0.17 מ"מ) לתבשיל תרבית תאים 35 מ"מ. מעיל את הכיסוי עם 400 μL של פיברונקטין כי כבר מדולל ב PBS לריכוז הסופי של 2.5 מיקרוגרם / מ"ל.
הערה: מספר הכיסויים הנדרשים לבדיקה נקבע לפי מספר התנאים הניסיוניים וההעתק הטכני. - מניחים את צלחת 35 מ"מ עם כיסוי מצופה fibronectin לתוך 37 °C (60 °F), 5% CO2 חממה במשך 1 שעה.
- מוציאים את המנה עם הכיסוי מהחממה. לשאוף את fibronectin ולשטוף את הכיסוי עם PBS על ידי צינור בעדינות סביב מכסה פעמיים עד שלוש פעמים.
- לשאוף את התקשורת תרבות התא מן המנה 10 ס"מ של תאים לשטוף את המנה עם PBS.
- הוסף 650 μL של 0.05% טריפסין-EDTA לצלחת 90% confluent של תאים, הטיית המנה כדי להפיץ את האנזים באופן שווה. מניחים את המנה עם טריפסין לתוך האינקובטור במשך דקה אחת.
- מוציאים את המנה עם התאים מהחממה. הוסף 10 מ"ל של מדיה תרבות התא לתוך צינור צנטריפוגה 15 מ"ל. במהירות להוסיף עוד 10 מ"ל של מדיה לתוך המנה כדי להרוות את טריפסין.
- פיפטה 1 מ"ל של התאים טריפסיניזציה לתוך צינור צנטריפוגה 15 מ"ל על מנת לדלל את התאים. פיפטה את התוכן של הצינור למעלה ולמטה כדי להבטיח הפצה שווה של תאים בתוך התקשורת. עבור סוגי תאים עם נטייה גבוהה לצבירה, סינון תאים באמצעות מסננת תאים (גודל רשת שינוי של 100 מיקרומטר) מומלץ למזער את המופע של גוש תאים.
- מן הצינור, פיפטה 500 - 1000 μL של תאים מדוללים לתוך צלחת 35 מ"מ המכיל את coverslip.
- בעדינות לנער את המנה כדי להפיץ את התאים באופן שווה. ודא שהכיסוי נמצא במפגש של ~ 10% (~ 50,000 תאים / מ"ל) והתאם את עוצמת הקול של תאים מדוללים לפי הצורך.
הערה: המטרה של תאים במפגש כה נמוך היא להבטיח שיש 1-2 תאים מקוטבים בכל שדה ראייה שישמשו כדי למקד את המטרה לאורך כל הרכישה המתפשטת של התא. - מעבר 1/5 של התאים הנותרים בצלחת 10 ס"מ לתוך צלחת אחת 6 ס"מ לכל מצב טיפול. מניחים את הכלים המעבר ואת צלחת 35 מ"מ עם כיסוי לתוך האינקובטור לילה.
הערה: אלה יהיו התאים שינותחו להפצת דינמיקה.
2. דגירה של סמים והתאוששות תאים
- הוסף 5 מ"ל של מדיה תרבות התא לתוך כל אחד משני צינורות צנטריפוגה 15 מ"ל, ו 20 מ"ל של פנול אדום חינם DMEM לתוך כל אחד משני צינורות צנטריפוגה 50 מ"ל.
הערה: מספר זוגות הצינור (15 מ"ל + 50 מ"ל) צריך להתאים למספר התנאים הניסיוניים. - כדי לבדוק את החשיבות של Arp2/3 להתפשטות תאים, פיפטה או מעכב תרופתי של Arp2/3, CK-666, או טיפול הבקרה, כגון DMSO, לתוך כל זוג של צינורות צנטריפוגה עד הריכוז הרצוי.
- מוציאים את הכלים 6 ס"מ (ראו שלב 1.11) מהחממה ושואבים את התקשורת. לשטוף את הכלים עם PBS חם.
- הוסף את התוכן של CK-666- או DMSO בתוספת 15 צינורות צנטריפוגה מ"ל לתוך כל אחד הכלים מעבר. מתייגים כל מנה עם הטיפול התרופתי הנכון ומניחים את הכלים באינקובטור למשך שעה.
- מוציאים את הכלים מהחממה ושואבים את התקשורת. לשטוף את הכלים עם PBS חם על מנת להסיר ביסודיות את כל המדיה האדומה פנול הנותרים.
- מוסיפים 230 μL של 0.05% טריפסין-EDTA לכל מנה 6 ס"מ ותאי דגירה במשך דקה אחת.
הערה: אם ישים, טריפסין יכול להיות מוחלף על ידי חוסם הידבקות תאים שאינו פרוטאוליטי. - מוציאים את הכלים מהחממה. עבור כל טיפול, להוסיף 5 מ"ל של DMEM ללא אדום פנול בתוספת סמים לתוך צינור צנטריפוגה 15 מ"ל המיועד "Tube B". הוסף 5 מ"ל נוספים של אותה מדיה לתוך המנה הרלוונטית כדי להרוות את טריפסין. מעבירים את תכולת המנה לצינור צנטריפוגה של 15 מ"ל המוגדר כ"שפופרת A".
- העבר 1 מ"ל של תאים מצינור A לתוך Tube B. חזור על כל טיפול.
- מניחים צינורות A ו- B לתוך האינקובטור במשך 45 דקות כדי לאפשר לתאים להתאושש טריפסיניזציה. שחררו מעט את המכסה של צינורות הצנטריפוגה לפני שהם מניחים אותם באינקובטור כדי לאפשר חדירת CO2.
הערה: משך זמן ההחלמה עשוי להשתנות עבור סוגי תאים שונים. אמנם בניסויים שלנו התאוששות באורך 45 דקות הייתה השפעה זניחה על הכדאיות של התא, סוגים מסוימים של תאים עשויים לעבור anoikis כאשר נשמר בהשעיה במשך זמן רב מדי. לכן, אנו ממליצים לקבוע את זמן ההחלמה האופטימלי באופן אמפירי. זמן ההחלמה האופטימלי מאפשר התפשטות תאים מהירה וסנכרונית ללא תאים מתים או אפופטיים במדגם.
3. הכנה קאמרית מגנטית
- ודא כי כל החלקים של תא 1 גם Chamlide תא מגנטי שיכול להכיל כיסוי 22 מ"מ x 22 מ"מ מרובע נוקו לפני השימוש.
- מוציאים את מנת ה-35 מ"מ עם הכיסוי (ראו שלב 1.11) מהחממה. שאפו את מדיית תרבות התאים ושטפו את הכיסוי עם PBS חם.
- מוציאים את הכיסוי מהצלחת 35 מ"מ באמצעות זוג מלקחיים ומניחים בעדינות את הכיסוי על הצלחת התחתונה של התא המגנטי.
- מניחים את אטם הסיליקון על גבי הכיסוי.
הערה: אטם סיליקון ממוקם בצורה לא נכונה הוא הגורם השכיח ביותר לתא מגנטי דולף. ודא כי האטם נשען על כניסתו של הלוח התחתון ואינו עולה מעבר לשקע. - מחברים את הגוף הראשי לצלחת התחתונה.
הערה: עשה את החלק הזה לאט מאוד. עצה טובה היא להחזיק את הצלחת התחתונה ביד אחת תוך הנחת הגוף העיקרי על גבי. זה מבטיח כי המגנטים של הגוף הראשי לא להרים את הצלחת התחתונה למעלה, אשר עלול לעקור ולפצח את הכיסוי. - הוסף 1 מ"ל של DMEM ללא סמים פנול אדום לתא המגנטי. קח רקמה ללא מוך בזהירות לטבול את המתחם בין הגוף הראשי ואת הצלחת התחתונה על מנת לבדוק את כל דליפות.
הערה: אם יש דליפה, שאפו במהירות את המדיה והמשיכו שוב משלב 3.4. - מנמיכים את הכיסוי השקוף על הגוף הראשי כדי להקיף את התא המגנטי.
- לרסס רקמת מעבדה תחילה במים ולנגב את החלק התחתון של התא המגנטי (כיסוי, לא את החלק מתכת). לאחר מכן, לרסס רקמת מעבדה שנייה עם כמות קטנה של 70% אתנול ולנגב, נזהר לא לפצח את הכיסוי.
4. רכישת תמונה
- מחממים את החממה העליונה של הבמה ואת התנור האובייקטיבי ל-37 מעלות צלזיוס ומגדירים את רמתה-CO 2 באינקובטור העליון של הבמה ל-5%.
הערה: אם החממה העליונה בשלב אינו מחובר לאספקת CO2, יש להשלים את מדיית תרבות התאים עם 25 mM HEPES כדי לשמור על pH קבוע 7.4. - החל כמות מספקת של שמן טבילה על 60X מחומם מראש, 1.4 N.A. שמן המטרה.
הערה: אנו משתמשים במטרה טבילה בשמן 60X, 1.4 N.A. בפרוטוקול זה בגלל שדה הראייה הגדול למדי שלה ויעילות איסוף אור יוצאת מן הכלל. אם נדרש שדה תצוגה גדול יותר, ניתן להשתמש במטרת הגדלה נמוכה יותר (למשל, 20x) כל עוד יחס האות לרעש של התמונות גדול מ- 2.5. - הביאו הן את התא המגנטי שהושלם והן את Tube B (שלב 2.9) למיקרוסקופ הקונפוקל. מניחים את התא המגנטי על האינקובטור העליון של הבמה.
הערה: מניחים את התא המגנטי בעדינות על הבמה כדי למנוע יצירת בועות בשמן הטבילה. - הגדר את המוקד לתאי הפלואורסצנט באמצעות ערוץ GFP. ודא שקצה התא חד ומוגדר היטב.
- הסר את הכיסוי השקוף של התא המגנטי ופיפט 500 μL מצינור B לתוך התא המגנטי. מניחים את הכיסוי השקוף בחזרה על גבי התא המגנטי.
- כדי לזהות תאים אידיאליים לניתוח הפצת תאים, חפש "הילות" של תאים שעדיין לא נקשרו לכיסוי אך אינם מתגלגלים עוד. תאים הנמצאים בשלבים המוקדמים ביותר של חיבור coverslip הם גם מועמדים נהדרים, אבל רכישת תמונה חייבת להיות מהירה על מנת ללכוד התפשטות.
- קבע את תצורת רכישת התמונה לשגות בזמן עבור הערוץ הירוק כך שיכלול ארבעה שדות תצוגה, בתמונה במרווחי זמן של 6 שניות.
הערה: בשל השונות הגבוהה של מהירות בליטה lamellipodia בין סוגי תאים שונים, קצב המסגרות האופטימלי צריך להיקבע באופן אמפירי. מרווח ההדמיה של 6 שניות בשימוש בניסויים שלנו הוא נקודת התחלה טובה לניתוח של תאים mesenchymal ואפיתל רבים. עם זאת, תאים המתפשטים מהר מאוד(למשל,תאי חיסון) עשויים לדרוש קצב פריימים גבוה בהרבה (מרווח הדמיה קצר יותר). קצב הפריימים האופטימלי לסרטים המתפשטים על-ידי תאים מבטיח תזוזה של 2-5 פיקסלים מקצה התא הבולט בין המסגרות הבאות. בהתחשב בדיוק של התאמת עקומה המשמשת לזיהוי הרמה של התפשטות תאים, קצב הפריימים האופטימלי צריך גם להבטיח 50-100 מדידות של תזוזת קצה התא במהלך שלב ההתפשטות המהיר של התפשטות התא. יש להתאים את מספר שדות הראייה בהתאם לזמן החשיפה, המרחק בין נקודות הרכישה ומהירות תנועת הבמה. מומלץ למשתמשים לקבוע את המספר המרבי של שדות תצוגה שניתן לרכוש באמצעות קצב הפריימים הרצוי. - לאחר זיהוי שדה ראייה מתאים, שמרו את הקואורדינטות X ו- Y של שלב המיקרוסקופ. המשך בזיהוי שלושה שדות תצוגה אחרים הקרובים יחסית זה לזה על ה- coverslip. שמור את הקואורדינטות של שלב המיקרוסקופ עבור כל שדה ראייה רצוי.
הערה: מומלץ מאוד למטב את נתיב תנועת הבמה בין שדות הראייה על מנת למזער כל תנועת מדגם מיותרת. אופטימיזציה כזו יכולה להתבצע באופן ידני או אוטומטי. תנועת מדגם מוגזמת מאטה את הרכישה ועלולה לגרום לתאים להתגלגל מחוץ לתצוגה כשהם יורדים. - רכוש תמונות למשך 15 דקות בקצב פריימים של 6 שניות ושמור את הקבצים. אם נדרשות רכישות נוספות, חזור על הפעולה החל משלב 4.6.
5. ניתוח של אזור התא, מעגליות ודינמיקת בליטה במהלך הפצת תאים
- הכנת תמונות לעיבוד וניתוח נתונים
הערה: התוכנה דורשת תמונה בפורמט .tiff וגודל פיקסל כפרמטרים של הקלט. ניתן למלא את שתי הדרישות באמצעות תוכנת הרכישה או פיג'י (בפרוטוקול זה). אם דרישות אלה מתקיימות, המשך לשלב 5.2.- התקן את הגירסה העדכנית ביותר של יישום פיג'י (https://imagej.net/Fiji/Downloads).
- פתח תמונה לשגות בזמן באמצעות פיג'י.
- העתיקו את גודל הפיקסל של התמונה באמצעות בחירה באפשרות 'מאפייני תמונה >'. העתק והדבק את גודל הפיקסל ב- μm לפנקס רשימות/Word.
- לניתוח אזור התפשטות התא והמחזוריות, שמרו את התמונה לשגות בזמן כערימה של תמונת tiff. תוכנת ניתוח הבנייה המותאמת אישית אינה תומכת בתבניות קובץ קנייניות. שמור את מחסנית התמונה בודדת של tiff של תאים על-ידי בחירה באפשרות > שמירה בשם >- Tiff.
- התקן את IDE פייתון (Spyder) ואת החבילות הדרושות (PySimpleGUI ו tifffile) לעיבוד וניתוח נתונים.
הערה: ההתקנה של פייתון וחבילות נדרשת רק עבור ההתקנה הראשונית.- הסרטים לשגות זמן ינותח ספיידר IDE באמצעות תסריט פייתון לבנות בהתאמה אישית. כדי להוריד את ספיידר IDE, להוריד את מפיץ אנקונדה (https://www.anaconda.com/products/individual) הכולל Spyder IDE ואת רוב הספריות הדרושות וחבילות עבור ניתוח זה.
- התקן את אנקונדה ושגר את ספיידר דרך נווט אנקונדה.
- בכרטיסיית המסוף IPython (הממוקמת בחלק השמאלי התחתון של Spyder), העתק והדבק את הפקודה הבאה: pip install PySimpleGUI והקש על מקש enter. הפעלת פקודה זו תתקין את החבילה הדרושה לאתחול ממשק המשתמש הגרפי (GUI).
- באותו מסוף, העתק והדבק את הפקודה הבאה: pip install tifffile והקש על מקש Enter. הפעלת פקודה זו תתקין את החבילה הדרושה לשמירת תמונות כקבצי tiff.
- הורד את כל סקריפטי פייתון מהקבצים המשלימים או את הסקריפטים המעודכנים ביותר מ- GitHub at: https://github.com/ernestiu/Cell-spreading-analysis.git
- כימות גורמי אזור תא וצורת תא במהלך הפצת תאים
- פתח את סקריפט הניתוח הראשי "cell_spreading_GUI.py" על ידי בחירת אפשרות הקובץ הפתוח בחלונית העליונה של Spyder או באמצעות קיצור הדרך Ctrl + O.
- פתח את ממשק המשתמש הגרפי של ניתוח הפצת התאים על-ידי בחירה באפשרות "הפעל קובץ" בחלונית העליונה או באמצעות קיצור הדרך F5.
- לחצו על הכרטיסייה 'אזור התפשטות תא' (איור 3A).
- בחר את תמונת ה- tiff שיש לנתח באמצעות לחצן עיון.
הערה: הקובץ שנבחר חייב להיות קובץ tiff. - ציין את ספריית היעד שבה יישמרו פלטי נתונים (למשל, מסיכות תאים, ערכים).
- ציין את הגדרות פלט הנתונים:
- שמירת מסיכות: שמור מסיכות תא שנוצרו במהלך תהליך הפילוח.
- ייצוא נתונים: יצא גיליון אלקטרוני של Excel (.xlsx) המכיל את כל נתוני הניתוח לתיקיית היעד.
אזור תא, מעגליות ויחס גובה-רוחב של כל התאים המתפשטים יישמרו כגיליון אלקטרוני של Excel בתיקיית היעד. אזור התא מחושב כ: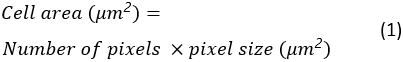
מעגליות התא היא מדד לכמה קרוב התא לתא עגול לחלוטין. הוא מחושב כ: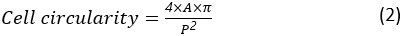
כאשר A ו- P הם אזור התא והיקף התא, בהתאמה. יחס הגובה-רוחב של התא מייצג את גודל התא המוארך. לתא מתפשט צריך להיות יחס גובה-רוחב קרוב ל- 1. יחס הגובה-רוחב מחושב כ: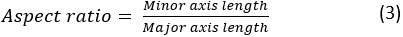
- שמירת קווי מתאר: שמור את קווי המתאר של גבול התא שכבת-על של תמונות בתיקיית היעד.
- ציון הגדרות הפילוח
- הצג פילוח: הצג את תוצאת הפילוח במסוף Spyder במהלך תהליך הניתוח.
- אזור התא הקטן ביותר (μm2): הזן את הערך המינימלי עבור אזור התא, כולל ערכי אזור של תאים בשלבים ההתחלה של הקובץ המצורף. אובייקטים עם שטח קטן מסף זה לא ייחשבו כתאים מתפשטים. מספר זה ישפיע על תהליך הפילוח.
- ציין את פרמטרי התמונה.
- מרווח זמן לרכישה (ים): הזן את תדירות רכישת התמונה בשניות.
- גודל פיקסל (μm): הזן את גודל הפיקסל שנרשם בעת הכנת תמונות לניתוח.
- עומק סיבית תמונה: הזן את עומק הסיביות של המצלמה/הגלאי.
- לחץ על הפעל. אם מתעוררת שגיאה, תופיע הודעת שגיאה במסוף של Spyder. אחרת, תהליך ניתוח התמונה יוצג במסוף.
הערה: התמונה הראשונה שתופיע במקטע מסוף/התוויית נתונים (בהתאם להגדרות Spyder) מציגה את כל התאים שזוהו בשדה התצוגה. תיבות ירוקות הממוקמות סביב התאים מציינות תאים מתפשטים המתאימים לפילוח וניתוח. תיבות אפורות מציינות תאים שאינם מתאימים לניתוח. המספר הכולל של תאים מתפשטים שזוהו יופיע גם בכרטיסיה מסוף. התוכנה מתווה את אזור התא (בכחול) ואת מעגליות התא (באדום) כפונקציה של זמן. גרפים אלה מאפשרים למשתמשים להעריך את הדיוק של פילוח תאים. פילוח מוצלח מניב עקומה הגדלה באופן מונוטוני עבור אזור התא. כדי להשיג עקומה מייצגת של אזור התפשטות התא, יש להסיר את שלב ההשהיה מהגרף באופן ידני. שלב ההשהיה כולל מדידות של אזור התא לפני שהתא מתחיל להתפשט. שלב ההשהיה מצוין על-ידי תנודות מהירות, כפי שמיוצגות בעלילה של אזור התא(איור 3C מימין).
6. לכמת את הדינמיקה של קצה התא במהלך הפצת תאים באמצעות קימוגרפים
- לפני הפעלת הניתוח, חתוך את הסרטים הגולמיים של תאים מתפשטים כדי ליצור סדרות זמן של תאים מתפשטים בודדים.
- השתמש בכלי מלבן בסרגל הכלים של פיג'י כדי לבחור באופן ידני אזור מעניין (ROI) המכמס תא בודד. (כדי להבטיח שההחללה תקציר מלא של התא המתפשט, השתמש בפונקציית הגלילה כדי לבדוק את ההחזר על ההשקעה בכל נקודות הזמן).
- לחץ באמצעות לחצן העכבר הימני על ההחזר על ההשקעה ובחר שכפל.
- בדוק מחסנית כפולה ולחץ על אישור.
- פתח את קובץ ה- Script הראשי של הניתוח "cell_spreading_GUI.py" על-ידי בחירה בלחצן פתח קובץ בסרגל הכלים של Spyder או באמצעות קיצור הדרך Ctrl + O. אם ממשק ה- GUI כבר נפתח, עבור ישירות לשלב 6.3.
- פתחו את ממשק המשתמש הגרפי של ניתוח הפצת התאים באמצעות בחירה באפשרות 'הפעל קובץ' בחלונית העליונה או באמצעות קיצור הדרך F5 (איור 3B).
- לחץ על הכרטיסיה מחולל & ניתוח קימוגרף.
- השתמש בלחצן עיון כדי לבחור את תמונת ה- tiff עבור הניתוח.
הערה: תבניות קובץ קנייניות, למשל, nd2, lif, zen, אינן נתמכות על-ידי קובץ ה- Script. - ציין את תיקיית היעד לשמירת נתוני הפלט (מסיכות וערכים של תאים).
- ציין את הגדרות הפלט.
- ייצוא נתונים: יצא גיליון אלקטרוני של Excel (.xlsx) לתיקיית היעד המכילה מיקומי קצה תא יחסיים ואירועי נסיגה של הקימוגרפים.
- ציין את פרמטרי התמונה:
- מרווח זמן לרכישה (ים): הזן את תדירות רכישת התמונה בשניות.
- גודל פיקסל (μm): הזן את גודל הפיקסל שנרשם בעת הכנת תמונות לניתוח בשלב 5.1.3.
- אזור התא הקטן ביותר (μm2): הזן את הערך המינימלי עבור אזור התא, כולל ערכי אזור של תאים בשלבים ההתחלה של הקובץ המצורף. אובייקטים עם שטח קטן מסף זה לא ייחשבו כתאים מתפשטים. מספר זה ישפיע על תהליך הפילוח.
- עומק סיבית תמונה: הזן את עומק הסיביות של המצלמה/הגלאי.
- לחץ על הפעל. אם מתעוררת שגיאה, תופיע הודעת שגיאה במסוף של Spyder. אחרת, סיכום של כימותי דינמיקת הבלטה יוצג במסוף. יהיו 4 זוגות של מדידות תדר נסיגה ומהירות בליטה, אשר מופקים 4 kymographs שנוצר החלק העליון, התחתון, השמאלי והימני של התא. תדירות הנסיגה מחושבת כ:
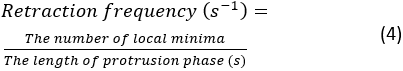
הערה: מספר זה מדגים באיזו תדירות הלמליפודיום נסוג במהלך ההתפשטות. מהירות הבליטה הממוצעת נמדדת על ידי השיפוע בין תחילת הבליטה לנקודת הרמה בקימוגרף. דמות קימוגרף מסכמת תוצג במסוף לאחר הפילוח. כדי לשמור את דמות הסיכום, לחץ באמצעות לחצן העכבר הימני על האיור ושמור את התמונה.
תוצאות
הפרוטוקול לעיל מתאר את ההליכים הניסיוניים להדמיית תאים חיים של תאים מתפשטים וכלי חישובי לניתוח כמותי של דינמיקת הפצת תאים. הכלי החישובי יכול לשמש בפורמט בעל תפוקה נמוכה או גבוהה כדי לזהות את השחקנים המולקולריים המסדירים את מכונות הפולמליזציה של האקטין בקצה המוביל של התא.
?...
Discussion
מבחני התפשטות התא המתוארים מאפשרים מעקב מתמשך אחר שינויים מורפולוגיים(למשל, גודל וצורה של תאים) ותנועות קצה תא(כלומר, מהירות בליטה ותדירות נסיגה), שהן תכונות חסרות ברוב הפרוטוקולים המתפשטים על ידי תאים19,24. בעוד תא סוף נפוץ מתפשט מבחנים מאפשרים קבי?...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי קרן קונוט פרס חוקר חדש S.P., קרן קנדה לחדשנות, NSERC דיסקברי גרנט התוכנית (מענקים RGPIN-2015-05114 ו RGPIN-2020-05881), אוניברסיטת מנצ'סטר ואוניברסיטת טורונטו קרן מחקר משותף, ואוניברסיטת טורונטו XSeed תוכנית.
Materials
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
References
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved