Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse quantitative de la dynamique des bords cellulaires pendant la propagation cellulaire
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Dans ce protocole, nous présentons les procédures expérimentales d’un essai de propagation cellulaire qui est basé sur la microscopie à cellules vivantes. Nous fournissons un outil de calcul open-source pour la segmentation impartiale des cellules étiquetées fluorescentement et l’analyse quantitative de la dynamique de lamellipodia pendant la propagation cellulaire.
Résumé
La propagation cellulaire est un processus dynamique dans lequel une cellule suspendue dans le corps se fixe à un substrat et s’aplatit d’une forme arrondie à une forme mince et étalée. Après l’attachement cellule-substrat, la cellule forme une mince feuille de lamellipodie émanant du corps cellulaire. Dans la lamellipodie, les monomères globulaires d’actine (G-actin) polymérisent dans un maillage filamenteux dense d’actine (F-actin) qui pousse contre la membrane plasmatique, fournissant ainsi les forces mécaniques nécessaires à la propagation de la cellule. Notamment, les acteurs moléculaires qui contrôlent la polymérisation de l’actine dans la lamellipodie sont essentiels pour de nombreux autres processus cellulaires, tels que la migration cellulaire et l’endocytose.
Depuis que les cellules qui se propagent forment une lamellipodie continue qui s’étend sur toute la périphérie cellulaire et se dilatent constamment vers l’extérieur, les analyses de propagation cellulaire sont devenues un outil efficace pour évaluer la cinétique des saillies lamellipodial. Bien que plusieurs implémentations techniques de l’analyse de propagation cellulaire aient été développées, il manque actuellement une description détaillée du flux de travail, qui comprendrait à la fois un protocole étape par étape et des outils informatiques pour l’analyse des données. Ici, nous décrivons les procédures expérimentales de l’analyse de propagation cellulaire et présentons un outil open-source pour l’analyse quantitative et impartiale de la dynamique du bord cellulaire lors de la propagation. Lorsqu’il est combiné avec des manipulations pharmacologiques et / ou des techniques de silencing des gènes, ce protocole est approprié à un écran à grande échelle des acteurs moléculaires régulant les saillies lamellipodial.
Introduction
Les saillies lamellipodial sont des structures cytosquelettiques proéminentes formées à l’avant d’une cellule migrante. Dans la lamellipodia, la polymérisation de l’actine à l’aide du complexe Arp2/3 et des formins crée un maillage d’actine ram ramifère à croissance rapide qui pousse contre la membraneplasmatique 1,2. La force de poussée générée par le maillage actin propulse physiquement la cellule versl’avant 1,3,4,5. L’épuisement du complexe Arp2/3 ou la perturbation des voies de signalisation essentielles aux saillies lamellipodiales nuisent souvent à la migration cellulaire6, 7. Bien que la migration des cellules lamellipodia-déficientes ait égalementété rapportée 8,9,l’importance de la lamellipodia dans la migration cellulaire est évidente car l’épuisement de cette structure saillante perturbe la capacité de la cellule à se déplacer par des microenvironnements biologiquescomplexes 6,10.
Un obstacle majeur à la compréhension de la régulation de la lamellipodia dans les cellules migration est la variabilité naturelle de la cinétique de saillie lamellipodial, la taille et la forme11,12,13,14. En outre, des études récentes ont démontré que la lamellipodia présentent des comportements complexes saillants, y compris fluctuants, périodiques et accélérant les saillies14,15. Par rapport à la lamellipodie très variable des cellules migratives6,16, lamellipodie formée pendant la propagation cellulaire sont plusuniformes 12. Depuis l’activité saillante de la propagation et la migration des cellules est entraînée par des assemblages macromoléculaires identiques, qui comprennent un réseau d’actine ram ramée, faisceaux d’actomyosine contractile, et à base d’intégrine adhérencescellulaires matrice 17,18, cellules de propagation ont été largement utilisés comme un modèle pour étudier la régulation de la dynamique lamellipodia.
La propagation cellulaire est un processus mécanochimique dynamique où une cellule en suspension adhère d’abord à un substrat par des adhérences à base d’intégrine17,19,20, puis se propage en étendant les saillies à base d’actine21,22,23. Pendant la phase de propagation, la lamellipodia émanant du corps cellulaire dépasse isotropiquement et constamment avec peu ou pas de rétractation oud’étalon 12. Les protocoles de propagation cellulaire les plus couramment utilisés sont les analyses de points de terminaison, où les cellules de propagation sont fixées à divers moments aprèsle placage 19,24. Ces analyses, bien que rapides et simples, sont limitées dans leur puissance diagnostique pour détecter des changements dans les dispositifs dynamiques de lamellipodia. Pour déterminer les mécanismes moléculaires qui contrôlent la dynamique de la lamellipodia, le groupe sheetz a été le pionnier de l’utilisation de l’analyse quantitative des cellules vivantes de propagation et a découvert de nombreuses propriétés fondamentales des saillies de bord cellulaire11,12,22. Ces études ont démontré que l’essai de propagation des cellules vivantes est une technique robuste et puissante dans la boîte à outils d’un laboratoire de biologie cellulaire. Malgré cela, un protocole détaillé et un outil de calcul open-source pour un test de propagation des cellules vivantes ne sont actuellement pas disponibles pour la communauté de biologie cellulaire. À cette fin, notre protocole décrit les procédures d’imagerie des cellules de propagation en direct et fournit un outil automatisé d’analyse d’image. Pour valider cette méthode, nous avons utilisé l’inhibition arp2/3 comme traitement expérimental et avons montré que l’inhibition de la fonction du complexe Arp2/3 n’arrêtait pas la propagation cellulaire, mais causait une réduction significative de la vitesse de saillie cellulaire, ainsi que la stabilité des saillies du bord cellulaire, donnant lieu à des bords de cellules déchiquetés. Ces données démontrent que la combinaison de l’imagerie à cellules vivantes et de l’analyse automatisée de l’image est un outil utile pour analyser la dynamique des bords cellulaires et identifier les composants moléculaires qui régulent la lamellipodie.
Protocole
1. Ensemencement cellulaire
REMARQUE : Le protocole décrit de propagation cellulaire a été exécuté utilisant des fibroblastes embryonnaires de souris (MEFs) exprimant PH-Akt-GFP (un marqueur fluorescent pour PIP3/PI(3,4)P2). Cette lignée cellulaire a été générée par l’intégration génomique d’une construction d’expression pour PH-Akt-GFP (Addgene #21218) par l’édition de gènes à 200 200 %. Cependant, d’autres marqueurs fluorescents qui sont exprimés transitoirement ou intégrés dans le génome peuvent également être utilisés dans cet essai. Pour une segmentation optimale de l’image, nous vous recommandons d’utiliser des marqueurs fluorescents qui sont répartis uniformément dans le cytoplasme, par exemple, le GFP cytostolique.
- Culture d’un plat de 10 cm de cellules à 90% confluence.
- Une fois que les cellules ont atteint la confluence appropriée, placez un coverslip de 22 mm x 22 mm (#1,5 ; épaisseur de 0,17 mm) dans un plat de culture cellulaire de 35 mm. Enduire le coverslip de 400 μL de fibronectine diluée dans PBS jusqu’à une concentration finale de 2,5 μg/mL.
REMARQUE : Le nombre de coverslips requis pour l’essai est déterminé par le nombre de conditions expérimentales et la réplique technique. - Placer le plat de 35 mm avec le couvercle recouvert de fibronectine dans un incubateur de 37 °C, 5 % de CO2 pendant 1 heure.
- Retirer le plat avec le coverslip de l’incubateur. Aspirer la fibronectine et laver le coverslip avec PBS en pipetting doucement autour de la coverslip deux à trois fois.
- Aspirer les supports de culture cellulaire à partir du plat de 10 cm de cellules et laver le plat avec PBS.
- Ajouter 650 μL de trypsine-EDTA de 0,05 % au plat confluent de cellules à 90 %, en inclinant le plat pour répartir uniformément l’enzyme. Placer le plat avec la trypsine dans l’incubateur pendant 1 minute.
- Retirer le plat avec les cellules de l’incubateur. Ajouter 10 mL de culture cellulaire dans un tube de centrifugeuse de 15 mL. Ajouter rapidement 10 mL de médias dans le plat pour étancher la trypsine.
- Pipette 1 mL des cellules trypsinisées dans le tube centrifugeuse de 15 mL afin de diluer les cellules. Pipette le contenu du tube de haut en bas pour assurer une distribution égale des cellules dans les médias. Pour les types de cellules à forte propension à l’agrégation, il est recommandé de filtrer les cellules à travers une passoire cellulaire (taille de maille de 100 μm) afin de minimiser l’occurrence de l’agglutination cellulaire.
- Du tube, pipette 500 - 1000 μL de cellules diluées dans le plat de 35 mm contenant le coverslip.
- Agiter doucement le plat pour répartir uniformément les cellules. Assurez-vous que le coverslip est à une confluence d’environ 10 % (~50 000 cellules/mL) et ajustez le volume des cellules diluées au besoin.
REMARQUE : Le but d’avoir des cellules à une confluence si faible est de s’assurer qu’il y a 1-2 cellules polarisées dans chaque champ de vision qui seront utilisées pour concentrer l’objectif tout au long de l’acquisition de propagation cellulaire. - Passage 1/5 des cellules restantes dans le plat de 10 cm dans un plat de 6 cm par condition de traitement. Placez les plats de passage et le plat de 35 mm avec le coverslip dans l’incubateur pendant la nuit.
REMARQUE : Ce sont les cellules qui seront analysées pour la dynamique de propagation.
2. Incubation de médicaments et récupération cellulaire
- Ajouter 5 mL de culture cellulaire dans chacun des deux tubes de centrifugeuse de 15 mL et 20 mL de DMEM sans phénol dans chacun des deux tubes de centrifugeuse de 50 mL.
REMARQUE : Le nombre de paires de tubes (15 mL + 50 mL) doit correspondre au nombre de conditions expérimentales. - Pour tester l’importance de l’Arp2/3 pour la propagation cellulaire, pipette soit l’inhibiteur pharmacologique d’Arp2/3, CK-666, ou le traitement de contrôle, comme DMSO, dans chaque paire de tubes de centrifugeuse jusqu’à la concentration désirée.
- Retirer les plats de 6 cm (voir étape 1.11) de l’incubateur et aspirer les supports. Laver la vaisselle avec du PBS chaud.
- Ajouter le contenu des tubes de centrifugeuse CK-666- ou DMSO-complétés de 15 mL dans chacun des plats adoptés. Étiquetez chaque plat avec le traitement médicamenteux correct et placez les plats dans l’incubateur pendant une heure.
- Retirer la vaisselle de l’incubateur et aspirer les supports. Laver la vaisselle avec du PBS chaud afin d’enlever soigneusement tous les supports rouges de phénol restants.
- Ajouter 230 μL de trypsine-EDTA de 0,05 % à chaque plat de 6 cm et incuber les cellules pendant 1 minute.
REMARQUE : Le cas échéant, la trypsine peut être remplacée par un bloqueur d’adhérence cellulaire non protéolytique. - Retirer la vaisselle de l’incubateur. Pour chaque traitement, ajouter 5 mL de phénol rouge sans drogue DMEM dans un tube centrifugeuse de 15 mL désigné comme « Tube B ». Ajouter 5 mL supplémentaires du même média dans le plat concerné pour étancher la trypsine. Transférer le contenu du plat dans un tube de centrifugeuse de 15 mL désigné comme « tube A ».
- Transférer 1 mL de cellules du tube A dans le tube B. Répétez pour chaque traitement.
- Placez les tubes A et B dans l’incubateur pendant 45 minutes pour permettre aux cellules de se remettre de la trypsinisation. Relâchez légèrement le bouchon des tubes de centrifugeuse avant de les placer dans l’incubateur pour permettre la pénétration du CO2.
REMARQUE : La durée du temps de récupération peut varier selon les types de cellules. Bien que dans nos expériences la récupération de 45 minutes ait eu un effet négligeable sur la viabilité cellulaire, certains types de cellules peuvent subir des anoikis lorsqu’ils sont maintenus en suspension trop longtemps. Par conséquent, nous recommandons de déterminer le temps de récupération optimal empiriquement. Le temps de récupération optimal permet une propagation rapide et synchrone des cellules sans cellules mortes ou apoptotiques dans l’échantillon.
3. Préparation de chambre magnétique
- Assurez-vous que toutes les parties d’une chambre magnétique à cellules chamlide de 1 puits pouvant accueillir un couvercle carré de 22 mm x 22 mm ont été nettoyées avant utilisation.
- Retirer le plat de 35 mm avec le coverslip (voir étape 1.11) de l’incubateur. Aspirez les supports de culture cellulaire et lavez le coverslip avec du PBS chaud.
- Retirer le coverslip du plat de 35 mm à l’aide d’une paire de forceps et déposer délicatement le coverslip sur la plaque inférieure de la chambre magnétique.
- Placez le joint en silicone sur le dessus du coverslip.
REMARQUE : Un joint en silicone mal placé est la cause la plus fréquente d’une chambre magnétique qui fuit. Assurez-vous que le joint repose dans l’indent de la plaque inférieure et ne s’élève pas au-delà de l’indent. - Fixez le corps principal sur la plaque inférieure.
REMARQUE : Faites cette partie très lentement. Une bonne astuce est de maintenir la plaque inférieure d’une main tout en plaçant le corps principal sur le dessus. Cela garantit que les aimants du corps principal ne soulèvent pas la plaque inférieure vers le haut, ce qui pourrait potentiellement déplacer et casser le coverslip. - Ajouter 1 mL de phénol rouge sans drogue DMEM à la chambre magnétique. Prenez un tissu sans peluche et tamponnez soigneusement l’enceinte entre le corps principal et la plaque inférieure afin de vérifier les fuites.
REMARQUE : En cas de fuite, aspirez rapidement les médias et procédez à nouveau à partir de l’étape 3.4. - Abaissez le couvercle transparent sur le corps principal pour enfermer la chambre magnétique.
- Vaporisez d’abord un tissu de laboratoire avec de l’eau et essuyez le fond de la chambre magnétique (le coverslip, pas la partie métallique). Ensuite, vaporisez un deuxième tissu de laboratoire avec une petite quantité de 70% d’éthanol et essuyez, en prenant soin de ne pas casser le coverslip.
4. Acquisition d’images
- Préchauffer l’incubateur supérieur de scène et le réchauffeur objectif à 37 °C et régler le niveau de CO2 dans l’incubateur supérieur de scène à 5 %.
REMARQUE : Si l’incubateur supérieur de scène n’est pas relié à un approvisionnement en CO2, les supports de culture cellulaire doivent être complétés par 25 mM HEPES pour maintenir le pH constant 7,4. - Appliquez une quantité suffisante d’huile d’immersion sur l’objectif d’huile préchauffé 60X et 1,4 N.A.
REMARQUE : Nous utilisons un objectif d’immersion d’huile de 60 X, 1,4 N.A. dans ce protocole en raison de son champ de vision raisonnablement grand et de son efficacité exceptionnelle en matière de collecte de lumière. Si un champ de vision plus large est nécessaire, un objectif de grossissement inférieur(p. ex.,20x) peut être utilisé tant que le rapport signal-bruit des images est supérieur à 2,5. - Apportez la chambre magnétique terminée et le tube B (étape 2.9) au microscope confocal. Placez la chambre magnétique sur l’incubateur supérieur de la scène.
REMARQUE : Placez doucement la chambre magnétique sur scène pour éviter de créer des bulles dans l’huile d’immersion. - Mettez l’accent sur les cellules fluorescentes à l’aide du canal GFP. Assurez-vous que le bord de la cellule est tranchant et bien défini.
- Retirez le couvercle transparent de la chambre magnétique et pipette 500 μL du tube B dans la chambre magnétique. Placez le couvercle transparent sur le dessus de la chambre magnétique.
- Pour identifier les cellules idéales pour l’analyse de la propagation cellulaire, recherchez des « halos » de cellules qui n’ont pas encore été attachés au coverslip mais qui ne roulent plus. Les cellules qui en sont aux premiers stades de l’attachement de coverslip sont également de grands candidats, mais l’acquisition d’image doit être rapide afin de capturer la diffusion.
- Configurez l’acquisition d’images en accéléré pour le canal vert pour inclure quatre champs de vision, photographiés à intervalles de 6 secondes.
REMARQUE : En raison de la grande variabilité de la vitesse de saillie de lamellipodia entre différents types de cellules, le taux optimal d’image devrait être déterminé empiriquement. L’intervalle d’imagerie de 6 secondes utilisé dans nos expériences est un bon point de départ pour l’analyse de nombreuses cellules mésenchymales et épithéliales. Toutefois, les cellules qui se propagent très rapidement(p. ex.,les cellules immunitaires) peuvent nécessiter un taux d’image beaucoup plus élevé (intervalle d’imagerie plus court). La vitesse d’image optimale pour les films d’épandage cellulaire assure un déplacement de 2-5 pixels du bord de la cellule saillante entre les images suivantes. Compte tenu de la précision de l’ajustement de courbe utilisé pour identifier le plateau de propagation cellulaire, le taux d’image optimal devrait également assurer 50-100 mesures du déplacement du bord cellulaire au cours de la phase d’expansion rapide de la propagation cellulaire. Le nombre de champs de vision doit être ajusté en fonction du temps d’exposition, de la distance entre les points d’acquisition et de la vitesse de déplacement de la scène. Il est conseillé aux utilisateurs de déterminer le nombre maximum de champs de vision qui peuvent être acquis avec le taux d’image souhaité. - Après avoir identifié un champ de vision approprié, enregistrez les coordonnées X et Y de l’étape du microscope. Procédez à l’identification de trois autres champs de vision qui sont relativement proches les uns des autres sur le coverslip. Enregistrez les coordonnées de l’étape microscope pour chaque champ de vision désiré.
REMARQUE : Il est fortement recommandé d’optimiser la trajectoire de mouvement de la scène entre les champs de vision afin de minimiser tout mouvement inutile de l’échantillon. Une telle optimisation peut être effectuée manuellement ou automatiquement. Un mouvement excessif de l’échantillon ralentit l’acquisition et peut entraîner le déploiement des cellules hors de vue pendant qu’elles descendent. - Obtenez des images pendant 15 minutes à une vitesse d’image de 6 secondes et enregistrez les fichiers. Si d’autres acquisitions sont nécessaires, répétez à partir de l’étape 4.6.
5. Analyse de la zone cellulaire, de la circularité et de la dynamique de saillie lors de la propagation cellulaire
- Préparer des images pour le traitement et l’analyse des données
REMARQUE : Le logiciel nécessite une image en format .tiff et une taille de pixel comme paramètres d’entrée. Les deux exigences peuvent être remplies à l’aide du logiciel d’acquisition ou fidjien (dans ce protocole). Si ces exigences sont remplies, passez à l’étape 5.2.- Installez la dernière version de l’application Fidji (https://imagej.net/Fiji/Downloads).
- Ouvrez une image en accéléré à l’aide de Fidji.
- Copiez la taille pixel de l’image en sélectionnant l’image > propriétés. Copiez et coller la taille des pixels en μm à Notepad/Word.
- Pour l’analyse de la zone de propagation cellulaire et de la circularité, enregistrez l’image en accéléré sous forme de pile d’images tiff. Le logiciel d’analyse sur mesure ne prend pas en charge les formats de fichiers propriétaires. Enregistrez la pile d’images de tiff de cellule individuelle en sélectionnant > enregistrer > Tiff.
- Installez le Python IDE (Spyder) et les paquets nécessaires (PySimpleGUI et tifffile) pour le traitement et l’analyse des données.
REMARQUE : L’installation de Python et de paquets n’est requise que pour la configuration initiale.- Les films en accéléré seront analysés dans le Spyder IDE à l’aide d’un script Python sur mesure. Pour télécharger le Spyder IDE, téléchargez le distributeur Anaconda (https://www.anaconda.com/products/individual) qui comprend Spyder IDE et la plupart des bibliothèques et paquets nécessaires pour cette analyse.
- Installez Anaconda et lancez Spyder via Anaconda Navigator.
- Dans l’onglet console IPython (situé dans la section inférieure droite de Spyder), copiez et coller la commande suivante : pip installez PySimpleGUI et appuyez sur la clé d’entrée. L’exécution de cette commande installera le paquet nécessaire pour lancer l’interface utilisateur graphique (GUI).
- Dans la même console, copiez et coller la commande suivante : installez le tifffile pip et appuyez sur la touche Enter. L’exécution de cette commande installera le paquet nécessaire pour enregistrer des images sous forme de fichiers tiff.
- Téléchargez tous les scripts Python à partir des fichiers supplémentaires ou des scripts les plus mis à jour de GitHub à : https://github.com/ernestiu/Cell-spreading-analysis.git
- Quantifier la zone cellulaire et les facteurs de forme cellulaire lors de la propagation cellulaire
- Ouvrez le script d’analyse principal « cell_spreading_GUI.py » en sélectionnant l’option open file dans le panneau supérieur de Spyder ou en utilisant le raccourci Ctrl + O.
- Ouvrez l’interface graphique d’analyse de diffusion cellulaire en sélectionnant « Exécuter le fichier » dans le panneau supérieur ou en utilisant le raccourci F5.
- Cliquez sur l’onglet Zone de propagation cellulaire( Figure 3A).
- Sélectionnez l’image tiff à analyser à l’aide du bouton parcourir.
REMARQUE : Le fichier sélectionné doit être un fichier tiff. - Spécifiez l’annuaire de destination où les sorties de données (p. ex., masques cellulaires, valeurs) seront enregistrées.
- Spécifiez les paramètres de sortie des données :
- Enregistrer les masques : enregistrez les masques cellulaires générés pendant le processus de segmentation.
- Données d’exportation : Exportez une feuille de calcul Excel (.xlsx) qui contient toutes les données d’analyse dans le dossier destination.
La zone cellulaire, la circularité et le rapport d’aspect de toutes les cellules de propagation seront enregistrés sous forme de feuille de calcul Excel dans le dossier destination. La zone cellulaire est calculée comme :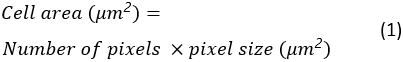
La circularité cellulaire est une mesure de la proximité d’une cellule avec une cellule parfaitement ronde. Il est calculé comme :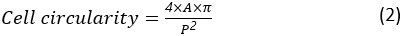
où A et P sont la zone cellulaire et le périmètre cellulaire, respectivement. Le rapport d’aspect de la cellule représente la façon dont la cellule est allongée. Une cellule de propagation doit avoir un rapport d’aspect proche de 1. Le rapport d’aspect est calculé comme :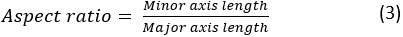
- Enregistrer les contours : enregistrez les images de superposition du contour de la limite cellulaire dans le dossier destination.
- Spécifiez les paramètres de segmentation
- Afficher la segmentation : Afficher le résultat de segmentation de la console Spyder pendant le processus d’analyse.
- Plus petite zone cellulaire (μm2): Entrez la valeur minimale pour la zone cellulaire, y compris les valeurs de zone des cellules dans les premiers stades de l’attachement. Les objets dont la zone est inférieure à ce seuil ne seront pas considérés comme des cellules qui se propagent. Ce nombre affectera le processus de segmentation.
- Spécifiez les paramètres de l’image.
- Intervalle d’acquisition (s) : Entrez la fréquence de l’acquisition de l’image en quelques secondes.
- Taille du pixel (μm) : Entrez la taille des pixels enregistrée lors de la préparation des images aux fins d’analyse.
- Profondeur du bit d’image : Entrez la profondeur du bit de la caméra/détecteur.
- Cliquez sur Exécuter. En cas d’erreur, un message d’erreur apparaîtra dans la console de Spyder. Dans le cas contraire, le processus d’analyse d’image sera affiché dans la console.
REMARQUE : La première image à apparaître dans la section console/Parcelles (selon les paramètres spyder) affiche toutes les cellules identifiées dans le champ de vision. Les boîtes vertes placées autour des cellules indiquent la propagation des cellules qui conviennent à la segmentation et à l’analyse. Les boîtes grises indiquent les cellules qui ne conviennent pas à l’analyse. Le nombre total de cellules de propagation identifiées apparaîtra également dans l’onglet Console. Le logiciel trace la zone cellulaire (en bleu) et la circularité cellulaire (en rouge) en fonction du temps. Ces graphiques permettent aux utilisateurs d’évaluer l’exactitude de la segmentation cellulaire. Une segmentation réussie donne une courbe monotone croissante pour la zone cellulaire. Pour obtenir une courbe représentative de la zone de propagation cellulaire, la phase de décalage doit être retirée manuellement du graphique. La phase de décalage comprend des mesures de la zone cellulaire avant que la cellule commence à se propager. La phase de décalage est indiquée par des fluctuations rapides, telles que représentées dans la parcelle de la zone cellulaire(figure 3C à droite).
6. Quantifier la dynamique du bord cellulaire pendant la propagation cellulaire à l’aide de kymographes
- Avant d’exécuter l’analyse, recadrer les films bruts des cellules qui se propagent pour créer des séries de temps de cellules de propagation individuelles.
- Utilisez l’outil Rectangle dans la barre d’outils Fidji pour sélectionner manuellement une région d’intérêt (ROI) qui encapsule une seule cellule. (Pour vous assurer que le retour sur investissement encapsule entièrement la cellule d’épandage, utilisez la fonction de défilement pour inspecter le retour sur investissement à tout moment.)
- Cliquez à droite sur le retour sur investissement et sélectionnez Duplicata.
- Vérifiez la pile en double et cliquez sur OK.
- Ouvrez le script d’analyse principal « cell_spreading_GUI.py » en sélectionnant le bouton Open File dans la barre d’outils de Spyder ou en utilisant le raccourci Ctrl + O. Si l’interface graphique a déjà été ouverte, passez directement à l’étape 6.3.
- Ouvrez l’interface graphique d’analyse de diffusion cellulaire en sélectionnant le fichier Run dans le panneau supérieur ou en utilisant le raccourci F5 (Figure 3B).
- Cliquez sur l’onglet Générateur kymograph et analyse.
- Utilisez le bouton parcourir pour sélectionner l’image tiff pour l’analyse.
REMARQUE : Les formats de fichiers propriétaires, par exemple, nd2, lif, zen, ne sont pas pris en charge par le script. - Spécifiez le dossier destination pour enregistrer les données de sortie (masques cellulaires et valeurs).
- Spécifiez les paramètres de sortie.
- Données d’exportation : Exportez une feuille de calcul Excel (.xlsx) vers le dossier destination qui contient des positions relatives du bord cellulaire et des événements de rétractation des kymographes.
- Spécifiez les paramètres de l’image :
- Intervalle d’acquisition (s) : Entrez la fréquence d’acquisition de l’image en quelques secondes.
- Taille du pixel (μm) : Entrez la taille des pixels enregistrée lors de la préparation des images pour l’analyse dans l’étape 5.1.3.
- Plus petite zone cellulaire (μm2): Entrez la valeur minimale pour la zone cellulaire, y compris les valeurs de zone des cellules dans les premiers stades de l’attachement. Les objets dont la zone est inférieure à ce seuil ne seront pas considérés comme des cellules qui se propagent. Ce nombre affectera le processus de segmentation.
- Profondeur du bit d’image : Entrez la profondeur du bit de la caméra/détecteur.
- Cliquez sur Exécuter. En cas d’erreur, un message d’erreur apparaîtra dans la console de Spyder. Sinon, un résumé des quantifications de la dynamique de saillie sera affiché dans la console. Il y aura 4 paires de mesures de fréquence de rétractation et de vitesse de saillie, qui sont extraites de 4 kymographs générés à partir des parties supérieure, inférieure, gauche et droite de la cellule. La fréquence de rétractation est calculée comme :
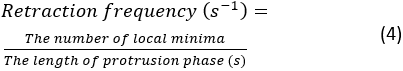
REMARQUE : Ce nombre montre à quelle fréquence le lamellipodium se rétracte au cours de la propagation. La vitesse moyenne de saillie est mesurée par la pente entre le début de la saillie et le point de plateau sur le kymograph. Une figure kymograph résumée sera affichée dans la console après la segmentation. Pour enregistrer le chiffre récapitulatif, cliquez à droite sur la figure et enregistrez l’image.
Résultats
Le protocole ci-dessus décrit les procédures expérimentales pour l’imagerie des cellules vivantes des cellules de propagation et un outil de calcul pour l’analyse quantitative de la dynamique de propagation cellulaire. L’outil de calcul peut être utilisé dans un format à faible ou à haut débit pour identifier les acteurs moléculaires régulant les machines de polymérisation de l’actine à l’avant-garde cellulaire.
La représentation schématique des procédures expérimenta...
Discussion
L’analyse de propagation cellulaire décrite permet le suivi continu des changements morphologiques(p. ex., taille et forme des cellules) et des mouvements des bords cellulaires(c.-à-d. vitesse de saillie et fréquence de rétractation), qui sont des caractéristiques manquantes dans la plupart des protocoles depropagation cellulaire 19,24. Bien que les analyses de propagation cellulaires à point final couramment utilisées permettent de dé...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ces travaux ont été appuyés par le Prix du nouvel enquêteur du Fonds Connaught à S.P., la Fondation canadienne pour l’innovation, le Programme de subventions à la découverte du CSERN (subventions RGPIN-2015-05114 et RGPIN-2020-05881), le Fonds conjoint de recherche de l’Université de Manchester et de l’Université de Toronto et le Programme XSeed de l’Université de Toronto.
matériels
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
Références
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon