Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Количественный анализ динамики края клетки во время распространения клеток
* Эти авторы внесли равный вклад
В этой статье
Резюме
В этом протоколе мы представляем экспериментальные процедуры анализа распространения клеток, основанного на живоклеточной микроскопии. Мы предоставляем вычислительный инструмент с открытым исходным кодом для объективной сегментации флуоресцентно помеченных клеток и количественного анализа динамики ламеллиподии во время распространения клеток.
Аннотация
Распространение клеток представляет собой динамический процесс, при котором ячейка, подвешенная в средствах массовой информации, прикрепляется к субстрату и выравнивается от округлой до тонкой и распределимой формы. Следуя клеточному субстрату, клетка образует тонкий лист ламеллиподии, исходящий из клеточного тела. В ламеллиподии, шаровые актин (G-actin) мономеры полимеризируются в плотный нитементный актин (F-актин) сетки, которая толкает против плазменной мембраны, тем самым обеспечивая механические силы, необходимые для клетки распространяться. Примечательно, что молекулярные игроки, которые контролируют актин полимеризации в lamellipodia имеют важное значение для многих других клеточных процессов, таких как миграция клеток и эндоцитоз.
Так как распространяющиеся клетки образуют непрерывную ламеллиподию, которая охватывает всю периферию клеток и постоянно расширяется наружу, распространяющие клетки анализы стали эффективным инструментом оценки кинетики ламеллиподиальных выступов. Хотя было разработано несколько технических реализаций анализа распространения клеток, в настоящее время отсутствует подробное описание рабочего процесса, которое будет включать как пошаговой протокол, так и вычислительные инструменты для анализа данных. Здесь мы описываем экспериментальные процедуры анализа распространения клеток и представляем инструмент с открытым исходным кодом для количественного и беспристрастного анализа динамики края клетки во время распространения. В сочетании с фармакологическими манипуляциями и/или генно-заглушающих методов, этот протокол является подложным к крупномасштабному экрану молекулярных игроков, регулирующих lamellipodial выступы.
Введение
Ламеллиподные выступы являются выдающимися цитоскелетными структурами, образованными в передней части мигрирующей клетки. В lamellipodia, полимеризация актина с помощью комплекса Arp2/3 и формин создает быстрорастущие разветвленной актинные сетки, которая толкает против плазменноймембраны 1,2. Сила нажатия, генерируемая актиальной сеткой, физически продвигаетячейку вперед 1,3,4,5. Истощение комплекса Arp2/3 или нарушение сигнальных путей, необходимых для ламеллиподных выступов, часто ухудшаетмиграцию клеток 6, 7. Хотя миграция ламеллиподии-дефицитных клетоктакже сообщалось 8,9, важность lamellipodia в миграции клеток очевидна, как истощение этой протрусской структуры возмущает способность клетки двигаться через сложные биологические микроокноронности6,10.
Основным препятствием для понимания регулирования lamellipodia в мигрирующих клетках является естественная изменчивость в lamellipodial выступ кинетики,размер, и форма 11,12,13,14. Кроме того, недавние исследования показали, что ламеллиподия обладают сложным протрусивным поведением, включая колебания, периодические иускоряющиеся выступы 14,15. По сравнению с весьма переменной lamellipodia мигрирующихклеток 6,16, lamellipodia формируется во время распространения клеток являются болееравномерной 12. Так как протрусическая активность распространяющихся и мигрирующих клеток определяется идентичными макромолекулярными сборками, которые включают разветвленную актиновую сеть, контрактильные актомиозинные пучки и интегрин-матрицы17,18, распространяющиеся клетки широко используются в качестве модели для изучения регулирования динамики ламеллиподии.
Распространение клеток является динамическим механохимическим процессом, где клетка в подвеске сначала прилипает к субстрату через интегрин на основе спаек17,19,20, а затем распространяется путем расширения актин-основевыступов 21,22,23. Во время фазы распространения, lamellipodia, вытекающих из клеточного тела выступают изотропически и настойчиво практически без опрокидывания или срыва12. Наиболее часто используемые протоколы распространения клеток являются анализом конечных точек, где распространяющиеся ячейки фиксируются в разное времяпосле покрытия 19,24. Эти анализы, хотя и быстро и просто, ограничены в своей диагностической мощности для обнаружения изменений в динамических особенностей lamellipodia. Чтобы определить молекулярные механизмы, которые контролируют динамику ламеллиподии, группа Sheetz впервые использовал количественный анализ живых распространяющих клеток и обнаружил много фундаментальных свойстввыступов края клетки 11,12,22. Эти исследования показали, что анализ распространения живых клеток является надежным и мощным методом в арсенале лаборатории клеточной биологии. Несмотря на это, подробный протокол и вычислительный инструмент с открытым исходным кодом для анализа распространения живой клетки в настоящее время недоступны для сообщества клеточной биологии. С этой целью в нашем протоколе излагаются процедуры визуализации живых распространяющихся клеток и предоставляется автоматизированный инструмент анализа изображений. Для проверки этого метода мы использовали ингибирование Arp2/3 в качестве экспериментального лечения и показали, что ингибирование функции комплекса Arp2/3 не арестовало распространение клеток, но вызвало значительное снижение скорости выпячивания клеток, а также стабильность выступов края клетки, что привело к зубчатым краям клеток. Эти данные показывают, что сочетание живой клетки изображения и автоматизированного анализа изображений является полезным инструментом для анализа динамики края клетки и выявления молекулярных компонентов, которые регулируют lamellipodia.
протокол
1. Посев клеток
ПРИМЕЧАНИЕ: Описанный протокол распространения клеток был выполнен с использованием эмбриональных фибробластов мыши (MEFs), выражаюющих PH-Akt-GFP (флуоресцентный маркер для PIP3/PI(3,4)P2). Эта клеточная линия была создана путем геномной интеграции построения экспрессии для PH-Akt-GFP (Addgene #21218) при посредничестве CRISPR редактирования генов. Тем не менее, другие флуоресцентные маркеры, которые выражаются временно или интегрированы в геном также могут быть использованы в этом анализе. Для оптимальной сегментации изображения мы рекомендуем использовать флуоресцентные маркеры, равномерно распределенные в цитоплазме, например, цитопзолированный GFP.
- Культура 10 см блюдо клеток до 90% слияния.
- После того, как клетки достигли надлежащего слияния, поместите 22 мм х 22 мм крышки (#1,5; 0,17 мм толщиной) в 35 мм клеточной культуры блюдо. Пальто крышку с 400 йл фибронектина, который был разбавлен в PBS до окончательной концентрации 2,5 мкг/мл.
ПРИМЕЧАНИЕ: Количество обложек, необходимых для анализа, определяется количеством экспериментальных условий и технической реплики. - Поместите 35-мм блюдо с фибронектин покрытием крышки в 37 градусов по Цельсию, 5% CO2 инкубатор в течение 1 часа.
- Удалите блюдо с крышкой из инкубатора. Аспирировать фибронектин и мыть крышки с PBS, мягко pipetting вокруг крышки два-три раза.
- Аспирировать клеточной культуры СМИ из 10 см блюдо клеток и мыть блюдо с PBS.
- Добавьте 650 йл 0,05% трипсина-ЭДТА к 90% стечению клеток, наклоняя блюдо, чтобы равномерно распределить фермент. Поместите блюдо с трипсином в инкубатор на 1 минуту.
- Удалите блюдо с клетками из инкубатора. Добавьте 10 мл средств массовой информации клеточной культуры в центрифугу 15 мл. Быстро добавьте еще 10 мл мультимедиа в блюдо, чтобы утолить трипсин.
- Pipette 1 мл трипсинизированных клеток в 15 мл центрифуги трубки для того, чтобы разбавить клетки. Pipette содержимое трубки вверх и вниз, чтобы обеспечить равномерное распределение клеток в средствах массовой информации. Для типов клеток с высокой склонностью к агрегации рекомендуется фильтровать клетки через клеточный ситечко (размер сетки 100 мкм), чтобы свести к минимуму возникновение слипания клеток.
- Из трубки, пипетка 500 - 1000 МКЛ разбавленных клеток в 35 мм блюдо, содержащее крышку.
- Аккуратно встряхните блюдо, чтобы равномерно распределить клетки. Убедитесь, что крышки находится на 10% слияние (50000 клеток / мл) и регулировать объем разбавленных клеток по мере необходимости.
ПРИМЕЧАНИЕ: Цель наличия клеток с таким низким уровнем слияния заключается в обеспечении наличия 1-2 поляризованных клеток в каждой области зрения, которые будут использоваться для фокусировки цели на протяжении всей приобретения распространения клеток. - Прохождение 1/5 из оставшихся клеток в 10 см блюдо в один 6 см блюдо на лечение состоянии. Поместите проходные блюда и 35 мм блюдо с крышкой в инкубатор на ночь.
ПРИМЕЧАНИЕ: Это будут клетки, которые будут проанализированы для распространения динамики.
2. Инкубация наркотиков и восстановление клеток
- Добавьте 5 мл средств массовой информации клеточной культуры в каждую из двух трубок центрифуги 15 мл и 20 мл фенола красного свободного DMEM в каждую из двух трубок центрифуги 50 мл.
ПРИМЕЧАНИЕ: Количество трубчатых пар (15 мл и 50 мл) должно соответствовать количеству экспериментальных условий. - Чтобы проверить важность Arp2/3 для распространения клеток, пипетка либо фармакологический ингибитор Arp2/3, CK-666, или контрольное лечение, такие как DMSO, в каждой паре центрифуг трубки до желаемой концентрации.
- Удалите проходные 6-сантиметровые блюда (см. Шаг 1.11) из инкубатора и аспирировать средства массовой информации. Вымойте посуду теплым PBS.
- Добавьте содержимое CK-666- или DMSO-дополненных 15 мл центрифуг трубки в каждом из проходных блюд. Этикетка каждое блюдо с правильной медикаментозной обработки и поместить блюда в инкубатор в течение одного часа.
- Удалите посуду из инкубатора и аспирировать средства массовой информации. Вымойте посуду теплым PBS для того, чтобы тщательно удалить все оставшиеся фенол красные средства массовой информации.
- Добавьте 230 МКЛ 0,05% трипсина-ЭДТА к каждому 6-сантиметровому блюду и инкубировать клетки в течение 1 минуты.
ПРИМЕЧАНИЕ: Если это применимо, трипсин может быть заменен не протеолитическим блокатором адгезии клеток. - Удалите посуду из инкубатора. Для каждого лечения добавьте 5 мл лекарственно-дополненного фенола красного свободного DMEM в 15 мл центрифуги трубки, обозначенной как "Tube B". Добавьте еще 5 мл одного и того же средства массовой информации в соответствующее блюдо, чтобы утолить трипсин. Перенесите содержимое блюда в центрифугу 15 мл, обозначенную как "Tube A".
- Передача 1 мл клеток из трубки А в трубку B. Повторите для каждого лечения.
- Поместите трубки A и B в инкубатор на 45 минут, чтобы клетки мыкновяются после трипсинизации. Слегка ослабьте крышку центрифуг трубки перед размещением их в инкубаторе, чтобы позволить CO2 пенетранса.
ПРИМЕЧАНИЕ: Продолжительность времени восстановления может варьироваться для различных типов клеток. Хотя в наших экспериментах 45-минутное восстановление имело незначительное влияние на жизнеспособность клеток, некоторые типы клеток могут подвергаться анойкису при слишком длительном приостановлении. Поэтому мы рекомендуем эмпирически определить оптимальное время восстановления. Оптимальное время восстановления позволяет быстро и синхронно распространять клетки без мертвых или апоптотических клеток в образце.
3. Подготовка магнитной камеры
- Убедитесь, что все части 1 Ну Chamlide Cell Магнитная камера, которая может вместить 22 мм х 22 мм квадратных крышки были очищены перед использованием.
- Удалите 35-мм блюдо с крышкой (см. Шаг 1.11) из инкубатора. Аспирировать клеточной культуры средств массовой информации и мыть крышки с теплой PBS.
- Снимите крышку с 35-мм тарелки с помощью пары типсов и аккуратно положите крышку на нижнюю пластину магнитной камеры.
- Поместите силиконовую прокладку поверх крышки.
ПРИМЕЧАНИЕ: Неправильно размещены силиконовые прокладки является наиболее распространенной причиной вытекающей магнитной камеры. Убедитесь, что прокладка лежит в отступе нижней пластины и не поднимается за пределы отступа. - Прикрепите основное тело к нижней пластине.
ПРИМЕЧАНИЕ: Делайте эту часть очень медленно. Хороший совет заключается в том, чтобы держать вниз нижнюю пластину с одной стороны, поместив основной орган на вершине. Это гарантирует, что магниты основного тела не поднимают нижнюю пластину вверх, что потенциально может вытеснить и взломать крышку. - Добавьте 1 мл лекарственно-дополненного фенола красного свободного DMEM в магнитную камеру. Возьмите ткань без ворса и тщательно мазок корпус между основным телом и нижней пластины для того, чтобы проверить на наличие утечек.
ПРИМЕЧАНИЕ: Если есть утечка, быстро аспирировать средства массовой информации и перейти снова от шага 3.4. - Опустите прозрачную крышку на основной корпус, чтобы охватить магнитную камеру.
- Спрей лабораторной ткани сначала с водой и протрите дно магнитной камеры (крышки, а не металлическая часть). После этого распылите вторую лабораторную ткань с небольшим количеством 70% этанола и протрите, будьте осторожны, чтобы не взломать крышку.
4. Приобретение изображений
- Разогреть верхний инкубатор и объективный обогреватель до 37 градусов по Цельсию и установить уровень CO2 в верхней стадии инкубатора до 5%.
ПРИМЕЧАНИЕ: Если верхний инкубатор этапа не подключен к поставке CO2, средства культуры клеток должны быть дополнены 25 мМ HEPES для поддержания постоянного рН 7.4. - Нанесите достаточное количество масла погружения на предварительно разогретую нефтяную цель 60X, 1.4 N.A.
ПРИМЕЧАНИЕ: Мы используем цель погружения нефти 60X, 1.4 N.A. в этот протокол из-за его достаточно большого поля зрения и выдающейся эффективности сбора света. Если требуется большее поле зрения,то можно использовать более низкую цель увеличения (например, 20x) до тех пор, пока соотношение сигнала к шуму изображений превышает 2,5. - Принесите как завершенную магнитную камеру, так и трубку B (шаг 2.9) к конфокальцу. Поместите магнитную камеру на верхнюю ступень инкубатора.
ПРИМЕЧАНИЕ: Поместите магнитную камеру мягко на сцене, чтобы избежать создания пузырьков в погружении нефти. - Установите фокус на флуоресцентные клетки с помощью канала GFP. Убедитесь, что край ячейки острый и четко определен.
- Удалите прозрачную крышку магнитной камеры и пипетку 500 МКЛ из трубки B в магнитную камеру. Поместите прозрачную крышку обратно на верхней части магнитной камеры.
- Чтобы определить клетки, идеально подходит для анализа распространения клеток, поиск "галос" клеток, которые до сих пор не прикреплены к крышке, но больше не прокатки вокруг. Клетки, которые находятся на самых ранних стадиях присоединения крышки также большие кандидаты, но изображение приобретение должно быть быстрым для того, чтобы захватить распространения.
- Настройте покадровую съемку изображения для зеленого канала, чтобы включить четыре поля зрения, изображенные с интервалом в 6 секунд.
ПРИМЕЧАНИЕ: Из-за высокой изменчивости скорости выпячивания lamellipodia между различными типами клеток, оптимальная частота кадров должна быть определена эмпирически. Интервал изображения 6 секунд, используемых в наших экспериментах, является хорошей отправной точкой для анализа многих мезенхимальных и эпителиальных клеток. Тем не менее, клетки, которые распространяются очень быстро(например,иммунные клетки) может потребовать гораздо более высокой частоты кадров (короткий интервал изображения). Оптимальная частота кадров для фильмов, распространяющих ячейки, обеспечивает смещение 2-5 пикселей выступающего края ячейки между последующими кадрами. Учитывая точность установки кривой, используемой для определения плато распространения клеток, оптимальная частота кадров должна также обеспечить 50-100 измерений смещения края ячейки во время быстрой фазы расширения распространения клеток. Количество полей зрения должно быть скорректировано в зависимости от времени экспозиции, расстояния между точками приобретения и скорости движения этапа. Пользователям рекомендуется определить максимальное количество полей зрения, которые могут быть приобретены с желаемой частотой кадров. - После определения подходящего поля зрения сохраните координаты X и Y стадии микроскопа. Продолжить с определением трех других областей зрения, которые находятся относительно близко друг к другу на обложке. Сохраните координаты стадии микроскопа для каждого желаемого поля зрения.
ПРИМЕЧАНИЕ: Настоятельно рекомендуется оптимизировать путь движения между полями зрения, чтобы свести к минимуму ненужные движения выборки. Такая оптимизация может быть выполнена как вручную, так и автоматически. Чрезмерное движение выборки замедляет приобретение и может привести к тому, что клетки будут выкатываться из поле зрения при их нисходящем спуске. - Приобретайте изображения в течение 15 минут с 6-секундной частотой кадров и сохраните файлы. Если требуется больше приобретений, повторите, начиная со шага 4.6.
5. Анализ клеточной области, круговорота и динамики выступов при распространении клеток
- Подготовка изображений для обработки и анализа данных
ПРИМЕЧАНИЕ: Программное обеспечение требует изображения в .tiff и размера пикселя в качестве входных параметров. Оба требования могут быть выполнены с помощью программного обеспечения приобретения или Фиджи (в этом протоколе). Если эти требования будут выполнены, перейти к шагу 5.2.- Установите последнюю версию приложения Фиджи (https://imagej.net/Fiji/Downloads).
- Откройте изображение замедленного действия с помощью Фиджи.
- Скопируйте размер пикселя изображения, выбрав Image > Properties. Копировать и вставлять размер пикселя в мкм на блокнот / слово.
- Для анализа области распространения клеток и круговорота сохраните покадровое изображение в качестве стека изображений tiff. Программное обеспечение для анализа пользовательских сборк не поддерживает собственные форматы файлов. Сохраните отдельный стек изображений ячейки tiff, выбрав файл > сохранить как > Tiff.
- Установите Python IDE (Spyder) и необходимые пакеты(PySimpleGUI и tifffile)для обработки и анализа данных.
ПРИМЕЧАНИЕ: Установка Python и пакетов требуется только для первоначальной настройки.- Фильмы замедленного действия будут проанализированы в Spyder IDE с помощью пользовательского сценария Python. Чтобы загрузить Spyder IDE, загрузите дистрибьютора Anaconda (https://www.anaconda.com/products/individual), который включает Spyder IDE и большинство необходимых библиотек и пакетов для этого анализа.
- Установите Anaconda и запустить Spyder через Anaconda Navigator.
- Во вкладке консоли IPython (расположен в правом нижнем разделе Spyder) скопируйте и вставьте следующую команду: установите пипс PySimpleGUI и нажмите клавишу входа. Запуск этой команды установит пакет, необходимый для инициирования графического пользовательского интерфейса (GUI).
- В той же консоли, копировать и вставлять следующую команду: пип установить tifffile и нажмите клавишу Enter. Запуск этой команды установит пакет, необходимый для сохранения изображений в качестве файлов tiff.
- Загрузите все скрипты Python из дополнительных файлов или самых обновленных скриптов с GitHub по https://github.com/ernestiu/Cell-spreading-analysis.git
- Количественная оценка площади клеток и факторов формы клеток во время распространения клеток
- Откройте основной сценарий анализа "cell_spreading_GUI.py", выбрав вариант открытого файла в верхней панели Spyder или используя ярлык Ctrl и O.
- Откройте графический интерфейс анализа распространения ячеей, выбрав "Run file" в верхней панели или используя ярлык F5.
- Нажмите на вкладку Область распространения клеток(рисунок 3A).
- Выберите изображение tiff для анализа с помощью кнопки просмотра.
ПРИМЕЧАНИЕ: Выбранный файл должен быть файлом tiff. - Укажите каталог назначения, в котором будут сохранены выходные данные (например, маски ячеей, значения).
- Укажите параметры вывода данных:
- Сохранить маски: Сохранить клеточные маски, созданные в процессе сегментации.
- Экспортные данные: Экспорт электронной таблицы Excel (.xlsx), которая содержит все данные анализа в папку назначения.
Область ячейки, круговорот и соотношение аспектов всех распространяющих ячеек будут сохранены в качестве электронной таблицы Excel в папке назначения. Область ячейки рассчитывается как: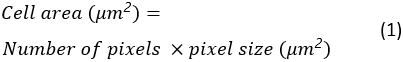
Круговая кругия клеток является мерой того, насколько близко клетка к идеально круглой клетке. Он рассчитывается как: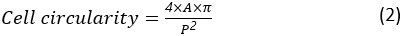
где A и P являются областью ячейки и периметром ячейки, соответственно. Соотношение сторон ячейки представляет, насколько вытянута клетка. Распространяющаяся ячейка должна иметь соотношение сторон близко к 1. Соотношение сторон рассчитывается как: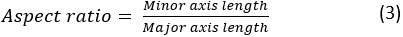
- Сохранить контуры: Сохранить контур границы ячейки наложения изображений в папке назначения.
- Укажите параметры сегментации
- Показать сегментацию: Показать результат сегментации консоли Spyder во время процесса анализа.
- Самая маленькая область ячейки (м2):Введите минимальное значение для области ячейки, включая значения области клеток на начальных стадиях вложения. Объекты с площадью, меньшей этого порога, не будут рассматриваться как распространяющиеся ячейки. Это число повлияет на процесс сегментации.
- Укажите параметры изображения.
- Интервал приобретения (ы): Введите частоту получения изображения в считанные секунды.
- Размер пикселя (мкм): Введите размер пикселя, который был записан при подготовке изображений для анализа.
- Глубина бита изображения: Введите бит глубину камеры / детектора.
- Нажмите Вы запустите. Если возникнет ошибка, сообщение об ошибке появится в консоли Spyder. В противном случае процесс анализа изображения будет показан на консоли.
ПРИМЕЧАНИЕ: Первое изображение, появиваеся в разделе консоль/Сюжеты (в зависимости от настроек Spyder), показывает все ячейки, идентифицированные в поле зрения. Зеленые ящики, расположенные вокруг ячеек, указывают на распространение клеток, пригодных для сегментации и анализа. Серые ящики указывают на ячейки, которые не подходят для анализа. Общее количество идентифицированных распространяющих ячеек также появится во вкладке Консоль. Программное обеспечение отображает область ячейки (синим цветом) и круговую ячейку (красным цветом) как функцию времени. Эти графики позволяют пользователям оценить точность сегментации клеток. Успешная сегментация дает монотонно увеличивающейся кривой для клеточной области. Чтобы получить репрезентативную кривую области распространения клеток, фаза задержки должна быть удалена из графика вручную. Фаза отставания включает измерения площади клетки до того, как клетка начнет распространяться. Фаза задержки указывается быстрыми колебаниями, как это представлено на участке области ячейки(рисунок 3C справа).
6. Количественная динамика края клетки во время распространения клеток с помощью кимографов
- Перед запуском анализа, урожай сырые фильмы распространения клеток для создания времени серии отдельных распространяющих клеток.
- Используйте инструмент Rectangle в баре инструментов Фиджи, чтобы вручную выбрать область интереса (ROI), которая инкапсулирует одну ячейку. (Чтобы убедиться, что рентабельность инвестиций полностью инкапсулирует распространяющиеся ячейки, используйте функцию прокрутки для проверки рентабельности инвестиций в любое время.)
- Нажмите правой кнопкой мыши на roi и выберите Duplicate.
- Проверьте дубликат стека и нажмите OK.
- Откройте основной сценарий анализа "cell_spreading_GUI.py", выбрав кнопку Open File в баре инструментов Spyder или используя ярлык Ctrl и O. Если графический интерфейс уже открыт, перейдите непосредственно к шагу 6.3.
- Откройте графический интерфейс анализа распространения ячеей, выбрав файл Run в верхней панели или используя ярлык F5 (рисунок 3B).
- Нажмите на вкладку генератора Kymograph и анализа.
- Используйте кнопку просмотра, чтобы выбрать изображение tiff для анализа.
ПРИМЕЧАНИЕ: Собственные форматы файлов, например, nd2, lif, zen, не поддерживаются скриптом. - Укажите папку назначения для сохранения выходных данных (маски ячейки и значения).
- Укажите параметры вывода.
- Экспортные данные: Экспорт электронной таблицы Excel (.xlsx) в папку назначения, которая содержит относительные позиции края ячейки и события опровержения кимографов.
- Укажите параметры изображения:
- Интервал приобретения (ы): Введите частоту приобретения изображения в считанные секунды.
- Размер пикселя (мкм): Введите размер пикселя, который был записан при подготовке изображений для анализа в шаге 5.1.3.
- Самая маленькая область ячейки (м2):Введите минимальное значение для области ячейки, включая значения области клеток на начальных стадиях вложения. Объекты с площадью, меньшей этого порога, не будут рассматриваться как распространяющиеся ячейки. Это число повлияет на процесс сегментации.
- Глубина бита изображения: Введите бит глубину камеры / детектора.
- Нажмите Вы запустите. Если возникнет ошибка, сообщение об ошибке появится в консоли Spyder. В противном случае на консоли будет показано резюме количественных данных динамики выступов. Там будет 4 пары частоты опрокидывания и измерения скорости выступов, которые извлекаются из 4 кимографов, генерируемых из верхней, нижней, левой и правой частей клетки. Частота опровержения рассчитывается как:
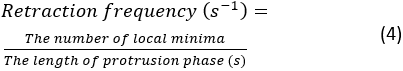
ПРИМЕЧАНИЕ: Это число показывает, как часто lamellipodium втягивается в течение распространения. Средняя скорость выступа измеряется наклоном между началом выступа и точкой плато на кимографе. После сегментации на консоли будет показана краткая цифра кимографа. Чтобы сохранить сводную цифру, нажмите правой кнопкой мыши на рисунке и сохраните изображение.
Результаты
Вышеупомянутый протокол описывает экспериментальные процедуры для живоклеточной визуализации распространяющихся клеток и вычислительный инструмент для количественного анализа динамики распространения клеток. Вычислительный инструмент может быть использован в формате низкой или...
Обсуждение
Описанный анализ распространения клеток позволяет непрерывно отслеживать морфологические изменения (например,размер клетки и форму) и движения края клетки (т.е. скорость выпячивание и частота опрокидывания), которые являются функциями, отсутствующими в большинстве протоколов
Раскрытие информации
Авторов нечего раскрывать.
Благодарности
Эта работа была поддержана Connaught Фонд Новый следователь премии S.P., Канадский фонд инноваций, NSERC Discovery Грант программы (гранты RGPIN-2015-05114 и RGPIN-2020-05881), Манчестерский университет и Университет Торонто Совместный исследовательский фонд, и Университет Торонто XSeed программы.
Материалы
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
Ссылки
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены