Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Quantitative Analyse der Zellkantendynamik während der Zellstreuung
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
In diesem Protokoll stellen wir die experimentellen Verfahren eines Zellstreu-Assays vor, der auf der Live-Zellmikroskopie basiert. Wir bieten ein Open-Source-Rechenwerkzeug für die unvoreingenommene Segmentierung fluoreszierend markierter Zellen und die quantitative Analyse der Lamellipodie-Dynamik während der Zellstreuung.
Zusammenfassung
Die Zellstreuung ist ein dynamischer Prozess, bei dem eine in Medien aufgehängte Zelle an einem Substrat befestigt wird und sich von einer abgerundeten zu einer dünnen und ausgebreiteten Form abflacht. Nach der Zellsubstrat-Anhaftung bildet die Zelle ein dünnes Blatt Lamellipodie, das aus dem Zellkörper ausgeht. In der Lamellipodia polymerisieren kugelförmige Actin-Monomere (G-Actin) in ein dichtes fadenförmiges Actin-Netz (F-Actin), das gegen die Plasmamembran drückt und so die mechanischen Kräfte bereitstellt, die für die Ausbreitung der Zelle erforderlich sind. Insbesondere die molekularen Akteure, die die Actin-Polymerisation in Lamellipodien steuern, sind für viele andere zelluläre Prozesse, wie Zellmigration und Endozytose, von wesentlicher Bedeutung.
Da sich streuende Zellen zu einer kontinuierlichen Lamellipodie bilden, die sich über die gesamte Zellperipherie erstrecken und sich dauerhaft nach außen ausdehnen, sind Zellstreuungstests zu einem effizienten Werkzeug geworden, um die Kinetik lamellipodischer Vorsprünge zu bewerten. Obwohl mehrere technische Implementierungen des Zellstreu-Assays entwickelt wurden, fehlt derzeit eine detaillierte Beschreibung des Workflows, die sowohl ein Schritt-für-Schritt-Protokoll als auch Rechenwerkzeuge für die Datenanalyse umfassen würde. Hier beschreiben wir die experimentellen Verfahren des Zellstreu-Assays und präsentieren ein Open-Source-Tool zur quantitativen und unvoreingenommenen Analyse der Zellkantendynamik während der Ausbreitung. In Kombination mit pharmakologischen Manipulationen und/oder Gen-Silencing-Techniken ist dieses Protokoll für einen groß angelegten Bildschirm von molekularen Akteuren zugänglich, die lamellipodiale Vorsprünge regulieren.
Einleitung
Lamellipodiale Vorsprünge sind prominente zytoskelettale Strukturen, die an der Vorderseite einer wandernden Zelle gebildet werden. In lamellipodien erzeugt die Polymerisation von Actin mit Hilfe des Arp2/3-Komplexes und der Formine ein schnell wachsendes verzweigtes Aktin-Netz, das gegen die Plasmamembran1,2drückt. Die schubsende Kraft, die durch das Aktin-Netz erzeugt wird, treibt die Zelle physisch nach vorne1,3,4,5. Erschöpfung des Arp2/3-Komplexes oder Störung der Signalwege, die für lamellipodiale Vorsprünge unerlässlich sind, beeinträchtigen häufig die Zellmigration6, 7. Obwohl die Migration von Lamellipodie-defiziden Zellen ebenfalls berichtet wurde8,9, ist die Bedeutung der Lamellipodie bei der Zellmigration offensichtlich, da die Erschöpfung dieser protrusiven Struktur die Fähigkeit der Zelle stört, sich durch komplexe biologische Mikroumgebungen zu bewegen6,10.
Ein großes Hindernis für das Verständnis der Regulation von Lamellipodie n. B. in wandernden Zellen ist die natürliche Variabilität in der lamellipodialen Protrusionskinetik, Größe und Form11,12,13,14. Darüber hinaus haben neuere Studien gezeigt, dass Lamellipodie komplexe protrusive Verhaltensweisen aufweisen, einschließlich schwankender, periodischer und beschleunigender Vorsprünge14,15. Im Vergleich zu den hochvariablen Lamellipodien der wandernden Zellen6,16sind Lamellipodien, die bei der Zellausbreitung gebildet werden, gleichmäßiger12. Da die protrusive Aktivität der Sichtung und Migration von Zellen durch identische makromolekulare Baugruppen angetrieben wird, die ein verzweigtes Aktin-Netzwerk, kontraktile Actomyosin-Bündel und integrinbasierte Zellmatrix-Adhäsionen17,18umfassen, wurden Streuzellen weithin als Modell für die Untersuchung der Regulation der Lamellipodie-Dynamik verwendet.
Zellstreuung ist ein dynamischer mechanochemischer Prozess, bei dem eine Zelle in Suspension zuerst durch integrinbasierte Adhäsionen17,19,20 an einem Substrat haftet und sich dann durch Verlängerung der aktinbasierten Vorsprünge21,22,23ausbreitet. Während der Ausbreitungsphase ragen Lamellipodieaus dem Zellkörper istisch und anhaltend heraus, ohne dass es sich um ein Zurückziehen oder Stocken12geht. Die am häufigsten verwendeten Zellstreuprotokolle sind Endpunkt-Assays, bei denen Streuzellen nach der Beschichtung19,24zu verschiedenen Zeitpunkten fixiert werden. Diese Assays sind zwar schnell und einfach, aber in ihrer diagnostischen Leistungsfähigkeit begrenzt, um Veränderungen in den dynamischen Merkmalen der Lamellipodie zu erkennen. Um die molekularen Mechanismen zu bestimmen, die die Lamellipodie-Dynamik steuern, war die Sheetz-Gruppe Vorreiter bei der quantitativen Analyse von lebenden Streuzellen und deckte viele grundlegende Eigenschaften von Zellrandvorsprüngenauf 11,12,22. Diese Studien haben gezeigt, dass der Live-Zell-Verbreitungstest eine robuste und leistungsstarke Technik in der Werkzeugkiste eines Zellbiologielabors ist. Trotzdem sind ein detailliertes Protokoll und Ein-Quell-Rechentool für einen Live-Zell-Verbreitungstest derzeit für die Zellbiologie-Community nicht verfügbar. Zu diesem Zweck skizziert unser Protokoll die Verfahren zur Abbildung von lebenden Streuzellen und stellt ein automatisiertes Bildanalysetool zur Verfügung. Um diese Methode zu validieren, verwendeten wir die Arp2/3-Hemmung als experimentelle Behandlung und zeigten, dass die Hemmung der Funktion des Arp2/3-Komplexes die Zellausbreitung nicht festhielt, sondern eine signifikante Verringerung der Zellvorsprunggeschwindigkeit sowie die Stabilität von Zellkantenvorsprüngen verursachte, was zu gezackten Zellkanten führte. Diese Daten zeigen, dass die Kombination von Live-Zell-Bildgebung und automatisierter Bildanalyse ein nützliches Werkzeug ist, um die Zellkantendynamik zu analysieren und molekulare Komponenten zu identifizieren, die Lamellipodie regulieren.
Protokoll
1. Zellsaatierung
HINWEIS: Das beschriebene Zellstreuprotokoll wurde mit embryonalen Fibroblasten (MEFs) der Maus durchgeführt, die PH-Akt-GFP (ein fluoreszierender Marker für PIP3/PI(3,4)P2) exdrücken. Diese Zelllinie wurde durch die genomische Integration eines Expressionskonstrukts für PH-Akt-GFP (Addgene #21218) durch CRISPR-vermittelte Genbearbeitung erzeugt. In diesem Test können jedoch auch andere fluoreszierende Marker verwendet werden, die vorübergehend exprimiert oder in das Genom integriert sind. Für eine optimale Bildsegmentierung empfehlen wir die Verwendung von Fluoreszenzmarkern, die gleichmäßig im Zytoplasma verteilt sind, z.B. zytosolisches GFP.
- Kultur eine 10 cm Schale von Zellen zu 90% Koninfluenza.
- Sobald die Zellen die richtige Konfluenz erreicht haben, legen Sie einen 22 mm x 22 mm Abdeckschlupf (#1,5; 0,17 mm Dicke) in eine 35 mm Zellkulturschale. Beschichten Sie den Deckelschlupf mit 400 l Fibronectin, das in PBS auf eine Endkonzentration von 2,5 g/ml verdünnt wurde.
HINWEIS: Die Anzahl der für den Test erforderlichen Abdeckungen wird durch die Anzahl der experimentellen Bedingungen und die technische Nachbildung bestimmt. - Die 35-mm-Schale mit dem fibronectinbeschichteten Deckel in einen 37 °C, 5% CO2-Inkubator für 1 Stunde legen.
- Entfernen Sie die Schale mit dem Deckel aus dem Inkubator. Das Fibronectin ansaugen und den Deckelrutsch mit PBS waschen, indem man den Deckel zwei- bis dreimal sanft umgibt.
- Die Zellkulturmedien aus der 10 cm-Zellschale ansaugen und mit PBS waschen.
- Fügen Sie der 90%igen konfluenten Schale der Zellen 650 l von 0,05% Trypsin-EDTA hinzu und kippen Sie die Schale, um das Enzym gleichmäßig zu verteilen. Legen Sie das Gericht mit dem Trypsin für 1 Minute in den Brutkasten.
- Entfernen Sie die Schale mit den Zellen aus dem Inkubator. Fügen Sie 10 ml Zellkulturmedien in ein 15 ml Zentrifugenrohr ein. Fügen Sie schnell weitere 10 ml Medien in die Schale, um das Trypsin zu löschen.
- Pipette 1 ml der trypsinisierten Zellen in das 15 ml Zentrifugenrohr, um die Zellen zu verdünnen. Pipette den Inhalt der Röhre nach oben und unten, um eine gleichmäßige Verteilung der Zellen innerhalb der Medien zu gewährleisten. Bei Zelltypen mit hoher Aggregationsneigung wird empfohlen, Zellen durch ein Zellsieb (100 m Netzgröße) zu filtern, um das Auftreten von Zellverklumpungen zu minimieren.
- Aus dem Rohr, Pipette 500 - 1000 l verdünnte Zellen in die 35 mm Schale mit dem Deckelschlupf.
- Schütteln Sie die Schale vorsichtig, um die Zellen gleichmäßig auszubreiten. Stellen Sie sicher, dass der Deckschein bei einer Konfluenz von 10 % (50.000 Zellen/ml) liegt, und passen Sie das Volumen der verdünnten Zellen nach Bedarf an.
HINWEIS: Der Zweck, Zellen mit einer so geringen Konfluenz zu haben, besteht darin, sicherzustellen, dass sich in jedem Sichtfeld 1-2 polarisierte Zellen befinden, die verwendet werden, um das Ziel während der gesamten Zellstreuung zu fokussieren. - Durchgang 1/5 der verbleibenden Zellen in der 10 cm Schale in eine 6 cm Schale pro Behandlungsbedingung. Legen Sie die durchzogenen Gerichte und die 35 mm Schale mit dem Deckelrutsch über Nacht in den Brutkasten.
HINWEIS: Dies sind die Zellen, die für die Verbreitung von Dynamik analysiert werden.
2. Drogeninkubation und Zellrückgewinnung
- Fügen Sie jeweils 5 ml Zellkulturmedien in zwei 15 ml Zentrifugenröhren und 20 ml phenolrotfreies DMEM in je zwei 50 ml Zentrifugenröhren ein.
HINWEIS: Die Anzahl der Röhrenpaare (15 ml + 50 ml) sollte der Anzahl der Versuchsbedingungen entsprechen. - Um die Bedeutung von Arp2/3 für die Zellstreuung zu testen, pipette entweder der pharmakologische Inhibitor von Arp2/3, CK-666 oder die Kontrollbehandlung, wie DMSO, in jedes Paar zentrifugenrohre bis zur gewünschten Konzentration.
- Entfernen Sie die 6 cm langen Gerichte (siehe Schritt 1.11) aus dem Inkubator und aspirieren Sie die Medien. Waschen Sie das Geschirr mit warmen PBS.
- Fügen Sie den Inhalt der CK-666- oder DMSO-ergänzten 15 ml Zentrifugenrohre in jedes der durchzogenen Gerichte ein. Beschriften Sie jedes Gericht mit der richtigen medikamentösen Behandlung und legen Sie das Geschirr für eine Stunde in den Brutkasten.
- Entfernen Sie die Gerichte aus dem Inkubator und aspirieren Sie die Medien. Waschen Sie das Geschirr mit warmen PBS, um alle verbleibenden phenolroten Medien gründlich zu entfernen.
- Fügen Sie 230 l von 0,05% Trypsin-EDTA zu jeder 6 cm Schale hinzu und brüten Zellen für 1 Minute.
HINWEIS: Gegebenenfalls kann Trypsin durch einen nicht-proteolytischen Zellhaftungsblocker ersetzt werden. - Entfernen Sie die Gerichte aus dem Inkubator. Fügen Sie für jede Behandlung 5 ml medikamentenergänztes phenolrotes DMEM in ein 15 ml Zentrifugenrohr ein, das als "Tube B" bezeichnet wird. Fügen Sie dem entsprechenden Gericht weitere 5 ml des gleichen Mediums hinzu, um das Trypsin zu löschen. Übertragen Sie den Inhalt der Schale in ein 15 ml Zentrifugenrohr, das als "Tube A" bezeichnet wird.
- Übertragen Sie 1 ml Zellen aus Tube A in Tube B. Für jede Behandlung wiederholen.
- Legen Sie die Röhren A und B für 45 Minuten in den Inkubator, damit sich die Zellen von der Trypsinisierung erholen können. Lösen Sie die Kappe der Zentrifugenrohre leicht, bevor Sie sie in den Inkubator legen, um CO 2-Penetranz zu ermöglichen.
HINWEIS: Die Dauer der Wiederherstellungszeit kann für verschiedene Zelltypen variieren. Obwohl in unseren Experimenten eine 45-minütige Erholung eine vernachlässigbare Wirkung auf die Zelllebensfähigkeit hatte, können einige Zelltypen anoikis unterzogen werden, wenn sie zu lange in Suspension gehalten werden. Daher empfehlen wir, die optimale Erholungszeit empirisch zu bestimmen. Die optimale Erholungszeit ermöglicht eine schnelle und synchrone Zellausbreitung ohne tote oder apoptotische Zellen in der Probe.
3. Magnetische Kammervorbereitung
- Stellen Sie sicher, dass alle Teile einer 1 Well Chamlide Cell Magnetkammer, die einen 22 mm x 22 mm quadratischen Abdeckungsslip aufnehmen kann, vor dem Gebrauch gereinigt wurden.
- Entfernen Sie die 35-mm-Schale mit dem Deckel (siehe Schritt 1.11) aus dem Inkubator. Aspirieren Sie die Zellkulturmedien und waschen Sie den Coverslip mit warmen PBS.
- Entfernen Sie den Deckelrutsch mit einem Zangenpaar von der 35-mm-Schale und legen Sie den Deckelrutsch vorsichtig auf die Bodenplatte der Magnetkammer.
- Legen Sie die Silikondichtung auf den Deckel.
HINWEIS: Eine unsachgemäß platzierte Silikondichtung ist die häufigste Ursache für eine undichte Magnetkammer. Stellen Sie sicher, dass die Dichtung im Einzug der Bodenplatte ruht und nicht über den Einzug hinaussteigt. - Befestigen Sie den Hauptkörper auf der Bodenplatte.
HINWEIS: Machen Sie diesen Teil sehr langsam. Eine gute Spitze ist, die Bodenplatte mit einer Hand zu halten, während der Hauptkörper oben platziert wird. Dadurch wird sichergestellt, dass die Magnete des Hauptkörpers die Bodenplatte nicht nach oben heben, was den Deckelrutsch möglicherweise verdrängen und knacken könnte. - Fügen Sie 1 ml medikamentenergänztes phenolrotes DMEM in die Magnetkammer. Nehmen Sie ein fusselfreies Gewebe und tupferen Sie sorgfältig das Gehäuse zwischen dem Hauptkörper und der Bodenplatte, um auf Leckagen zu überprüfen.
HINWEIS: Wenn es lecken, schnell die Medien aspirieren und von Schritt 3.4 wieder fortfahren. - Senken Sie die transparente Abdeckung auf den Hauptkörper, um die magnetische Kammer einzuschließen.
- Sprühen Sie zuerst ein Laborgewebe mit Wasser und wischen Sie den Boden der Magnetkammer ab (den Deckel, nicht das Metallteil). Danach sprühen Sie ein zweites Laborgewebe mit einer kleinen Menge von 70% Ethanol und wischen Sie, wobei Sie darauf achten, den Deckelrutsch nicht zu knacken.
4. Bildaufnahme
- Den Bühnen-Top-Inkubator und die Objektivheizung auf 37 °C vorheizen und denCO2-Gehalt im Bühnen-Top-Inkubator auf 5% einstellen.
HINWEIS: Wenn der Bühnen-Top-Inkubator nicht an eineCO2-Versorgung angeschlossen ist, sollten die Zellkulturmedien um 25 mM HEPES ergänzt werden, um einen konstanten pH-Wert von 7,4 beizubehalten. - Tragen Sie eine ausreichende Menge Tauchöl auf das vorgewärmte 60X, 1.4 N.A. Ölobjektiv auf.
HINWEIS: Wir verwenden ein 60X, 1.4 N.A. Öl Immersion Objektiv in diesem Protokoll wegen seiner relativ großen Sichtfeld und hervorragende Lichtsammlung Effizienz. Wenn ein größeres Sichtfeld erforderlich ist, kann ein niedrigeres Vergrößerungsziel(z.B.20x) verwendet werden, solange das Signal-Rausch-Verhältnis der Bilder größer als 2,5 ist. - Bringen Sie sowohl die fertige Magnetkammer als auch Tube B (Schritt 2.9) zum konfokalen Mikroskop. Legen Sie die Magnetkammer auf den Bühnen-Inkubator.
HINWEIS: Legen Sie die Magnetische Kammer sanft auf die Bühne, um Blasen im Tauchöl zu vermeiden. - Legen Sie den Fokus auf die fluoreszierenden Zellen mithilfe des GFP-Kanals fest. Stellen Sie sicher, dass die Zellenkante scharf und gut definiert ist.
- Entfernen Sie die transparente Abdeckung der Magnetkammer und Pipette 500 l aus Tube B in die Magnetkammer. Legen Sie die transparente Abdeckung wieder auf die Magnetkammer.
- Um Zellen zu identifizieren, die für die Zellstreuanalyse ideal sind, suchen Sie nach "Halos" von Zellen, die noch nicht an den Deckzettel anhängen müssen, aber nicht mehr herumrollen. Zellen, die sich in den frühesten Stadien der Coverslip-Befestigung befinden, sind ebenfalls großartige Kandidaten, aber die Bildaufnahme muss schnell erfolgen, um die Ausbreitung zu erfassen.
- Konfigurieren Sie die Zeitraffer-Bildaufnahme für den grünen Kanal so, dass sie vier Sichtfelder enthält, die in 6-Sekunden-Intervallen abgebildet sind.
HINWEIS: Aufgrund der hohen Variabilität der Lamellipodia-Vorsprunggeschwindigkeit zwischen verschiedenen Zelltypen sollte die optimale Bildrate empirisch bestimmt werden. Das in unseren Experimenten verwendete Bildgebungsintervall von 6 Sekunden ist ein guter Ausgangspunkt für die Analyse vieler mesenchymaler und epitheliale Zellen. Zellen, die sich sehr schnell ausbreiten(z. B.Immunzellen), können jedoch eine viel höhere Bildrate (kürzeres Bildgebungsintervall) erfordern. Die optimale Bildrate für Zellstreufilme sorgt für eine Verschiebung der hervorstehenden Zellkante zwischen nachfolgenden Frames um 2-5 Pixel. Unter Berücksichtigung der Genauigkeit der Kurvenanpassung, die zur Identifizierung des Plateaus der Zellstreuung verwendet wird, sollte die optimale Bildrate auch 50-100 Messungen der Zellkantenverschiebung während der schnellen Expansionsphase der Zellstreuung sicherstellen. Die Anzahl der Sichtfelder sollte abhängig von der Belichtungszeit, dem Abstand zwischen Erfassungspunkten und der Stufenbewegungsgeschwindigkeit angepasst werden. Benutzern wird empfohlen, die maximale Anzahl von Ansichtsfeldern zu bestimmen, die mit der gewünschten Bildrate erfasst werden können. - Nachdem Sie ein geeignetes Sichtfeld identifiziert haben, speichern Sie die X- und Y-Koordinaten der Mikroskopstufe. Identifizieren Sie drei weitere Sichtfelder, die auf dem Deckblatt relativ nahe beieinander liegen. Speichern Sie die Koordinaten der Mikroskopstufe für jedes gewünschte Sichtfeld.
HINWEIS: Es wird dringend empfohlen, den Bewegungspfad zwischen Ansichtsfeldern zu optimieren, um unnötige Probenbewegungen zu minimieren. Eine solche Optimierung kann entweder manuell oder automatisch durchgeführt werden. Übermäßige Probenbewegungen verlangsamen die Erfassung und können dazu führen, dass Zellen beim Absteigen aus der Ansicht rollen. - Erfassen Sie Bilder für 15 Minuten mit einer Bildrate von 6 Sekunden und speichern Sie die Dateien. Wenn weitere Akquisitionen erforderlich sind, wiederholen Sie dies mit Schritt 4.6.
5. Analyse der Zellfläche, Zirkularität und Vorsprungdynamik während der Zellausbreitung
- Vorbereiten von Bildern für die Datenverarbeitung und -analyse
HINWEIS: Die Software benötigt ein Bild im .tiff Format und eine Pixelgröße als Eingabeparameter. Beide Anforderungen können mit der Erfassungssoftware oder Fidschi (in diesem Protokoll) erfüllt werden. Wenn diese Anforderungen erfüllt sind, fahren Sie mit Schritt 5.2 fort.- Installieren Sie die neueste Version der Fidschi-Anwendung (https://imagej.net/Fiji/Downloads).
- Öffnen Sie ein Zeitrafferbild mit Fidschi.
- Kopieren Sie die Pixelgröße des Bildes, indem Sie Bild > Eigenschaftenauswählen. Kopieren Sie die Pixelgröße in 'm' und fügen Sie sie in Notepad/Word ein.
- Speichern Sie für die Analyse des Zellstreubereichs und der Zirkularität das Zeitrafferbild als Tiff-Bildstapel. Die benutzerdefinierte Analysesoftware unterstützt keine proprietären Dateiformate. Speichern Sie den einzelnen Zell-Tiff-Bildstapel, indem Sie Datei > Speichern als > Tiffauswählen.
- Installieren Sie die Python IDE (Spyder) und die notwendigen Pakete (PySimpleGUI und tifffile) für die Datenverarbeitung und -analyse.
HINWEIS: Die Installation von Python und Paketen ist nur für die Ersteinrichtung erforderlich.- Die Zeitrafferfilme werden in der Spyder-IDE mithilfe eines benutzerdefinierten Python-Skripts analysiert. Um die Spyder IDE herunterzuladen, laden Sie den Anaconda-Distributor (https://www.anaconda.com/products/individual) herunter, der Spyder IDE und die meisten der für diese Analyse erforderlichen Bibliotheken und Pakete enthält.
- Installieren Sie Anaconda und starten Sie Spyder über Anaconda Navigator.
- Kopieren Sie auf der Registerkarte IPython-Konsole (im unteren rechten Bereich von Spyder) den folgenden Befehl: pip install PySimpleGUI installieren und die Eingabetaste drücken. Wenn Sie diesen Befehl ausführen, wird das Paket installiert, das zum Initiieren der grafischen Benutzeroberfläche (GUI) erforderlich ist.
- Kopieren Sie in derselben Konsole den folgenden Befehl: pip install tifffile, und drücken Sie die Eingabetaste. Wenn Sie diesen Befehl ausführen, wird das Paket installiert, das zum Speichern von Images als tiff-Dateien erforderlich ist.
- Laden Sie alle Python-Skripte aus den zusatzdateien oder die aktuellsten Skripte von GitHub herunter unter: https://github.com/ernestiu/Cell-spreading-analysis.git
- Quantifizierung von Zellflächen- und Zellformfaktoren während der Zellausbreitung
- Öffnen Sie das Hauptanalyseskript "cell_spreading_GUI.py", indem Sie die Option "Offene Datei" im oberen Bereich von Spyder auswählen oder die Tastenkombination Strg + Overwenden.
- Öffnen Sie die GUI für die Zellverteilungsanalyse, indem Sie im oberen Bereich "Datei ausführen" auswählen oder die Tastenkombination F5verwenden.
- Klicken Sie auf die Registerkarte Zellverteilungsbereich (Abbildung 3A).
- Wählen Sie das zu analysierende Tiff-Bild mit der Schaltfläche "Durchsuchen" aus.
HINWEIS: Die ausgewählte Datei muss eine tiff-Datei sein. - Geben Sie das Zielverzeichnis an, in dem Datenausgaben (z. B. Zellmasken, Werte) gespeichert werden sollen.
- Geben Sie die Datenausgabeeinstellungen an:
- Masken speichern: Speichern von Zellenmasken, die während des Segmentierungsprozesses generiert wurden.
- Exportieren von Daten: Exportieren Sie eine Excel-Tabelle (.xlsx), die alle Analysedaten enthält, in den Zielordner.
Zellfläche, Zirkularität und Seitenverhältnis aller Streuzellen werden als Excel-Tabelle im Zielordner gespeichert. Der Zellbereich wird wie folgt berechnet: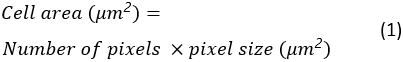
Die Zellzirkularität ist ein Maß dafür, wie nah eine Zelle an einer perfekt runden Zelle ist. Er wird wie folgt berechnet: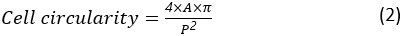
wobei A und P der Zellbereich bzw. der Zellumfang sind. Das Seitenverhältnis der Zelle stellt dar, wie langgestreckt die Zelle ist. Eine Streuzelle sollte ein Seitenverhältnis nahe 1 aufweisen. Das Seitenverhältnis wird wie folgt berechnet: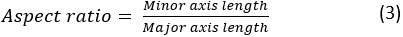
- Konturen speichern: Speichern Sie die Zellbegrenzungskontur-Überlagerungsbilder im Zielordner.
- Angeben der Segmentierungseinstellungen
- Segmentierung anzeigen: Zeigen Sie das Segmentierungsergebnis in der Spyder-Konsole während des Analyseprozesses an.
- Kleinster Zellbereich (m2): Geben Sie den Mindestwert für die Zellfläche ein, einschließlich der Flächenwerte von Zellen in den Anfangsstadien der Anhaftung. Objekte mit einem Bereich, der kleiner als dieser Schwellenwert ist, werden nicht als Streuzellen betrachtet. Diese Zahl wirkt sich auf den Segmentierungsprozess aus.
- Geben Sie die Bildparameter an.
- Erfassungsintervall (s): Geben Sie die Häufigkeit der Bildaufnahme in Sekunden ein.
- Pixelgröße (m): Geben Sie die Pixelgröße ein, die beim Vorbereiten von Bildern für die Analyse aufgezeichnet wurde.
- Bildbittiefe: Geben Sie die Bittiefe der Kamera/des Detektors ein.
- Klicken Sie auf Ausführen. Wenn ein Fehler auftritt, wird eine Fehlermeldung in der Spyder-Konsole angezeigt. Andernfalls wird der Bildanalyseprozess in der Konsole angezeigt.
HINWEIS: Das erste Bild, das im Abschnitt Konsole/Plots angezeigt wird (abhängig von den Spyder-Einstellungen), zeigt alle zellen, die im Sichtfeld identifiziert wurden. Grüne Felder, die um die Zellen gelegt werden, zeigen sich auf streuende Zellen, die für die Segmentierung und Analyse geeignet sind. Graue Felder zeigen Zellen an, die nicht für die Analyse geeignet sind. Die Gesamtzahl der identifizierten Verteilungszellen wird auch auf der Registerkarte Konsole angezeigt. Die Software zeichnet den Zellbereich (in blau) und die Zellzirkularität (in rot) als Funktion der Zeit. Mit diesen Diagrammen können Benutzer die Genauigkeit der Zellsegmentierung bewerten. Eine erfolgreiche Segmentierung ergibt eine monoton steigende Kurve für die Zellfläche. Um eine repräsentative Kurve des Zellstreubereichs zu erhalten, sollte die Verzögerungsphase manuell aus dem Diagramm entfernt werden. Die Verzögerungsphase umfasst Messungen der Zellfläche, bevor sich die Zelle ausbreitet. Die Verzögerungsphase wird durch schnelle Schwankungen angezeigt, wie sie im Zellflächendiagramm dargestellt sind(Abbildung 3C rechts).
6. Quantifizierung der Zellkantendynamik während der Zellstreuung mit Kymographen
- Schneiden Sie vor der Analyse die Rohfilme von sich ausbreitenden Zellen zu, um Zeitreihen einzelner Sichtzellen zu erstellen.
- Verwenden Sie das Rechteck-Werkzeug in der Werkzeugleiste Fidschi, um manuell einen Bereich von Interesse (ROI) auszuwählen, der eine einzelne Zellekapselt. (Um sicherzustellen, dass der ROI die Streuzelle vollständig kapselt, verwenden Sie die Bildlauffunktion, um den ROI jederzeit zu überprüfen.)
- Klicken Sie mit der rechten Maustaste auf den ROI und wählen Sie Duplizierenaus.
- Überprüfen Sie Duplicate Stack und klicken Sie auf OK.
- Öffnen Sie das Hauptanalyseskript "cell_spreading_GUI.py", indem Sie die Schaltfläche Datei öffnen in der Symbolleiste von Spyder auswählen oder die Tastenkombination Strg + Overwenden. Wenn die GUI bereits geöffnet wurde, gehen Sie direkt zu Schritt 6.3.
- Öffnen Sie die GUI für die Zellstreuanalyse, indem Sie Datei im oberen Bereich ausführen auswählen oder die Tastenkombination F5 (Abbildung 3B) verwenden.
- Klicken Sie auf die Registerkarte Kymographgenerator & Analyse.
- Verwenden Sie die Schaltfläche "Durchsuchen", um das tiff-Bild für die Analyse auszuwählen.
HINWEIS: Proprietäre Dateiformate, z.B. nd2, lif, zen, werden vom Skript nicht unterstützt. - Geben Sie den Zielordner an, um die Ausgabedaten zu speichern (Zellenmasken und Werte).
- Geben Sie die Ausgabeeinstellungen an.
- Exportieren von Daten: Exportieren Sie eine Excel-Tabelle (.xlsx) in den Zielordner, der relative Zellenkantenpositionen und Rückzugsereignisse der Kymographen enthält.
- Geben Sie die Bildparameter an:
- Erfassungsintervall (s): Geben Sie die Bildaufnahmehäufigkeit in Sekunden ein.
- Pixelgröße ('m') : Geben Sie die Pixelgröße ein, die beim Vorbereiten von Bildern für die Analyse in Schritt 5.1.3 aufgezeichnet wurde.
- Kleinster Zellbereich (m2): Geben Sie den Mindestwert für die Zellfläche ein, einschließlich der Flächenwerte von Zellen in den Anfangsstadien der Anhaftung. Objekte mit einem Bereich, der kleiner als dieser Schwellenwert ist, werden nicht als Streuzellen betrachtet. Diese Zahl wirkt sich auf den Segmentierungsprozess aus.
- Bildbittiefe: Geben Sie die Bittiefe der Kamera/des Detektors ein.
- Klicken Sie auf Ausführen. Wenn ein Fehler auftritt, wird eine Fehlermeldung in der Spyder-Konsole angezeigt. Andernfalls wird eine Zusammenfassung der Protrusionsdynamik-Quantifizierungen in der Konsole angezeigt. Es wird 4 Paare von Rückzugsfrequenz und Vorsprunggeschwindigkeit Messungen, die aus 4 Kymographen aus dem oberen, unteren, linken und rechten Teil der Zelle generiert extrahiert werden. Die Rückzugshäufigkeit wird wie folgt berechnet:
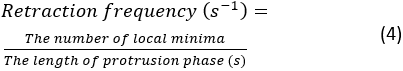
HINWEIS: Diese Zahl zeigt, wie häufig sich das Lamellipodium im Laufe der Ausbreitung zurückzieht. Die durchschnittliche Vorsprunggeschwindigkeit wird durch die Steigung zwischen dem Beginn des Vorsprungs und dem Plateaupunkt auf dem Kymograph gemessen. Eine zusammenfassende Kymographenfigur wird nach der Segmentierung in der Konsole angezeigt. Um die Zusammenfassungzulage zu speichern, klicken Sie mit der rechten Maustaste auf die Abbildung und speichern Sie das Bild.
Ergebnisse
Das obige Protokoll beschreibt die experimentellen Verfahren zur Live-Zell-Bildgebung von Streuzellen und ein Rechenwerkzeug für die quantitative Analyse der Zellstreudynamik. Das Rechenwerkzeug kann in einem Format mit niedrigem oder hohem Durchsatz verwendet werden, um die molekularen Akteure zu identifizieren, die die Aktinpolymerisationsmaschinerie an der Zellvorderkante regulieren.
Die schematische Darstellung der experimentellen Verfahren ist in Abbildung 1...
Diskussion
Der beschriebene Zellstreutest ermöglicht die kontinuierliche Verfolgung morphologischer Veränderungen(z. B. Zellgröße und -form) und Zellkantenbewegungen(d. h. Vorsprunggeschwindigkeit und Rückzugshäufigkeit), die in den meisten Zellstreuprotokollen19,24fehlen. Während häufig verwendete Endpunktzellstreuungs-Assays die Bestimmung der Zellstreugeschwindigkeit ermöglichen, können diese Assays die zeitliche Dynamik von Zellkantenbewegung...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch den Connaught Fund New Investigator Award an S.P., Canada Foundation for Innovation, NSERC Discovery Grant Program (Grants RGPIN-2015-05114 und RGPIN-2020-05881), University of Manchester und University of Toronto Joint Research Fund und University of Toronto XSeed Program unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
Referenzen
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten