É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise quantitativa da dinâmica da borda celular durante a disseminação celular
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Neste protocolo, apresentamos os procedimentos experimentais de um ensaio de difusão celular que é baseado em microscopia de células vivas. Fornecemos uma ferramenta computacional de código aberto para a segmentação imparcial de células fluorescentes rotuladas e análise quantitativa da dinâmica da lamellipodia durante a disseminação celular.
Resumo
A disseminação celular é um processo dinâmico no qual uma célula suspensa em mídia se conecta a um substrato e se achata de uma forma arredondada para uma forma fina e espalhada. Seguindo o acessório de substrato celular, a célula forma uma fina folha de lamellipodia emanando do corpo celular. Na lamellipodia, os monômeros globulares (G-actin) polimerizam-se em uma densa malha de ato desordenada (F-actin) que empurra contra a membrana plasmática, fornecendo assim as forças mecânicas necessárias para que a célula se espalhe. Notavelmente, os atores moleculares que controlam a polimerização de actina na lamellipodia são essenciais para muitos outros processos celulares, como migração celular e endocitose.
Uma vez que a disseminação de células forma lamellipodia contínua que abrange toda a periferia celular e se expande persistentemente para fora, ensaios de disseminação celular tornaram-se uma ferramenta eficiente para avaliar a cinética das protrusões lamellipodial. Embora várias implementações técnicas do ensaio de disseminação de células tenham sido desenvolvidas, uma descrição detalhada do fluxo de trabalho, que incluiria um protocolo passo a passo e ferramentas computacionais para análise de dados, está atualmente em falta. Aqui, descrevemos os procedimentos experimentais do ensaio de disseminação celular e apresentamos uma ferramenta de código aberto para análise quantitativa e imparcial da dinâmica da borda celular durante a disseminação. Quando combinado com manipulações farmacológicas e/ou técnicas de silenciamento de genes, este protocolo é favorável a uma tela em larga escala de jogadores moleculares que regulam saliências lamellipodiais.
Introdução
As saliências lamellipodias são estruturas citoesqueléticos proeminentes formadas na frente de uma célula migratória. Em lamellipodia, a polimerização do actin com o auxílio do complexo Arp2/3 e formins cria um trabalho de malha de actina ramificada de rápido crescimento que empurra contra a membrana plasmática1,2. A força de empurrão gerada pelo trabalho de malha de actina impulsiona fisicamente a célula para a frente1,3,4,5. Esgotamento do complexo Arp2/3 ou interrupção de vias de sinalização essenciais para saliências lamellipodias muitas vezes prejudicam a migração celular6, 7. Embora a migração de células deficientes de lamellipodia também tenha sido relatada8,9, a importância da lamellipodia na migração celular é evidente como o esgotamento dessa estrutura protrusiva perturba a capacidade da célula de se mover através de microambientes biológicos complexos6,10.
Um grande empecilho para entender a regulação da lamellipodia em células migratórias é a variabilidade natural na cinética de protrusão lamellipodial, tamanho e forma11,12,13,14. Além disso, estudos recentes demonstraram que a lamellipodia apresenta comportamentos salitivos complexos, incluindo saliências flutuantes, periódicas e aceleradas14,15. Em comparação com a lamellipodia altamente variável das células migratórias6,16, a lamellipodia formada durante a disseminação celular são mais uniformes12. Uma vez que a atividade strusiva de células disseminadas e migratórias é impulsionada por conjuntos macromoleculares idênticos, que incluem uma rede de actina ramificada, feixes de actomyosina contracttil e aderências de matriz celular baseada em integrin17,18, células disseminadas têm sido amplamente utilizadas como modelo para investigar a regulação da dinâmica da lamellipodia.
A disseminação celular é um processo mecanoquímico dinâmico onde uma célula em suspensão primeiro adere a um substrato através de aderências baseadas em integrin17,19,20 e, emseguida,se espalha estendendo as saliências baseadas em actina21,22,23. Durante a fase de disseminação, a lamellipodia emanando do corpo celular protuberante e persistentemente com pouca ou nenhuma retração ou paralisação12. Os protocolos de disseminação de células mais usados são os ensaios de pontos finais, onde as células de disseminação são fixadas em vários momentos após o revestimento19,24. Estes ensaios, embora rápidos e simples, são limitados em seu poder de diagnóstico para detectar alterações nas características dinâmicas da lamellipodia. Para determinar os mecanismos moleculares que controlam a dinâmica da lamellipodia, o grupo Sheetz foi pioneiro no uso da análise quantitativa de células vivas e descobriu muitas propriedades fundamentais das saliências de borda celular11,12,22. Esses estudos demonstraram que o ensaio de disseminação de células vivas é uma técnica robusta e poderosa na caixa de ferramentas de um laboratório de biologia celular. Apesar disso, um protocolo detalhado e uma ferramenta computacional de código aberto para um ensaio de espalhamento de células vivas estão atualmente indisponíveis para a comunidade de biologia celular. Para isso, nosso protocolo descreve os procedimentos de imagens ao vivo espalhando células e fornece uma ferramenta automatizada de análise de imagens. Para validar este método, utilizamos a inibição de Arp2/3 como tratamento experimental e mostramos que inibir a função do complexo Arp2/3 não dedundo a propagação celular, mas causou uma redução significativa na velocidade de saliência celular, bem como a estabilidade das saliências da borda celular, dando origem a bordas celulares irregulares. Esses dados demonstram que a combinação de imagens de células vivas e análise automatizada de imagens é uma ferramenta útil para analisar a dinâmica das bordas celulares e identificar componentes moleculares que regulam a lamellipodia.
Protocolo
1. Semeadura celular
NOTA: O protocolo de disseminação de células descrito foi realizado utilizando fibroblastos embrionários de camundongos (MEFs) expressando PH-Akt-GFP (um marcador fluorescente para PIP3/PI(3,4)P2). Esta linha celular foi gerada pela integração genomicamente de uma construção de expressão para PH-Akt-GFP (Addgene #21218) pela edição de genes mediada pelo CRISPR. No entanto, outros marcadores fluorescentes que são expressos transitoriamente ou integrados no genoma também podem ser usados neste ensaio. Para a segmentação de imagem ideal, recomendamos o uso de marcadores fluorescentes que são distribuídos uniformemente no citoplasma, por exemplo, GFP citosolic.
- Cultura um prato de 10 cm de células para 90% de confluência.
- Uma vez que as células tenham atingido a confluência adequada, coloque uma tampa de 22 mm x 22 mm (#1,5; 0,17 mm de espessura) em um prato de cultura celular de 35 mm. Cubra com 400 μL de fibronectina que foi diluída em PBS para uma concentração final de 2,5 μg/mL.
NOTA: O número de tampas necessárias para o ensaio é determinado pelo número de condições experimentais e réplica técnica. - Coloque o prato de 35 mm com a tampa revestida de fibronectina em uma incubadora de 37 °C, 5% DE CO2 por 1 hora.
- Retire o prato com a tampa da incubadora. Aspire a fibronectina e lave a tampa com PBS, encanar suavemente ao redor da tampa de duas a três vezes.
- Aspire a mídia de cultura celular a partir do prato de 10 cm de células e lave o prato com PBS.
- Adicione 650 μL de 0,05% de trypsin-EDTA ao prato 90% confluente de células, inclinando o prato para distribuir uniformemente a enzima. Coloque o prato com o trypsin na incubadora por 1 minuto.
- Remova o prato com as células da incubadora. Adicione 10 mL de mídia de cultura celular em um tubo de centrífuga de 15 mL. Adicione rapidamente mais 10 mL de mídia no prato para saciar o trypsin.
- Pipeta 1 mL das células experimentpsinizadas no tubo centrífuga de 15 mL, a fim de diluir as células. Pipeta o conteúdo do tubo para cima e para baixo para garantir uma distribuição uniforme das células dentro da mídia. Para tipos de células com alta propensão à agregação, a filtragem de células através de um coador de células (tamanho de malha de 100 μm) é recomendada para minimizar a ocorrência de aglomerados celulares.
- Do tubo, pipeta 500 - 1000 μL de células diluídas no prato de 35 mm contendo o deslizamento de tampa.
- Agite suavemente o prato para espalhar uniformemente as células. Certifique-se de que o deslizamento de tampa esteja em uma confluência de ~10% (~50.000 células/mL) e ajuste o volume de células diluídas conforme necessário.
NOTA: O objetivo de ter células em uma confluência tão baixa é garantir que haja 1-2 células polarizadas em cada campo de visão que serão usadas para focar o objetivo durante toda a aquisição de difusão celular. - Passagem 1/5 das células restantes no prato de 10 cm em um prato de 6 cm por condição de tratamento. Coloque os pratos perequiclados e o prato de 35 mm com a tampa na incubadora durante a noite.
NOTA: Estas serão as células que serão analisadas para difundir dinâmicas.
2. Incubação de Medicamentos e Recuperação celular
- Adicione 5 mL de mídia de cultura celular em cada um dos dois tubos de centrífugas de 15 mL, e 20 mL de DMEM livre de fenol vermelho em cada um dos dois tubos de centrífuga de 50 mL.
NOTA: O número de pares de tubos (15 mL + 50 mL) deve corresponder ao número de condições experimentais. - Para testar a importância do Arp2/3 para a disseminação celular, pipeta seja o inibidor farmacológico de Arp2/3, CK-666, ou o tratamento de controle, como o DMSO, em cada par de tubos de centrífuga até a concentração desejada.
- Retire os pratos de 6 cm (ver Passo 1.11) da incubadora e aspire a mídia. Lave os pratos com PBS quente.
- Adicione o conteúdo dos tubos de centrífugas de CK-666 ou DMSO em cada um dos pratos com passagem. Rotule cada prato com o tratamento correto da droga e coloque os pratos na incubadora por uma hora.
- Retire os pratos da incubadora e aspire a mídia. Lave os pratos com PBS quente, a fim de remover completamente toda a mídia vermelha fenol restante.
- Adicione 230 μL de 0,05% de trypsin-EDTA a cada prato de 6 cm e incubar células por 1 minuto.
NOTA: Se aplicável, a trippsina pode ser substituída por um bloqueador de adesão celular não proteolítica. - Retire os pratos da incubadora. Para cada tratamento, adicione 5 mL de DMEM livre de fenol suplementado com drogas em um tubo de centrífuga de 15 mL designado como "Tubo B". Adicione mais 5 mL da mesma mídia no prato relevante para saciar o trypsin. Transfira o conteúdo do prato para um tubo de centrífuga de 15 mL designado como "Tubo A".
- Transfira 1 mL de células do tubo A para o tubo B. Repita para cada tratamento.
- Coloque os tubos A e B na incubadora por 45 minutos para permitir que as células se recuperem da experimentação. Solte ligeiramente a tampa dos tubos de centrífuga antes de colocá-los na incubadora para permitir a penetração de CO2.
NOTA: A duração do tempo de recuperação pode variar para diferentes tipos de células. Embora em nossos experimentos a recuperação de 45 minutos tenha tido um efeito insignificante na viabilidade celular, alguns tipos de células podem sofrer anoikis quando mantidos em suspensão por muito tempo. Por isso, recomendamos determinar o tempo ideal de recuperação empiricamente. O tempo ideal de recuperação permite que células rápidas e síncrogas se espalhem sem células mortas ou apoptóticas na amostra.
3. Preparação da Câmara Magnética
- Certifique-se de que todas as partes de uma Câmara Magnética celular de 1 poço que possa acomodar uma cobertura quadrada de 22 mm x 22 mm foram limpas antes do uso.
- Retire o prato de 35 mm com o deslizamento (ver Passo 1.11) da incubadora. Aspire a mídia de cultura celular e lave o deslizamento com PBS quente.
- Remova a mancha de cobertura da placa de 35 mm usando um par de fórceps e coloque suavemente a mancha na placa inferior da câmara magnética.
- Coloque a junta de silicone em cima da tampa.
NOTA: Uma junta de silicone mal colocada é a causa mais comum de uma câmara magnética com vazamento. Certifique-se de que a junta repousa no travessão da placa inferior e não suba além do travessão. - Coloque o corpo principal na placa inferior.
NOTA: Faça esta parte muito lentamente. Uma boa dica é segurar a placa inferior com uma mão enquanto coloca o corpo principal em cima. Isso garante que os ímãs do corpo principal não levantem a placa inferior para cima, o que poderia potencialmente deslocar e quebrar a tampa. - Adicione 1 mL de DMEM livre de fenol suplementado a medicamentos à câmara magnética. Pegue um tecido sem fiapos e desça cuidadosamente o invólucro entre o corpo principal e a placa inferior, a fim de verificar se há vazamentos.
NOTA: Se houver vazamento, aspire rapidamente a mídia e prossiga novamente a partir da etapa 3.4. - Abaixe a tampa transparente sobre o corpo principal para fechar a câmara magnética.
- Pulverize um tecido de laboratório primeiro com água e limpe o fundo da câmara magnética (o deslizamento de cobertura, não a parte metálica). Em seguida, pulverize um segundo tecido de laboratório com uma pequena quantidade de 70% de etanol e limpe, tomando cuidado para não quebrar a tampa.
4. Aquisição de Imagens
- Pré-aqueça a incubadora superior do estágio e o aquecedor objetivo para 37 °C e coloque o nível de CO2 na incubadora superior do estágio para 5%.
NOTA: Se a incubadora superior do estágio não estiver conectada a uma fonte de CO2, a mídia de cultura celular deve ser complementada com HEPES de 25 mM para manter o pH constante 7.4. - Aplique uma quantidade suficiente de óleo de imersão ao objetivo de óleo pré-aquecido 60X, 1.4 N.A.
NOTA: Usamos um objetivo de imersão de óleo de 60X, 1.4 N.A. neste protocolo devido ao seu campo de visão razoavelmente grande e excelente eficiência de coleta de luz. Se for necessário um campo de visão maior, um objetivo de ampliação mais baixo (por exemplo,20x) pode ser usado desde que a relação sinal-ruído das imagens seja superior a 2,5. - Leve tanto a câmara magnética completa quanto o tubo B (Passo 2.9) para o microscópio confocal. Coloque a câmara magnética na incubadora superior do palco.
NOTA: Coloque a câmara magnética suavemente no palco para evitar a criação de bolhas no óleo de imersão. - Defina o foco para as células fluorescentes usando o canal GFP. Certifique-se de que a borda da célula está afiada e bem definida.
- Remova a tampa transparente da câmara magnética e a pipeta de 500 μL do tubo B para a câmara magnética. Coloque a tampa transparente de volta em cima da câmara magnética.
- Para identificar células ideais para análise de difusão celular, procure por "halos" de células que ainda não se anexaram ao deslizamento de tampas, mas não estão mais rolando. As células que estão nos estágios iniciais do anexo coverlip também são grandes candidatos, mas a aquisição de imagens deve ser rápida para capturar a propagação.
- Configure a aquisição de imagem de lapso de tempo para o canal verde para incluir quatro campos de exibição, visualizados em intervalos de 6 segundos.
NOTA: Devido à alta variabilidade da velocidade de saliência lamellipodia entre diferentes tipos de células, a taxa de quadros ideal deve ser determinada empiricamente. O intervalo de imagem de 6 segundos usado em nossos experimentos é um bom ponto de partida para a análise de muitas células mesenquimais e epiteliais. No entanto, as células que se espalham muito rapidamente (por exemplo,células imunes) podem exigir uma taxa de quadros muito maior (menor intervalo de imagem). A taxa de quadros ideal para filmes de espalhamento de células garante um deslocamento de 2-5 pixels da borda da célula saliente entre os quadros subsequentes. Considerando a precisão do encaixe da curva usado para identificar o platô de propagação celular, a taxa de quadro ideal também deve garantir 50-100 medições do deslocamento da borda celular durante a fase de expansão rápida da expansão celular. O número de campos de visão deve ser ajustado dependendo do tempo de exposição, da distância entre os pontos de aquisição e da velocidade de movimento do estágio. Os usuários são aconselhados a determinar o número máximo de campos de visão que podem ser adquiridos com a taxa de quadros desejada. - Depois de identificar um campo de visão adequado, salve as coordenadas X e Y do estágio do microscópio. Proceda com a identificação de outros três campos de visão que estão relativamente próximos um do outro no deslizamento de cobertura. Guarde as coordenadas do estágio do microscópio para cada campo de visão desejado.
NOTA: Recomenda-se otimizar fortemente o caminho de movimento do palco entre os campos de visão, a fim de minimizar qualquer movimento amostral desnecessário. Essa otimização pode ser realizada manualmente ou automaticamente. O movimento excessivo da amostra retarda a aquisição e pode fazer com que as células se distribuam fora de vista à medida que estão descendo. - Adquira imagens por 15 minutos a uma taxa de quadro de 6 segundos e salve os arquivos. Se for necessário mais aquisições, repita a partir da Etapa 4.6.
5. Análise da área celular, circularidade e dinâmica de saliência durante a disseminação celular
- Prepare imagens para processamento e análise de dados
NOTA: O software requer uma imagem em formato .tiff e um tamanho de pixel como parâmetros de entrada. Ambos os requisitos podem ser cumpridos usando o software de aquisição ou Fiji (neste protocolo). Se esses requisitos forem cumpridos, proceda à etapa 5.2.- Instale a versão mais recente do aplicativo Fiji (https://imagej.net/Fiji/Downloads).
- Abra uma imagem de lapso de tempo usando Fiji.
- Copie o tamanho do pixel da imagem selecionando Propriedades > de imagem. Copie e cole o tamanho do pixel em μm para Bloco de Notas/Word.
- Para a análise da área de disseminação celular e da circularidade, salve a imagem de lapso de tempo como uma pilha de imagens de tiff. O software de análise de compilação personalizada não suporta formatos de arquivo proprietários. Salve a pilha de imagens de tiff de células individuais selecionando Arquivo > Salvar como > Tiff.
- Instale o Python IDE (Spyder) e os pacotes necessários (PySimpleGUI e tifffile) para processamento e análise de dados.
NOTA: A instalação do Python e dos pacotes só é necessária para a configuração inicial.- Os filmes de lapso de tempo serão analisados no Spyder IDE usando um script Python de compilação personalizada. Para baixar o Spyder IDE, baixe o distribuidor Anaconda (https://www.anaconda.com/products/individual) que inclui Spyder IDE e a maioria das bibliotecas e pacotes necessários para esta análise.
- Instale a Anaconda e inicie o Spyder através do Anaconda Navigator.
- Na aba do console IPython (localizada na seção inferior direita do Spyder), copie e cole o seguinte comando: indique instale PySimpleGUI e pressione a tecla enter. A execução deste comando instalará o pacote necessário para iniciar a interface gráfica do usuário (GUI).
- No mesmo console, copie e cole o seguinte comando: pip instale o arquivo e pressione a tecla Enter. A execução deste comando instalará o pacote necessário para salvar imagens como arquivos de tiff.
- Baixe todos os scripts python dos arquivos suplementares ou dos scripts mais atualizados do GitHub em: https://github.com/ernestiu/Cell-spreading-analysis.git
- Quantificar a área celular e os fatores de forma celular durante a disseminação celular
- Abra o script principal de análise "cell_spreading_GUI.py" selecionando a opção de arquivo aberto no painel superior do Spyder ou usando o atalho Ctrl + O.
- Abra a gui de análise de expansão da célula selecionando "Executar arquivo" no painel superior ou usando o atalho F5.
- Clique na guia Área de Difusão celular (Figura 3A).
- Selecione a imagem de tiff a ser analisada usando o botão de navegação.
NOTA: O arquivo selecionado deve ser um arquivo de tiff. - Especifique o diretório de destino onde as saídas de dados (por exemplo, máscaras de células, valores) serão salvas.
- Especifique as configurações de saída de dados:
- Salve máscaras: Salve máscaras celulares geradas durante o processo de segmentação.
- Dados de exportação: Exporte uma planilha excel (.xlsx) que contenha todos os dados de análise para a pasta de destino.
A área celular, a circularidade e a proporção de todas as células em expansão serão salvas como uma planilha do Excel na pasta de destino. A área celular é calculada como: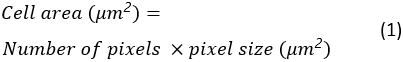
A circularidade celular é uma medida de quão perto uma célula está de uma célula perfeitamente redonda. É calculado como: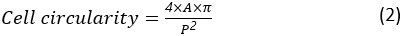
onde A e P são a área celular e o perímetro celular, respectivamente. A proporção da célula representa o quão alongada a célula é. Uma célula que se espalha deve ter uma proporção próxima de 1. A proporção é calculada como: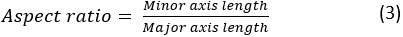
- Salve contornos: Salve as imagens de sobreposição do contorno do limite da célula na pasta de destino.
- Especifique as configurações de segmentação
- Mostrar segmentação: Mostrar o resultado de segmentação no console Spyder durante o processo de análise.
- Menor área celular (μm2): Digite o valor mínimo para a área celular, incluindo valores de área das células nos estágios iniciais de apego. Objetos com uma área menor do que este limiar não serão considerados como células disseminoras. Esse número afetará o processo de segmentação.
- Especifique os parâmetros de imagem.
- Intervalo de aquisição (s): Digite a frequência da aquisição da imagem em segundos.
- Tamanho do pixel (μm): Digite o tamanho do pixel que foi registrado ao preparar imagens para análise.
- Profundidade da broca da imagem: Digite a profundidade do bit da câmera/detector.
- Clique em Executar. Se um erro surgir, uma mensagem de erro aparecerá no console do Spyder. Caso contrário, o processo de análise de imagem será mostrado no console.
NOTA: A primeira imagem a aparecer na seção console/Plots (dependendo das configurações spyder) mostra todas as células identificadas no campo de exibição. Caixas verdes colocadas ao redor das células indicam células disseminar que são adequadas para segmentação e análise. Caixas cinzentas indicam células que não são adequadas para análise. O número total de células de espalhamento identificadas também aparecerá na guia Console. O software traça a área celular (em azul) e a circularidade celular (em vermelho) em função do tempo. Esses gráficos permitem que os usuários avaliem a precisão da segmentação celular. Uma segmentação bem sucedida produz uma curva monotonicamente crescente para a área celular. Para obter uma curva representativa da área de propagação celular, a fase de defasagem deve ser removida manualmente do gráfico. A fase de defasagem inclui medidas da área celular antes que a célula comece a se espalhar. A fase de defasagem é indicada por flutuações rápidas, conforme representado no enredo da área celular(Figura 3C à direita).
6. Quantifique a dinâmica da borda celular durante a disseminação celular usando kymografos
- Antes de executar a análise, corte os filmes crus de células disseminados para criar séries temporidas de células espalhadas individuais.
- Use a ferramenta Retângulo na barra de ferramentas Fiji para selecionar manualmente uma região de interesse (ROI) que encapsula uma única célula. (Para garantir que o ROI encapsula totalmente a célula de disseminação, use a função de pergaminho para inspecionar o ROI em todos os pontos de tempo.)
- Clique com o botão direito do mouse no ROI e selecione Duplicar.
- Verifique a pilha duplicada e clique em OK.
- Abra o script principal de análise "cell_spreading_GUI.py" selecionando o botão Arquivo Aberto na barra de ferramentas do Spyder ou usando o atalho Ctrl + O. Se a GUI já foi aberta, vá diretamente para o Passo 6.3.
- Abra a gui de análise de expansão da célula selecionando o arquivo Executar no painel superior ou usando o atalho F5 (Figura 3B).
- Clique na guia gerador &analysis do gerador e análise de Kymograph.
- Use o botão de navegação para selecionar a imagem de tiff para a análise.
NOTA: Formatos de arquivo proprietários, por exemplo, nd2, lif, zen, não são suportados pelo script. - Especifique a pasta de destino para salvar os dados de saída (máscaras e valores de células).
- Especifique as configurações de saída.
- Dados de exportação: Exporte uma planilha excel (.xlsx) para a pasta de destino que contém posições relativas de borda celular e eventos de retração dos kymographs.
- Especifique os parâmetros de imagem:
- Intervalo de aquisição (s): Digite a frequência de aquisição de imagens em segundos.
- Tamanho do pixel (μm): Digite o tamanho do pixel que foi registrado ao preparar imagens para a análise no Passo 5.1.3.
- Menor área celular (μm2): Digite o valor mínimo para a área celular, incluindo valores de área das células nos estágios iniciais de apego. Objetos com uma área menor do que este limiar não serão considerados como células disseminoras. Esse número afetará o processo de segmentação.
- Profundidade da broca da imagem: Digite a profundidade do bit da câmera/detector.
- Clique em Executar. Se um erro surgir, uma mensagem de erro aparecerá no console do Spyder. Caso contrário, um resumo das quantificações da dinâmica de saliência será mostrado no console. Haverá 4 pares de frequência de retração e medidas de velocidade de saliência, que são extraídos de 4 kymografos gerados das partes superior, inferior, esquerda e direita da célula. A frequência de retração é calculada como:
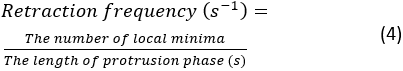
NOTA: Este número demonstra com que frequência o lamellipodium se retrai ao longo da propagação. A velocidade média de saliência é medida pela inclinação entre o início da saliência e o ponto de planalto no kymograph. Uma figura de kymograph sumária será mostrada no console após a segmentação. Para salvar a figura de resumo, clique com o botão direito do mouse na figura e salve a imagem.
Resultados
O protocolo acima descreve os procedimentos experimentais para a imagem de células vivas de células disseminadas e uma ferramenta computacional para a análise quantitativa da dinâmica de disseminação celular. A ferramenta computacional pode ser usada em um formato de baixo ou alto rendimento para identificar os atores moleculares que regulam o maquinário de polimerização de actina na borda principal da célula.
A representação esquemática dos procedimentos experimentais é retratad...
Discussão
O ensaio de disseminação de células descrito permite o rastreamento contínuo de alterações morfológicas (por exemplo, tamanho e forma da célula) e movimentos de borda celular(ou seja, velocidade de saliência e frequência de retração), que são características ausentes na maioria dos protocolos de disseminaçãocelular 19,24. Embora os ensaios de disseminação de células de ponto final comumente usados permitam a determinação da ...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pelo Connaught Fund New Investigator Award to S.P., Canada Foundation for Innovation, NSERC Discovery Grant Program (bolsas RGPIN-2015-05114 e RGPIN-2020-05881), University of Manchester and University of Toronto Joint Research Fund e University of Toronto XSeed Program.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
Referências
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados