このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
細胞拡散時の細胞エッジダイナミクスの定量的解析
要約
本プロトコルでは、生細胞顕微鏡に基づく細胞拡散アッセイの実験手順を提示する。蛍光標識細胞の偏りのないセグメンテーションや細胞拡散時のラメリポディアダイナミクスの定量分析のためのオープンソース計算ツールを提供します。
要約
細胞の拡散は、媒体に懸濁した細胞が基板に付着し、丸みを帯びた薄い広がり形状に平坦化する動的なプロセスです。細胞基板の付着に続いて、細胞は細胞体から発せられる薄いラメリポディアのシートを形成する。ラメリポディアでは、球状アクチン(G-アクチン)モノマーが、細胞膜に押し出される緻密な糸状アクチン(F-アクチン)メッシュワークに重合し、細胞が広がるのに必要な機械的力を提供する。特に、ラメリポディアにおけるアクチン重合を制御する分子奏者は、細胞移動やエンドサイトーシスなどの他の多くの細胞プロセスに不可欠です。
広がる細胞は、細胞周辺全体に広がる連続的なラメリポディアを形成し、持続的に外側に拡大するので、細胞拡散アッセイは、ラメリポジア性突起の運動を評価するための効率的なツールとなっています。細胞拡散アッセイのいくつかの技術的実装が開発されているが、データ分析のためのステップバイステップのプロトコルと計算ツールの両方を含むワークフローの詳細な説明は、現在欠けている。ここでは、細胞拡散アッセイの実験手順を説明し、広がる際の細胞エッジダイナミクスの定量的かつ公平な分析のためのオープンソースツールを提示する。薬理学的操作および/または遺伝子サイレンシング技術と組み合わせると、このプロトコルは、ラメリポアル突起を調節する分子プレーヤーの大規模なスクリーンに適しています。
概要
ラメリポジア性突起は、移動細胞の前部に形成される顕著な細胞骨格構造である。ラメリポディアでは、Arp2/3複合体およびフォルミンの助けを借りてアクチンを重合すると、血漿膜1,2に対して押し出される急速に成長する分岐アクチンメッシュワークが作成される。アクチンメッシュワークによって生じる押し出し力は、細胞を1、3、4、5に物理的に推進する。ラメリポジア突起に不可欠なArp2/3複合体またはシグナル伝達経路の破壊の枯渇は、しばしば細胞の移動を損なう6,7。 また、ラメリポディア欠損細胞の移動も報告されているが、8,9、細胞遊離におけるラメリポディアの重要性は、この突起構造の枯渇が、細胞が複雑な生物学的微小環境6,10を移動する能力を妨げるとして明らかである。
細胞の移動におけるラメリポディアの調節を理解する大きな障害は、ラメリポアル突起動態、サイズ、および形状11、12、13、14における自然変動である。さらに、最近の研究では、ラメリポディアは、変動、周期的、および加速する突起14,15を含む複雑な突起行動を示すことが実証されている。細胞6,16を移動させる非常に可変的なラメリポディアと比較して、細胞の広がりの間に形成されるラメリポディアはより均一である12。細胞の広がりと移動の突起活性は、分岐アクチンネットワーク、収縮アクトミオシン束、およびインテグリンベースの細胞マトリックス接着を含む同一の高分子集合体によって駆動されるため、細胞の広がりは、ラメリポディアダイナミクスの調節を調べるためのモデルとして広く用いられてきた。
細胞拡散は、懸濁液中の細胞がまずインテグリン系付着17、19、20を介して基質に付着し、次いでアクチンベースの突起部21、22、23を伸ばして広がる動的メカノケミカルプロセスである。 広がり段階の間、細胞体から発するラメリポディアは、引き込みや失速をほとんどあるいは全く伴わない、等熱帯かつ持続的に突出する。最も一般的に使用される細胞拡散プロトコルはエンドポイントアッセイであり、広がる細胞はめっき19、24の後のさまざまな時間に固定される。これらのアッセイは、迅速かつ簡単ではあるが、ラメリポディアの動的特徴の変化を検出するために診断力に制限されている。ラメリポディアダイナミクスを制御する分子機構を決定するために、Sheetzグループは、生きた広がり細胞の定量分析の使用を開拓し、細胞エッジ突起11、12、22の多くの基本的特性を明らかにした。これらの研究は、生細胞拡散アッセイが細胞生物学研究所のツールボックスにおける堅牢で強力な技術であることを実証した。それにもかかわらず、ライブセル拡散アッセイのための詳細なプロトコルとオープンソース計算ツールは、現在、細胞生物学コミュニティでは利用できません。この作業を行うために、当社のプロトコルは、ライブ拡散セルのイメージング手順を概説し、自動画像解析ツールを提供します。この方法を検証するために、実験的な治療法としてArp2/3阻害を用い、Arp2/3複合体の機能を阻害しても細胞拡散は阻止されず、細胞突起速度の大幅な低下、細胞端突起の安定性が著しく低下し、細胞縁部がギザギザに生じることを示した。これらのデータは、ライブセルイメージングと自動画像解析の組み合わせが、細胞エッジダイナミクスを分析し、ラメリポディアを調節する分子成分を同定するのに有用なツールであることを示しています。
プロトコル
1. 細胞の播種
注:記載された細胞拡散プロトコルは、PH-Akt-GFP(PIP3/PI(3,4)P2の蛍光マーカー)を発現するマウス胚性線維芽細胞(MEF)を用いて行った。この細胞株は、CRISPR媒介遺伝子編集によるPH-Akt-GFP(Addgene #21218)の発現構築物をジェノミスティックに統合して生成した。しかしながら、ゲノム中で一過性または一体に発現される他の蛍光マーカーも、このアッセイに使用することができる。最適な画像セグメンテーションのために、細胞質に均一に分布する蛍光マーカー(例えば、細胞質GFP)を使用することを推奨します。
- 培養細胞の10cm皿を90%合流する。
- 細胞が適切な合流度を達成したら、22 mm x 22 mm のカバースリップ(#1.5; 0.17 mm 厚さ)を 35 mm の細胞培養皿に入れます。PBSで希釈したフィブロネクチン400μLでカバースリップを最終濃度2.5μg/mLにコーティングします。
注: アッセイに必要なカバーリップの数は、実験条件と技術レプリカの数によって決まります。 - フィブロネクチンコーティングカバースリップ付きの35mm皿を37°C、5%CO2インキュベーターに1時間置きます。
- インキュベーターからカバースリップで皿を取り除きます。フィブロネクチンを吸引し、カバースリップを2~3回軽くピペットでPBSで洗います。
- 細胞の10cm皿から細胞培養培地を吸引し、PBSで洗う。
- 0.05%のトリプシンEDTAの650 μLを細胞の90%コンフルエント皿に加え、皿を傾けて酵素を均等に分配します。トリプシンと一緒に皿をインキュベーターに1分間入れます。
- インキュベーターから細胞と皿を取り除きます。15 mL遠心分離チューブに10 mLの細胞培養培地を加えます。すぐにトリプシンをクエンチするために皿に別の10 mLのメディアを追加します。
- トリプシン化した細胞のピペット1mLを細胞を希釈するために15mL遠心分離管に入った。管の内容物を上下にピペットして、メディア内の細胞の均等な分布を保証します。凝集傾向の高い細胞タイプでは、細胞凝集の発生を最小限に抑えるために、細胞ストレーナー(100 μmメッシュサイズ)を通して細胞をフィルタリングすることが推奨されます。
- チューブから、ピペット500-1000μLの希釈された細胞をカバースリップを含む35mm皿に入れた。
- 皿を軽く振って細胞を均等に広げます。カバースリップが約10%の合流度(約50,000細胞/mL)であることを確認し、必要に応じて希釈された細胞の体積を調整します。
注: このような低い合流度で細胞を持つことの目的は、細胞の広がり獲得全体にわたって目的を集中させるために使用される各視野に1-2の偏光細胞があることを保証することです。 - 10cm皿の残りの細胞の1/5を処理条件ごとに1つの6cm皿に1個にする。通路を通した皿と35mmの皿を蓋付きでインキュベーターに一晩置きます。
注: これらは、ダイナミクスを広げる解析対象となるセルになります。
2. 薬物インキュベーションと細胞回復
- 2つの15 mL遠心分離管のそれぞれに5mLの細胞培養培地を加え、20 mLのフェノールレッドフリーDMEMを2つの50 mL遠心分離管のそれぞれに加えます。
注: チューブペアの数(15 mL + 50 mL)は実験条件の数に対応する必要があります。 - 細胞の広がりにArp2/3の重要性をテストするために、ピペットはArp2/3、CK-666の薬理学的阻害剤、またはDMSOなどの対照処理を、所望の濃度までの遠心管の各ペアに入れる。
- インキュベーターから継がれた6cmの皿(ステップ1.11を参照)を取り出し、メディアを吸引します。温かいPBSで皿を洗います。
- CK-666-またはDMSO補数15 mL遠心チューブの内容物を各継ぎ皿に加えます。各料理に適切な薬物処理のラベルを付け、皿をインキュベーターに1時間置きます。
- インキュベーターから食器を取り出し、メディアを吸引します。残りのフェノールレッドメディアをすべて完全に取り除くために、温かいPBSで皿を洗います。
- 0.05%のトリプシンEDTAを6cmの皿に230 μL加え、細胞を1分間インキュベートします。
注:該当する場合、トリプシンは非タンパク質分解細胞接着ブロッカーに置き換えることができます。 - インキュベーターから食器を取り除きます。各治療に対して、「チューブB」として指定された15 mL遠心分離管に、薬物補充フェノールレッドフリーDMEMを5 mL加えます。関連する皿に同じ培地の5 mLを追加して、トリプシンをクエンチします。「チューブA」と指定された15 mL遠心分離管に皿の内容物を移します。
- チューブAからチューブBに1mLの細胞を移す。
- チューブAとBをインキュベーターに45分間入れ、細胞がトリプシン化から回復できるようにします。遠心分離管のキャップを少し緩めてからインキュベーターに入れ、CO2 の浸透を可能にします。
注: 回復時間は、セルの種類によって異なる場合があります。我々の実験では、45分間の回復は細胞の生存率にごくわずかな影響を及ぼしたが、一部の細胞タイプは、あまりにも長い間懸濁液中で維持されるとアノイキスを受ける可能性がある。そのため、最適な回復時間を経験的に決定することをお勧めします。最適な回復時間により、サンプル中に死細胞やアポトーシス細胞を使い、高速かつ同期的な細胞拡散が可能になります。
3. 磁気チャンバの準備
- 22 mm x 22 mm の四角いカバースリップを収容できる 1 ウェル・チャムライド・セル磁気チャンバーのすべての部品が使用前にクリーニングされていることを確認してください。
- 35 mm 皿をカバースリップで取り外します(ステップ 1.11 を参照)。細胞培養培地を吸引し、カバースリップを温かいPBSで洗浄する。
- 鉗子のペアを使用して35 mm皿からカバースリップを取り出し、磁気チャンバーの底板にカバースリップをそっと置きます。
- カバースリップの上にシリコーンガスケットを置きます。
注:不適切に配置されたシリコンガスケットは、漏れやすい磁気チャンバの最も一般的な原因です。ガスケットが底板のインデントに置かれ、インデントを超えて上昇しないようにしてください。 - 本体を底板に取り付けます。
注:この部分は非常にゆっくりと行ってください。良いヒントは、本体を上に置きながら、片手で底板を押さえます。これにより、本体の磁石が底板を持ち上げないようにし、カバースリップを置き換えて割る可能性があります。 - 磁性チャンバに薬物補充フェノールレッドフリーDMEMの1 mLを加えます。糸くずのないティッシュを取り、漏れを確認するために本体と底板の間にエンクロージャを慎重にダブる。
注:漏れがある場合は、すぐにメディアを吸引し、ステップ3.4からもう一度進んでください。 - 透明カバーを本体に下げて、磁気チャンバを囲みます。
- 最初に実験室のティッシュに水を吹き付け、磁気チャンバーの底を拭きます(金属部分ではなくカバースリップ)。その後、少量の70%エタノールで第2の実験室組織をスプレーし、カバースリップを割れないように注意してください。
4. 画像取得
- ステージトップインキュベーターと目的ヒーターを37°Cに予熱し、ステージトップインキュベーターのCO2 レベルを5%に設定します。
注:ステージトップインキュベーターがCO2 供給に接続されていない場合、細胞培養培地は、一定のpH 7.4を維持するために25 mM HEPESで補充する必要があります。 - 十分な量の浸漬油を、あらかじめ温めた60X、1.4 N.A.油の目的に適用します。
注:私たちは、合理的に大きな視野と優れた光収集効率のために、このプロトコルで60X、1.4 N.A.油浸漬目的を使用しています。より大きな視野が必要な場合、画像の信号対雑音比が2.5を超える限り、より低い倍率の目的(例えば、20倍)を使用することができます。 - 完成した磁気チャンバとチューブB(ステップ2.9)の両方を共焦点顕微鏡に持って行きます。磁気チャンバーをステージトップインキュベーターに置きます。
注:浸漬油に泡を作らないように、ステージ上に静かに磁性チャンバーを置きます。 - GFPチャネルを使用して蛍光細胞に焦点を当てます。セルの端がシャープで、適切に定義されていることを確認します。
- チューブBから磁気チャンバに、磁気チャンバとピペット500μLの透明カバーを取り外します。透明カバーを磁気チャンバの上に戻します。
- 細胞拡散解析に最適な細胞を特定するには、カバースリップにまだ付着していないが、もはや転がり回っていない細胞の「ハロー」を検索します。カバースリップアタッチメントの初期段階にあるセルも優れた候補ですが、画像の取得は、拡散をキャプチャするために迅速に行う必要があります。
- 緑色のチャネルのタイムラプス画像取得を、6秒間隔で画像化された4つの視野を含むように設定します。
注: 異なる細胞タイプ間のラメリポディア突起速度のばらつきが高いため、最適なフレームレートは経験的に決定する必要があります。我々の実験で使用される6秒のイメージング間隔は、多くの間葉細胞および上皮細胞の分析の出発点として良い。しかし、非常に迅速に広がる細胞(例えば、免疫細胞)は、はるかに高いフレームレート(短いイメージング間隔)を必要とするかもしれません。セル拡散ムービーの最適なフレームレートにより、後続のフレーム間の突出したセルエッジの 2 ~ 5 ピクセルの変位が保証されます。細胞拡散の高原を識別するために使用されるカーブフィッティングの精度を考慮すると、最適なフレームレートはまた、細胞の広がりの急速な膨張段階の間に細胞エッジ変位の50-100測定を確実にする必要があります。視野数は、露光時間、取得点間の距離、およびステージの移動速度に応じて調整する必要があります。ユーザーは、目的のフレームレートで獲得できる視野の最大数を決定することをお勧めします。 - 適切な視野を特定した後、顕微鏡ステージのX座標とY座標を保存します。カバースリップ上で互いに比較的近い他の3つの視野を特定します。すべての所望の視野のための顕微鏡の段階の座標を保存しなさい。
注: 不要なサンプルの動きを最小限に抑えるために、視野間のステージ移動パスを最適化することを強くお勧めします。このような最適化は、手動または自動で実行できます。過度のサンプルの動きは取得速度を低下させ、細胞が降下するにともなって見えなくなることがあります。 - 6秒のフレームレートで15分間画像を取得し、ファイルを保存します。さらに取得が必要な場合は、ステップ 4.6 から繰り返します。
5. 細胞の広がり時の細胞面積、円形、突起ダイナミクスの解析
- データ処理と分析のための画像の準備
注:ソフトウェアは、.tiff形式の画像と入力パラメータとしてピクセルサイズが必要です。両方の要件は、取得ソフトウェアまたはフィジー(このプロトコルで)を使用して満たすことができます。これらの要件が満たされている場合は、ステップ 5.2 に進みます。- フィジー アプリケーション (https://imagej.net/Fiji/Downloads) の最新バージョンをインストールします。
- フィジーを使用してタイムラプス画像を開きます。
- [イメージの>プロパティ] を選択して、イメージのピクセル サイズ をコピーします。ピクセルサイズをコピーして、メモ帳/Wordにμmで貼り付けます。
- セルの広がり領域と円形の解析のために、タイムラプス画像をtiff画像スタックとして保存します。カスタム ビルド分析ソフトウェアは、独自のファイル形式をサポートしていません。[ファイル] > [TIFF] を選択して、個々のセル tiff イメージ スタック >保存します。
- データ処理および分析のために、Python IDE (スパイダー) と必要なパッケージ(PySimpleGUI および tifffile) をインストールします。
注: Python とパッケージのインストールは、初期設定でのみ必要です。- タイムラプスムービーは、カスタムビルドのPythonスクリプトを使用してSpyder IDEで分析されます。Spyder IDE をダウンロードするには、Spyder IDE とこの分析に必要なライブラリとパッケージのほとんどを含む Anaconda ディストリビューター (https://www.anaconda.com/products/individual) をダウンロードします。
- アナコンダをインストールし、アナコンダナビゲーターを介してスパイダーを起動します。
- IPython コンソール タブ (スパイダーの右下のセクションにあります) で、次のコマンドをコピーして貼り付けます: Pip インストール PySimpleGUI と Enter キーを押します。このコマンドを実行すると、グラフィカル・ユーザー・インターフェース (GUI) の開始に必要なパッケージがインストールされます。
- 同じコンソールで、次のコマンドをコピーして貼り付けます: pip は、tifffile をインストールし 、Enter キーを押します。このコマンドを実行すると、イメージを tiff ファイルとして保存するために必要なパッケージがインストールされます。
- 補助ファイルまたは GitHub から最新のスクリプトからすべての Python スクリプトをダウンロードする: https://github.com/ernestiu/Cell-spreading-analysis.git
- 細胞の広がり時の細胞面積と細胞形状因子の定量化
- Spyder の上部パネルで開いているファイルオプションを選択するか、ショートカット Ctrl + Oを使用して、メインの分析スクリプト 「cell_spreading_GUI.py」を開きます。
- 上部パネルで「ファイルを実行」を選択するか、ショートカット F5を使用して、セル拡散解析 GUI を開きます。
- [ セルの広がり範囲 ] タブ (図 3A)をクリックします。
- 参照ボタンを使用して、分析する tiff イメージを選択します。
注: 選択したファイルは tiff ファイルである必要があります。 - データ出力先のディレクトリ(セルマスク、値など)を指定します。
- データ出力設定を指定します。
- マスクを保存: セグメンテーションプロセス中に生成されたセルマスクを保存します。
- データのエクスポート: すべての分析データを含む Excel スプレッドシート (.xlsx) をエクスポート先のフォルダーにエクスポートします。
セル領域、すべての展開セルの円形度と縦横比は、コピー先のフォルダに Excel スプレッドシートとして保存されます。セル領域は次のように計算されます。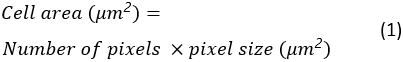
セルの円形度は、セルが完全に丸いセルにどれだけ近いかの尺度です。これは次のように計算されます。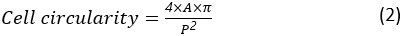
ここで、A と P はそれぞれセル領域とセルの周長です。セルの縦横比は、セルの伸長を表します。広がるセルは、1 に近いアスペクト比を持つ必要があります。アスペクト比は次のように計算されます。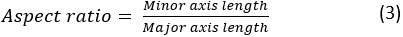
- コンターを保存: セル境界の輪郭オーバーレイ画像を保存先フォルダに保存します。
- セグメンテーション設定を指定する
- セグメンテーションの表示: 分析プロセス中に Spyder コンソールでセグメンテーション結果を表示します。
- 最小セル領域 (μm2):結合の開始段階のセルの面積値を含む、セル領域の最小値を入力します。この閾値より小さい領域を持つオブジェクトは、広がるセルとは見なされません。この数はセグメンテーションプロセスに影響します。
- イメージのパラメータを指定します。
- 取得間隔(秒単位):画像取得の頻度を秒単位で入力します。
- ピクセルサイズ(μm):解析用の画像を準備する際に記録されたピクセルサイズを入力します。
- イメージのビット深度: カメラ/検出器のビット深度を入力します。
- [ ファイルを実行 ]をクリックします。エラーが発生すると、Spyder のコンソールにエラー メッセージが表示されます。それ以外の場合は、イメージ分析プロセスがコンソールに表示されます。
注: コンソール/プロットセクションに表示される最初の画像(Spyder の設定に応じて)には、視野に示されているすべてのセルが表示されます。セルの周囲に配置された緑色のボックスは、セグメンテーションと分析に適した広がるセルを示します。灰色のボックスは、分析に適さないセルを示します。識別された広がりセルの総数は、[コンソール]タブにも表示されます。ソフトウェアは、セル領域(青)とセルの円形度(赤)を時間の関数としてプロットします。これらのグラフを使用すると、セルセグメンテーションの精度を評価できます。セグメンテーションが成功すると、細胞領域の単調に増加する曲線が得られます。セルの広がり領域の代表的な曲線を得るためには、ラグフェーズを手動でグラフから削除する必要があります。ラグフェーズには、細胞が広がる前のセル領域の測定値が含まれます。ラグフェーズは、セル領域プロット(図3C 右)に示されているように、速い変動によって示されます。
6. キモグラフを使用した細胞拡散時の細胞エッジダイナミクスの定量化
- 分析を実行する前に、細胞を広げる生のムービーをトリミングして、個々の広がるセルの時系列を作成します。
- フィジーのツールバーの [四角形 ] ツールを使用して、 単一のセルをカプセル化する対象領域 (ROI) を手動で選択します。(ROI が拡散セルを完全にカプセル化するようにするには、スクロール関数を使用して、すべての時点で ROI を検査します。
- ROI を右クリックし、[ 複製] を選択します。
- [重複スタック] をオンにし、[OK]をクリックします。
- Spyder のツール バーで [ファイルを開く ] ボタンを選択するか、ショートカット Ctrl + Oを使用して、メイン分析スクリプト "cell_spreading_GUI.py" を開きます。GUI がすでに開かれている場合は、ステップ 6.3 に直接進みます。
- 上部パネルで[ファイルを実行]を選択するか、ショートカットF5(図3B)を使用して、セル拡散解析GUIを開きます。
- [キモグラフ ジェネレータと解析] タブをクリックします。
- 参照ボタンを使用して、解析用の tiff イメージを選択します。
注: 独自のファイル形式、 例えば 、nd2、lif、zenは、スクリプトではサポートされていません。 - 出力データを保存する保存先フォルダ(セルマスクと値)を指定します。
- 出力設定を指定します。
- データのエクスポート: Excel スプレッドシート (.xlsx) を、相対セル エッジ位置と、キモグラフの取り消しイベントを含むエクスポート先のフォルダーにエクスポートします。
- イメージのパラメータを指定します。
- 取得間隔(秒):画像取得頻度を秒単位で入力します。
- ピクセルサイズ(μm):ステップ5.1.3で分析用の画像を準備する際に記録されたピクセルサイズを入力します。
- 最小セル領域 (μm2):結合の開始段階のセルの面積値を含む、セル領域の最小値を入力します。この閾値より小さい領域を持つオブジェクトは、広がるセルとは見なされません。この数はセグメンテーションプロセスに影響します。
- イメージのビット深度: カメラ/検出器のビット深度を入力します。
- [ ファイルを実行 ]をクリックします。エラーが発生すると、Spyder のコンソールにエラー メッセージが表示されます。それ以外の場合は、突起ダイナミクスの定量の概要がコンソールに表示されます。4組の引き込み周波数と突起速度測定があり、細胞の上下左右の部分から生成された4つのカイモグラフから抽出されます。引き込み頻度は次のように計算されます。
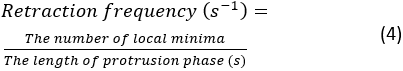
注:この数字は、ラメリ表彰台が拡散の過程で撤回する頻度を示しています。平均突起速度は、突起の始まりと、キモグラフ上のプラトー点との間の斜面によって測定されます。分割後にコンソールにサマリーカイモグラフの図が表示されます。サマリー図を保存するには、図を右クリックして画像を保存します。
結果
上記のプロトコルは、細胞拡散の生細胞イメージングのための実験手順と、細胞拡散ダイナミクスの定量的分析のための計算ツールを説明する。計算ツールは、低スループットまたはハイスループット形式で使用して、セルのリーディングエッジでアクチン重合機械を調節する分子プレーヤーを識別することができます。
実験手順の概略図を図 1に...
ディスカッション
記載された細胞拡散アッセイは、形態変化(例えば、細胞サイズおよび形状)および細胞エッジの動き(すなわち、突起速度および引き込み頻度)の連続的追跡を可能にする。これは、ほとんどの細胞拡散プロトコル19、24において欠けている特徴である。一般的に使用されるエンドポイント細胞拡散アッセイは細胞拡散速度の決定を可能に...
開示事項
著者らは開示するものは何もない。
謝辞
この研究は、コノート基金の新調査官賞(S.P.、カナダイノベーション財団、NSERCディスカバリー補助金プログラム)(RGPIN-2015-05114とRGPIN-2020-05881)、マンチェスター大学とトロント大学共同研究基金、トロント大学XSeedプログラムによって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin (0.05%), 0.53 mM EDTA | Wisent Bioproducts | 325-042-CL | |
| 10.0 cm Petri Dish, Polystyrene, TC Treated, Vented | Starstedt | 83.3902 | |
| 15 mL High Clarity PP Centrifuge Tube, Conical Bottom, with Dome Seal Screw Cap, Sterile | Falcon | 352097 | |
| 1-Well Chamlide CMS for 22 mm x 22 mm Coverslip | Quorum Technologies | CM-S22-1 | |
| 35 mm TC-treated Easy-Grip Style Cell Culture Dish | Falcon | 353001 | |
| 50 mL Centrifuge Tube, Transparent, Plug Seal | Nest | 602002 | |
| 6.0 cm Cell Culture Dishes Treated for Increased Cell Attachment, Sterile | VWR | 10861-658 | |
| Arp2/3 Complex Inhibitor I, CK-666 | Millipore Sigma | 182515 | |
| Camera, Prime 95B-25MM | Photometrics | ||
| Dimethyl Sulfoxide, Sterile | BioShop | DMS666 | |
| DMEM, 1x, 4.5 g/L Glucose, with L-Glutamine, Sodium Pyruvate and Phenol Red | Wisent Bioproducts | 319-005 CL | |
| DMEM/F-12, HEPES, No Phenol Red | Gibco | 11039021 | |
| D-PBS, 1X | Wisent Bioproducts | 311-425 CL | |
| Fetal Bovine Serum | Wisent Bioproducts | 080-110 | |
| Fiji Software | ImageJ | ||
| HEPES (1 M) | Gibco | 15630080 | |
| Human Plasma Fibronectin Purified Protein 1 mg | Millipore Sigma | FC010 | |
| Immersion Oil | Cargille | 16241 | |
| L-Glutamine Solution (200 mM) | Wisent Bioproducts | 609-065-EL | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140050 | |
| Micro Cover Glasses, Square, No. 11/2 22 x 22 mm | VWR | CA48366-227-1 | |
| Microscope Body, Eclipse Ti2-E | Nikon | ||
| Objective, CFI Plan Apo Lambda 60X Oil | Nikon | MRD01605 | |
| Penicillin-Streptomycin | Sigma | P4333 | |
| Spinning Disk, Crest Light V2 | CrestOptics | ||
| Spyder | Anaconda | ||
| Stage top incubator | Tokai Hit | ||
| Statistics Software, Prism | GraphPad | ||
| Tweezers, Style 2 | Electron Microscopy Sciences | 78326-42 |
参考文献
- Mullins, R. D., Heuser, J. A., Pollard, T. D. The interaction of Arp2/3 complex with actin: Nucleation, high affinity pointed end capping, and formation of branching networks of filaments. Proceedings of the National Academy of Sciences. 95 (11), 6181-6186 (1998).
- Yang, C., Czech, L., Gerboth, S., Kojima, S., Scita, G., Svitkina, T. Novel Roles of Formin mDia2 in Lamellipodia and Filopodia Formation in Motile Cells. PLoS Biology. 5 (11), 317 (2007).
- Mogilner, A., Oster, G. Cell motility driven by actin polymerization. Biophysical Journal. 71 (6), 3030-3045 (1996).
- Mogilner, A., Oster, G. Force Generation by Actin Polymerization II: The Elastic Ratchet and Tethered Filaments. Biophysical Journal. 84 (3), 1591-1605 (2003).
- Pollard, T. D., Borisy, G. G. Cellular Motility Driven by Assembly and Disassembly of Actin Filaments. Cell. 112 (4), 453-465 (2003).
- Wu, C., et al. Arp2/3 is critical for lamellipodia and response to extracellular matrix cues but is dispensable for chemotaxis. Cell. 148 (5), 973-987 (2012).
- Steffen, A., et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. Journal of cell science. 126, 4572-4588 (2013).
- Gupton, S. L., et al. Cell migration without a lamellipodium. The Journal of Cell Biology. 168 (4), 619-631 (2005).
- Dimchev, V., et al. Induced Arp2/3 Complex Depletion Increases FMNL2/3 Formin Expression and Filopodia Formation. Frontiers in Cell and Developmental Biology. 9, 634708 (2021).
- Leithner, A., et al. Diversified actin protrusions promote environmental exploration but are dispensable for locomotion of leukocytes. Nature cell biology. 18 (11), 1253-1259 (2016).
- Giannone, G., Dubin-Thaler, B. J., Döbereiner, H. -. G., Kieffer, N., Bresnick, A. R., Sheetz, M. P. Periodic Lamellipodial Contractions Correlate with Rearward Actin Waves. Cell. 116 (3), 431-443 (2004).
- Dubin-Thaler, B. J., et al. Quantification of Cell Edge Velocities and Traction Forces Reveals Distinct Motility Modules during Cell Spreading. PLoS ONE. 3 (11), 3735 (2008).
- Suraneni, P., Rubinstein, B., Unruh, J. R., Durnin, M., Hanein, D., Li, R. The Arp2/3 complex is required for lamellipodia extension and directional fibroblast cell migration. The Journal of cell biology. 197 (2), 239-251 (2012).
- Wang, C., et al. Deconvolution of subcellular protrusion heterogeneity and the underlying actin regulator dynamics from live cell imaging. Nature Communications. 9 (1), 1688 (2018).
- Dimchev, G., et al. Lamellipodin tunes cell migration by stabilizing protrusions and promoting adhesion formation. Journal of cell science. 133 (7), 239020 (2020).
- Burnette, D. T., et al. A role for actin arcs in the leading-edge advance of migrating cells. Nature cell biology. 13 (4), 371-381 (2011).
- Yamada, K. M., Kennedy, D. W. Dualistic nature of adhesive protein function: fibronectin and its biologically active peptide fragments can autoinhibit fibronectin function. The Journal of Cell Biology. 99 (1), 29-36 (1984).
- Cai, Y., et al. Nonmuscle Myosin IIA-Dependent Force Inhibits Cell Spreading and Drives F-Actin Flow. Biophysical Journal. 91 (10), 3907-3920 (2006).
- Humphries, M. J. Cell adhesion assays. Molecular Biotechnology. 18 (1), 57-61 (2001).
- Cavalcanti-Adam, E. A., Volberg, T., Micoulet, A., Kessler, H., Geiger, B., Spatz, J. P. Cell Spreading and Focal Adhesion Dynamics Are Regulated by Spacing of Integrin Ligands. Biophysical Journal. 92 (8), 2964-2974 (2007).
- Dubin-Thaler, B. J., Giannone, G., Döbereiner, H. -. G., Sheetz, M. P. Nanometer Analysis of Cell Spreading on Matrix-Coated Surfaces Reveals Two Distinct Cell States and STEPs. Biophysical Journal. 86 (3), 1794-1806 (2004).
- Gauthier, N. C., Fardin, M. A., Roca-Cusachs, P., Sheetz, M. P. Temporary increase in plasma membrane tension coordinates the activation of exocytosis and contraction during cell spreading. Proceedings of the National Academy of Sciences. 108 (35), 14467-14472 (2011).
- Wolfenson, H., Iskratsch, T., Sheetz, M. P. Early Events in Cell Spreading as a Model for Quantitative Analysis of Biomechanical Events. Biophysical Journal. 107 (11), 2508-2514 (2014).
- Guan, J. -. L., Berrier, A. L., LaFlamme, S. E. Cell Migration, Developmental Methods and Protocols. Methods in molecular biology. 294, 55-68 (2004).
- Raucher, D., et al. Phosphatidylinositol 4,5-Bisphosphate Functions as a Second Messenger that Regulates Cytoskeleton-Plasma Membrane Adhesion. Cell. 100 (2), 221-228 (2000).
- Machacek, M., Danuser, G. Morphodynamic Profiling of Protrusion Phenotypes. Biophysical Journal. 90 (4), 1439-1452 (2006).
- Zack, G. W., Rogers, W. E., Latt, S. A. Automatic measurement of sister chromatid exchange frequency. The journal of histochemistry and cytochemistry official journal of the Histochemistry Society. 25 (7), 741-753 (1977).
- Bardsley, W. G., Aplin, J. D. Kinetic analysis of cell spreading. I. Theory and modelling of curves. Journal of cell science. 61, 365-373 (1983).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved