Method Article
Sistema OnePot PURE Cell-Free
En este artículo
Resumen
Presentamos un método rápido y rentable para producir el sistema TX-TL recombinante PURE libre de células utilizando equipos de laboratorio estándar.
Resumen
El sistema definido de transcripción-traducción PURE (síntesis de proteínas utilizando elementos recombinantes) proporciona un chasis atractivo para la biología sintética libre de células. Desafortunadamente, los sistemas disponibles comercialmente son costosos y su capacidad de ajuste es limitada. En comparación, un enfoque casero se puede personalizar en función de las necesidades del usuario. Sin embargo, la preparación de sistemas caseros requiere mucho tiempo y es ardua debido a la necesidad de ribosomas, así como 36 purificaciones de proteínas a mediana escala. La racionalización de la purificación de proteínas mediante el coculturización y la copurificación permite minimizar los requisitos de tiempo y mano de obra. Aquí, presentamos un método fácil, ajustable, rentable y de tiempo para producir todos los componentes del sistema PURE en 1 semana, utilizando equipos de laboratorio estándar. Además, el rendimiento del OnePot PURE es comparable al de los sistemas disponibles comercialmente. El método de preparación OnePot PURE amplía la accesibilidad del sistema PURE a más laboratorios debido a su simplicidad y rentabilidad.
Introducción
Los sistemas de transcripción-traducción sin células (TX-TL) constituyen una plataforma prometedora para investigar e ingeniería de sistemas biológicos. Proporcionan condiciones de reacción simplificadas y sintonizables, ya que ya no dependen de procesos que sostienen la vida, incluido el crecimiento, la homeostasis o los mecanismos reguladores1. Por lo tanto, se anticipa que los sistemas libres de células contribuirán a la investigación de los sistemas biomoleculares, ofrecerán un marco para probar estrategias racionales de biodiseño2y proporcionarán un chasis para una futura célula sintética3,4. El sistema PURE totalmente recombinante ofrece un chasis especialmente atractivo debido a su composición definida y mínima, así como a su capacidad de ajuste y puesta a punto5.
Desde que se estableció el primer sistema PURE funcional y totalmente recombinante en 20015,se han realizado esfuerzos para ampliar los límites del sistema y optimizar la composición del sistema para mejorar los rendimientos del sistema6,7,8,permitir la regulación transcripcional9,la membrana10, 11 y la síntesis secretora de proteínas12,y facilitar el plegamientode proteínas 13,14 . Hoy en día, hay tres sistemas disponibles comercialmente: PUREfrex (GeneFrontier), PURExpress (NEB) y Magic PURE (Creative Biolabs). Sin embargo, esos sistemas son costosos, su composición exacta es propietaria y, por lo tanto, desconocida, y la adaptabilidad es limitada.
Los sistemas PURE preparados internamente demostraron ser la opción más rentable y sintonizable15,16. Sin embargo, los 37 pasos de purificación requeridos para las fracciones de proteínas y ribosomas consumen mucho tiempo y son tediosos. Se han hecho varios intentos para mejorar la eficiencia de la preparación del sistema PURE17,18,19. Recientemente demostramos que es posible cocultivar y copurpurar todas las proteínas no ribosómicas requeridas presentes en el sistema PURE. Este método OnePot ha demostrado ser rentable y eficiente en el tiempo, reduciendo el tiempo de preparación de varias semanas a 3 días hábiles. El enfoque genera un sistema PURE con una capacidad de producción de proteínas comparable al sistema PURExpress disponible comercialmente20. Contrariamente a los enfoques anteriores para simplificar la preparación PURE17,18,19, en el enfoque OnePot todas las proteínas todavía se expresan en cepas separadas. Esto permite al usuario ajustar la composición del sistema OnePot PURE simplemente omitiendo o agregando cepas específicas o ajustando los volúmenes de inoculación, generando así sistemas PURE de abandono o alterando las proporciones de proteínas finales, respectivamente.
El protocolo presentado aquí proporciona un método detallado para crear el sistema OnePot PURE como se describió anteriormente20,aunque β-mercaptoetanol fue reemplazado por tris(2-carboxietilo)fosfina (TCEP). Además, se describen dos métodos para la purificación de ribosomas: la purificación tradicional de ribosomas sin etiquetas utilizando interacción hidrofóbica y cojín de sacarosa, adaptado de Shimizu et al.15, y la purificación de ribosomas Ni-NTA basada en Wang et al.18 y Ederth et al.21 pero significativamente modificada. Este último método facilita aún más la preparación del sistema PURE y lo hace accesible a más laboratorios, ya que solo se requiere un equipo de laboratorio estándar.
El protocolo experimental resume la preparación de un versátil sistema TX-TL libre de células PURE para proporcionar una plataforma sin células simple, sintonizable y rentable, que se puede preparar utilizando equipos de laboratorio estándar en una semana. Además de introducir la composición PURE estándar, indicamos cómo y dónde se puede ajustar, con un enfoque principal en los pasos críticos en el protocolo para garantizar la funcionalidad del sistema.
Protocolo
NOTA: Este protocolo describe la preparación del sistema TX-TL libre de células a partir de componentes recombinantes. Para mayor comodidad, el trabajo se divide en cinco partes. La primera parte describe los pasos de preparación, que deben hacerse antes de comenzar el protocolo. La segunda parte describe la preparación de la solución de proteína OnePot. La tercera parte describe las purificaciones de ribosomas, la cuarta parte detalla la preparación de la solución energética y la última parte proporciona un manual para configurar una reacción PURA. Para mayor comodidad, los protocolos se dividen en días y se resumen en horarios diarios en la Tabla 1. Siguiendo el cronograma, todo el sistema puede ser preparado en 1 semana por una persona.
1. Trabajo preliminar
- Preparar los medios de cultivo bacteriano y los suplementos de medios como se describe en la Tabla suplementaria 1. Prepare y esterilice los materiales requeridos, incluidas las puntas de pipeta, 96 placas de pozo profundo.
- Preparación de la cepa
- Transformar las deformaciones de expresión indicadas en la Tabla 2 con los vectores de expresión correspondientes utilizando el método de choque térmico.
- Agregue plásmido purificado a las bacterias químicamente competentes e incube en hielo durante 20-30 min.
- Coloque la mezcla a 42 °C durante 30 s (choque térmico) y luego vuelva a colocarla sobre hielo durante 2 min.
- Pipete 20 μL de la bacteria directamente sobre placas de agar que contienen ampicilina (AMP) e incubar a 37 °C durante la noche. Guarde las placas a 4 °C durante un tiempo de hasta 1 semana.
- Inocular 3 ml de medios LB que contengan AMP con una sola colonia de bacterias de las placas de agar. Incubar a 37 °C mientras se agita a 260 rpm durante la noche.
- Mezclar 250 μL del cultivo con 250 μL de glicerol al 50% (v/v) y almacenar a -80 °C.
NOTA: Para una preparación más rápida en el futuro, almacene las cepas en una placa de 96 pozos como existencias de glicerol.
- Confirmar todas las transformaciones vectoriales por PCR de colonias y secuenciación. Secuenciar el gen, la región promotora y el sitio de unión al ribosoma.
- Transformar las deformaciones de expresión indicadas en la Tabla 2 con los vectores de expresión correspondientes utilizando el método de choque térmico.
- Prueba de expresión
- Inocular 300 μL de medios LB que contengan AMP con alrededor de 1 μL de las existencias de glicerol preparadas en una placa de pozo profundo de 1,3 mL. Selle la placa con una membrana transpirable y luego incube a 37 ° C mientras agita a 260 rpm durante la noche.
NOTA: Todas las expresiones se realizan por separado en este punto. - Inocular 300 μL de medios LB frescos que contengan AMP con 1 μL de los cultivos nocturnos. Incubar a 37 °C mientras se agita a 260 rpm durante la noche. Después de 2 h, inducir las células con 100 μM de isopropil β-D-1-tiogalactopyranoside (IPTG) y crecer durante 3 h adicionales.
- Mezclar 10 μL del cultivo con 10 μL de tampón 2x Laemmli y calentar a 95 °C durante 10 min. Gire las muestras durante 1 minuto con una centrífuga de mesa y cargue 10 μL del sobrenadante en un gel PAGE. Ejecute el gel en tampón Tris/Glycine/SDS a 200 V durante 30 min. Enjuáguelo bien con agua desionizada. Cubra el gel con una tinción de proteína Coomassie e incube durante 1 h. Desastinta el gel en agua si es necesario (resultados representativos para la prueba de expresión en la Figura 1).
NOTA: Use geles PAGE degradados (4%-15% o 4%-20%) para lograr una buena separación.
- Inocular 300 μL de medios LB que contengan AMP con alrededor de 1 μL de las existencias de glicerol preparadas en una placa de pozo profundo de 1,3 mL. Selle la placa con una membrana transpirable y luego incube a 37 ° C mientras agita a 260 rpm durante la noche.
- Restauración y limpieza de resina IMAC Sepharose
- Preparación de columnas.
- Mezclar bien la resina de Sepharose por vórtice.
- Pipetee la cantidad requerida de resina en una columna de flujo de gravedad vacía.
NOTA: La cantidad de resina requerida varía entre la purificación de His-ribosoma y la purificación de proteínas y se especifica en las secciones respectivas. - Lavar la resina con 30 ml de agua desionizada.
- Proceda con la recarga de columna como se especifica en la sección 1.4.4.
NOTA: Siempre deje pasar todo el líquido a través de la columna antes de continuar con el siguiente paso. Sin embargo, asegúrese de que la columna nunca se seque. Siempre que pase cualquier líquido a través de la columna, asegúrese de detener el flujo o continúe con el siguiente paso tan pronto como el líquido llegue a la resina.
- Restauración.
- Lavar la columna con 30 ml de agua desionizada.
- Aplicar 10 ml de una solución de EDTA de 0,2 M y NaCl de 0,5 M.
- Agregue 30 ml de una solución de NaCl de 0,5 M.
- Lavar la columna con 50 ml de agua desionizada.
- Almacenar en etanol al 20% (v/v) a 4 °C o continuar con el siguiente paso.
- Limpieza.
PRECAUCIÓN: Use equipo de protección.- Lavar la columna con 30 mL de 0,5 M de NaOH.
- Lavar la columna con 30 ml de agua desionizada.
- Lavar la columna con 30 mL de ácido acético de 0,1 M.
- Lavar la columna con 30 ml de agua desionizada.
- Lavar la columna con 30 ml de etanol al 70% (v/v).
- Lavar la columna con 50 ml de agua desionizada.
- Almacenar en etanol al 20% (v/v) a 4 °C o continuar con el siguiente paso.
- Recarga.
- Agregue 10 ml de solución de sulfato de níquel de 0,1 M a la columna.
PRECAUCIÓN: El sulfato de níquel es tóxico. Los residuos de sulfato de níquel deben desecharse con las precauciones indicadas por el proveedor. - Lavar la columna con 50 ml de agua desionizada.
- Conservar en etanol al 20% ((v/v)) a 4 °C o continuar con el equilibrio de la columna.
NOTA: Si la columna se almacena en etanol entre pasos, asegúrese de eliminar todos los rastros de etanol lavando la columna con agua.
- Agregue 10 ml de solución de sulfato de níquel de 0,1 M a la columna.
- Preparación de columnas.
2. Expresión y purificación de la solución de proteína OnePot
NOTA: El protocolo consta de tres partes divididas en días(Figura 2). Un procedimiento de preparación ideal produce 1,5 ml de solución de proteína OnePot de 13,5 mg/ml, que corresponde a más de mil reacciones PURAS de 10 μL. Sin embargo, la cantidad y la concentración ideal de la solución variarán de un lote a otro. Los usuarios experimentados pueden realizar múltiples preparaciones onePot PURE a la vez.
Día 1:
- Preparar medios de cultivo bacterianos y suplementos de medios como se describe en la Tabla suplementaria 1.
- Prepare y esterilice los materiales requeridos, incluidas las puntas de pipeta, dos placas de 96 pozos profundos y un matraz Erlenmeyer desconcertado de 1 L.
- Preparar tampones y suplementos como se describe en la Tabla suplementaria 2. El filtro esteriliza todos los tampones utilizando filtros de tapa de botella (0,45 μm) y guárdalos a 4 °C. Complemente todos los tampones con 1 mM TCEP justo antes de su uso, a menos que se indique lo contrario.
- Utilice 2 ml de resina de sefalrosa para la purificación de proteínas OnePot. Prepare la columna como se describe en la sección 1.4.
- Para preparar los cultivos iniciadores, combine 20 ml de medios LB con 20 μL de AMP. En una placa estéril de 96, 1.3 ml de pozo profundo, agregue 300 μL del medio en 35 pozos. Inocular cada uno de ellos con su respectiva cepa, excepto el factor de elongación termoestable (EF-Tu), y sellar la placa con una membrana transpirable.
NOTA: Inocule la placa usando un replicador de 96 pozos (ver Tabla de Materiales). El volumen del pozo de la placa del pozo profundo y el volumen del cultivo iniciador son esenciales. Los volúmenes de medios más grandes o los volúmenes de pozos más pequeños conducirán a una densidad bacteriana diferente debido a las inconsistencias de la esteación. - Para el cultivo EF-Tu, inocular 3 ml de medios LB en un tubo de cultivo de 14 ml con una tapa a presión. Un solo 3 ml de cultivo para EF-Tu es suficiente para un cultivo de expresión OnePot.
- Incubar a 37 °C mientras se agita a 260 rpm durante la noche.
Día 2:
NOTA: Realice todos los pasos a temperatura ambiente a menos que se indique lo contrario.
- Transfiera 500 ml de medios LB y 500 μL de AMP al matraz desconcertado estéril.
- Inocular el cultivo OnePot PURE con 1675 μL del cultivo EF-Tu y 55 μL de cada uno de los cultivos de la placa de pozo profundo (Tabla 2).
NOTA: Durante este paso, la composición general de la proteína se puede ajustar ajustando las proporciones de inoculación. Asegúrese de que el volumen total de inoculación permanezca constante a 3,6 ml.
OPCIONAL: Para confirmar que todas las cepas han crecido durante la noche, mida la densidad óptica de los cultivos nocturnos a 600 nM (OD600)en una placa de 96 pozos utilizando un lector de placas. Utilice una dilución de 10x para la medición de la densidad óptica. - Incubar el cultivo durante 2 h a 37 °C con una agitación de 260 rpm, o hasta que el OD600 del cultivo alcance 0.2-0.3.
- Inducir el cultivo con 500 μL de 0,1 mM IPTG y crecer durante 3 h adicionales.
- Cosechar las células por centrifugación a 4 °C y 3220 x g durante 10 min y almacenar el pellet celular a -80 °C hasta su uso posterior.
NOTA: Para optimizar el tiempo, prepare la solución energética descrita en la sección 4 durante las horas de incubación del día 2(Tabla 1).
Día 3:
- Mida las cantidades de tampones necesarios para la purificación descrita en los pasos a continuación y agregue TCEP a todos ellos como se indica en la Tabla suplementaria 2. Almacene los tampones restantes sin TCEP a 4 °C para futuras purificaciones.
- Equilibrar la columna cargada (sección 2.4) con 30 ml de tampón A. Después de que hayan pasado 25 ml de búfer A, cierre la columna desde la parte inferior. En paralelo, continúe con los pasos 2.15-2.17.
- Descongele las células y use una pipeta serológica para resuspend el pellet celular en 7.5 ml de tampón A.
- Lise las celdas utilizando un sonicador de sonda de 130 vatios (consulte la Tabla de materiales,diámetro de la punta de la sonda: 6 mm) con los siguientes parámetros: pulso de encendido de 4 x 20 s, pulso de apagado de 20 s, amplitud del 70%. Si la sonicación tiene éxito, la solución se oscurecerá (Figura 2).
NOTA: Asegúrese de mantener las células en hielo durante la sonicación. Coloque la sonda lo suficientemente profunda en la solución sin tocar el tubo. Si se genera una gran cantidad de espuma, la transferencia de energía se amortiguará. En ese caso, deje que la espuma se asiente, baje la sonda más profundamente en la solución y extienda el tiempo de sonicación. - Retire los restos celulares por centrifugación a 21130 x g durante 20 min a 4 °C inmediatamente después de la sonicación. Mantenga el lisate en hielo.
- Agregue el sobrenadante a la columna equilibrada. Cierre la columna desde la parte superior y asegúrese de que no haya fugas. Incubar la columna durante 3 h a 4 °C en rotación utilizando un rotador de tubo.
- Elute los componentes no unidos de la columna y lávelos con 25 ml de tampón A.
- Lavar la columna con 25 mL de tampón de imidazol de 25 mM (23,95 mL de tampón A y 1,25 mL de tampón B).
- Elute las proteínas con 5 mL de tampón de imidazol de 450 mM (0,5 mL de tampón A y 4,5 mL de tampón B). Mantenga las proteínas eluidas en hielo en todo momento.
- Diluya el eluido con 25 ml de tampón HT, mantenga la mezcla en hielo. Añadir 15 mL a un filtro centrífugo de 15 mL y concentrar a un volumen de 1,5 mL. Agregue los 15 ml restantes al filtro con la solución concentrada y concéntrese a 1,5 ml una vez más.
- Añadir 10 ml de tampón HT a la muestra concentrada y concentrar a 1 ml. Añadir una cantidad igual de tampón B y conservar a -80 °C hasta su posterior uso.
NOTA: Una ronda de intercambio/concentración toma aproximadamente 60 minutos de giro a 3220 x g a 4 °C. - Durante el intercambio de búfer, restaure la columna como se especifica en la sección 1.4.
Día 4:
- Mida la concentración de proteína utilizando el ensayo de Bradford descrito por el proveedor. Concentrar la muestra con un filtro centrífugo de corte de 0,5 mL a 20 mg/mL.
NOTA: Diluya la solución de proteína 25 veces o 50 veces antes de las mediciones de concentración para evitar sobresaturar el ensayo de Bradford. - Para establecer la concentración ideal de proteínas, realice una prueba de expresión en esta etapa (sección 5.2) con diferentes concentraciones de la solución proteica. Para realizar la valoración, mantenga constante el volumen total de la solución y pipetee la solución de proteína OnePot, incluido el tampón B de stock, en cinco proporciones diferentes(Tabla suplementaria 7).
- Verifique la composición de la proteína OnePot PURE usando SDS-PAGE (Figura 3A). Diluir 2,5 μL de la muestra con 7,5 μL de agua, mezclar con 10 μL de tampón 2x Laemmli y luego cargar 5 μL y 2,5 μL de las muestras en el gel. Ejecute SDS-PAGE como se especifica en la sección 1.3.3.
- Alícuota la solución proteica en alícuotas de 50 μL después de verificar la expresión y ajustar la concentración. Guarde la solución de proteína OnePot PURE a -80 °C hasta su uso posterior.
NOTA: Si se sospecha que un componente proteico no está presente o está presente en una concentración inferior a la esperada en el OnePot PURE, realice los siguientes pasos. - Compruebe si el cultivo nocturno de la cepa respectiva ha crecido a un ritmo comparable al de los otros cultivos mediante la realización de mediciones de densidad óptica (OD600)de todos los cultivos.
- Realice una prueba de expresión adicional de la cepa específica para verificar la expresión de la proteína sospechosa.
3. Solución de ribosoma
NOTA: Se introducen dos estrategias diferentes de purificación de ribosomas, una para los ribosomas marcados con hexahistidina y otra para los ribosomas no marcados. La principal ventaja del método de purificación que utiliza His-purification en una columna de flujo de gravedad Ni-NTA de afinidad estándar es que la purificación es fácil, rápida y no requiere equipo de laboratorio adicional, como un sistema FPLC y una ultracentrífuga. Sin embargo, la capacidad de producción de proteínas en las reacciones OnePot PURE es de alrededor de un tercio en comparación con los ribosomas sin etiquetas. Por lo tanto, elija el método para la producción de ribosomas en función de si un alto rendimiento es importante para la aplicación dada.
-
Purificación de ribosomas marcada por His
NOTA: Este protocolo utiliza la cepa E. coli RB1, un regalo del profesor Wang (Universidad de Columbia, EE.UU.)18. Esta cepa tiene una inserción genómica de una etiqueta de hexahistidina en el extremo C de la proteína ribosómica 50S (L7 / L12), lo que permite la purificación utilizando una columna de flujo de gravedad Ni-NTA. El rendimiento habitual es de alrededor de 0,5 ml de ribosomas de 3,45 μM, que es suficiente para más de quinientas reacciones PURAS de 10 μL.
Día 1:
- Preparar medios de cultivo bacterianos y suplementos de medios como se describe en la Tabla suplementaria 1.
- Prepare y esterilice los materiales necesarios, incluidas las puntas de pipeta, un matraz Erlenmeyer de 5 L y un matraz Erlenmeyer de 100 ml.
- Preparar tampones y suplementos como se describe en la Tabla suplementaria 2. El filtro esteriliza todos los tampones utilizando filtros de tapa de botella (0,45 μm) y guárdalos a 4 °C.
Día 2:
- Pipetear 5 ml de resina a una columna y preparar la columna como se especifica en el punto 1.4.
NOTA: Debido al mayor volumen de la resina, la restauración y purificación toman mucho más tiempo. Use una columna diferente para la purificación del ribosoma para evitar la contaminación cruzada y límpielo a fondo antes de la purificación. - Preparar un cultivo nocturno de la cepa rb1 de E. coli inoculando 35 ml de medios LB en un matraz Erlenmeyer de 100 ml. Incubar a 37 °C mientras se agita a 260 rpm.
Día 3:
NOTA: Realice todos los pasos a temperatura ambiente a menos que se indique lo contrario.
- Agregue 2 L de medios LB en un matraz estéril de 5 L, inocule con 12 ml del cultivo nocturno y luego incube durante 3-4 h a 37 ° C mientras agita a 260 rpm.
NOTA: Alternativamente, realice el cultivo bacteriano en 4 x 500 ml de cultivos en matraces desconcertados de 1 L. - Pellet las células por centrifugación durante 10 min a 3220 x g y 4 °C. Conservar a -80 °C hasta su uso posterior.
Día 4:
- Equilibrar la columna preparada en la etapa 3.1.4. con 30 ml de tampón de lisis.
- Resuspend el pellet en 20 ml de tampón de lisis utilizando una pipeta serológica.
- Lise las celdas con un sonicador de sonda de 130 vatios (consulte la Tabla de materiales,diámetro de la punta de la sonda: 6 mm) sobre hielo con los siguientes parámetros: pulso de 11 x 20 s encendido; Pulso de 20 s apagado, 70% de amplitud (consulte el paso 2.16 para obtener detalles del procedimiento).
- Inmediatamente después de la sonicación, retire los restos celulares por centrifugación durante 20 min a 21130 x g a 4 °C. Mantenga el lisate en hielo.
- Cargue el sobrenadante a las columnas y déjelo pasar.
- Lave la columna con las siguientes mezclas de tampones de lisis y elución.
- Lavar 0: utilizar 30 ml de tampón de lisis.
- Lavado 1: use 30 mL de imidazol de 5 mM (29 mL de tampón de lisis, 1 mL de tampón de elución).
- Lavado 2: use 60 ml de imidazol de 25 mM (50 ml de tampón de lisis, 10 ml de tampón de elución).
- Lavado 3: use 30 ml de imidazol de 40 ml de imidazol (22 ml de tampón de lisis, 8 ml de tampón de elución).
- Lavado 4: use 30 ml de imidazol de 60 ml (18 ml de tampón de lisis, 12 ml de tampón de elución).
- Elute los ribosomas con 7,5 mL del tampón de elución. Mantenga las proteínas eluidas en hielo en todo momento.
- Añadir 22 μL de β-mercaptoetanol puro a 45 ml de tampón de ribosoma.
PRECAUCIÓN: β-mercaptoetanol es tóxico. Tome precauciones de seguridad y trabaje en una campana extractora de humos. - Añadir el eluyuga a un filtro centrífugo de 15 mL y concentrar a 1 mL.
- Añadir 15 ml de tampón de ribosoma a la muestra concentrada y concentrar de nuevo a 1 ml.
NOTA: Repita el paso anterior dos veces. - Conservar a -80 °C hasta su uso posterior.
NOTA: Una ronda de intercambio/concentración toma aproximadamente 60 min de centrifugación a 3220 x g a 4 °C. - Durante el intercambio de búfer, restaure la columna como se especifica en la sección 1.4.
Día 5:
- Determinar la concentración de ribosomas midiendo la absorbancia a 260 nM de una muestra diluida 1:100 en tampón de ribosomas. Un valor de absorbancia de 10 de la solución diluida corresponde a 23 μM de solución sin diluir como se describió anteriormente16.
- Implementar una concentración final de stock de 3,45 μM. Para ajustar la concentración, diluya los ribosomas con tampón de ribosomas o concéntrelos aún más por centrifugación a 14000 x g en un filtro centrífugo de 3 kDa 0,5 ml a 4 °C.
NOTA: Para lograr una expresión óptima del sistema, realice una titulación de la concentración de ribosomas (sección 5.2, Tabla suplementaria 7). - Verifique la composición del ribosoma utilizando SDS-PAGE (Figura 3A) como se especifica en la sección 1.3.3. Diluya 2,5 μL de la muestra con 7,5 μL de agua, mezcle con 10 μL de 2x tampón Laemmli y luego cargue 5 μL y 2,5 μL de las muestras en el gel.
-
Purificación de ribosomas sin etiquetas
NOTA: La purificación de ribosomas sin etiquetas se realiza utilizando un sistema FPLC(Tabla de Materiales)y se basa en cromatografía de interacción hidrofóbica utilizando columnas de butilo de 2 x 5 ml(Tabla de Materiales). Aunque los ribosomas pueden purificarse de cualquier cepa, el uso de la cepa E. coli A19 (E. coli Genetic Resources en Yale CGSC) es ventajoso debido a su deleción de la RNasa I22. Realice la purificación a 4 °C en una cámara frigorífica o en un gabinete de enfriamiento. El rendimiento habitual es de alrededor de 0,5 mL de ribosomas de 10 μM, lo que corresponde a más de quinientas reacciones PURAS de 10 μL.
Día 1:
- Preparar medios de cultivo bacterianos y suplementos de medios como se describe en la Tabla suplementaria 1.
- Prepare y esterilice los materiales requeridos, incluidas las puntas de pipeta, el matraz Erlenmeyer de 5 L y el matraz Erlenmeyer de 100 ml.
- Preparar tampones y suplementos como se describe en la Tabla suplementaria 2. El filtro esteriliza todos los tampones utilizando filtros de tapa de botella (0,45 μm) y guárdalos a 4 °C.
Día 2:
- Para preparar un cultivo nocturno de la cepa E. coli A19, inocular 35 ml de lb medios en un matraz Erlenmeyer de 100 ml. Incubar a 37 °C mientras se agita a 260 rpm.
Día 3:
- Transfiera 2 L de medios LB al matraz desconcertado estéril de 5 L, inocule con 30 ml del cultivo nocturno y luego incube durante 3-4 h a 37 ° C mientras agita a 200 rpm.
- Pellet de las células por centrifugación a 4000 x g durante 15 min a 4 °C. Vuelva a utilizar el pellet en 25 ml de tampón de suspensión y guárdelo a -80 °C hasta su uso posterior.
Día 4:
- Realice los pasos 3.2.8-3.2.12 en paralelo con los pasos 3.2.13-3.2.19.
- Descongelar y lisar las células utilizando un sonicador de sonda de 130 vatios (ver Tabla de materiales y diámetro de la punta de la sonda: 6 mm) sobre hielo con los siguientes parámetros: pulso de 12 x 20 s encendido; Pulso de apagado de 20 s, amplitud del 70% (consulte los detalles del procedimiento del paso 2.16).
- Retire inmediatamente los restos celulares por centrifugación a 20000 x g durante 20 min a 4 °C.
- Aspira el sobrenadante y mide el volumen. Agregue un volumen igual de tampón de suspensión (alto contenido de sal) para ajustar la concentración final de sulfato de amonio a 1,5 M y mezcle bien.
- Retirar el precipitado por centrifugación a 20000 x g durante 20 min a 4 °C.
- Filtre el sobrenadante utilizando un filtro de jeringa de membrana de polietersulfona de 0,45 μm antes de la purificación de FPLC y recoja el filtrado en una botella de vidrio de 100 ml. Mantenga el sobrenadante a 4 °C en todo momento.
- Configure el sistema FPLC para la purificación de cromatografía de interacción hidrofóbica utilizando una columna doble de butilo (2 x 5 ml) de la siguiente manera. Para esta configuración, un volumen de columna (CV) se refiere a un volumen de 10 ml.
- Se necesitarán tres entradas: dos como líneas de amortiguación y una como línea de muestra. Debido a la configuración predeterminada del purificador, es conveniente elegir las líneas A1 y B1 para el búfer C y el búfer D, respectivamente, y la línea A2 como línea de muestra. Aplique un caudal predeterminado de 4 ml/min, excepto para lavados con bomba (10 ml/min) o a menos que se indique lo contrario.
NOTA: Como TCEP es un reactivo costoso, agregue la cantidad correspondiente a los búferes C y D solo después de la etapa de equilibrio. - Realice un lavado de la bomba del sistema en etanol al 20% ((v / v)) para limpiar el sistema y eliminar la contaminación potencial de las purificaciones anteriores. Ajuste manualmente un caudal de 0,2 ml/min y monte la columna. Detenga el flujo.
- Ejecute un sistema de lavado de bomba con agua. Lavar la columna con 3 CV de agua.
- Equilibrio: coloque las entradas A1 y A2 en el tampón C y la entrada B1 en el tampón D sin TCEP. Ejecute un lavado de bomba y equilibre la columna con 4 CV de tampón C.
- Agregue TCEP a los búferes C y D.
- Prepare tubos de 15 ml o tubos colectores de fracción redonda transparentes al colector de fracciones para recolectar fracciones de elución de 4-5 ml.
- Carga: Coloque la entrada A2 en la botella con la muestra filtrada. Cargue aproximadamente el 90% del volumen de la muestra en la columna. Diluya la muestra con 20 ml de tampón C que contenga TCEP y cargue 10 ml de la muestra en la columna. Repita el paso de dilución al menos dos veces y cargue la mayor cantidad de muestra posible en la columna. Es fundamental asegurarse de que no se absorba aire en la máquina.
- Paso de lavado 1: lavar con 3 CV de tampón C para eliminar los componentes no unidos.
- Paso de lavado 2: lavar con 5 CV de tampón C al 80% y búfer D al 20%.
- Elución: eluir el producto aplicando el 50% del tampón C y el 50% del tampón D, con un volumen total de elución de 5 CV. Recoger esta fracción en los tubos colectores.
- Paso de lavado 3: Elute todos los contaminantes que interactúan fuertemente utilizando el tampón D 100% con un volumen total de 5 CV.
- Analizar el espectro de absorción de la fracción muestral a 260 o 280 nM (Figura 4). El primer pico muestra las proteínas no absorbidas eluidas durante la carga y el primer paso de lavado; el segundo pico muestra contaminantes que han sido eluidos durante el segundo paso de lavado. El tercer pico monitorea el producto final, y el último pico muestra los contaminantes que interactúan fuertemente. Agrupar todas las fracciones de muestra correspondientes al tercer pico para su posterior procesamiento. Mantenga las proteínas eluidas en hielo en todo momento.
- Superponga suavemente la fracción recuperada sobre 15 ml del tampón del cojín en cuatro tubos de ultracentrifugación de policarbonato. Agregue un máximo de 15 ml de la muestra a 15 ml del tampón de cojín. Asegúrese de equilibrar bien el peso del tubo. Pellet de los ribosomas por ultracentrifugación a 100000 x g a 4 °C durante 16 h.
NOTA: Asegúrese de que no haya grietas en los tubos de ultracentrifugación. - Limpie y restablezca la columna de la siguiente manera. Un caudal de 5 mL/min funciona bien. Coloque todas las entradas en el agua y ejecute un lavado con bomba. Lavar la columna con 2 CV de agua.
- Coloque la entrada en una solución de NaOH de 0,5 M, realice un lavado con bomba y, posteriormente, lave la columna con 3 CV de NaOH.
- Coloque la entrada en agua, realice un lavado con bomba y luego lave la columna en 2 CV de agua.
- Coloque la entrada a una solución de ácido acético de 0,1 M, realice un lavado con bomba y, posteriormente, lave la columna con 3 CV de solución de ácido acético.
- Lave con bomba y lave la columna con 2 CV de agua.
- Coloque todas las entradas en etanol al 20% ((v/v)), ejecute un paso de lavado de la bomba y almacene la columna en etanol al 20% ((v/v)) lavándola con 3 CV de una solución de etanol al 20% ((v/v)).
NOTA: Asegúrese de que el sistema nunca se seque ni absorba aire. Nunca aplique tampón directamente al etanol, o etanol al tampón. Siempre agregue un paso intermedio de lavado con agua, ya que de lo contrario existe el riesgo de que los precipitados obstruyan la columna. Asegúrese de agregar suficientes tubos de recolección de muestras.
Día 5:
- Deseche el sobrenadante y con cuidado, sin molestar el gránulo translúcido, lave cada gránulo con 0,5 ml de tampón de ribosoma helado. Repita este paso dos veces.
- Resusped cada uno de los gránulos transparentes en 100 μL de tampón ribosómico sobre hielo utilizando una barra de agitación magnética (3 mM de diámetro, 10 mM de longitud) en un agitador magnético utilizando la velocidad más baja posible. Recoja los ribosomas resuspendidos y lave los tubos con 50 μL adicionales de tampón de ribosomas.
NOTA: El pellet translúcido es difícil de ver. Por lo tanto, lave cuidadosamente el pellet desde los lados del tubo. - Determine la concentración de ribosomas midiendo la absorbancia a 260 nM de la muestra diluida en una proporción de 1:100 en tampón de ribosomas. Una absorbancia de 10 de la solución diluida corresponde a 23 μM de solución sin diluir como se describió anteriormente16.
- Implementar una concentración final de stock de 10 μM. Para ajustar la concentración, diluya los ribosomas con tampón de ribosomas o concéntrelos aún más mediante centrifugación a 14000 x g en un filtro centrífugo de 3 kDa a 4 °C.
NOTA: Para lograr una expresión óptima del sistema, realice la titulación del ribosoma (sección 5.2, Tabla suplementaria 7). - Verifique la composición del ribosoma con SDS-PAGE (Figura 3A) como se especifica en la sección 1.3.3. Diluya 2,5 μL de la muestra con 7,5 μL de agua, mezcle con 10 μL de 2x tampón Laemmli y luego cargue 5 μL y 2,5 μL de las muestras en el gel.
4. Solución energética
NOTA: La composición de la solución de energía 2.5x presentada aquí es un ejemplo de una solución que funcionó bien para una reacción TX-TL estándar. Para optimizar el tiempo, prepare la solución energética durante el día 2. La preparación de la solución de aminoácidos se explica en detalle, seguida del procedimiento de preparación final.
- Solución de aminoácidos
NOTA: Prepare la solución de aminoácidos a granel. Preparar la cantidad de soluciones de almacenamiento de aminoácidos requeridas para un volumen final de al menos 2000 μL reducirá el error de pesaje para las cantidades que de otro modo serían muy pequeñas. La concentración total de la solución de aminoácidos está limitada por la solubilidad de los aminoácidos y las respectivas concentraciones de solución stock. Para el sistema PURE estándar, prepare una solución con una concentración final de 3,25 mM. Utilice la tabla de cálculo de la solución de aminoácidos (Tabla suplementaria 3) como plantilla. Use cisteína en forma de sal para garantizar una solubilidad suficiente. Evite el uso de métodos de preparación de aminoácidos a base de KOH. Es posible pesar directamente las cantidades exactas de aminoácidos en la solución final de aminoácidos sin preparar la solución de stock para todos los aminoácidos. Sin embargo, esto es más desafiante y menos preciso.- Preparar soluciones de stock para cada aminoácido como se describe en la Tabla suplementaria 3,excepto para la tirosina.
NOTA: Debido a las diferentes solubilidades de los aminoácidos en agua, las respectivas concentraciones sugeridas de la solución stock difieren. - La masa mínima [mg] proporciona la masa mínima aproximada requerida para obtener una cantidad suficiente de solución madre para el volumen total objetivo, como referencia.
NOTA: La masa mínima se calcula con un excedente del 10%. - Para una preparación más fácil de las soluciones, no pese la cantidad exacta de aminoácido, sino que, para la masa en cuestión, ajuste la cantidad de agua para lograr la concentración deseada. Calcule la cantidad de agua desionizada (Agua para agregar [μL]) necesaria, basada en la masa real llenada (celdas de color amarillo claro) y la concentración deseada utilizando la hoja de cálculo en la Tabla suplementaria 3.
- Solubiliza las soluciones de aminoácidos por vórtice hasta que todo el precipitado se haya disuelto. Las soluciones individuales de aminoácidos se pueden almacenar a -20 °C durante varias semanas.
NOTA: Algunos aminoácidos son difíciles de disolver en agua; el proceso puede llevar algún tiempo. - Pesar la cantidad exacta de tirosina requerida para obtener una concentración final de 3,25 mM directamente en el tubo para la solución de aminoácidos.
NOTA: La tirosina es muy difícil de disolver en agua. Agréguelo directamente en lugar de preparar una solución de stock. - Agregue las cantidades correspondientes de soluciones madre de aminoácidos y agua como se indica en el volumen final para agregar la columna [μL] (células de color azul claro) y vórtice bien la solución. Guarde la solución de aminoácidos completa a -80 °C hasta su uso posterior.
- Preparar soluciones de stock para cada aminoácido como se describe en la Tabla suplementaria 3,excepto para la tirosina.
- Preparación de la solución energética
NOTA: En total, la solución energética 2.5x contiene 0.75 mM de cada aminoácido, 29.5 mM de acetato de magnesio, 250 mM de glutamato de potasio, 5 mM de ATP y GTP cada uno, 2.5 mM de CTP, UTP y TCEP, respectivamente, 8.75 mg / ml de ARNt de E. coli MRE 600, 50 mM de fosfato de creatina, 0.05 mM de ácido folínico, 5 mM de espermidina, y 125 mM de HEPES. Los usuarios primerizos preparan la solución energética en pequeños lotes de 200 μL. Almacene las soluciones individuales preparadas de acuerdo con la Tabla suplementaria 4 a -20 °C o -80 °C para su uso posterior.- Descongele todas las soluciones acuosas mencionadas en la Tabla suplementaria 5 sobre hielo.
- Mientras tanto, prepare las soluciones de stock para los componentes restantes enumerados en la Tabla suplementaria 4. Mantenga todas las soluciones en hielo después de la preparación.
NOTA: Agregue 500 μL de RNasa y agua libre de DNasa directamente al vial para disolver los ARNt liofilizados. Mezclar bien mediante vórtice suave; limitar el pipeteo para evitar la introducción de RNasas. - Añadir los volúmenes calculados (Tabla suplementaria 5) de soluciones de stock y agua y mezclar bien utilizando un vórtice. Mantenga la solución en hielo en todo momento.
- Mida el pH de la solución mediante pipeteo de 1 μL en una tira de pH, para asegurarse de que el pH de la solución sea neutro.
- Alícuota la solución energética a 50-100 μL por tubo sobre hielo y guárdala a -80 °C hasta su uso posterior. Durante la alícuota, vórtice el stock principal con frecuencia para evitar que los componentes se precipiten.
NOTA: Opcionalmente, realice un ensayo de actividad de la solución energética recién fabricada frente a soluciones energéticas comerciales, por ejemplo, la Solución A en PURExpress. Si se observa un rendimiento significativamente menor del sistema con la solución energética, la optimización de las concentraciones de iones, especialmente los iones de magnesio, por titulación (5-20 mM) puede ser ventajosa.
5. Reacción OnePot PURE
- Plantilla de ADN
NOTA: Las proteínas codificadas aguas abajo del promotor T7 se pueden expresar en PURE a partir de ADN lineal o circular. Al generar una plantilla de ADN lineal utilizando la extensión PCR, se pueden omitir los tediosos pasos de clonación. Las plantillas lineales para este estudio fueron generadas por PCR como se describe a continuación, utilizando una ADN polimerasa de alta fidelidad (Tabla de Materiales). Las secuencias de imprimación, las temperaturas de fusión y los ajustes del termociclador utilizados en este estudio se especifican en la Tabla suplementaria 6. La preparación de la plantilla de ADN no está incluida en el horario diario.- Configure una reacción de PCR según lo recomendado por el proveedor de polimerasa.
NOTA: Los parámetros optimizados para una ADN polimerasa de alta fidelidad(Tabla de Materiales)se dan en la Tabla Suplementaria 6. - Amplificar el gen objetivo (por ejemplo, eGFP) como una plantilla lineal de un plásmido o genoma utilizando cebadores específicos del gen (500 nM) (para los parámetros, véase la Tabla suplementaria 6).
- La amplificación genera extensiones cortas para proporcionar secuencias de recocido para los siguientes pasos de PCR de extensión.
- Revise el amplicón en un gel de agarosa para obtener el tamaño y la pureza correctos.
- Utilice el ADN amplificado como plantilla para los pasos de extensión posteriores. Configure una reacción de al menos 50 μL.
- Ejecute 10 ciclos de amplificación por PCR con los cebadores de extensión (2,5 nM). Después de completar los ciclos de amplificación, agregue inmediatamente los cebadores finales (500 nM) a la misma reacción y ejecute 30 ciclos para amplificar el producto de PCR extendido. Encuentre las temperaturas de fusión y las secuencias de imprimación en la Tabla suplementaria 6.
- Purifique los fragmentos de ADN utilizando un kit de purificación de ADN y eluye el ADN en agua libre de nucleasas en lugar de EDTA que contiene tampón de elución.
- Verifique la plantilla lineal en un gel de agarosa para obtener el tamaño y la pureza correctos.
- Mida la concentración de ADN en ng/μL utilizando un espectrofotómetro UV-Vis.
- Configure una reacción de PCR según lo recomendado por el proveedor de polimerasa.
- Configuración de la reacción PURE
NOTA: La composición final de la reacción es 1x solución de energía, ribosomas sin etiquetas o ribosomas His-tag, proteínas OnePot PURE y plantilla de ADN. La relación de volumen de reacción comprende una solución de energía del 40%, una solución de proteína y ribosoma del 30%, y un 30% de ADN y agua. Los volúmenes de reacción típicos varían entre 5 μL y 25 μL. Cuantificar la expresión de una proteína fluorescente continuamente en un lector de placas. Usa un Lys verde in vitro Translation Labeling System, que incorpora residuos de lisina etiquetados fluorescentemente en proteínas recién sintetizadas, para verificar la expresión de proteínas no fluorescentes en un gel SDS-PAGE. Una plantilla de reacción de ejemplo se da en Cuadro suplementario 7 para ayudar a establecer una reacción de expresión libre de células PURAS. Las celdas en amarillo indican los valores de entrada del usuario, y las celdas en naranja indican reactivos adicionales que se agregarán opcionalmente a la reacción. Mantenga las relaciones de volumen de los componentes precisas para garantizar el equilibrio iónico correcto. Por ejemplo, para lograr una mayor concentración de proteínas, aumente la concentración de solución de proteína OnePot; sin embargo, no aumente el volumen de solución de proteína agregada a la reacción.- Rellene la concentración [ng/μL] y la longitud [pares de bases] del ADN en las celdas amarillas correspondientes en la hoja de cálculo. Use 2-10 nM de ADN para la reacción.
- Rellene el volumen de reacción total deseado en μL.
- Retire los reactivos necesarios del congelador y descongelarlos en hielo.
NOTA: La recongelación de los componentes es posible sin una disminución de la funcionalidad. Sin embargo, minimice el número de ciclos de congelación-descongelación y el tiempo que las muestras se almacenan en hielo tanto como sea posible. - Pipetear las cantidades calculadas de agua, ADN y solución de energía a un lado del tubo de PCR o a una esquina de un pozo en la placa de 384 pozos. Agregue la cantidad requerida de cualquier reactivo adicional en el mismo lado. Minimice el número de muestras por experimento para evitar la evaporación de la muestra y el sesgo de la hora de inicio experimental.
NOTA: Es crucial mantener el componente energético físicamente separado de los componentes proteicos para evitar el consumo prematuro de las fuentes de energía y menores rendimientos. - Pipetear las cantidades calculadas de proteína y solución de ribosoma al otro lado de un tubo de PCR o a la esquina opuesta de la placa de 384 pozos.
NOTA: Utilice mezclas maestras siempre que sea posible para reducir el impacto de los errores de pipeteo. Después de las pruebas iniciales, las soluciones de ribosoma y proteína se pueden mezclar y almacenar como una sola solución. - Girar durante un corto tiempo (30 s) para fusionar los componentes de reacción. Para evitar la evaporación durante los experimentos de lector de placas, agregue 35 μL de cera líquida y selle la placa con un sellador transparente (consulte la Tabla de materiales).
- Incubar durante un mínimo de 3 h a 37 °C.
- Para la lectura en un lector de placas, mida la intensidad de fluorescencia en la longitud de onda requerida cada 2 minutos (los resultados representativos se muestran en la Figura 3B).
- Realice los pasos siguientes para las muestras etiquetadas con Green Lys.
- Después de la expresión libre de células, incubar la muestra con 0,16 μg/μL de RNasa A durante 30 min a 37 °C para eliminar el fondo fluorescente del kit de etiquetado Green Lys.
NOTA: Utilice RNasa A, ya que otros tipos de RNasas no eliminan el fondo lo suficientemente bien. - Visualice la expresión de proteínas ejecutando SDS-PAGE como se especifica en la sección 1.3.3. Lave el gel sin teñir suavemente en agua desionizada y visualívelo en un imager fluorescente utilizando una longitud de onda de excitación de 488 nm.
- Posteriormente, manche el gel utilizando métodos convencionales de tinción de Coomassie. Para conocer los parámetros adecuados, véase la sección 1.3.3.
NOTA: Realice una titulación de la solución proteica con la concentración recomendada de ribosoma y, si es necesario, titule los ribosomas con la concentración óptima de proteína OnePot después. Utilice el kit comercial PURExpress ΔRibosome como control positivo. La solución A, factor mix y la solución de ribosoma corresponden a la energía preparada, la solución de proteína OnePot y los ribosomas purificados, respectivamente.
Resultados
El protocolo anterior está diseñado para facilitar el establecimiento del sistema TX-TL libre de células PURE en cualquier laboratorio. El protocolo incluye una descripción detallada de la preparación de las tres partes distintas del sistema PURE: la proteína OnePot, el ribosoma y la solución energética. En la Tabla 1se muestra una programación diaria detallada, que optimiza el flujo de trabajo. El flujo de trabajo está optimizado para la purificación de ribosomas marcados con His, y los marcos de tiempo pueden diferir ligeramente si se realiza la purificación de ribosomas sin etiquetas. Una preparación proporciona una cantidad suficiente de PURE para un mínimo de quinientas reacciones de 10 μL. Además, las soluciones preparadas son estables durante más de un año a -80 °C y pueden soportar múltiples ciclos de congelación-descongelación.
Los niveles adecuados de sobreexpresión para todas las cepas son cruciales para la funcionalidad de la solución proteica final. La Figura 1 muestra una sobreexpresión exitosa en las 36 cepas individuales utilizadas posteriormente para la preparación de proteínas OnePot. La variación en las intensidades de banda de las proteínas sobreres expresadas ocurrió muy probablemente debido a un sesgo en los volúmenes de carga en el gel SDS-PAGE. Los tamaños de proteína esperados se resumen en la Tabla 2. GlyRS y PheRS consisten en dos subunidades de varios pesos moleculares; las 34 proteínas restantes consisten en una sola subunidad. La clave de la simplicidad y la eficacia del tiempo de este protocolo es el paso de coculturización y co-purificación (Figura 2). La solución de proteína OnePot se preparó aumentando la proporción de cepa EF-Tu con respecto a todas las demás cepas de expresión. La composición global de las proteínas finales fue analizada por SDS-PAGE (Figura 3A). A partir de los geles (carriles 2, 3), se nota que EF-Tu (43,3 kDa) está presente en una concentración más alta en comparación con las otras proteínas, como se esperaba. Si bien el gel proporciona una buena primera indicación de las proporciones de expresión de proteínas, es difícil determinar si y en qué nivel se expresó cada proteína individual. Por lo tanto, es muy recomendable confirmar la sobreexpresión en cada cepa antes de coculturar, como se muestra arriba.
El ribosoma de E. coli es una máquina molecular compleja compuesta por más de 50 subunidades de proteínas individuales23. En la Figura 4se muestra un espectro de absorción representativo a 260 nm para la purificación de ribosomas sin etiquetas; el tercer pico es característico de la elución exitosa del ribosoma. Para ambos métodos de purificación de ribosomas, se observó el patrón de funcionamiento esperado en el gel SDS-PAGE (Figura 3A)18. Observamos contaminaciones para ambas purificaciones, aunque en pequeñas cantidades (<10%). En particular, diferentes contaminantes estaban presentes en los ribosomas sin etiquetas (carriles 5, 6) e hisetiquetados (carriles 11, 12) debido a la variación en el método. Para referencia del usuario, también se incluyen los geles SDS-PAGE para los sistemas combinados (carriles 8, 9 y 14, 15).
Por último, se compara el rendimiento de los sistemas preparados (Figura 3) utilizando las diferentes variantes de ribosomas. Los cursos de tiempo de expresión in vitro de eGFP muestran que ambos sistemas PURE son funcionales y producen eGFP fluorescente. Sin embargo, la solución de proteína OnePot combinada con los ribosomas marcados con His, utilizando la concentración de ribosomas optimizada por titulación, produjo solo un tercio del nivel de expresión de la versión de ribosoma no etiquetada(Figura 3B). Se observaron resultados similares cuando se expresaron y etiquetaron tres proteínas de diferentes tamaños utilizando el sistema de etiquetado in vitro Green Lys tRNA(Figura 3C). Como se ve en el gel fluorescente, los productos de longitud completa se expresaron con éxito en ambos sistemas; sin embargo, solo alrededor de la mitad del nivel de expresión se logró con el sistema de ribosoma His-tag. Además del etiquetado de fluorescencia, las bandas esperadas para las tres proteínas se distinguen en un gel teñido de Coomassie(Figura 3D). Los resultados muestran que el sistema de expresión introducido, que se puede preparar en una semana en un laboratorio con equipo estándar, se puede utilizar para la expresión in vitro de proteínas codificadas aguas abajo del promotor T7 a partir de plantillas lineales.

Figura 1: Resultados representativos para la prueba de sobreexpresión para todas las cepas de expresión del sistema PURE. Los números y tamaños de proteínas PURAS se resumen en la Tabla 2. Los números de proteína 21, 24 y 27 están marcados con una estrella para una mejor visualización. Haga clic aquí para ver una versión más grande de esta figura.
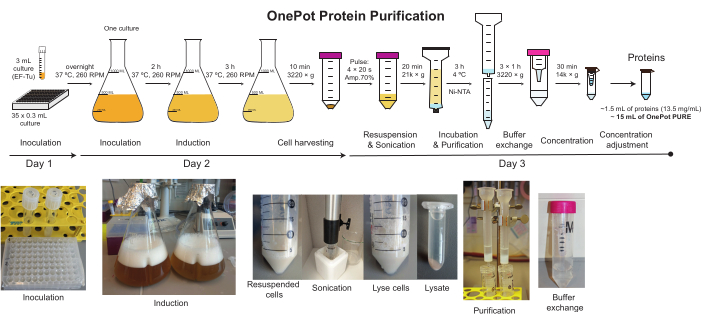
Figura 2: Purificación de proteínas OnePot. La representación esquemática y las fotografías correspondientes de todos los pasos involucrados en la producción de la solución de proteína OnePot. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Rendimiento de los sistemas preparados utilizando las diferentes variantes de ribosomas. (A) Geles SDS-PAGE teñidos de azul coomassie de la solución proteica OnePot (carriles 2, 3), ribosomas sin etiquetas sin solución proteica (carriles 5, 6) y con solución proteica (carriles 8, 9), ribosomas his-etiquetados sin solución proteica (carriles 11, 12) y con solución proteica (carriles 14, 15). Se cargaron dos concentraciones diferentes por muestra. (B) Comparación de la expresión de eGFP de ribosomas marcados con His y ribosomas sin etiquetas. La intensidad de fluorescencia de la expresión in vitro de eGFP se controla a lo largo del tiempo para una reacción PURA utilizando ribosomas sin etiquetas (1,8 μM, azul) y ribosomas marcados con His (0,62 μM, rojo). Las concentraciones de la plantilla lineal y la solución de proteína OnePot fueron de 4 nM y 2 mg/ml, respectivamente. Los paneles (C) y (D) muestran el gel SDS-PAGE de proteínas sintetizadas en OnePot con tag-free (1.8 μM, azul, carriles 3, 4, 5) y ribosomas His-tag (0.62 μM, rojo, carriles 6, 7, 8) etiquetados con un kit de etiquetado in vitro GreenLys (C) y teñidos con azul Coomassie (D), respectivamente. Las flechas negras indican las bandas esperadas de proteínas sintetizadas: eGFP (26,9 kDa), ArgRS (64,7 kDa), T7 RNAP (98,9 kDa). Las concentraciones de la plantilla lineal y de la solución de proteína OnePot fueron de 4 nM y 1,6 mg/ml, respectivamente. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Espectros de absorbancia a 260 nm. Resultados representativos de espectros de absorbancia a 260 nm durante la purificación por interacción hidrofóbica de ribosomas sin etiquetas. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Un calendario diario optimizado para la preparación de todas las soluciones OnePot PURE. Haga clic aquí para descargar esta tabla.
Tabla 2: Lista de proteínas PURAS Haga clic aquí para descargar esta Tabla.
Tabla suplementaria 1: Reactivos. La tabla enumera concentraciones, volúmenes y otros detalles específicos de los reactivos y componentes utilizados durante este estudio. Haga clic aquí para descargar esta tabla.
Cuadro complementario 2: Colchones. La hoja de cálculo enumera las composiciones tampón exactas para las purificaciones de proteínas, ribosomas sin etiquetas y ribosomas His-tag, así como las concentraciones de las soluciones de stock utilizadas para su preparación. Además, calcula las cantidades requeridas de componentes en función del volumen del búfer. Haga clic aquí para descargar esta tabla.
Tabla complementaria 3: Cálculos de aminoácidos. La hoja de cálculo enumera los aminoácidos y sus concentraciones de solución de stock recomendadas requeridas para la solución energética. Calcula la cantidad de agua que se agregará a cada aminoácido en función de la masa pesada real, y también calcula el volumen de la solución de aminoácidos que se agregará a la mezcla final de aminoácidos. Haga clic aquí para descargar esta tabla.
Tabla complementaria 4: Soluciones de stock para la solución energética. La tabla enumera las concentraciones y volúmenes de soluciones de stock necesarias para la solución energética e indica más detalles, incluidas las condiciones de almacenamiento. Haga clic aquí para descargar esta tabla.
Cuadro complementario 5: Solución energética. La tabla enumera los componentes de la solución energética y sus concentraciones recomendadas. Además, calcula sus volúmenes requeridos para ser agregados a la solución final en función de sus concentraciones de solución de stock y el volumen de la solución de energía. Haga clic aquí para descargar esta tabla.
Cuadro complementario 6: PCR. La tabla enumera las secuencias y concentraciones de los cebadores utilizados para la extensión PCR e indica las temperaturas de fusión y los pasos del termociclador optimizados para una ADN polimerasa de alta fidelidad. Haga clic aquí para descargar esta tabla.
Tabla complementaria 7: Reacción PURA. La hoja de cálculo muestra un ejemplo de configuración de una reacción PURE. Enumera las concentraciones y volúmenes utilizados de los componentes para una reacción PURA utilizando ribosomas sin etiquetas o ribosomas His-tag. Además, calcula las proporciones de volumen para las valoraciones de proteínas y ribosomas. Haga clic aquí para descargar esta tabla.
Discusión
El protocolo presentado aquí describe un método simple, efectivo en tiempo y costo para preparar un sistema de expresión PURE versátil20 basado en la composición estándar15. Al utilizar el protocolo junto con los horarios diarios suministrados(Tabla 1),todos los componentes se pueden preparar en 1 semana y producen cantidades suficientes para reacciones PURE de hasta quinientas 10 μL. Dado que las proteínas utilizadas en este protocolo están sobreexpresadas a partir de plásmidos de copia alta y tienen baja toxicidad para E. coli,se observan buenos niveles de expresión para todas las proteínas requeridas(Figura 1). Esto permite el fácil ajuste de las cepas, y por lo tanto también la composición de proteínas en los cocultivos, simplemente modificando las proporciones de las cepas de inoculación20. Además de las proteínas ribosómicas, la concentración de EF-Tu demostró ser de fundamental importancia para los rendimientos de expresión6. Por el contrario, los cambios en la concentración de los otros componentes de la proteína tuvieron un impacto relativamente bajo en la robustez del sistema PURE7,24. Por lo tanto, ajustando la relación de inoculación de EF-Tu con respecto a todos los demás componentes, se puede lograr una composición comparable a la composición PURE estándar, y se puede lograr un sistema PURE con un rendimiento similar20. Al preparar la solución proteica, es crucial asegurarse de que todas las cepas crezcan bien y sobreexpresen la proteína codificada después de la inducción(Figura 1).
La función de ribosoma es clave para el rendimiento general del sistema PURE24. En este protocolo, se demuestran dos métodos diferentes para preparar la solución de ribosoma, es decir, la purificación de ribosomas sin etiquetas y con etiqueta His. La purificación de ribosomas sin etiquetas se basa en cromatografía de interacción hidrofóbica seguida de centrifugación con un cojín de sacarosa, que requiere acceso a un sistema de purificación FPLC y una ultracentrífuga15. Por el contrario, el método que utiliza ribosomas marcados con His18 y la purificación por cromatografía de afinidad de flujo por gravedad no requiere equipo especializado y se puede realizar en la mayoría de los laboratorios. Este último método, por lo tanto, aporta ventajas como la simplicidad y la accesibilidad. Sin embargo, observamos un rendimiento de síntesis significativamente menor cuando se utilizaron los ribosomas marcados con His en el OnePot PURE en comparación con la variante sin etiquetas (Figura 3). Según el tipo de aplicación, este menor rendimiento puede ser aceptable.
La solución energética proporciona los componentes de bajo peso molecular y los ARNt necesarios para alimentar las reacciones TX-TL in vitro. Este protocolo proporciona una receta para una solución energética típica, que se puede ajustar fácilmente en función de las necesidades del usuario. Junto con el ARNt, el NTP y el fosfato de creatina, la abundancia y concentración de iones Mg2+ han sido cruciales para el rendimiento general del sistema PURE8,ya que son cofactores críticos para la transcripción y traducción. En algunos casos, la titulación de iones puede, por lo tanto, mejorar en gran medida el rendimiento general de PURE. La integridad del ADN es crucial para el rendimiento de PURE. Por lo tanto, la secuencia verifica la región promotora, el sitio de unión al ribosoma y el gen objetivo y garantiza que una concentración adecuada de ADN (<2 nM) ayudará a solucionar los problemas que puedan surgir al configurar una reacción PURE.
El sistema PURE es un sistema TX-TL mínimo y, por lo tanto, aplicaciones específicas pueden requerir ajustes adicionales25. Estos pueden incluir la incorporación de diferentes ARN polimerasas9,26,chaperonas13y factores proteicos como EF-P o ArfA8. Aunque las cepas de expresión para estas proteínas se pueden incluir en los cocultivos, agregarlas por separado al sistema preparado puede proporcionar un mejor control de los niveles de proteínas requeridos. Además, la inclusión de vesículas es esencial para la producción de proteínas de membrana10,11. La oxidación en lugar de reducir los ambientes y una isomerasa de enlace disulfuro facilitan la formación adecuada de enlaces disulfuro, que son, por ejemplo, necesarios para las proteínas secretoras12.
Es esencial asegurarse de que los componentes adicionales no interfieran con la reacción. Los factores más importantes a los que debe prestar atención al configurar una reacción o agregar otros componentes se enumeran a continuación. Asegúrese de que no se utilicen tampones incompatibles ni se alteren las concentraciones de iones. Evite las soluciones que contengan glicerol, altas concentraciones de potasio, magnesio, iones de calcio, osmolatos, pirofosfato, antibióticos o EDTA, tanto como sea posible. Por ejemplo, reemplazar un tampón de elución con agua durante la purificación del ADN puede ser beneficioso, ya que el EDTA es un aditivo común en este tampón. Suministrar a las soluciones moléculas adicionales cargadas negativamente como NTP o dNTP requiere ajustar la concentración de magnesio8,ya que las moléculas cargadas negativamente se comportan como agentes quelantes y se unen a moléculas cargadas positivamente. Un pH neutro es ideal para la reacción. En consecuencia, todos los componentes deben amortiguarse al pH correspondiente; esto es especialmente importante para moléculas altamente ácidas o básicas como los NTP. Por último, la temperatura y el volumen son parámetros clave para la reacción. Para lograr un buen rendimiento, se debe implementar una temperatura de alrededor de 37 ° C, ya que las temperaturas por debajo de 34 ° C reducirán significativamente el rendimiento27.
Es relevante tener en cuenta que antes de preparar el OnePot PURE, se debe considerar la aplicación de destino y los requisitos asociados, como el volumen, la pureza, la facilidad de modificación y la inclusión u omisión de componentes. Para muchas aplicaciones, el sistema será una excelente opción, pero otras pueden requerir rendimientos, capacidad de ajuste y otros factores, que el sistema OnePot no puede proporcionar. Independientemente, el protocolo introducido será beneficioso para la preparación de cualquier sistema casero, ya que aquí se resumen todos los pasos críticos para dicha preparación.
Una de las principales ventajas del sistema OnePot es su compatibilidad con el sistema PURExpress disponible comercialmente, que ofrece la posibilidad de probar la funcionalidad y la integridad de todos los componentes por separado mediante la sustitución secuencial de cada componente PURExpress por su equivalente OnePot. Las ventajas del sistema OnePot PURE, como la capacidad de ajuste y la preparación fácil, rápida y rentable, harán que tx-TL sin células sea accesible a más laboratorios en todo el mundo y contribuirán a expandir la implementación de esta poderosa plataforma en biología sintética libre de células.
Divulgaciones
Los autores declaran que no tienen intereses financieros contrapuestos.
Agradecimientos
Este trabajo fue apoyado por el Consejo Europeo de Investigación en el marco de la Subvención del Programa de Investigación e Innovación Horizonte 2020 de la Unión Europea 723106, una Subvención de la Fundación Nacional de Ciencias de Suiza (182019) y EPFL.
Materiales
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS buffer | Bio-Rad Laboratories | 1610732 | |
| 15 mL centrifuge tubes | VWR International | 525-0309 | |
| 384-well Black Assay Plates | Corning | 3544 | |
| 4-20% Mini-PROTEANRTM TGXTM Precast Protein Gels | Bio-Rad Laboratories | 4561096 | |
| 50 mL centrifuge tubes | VWR International | 525-0304 | |
| 96-Well Polypropylene DeepWell plate | Nunc | 260252 | |
| Acetic acid, 99.8 % | Acros | 222140010 | |
| Äkta purifier | GE Healthcare | purification of tag free ribosomes | |
| AMICON ULTRA 0.5 mL - 3 KDa | Merck Millipore | UFC500324 | |
| AMICON ULTRA 15 mL - 3 KDa | Merck Millipore | UFC900324 | |
| Amino acids | Sigma-Aldrich | LAA21-1KT | |
| Ammonium chloride | Sigma-Aldrich | 09718-250G | |
| Ammonium sulfate | Sigma-Aldrich | A4418 | |
| Ampicillin | Condalab | 6801 | |
| BenchMark Fluorescent Protein Standard | ThermoFisher | LC5928 | |
| Breathe-Easy sealing membrane | Diversified Biotech | Z380059-1PAK | |
| Centrifuge tubes polycarbonate | Beckman | 355631 | purification of tag free ribosomes |
| Chill-out Liquid Wax | Bio-Rad Laboratories | CHO1411 | |
| Creatine phosphate | Sigma-Aldrich | 27920 | |
| DNA Clean & Concentrator-25 (Capped) | Zymo | ZYM-D4034-200TS | |
| DTT | SantaCruz Biotech | sc-29089B | |
| Econo-Pac Chromatography Columns | Bio-Rad Laboratories | 7321010 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | 03609-250G | |
| Eppendorf Protein LoBind microcentrifuge tubes | VWR International / Eppendorf | 525-0133 | |
| Falcon 14 mL Round Bottom Polystyrene Test Tube, with Snap Cap | Falcon | 352051 | |
| Flasks, baffled 1000 mL 4 baffles, borosilicate glass | Scilabware | 9141173 | |
| FluoroTect Green Lys in vitro Translation Labeling System | Promega | L5001 | optional |
| Folinic acid | Sigma-Aldrich | PHR1541 | |
| Glycerol | Sigma-Aldrich | G7757-1L | |
| HEPES | Gibco | 15630-056 | |
| HiTrap Butyl HP Column | GE Healthcare | 28411005 | purification of tag free ribosomes |
| IMAC Sepharose 6 Fast Flow | GE Healthcare | 17-0921-07 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| InstantBlue | Expedeon | ISB1L-1L | |
| IPTG (Isopropyl-beta-D-thiogalactoside) | Alfa Aesar | B21149.03 | |
| Laemmli buffer (2x), sample buffer | Sigma-Aldrich | S3401-1VL | |
| Lysogeny broth (LB) media | AppliChem | A0954 | |
| Magnesium acetate | Sigma-Aldrich | M0631 | |
| Magnesium chloride | Honeywell Fluka | 63020-1L | |
| Nickel Sulfate | Alfa Aesar | 15414469 | |
| NTP | ThermoFisher | R0481 | |
| Phusion High-Fidelity DNA Polymerase (2 U/µL) | ThermoFisher | F530S | |
| Potassium chloride | Sigma-Aldrich | P5405-1KG | |
| Potassium glutamate | Sigma-Aldrich | 49601 | |
| PURExpress In Vitro Protein Synthesis Kit | NEB | E6800S | |
| PURExpress Δ Ribosome Kit | NEB | E3313S | |
| Quick Start Bradford 1x Dye Reagent | Bio-Rad Laboratories | 5000205 | |
| Rapid-Flow Sterile Single Use Vacuum Filter Units | ThermoFisher | 564-0020 | |
| RNaseA solution | Promega | A7973 | |
| SealPlate film | Excel Scientific | Z369659-100EA | |
| Sodium hydroxide | Sigma-Aldrich | 6203 | |
| Spermidine | Sigma-Aldrich | S2626 | |
| Sucrose | Sigma-Aldrich | 84097 | |
| TCEP (Tris(2-carboxyethyl)phosphin -hydrochlorid) | Sigma-Aldrich | 646547-10X1mL | |
| Thickwall Polycarbonate Tube | Beckman | 355631 | |
| Trichloroacetic acid | Sigma-Aldrich | T0699 | |
| Tris base | ThermoFisher | BP152-500 | |
| tRNA | Roche | 10109541001 | |
| Ultracentrifuge Optima L-80 | Beckman | purification of tag free ribosomes | |
| Whatman GD/X syringe filters | GE Whatman | WHA68722504 | |
| β-mercaptoethanol | Sigma-Aldrich | M6250-100mL |
Referencias
- Laohakunakorn, N., et al. Bottom-up construction of complex biomolecular systems with cell-free synthetic biology. Frontiers in Bioengineering and Biotechnology. 8, 213 (2020).
- Laohakunakorn, N. Cell-free systems: a proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788 (2020).
- Cho, E., Lu, Y. Compartmentalizing cell-free systems: toward creating life-like artificial cells and beyond. ACS Synthetic Biology. 9 (11), 2881-2901 (2020).
- Gaut, N. J., Adamala, K. P. Reconstituting natural cell elements in synthetic cells. Advanced Biology (Weinh). 5 (3), 2000188 (2021).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nature Biotechnology. 19 (8), 751-755 (2001).
- Li, J., Gu, L., Aach, J., Church, G. M. Improved cell-free RNA and protein synthesis system. PLoS One. 9 (9), 106232 (2014).
- Kazuta, Y., Matsuura, T., Ichihashi, N., Yomo, T. Synthesis of milligram quantities of proteins using a reconstituted in vitro protein synthesis system. Journal of Bioscience and Bioengineering. 118 (5), 554-557 (2014).
- Li, J., et al. Dissecting limiting factors of the protein synthesis using recombinant elements (PURE) system. Translation. 5 (1), 1327006 (2017).
- de Maddalena, L. L., et al. GreA and GreB enhance expression of Escherichia coli RNA polymerase promoters in a reconstituted transcription-translation system. ACS Synthethic Biology. 5 (9), 929-935 (2016).
- Kuruma, Y., Ueda, T. The PURE system for the cell-free synthesis of membrane proteins. Nature Protocols. 10 (9), 1328-1344 (2015).
- Jacobs, M. L., Boyd, M. A., Kamat, N. P. Diblock copolymers enhance folding of a mechanosensitive membrane protein during cell-free expression. Proceedings of the National Academy of Sciences of the Unites States of America. 116 (10), 4031-4036 (2019).
- Shimizu, Y., Kanamori, T., Ueda, T. Protein synthesis by pure translation systems. Methods. 36 (3), 299-304 (2005).
- Niwa, T., Kanamori, T., Ueda, T., Taguchi, H. Global analysis of chaperone effects using a reconstituted cell-free translation system. Proceedings of the National Academy of Sciences of the Unites States of America. 109 (23), 8937-8942 (2012).
- Niwa, T., et al. Large-scale analysis of macromolecular crowding effects on protein aggregation using a reconstituted cell-free translation system. Frontiers in Microbiology. 6, 1113 (2015).
- Shimizu, Y., Ueda, T. PURE technology. Methods in molecular biology. 607, 11-21 (2010).
- Horiya, S., Bailey, J. K., Krauss, I. J. Directed evolution of glycopeptides using mRNA display. Methods in Enzymology. 597, 83-141 (2017).
- Villarreal, F., et al. Synthetic microbial consortia enable rapid assembly of pure translation machinery. Nature Chemical Biology. 14 (1), 29-35 (2018).
- Wang, H. H., et al. Multiplexed in vivo His-tagging of enzyme pathways for in vitro single-pot multienzyme catalysis. ACS Synthetic Biology. 1 (2), 43-52 (2012).
- Shepherd, T., et al. De Novo Design and synthesis of a 30-cistron translation-factor module. Nucleic Acids Research. 45 (18), 10895-10905 (2017).
- Lavickova, B., Maerkl, S. J. A Simple, robust, and low-cost method to produce the PURE cell-free system. ACS Synthetic Biology. 8 (2), 455-462 (2019).
- Ederth, J., Mandava, C. S., Dasgupta, S., Sanyal, S. A single-step method for purification of active his-tagged ribosomes from a genetically engineered Escherichia coli. Nucleic Acids Research. 37 (2), 15 (2009).
- Gesteland, R. F. Isolation and characterization of ribonuclease I mutants of Escherichia coli. Journal of Molecular Biology. 16 (1), 67-84 (1996).
- Li, J., et al. Cogenerating synthetic parts toward a self-replicating system. ACS Synthetic Biology. 6 (7), 1327-1336 (2017).
- Matsuura, T., Kazuta, Y., Aita, T., Adachi, J., Yomo, T. Quantifying epistatic interactions among the components constituting the protein translation system. Molecular Systems Biology. 5, 297 (2009).
- Libicher, K., Hornberger, R., Heymann, M., Mutschler, H. In vitro self-replication and multicistronic expression of large synthetic genomes. Nature Communications. 11 (1), 904 (2020).
- Niederholtmeyer, H., Stepanova, V., Maerkl, S. J. Implementation of cell-free biological networks at steady state. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 15985-15990 (2013).
- Lavickova, B., Laohakunakorn, N., Maerkl, S. J. A partially self-regenerating synthetic cell. Nature Communications. 11 (1), 6340 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados