Method Article
Sistema OnePot PURE Cell-Free
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Presentiamo un metodo rapido ed economico per produrre il sistema TX-TL ricombinante PURE cell-free utilizzando apparecchiature di laboratorio standard.
Abstract
Il sistema di trascrizione-traduzione DEFINITO PURE (sintesi proteica utilizzando elementi ricombinanti) fornisce uno chassis accattivante per la biologia sintetica senza cellule. Sfortunatamente, i sistemi disponibili in commercio sono costosi e la loro sintonizzazione è limitata. In confronto, un approccio fatto in casa può essere personalizzato in base alle esigenze dell'utente. Tuttavia, la preparazione di sistemi fatti in casa è dispendiosa in termini di tempo e ardua a causa della necessità di ribosomi e di 36 purificazioni proteiche su media scala. La razionalizzazione della purificazione delle proteine mediante coculturazione e co-purificazione consente di ridurre al minimo i requisiti di tempo e manodopera. Qui, presentiamo un metodo semplice, regolabile, rapido ed economico per produrre tutti i componenti del sistema PURE entro 1 settimana, utilizzando apparecchiature di laboratorio standard. Inoltre, le prestazioni di OnePot PURE sono paragonabili ai sistemi disponibili in commercio. Il metodo di preparazione OnePot PURE espande l'accessibilità del sistema PURE a più laboratori grazie alla sua semplicità ed economicità.
Introduzione
I sistemi di trascrizione-traduzione senza cellule (TX-TL) costituiscono una piattaforma promettente per lo studio e l'ingegneria dei sistemi biologici. Forniscono condizioni di reazione semplificate e sintonizzabili, in quanto non si basano più su processi di sostegno vitale, tra cui crescita, omeostasi o meccanismi di regolazione1. Pertanto, si prevede che i sistemi privi di cellule contribuiranno allo studio dei sistemi biomolecolari, offriranno un quadro per testare strategie di biodesign razionale2e forniranno un telaio per una futura cellula sintetica3,4. Il sistema PURE completamente ricombinante offre uno chassis particolarmente accattivante grazie alla sua composizione definita e minimale, nonché alla sua regolabilità e sintonizzazione5.
Da quando il primo sistema PURE funzionale e completamente ricombinante è stato istituito nel 20015,sono stati fatti sforzi per espandere i limiti del sistema e ottimizzare la composizione del sistema per migliorare i rendimenti del sistema6,7,8,consentire la regolazione trascrizionale9,la membrana10, 11 e la sintesi proteica secretoria12e facilitare il ripiegamento proteico13,14 . Al giorno d'oggi, ci sono tre sistemi disponibili in commercio: PUREfrex (GeneFrontier), PURExpress (NEB) e Magic PURE (Creative Biolabs). Tuttavia, questi sistemi sono costosi, la loro composizione esatta è proprietaria e quindi sconosciuta e l'adattabilità è limitata.
I sistemi PURE preparati internamente si sono rivelati l'opzione più economica e sintonizzabile15,16. Tuttavia, le 37 fasi di purificazione richieste per le frazioni proteiche e ribosomiche richiedono molto tempo e sono noiose. Sono stati fatti diversi tentativi per migliorare l'efficienza della preparazione del sistema PURE17,18,19. Recentemente abbiamo dimostrato che è possibile co-coltura e co-purificare tutte le proteine non ribosomiali necessarie presenti nel sistema PURE. Questo metodo OnePot ha dimostrato di essere conveniente ed efficiente in termini di tempo, riducendo i tempi di preparazione da diverse settimane a 3 giorni lavorativi. L'approccio genera un sistema PURE con una capacità di produzione di proteine paragonabile al sistema PURExpress20disponibile in commercio . Contrariamente ai precedenti approcci per semplificare la preparazione PURE17,18,19,nell'approccio OnePot tutte le proteine sono ancora espresse in ceppi separati. Ciò consente all'utente di regolare la composizione del sistema OnePot PURE semplicemente omettendo o aggiungendo ceppi specifici o regolando i volumi di inoculazione, generando così sistemi PURE dropout o alterando rispettivamente i rapporti proteici finali.
Il protocollo qui presentato fornisce un metodo dettagliato per la creazione del sistema OnePot PURE come descritto in precedenza20, sebbene β-mercaptoetanolo sia stato sostituito con tris (2-carbossietil) fosfina (TCEP). Inoltre, vengono descritti due metodi per la purificazione dei ribosomi: la tradizionale purificazione del ribosoma senza tag utilizzando l'interazione idrofobica e il cuscino di saccarosio, adattata da Shimizu et al.15e la purificazione del ribosoma Ni-NTA basata su Wang et al.18 e Ederth et al.21 ma significativamente modificata. Quest'ultimo metodo facilita ulteriormente la preparazione del sistema PURE e lo rende accessibile a più laboratori, poiché sono necessarie solo attrezzature di laboratorio standard.
Il protocollo sperimentale riassume la preparazione di un versatile sistema TX-TL privo di celle PURE per fornire una piattaforma senza celle semplice, sintonizzabile ed economica, che può essere preparata utilizzando apparecchiature di laboratorio standard entro una settimana. Oltre a introdurre la composizione standard PURE, indichiamo come e dove può essere regolata, con particolare attenzione ai passaggi critici del protocollo per garantire la funzionalità del sistema.
Protocollo
NOTA: questo protocollo descrive la preparazione di un sistema TX-TL senza celle da componenti ricombinanti. Per comodità, il lavoro è separato in cinque parti. La prima parte descrive le fasi di preparazione, che dovrebbero essere eseguite prima di iniziare il protocollo. La seconda parte descrive la preparazione della soluzione proteica OnePot. La terza parte descrive le purificazioni dei ribosomi, la quarta parte descrive in dettaglio la preparazione della soluzione energetica e l'ultima parte fornisce un manuale per l'impostazione di una reazione PURE. Per comodità, i protocolli sono divisi in giorni e riassunti in programmi giornalieri nella Tabella 1. Seguendo il programma, l'intero sistema può essere preparato in 1 settimana da una persona.
1. Lavori preliminari
- Preparare i supporti di coltura batterica e gli integratori di supporto come descritto nella tabella supplementare 1. Preparare e sterilizzare i materiali necessari, comprese le punte delle pipette, 96 piastre a pozzo profondo.
- Preparazione del ceppo
- Trasformare le deformazioni di espressione indicate nella Tabella 2 con i corrispondenti vettori di espressione utilizzando il metodo dello shock termico.
- Aggiungere plasmide purificato ai batteri chimicamente competenti e incubare sul ghiaccio per 20-30 min.
- Posizionare la miscela a 42 °C per 30 s (shock termico) e quindi rimetterla sul ghiaccio per 2 minuti.
- Pipettare 20 μL dei batteri direttamente su piastre di agar contenenti ampicillina (AMP) e incubare a 37 °C durante la notte. Conservare i piatti a 4 °C per un massimo di 1 settimana.
- Inoculare 3 ml di mezzi LB contenenti AMP con una singola colonia di batteri dalle piastre di agar. Incubare a 37 °C agitando a 260 giri/min durante la notte.
- Miscelare 250 μL di coltura con 250 μL di glicerolo al 50% (v/v) e conservare a -80 °C.
NOTA: per una preparazione più rapida in futuro, conservare i ceppi in una piastra a 96 pozzetti come scorte di glicerolo.
- Confermare tutte le trasformazioni vettoriali mediante PCR e sequenziamento della colonia. Sequenziare il gene, la regione del promotore e il sito di legame del ribosoma.
- Trasformare le deformazioni di espressione indicate nella Tabella 2 con i corrispondenti vettori di espressione utilizzando il metodo dello shock termico.
- Test di espressione
- Inoculare 300 μL di mezzi LB contenenti AMP con circa 1 μL delle scorte di glicerolo preparate in una piastra profonda da 1,3 mL. Sigillare la piastra con una membrana traspirante e quindi incubare a 37 °C agitando a 260 giri/min durante la notte.
NOTA: a questo punto vengono eseguite tutte le espressioni separatamente. - Inoculare 300 μL di mezzi LB freschi contenenti AMP con 1 μL di colture overnight. Incubare a 37 °C agitando a 260 giri/min durante la notte. Dopo 2 ore, indurre le cellule con 100 μM di isopropil β-D-1-tiogalattaccoranoside (IPTG) e crescere per ulteriori 3 ore.
- Miscelare 10 μL della coltura con 10 μL di 2x tampone Laemmli e riscaldare a 95 °C per 10 min. Ruotare i campioni per 1 minuto usando una centrifuga da tavolo e caricare 10 μL del surnatante su un gel PAGE. Eseguire il gel nel tampone Tris/Glicina/SDS a 200 V per 30 min. Risciacquare bene con acqua deionizzata. Coprire il gel con una macchia proteica Coomassie e incubare per 1 ora. Se necessario, detenere il gel in acqua (risultati rappresentativi per la prova di espressione nella Figura 1).
NOTA: utilizzare gel PAGE sfumati (4%-15% o 4%-20%) per ottenere una buona separazione.
- Inoculare 300 μL di mezzi LB contenenti AMP con circa 1 μL delle scorte di glicerolo preparate in una piastra profonda da 1,3 mL. Sigillare la piastra con una membrana traspirante e quindi incubare a 37 °C agitando a 260 giri/min durante la notte.
- Restauro e pulizia della resina di sefarosio IMAC
- Preparazione della colonna.
- Mescolare bene la resina di sefarosio vorticosamente.
- Pipettare la quantità necessaria di resina in una colonna di flusso gravitazionale vuota.
NOTA: La quantità di resina richiesta varia tra la purificazione dell'his-ribosoma e la purificazione delle proteine ed è specificata nelle rispettive sezioni. - Lavare la resina con 30 ml di acqua deionizzata.
- Procedere con la ricarica della colonna come specificato al punto 1.4.4.
NOTA: lasciare sempre passare tutto il liquido attraverso la colonna prima di continuare con il passaggio successivo. Tuttavia, assicurarsi che la colonna non si esaurisca mai. Ogni volta che si fa passare un liquido attraverso la colonna, assicurarsi di interrompere il flusso o continuare al passaggio successivo non appena il liquido raggiunge la resina.
- Restauro.
- Lavare la colonna con 30 ml di acqua deionizzata.
- Applicare 10 mL di una soluzione di EDTA da 0,2 M e 0,5 M naCl.
- Aggiungere 30 mL di una soluzione naCl da 0,5 M.
- Lavare la colonna con 50 ml di acqua deionizzata.
- Conservare in etanolo al 20% (v/v) a 4 °C o continuare con il passaggio successivo.
- Pulitura.
ATTENZIONE: Indossare dispositivi di protezione.- Lavare la colonna con 30 mL di 0,5 M NaOH.
- Lavare la colonna con 30 ml di acqua deionizzata.
- Lavare la colonna con 30 mL di acido acetico 0,1 M.
- Lavare la colonna con 30 ml di acqua deionizzata.
- Lavare la colonna con 30 ml di etanolo al 70% (v/v).
- Lavare la colonna con 50 ml di acqua deionizzata.
- Conservare in etanolo al 20% (v/v) a 4 °C o continuare con il passaggio successivo.
- Ricarica.
- Aggiungere 10 mL di soluzione di solfato di nichel 0,1 M alla colonna.
ATTENZIONE: Il solfato di nichel è tossico. I rifiuti di solfato di nichel devono essere scartati con le precauzioni indicate dal fornitore. - Lavare la colonna con 50 ml di acqua deionizzata.
- Conservare in etanolo al 20% ((v/v)) a 4 °C o continuare con l'equilibratura della colonna.
NOTA: se la colonna viene conservata in etanolo tra un passaggio e l'altro, assicurarsi di rimuovere tutte le tracce di etanolo lavando la colonna con acqua.
- Aggiungere 10 mL di soluzione di solfato di nichel 0,1 M alla colonna.
- Preparazione della colonna.
2. Espressione e purificazione della soluzione proteica OnePot
NOTA: Il protocollo è composto da tre parti divise in giorni (Figura 2). Una procedura di preparazione ideale produce 1,5 mL di 13,5 mg/mL di soluzione proteica OnePot, che corrisponde a più di mille reazioni PURE da 10 μL. Tuttavia, la quantità e la concentrazione ideale della soluzione varieranno da lotto a lotto. Gli utenti esperti possono eseguire più preparazioni OnePot PURE alla volta.
Giorno 1:
- Preparare i supporti di coltura batterica e gli integratori di supporto come descritto nella tabella supplementare 1.
- Preparare e sterilizzare i materiali necessari, comprese le punte delle pipette, due piastre da 96 pozzi profondi e un pallone Erlenmeyer sconcertato da 1 L.
- Preparare tamponi e supplementi come descritto nella tabella supplementare 2. Il filtro sterilizza tutti i tamponi utilizzando filtri tappo bottiglia (0,45 μm) e li conserva a 4 °C. Integrare tutti i tamponi con 1 mM TCEP subito prima dell'uso, se non diversamente indicato.
- Utilizzare 2 ml di resina di sefarosio per la purificazione delle proteine OnePot. Preparare la colonna come descritto al paragrafo 1.4.
- Per preparare le colture iniziali, combinare 20 ml di supporti LB con 20 μL di AMP. In una piastra sterile da 96, 1,3 mL di profondità, aggiungere 300 μL del mezzo in 35 pozzetti. Inoculare ciascuno di essi con il rispettivo ceppo, ad eccezione del fattore di allungamento termo instabile (EF-Tu), e sigillare la piastra con una membrana traspirante.
NOTA: Inoculare la piastra utilizzando un replicatore a 96 pozzetti (vedi Tabella dei materiali). Il volume del pozzo della piastra del pozzo profondo e il volume della coltura di avviamento sono essenziali. Volumi di supporti più grandi o volumi di pozzi più piccoli porteranno a una diversa densità batterica a causa di incongruenze di aerazione. - Per la coltura EF-Tu, inoculare 3 mL di materiale LB in un tubo di coltura da 14 mL con tappo a scatto. Un singolo 3 mL di coltura per EF-Tu è sufficiente per una cultura di espressione OnePot.
- Incubare a 37 °C agitando a 260 giri/min durante la notte.
Giorno 2:
NOTA: Eseguire tutti i passaggi a temperatura ambiente se non diversamente indicato.
- Trasferire 500 mL di lb e 500 μL di AMP nel matraccio sterile sconcertato.
- Inoculare la coltura OnePot PURE con 1675 μL della coltura EF-Tu e 55 μL di ciascuna delle colture dalla piastra del pozzo profondo (Tabella 2).
NOTA: Durante questa fase, la composizione proteica complessiva può essere regolata regolando i rapporti di inoculazione. Assicurarsi che il volume complessivo di inoculazione rimanga costante a 3,6 ml.
FACOLTATIVO: Per confermare che tutti i ceppi sono cresciuti durante la notte, misurare la densità ottica delle colture notturne a 600 nM (OD600)in una piastra a 96 pozzetti utilizzando un lettore di piastre. Utilizzare una diluizione di 10 volte per la misurazione della densità ottica. - Incubare la coltura per 2 ore a 37 °C con un scuotimento di 260 giri/min, o fino a quando l'OD600 della coltura raggiunge 0,2-0,3.
- Indurre la coltura con 500 μL di 0,1 mM IPTG e crescere per ulteriori 3 ore.
- Raccogliere le celle mediante centrifugazione a 4 °C e 3220 x g per 10 minuti e conservare il pellet cellulare a -80 °C fino a nuovo utilizzo.
NOTA: Per ottimizzare i tempi, preparare la soluzione energetica descritta nel paragrafo 4 durante i tempi di incubazione del giorno 2 (Tabella 1).
Giorno 3:
- Misurare le quantità di tamponi necessari per la purificazione descritte nelle fasi seguenti e aggiungere TCEP a tutti loro come indicato nella tabella supplementare 2. Conservare i tamponi rimanenti senza TCEP a 4 °C per future purificazioni.
- Equilibrare la colonna carica (punto 2.4) con 30 mL di tampone A. Dopo aver attraversato 25 mL di buffer A, chiudere la colonna dal basso. In parallelo, continuare con i passaggi 2.15-2.17.
- Scongelare le celle e utilizzare una pipetta sierologica per ricaspendare il pellet cellulare in 7,5 ml di tampone A.
- Lisi delle celle utilizzando un sonicatore a sonda da 130 watt (vedi Tabella dei materiali,diametro punta sonda: 6 mm) con i seguenti parametri: 4 x 20 s impulso acceso, 20 s impulso spento, 70% di ampiezza. Se la sonicazione ha successo, la soluzione diventerà più scura (Figura 2).
NOTA: Assicurarsi di mantenere le cellule sul ghiaccio durante la sonicazione. Posizionare la sonda abbastanza in profondità nella soluzione senza toccare il tubo. Se viene generata una grande quantità di schiuma, il trasferimento di energia verrà smorzato. In tal caso, lasciare che la schiuma si depositi, abbassare la sonda più in profondità nella soluzione e prolungare il tempo di sonicazione. - Rimuovere i detriti cellulari mediante centrifugazione a 21130 x g per 20 minuti a 4 °C immediatamente dopo la sonicazione. Mantenere il lisirato sul ghiaccio.
- Aggiungete il surnatante alla colonna equilibrata. Chiudi la colonna dall'alto e assicurati che non ci siano perdite. Incubare la colonna per 3 ore a 4 °C in rotazione utilizzando un rotatore a tubo.
- Eluire i componenti non legati dalla colonna e lavare con 25 ml di tampone A.
- Lavare la colonna con 25 mL di tampone imidazolo da 25 mM (23,95 mL di tampone A e 1,25 mL di tampone B).
- Eluire le proteine con 5 mL di tampone imidazolo da 450 mM (0,5 mL di tampone A e 4,5 mL di tampone B). Mantenere le proteine eluite sul ghiaccio in ogni momento.
- Diluire l'eluato con 25 ml di tampone HT, mantenere la miscela sul ghiaccio. Aggiungere 15 mL a un filtro centrifugo da 15 mL e concentrare fino a un volume di 1,5 mL. Aggiungere i restanti 15 mL al filtro con la soluzione concentrata e concentrare nuovamente a 1,5 mL.
- Aggiungere 10 mL di tampone HT al campione concentrato e concentrare a 1 mL. Aggiungere una quantità uguale di buffer di riserva B e conservare a -80 °C fino a nuovo utilizzo.
NOTA: Un giro di scambio/concentrazione richiede circa 60 minuti di rotazione a 3220 x g a 4 °C. - Durante lo scambio del buffer, ripristinare la colonna come specificato nella sezione 1.4.
Giorno 4:
- Misurare la concentrazione proteica utilizzando il test Bradford come descritto dal fornitore. Concentrare il campione con un filtro centrifugo cutoff da 0,5 mL di 3 kDa a 20 mg/mL.
NOTA: Diluire la soluzione proteica 25 volte o 50 volte prima delle misurazioni della concentrazione per evitare di sovrasaturare il saggio di Bradford. - Per stabilire la concentrazione proteica ideale, eseguire un test di espressione in questa fase (paragrafo 5.2) con diverse concentrazioni della soluzione proteica. Per eseguire la titolazione, mantenere costante il volume totale della soluzione e pipettare la soluzione proteica OnePot, incluso il buffer di riserva B, a cinque diversi rapporti (Tabella supplementare 7).
- Verificare la composizione proteica OnePot PURE utilizzando SDS-PAGE (Figura 3A). Diluire 2,5 μL del campione con 7,5 μL di acqua, mescolare con 10 μL di 2x tampone Laemmli e quindi caricare 5 μL e 2,5 μL dei campioni sul gel. Eseguire SDS-PAGE come specificato nella sezione 1.3.3.
- Aliquotare la soluzione proteica in aliquote da 50 μL dopo aver verificato l'espressione e regolato la concentrazione. Conservare la soluzione proteica OnePot PURE a -80 °C fino a nuovo utilizzo.
NOTA: Se si sospetta che un componente proteico non sia presente o sia presente in una concentrazione inferiore al previsto in OnePot PURE, eseguire i seguenti passaggi. - Verificare se la coltura notturna del rispettivo ceppo è cresciuta a un ritmo comparabile a quello delle altre colture eseguendo misurazioni ottiche della densità (OD600) di tutte le colture.
- Eseguire un ulteriore test di espressione del ceppo specifico per verificare l'espressione della proteina sospetta.
3. Soluzione di ribosoma
NOTA: vengono introdotte due diverse strategie di purificazione dei ribosomi, una per i ribosomi marcati con esastidina e una per i ribosomi non marcati. Il principale vantaggio del metodo di purificazione che utilizza his-purification su una colonna di flusso gravitazionale Ni-NTA di affinità standard è che la purificazione è facile, veloce e non richiede ulteriori attrezzature di laboratorio, come un sistema FPLC e un ultracentrifuga. Tuttavia, la capacità di produzione di proteine nelle reazioni OnePot PURE è di circa un terzo rispetto ai ribosomi privi di tag. Pertanto, scegliere il metodo per la produzione di ribosomi in base al fatto che un alto rendimento sia importante per l'applicazione data.
-
Purificazione del ribosoma con etichetta His
NOTA: Questo protocollo utilizza il ceppo E. coli RB1, un dono del Professor Wang (Columbia University, USA)18. Questo ceppo ha un inserimento genomico di un tag esastidina sul terminale C della proteina ribosomiale 50S (L7/L12), consentendo la purificazione utilizzando una colonna di flusso gravitazionale Ni-NTA. La resa abituale è di circa 0,5 ml di ribosomi da 3,45 μM, che è sufficiente per più di cinquecento 10 μL di reazioni PURE.
Giorno 1:
- Preparare i supporti di coltura batterica e gli integratori di supporto come descritto nella tabella supplementare 1.
- Preparare e sterilizzare i materiali necessari, comprese le punte delle pipette, un pallone Erlenmeyer da 5 L e un pallone Erlenmeyer da 100 ml.
- Preparare tamponi e supplementi come descritto nella tabella supplementare 2. Il filtro sterilizza tutti i tamponi utilizzando filtri tappo bottiglia (0,45 μm) e li conserva a 4 °C.
Giorno 2:
- Pipettare 5 mL di resina su una colonna e preparare la colonna come specificato al punto 1.4.
NOTA: A causa del maggiore volume della resina, il restauro e la purificazione richiedono molto più tempo. Utilizzare una colonna diversa per la purificazione dei ribosomi per evitare la contaminazione incrociata e pulirla accuratamente prima della purificazione. - Preparare una coltura notturna del ceppo E. coli RB1 inoculando 35 mL di lb in un matraccio Erlenmeyer da 100 mL. Incubare a 37 °C agitando a 260 giri/min.
Giorno 3:
NOTA: Eseguire tutti i passaggi a temperatura ambiente se non diversamente indicato.
- Aggiungere 2 L di lb in un matraccio sterile da 5 L, inoculare con 12 ml di coltura notturna e quindi incubare per 3-4 ore a 37 °C agitando a 260 giri/min.
NOTA: In alternativa, eseguire la coltura batterica in 4 x 500 mL di colture in flaconi sconcertati da 1 L. - Pellet le celle mediante centrifugazione per 10 min a 3220 x g e 4 °C. Conservare a -80 °C fino a nuovo utilizzo.
Giorno 4:
- Equilibrare la colonna preparata al punto 3.1.4. con 30 ml di tampone di lisi.
- Risusciendi il pellet in 20 ml di tampone di lisi utilizzando una pipetta sierologica.
- Lisire le celle con un sonicatore a sonda da 130 watt (vedi Tabella dei materiali,diametro punta sonda: 6 mm) su ghiaccio con i seguenti parametri: 11 x 20 s impulso acceso; 20 s impulso spento, ampiezza del 70% (vedere il passaggio 2.16 per i dettagli della procedura).
- Immediatamente dopo la sonicazione, rimuovere i detriti cellulari mediante centrifugazione per 20 minuti a 21130 x g a 4 °C. Mantenere il lisirato sul ghiaccio.
- Carica il surnatante sulle colonne e lascialo passare.
- Lavare la colonna con le seguenti miscele di tamponi di lisi ed eluizione.
- Lavare 0: utilizzare 30 ml di tampone di lisi.
- Lavaggio 1: utilizzare 30 mL di imidazolo da 5 mM (29 mL di tampone di lisi, 1 mL di tampone di eluizione).
- Lavaggio 2: utilizzare 60 mL di imidazolo da 25 mM (50 mL di tampone di lisi, 10 mL di tampone di eluizione).
- Lavare 3: utilizzare 30 mL di imidazolo da 40 mM (22 mL di tampone di lisi, 8 mL di tampone di eluizione).
- Lavaggio 4: utilizzare 30 mL di imidazolo da 60 mM (18 mL di tampone di lisi, 12 mL di tampone di eluizione).
- Eluire i ribosomi con 7,5 ml del tampone di eluizione. Mantenere le proteine eluite sul ghiaccio in ogni momento.
- Aggiungere 22 μL di β-mercaptoetanolo puro a 45 mL di tampone ribosomico.
ATTENZIONE: β-mercaptoetanolo è tossico. Prendere precauzioni di sicurezza e lavorare in una cappa aspirante. - Aggiungere l'eluato a un filtro centrifugo da 15 ml e concentrare a 1 mL.
- Aggiungere 15 mL di tampone ribosoma al campione concentrato e concentrare nuovamente a 1 mL.
NOTA: ripetere due volte il passaggio precedente. - Conservare a -80 °C fino a nuovo utilizzo.
NOTA: Un ciclo di scambio/concentrazione richiede circa 60 minuti di centrifugazione a 3220 x g a 4 °C. - Durante lo scambio del buffer, ripristinare la colonna come specificato nella sezione 1.4.
Giorno 5:
- Determinare la concentrazione di ribosomi misurando l'assorbanza a 260 nM di un campione diluito 1:100 in tampone ribosomi. Un valore di assorbanza di 10 della soluzione diluita corrisponde a 23 μM di soluzione non diluita come descritto in precedenza16.
- Implementare una concentrazione finale di stock di 3,45 μM. Per regolare la concentrazione, diluire i ribosomi con tampone ribosomiale o concentrarli ulteriormente mediante centrifugazione a 14000 x g in un filtro centrifugo da 3 kDa 0,5 mL a 4 °C.
NOTA: Per ottenere un'espressione ottimale del sistema, eseguire una titolazione della concentrazione di ribosomi (paragrafo 5.2, Tabella supplementare 7). - Verificare la composizione del ribosoma utilizzando SDS-PAGE (Figura 3A) come specificato al paragrafo 1.3.3. Diluire 2,5 μL del campione con 7,5 μL di acqua, mescolare con 10 μL di 2x tampone Laemmli, quindi caricare 5 μL e 2,5 μL dei campioni sul gel.
-
Purificazione del ribosoma senza tag
NOTA: La purificazione dei ribosomi senza tag viene eseguita utilizzando un sistema FPLC(Tabella dei materiali)e si basa sulla cromatografia di interazione idrofobica utilizzando 2 colonne di butile da 5 ml(Tabella dei materiali). Sebbene i ribosomi possano essere purificati da qualsiasi ceppo, l'uso del ceppo E. coli A19 (E. coli Genetic Resources at Yale CGSC) è vantaggioso a causa della sua delezione della RNasi I22. Eseguire la purificazione a 4 °C in una cella frigorifera o in un armadio di raffreddamento. La resa abituale è di circa 0,5 ml di ribosomi da 10 μM, che corrisponde a più di cinquecento reazioni PURE da 10 μL.
Giorno 1:
- Preparare i supporti di coltura batterica e gli integratori di supporto come descritto nella tabella supplementare 1.
- Preparare e sterilizzare i materiali necessari, comprese le punte delle pipette, il matraccio Erlenmeyer da 5 L e il matraccio Erlenmeyer da 100 ml.
- Preparare tamponi e supplementi come descritto nella tabella supplementare 2. Il filtro sterilizza tutti i tamponi utilizzando filtri tappo bottiglia (0,45 μm) e li conserva a 4 °C.
Giorno 2:
- Per preparare una coltura notturna del ceppo E. coli A19, inoculare 35 mL di lb in un matraccio Erlenmeyer da 100 mL. Incubare a 37 °C agitando a 260 giri/min.
Giorno 3:
- Trasferire 2 L di lb media nel matraccio sterile sconcertato da 5 L, inoculare con 30 ml di coltura notturna e quindi incubare per 3-4 ore a 37 °C agitando a 200 giri/min.
- Pellet le celle mediante centrifugazione a 4000 x g per 15 min a 4 °C. Risuscisciare il pellet in 25 mL di tampone di sospensione e conservare a -80 °C fino a nuovo utilizzo.
Giorno 4:
- Eseguire i passaggi 3.2.8-3.2.12 in parallelo con i passaggi 3.2.13-3.2.19.
- Scongelare e lisi le celle utilizzando un sonicatore sonda da 130 watt (vedi Tabella dei materiali e diametro della punta della sonda: 6 mm) su ghiaccio con i seguenti parametri: 12 x 20 s impulso acceso; 20 s pulse off, 70% di ampiezza (vedere i dettagli della procedura del passaggio 2.16).
- Rimuovere immediatamente i detriti cellulari mediante centrifugazione a 20000 x g per 20 min a 4 °C.
- Aspirare il surnatante e misurare il volume. Aggiungere un volume uguale di tampone di sospensione (sale alto) per regolare la concentrazione finale di solfato di ammonio a 1,5 M e mescolare bene.
- Rimuovere il precipitato per centrifugazione a 20000 x g per 20 minuti a 4 °C.
- Filtrare il surnatante utilizzando un filtro a siringa a membrana di polietereulfone da 0,45 μm prima della purificazione FPLC e raccogliere il filtrato in un flacone di vetro da 100 ml. Mantenere il surnatante a 4 °C in ogni momento.
- Impostare il sistema FPLC per la purificazione della cromatografia a interazione idrofobica utilizzando una doppia colonna butinica (2 x 5 ml) come segue. Per questa configurazione, un volume di colonna (CV) si riferisce a un volume di 10 ml.
- Saranno necessarie tre insenature: due come linee tampone e una come linea campione. A causa delle impostazioni predefinite del purificatore, è conveniente scegliere le linee A1 e B1 per il buffer C e il buffer D, rispettivamente, e la linea A2 come linea del campione. Applicare una portata predefinita di 4 mL/min, ad eccezione dei lavaggi della pompa (10 mL/min) o se non diversamente indicato.
NOTA: poiché il TCEP è un reagente costoso, aggiungere la quantità corrispondente ai buffer C e D solo dopo la fase di bilanciamento. - Eseguire un lavaggio con pompa di sistema in etanolo al 20% ((v / v)) per pulire il sistema e rimuovere potenziali contaminazioni da precedenti purificazioni. Impostare manualmente una portata di 0,2 ml/min e montare la colonna. Arrestare il flusso.
- Eseguire un lavaggio della pompa del sistema con acqua. Lavare la colonna con 3 CV di acqua.
- Equilibratione: posizionare gli insenature A1 e A2 nel tampone C e l'ingresso B1 nel tampone D senza TCEP. Eseguire un lavaggio della pompa ed equilibrare la colonna con 4 CV di buffer C.
- Aggiungere TCEP ai buffer C e D.
- Preparare tubi da 15 mL o tubi collettori di frazione rotonda trasparenti al collettore di frazione per raccogliere frazioni di eluizione da 4-5 ml.
- Caricamento: Posizionare l'ingresso A2 nel flacone con il campione filtrato. Caricare circa il 90% del volume del campione sulla colonna. Diluire il campione con 20 mL di tampone C contenente TCEP e caricare 10 mL del campione sulla colonna. Ripetere la fase di diluizione almeno due volte e caricare il maggior numero possibile di campioni sulla colonna. È fondamentale assicurarsi che nessuna aria venga aspirata nella macchina.
- Fase di lavaggio 1: lavare con 3 CV di tampone C per rimuovere i componenti non legati.
- Fase di lavaggio 2: lavare con 5 CV di tampone C all'80% e tampone D al 20%.
- Eluizione: eluire il prodotto applicando il 50% del tampone C e il 50% del tampone D, con un volume totale di eluizione di 5 CV. Raccogliere questa frazione nei tubi del collettore.
- Fase di lavaggio 3: Eluire tutti i contaminanti fortemente interagenti utilizzando il tampone D al 100% con un volume totale di 5 CV.
- Analizzare lo spettro di assorbimento della frazione campione a 260 o 280 nM (Figura 4). Il primo picco mostra le proteine non assorbite eluite durante il carico e la prima fase di lavaggio; il secondo picco mostra i contaminanti che sono stati eluiti durante la seconda fase di lavaggio. Il terzo picco monitora il prodotto finale e l'ultimo picco mostra i contaminanti fortemente interagenti. Raggruppate tutte le frazioni campione corrispondenti al terzo picco per un'ulteriore elaborazione. Mantenere le proteine eluite sul ghiaccio in ogni momento.
- Sovrapporre delicatamente la frazione recuperata su 15 mL del tampone del cuscino in quattro tubi di ultracentrifugazione in policarbonato. Aggiungere un massimo di 15 mL del campione a 15 mL del tampone del cuscino. Assicurati di bilanciare bene il peso del tubo. Pellet i ribosomi mediante ultracentrifugazione a 100000 x g a 4 °C per 16 ore.
NOTA: Assicurarsi che non siano presenti crepe nei tubi di ultracentrifugazione. - Pulire e reimpostare la colonna come segue. Una portata di 5 ml/min funziona bene. Posizionare tutte le insenature nell'acqua ed eseguire un lavaggio a pompa. Lavare la colonna con 2 CV di acqua.
- Posizionare l'ingresso in una soluzione di NaOH da 0,5 M, eseguire un lavaggio a pompa e successivamente lavare la colonna con 3 CV di NaOH.
- Posizionare l'ingresso in acqua, eseguire un lavaggio a pompa e quindi lavare la colonna in 2 CV di acqua.
- Posizionare l'ingresso in una soluzione di acido acetico 0,1 M, eseguire un lavaggio a pompa e successivamente lavare la colonna con 3 CV di soluzione di acido acetico.
- Pompa lavare e lavare la colonna con 2 CV di acqua.
- Posizionare tutte le entrate in etanolo al 20% ((v/v)), eseguire una fase di lavaggio della pompa e conservare la colonna in etanolo al 20% ((v/v)) lavandolo con 3 CV di una soluzione di etanolo al 20% ((v/v)).
NOTA: assicurarsi che il sistema non si asciughi o non risucchi l'aria. Non applicare mai il tampone direttamente all'etanolo o l'etanolo al tampone. Aggiungere sempre una fase di lavaggio ad acqua nel mezzo, altrimenti c'è il rischio di precipitati che intasano la colonna. Assicurati di aggiungere abbastanza provette per la raccolta dei campioni.
Giorno 5:
- Scartare il surnatante e con attenzione, senza disturbare il pellet traslucido, lavare ogni pellet con 0,5 ml di tampone ribosoma ghiacciato. Ripetere questo passaggio due volte.
- Gravare ciascuno dei pellet trasparenti in 100 μL di tampone ribosoma su ghiaccio utilizzando una barra di agitazione magnetica (diametro 3 mM, lunghezza 10 mM) su un agitatore magnetico utilizzando la velocità più bassa possibile. Raccogliere i ribosomi riconsoppiati e lavare i tubi con ulteriori 50 μL di tampone ribosoma.
NOTA: Il pellet traslucido è difficile da vedere. Pertanto, lavare accuratamente il pellet dai lati del tubo. - Determinare la concentrazione di ribosomi misurando l'assorbanza a 260 nM del campione diluito in un rapporto di 1:100 nel tampone ribosomico. Un'assorbanza di 10 della soluzione diluita corrisponde a 23 μM di soluzione non diluita come descritto in precedenza16.
- Implementare una concentrazione finale di stock di 10 μM. Per regolare la concentrazione, diluire i ribosomi con tampone di ribosomi o concentrarli ulteriormente mediante centrifugazione a 14000 x g in un filtro centrifugo da 3 kDa a 4 °C.
NOTA: Per ottenere un'espressione ottimale del sistema, eseguire la titolazione dei ribosomi (paragrafo 5.2, Tabella supplementare 7). - Verificare la composizione del ribosoma con SDS-PAGE (Figura 3A) come specificato al paragrafo 1.3.3. Diluire 2,5 μL del campione con 7,5 μL di acqua, mescolare con 10 μL di 2x tampone Laemmli, quindi caricare 5 μL e 2,5 μL dei campioni sul gel.
4. Soluzione energetica
NOTA: La composizione per la soluzione energetica 2,5x qui introdotta è un esempio di una soluzione che ha funzionato bene per una reazione TX-TL standard. Per ottimizzare i tempi, preparare la soluzione energetica durante il giorno 2. La preparazione della soluzione di aminoacidi è spiegata in dettaglio, seguita dalla procedura di preparazione finale.
- Soluzione di aminoacidi
NOTA: Preparare la soluzione di aminoacidi alla rinfusa. La preparazione della quantità di soluzioni di materiale amminoacidico necessaria per un volume finale di almeno 2000 μL ridurrà l'errore di pesatura per le quantità altrimenti molto piccole. La concentrazione complessiva della soluzione di amminoacidi è limitata dalla solubilità degli amminoacidi e dalle rispettive concentrazioni di soluzioni stock. Per il sistema STANDARD PURE, preparare una soluzione con una concentrazione finale di 3,25 mM. Utilizzare la tabella di calcolo della soluzione di amminoacidi (Tabella supplementare 3) come modello. Utilizzare la cisteina sotto forma di sale per garantire una solubilità sufficiente. Evitare l'uso di metodi di preparazione degli aminoacidi a base di KOH. È possibile pesare direttamente le quantità esatte di amminoacidi nella soluzione finale di amminoacidi senza preparare una soluzione stock per tutti gli amminoacidi. Tuttavia, questo è più impegnativo e meno preciso.- Preparare soluzioni stock per ogni amminoacido come descritto nella tabella supplementare 3, ad eccezione della tirosina.
NOTA: A causa delle diverse solubilità degli amminoacidi in acqua, le rispettive concentrazioni suggerite della soluzione stock differiscono. - La massa minima [mg] fornisce la massa minima approssimativa necessaria per ottenere una quantità sufficiente di soluzione stock per il volume complessivo target, come riferimento.
NOTA: La massa minima è calcolata con un surplus del 10%. - Per una preparazione più semplice delle soluzioni, non pesare la quantità esatta di aminoacidi, ma invece, per la massa a portata di mano, regolare la quantità di acqua per ottenere la concentrazione desiderata. Calcolare la quantità di acqua deionizzata (Acqua da aggiungere [μL]) necessaria, in base alla massa effettiva riempita (celle giallo chiaro) e alla concentrazione desiderata utilizzando il foglio di calcolo nella Tabella supplementare 3.
- Solubilizzare le soluzioni di materiale amminoacidico vorticosamente fino a quando tutto il precipitato si è dissolto. Le singole soluzioni di aminoacidi possono essere conservate a -20 °C per diverse settimane.
NOTA: Alcuni aminoacidi sono difficili da sciogliere in acqua; il processo potrebbe richiedere del tempo. - Pesare la quantità esatta di tirosina necessaria per ottenere una concentrazione finale di 3,25 mM direttamente nel tubo per la soluzione di amminoacidi.
NOTA: La tirosina è molto difficile da sciogliere in acqua. Aggiungilo direttamente invece di preparare una soluzione stock. - Aggiungere le quantità corrispondenti di soluzioni di materiale amminoacidico e acqua come indicato nel volume finale per aggiungere la colonna [μL] (celle azzurre) e vorticosamente bene la soluzione. Conservare la soluzione di aminoacidi completata a -80 °C fino a nuovo utilizzo.
- Preparare soluzioni stock per ogni amminoacido come descritto nella tabella supplementare 3, ad eccezione della tirosina.
- Preparazione della soluzione energetica
NOTA: In totale, la soluzione energetica 2,5x contiene 0,75 mM di ciascun amminoacido, 29,5 mM di acetato di magnesio, 250 mM di glutammato di potassio, 5 mM di ATP e GTP ciascuno, 2,5 mM di CTP, UTP e TCEP, rispettivamente, 8,75 mg/mL di tRNA da E. coli MRE 600, 50 mM di creatina fosfato, 0,05 mM di acido folinico, 5 mM di spermidina e 125 mM di HEPES. Gli utenti per la prima volta preparano la soluzione energetica in piccoli lotti da 200 μL. Conservare le singole soluzioni preparate secondo la tabella supplementare 4 a -20 °C o -80 °C per un uso successivo.- Scongelare tutte le soluzioni acquose menzionate nella tabella complementare 5 sul ghiaccio.
- Nel frattempo, preparare le soluzioni di riserva per i restanti componenti elencati nella tabella supplementare 4. Conservare tutte le soluzioni sul ghiaccio dopo la preparazione.
NOTA: Aggiungere 500 μL di RNasi e acqua priva di DNasi direttamente al flaconcino per sciogliere i tRNA liofilizzati. Mescolare bene con un leggero vortice; limitare il pipettaggio per evitare l'introduzione di RNasi. - Aggiungere i volumi calcolati (Tabella supplementare 5) di soluzioni stock e acqua e mescolare bene usando un vortice. Mantenere la soluzione sul ghiaccio in ogni momento.
- Misurare il pH della soluzione pipettando 1 μL su una striscia di pH, per assicurarsi che il pH della soluzione sia neutro.
- Aliquotare la soluzione energetica a 50-100 μL per tubo su ghiaccio e conservare a -80 °C fino a nuovo utilizzo. Durante l'aliquotazione, vortice il calcio principale frequentemente per evitare che i componenti precipitino.
NOTA: Facoltativamente, condurre un test di attività della soluzione energetica di nuova soluzione rispetto a soluzioni energetiche commerciali, ad esempio la soluzione A in PURExpress. Se si osservano prestazioni significativamente inferiori del sistema con la soluzione energetica, l'ottimizzazione delle concentrazioni di ioni, in particolare ioni magnesio, mediante titolazione (5-20 mM) può essere vantaggiosa.
5. Reazione OnePot PURE
- Modello di DNA
NOTA: Le proteine codificate a valle del promotore T7 possono essere espresse in PURE da DNA lineare o circolare. Generando un modello di DNA lineare utilizzando l'estensione PCR, è possibile omettere noiose fasi di clonazione. I modelli lineari per questo studio sono stati generati dalla PCR come descritto di seguito, utilizzando una DNA polimerasi ad alta fedeltà (Tabella dei materiali). Le sequenze di primer, le temperature di fusione e le impostazioni del termociclatore utilizzate in questo studio sono specificate nella tabella supplementare 6. La preparazione del modello di DNA non è inclusa nel programma giornaliero.- Impostare una reazione PCR come raccomandato dal fornitore di polimerasi.
NOTA: I parametri ottimizzati per una DNA polimerasi ad alta fedeltà(Tabella dei materiali)sono riportati nella Tabella supplementare 6. - Amplificare il gene bersaglio (ad esempio, eGFP) come modello lineare da un plasmide o genoma utilizzando primer gene-specifici (500 nM) (per i parametri, vedere tabella supplementare 6).
- L'amplificazione genera brevi estensioni per fornire sequenze di ricottura per i seguenti passaggi di estensione PCR.
- Controllare l'amplicon su un gel di acarosio per le dimensioni e la purezza corrette.
- Usa il DNA amplificato come modello per i successivi passaggi di estensione. Impostare una reazione di almeno 50 μL.
- Eseguire 10 cicli di amplificazione PCR con i primer di estensione (2,5 nM). Dopo aver completato i cicli di amplificazione, aggiungere immediatamente i primer finali (500 nM) alla stessa reazione ed eseguire 30 cicli per amplificare il prodotto PCR esteso. Trova le temperature di fusione e le sequenze di primer nella Tabella supplementare 6.
- Purificare i frammenti di DNA utilizzando un kit di purificazione del DNA ed eluire il DNA in acqua priva di nucleasi invece di EDTA contenente tampone di eluizione.
- Controllare il modello lineare su un gel di acarosio per dimensioni e purezza corrette.
- Misurare la concentrazione di DNA in ng/μL utilizzando uno spettrofotometro UV-Vis.
- Impostare una reazione PCR come raccomandato dal fornitore di polimerasi.
- Impostazione della reazione PURE
NOTA: La composizione finale della reazione è 1x soluzione energetica, ribosomi privi di tag o ribosomi His-tag, proteine OnePot PURE e modello di DNA. Il rapporto del volume di reazione comprende il 40% di soluzione energetica, il 30% di proteine e soluzione di ribosomi e il 30% di DNA e acqua. I volumi di reazione tipici variano tra 5 μL e 25 μL. Quantificare continuamente l'espressione di una proteina fluorescente su un lettore di piastre. Usa un Green Lys in vitro Translation Labeling System, che incorpora residui di lisina etichettati fluorescentemente in proteine di nuova sintesi, per verificare l'espressione di proteine non fluorescenti su un gel SDS-PAGE. Un modello di reazione di esempio è fornito in Tabella complementare 7 per aiutare a stabilire una reazione di espressione libera da cellule PURE. Le celle in giallo indicano i valori di input dell'utente e le celle in arancione indicano reagenti aggiuntivi da aggiungere facoltativamente alla reazione. Mantenere precisi i rapporti di volume dei componenti per garantire il corretto equilibrio ioio. Ad esempio, per ottenere una maggiore concentrazione proteica, aumentare la concentrazione della soluzione proteica OnePot; tuttavia, non aumentare il volume della soluzione proteica aggiunta alla reazione.- Inserisci la concentrazione [ng/μL] e la lunghezza [coppie di basi] del DNA nelle corrispondenti cellule gialle nel foglio di calcolo. Utilizzare 2-10 nM di DNA per la reazione.
- Riempire il volume totale di reazione desiderato in μL.
- Rimuovere i reagenti necessari dal congelatore e scongelarli sul ghiaccio.
NOTA: il ricongelamento dei componenti è possibile senza una diminuzione della funzionalità. Tuttavia, ridurre al minimo il numero di cicli di congelamento-disgelo e il tempo in cui i campioni vengono conservati sul ghiaccio il più possibile. - Pipettare le quantità calcolate di acqua, DNA e soluzione energetica su un lato del tubo PCR o su un angolo di un pozzo sulla piastra a 384 pozzetti. Aggiungere la quantità richiesta di qualsiasi reagente aggiuntivo sullo stesso lato. Ridurre al minimo il numero di campioni per esperimento per evitare l'evaporazione del campione e la distorsione del tempo di inizio sperimentale.
NOTA: È fondamentale mantenere la componente energetica fisicamente separata dalle componenti proteiche per evitare il consumo prematuro delle fonti energetiche e rese inferiori. - Pipettare le quantità calcolate di proteine e soluzione di ribosomi all'altro lato di un tubo PCR o all'angolo opposto della piastra a 384 pozzetti.
NOTA: utilizzare le miscele master quando possibile per ridurre l'impatto degli errori di pipettaggio. Dopo i test iniziali, le soluzioni di ribosomi e proteine possono essere miscelate e conservate come un'unica soluzione. - Ruotare per un breve periodo (30 s) per unire i componenti della reazione. Per evitare l'evaporazione durante gli esperimenti di lettura di piastre, aggiungere 35 μL di cera liquida e sigillare la piastra con un sigillante trasparente (vedi Tabella dei materiali).
- Incubare per un minimo di 3 ore a 37 °C.
- Per la lettura su un lettore di piastre, misurare l'intensità di fluorescenza alla lunghezza d'onda richiesta ogni 2 minuti (i risultati rappresentativi sono mostrati nella Figura 3B).
- Eseguire la procedura seguente per i campioni etichettati Green Lys.
- Dopo l'espressione cell-free, incubare il campione con 0,16 μg/μL di RNasi A per 30 minuti a 37 °C per rimuovere lo sfondo fluorescente del kit di etichettatura Green Lys.
NOTA: utilizzare la RNasi A, poiché altri tipi di RNasi non rimuovono sufficientemente lo sfondo. - Visualizzare l'espressione proteica eseguendo SDS-PAGE come specificato al paragrafo 1.3.3. Lavare delicatamente il gel non macchiato in acqua deionizzata e immaginarlo su un imager fluorescente utilizzando una lunghezza d'onda di eccitazione di 488 nm.
- Successivamente, colorare il gel utilizzando i metodi di colorazione Coomassie convenzionali. Per i parametri appropriati si veda il punto 1.3.3.
NOTA: Eseguire una titolazione della soluzione proteica con la concentrazione di ribosomi raccomandata e, se necessario, titolare i ribosomi con la concentrazione ottimale di proteina OnePot in seguito. Utilizzare il kit commerciale PURExpress ΔRibosome come controllo positivo. La soluzione A, il Factor Mix e la soluzione ribosoma corrispondono rispettivamente all'energia preparata, alla soluzione proteica OnePot e ai ribosomi purificati.
Risultati
Il protocollo di cui sopra è progettato per facilitare la creazione del sistema TX-TL privo di cellule PURE in qualsiasi laboratorio. Il protocollo include una descrizione dettagliata della preparazione delle tre parti distinte del sistema PURE: la proteina OnePot, il ribosoma e la soluzione energetica. Una pianificazione giornaliera dettagliata, che ottimizza il flusso di lavoro, è mostrata nella Tabella 1. Il flusso di lavoro è ottimizzato per la purificazione dei ribosomi his-tagged e gli intervalli di tempo possono differire leggermente se viene eseguita la purificazione dei ribosomi senza tag. Un preparato fornisce una quantità sufficiente di PURE per un minimo di cinquecento reazioni da 10 μL. Inoltre, le soluzioni preparate sono stabili per più di un anno a -80 °C e possono resistere a più cicli di congelamento-disgelo.
Livelli di sovraespressione adeguati per tutti i ceppi sono cruciali per la funzionalità della soluzione proteica finale. La Figura 1 mostra una sovraespressione riuscita in tutti i 36 singoli ceppi utilizzati successivamente per la preparazione proteica OnePot. La variazione delle intensità di banda delle proteine sovra-espresse si è verificata molto probabilmente a causa di una distorsione nei volumi di carico sul gel SDS-PAGE. Le dimensioni proteiche previste sono riassunte nella Tabella 2. GlyRS e PheRS sono costituiti da due subunità di vari pesi molecolari; le restanti 34 proteine sono costituite da una singola subunità. La chiave della semplicità e dell'efficacia temporale di questo protocollo è la fase di coculturazione e co-purificazione (Figura 2). La soluzione proteica OnePot è stata preparata aumentando il rapporto tra il ceppo EF-Tu e tutti gli altri ceppi di espressione. La composizione complessiva delle proteine finali è stata analizzata da SDS-PAGE (Figura 3A). Dai gel (corsie 2, 3), si nota che EF-Tu (43,3 kDa) è presente in una concentrazione maggiore rispetto alle altre proteine, come previsto. Mentre il gel fornisce una buona prima indicazione dei rapporti di espressione proteica, è difficile determinare se e a quale livello ogni singola proteina è stata espressa. Pertanto, si consiglia vivamente di confermare la sovraespressione in ciascun ceppo prima della coculturazione, come mostrato sopra.
Il ribosoma E. coli è una macchina molecolare complessa composta da oltre 50 singole subunità proteiche23. Uno spettro di assorbimento rappresentativo a 260 nm per la purificazione del ribosoma senza tag è mostrato nella Figura 4; il terzo picco è caratteristico dell'eluizione ribososoma riuscita. Per entrambi i metodi di purificazione dei ribosomi, è stato osservato il modello di esecuzione previsto sul gel SDS-PAGE (Figura 3A)18. Abbiamo osservato contaminazioni per entrambe le purificazioni, anche se in piccole quantità (<10%). In particolare, diversi contaminanti erano presenti nei ribosomi tag-free (corsie 5, 6) e His-tagged (corsie 11, 12) a causa della variazione del metodo. Per riferimento dell'utente, sono inclusi anche i gel SDS-PAGE per i sistemi combinati (corsie 8, 9 e 14, 15).
Infine, vengono confrontate le prestazioni dei sistemi preparati (Figura 3) utilizzando le diverse varianti di ribosomi. I corsi temporali di espressione eGFP in vitro mostrano che entrambi i sistemi PURE sono funzionali e producono eGFP fluorescenti. Tuttavia, la soluzione proteica OnePot combinata con i ribosomi His-tagged, utilizzando la concentrazione di ribosomi ottimizzata dalla titolazione, ha prodotto solo un terzo del livello di espressione della versione ribosoma non marcata (Figura 3B). Risultati simili sono stati osservati quando tre proteine di diverse dimensioni sono state espresse ed etichettate utilizzando il sistema di etichettatura in vitro del tRNA Green Lys (Figura 3C). Come si vede sul gel fluorescente, i prodotti a figura intera sono stati espressi con successo in entrambi i sistemi; tuttavia, solo circa la metà del livello di espressione è stato raggiunto con il sistema di ribosomi His-tag. Oltre all'etichettatura di fluorescenza, le bande previste per tutte e tre le proteine sono distinguibili su un gel colorato di Coomassie (Figura 3D). I risultati mostrano che il sistema di espressione introdotto, che può essere preparato entro una settimana in un laboratorio con attrezzature standard, può essere utilizzato per l'espressione in vitro di proteine codificate a valle del promotore T7 da modelli lineari.

Figura 1: Risultati rappresentativi per il test di sovraespressione per tutti i ceppi di espressione del sistema PURE. I numeri e le dimensioni delle proteine PURE sono riassunti nella Tabella 2. I numeri proteici 21, 24 e 27 sono contrassegnati con una stella per una migliore visualizzazione. Fare clic qui per visualizzare una versione più grande di questa figura.
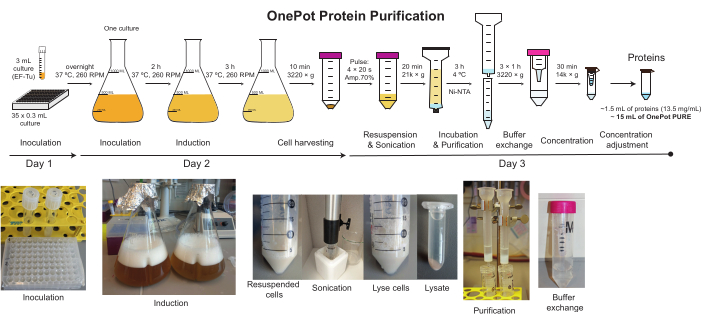
Figura 2: Purificazione delle proteine OnePot. La rappresentazione schematica e le fotografie corrispondenti di tutte le fasi coinvolte nella produzione della soluzione proteica OnePot. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Prestazioni dei sistemi preparati utilizzando le diverse varianti di ribosomi. (A) Gel SDS-PAGE colorati blu Coomassie della soluzione proteica OnePot (corsie 2, 3), ribosomi privi di tag senza soluzione proteica (corsie 5, 6) e con soluzione proteica (corsie 8, 9), ribosomi his-tagged senza soluzione proteica (corsie 11, 12) e con soluzione proteica (corsie 14, 15). Sono state caricate due diverse concentrazioni per campione. (B) Confronto dell'espressione eGFP di ribosomi con tag His e ribosomi privi di tag. L'intensità di fluorescenza dell'espressione di eGFP in vitro viene monitorata nel tempo per una reazione PURE utilizzando ribosomi privi di tag (1,8 μM, blu) e ribosomi his-tagged (0,62 μM, rosso). Le concentrazioni del modello lineare e della soluzione proteica OnePot erano rispettivamente di 4 nM e 2 mg/ml. I pannelli (C) e (D) mostrano il gel SDS-PAGE di proteine sintetizzate in OnePot con tag-free (1,8 μM, blu, corsie 3, 4, 5) e ribosomi His-tag (0,62 μM, rosso, corsie 6, 7, 8) etichettati con un kit di etichettatura in vitro GreenLys(C)e colorati con blu Coomassie (D), rispettivamente. Le frecce nere indicano le bande previste di proteine sintetizzate: eGFP (26,9 kDa), ArgRS (64,7 kDa), T7 RNAP (98,9 kDa). Le concentrazioni del modello lineare e della soluzione proteica OnePot erano rispettivamente di 4 nM e 1,6 mg/ml. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Spettri di assorbanza a 260 nm. Risultati rappresentativi degli spettri di assorbanza a 260 nm durante la purificazione dell'interazione idrofobica di ribosomi privi di tag. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 1: Un programma giornaliero ottimizzato per il tempo per la preparazione di tutte le soluzioni OnePot PURE. Fare clic qui per scaricare questa tabella.
Tabella 2: Elenco delle proteine PURE Fare clic qui per scaricare questa tabella.
Tabella supplementare 1: Reagenti. La tabella elenca concentrazioni, volumi e altri dettagli specifici dei reagenti e dei componenti utilizzati durante questo studio. Fare clic qui per scaricare questa tabella.
Tabella supplementare 2: Buffer. Il foglio di calcolo elenca le esatte composizioni tampone per le purificazioni di proteine, ribosomi privi di tag e ribosomi His-tag, nonché le concentrazioni delle soluzioni stock utilizzate per la loro preparazione. Inoltre, calcola le quantità richieste di componenti in base al volume del buffer. Fare clic qui per scaricare questa tabella.
Tabella supplementare 3: Calcoli degli amminoacidi. Il foglio di calcolo elenca gli amminoacidi e le loro concentrazioni raccomandate di soluzione stock necessarie per la soluzione energetica. Calcola la quantità di acqua da aggiungere a ciascun amminoacido in base alla massa pesata effettiva e calcola anche il volume della soluzione di amminoacidi da aggiungere alla miscela di amminoacidi finali. Fare clic qui per scaricare questa tabella.
Tabella complementare 4: Soluzioni stock per la soluzione energetica. La tabella elenca le concentrazioni e i volumi di soluzioni stock necessarie per la soluzione energetica e indica ulteriori dettagli, comprese le condizioni di stoccaggio. Fare clic qui per scaricare questa tabella.
Tabella complementare 5: Soluzione energetica. La tabella elenca i componenti della soluzione energetica e le loro concentrazioni raccomandate. Inoltre, calcola i volumi necessari da aggiungere alla soluzione finale in base alle concentrazioni della soluzione stock e al volume della soluzione energetica. Fare clic qui per scaricare questa tabella.
Tabella supplementare 6: PCR. La tabella elenca sequenze e concentrazioni dei primer utilizzati per la PCR di estensione e indica le temperature di fusione e i passaggi del termociclatore ottimizzati per una DNA polimerasi ad alta fedeltà. Fare clic qui per scaricare questa tabella.
Tabella supplementare 7: Reazione PURA. Il foglio di calcolo mostra un esempio di configurazione di una reazione PURE. Elenca le concentrazioni e i volumi utilizzati dei componenti per una reazione PURE utilizzando ribosomi privi di tag o ribosomi His-tag. Inoltre, calcola i rapporti di volume per le titolazioni di proteine e ribosomi. Fare clic qui per scaricare questa tabella.
Discussione
Il protocollo qui presentato descrive un metodo semplice, rapido ed economico per preparare un versatile sistema di espressione PURE20 basato sulla composizione standard15. Utilizzando il protocollo insieme ai programmi giornalieri forniti (Tabella 1), tutti i componenti possono essere preparati in 1 settimana e produrre quantità sufficienti per un massimo di cinquecento reazioni PURE da 10 μL. Poiché le proteine utilizzate in questo protocollo sono sovraespresse da plasmidi ad alta copia e hanno una bassa tossicità per E. coli, si osservano buoni livelli di espressione per tutte le proteine richieste (Figura 1). Ciò consente la facile regolazione dei ceppi, e quindi anche della composizione proteica nelle cocolture, semplicemente modificando i rapporti dei ceppi di inoculazione20. Oltre alle proteine ribosomiali, la concentrazione di EF-Tu si è rivelata di fondamentale importanza per i rendimenti di espressione6. Al contrario, i cambiamenti nella concentrazione degli altri componenti proteici hanno avuto un impatto relativamente basso sulla robustezza del sistema PURE7,24. Pertanto, regolando il rapporto di inoculazione di EF-Tu per quanto riguarda tutti gli altri componenti, è possibile ottenere una composizione simile alla composizione PURE standard e ottenere un sistema PURE con una resa similea 20. Nella preparazione della soluzione proteica, è fondamentale assicurarsi che tutti i ceppi crescano bene e sovraesprimano la proteina codificata dopo l'induzione (Figura 1).
La funzione ribosoma è fondamentale per le prestazioni complessive del sistema PURE24. In questo protocollo, vengono dimostrati due diversi metodi per preparare la soluzione di ribosoma, cioè la purificazione del ribosoma senza tag e con tag His. La purificazione del ribosoma tag-free si basa sulla cromatografia di interazione idrofobica seguita da centrifugazione con un cuscino di saccarosio, che richiede l'accesso a un sistema di purificazione FPLC e un ultracentrifuga15. Al contrario, il metodo che utilizza i ribosomi18 con tag His e la purificazione della cromatografia per affinità del flusso gravitazionale non richiede attrezzature specializzate e può essere eseguito nella maggior parte dei laboratori. Quest'ultimo metodo, quindi, porta vantaggi come la semplicità e l'accessibilità. Tuttavia, abbiamo osservato una resa di sintesi significativamente inferiore quando si utilizzano i ribosomi his-tagged in OnePot PURE rispetto alla variante senza tag (Figura 3). In base al tipo di applicazione, questa resa inferiore può essere accettabile.
La soluzione energetica fornisce i componenti a basso peso molecolare e i tRNA necessari per alimentare le reazioni TX-TL in vitro. Questo protocollo fornisce una ricetta per una tipica soluzione energetica, che può essere facilmente regolata in base alle esigenze dell'utente. Insieme a tRNA, NTP e creatina fosfato, l'abbondanza e la concentrazione di ioni Mg2+ sono state cruciali per le prestazioni complessive del sistema PURE8, in quanto sono cofattori critici per la trascrizione e la traduzione. In alcuni casi, la titolazione degli ioni può, quindi, migliorare notevolmente le prestazioni complessive di PURE. L'integrità del DNA è fondamentale per le prestazioni PURE. Pertanto, la sequenza che verifica la regione del promotore, il sito di legame del ribosoma e il gene bersaglio e garantisce che un'adeguata concentrazione di DNA (<2 nM) aiuterà a risolvere i problemi che possono sorgere durante la configurazione di una reazione PURE.
Il sistema PURE è un sistema TX-TL minimo e applicazioni specifiche possono quindi richiedere ulteriori regolazioni25. Questi possono includere l'incorporazione di diverse RNA polimerasi9,26,chaperoni13e fattori proteici come EF-P o ArfA8. Sebbene i ceppi di espressione per queste proteine possano essere inclusi nelle cocolture, aggiungerli separatamente al sistema preparato può fornire un migliore controllo dei livelli proteici richiesti. Inoltre, l'inclusione delle vescicole è essenziale per la produzione di proteine di membrana10,11. Ambienti ossidanti piuttosto che riducenti e un legame disolfuro isomerasi facilitano la corretta formazione del legame disolfuro, che sono, ad esempio, necessari per le proteine secretoria12.
È essenziale assicurarsi che eventuali componenti aggiuntivi non interferiscano con la reazione. I fattori più importanti a cui prestare attenzione quando si imposta una reazione o si aggiungono altri componenti sono elencati di seguito. Assicurarsi che non vengano utilizzati tamponi incompatibili né che le concentrazioni di ioni siano disturbate. Evitare soluzioni contenenti glicerolo, alte concentrazioni di potassio, magnesio, ioni calcio, osmoliti, pirofosfato, antibiotici o EDTA, per quanto possibile. Ad esempio, la sostituzione di un tampone di eluizione con acqua durante la purificazione del DNA può essere utile in quanto l'EDTA è un additivo comune in questo tampone. Fornire alle soluzioni ulteriori molecole caricate negativamente come NTP o dNTP richiede la regolazione della concentrazione di magnesio8, poiché le molecole caricate negativamente si comportano come agenti chelanti e legano molecole caricate positivamente. Un pH neutro è ideale per la reazione. Di conseguenza, tutti i componenti devono essere tamponati al pH corrispondente; ciò è particolarmente importante per molecole altamente acide o basiche come le NTP. Infine, temperatura e volume sono parametri chiave per la reazione. Per ottenere una buona resa, si dovrebbe implementare una temperatura intorno ai 37 °C, poiché temperature inferiori a 34 °C ridurranno significativamente la resa27.
È importante notare che prima di preparare OnePot PURE, è necessario considerare l'applicazione di destinazione e i requisiti associati, come volume, purezza, facilità di modifica e inclusione o omissione di componenti. Per molte applicazioni, il sistema sarà una scelta eccellente, ma altri potrebbero richiedere rendimenti, regolabilità e altri fattori, che il sistema OnePot non può fornire. Indipendentemente da ciò, il protocollo introdotto sarà utile per la preparazione di qualsiasi sistema fatto in casa, poiché tutti i passaggi critici per tale preparazione sono riassunti qui.
Uno dei principali vantaggi del sistema OnePot è la sua compatibilità con il sistema PURExpress disponibile in commercio, che offre la possibilità di testare la funzionalità e l'integrità di tutti i componenti separatamente sostituendo sequenzialmente ciascun componente PURExpress con il suo equivalente OnePot. I vantaggi del sistema OnePot PURE, come la sintonizzazione e la preparazione facile, veloce ed economica, renderanno tx-TL senza cellule accessibile a più laboratori in tutto il mondo e contribuiranno ad espandere l'implementazione di questa potente piattaforma nella biologia sintetica senza cellule.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto dal Consiglio europeo della ricerca nell'ambito della sovvenzione del programma di ricerca e innovazione Horizon 2020 dell'Unione europea 723106, una sovvenzione del Fondo nazionale svizzero per la ricerca scientifica (182019) e l'EPFL.
Materiali
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS buffer | Bio-Rad Laboratories | 1610732 | |
| 15 mL centrifuge tubes | VWR International | 525-0309 | |
| 384-well Black Assay Plates | Corning | 3544 | |
| 4-20% Mini-PROTEANRTM TGXTM Precast Protein Gels | Bio-Rad Laboratories | 4561096 | |
| 50 mL centrifuge tubes | VWR International | 525-0304 | |
| 96-Well Polypropylene DeepWell plate | Nunc | 260252 | |
| Acetic acid, 99.8 % | Acros | 222140010 | |
| Äkta purifier | GE Healthcare | purification of tag free ribosomes | |
| AMICON ULTRA 0.5 mL - 3 KDa | Merck Millipore | UFC500324 | |
| AMICON ULTRA 15 mL - 3 KDa | Merck Millipore | UFC900324 | |
| Amino acids | Sigma-Aldrich | LAA21-1KT | |
| Ammonium chloride | Sigma-Aldrich | 09718-250G | |
| Ammonium sulfate | Sigma-Aldrich | A4418 | |
| Ampicillin | Condalab | 6801 | |
| BenchMark Fluorescent Protein Standard | ThermoFisher | LC5928 | |
| Breathe-Easy sealing membrane | Diversified Biotech | Z380059-1PAK | |
| Centrifuge tubes polycarbonate | Beckman | 355631 | purification of tag free ribosomes |
| Chill-out Liquid Wax | Bio-Rad Laboratories | CHO1411 | |
| Creatine phosphate | Sigma-Aldrich | 27920 | |
| DNA Clean & Concentrator-25 (Capped) | Zymo | ZYM-D4034-200TS | |
| DTT | SantaCruz Biotech | sc-29089B | |
| Econo-Pac Chromatography Columns | Bio-Rad Laboratories | 7321010 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | 03609-250G | |
| Eppendorf Protein LoBind microcentrifuge tubes | VWR International / Eppendorf | 525-0133 | |
| Falcon 14 mL Round Bottom Polystyrene Test Tube, with Snap Cap | Falcon | 352051 | |
| Flasks, baffled 1000 mL 4 baffles, borosilicate glass | Scilabware | 9141173 | |
| FluoroTect Green Lys in vitro Translation Labeling System | Promega | L5001 | optional |
| Folinic acid | Sigma-Aldrich | PHR1541 | |
| Glycerol | Sigma-Aldrich | G7757-1L | |
| HEPES | Gibco | 15630-056 | |
| HiTrap Butyl HP Column | GE Healthcare | 28411005 | purification of tag free ribosomes |
| IMAC Sepharose 6 Fast Flow | GE Healthcare | 17-0921-07 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| InstantBlue | Expedeon | ISB1L-1L | |
| IPTG (Isopropyl-beta-D-thiogalactoside) | Alfa Aesar | B21149.03 | |
| Laemmli buffer (2x), sample buffer | Sigma-Aldrich | S3401-1VL | |
| Lysogeny broth (LB) media | AppliChem | A0954 | |
| Magnesium acetate | Sigma-Aldrich | M0631 | |
| Magnesium chloride | Honeywell Fluka | 63020-1L | |
| Nickel Sulfate | Alfa Aesar | 15414469 | |
| NTP | ThermoFisher | R0481 | |
| Phusion High-Fidelity DNA Polymerase (2 U/µL) | ThermoFisher | F530S | |
| Potassium chloride | Sigma-Aldrich | P5405-1KG | |
| Potassium glutamate | Sigma-Aldrich | 49601 | |
| PURExpress In Vitro Protein Synthesis Kit | NEB | E6800S | |
| PURExpress Δ Ribosome Kit | NEB | E3313S | |
| Quick Start Bradford 1x Dye Reagent | Bio-Rad Laboratories | 5000205 | |
| Rapid-Flow Sterile Single Use Vacuum Filter Units | ThermoFisher | 564-0020 | |
| RNaseA solution | Promega | A7973 | |
| SealPlate film | Excel Scientific | Z369659-100EA | |
| Sodium hydroxide | Sigma-Aldrich | 6203 | |
| Spermidine | Sigma-Aldrich | S2626 | |
| Sucrose | Sigma-Aldrich | 84097 | |
| TCEP (Tris(2-carboxyethyl)phosphin -hydrochlorid) | Sigma-Aldrich | 646547-10X1mL | |
| Thickwall Polycarbonate Tube | Beckman | 355631 | |
| Trichloroacetic acid | Sigma-Aldrich | T0699 | |
| Tris base | ThermoFisher | BP152-500 | |
| tRNA | Roche | 10109541001 | |
| Ultracentrifuge Optima L-80 | Beckman | purification of tag free ribosomes | |
| Whatman GD/X syringe filters | GE Whatman | WHA68722504 | |
| β-mercaptoethanol | Sigma-Aldrich | M6250-100mL |
Riferimenti
- Laohakunakorn, N., et al. Bottom-up construction of complex biomolecular systems with cell-free synthetic biology. Frontiers in Bioengineering and Biotechnology. 8, 213 (2020).
- Laohakunakorn, N. Cell-free systems: a proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788 (2020).
- Cho, E., Lu, Y. Compartmentalizing cell-free systems: toward creating life-like artificial cells and beyond. ACS Synthetic Biology. 9 (11), 2881-2901 (2020).
- Gaut, N. J., Adamala, K. P. Reconstituting natural cell elements in synthetic cells. Advanced Biology (Weinh). 5 (3), 2000188 (2021).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nature Biotechnology. 19 (8), 751-755 (2001).
- Li, J., Gu, L., Aach, J., Church, G. M. Improved cell-free RNA and protein synthesis system. PLoS One. 9 (9), 106232 (2014).
- Kazuta, Y., Matsuura, T., Ichihashi, N., Yomo, T. Synthesis of milligram quantities of proteins using a reconstituted in vitro protein synthesis system. Journal of Bioscience and Bioengineering. 118 (5), 554-557 (2014).
- Li, J., et al. Dissecting limiting factors of the protein synthesis using recombinant elements (PURE) system. Translation. 5 (1), 1327006 (2017).
- de Maddalena, L. L., et al. GreA and GreB enhance expression of Escherichia coli RNA polymerase promoters in a reconstituted transcription-translation system. ACS Synthethic Biology. 5 (9), 929-935 (2016).
- Kuruma, Y., Ueda, T. The PURE system for the cell-free synthesis of membrane proteins. Nature Protocols. 10 (9), 1328-1344 (2015).
- Jacobs, M. L., Boyd, M. A., Kamat, N. P. Diblock copolymers enhance folding of a mechanosensitive membrane protein during cell-free expression. Proceedings of the National Academy of Sciences of the Unites States of America. 116 (10), 4031-4036 (2019).
- Shimizu, Y., Kanamori, T., Ueda, T. Protein synthesis by pure translation systems. Methods. 36 (3), 299-304 (2005).
- Niwa, T., Kanamori, T., Ueda, T., Taguchi, H. Global analysis of chaperone effects using a reconstituted cell-free translation system. Proceedings of the National Academy of Sciences of the Unites States of America. 109 (23), 8937-8942 (2012).
- Niwa, T., et al. Large-scale analysis of macromolecular crowding effects on protein aggregation using a reconstituted cell-free translation system. Frontiers in Microbiology. 6, 1113 (2015).
- Shimizu, Y., Ueda, T. PURE technology. Methods in molecular biology. 607, 11-21 (2010).
- Horiya, S., Bailey, J. K., Krauss, I. J. Directed evolution of glycopeptides using mRNA display. Methods in Enzymology. 597, 83-141 (2017).
- Villarreal, F., et al. Synthetic microbial consortia enable rapid assembly of pure translation machinery. Nature Chemical Biology. 14 (1), 29-35 (2018).
- Wang, H. H., et al. Multiplexed in vivo His-tagging of enzyme pathways for in vitro single-pot multienzyme catalysis. ACS Synthetic Biology. 1 (2), 43-52 (2012).
- Shepherd, T., et al. De Novo Design and synthesis of a 30-cistron translation-factor module. Nucleic Acids Research. 45 (18), 10895-10905 (2017).
- Lavickova, B., Maerkl, S. J. A Simple, robust, and low-cost method to produce the PURE cell-free system. ACS Synthetic Biology. 8 (2), 455-462 (2019).
- Ederth, J., Mandava, C. S., Dasgupta, S., Sanyal, S. A single-step method for purification of active his-tagged ribosomes from a genetically engineered Escherichia coli. Nucleic Acids Research. 37 (2), 15 (2009).
- Gesteland, R. F. Isolation and characterization of ribonuclease I mutants of Escherichia coli. Journal of Molecular Biology. 16 (1), 67-84 (1996).
- Li, J., et al. Cogenerating synthetic parts toward a self-replicating system. ACS Synthetic Biology. 6 (7), 1327-1336 (2017).
- Matsuura, T., Kazuta, Y., Aita, T., Adachi, J., Yomo, T. Quantifying epistatic interactions among the components constituting the protein translation system. Molecular Systems Biology. 5, 297 (2009).
- Libicher, K., Hornberger, R., Heymann, M., Mutschler, H. In vitro self-replication and multicistronic expression of large synthetic genomes. Nature Communications. 11 (1), 904 (2020).
- Niederholtmeyer, H., Stepanova, V., Maerkl, S. J. Implementation of cell-free biological networks at steady state. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 15985-15990 (2013).
- Lavickova, B., Laohakunakorn, N., Maerkl, S. J. A partially self-regenerating synthetic cell. Nature Communications. 11 (1), 6340 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon